A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
תיוג קוולנטי עם דיתילפירוקרבונט לחקר מבנה חלבון מסדר גבוה יותר על ידי ספקטרומטריית מסה
In This Article
Summary
ההליכים הניסיוניים לביצוע תיוג קוולנטי מבוסס diethylpyrocarbonate עם זיהוי ספקטרומטרי מסה מתוארים. Diethylpyrocarbonate הוא פשוט מעורבב עם חלבון או חלבון קומפלקס של עניין, המוביל לשינוי של שאריות חומצות אמינו נגיש ממס. ניתן לזהות את השאריות ששונו לאחר עיכול פרוטאוליטי וניתוח כרומטוגרפיה נוזלית/ספקטרומטריית מסה.
Abstract
אפיון המבנה הגבוה יותר של החלבון חיוני להבנת תפקידו. ספקטרומטריית מסה (MS) התפתחה ככלי רב עוצמה למטרה זו, במיוחד עבור מערכות חלבון שקשה ללמוד בשיטות מסורתיות. כדי לחקור מבנה של חלבון על ידי טרשת נפוצה, תגובות כימיות ספציפיות מבוצעות בתמיסה המקודדת את המידע המבני של החלבון לתוך המסה שלו. גישה יעילה במיוחד היא להשתמש ריאגנטים כי covalently לשנות ממס נגיש חומצת אמינו שרשראות בצד. תגובות אלה מובילות לעליות מסה שניתן לבצע בהן לוקליזציה עם רזולוציה ברמת השאריות בשילוב עם עיכול פרוטאוליטי וספקטרומטריית מסה דו-מושבית. כאן, אנו מתארים את הפרוטוקולים הקשורים לשימוש diethylpyrocarbonate (DEPC) כמו מגיב תיוג covalent יחד עם זיהוי טרשת נפוצה. DEPC היא מולקולה אלקטרופילית מאוד המסוגלת לתייג עד 30% מהשאריות בחלבון הממוצע, ובכך לספק רזולוציה מבנית מצוינת. DEPC שימש בהצלחה יחד עם טרשת נפוצה כדי להשיג מידע מבני עבור חלבונים חד תחומיים קטנים, כגון β2-microglobulin, לחלבונים רב תחומיים גדולים, כגון נוגדנים חד שבטיים.
Introduction
חלבונים הם ביומולקולות חיוניות כמעט בכל תהליך פיזיולוגי. מגוון הפונקציות שחלבונים מבצעים אפשרי בגלל המבנים שהם מאמצים והאינטראקציות שיש להם עם ביומולקולות אחרות. כדי להבין את תפקוד החלבון ברמה עמוקה יותר, יש צורך בכלים ביוכימיים וביופיזיים כדי להבהיר את התכונות המבניות החשובות הללו ואת האינטראקציות. באופן מסורתי, קריסטלוגרפיה של קרני רנטגן, מיקרוסקופ אלקטרונים קריוגניים וספקטרוסקופיה מגנטית גרעינית (NMR) סיפקו את הפרטים האטומיים הרצויים כדי לחשוף את מבנה החלבון. עם זאת, מערכות חלבון רבות לא ניתן לחקור על ידי טכניקות אלה בגלל התנהגות התגבשות ירודה, זמינות חלבון מוגבלת, הטרוגניות מדגם מוגזם, או מגבלות משקל מולקולרי. כתוצאה מכך, נוצרו שיטות ניתוח חדשות יותר המתגברות על מגבלות אלה. בין הטכניקות המתעוררות שיכולות לספק מידע מבני חלבון הוא ספקטרומטריית מסה.
ספקטרומטריית מסה (MS) מודדת את יחס המסה לטעינה (m/z) של מולקולה, ולכן יש להשיג מידע מבני ברמה גבוהה יותר על ידי קידוד המידע המבני הרצוי למסת החלבון. פותחו מספר גישות לקידוד מידע זה, כולל חילופי מימן-דיטריום (HDX)1,2,3,4, הצלבה כימית (XL)5,6, ו תיוג covalent (CL)7,8,9,10. ב- HDX, עמוד השדרה אמיד מימן מוחלפים על ידי דיטריומים קצת יותר מסיביים בשיעורים התלויים בנגישות ממס ובהיקף מליטה H. ניתן לאתר את היקף ה-HDX על ידי עיכול מהיר של החלבון לרסיסי פפטיד לפני ההפרדה והמדודה של שברים אלה על ידי ספקטרומטר המסה או על ידי ניתוק החלבון בניסוי מלמעלה למטה. קביעת שער החליפין מספקת תובנה נוספת לגבי דינמיקת החלבון. HDX הוכיחה את עצמה ככלי רב ערך לאפיון מבנה החלבון למרות האתגרים הקשורים להחלפת גב והצורך בציוד מיוחד כדי למקסם את הרבייה. ב- XL-MS, חלבונים מגיבים עם ריאגנטים דו-תפקודיים המקשרים באופן קוולנטי שרשראות צד סמוכות לשאריות בתוך חלבון נתון או בין שני חלבונים. בעשותו כן, XL-MS יכול לספק אילוצי מרחק שניתן להשתמש בהם כדי לאפיין את מבנה החלבון. אזורי החלבון המקושרים זה לזה יכולים להיות מזוהים על ידי עיכול פרוטאוליטי ואחריו ניתוח כרומטוגרפיה נוזלית (LC)-MS. בעוד XL-MS הוא כלי רב תכליתי ששימש לחקר מגוון מתחמי חלבון, כולל בתוך תאים, זיהוי של מוצרי XL הוא מאתגר ודורש תוכנה מיוחדת.
CL-MS התפתח לאחרונה ככלי משלים ולעיתים אלטרנטיבי מבוסס טרשת נפוצה לחקר מבנה החלבון והאינטראקציות. ב- CL-MS, קומפלקס חלבונים או חלבונים משתנה בקובאליות עם מגיב מונו-פונקציונלי שיכול להגיב עם שרשראות צד חשופות ממס(איור 1). על ידי השוואת היקף השינוי של קומפלקס חלבון או חלבון בתנאים שונים, ניתן לחשוף שינויי קונפורמציה, אתרי קשירה וממשקי חלבון-חלבון. לאחר תגובת CL, מידע ספציפי לאתר, לעתים קרובות ברמת חומצת אמינו אחת, ניתן להשיג באמצעות זרימות עבודה פרוטאומיות טיפוסיות מלמטה למעלה שבהן חלבונים מתעכלים באופן פרוטאוליטי, שברי פפטיד מופרדים על ידי LC, ואתרים מותאמים מזוהים באמצעות טנדם MS (MS / MS). ההיסטוריה העשירה של הכימיה הביו-מצויגת הפכה ריאגנטים רבים לזמינים לניסויי CL-MS. ריאגנטים CL מתחלקים לשתי קטגוריות כלליות: (i) ספציפי ו-(ii) לא ספציפי. ריאגנטים ספציפיים מגיבים עם קבוצה פונקציונלית אחת (למשל, אמינים חינם)8,10 והם קלים ליישום, אך הם נוטים לספק מידע מבני מוגבל. ריאגנטים לא ספציפיים מגיבים עם מגוון רחב של שרשראות צד, אך לעתים קרובות דורשים ציוד מיוחד כגון לייזרים או מקורות synchrotron לייצר מינים אלה תגובתי מאוד. הידרוקסיל רדיקלים הם ריאגנט לא ספציפי הנפוץ ביותר, לאחר מיושם טביעת רגל רדיקלית הידרוקסיל (HRF)7,11,12,13 ניסויים לחקור מגוון רחב של חלבונים ותסביך חלבון תחת מגוון רחב של תנאים.
קבוצת המחקר שלנו השתמשה בהצלחה בריאגנט אחר יחסית לא ספציפי בשם diethylpyrocarbonate (DEPC) כדי לחקור מבנה חלבון ואינטראקציות בהקשר של ניסויים CL-MS14,15,16,17,18,19,20 , 21,22,23,24,25. DEPC מציעה את הפשטות של ריאגנטים תיוג ספציפיים (כלומר, אין צורך בציוד מיוחד כדי לבצע את התגובות), תוך תגובה עם עד 30% של חומצות אמינו בחלבון הממוצע. כריאגנט אלקטרופילי מאוד, DEPC מסוגל להגיב עם N-terminus ואת שרשראות הצד הנוקליופילי של ציסטאין, היסטידין, ליצין, טירוסין, סרין, ושאריות threonine. בדרך כלל, מוצר יחיד של תגובות אלה נוצר, וכתוצאה מכך עלייה המונית של 72.02 Da. סוג אחד זה של המוצר מנוגד ל-55 מוצרים שונים שניתן לייצר כאשר חלבונים מגיבים עם הידרוקסיל רדיקלים7. כימיה פשוטה כזו מאפשרת זיהוי של אתרים מסומנים.
כאן, אנו מספקים פרוטוקולים לשימוש CL-MS מבוסס DEPC ללמוד מבנה חלבון ואינטראקציות. מתוארים פרטים הקשורים להכנת ריאגנטים, תגובות של חלבון DEPC, תנאי עיכול חלבונים, פרמטרי LC-MS ו- MS/MS וניתוח נתונים. אנו גם מדגימים את התועלת של תיוג DEPC על ידי מתן תוצאות לדוגמה מאינטראקציות חלבון-מתכת, חלבון-ליגנד וחלבון-חלבון, כמו גם חלבונים העוברים שינויים מבניים בעת החימום.
Protocol
1. הכנת חלבונים ריאגנטים
הערה: פרוטוקול זה כולל זרימת עבודה לדוגמה לתיוג חלבון עם DEPC. תנאים מסוימים וריכוזי ריאגנט המפורטים עשויים להשתנות בהתאם לחלבון המועדף.
- הכינו את כל הפתרונות ריאגנטים בצינורות מיקרוצנטריפוגה 1.5 מ"ל.
- הכינו תמיסת חלבון של הריכוז הרצוי, בדרך כלל בטווח של עשרות מיקרומטר, במאגר חומצה פרופאנסולפונית (MOPS) של 10 מ"מ 3-(N-מורפולינו) ב- pH 7.4. לחלופין, הכינו פתרון חלבון קיים להחלפת חיץ ב- 10 mM pH 7.4 MOPS אם המדגם מכיל מאגר נוקליאופילי שיהיה תגובתי עם DEPC. מאגרים אחרים (למשל, מלוחים מאגר פוספט) ניתן להשתמש גם, כל עוד אין להם קבוצות פונקציונליות נוקליאופילית.
- הכן פתרון DEPC 100 מ"מ באצטוניטריל יבש (ACN) על ידי pipetting 1.45 μL של המניה 6.9 M DEPC פתרון לתוך 98.55 μL של ACN.
הערה: אין קבוצה אחת של ריכוזים שיעבדו עם כל חלבון, אם כי ניתן להעריך את הריכוזים האופטימליים על סמך מספר שאריות שלו ושל Lys23. לדוגמה, עם 50 μL של 50 μM β2-microglobulin פתרון, להגיב על החלבון עם 0.2 μL של DEPC 100 מ"מ עבור ריכוז DEPC הסופי של 200 μM (שווה 4x ריכוז החלבון) כדי להבטיח את היחס הטוחן הרצוי באמצעות דוגמה כללית זו (טבלה 1). תיוג DEPC הוא תגובת סדרשני, כך ששינוי ריכוז החלבון או ה- DEPC בתערובת התגובה ישנה את קצב התיוג. - הכן 1 M imidazole פתרון על ידי שקילה 10 מ ג של imidazole והתמוססות ב 146.9 μL של מים ברמה HPLC.
2. תיוג קוולנטי של חלבון שלם
- הגדר טמפרטורת אמבט מים ל 37 מעלות צלזיוס ולחכות לאמבטיה כדי להגיע לטמפרטורה יציבה.
הערה: ריכוזים ונפחים של ריאגנטים עבור פרוטוקול תיוג לדוגמה ניתן למצוא בטבלה 1. - בצינור מיקרוצנטריפוגה חדש, מערבבים מאגר MOPS ותמיסת חלבון בכמויות המופיעות בטבלה 1.
- לחלבון ולחוצץ מוסיפים 0.2 μL של פתרון DEPC, מקפידים לערבב כראוי את הפתרון שנוצר, ולאחר מכן מניחים את הצינור המכיל את תערובת התגובה לאמבט המים של 37 מעלות צלזיוס למשך דקה אחת.
הערה: נפח ACN הוסיף לא יעלה על 1% מנפח התגובה הכולל כדי למנוע הפרעה של מבנה החלבון במהלך תגובת התיוג. זמן התגובה תלוי המשתמש, אם כי תגובה של דקה אחת בתנאים לדוגמה ממזערת את ההארבל ואת ההידרו ליזה הפוטנציאלית של DEPC14. - לאחר 1 דקה, להסיר את הצינור המכיל את תערובת התגובה מאמבט המים להרוות את התגובה עם 1 μL של פתרון 1 M imidazole כדי לטהר את DEPC unreacted הנותרים.
הערה: הריכוז הסופי של imidazole בתערובת התגובה צריך שווה 50x הריכוז של DEPC בתערובת התגובה. פעולה זו תבטיח כי DEPC שנותר לא מנוטרל הוא נוקה.
3. הכנת תקציר חלבונים עבור LC-MS מלמטה למעלה
הערה: בחר תנאי עיכול נוחים לחלבון המעניין. צעדים נפוצים כרוכים בהפחתת החלבון והפחתתו של כל קשר דיסולפידי.
- פותחים את החלבון על ידי הוספת מגיב נפרש מתאים לתערובת התגובה.
הערה: סוכנים נפוצים כוללים ACN, אוריאה, ו guanidine הידרוכלוריד (GuHCl). - הכן פתרונות של Tris(2-carboxyethyl)פוספין (TCEP) ו iodoacetamide (IAM) על ידי שקילה 5 מ"ג של כל אחד והפסתם בצינורות microcentrifuge חדשים ב 174.4 ו 270.3 μL של 10 mM pH 7.4 MOPS מאגר, בהתאמה, עבור הפחתת ו alkylation צעדים.
- להפחית קשרים disulfide על ידי הוספת 2 μL של TCEP 100 מ"מ (ריכוז סופי של 2 מ"מ בתערובת תגובה) פתרון לתערובת התגובה ולהגיב במשך 3 דקות בטמפרטורת החדר.
הערה: הריכוז הסופי של TCEP צריך להיות שווה פי 40 מריכוז החלבון לכל קשר דיסולפיד הקיים בתמיסה. - אלקילאט הפחית ציסטאינים עם 4 μL של פתרון IAM 100 מ"מ (ריכוז סופי של 4 מ"מ בתערובת תגובה) במשך 30 דקות בחושך. IAM רגיש לאור ויתפרק תחת אור ישיר.
הערה: הריכוז הסופי של IAM בתמיסה צריך להיות כפול הריכוז המשמש TCEP, או 80x ריכוז החלבון לכל קשר דיסולפיד. - לעכל את החלבון עם אנזים מתאים כגון טריפסין או chymotrypsin. יחס אנזימים של 10:1 לעיכול של 3 שעות ב-37°C עם אנזים משותק בקצב רועד של 300 משיכות/דקה מספיק בדרך כלל לחלבונים עם תווית DEPC. ראה דיון.
- לאחר העיכול, להפריד את האנזים משותק מן הפפטידים מתעכל על ידי צנטריפוגה ב 12,000 סל"ד במשך 5 דקות.
- לנתח את המדגם מיד על ידי LC-MS / MS או פלאש להקפיא את המדגם עם חנקן נוזלי כדי למזער את השפלה מדגם ואובדן תווית. אחסן את הדגימות שהוקפאו בהבזק ב- < -20 °C עד מוכן לניתוח LC-MS/ MS.
4. ניתוח LC-MS/MS
הערה: ניתן להשתמש בפרמטרים סטנדרטיים של LC-MS/MS עבור פרוטאומיקה מלמטה למעלה כדי לזהות אתרים המסומנים בתווית על שברי הפפטיד הפרוטאוליטיים. דוגמה כללית מתוארת להלן.
- הפרד את הפפטידים בעלי התווית DEPC באמצעות שלב נייח C18 הפוך. השתמש בשלב נייד טיפוסי של LC של שני ממיסים: (A) מים + 0.1% חומצה פורמית ו- (B) ACN + 0.1% חומצה פורמית באמצעות שיפוע (למשל, איור 2) כדי להשיג את ההפרדה הטובה ביותר של פפטידים.
הערה: ניתן למטב את זמן ההפרדה בהתבסס על מורכבות הדגימה, וקצב זרימת הפאזה הנייד תלוי באם נעשה שימוש בנימים או בננו LC. - השתמש בספקטרומטר מסה המסוגל לבצע LC-MS ו- MS/MS מקוון כדי לזהות אתרי שינוי DEPC בפפטיד. בניסויים שלנו, השתמשנו בהצלחה במספר סוגים של ספקטרומטרים של מסה. כל ספקטרומטר מסה המסוגל לבצע באופן אוטומטי MS / MS של פפטידים רבים במהלך ניתוח LC-MS צריך להיות מתאים. הפרמטרים הרלוונטיים של MS כוללים: מתח מקור ESI = -4000 V עבור ESI רגיל; -2000 V עבור nanospray; רזולוציית אורביטראפ = 60,000; משך אי-הכללה דינאמי = 30 שניות; סוג הפעלה של MS/MS: CID, ETD או שניהם; טווח סריקה המונית = 200-2,000; בקרת הגברה אוטומטית = 4.0E5 (MS1 ב- Orbitrap) ו- 5.0E4 (MS2 במלכודת יון מרובע ליניארי).
- טען והזרק את דגימת החלבון המתעכלת המסומנת במערכת LC והתחל את רכישת LC-MS/MS. אם הדגימה הוקפאה, הפשר לפני הניתוח. להסיט את קולחים LC לפסולת במשך 5 הדקות הראשונות, כדי למנוע מלחים מוגזמים מלהיכנס למקור ESI.
הערה: לולאת הזרקת μL 5 מנוצלת בדרך כלל, ומאפשרת הזרקה של כ -2.5 מיקרוגרם חלבון ל- LC-MS / MS. הדבר תלוי בתנאי הטעינה של ה- LC כדי לא לסתום את מזרק הדגימה.
5. ניתוח נתונים
- זהה אתרי תוויות DEPC וכמת אזורי שיא פפטיד באמצעות תוכנה מתאימה עבור ספקטרומטר המסה המשמש.
- כלול תוספת DEPC (72.02 Da) ו carbamidomethylation (57.02 Da) כשינויים משתנים. פרמטרי חיפוש נוספים עבור ניתוח MS/MS הם כדלקמן: מחשוף מרבי שלא נענו = 3; סוגי יון קטע = b ו- y; סובלנות m/z מבשר = 10 עמודים לדקה (ערך זה צריך להיות גבוה יותר אם נעשה שימוש בספקטרומטר מסה של מלכודת יון מרובע); סטיית קטע m/z = 0.5 Da (ערך זה צריך להיות נמוך יותר אם ספקטרומטר מסה ברזולוציה גבוהה משמש לסריקת יון מוצר); טעינת קודמן = 1-4.
הערה: לאלגוריתמים שונים לחיפוש מסדי נתונים יש מערכות ניקוד שונות, ורבים מהם עלולים להתקשות בזיהוי פפטידים ששונו על-ידי DEPC מכיוון שרמות השינוי יכולות להיות נמוכות. התאמת חיתוך הציון עשויה להיות נחוצה כדי לזהות יותר פפטידים שכותרתו. אם כן, יש להשתמש בחקירה ידנית של נתוני MS/MS כדי לאמת פפטידים בעלי ניקוד נמוך. המכפלה של הידרוליזה של תווית DEPC אינה כלולה בחיפוש הנתונים מכיוון שה- DEPC שעבר הידרוליזה אינו מגיב עוד כלפי שרשראות צד נוקליאופיליות. - קבעו אחוזי שינוי ברמת השאריות בעזרת אזורי השיא הכרומטוגרפיים של הגרסאות שהשתנו ולא שונו של הפפטידים.
הערה: כל פפטיד המכיל את שאריות הריבית שהשתנו חייב להילקח בחשבון וכל מצבי החיוב הכלולים חייבים להיות נוכחים בכל הדגימות שנמדדו. פפטידים בעלי יעילות יינון שונה ו eluting בזמנים שונים גורם ערך זה להיות מידה יחסית ולא מוחלטת של שינוי של אתר מסוים.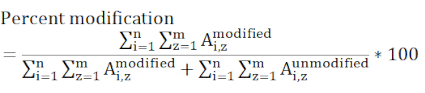
כאשרA i,z מייצג אזור שיא של כל פפטיד נתון (i) המכיל את שאריות העניין ושוקל את כל מצבי הטעינה שזוהו (z). - קבע אם שינוי תיוג בין פקד למדגם ניסיוני הוא משמעותי באמצעות הערכה סטטיסטית. שלוש מדידות משכפלות עבור כל מדגם הן אופייניות, ובדיקות t מנוצלות בדרך כלל עם מרווחי בר-סמך של 95 או 99%.
תוצאות
זיהוי אתרי שינוי ואחוזי שינוי של DEPC
תוספת המונית עקב תיוג קוולנטי ניתן למדוד ב (א) חלבון שלם ו (ב) רמות פפטיד8,9. ברמה ללא פגע, התפלגות של מיני חלבון עם מספר שונה של תוויות ניתן לקבל מניתוח ישיר או LC-MS של דגימות חלבון שכותרתו. כדי להשיג מידע מבני ברזולוציה ?...
Discussion
שלבים קריטיים
יש לשקול מספר נקודות לגבי תכנון ניסיוני כדי להבטיח תוצאות תיוג אמינות. ראשית, כדי למקסם את תיוג החלבון, יש צורך להימנע מאגרים עם קבוצות נוקליאופיליות חזקות (למשל, טריס) כי הם יכולים להגיב עם DEPC ולהוריד את היקף התיוג. זה גם מתקבל על הדעת כי מאגרים כאלה יכולים להגיב עם שארי...
Disclosures
למחברים אין מה לחשוף.
Acknowledgements
המחברים מכירים בתמיכת המכונים הלאומיים לבריאות (NIH) תחת גרנט R01 GM075092. ספקטרומטר מסת פיוז'ן Thermo Orbitrap המשמש לרכישת חלק מהנתונים המתוארים כאן נרכש בכספים ממענק המכונים הלאומיים לבריאות S10OD010645.
Materials
| Name | Company | Catalog Number | Comments |
| 1.5 mL microcentrifuge tube | Thermo Fisher Scientific | 3448 | |
| 3-(N-morpholino)propanesulfonic acid | Millipore Sigma | M1254 | |
| 3-(N-morpholino)propanesulfonic acid sodium salt | Millipore Sigma | M9381 | |
| Acclaim PepMap RSLC C18 Column | Thermo Scientific | 164537 | 300 μm x 15 cm, C18, 2 μm, 100 A |
| Acetonitrile | Fisher Scientific | A998-1 | |
| Diethylpyrocarbonate | Millipore Sigma | D5758 | |
| HPLC-grade water | Fisher Scientific | W5-1 | |
| Imidazole | Millipore Sigma | I5513 | |
| Immobilized chymotrypsin | ProteoChem | g4105 | |
| Immobilized trypsin, TPCK Treated | Thermo Fisher Scientific | 20230 | |
| Iodoacetamide | Millipore Sigma | I1149 | |
| Tris(2-carboxyethyl)phosphine | Millipore Sigma | C4706 |
References
- Katta, V., Chait, B. T., Carr, S. Conformational Changes in Proteins Probed by Hydrogen-exchange Electrospray-ionization. Rapid Communications in Mass Spectrometry. 5, 214-217 (1991).
- Wales, T. E., Engen, J. R. Hydrogen exchange mass spectrometry for the analysis of protein dynamics. Mass Spectrometry Reviews. 25, 158-170 (2006).
- Pirrone, G. F., Iacob, R. E., Engen, J. R. Applications of hydrogen/deuterium exchange MS from 2012 to 2014. Analytical Chemistry. 87, 99-118 (2015).
- Oganesyan, I., Lento, C., Wilson, D. J. Contemporary hydrogen deuterium exchange mass spectrometry. Methods. 144, 27-42 (2018).
- Sinz, A. Chemical cross-linking and mass spectrometry to map three-dimensional protein structures and protein-protein interactions. Mass Spectrometry Reviews. 25, 663-682 (2006).
- Holding, A. N. XL-MS: Protein cross-linking coupled with mass spectrometry. Methods. 89, 54-63 (2015).
- Xu, G., Chance, M. R. Hydroxyl radical-mediated modification of proteins as probes for structural proteomics. Chemical Reviews. 107, 3514-3543 (2007).
- Mendoza, V. L., Vachet, R. W. Probing Protein Structure by Amino Acid-Specific Covalent Labeling and Mass Spectrometry. Mass Spectrometry Reviews. 28, 785-815 (2009).
- Limpikirati, P., Liu, T., Vachet, R. W. Covalent labeling-mass spectrometry with non-specific reagents for studying protein structure and interactions. Methods. 144, 79-93 (2018).
- Liu, X. R., Zhang, M. M., Gross, M. L. Mass Spectrometry-Based Protein Footprinting for Higher-Order Structure Analysis: Fundamentals and Applications. Chemistry Reviews. 120, 4335 (2020).
- Maleknia, S. D., Brenowitz, M., Chance, M. R. Millisecond radiolytic modification of peptides by synchrotron X-rays identified by mass spectrometry. Analytical Chemistry. 71, 3965-3973 (1999).
- Aye, T. T., Low, T. Y., Sze, S. K. Nanosecond laser-induced photochemical oxidation method for protein surface mapping with mass spectrometry. Analytical Chemistry. 77, 5814-5822 (2005).
- Hambly, D. M., Gross, M. L. Laser flash photolysis of hydrogen peroxide to oxidize protein solvent-accessible residues on the microsecond timescale. Journal of the American Society of Mass Spectrometry. 16, 2057-2063 (2005).
- Mendoza, V. L., Vachet, R. W. Protein surface mapping using diethylpyrocarbonate with mass spectrometric detection. Analytical Chemistry. 80, 2895-2904 (2008).
- Mendoza, V. L., Antwi, K., Barón-rodríguez, M. A., Blanco, C., Vachet, R. W. Structure of the Pre-amyloid Dimer of β-2-microglobulin from Covalent Labeling and Mass Spectrometry. Biochemistry. 49, 1522-1532 (2010).
- Mendoza, V. L., Barón-Rodríguez, M. A., Blanco, C., Vachet, R. W. Structural insights into the pre-amyloid tetramer of β-2-microglobulin from covalent labeling and mass spectrometry. Biochemistry. 50, 6711-6722 (2011).
- Zhou, Y., Vachet, R. W. Increased protein structural resolution from diethylpyrocarbonate-based covalent labeling and mass spectrometric detection. Journal of the American Society of Mass Spectrometry. 23, 708-717 (2012).
- Borotto, N. B., et al. Investigating Therapeutic Protein Structure with Diethylpyrocarbonate Labeling and Mass Spectrometry. Analytical Chemistry. 87, 10627-10634 (2015).
- Liu, T., Marcinko, T. M., Kiefer, P. A., Vachet, R. W. Using Covalent Labeling and Mass Spectrometry To Study Protein Binding Sites of Amyloid Inhibiting Molecules. Analytical Chemistry. 89, 11583-11591 (2017).
- Limpikirati, P., et al. Covalent labeling and mass spectrometry reveal subtle higher order structural changes for antibody therapeutics. MAbs. 11, 463-476 (2019).
- Limpikirati, P., Pan, X., Vachet, R. W. Covalent Labeling with Diethylpyrocarbonate: Sensitive to the Residue Microenvironment, Providing Improved Analysis of Protein Higher Order Structure by Mass Spectrometry. Analytical Chemistry. 91, 8516-8523 (2019).
- Liu, T., Limpikirati, P., Vachet, R. W. Synergistic Structural Information from Covalent Labeling and Hydrogen-Deuterium Exchange Mass Spectrometry for Protein-Ligand Interactions. Analytical Chemistry. 91, 15248-15254 (2019).
- Pan, X., Limpikirati, P., Chen, H., Liu, T., Vachet, R. W. Higher-Order Structure Influences the Kinetics of Diethylpyrocarbonate Covalent Labeling of Proteins. Journal of the American Society of Mass Spectrometry. 31, 658-665 (2020).
- Limpikirati, P. K., Zhao, B., Pan, X., Eyles, S. J., Vachet, R. W. Covalent Labeling/Mass Spectrometry of Monoclonal Antibodies with Diethylpyrocarbonate: Reaction Kinetics for Ensuring Protein Structural Integrity. Journal of the American Society of Mass Spectrometry. 31, 1223-1232 (2020).
- Liu, T., Marcinko, T. M., Vachet, R. W. Protein-Ligand Affinity Determinations Using Covalent Labeling-Mass Spectrometry. Journal of the American Society of Mass Spectrometry. 31, 1544-1553 (2020).
- Srikanth, R., Mendoza, V. L., Bridgewater, J. D., Zhang, G., Vachet, R. W. Copper Binding to β-2-Microglobulin and its Pre-Amyloid Oligomers. Biochemistry. 48, 9871-9881 (2009).
- Lim, J., Vachet, R. W. Using mass spectrometry to study copper-protein binding under native and non-native conditions: β-2-microglobulin. Analytical Chemistry. 76, 3498-3504 (2004).
- Lindsley, C. W. Predictions and Statistics for the Best-Selling Drugs Globally and in the United States in 2018 and a Look Forward to 2024 Projections. ACS Chemical Neuroscience. 10, 1115 (2019).
- Floege, J., Ketteler, M. β2-Microglobulin-derived amyloidosis: An update. Kidney International. 59, 164 (2001).
- Antwi, K., et al. Cu (II) organizes β-2-microglobulin oligomers but is released upon amyloid formation. Protein Science. 17, 748-759 (2008).
- Dong, J., et al. Unique Effect of Cu(II) in the Metal-Induced Amyloid Formation of β-2-Microglobulin. Biochemistry. 53, 1263-1274 (2014).
- Marcinko, T. M., Drews, T., Liu, T., Vachet, R. W. Epigallocatechin-3-gallate Inhibits Cu(II)-Induced β-2-Microglobulin Amyloid Formation by Binding to the Edge of Its β-Sheets. Biochemistry. 59, 1093-1103 (2020).
- Zhou, Y., Vachet, R. W. Diethylpyrocarbonate Labeling for the Structural Analysis of Proteins: Label Scrambling in Solution and How to Avoid it. Journal of the American Society of Mass Spectrometry. 23, 899-907 (2012).
- Borotto, N. B., Degraan-Weber, N., Zhou, Y., Vachet, R. W. Label scrambling during CID of covalently labeled peptide ions. Journal of the American Society of Mass Spectrometry. 25, 1739-1746 (2014).
- Aprahamian, M. L., Chea, E. E., Jones, L. M., Lindert, S. Rosetta Protein Structure Prediction from Hydroxyl Radical Protein Footprinting Mass Spectrometry Data. Analytical Chemistry. 90, 7721-7729 (2018).
- Schmidt, C., et al. Surface Accessibility and Dynamics of Macromolecular Assemblies Probed by Covalent Labeling Mass Spectrometry and Integrative Modeling. Analytical Chemistry. 89, 1459-1468 (2017).
- Zheng, X., Wintrode, P. L., Chance, M. R. Complementary Structural Mass Spectrometry Techniques Reveal Local Dynamics in Functionally Important Regions of a Metastable Serpin. Structure. 16, 38-51 (2008).
- Pan, Y., Piyadasa, H., O'Neil, J. D., Konermann, L. Conformational dynamics of a membrane transport protein probed by H/D exchange and covalent labeling: The glycerol facilitator. Journal of Molecular Biology. 416, 400-413 (2012).
- Li, J., et al. Mapping the Energetic Epitope of an Antibody/Interleukin-23 Interaction with Hydrogen/Deuterium Exchange, Fast Photochemical Oxidation of Proteins Mass Spectrometry, and Alanine Shave Mutagenesis. Analytical Chemistry. 89, 2250-2258 (2017).
- Borotto, N. B., Zhang, Z., Dong, J., Burant, B., Vachet, R. W. Increased β-Sheet Dynamics and D-E Loop Repositioning Are Necessary for Cu(II)-Induced Amyloid Formation by β-2-Microglobulin. Biochemistry. 56, 1095-1104 (2017).
- Shi, L., Liu, T., Gross, M. L., Huang, Y. Recognition of Human IgG1 by Fcγ Receptors: Structural Insights from Hydrogen-Deuterium Exchange and Fast Photochemical Oxidation of Proteins Coupled with Mass Spectrometry. Biochemistry. 58, 1074-1080 (2019).
- Gerega, S. K., Downard, K. M. PROXIMO - A new docking algorithm to model protein complexes using data from radical probe mass spectrometry (RP-MS). Bioinformatics. 22, 1702-1709 (2006).
- Kamal, J. K. A., Chance, M. R. Modeling of protein binary complexes using structural mass spectrometry data. Protein Science. 17, 79-94 (2007).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved