需要订阅 JoVE 才能查看此. 登录或开始免费试用。
Method Article
分离、表征和总 DNA 提取以鉴定真菌异养植物中的内生真菌
Erratum Notice
摘要
本文旨在为植物相关内生真菌的分离、分离株的长期保存、形态学表征和总 DNA 提取提供详细和充分的方案,以便随后进行分子鉴定和宏基因组分析。
摘要
霉菌异养植物是菌根依赖性最极端的形式之一,完全丧失了自养能力。与任何其他重要资源一样重要,与这些植物密切相关的真菌对它们来说是必不可少的。因此,研究真菌异养物种的一些最相关的技术是能够研究相关真菌的技术,尤其是那些栖息在根部和地下器官中的真菌。在这种情况下,通常应用鉴定培养依赖性和培养依赖性内生真菌的技术。分离真菌内生菌为形态学鉴定它们、分析它们的多样性和维持接种物提供了一种用于兰花种子共生萌发的方法。然而,众所周知,有大量不可培养的真菌栖息在植物组织中。因此,不依赖文化的分子鉴定技术提供了更广泛的物种多样性和丰度。本文旨在为启动两个调查程序提供必要的方法支持:一个是依赖于文化的调查程序,另一个是独立的调查程序。关于培养依赖性方案,详细介绍了从收集地点到实验室设施收集和维护植物样品的过程,以及从真菌异养植物的地下和地上器官中分离丝状真菌,保留分离物的集合,通过载玻片培养方法对菌丝进行形态学表征,以及通过总DNA提取对真菌进行分子鉴定。包括不依赖培养的方法,详细程序包括收集植物样本进行宏基因组分析和使用商业试剂盒从叶绿素植物器官中提取总DNA。最后,还建议使用连续性方案(例如,聚合酶链反应 [PCR]、测序)进行分析,并在此处介绍技术。
引言
根据定义,内生真菌是那些栖息在植物器官和组织内部的不明显感染(即不会对其宿主造成伤害)的真菌1,2。这些真菌可以与寄主植物进行中性或有益的相互作用,可能赋予对病原体和不利环境条件的抵抗力,并可能有助于植物有益化合物(例如生长因子和其他植物激素)的合成1,3。菌根内生菌是与植物建立菌根关联的真菌,参与养分转移4.在兰科中,与菌根内生菌的相互作用是绝大多数物种种子萌发和该科所有植物幼苗建立的基础5.在这种情况下,真菌异养兰花代表了对其菌根伙伴的完全依赖性,因为它们依赖于这些真菌在其整个生命周期中的矿物质营养物质和碳化合物转移6.因此,在研究真菌异养生命策略时,分离和鉴定关联真菌是基础。此外,人们对真菌内生菌在真菌异养植物中的作用知之甚少,甚至对这些真菌的真正多样性知之甚少 7,8.
内生真菌的研究 可以通过不同的技术 进行,传统上被描述为不依赖培养或依赖,例如:(a)直接观察,(b)真菌分离和形态学和/或分子鉴定,以及(c)植物组织的总DNA提取和分子鉴定9。在直接观察(a)中,内生真菌可以通过光学或电子显微镜9在植物细胞和组织内部进行研究,因为Pena-Passos等人10详细介绍了不同的显微镜方案。通过分离方法(b),可以根据真菌内生菌的菌落、菌丝以及生殖或抗性结构形态来表征真菌内生菌。此外, 通过 分离技术,可以通过DNA提取、分子识别序列(条形码或指纹图谱)的扩增和测序11来对分离株进行分子鉴定。后一种技术 (c) 能够在植物组织内部(元条形码)中通过 DNA 提取对内生真菌进行分子鉴定,然后进行文库制备和测序12。
此外,真菌分离株可以使用来自自养或真菌异养兰花的种子进行共生发芽试验。这种应用的一个例子是 Sisti 等人进行的研究13,描述了 Pogoniopsis schenckii(一种真菌异养兰)的原生球茎发育的萌发和初始阶段,与其一些分离株(包括非菌根内生真菌)相关。Pena-Passos等人10在视频中详细介绍了应用的共生萌发方案。分离与不同植物器官相关的真菌可以对植物-真菌相互作用的性质进行不同的研究(例如,理解这种关联的生态或生理方面,以及调查从真菌到植物的营养转移)9。
第 1 节中介绍的方法基于地下器官样本的收集,因为这些器官在收集方面存在最大的困难,并且由于菌根内生菌定植于它们,因此它们引起了人们的极大兴趣。然而,包含的方案(步骤1.1和1.2)都可以应用于其他真菌异养植物器官(例如,根茎,花茎和果实)。步骤1.1中描述的收集方法被指定用于分离内生真菌(第2节)进行形态学表征(第4节和第5节)和/或总DNA提取以进行分离物鉴定(第6节)。另一方面,步骤1.2中描述的收集方法专门用于植物组织的总DNA提取,用于元条形码技术(第7节)。在第 3 节中,介绍了四种丝状真菌储存和保存方法,其中两种用于短期储存(3-6 个月),另外两种适合长期储存(>1 年)。形态学特征(第 4 节和第 5 节)可能与分子鉴定相关联,以加强它并提供有关真菌宏观和微观形态学的重要信息。 图1 总结了随后描述的集体方法。

图1:所提出方法的示意图。 通过培养依赖性和独立性方法进行植物收集和真菌分离、保存和分子鉴定。 请点击这里查看此图的较大版本.
研究方案
1. 植物样本采集
- 培养依赖性方法的样品采集
- 小心挖掘地下器官;这些可以是要收集的植物的根、茎、根茎或储存器官。除了高度压实的土壤外,还要手工收集这些样品。
注意:在此步骤中使用抹子或勺子等工具是不明智的,因为它会破坏真菌异养植物的脆弱结构,并可能导致组织受到非内生真菌的污染。 - 尽可能多地收集地下器官。将样品保存在冷却容器内的纸袋中(例如,聚苯乙烯泡沫盒或带冰的保温袋)。如果还收集了空中器官,请将它们与地下器官分开运输。
- 小心挖掘地下器官;这些可以是要收集的植物的根、茎、根茎或储存器官。除了高度压实的土壤外,还要手工收集这些样品。
- 非培养方法的样品采集
- 根据步骤 1.1.1 的说明中指出的相同建议,仔细挖掘要收集的植物的根部。
- 尽可能多地收集地下器官。将收集的样品保存在液氮内的冷冻管中(理想的选择)或使用冰包围的离心管(替代选择)。如果收集,请单独维护空中器官。
2. 与植物器官相关的内生真菌的分离14
注意:本节中使用的每种材料、溶液和试剂都必须是无菌的。已经灭菌的不能购买的应在121°C高压灭菌20分钟。
- 植物器官的浅表灭虫
- 在流水中清洗收集的样品(步骤1.1.2),并尽可能多地去除样品可能具有的基质和其他碎屑。
- 在层流罩内,将洗涤后的样品浸没在70%乙醇中1分钟(可以使用烧杯或玻璃罐)。
- 将样品转移到另一个装有2%活性氯的次氯酸钠的容器中3分钟。
- 将样品更换为装有70%乙醇的容器中,并保持浸没1分钟。然后,用蒸馏水在两个容器中依次清洗样品。
- 在培养基中安装植物片段
注意: 步骤 2.2 中详述的每个步骤都必须在层流罩内进行。- 安装前,用 19.5 g/L 马铃薯葡萄糖琼脂培养基 (PDA) + 7 g/L 细菌琼脂 + 3 mL/L 抗生素(例如链霉素、青霉素、四环素、氨苄西林)制备培养皿(直径 8-10 cm)。
- 在使用之前,将培养皿与培养基在36°C下保持24小时,以确保没有污染。丢弃被细菌或真菌菌落污染的餐具。
注意:在此步骤中,在培养基中添加抗生素是必不可少的,因为器官被可能抑制真菌生长的细菌高度感染。验证抗生素在高压灭菌期间是否可以加热;在将培养基冷却至36°C左右(在将其放入培养皿之前)后,必须添加一些抗生素。 - 在对样品进行表面消毒后,仍然在层流罩内,在PDA中,立即从用于清洗样品的最后一个容器中接种几滴蒸馏水。此步骤对于评估样品表面除虫的功效非常重要。
- 在空的高压灭菌培养皿中,放置样品并使用火焰手术刀和镊子将样品切成约 0.2 厘米的厚度。
- 如果需要,将圆柱形样品纵向切成两半,以放大与介质接触的表面。为了更好地暴露水果和其他更球形的器官,请彻底切碎或切片结构。种子也可能是重要的真菌来源,因此请确保切片的果实将它们暴露在培养基中。
注意:只有健康的器官,没有组织损伤或可能的疾病或致病性感染的信号,才应该用于真菌内生菌分离。 - 用PDA +抗生素在培养皿中分布地下器官的五个片段。确保碎片彼此尽可能远离,并且不要接触盘子边缘。不要在培养基上排列任何片段。准备每个安装器官的副本,以防污染。
- 用保鲜膜密封培养皿,并将它们在25-27°C的黑暗中储存(最好在培养箱中)5天。
- 隔离频率 (IF) 演算11
- 器官片段孵育 5 天后,根据呈现真菌菌落生长的孵育片段数除以孵育片段总数计算 IF,如以下公式所示:
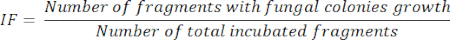
- 器官片段孵育 5 天后,根据呈现真菌菌落生长的孵育片段数除以孵育片段总数计算 IF,如以下公式所示:
- 通过条纹和传代培养纯化真菌分离株
注意: 步骤 2.4 中详述的每个步骤都必须在层流罩内进行。- 用5-7 g/L琼脂(AA;仅限细菌琼脂)制备培养皿(直径5厘米)。将培养皿在36°C下保持24小时,以消除可能被污染的培养皿。
- 用代码识别每个生长的真菌菌落,并在培养皿的底部划定其边缘(可以使用永久性记号笔完成)。通过颜色、生长模式、质地和边缘格式区分菌落。
- 用高压灭菌的木制牙签,使用细尖,从真菌菌落中回收少量菌丝体。最好集中在菌落边缘,并尽可能选择远离另一个菌落的区域,避免同时恢复多种类型的菌落。
- 使用同一根牙签,尖端有菌丝体,划出 AA,产生三个条纹(凹槽)。确保每个条纹与另一个条纹和培养皿边缘的距离为 1 厘米。写下适当的代码(使用贴纸和铅笔),密封培养皿,并在25-27°C的黑暗中保持孵育3天。
- 准备含有 39 g/L PDA 的培养皿(直径 8 厘米)。在这个阶段没有必要添加抗生素。
- 孵育 AA 培养皿后,在光线下(来自灯或窗户)仔细观察它们,旨在识别形成单个菌落的细小菌丝。使用培养皿底部的永久性标记划定每个培养皿中单个菌落的面积。
- 在层流罩中,使用高压灭菌的牙签切割含有菌落的培养基的一部分,并将切割的体积转移到新 PDA 培养皿的中心。
- 用分离物的代码识别培养皿,用保鲜膜密封培养皿,并在25-27°C的黑暗中保持7-14天。
3. 纯化真菌分离株的保存
注意:本节中使用的每种材料、溶液和试剂都必须是无菌的。已经灭菌的不能购买的应在121°C高压灭菌20分钟。
- 用卡斯特拉尼法或矿物油保存(3-6个月)11,15
- 用 0.5 mL 蒸馏水或含有 0.5 mL 矿物油(取决于所选方法)制备 2 mL 微量离心管。确保管子是空的,并将高压灭菌的水和油添加到层流罩内的管子中。
- 在层流罩中,将培养皿与已经在PDA(39g / L)中生长的纯化分离物放置7-14天。使用高压灭菌的牙签,切割含有菌丝体边缘的培养基的小长方体(上部区域为0.5cm×0.5cm)。
- 将四到六个长方体放入装有蒸馏水(Castellani方法)或矿物油的微量离心管中。根据需要将试管在25°C的黑暗中储存,并遵守方法的时间限制。
注意: 避免在试管中添加过多的长方体并使其充满,这可能会增加污染的机会。可以将试管保持冷藏状态,这可能有利于将某些分离物保存更长时间。Currah等人16 建议将热带兰花的菌根真菌分离株的复制品储存在冷藏和25°C下,因为它们在冷藏时可能会丢失。 - 回收一个长方体并将其放置在新PDA培养皿的中心,以生长储存的分离物。
- 未脱壳稻粒的冷冻保存(>1 年)17
- 将未去壳的米粒在流水中清洗并煮至稻壳开始打开。将煮熟的谷物分布在带有螺旋盖的玻璃试管中,高压灭菌器两次,中间间隔24小时。
- 在层流罩中,将培养皿与已经在PDA(39g / L)中生长的纯化分离物放置7-14天。使用高压灭菌的牙签,从菌丝体边缘恢复五个小菌丝碎片,并将它们接种在含有高压灭菌米粒的管中。
注意:在管的不同点和深度接种菌丝可保证分离物在更短的时间内定植未去壳的米粒。 - 将试管在25-27°C的黑暗中孵育14天。每 3 天涡旋一次,搅拌试管,以保持谷物个性化。
- 在观察米粒中的真菌生长后,将谷物分布在高压灭菌的培养皿中,该培养皿内衬有滤纸以吸收水分,在纸上,使谷物干燥。在25-27°C的黑暗中储存2-3天。
- 在底部用 1/3 硅胶和在二氧化硅上方用 1/3 玻璃棉制备冷冻管。最后,将 1/3 的米粒与生长的真菌菌丝体分布在一起。在-20°C储存24小时。
- 24小时后,根据需要将冷冻管储存在-80°C,观察方法的时间限制。
- 使用冷冻保护剂在蛭石中冷冻保存(>1 年)18
- 在蒸馏水中制备由0.2%酵母提取物和2%葡萄糖组成的液体培养基。将pH调节至5并高压灭菌。
- 将0.2g蛭石(使用精细粒度法)分布在带有螺旋盖的冷冻管中,并对其进行高压灭菌。向含有蛭石的冷冻管中加入 0.8 mL 高压灭菌液体培养基。
- 在层流罩中,将培养皿与已经在PDA(39g / L)中生长的纯化分离物放置7-14天。使用高压灭菌的牙签,从菌丝体边缘恢复三到五个菌丝片段,并沿着含有蛭石+液体培养基的管接种。请记住使用相应的分离代码识别冷冻管。
注意:在冷冻管的不同点和深度接种菌丝可保证分离物在更短的时间内定植蛭石。 - 将冷冻管储存在25-27°C的黑暗中,直到可以观察到大多数蛭石颗粒的定植,这通常需要大约14天。
- 在蒸馏水中制备由5%甘油和5%海藻糖组成的冷冻保护剂溶液,并将其高压灭菌。使用前让溶液冷却。
- 在冷冻管中的蛭石被相应的真菌分离物定植后,在每个管中分配0.4mL冷冻保护剂,并将冷冻管在4°C下冷藏48小时。之后,根据需要将冷冻管保持在-80°C,观察方法的时间限制。
4. 丝状真菌的宏观形态学特征(菌落形态学)
- 用39 g / L PDA保持每个培养皿中生长的菌丝体的照相记录7-14天。请记住记录菌落的两侧,上面和下面(反向)。如果盘子在第 6 节中很容易使用或没有维护,请在拍照时打开盘子以获得更好的照片。
- 如果对菌落形态的定量数据感兴趣,传代培养复制分离株并将其保持在相同的生长条件下并保持恒定的时间,以记录菌落直径并计算生长速率(通常以mm / h为单位)。
注:通过使用不同类型的培养基进行比较19 并考虑使用统计工具处理数据,可以获得更复杂的定量结果。 - 在立体显微镜下观察菌落,以识别形态特征并在放大镜下拍照。根据菌落的宏观形态特征评估菌落,详见结果部分。查阅不同的书目来源,以帮助对宏观形态学进行分类并将其与分子和/或形态学鉴定联系起来。
5. 丝状真菌的微形态学表征(菌丝形态学)
注意:讨论部分比较了微形态学技术,考虑了它们的可能用途和缺点。
- 用 39 g/L PDA 在培养皿上培养分离株 7-14 天。为了评估假定的兰花菌根真菌,在 17 g/L 玉米粉琼脂(CMA;或其他营养限制培养基)上生长分离株3-7 天 19.
- 挑逗支架
- 在层流罩内工作。将一滴选定的污渍(步骤5.5)放在干净的载玻片上。
- 使用高压灭菌的牙签或其他无菌材料,小心地从生长的分离物中取出一些菌丝,并将它们放入污渍滴中。放置盖玻片(最好以 45° 的初始角度以避免气泡)并在光学显微镜下进行分析。
- 胶带安装
注意:该技术通常应用于真菌培养物中,以便在第 6 节中易于使用或不维护,因为胶带不能高压灭菌,并且在执行该方法后可能会发生污染。- 将一滴选定的污渍(步骤5.5)放在干净的载玻片上。剪下一条透明胶带,其尺寸适合载玻片和中央污渍滴。
- 将条带的粘性表面引导到菌丝体表面。不要按压,并尝试收集一些菌丝,但不要粘太多菌丝。
- 将胶带粘在载玻片上,确保污渍与收集的菌丝接触。在胶带上方滴一滴水,然后盖玻片。在光学显微镜下分析载玻片。
- 丝状真菌20的载玻片培养
- 在一个大的培养皿(最好是直径>9厘米)内,放置:底部的滤纸,U形玻璃棒或适应装置(目的是为载玻片提供仰角),一个载玻片和两个盖玻片。较高的培养皿便于操作。高压灭菌这些培养皿试剂盒,每个分离株一个。
- 准备少量 39 g/L PDA 或 17 g/L CMA,加入 10 g/L 细菌琼脂,然后高压灭菌(一个培养皿的体积足以容纳 30 多个分离株)。玻片培养中使用的培养基必须比普通培养基更硬。高压灭菌器 100 mL 蒸馏水。
- 在层流罩内工作。将液体培养基放入培养皿中,形成一层约0.5厘米高的层。让介质凝固。一旦凝固,使用无菌手术刀切开尺寸为 1 cm x 1 cm 的培养基正方形。
- 打开层流罩内的培养皿套件,并使用火焰镊子组织培养皿内高压灭菌的材料。按编号顺序排列材料,如 图2A所示,将正方形培养基放在载玻片上。在将盖玻片放在培养基上之前,请继续执行步骤5.4.5。
- 使用高压灭菌的牙签从分离物中回收一些菌丝,并小心地擦拭放置在载玻片上的培养基的四个侧面。使用火焰镊子将高压灭菌的盖玻片放在培养基方块上。
- 使用无菌移液器吸头将高压灭菌的水放入滤纸中,以形成一个潮湿的腔室。使用一卷纸使纸张饱和,不要放过多的水。用保鲜膜密封培养皿,并用相应的隔离代码进行识别。将盘子在25-27°C的黑暗中保持3-7天。
- 评估载玻片和盖玻片中的菌丝生长。从每个载玻片培养试剂盒中产生两张载玻片,一张使用带有菌丝的载玻片和高压灭菌的盖玻片,第二张使用带有菌丝的盖玻片和另一张载玻片。
- 观察到菌丝生长后,小心地将培养基的正方形从载玻片上分离(图2B),使用选定的染色剂(步骤5.5)安装两个载玻片,并在光学显微镜下进行分析。请记住识别幻灯片上的隔离代码。要生产半永久性载玻片,请用透明指甲油密封盖玻片。
- 要产生永久性载玻片,染色后小心地从粘附的菌丝中洗涤染料,然后让载玻片干燥。使用快速安装介质进行安装(有关详细信息,请阅读 Pena-Passos 等人 10)。
注意:使用这种技术生产永久性载玻片的缺点是可以清洗未粘附在玻璃上的孢子和结构。
- 染色方法和菌丝观察
- 乳酚棉蓝 (LPCB)21:加入 20 mL 乳酸、40 mL 甘油和 20 mL 蒸馏水。通过轻轻加热将20g苯酚晶体溶解在该溶液中。溶解 0.05 g 甲基蓝(棉蓝,或 2 mL 1% 水溶液)。LPCB也可以很容易地购买已经准备好。
注意:苯酚具有剧毒和挥发性;只能在通风橱内操作它并戴上手套。 - 甲苯胺蓝O 10(TBO):在0.1M磷酸盐缓冲液(pH 6.8)中制备0.05%TBO。
- 刚果红22,23,24:在蒸馏水中制备 1% 刚果红的溶液并过滤。孵育 5-10 分钟。该染料也可用于荧光显微镜25。
- 在光学显微镜下观察和拍摄真菌菌丝时,请查阅不同的书目来源以帮助识别结构(查看讨论)。
- 乳酚棉蓝 (LPCB)21:加入 20 mL 乳酸、40 mL 甘油和 20 mL 蒸馏水。通过轻轻加热将20g苯酚晶体溶解在该溶液中。溶解 0.05 g 甲基蓝(棉蓝,或 2 mL 1% 水溶液)。LPCB也可以很容易地购买已经准备好。

图2:丝状真菌的载玻片培养程序 。 (A) 载玻片培养试剂盒的示意图配置,其中数字表示元件排列的顺序。(B)在载玻片和盖玻片中观察到菌丝生长后培养基的方块分离。 请点击这里查看此图的较大版本.
6. 从真菌分离株中提取总 DNA(自制方案26 与修改 27)
注意:本节中使用的每种材料、溶液和试剂都必须是无菌的。已经灭菌的不能购买的应在121°C高压灭菌20分钟。在整个实验过程中戴上手套,并在通风橱内执行某些阶段。
- 制备提取缓冲液:1%十二烷基硫酸钠(SDS),250mM NaCl,200mM Tris-HCl(pH 8.0)和25mM乙二胺四乙酸(EDTA)。将纯化的分离株在 39 g/L PDA 中生长 7-14 天。
- 用刮刀或勺子小心地从分离物中刮下菌丝体,并将碎片转移到高压灭菌的研钵中,避免将培养基与菌丝聚集体转移。使用瓷杵,用液氮将菌丝体研磨成细粉。不要让被磨碎的菌丝体融化,添加氮气以避免融化。
- 在 2 mL 微量离心管中加入 1 mL 提取缓冲液,并将研磨样品放置至 1.5 mL 标记。轻轻搅拌试管的内容物以使其均匀化。
- 在涡旋中搅拌管5秒,并将它们置于65°C的热块中20分钟。每7-10分钟通过倒置仔细均匀地使内容物均匀。
- 在4°C下以10,000× g 离心管10分钟。 将 800 μL 从上层相转移到新的 2 mL 微量离心管中,向管中加入 800 μL 苯酚,并通过倒置混合内容物。
注意:苯酚是一种剧毒和挥发性试剂;它可引起红斑、坏疽和组织坏死。吸入时,可引起呼吸困难和咳嗽。全身吸收会损害肝脏、肾脏和中枢神经系统。仅在通风橱内操作苯酚并戴上手套。
注意:从下一步开始,戴上手套并在通风橱内执行协议。 - 在4°C下以10,000× g 离心管10分钟,并将800μL从上相转移到新的2mL管中,小心避免劣等相内容物的转移。
- 向试管中加入 400 μL 苯酚和 400 μL 氯仿,倒置混合内容物。在4°C下以10,000× g 离心10分钟。
注意:氯仿是一种剧毒和挥发性试剂;它与皮肤接触时会引起刺激和伤害,如果吸入,会影响中枢神经和心肺系统、肝脏和肾脏。仅在通风橱内操作氯仿并戴上手套。 - 将 800 μL 或更少的高级相回收到新的 2 mL 试管中。加入800μL氯仿,倒置混合内容物,并在4°C下以10,000× g 离心10分钟。
注意:在此步骤中,不得将下相的残留物转移到新管中。因此,在恢复上相时要格外小心。 - 将 600-800 μL 从上层相转移到新的 1.5 mL 试管中,并加入 450 μL 异丙醇。倒置混合内容物,并在25°C孵育5分钟。
- 将试管在4°C下以10,000× g 离心5分钟,并使用微量移液管弃去上清液。小心不要丢弃沉积在管底部的颗粒。
- 加入 500 μL 80% 乙醇并离心 5 分钟。重复两次,以防颗粒未澄清。
- 使用微量移液管除去乙醇,并将沉淀在37°C干燥30-60分钟。加入 30-50 μL 去离子水,使用微量移液管洗脱沉淀。
- 将试管保持在4°C过夜以完全洗脱DNA,并将内容物冷冻在-20°C。
7. 从植物器官中提取用于元条形码方法的总 DNA(商业试剂盒)
注意:对于以下方法,有必要购买 材料表 中指示的商业试剂盒作为土壤 DNA 提取试剂盒。本节中使用的每种材料、溶液和试剂都必须是无菌的。已经灭菌的不能购买的应在121°C高压灭菌20分钟。强烈建议在整个实验过程中戴上手套,这些步骤可以在层流罩内进行。所描述的方案是从De Souza等人12修改而来的,来自制造商详细阐述的方案。
- 使用瓷杵和研钵,在液氮中研磨按照步骤1.2收集的根,将样品还原成细粉。将 0.3 g 研磨样品加入 PowerBead 管中,轻轻搅拌以均匀化。
- 向 PowerBead 管中加入 60 μL 溶液 C1(包含在试剂盒中),并通过倒置混合内容物。使用组织匀浆器和细胞裂解器(材料表),将管牢固地绑在足够的支架上,将支架耦合到涡流上,并以最大速度启动设备10-20分钟。
注意:如果C1溶液沉淀,请在60°C下加热直至完全溶解。 - 在25°C下以10,000× g 离心管30秒。 将 500 μL 上清液转移到 2 mL 微量离心管中。转移的内容物可能是颗粒物。
- 向试管中加入 250 μL 溶液 C2,并在涡旋中搅拌 5 秒。在4°C孵育5分钟,然后在25°C下以10,000× g 离心1-2分钟。
- 将超过 600 μL 的上清液转移到新的 2 mL 管中,避免沉淀。加入 200 μL 溶液 C3,在涡旋上搅拌 5 秒。
- 在4°C孵育5分钟,然后在25°C下以10,000× g 离心1分钟。 在此阶段,确保上清液不是颗粒。
- 将超过 750 μL 的上清液转移到新的 2 mL 试管中,避免沉淀。将溶液C4均匀化,向上清液中加入1,100μL溶液C4,并在涡旋中搅拌5秒。
- 从MB离心柱中的试管内容物中加载675μL,在过滤器上,并在25°C下以10,000× g 离心1分钟。 丢弃液体内容物。
- 重复上一步两次,直到处理完每个试管的所有内容物。然后,在管上层柱的过滤器中心加入500μL溶液C5,并在25°C下以10,000× g 离心30秒。
- 弃去液体内容物,并在与上一步相同的条件下再次离心。小心地将MB离心柱的上层柱转移到新的2 mL微量离心管中,避免滴落柱中的任何液体内容物。
- 将 85-100 μL 溶液 C6 加入过滤器中心并等待 1 分钟。在25°C下以10,000× g 离心30秒,弃去MB离心柱。将试管储存在-80°C。
8. 分光光度计中的DNA定量(查看材料表)
- 使用指示的分光光度计,打开软件。选择" 核酸"选项,选择 "DNA"选项,并将浓度设置为 ng/μL。
- 向分光光度计检测器中加入 1 μL 去离子水,并通过选择 空白选项进行校准。阅读后,用软纸轻轻擦拭检测器。
- 用要读取的样品的隔离代码命名现场样品,并将其 1 μL 放入设备检测器中。选择" 测量"选项;将生成一个图形和一个包含结果的表格。
- 重复步骤8.3,直到读取完所有样品。建议保存包含生成结果的表格以供记录和分析:选择 "报告 "选项和保存.xml文件的位置。
- 向检测器中加入 5 μL 去离子水,等待几分钟,然后用软纸轻轻擦拭。
结果
在分离方案中,考虑到最后一次洗涤时使用的水存在污染,并且在带有接种片段的培养皿中也检测到污染,根据污染物的类型,可以采取不同的措施(表1)。如果高度孢子化真菌污染物(也呈现加速生长)和强烈繁殖的细菌,对所选抗生素具有抗药性,则必须从一开始就重复此过程。相反,如果真菌污染物生长缓慢或没有孢子,则可以简单地避免在方案的后?...
讨论
植物样品的表面灭虫是所提出的协议中最关键的阶段之一。PDA培养皿中不会受到上次洗涤的液滴污染,这是非常可取的。细菌经常在隔离皿中被观察到为污染物,通常比空气中的孢子真菌更多,因为内生细菌在植物组织中也很常见3,11。因此,在安装器官碎片时,在培养基中添加抗生素是必不可少的。当组合不同的抗生素类型时,可以获得更好的效果,?...
披露声明
作者没有什么可披露的,也没有利益冲突。
致谢
我们感谢 FAPESP (2015/26479-6) 和 CNPq (447453/2014-9) 的资助。JLSM 感谢 CNPq 提供的生产力资助 (303664/2020-7)。MPP 感谢 Capes(硕士学位奖学金,流程 88887.600591/2021-00)和 CNPq。
材料
| Name | Company | Catalog Number | Comments |
| Adhesive tape | (from any company, for adhesive tape mount in micromorphological analyses) | ||
| Ampicillin | Sigma-Aldrich | A5354 | (for installation of plant fragments; other antibiotics may be used - check step 2.2.1) |
| Autoclave | (from any company, for materials sterilization in many steps) | ||
| Bacteriological agar | Sigma-Aldrich | A1296 | (for many steps) |
| C1, C2, C3, C4, C5, and C6 solutions | Qiagen | 12888-50 | (purchased with DNeasy PowerSoil kit) |
| Centrifuge | Merck/Eppendorf | 5810 G | (for total DNA extraction from fungal isolates) |
| Centrifuge tubes | Merck | CLS430828 | (for samples collection) |
| Chloroform | Sigma-Aldrich | C2432 | (for total DNA extraction from fungal isolates) |
| Congo red | Supelco | 75768 | (for hyphae staining) |
| Cryotubes | Merck | BR114831 | (for many steps) |
| Ethanol | Supelco | 100983 | It will be necessary to carry out the appropriate dilutions (for many steps) |
| Ethylenediaminetetraacetic acid (EDTA) | Sigma-Aldrich | 3609 | (for total DNA extraction from fungal isolates) |
| Filter paper | Merck | WHA10010155 | (for many steps) |
| Glass test tubes | Merck | CLS7082516 | (for cryopreservation in unhulled rice grains) |
| Glass wool | Supelco | 20411 | (for cryopreservation in unhulled rice grains) |
| Glucose | Sigma-Aldrich | G8270 | Or dextrose (for cryopreservation in vermiculite) |
| Glycerol | Sigma-Aldrich | G5516 | Or glycerin (for cryopreservation in vermiculite, for preparing LPCB) |
| Isopropanol | Sigma-Aldrich | 563935 | (for total DNA extraction from fungal isolates) |
| Lactic acid | Sigma-Aldrich | 252476 | (for preparing LPCB - hyphae staining) |
| Lactophenol blue solution (LPCB) | Sigma-Aldrich | 61335 | (for hyphae staining) |
| Laminar flow hood | (class I, from any company, for many steps) | ||
| Light microscope | (from any company, for hyphae observation) | ||
| MB Spin Columns | Qiagen | 12888-50 | (purchased with DNeasy PowerSoil kit) |
| Methyl blue (cotton blue) | Sigma-Aldrich | M5528 | (for preparing LPCB - hyphae staining) |
| Microcentrifuge tube (1.5 mL) | Merck | HS4323 | (for total DNA extraction from fungal isolates) |
| Microcentrifuge tube (2 mL) | Merck | BR780546 | (for many steps) |
| Mineral oil | (for preservation of fungal isolates) | ||
| Paper bags | Average size 150 mm x 200 mm (for samples collection) | ||
| Petri dish (Glass, 120 mm x 20 mm) | Merck/Pyrex | SLW1480/10D | (autoclavable, for fungi slide culture, prefer higher ones) |
| Petri dish (Glass, 50 mm x 17 mm) | Merck/Aldrich | Z740618 | (for purification of fungal isolates); alternatively: polystyrene petri dishes (sterile, γ-irradiated, non-autoclavable) |
| Petri dish (Glass, 80 mm x 15 mm) | Merck/Brand | BR455732 | (for installation of plant fragments); alternatively: polystyrene petri dishes (sterile, γ-irradiated, non-autoclavable) |
| Phenol | Sigma-Aldrich | P1037 | (for total DNA extraction from fungal isolates, for preparing LPCB) |
| Porcelain mortar | Sigma-Aldrich | Z247464 | (for total DNA extraction from fungal isolates) |
| Porcelain pestle | Sigma-Aldrich | Z247502 | (for total DNA extraction from fungal isolates) |
| Potato dextrose agar (PDA) | Millipore | P2182 | (for many steps) |
| PowerBead tubes | Qiagen | 12888-50 | (purchased with DNeasy PowerSoil kit) |
| Rapid mounting medium (Entellan) | Sigma-Aldrich | 1.0796 | (for fungi slide culture) |
| Silica gel | Supelco | 717185 | (for cryopreservation in unhulled rice grains) |
| Sodium chloride (NaCl) | Sigma-Aldrich | S9888 | (for total DNA extraction from fungal isolates) |
| Sodium dodecyl sulfate (SDS) | Sigma-Aldrich | L3771 | Lauryl sulfate sodium salt (for total DNA extraction from fungal isolates) |
| Sodium hypochlorite (w/ 2% active chlorine) | (commercial product, for superficial desinfestation) | ||
| Soil DNA extraction kit (DNeasy PowerSoil kit) | Qiagen | 12888-50 | (for total DNA extraction from plant organs) |
| Spectrophotometer - Nanodrop 2000/2000c | ThermoFisher Scientific | ND2000CLAPTOP | (for total DNA extraction from plant organs) |
| Stereomicroscope | (=dissecting microscope, from any company, for macromorphological analyses) | ||
| Tetracycline | Sigma-Aldrich | T7660 | (for installation of plant fragments) |
| Thermoblock | Merck/Eppendorf | EP5362000035 | (or from other companies) |
| Tissue homogenizer and cell lyzer | SPEX SamplePrep | 2010 Geno/Grinder - Automated Tissue Homogenizer and Cell Lyzer (for total DNA extraction from plant organs) | |
| Toluidine blue O | Sigma-Aldrich/Harleco | 364-M | (for hyphae staining) |
| Trehalose | Sigma-Aldrich | T9531 | (for cryopreservation in vermiculite) |
| Tris Base Solution (Tris) | Sigma-Aldrich | T1699 | (for total DNA extraction from fungal isolates) |
| Unhulled rice grains | (for cryopreservation) | ||
| U-shaped glass rod | (or an adaptation - check step 5.4.1, for fungi slide culture) | ||
| Vermiculite | Fine granulometry (for cryopreservation in vermiculite) | ||
| Vortexer | Sigma-Aldrich/BenchMixer | BMSBV1000 | (for total DNA extraction from fungal isolates) |
| Yeast extract | Sigma-Aldrich | Y1625 | (for cryopreservation in vermiculite) |
参考文献
- de Azevedo, J. L. Endophytic microorganisms. Ecologia Microbiana. , 117-137 (1998).
- Stone, J. K., Bacon, C. W., White, J. F. An overview of endophytic microbes: endophytism defined. Microbial Endophytes. , 17-44 (2000).
- Schulz, B., Boyle, C. What are Endophytes. Microbial Root Endophytes. , 1-13 (2006).
- Smith, S. E., Read, D. J. . Mycorrhizal Symbiosis. , (2008).
- Rasmussen, H. N., Dixon, K. W., Jersáková, J., Těšitelová, T. Germination and seedling establishment in orchids: a complex of requirements. Annals of Botany. 116 (3), 391-402 (2015).
- Rasmussen, H. N., Rasmussen, F. N. Orchid mycorrhiza: implications of a mycophagous life style. Oikos. 118 (3), 334-345 (2009).
- Ma, X., Kang, J., Nontachaiyapoom, S., Wen, T., Hyde, K. D. Non-mycorrhizal endophytic fungi from orchids. Current Science. 109 (1), 72-87 (2015).
- Favre-Godal, Q., Gourguillon, L., Lordel-Madeleine, S., Gindro, K., Choisy, P. Orchids and their mycorrhizal fungi: an insufficiently explored relationship. Mycorrhiza. 30 (1), 5-22 (2020).
- Sun, X., Guo, L. -. D. Endophytic fungal diversity: review of traditional and molecular techniques. Mycology. 3 (1), 65-76 (2012).
- Pena-Passos, M., Sisti, L. S., Mayer, J. L. S. Microscopy techniques for interpreting fungal colonization in mycoheterotrophic plants tissues and symbiotic germination of seeds. Journal of Visualized Experiments. (183), e63777 (2022).
- Araújo, W. L., et al. . Endophytic microorganisms: Theoretical and Practical Aspects of Isolation and Characterization. 1st ed. 1, 257 (2014).
- de Souza, R. S. C., et al. Unlocking the bacterial and fungal communities assemblages of sugarcane microbiome. Scientific Reports. 6, 28774 (2016).
- Sisti, L. S., et al. The role of non-mycorrhizal fungi in germination of the mycoheterotrophic orchid Pogoniopsis schenckii Cogn. Frontiers in Plant Science. 10, 1589 (2019).
- Araújo, W. L., et al. Variability and interactions between endophytic bacteria and fungi isolated from leaf tissues of citrus rootstocks. Canadian Journal of Microbiology. 47 (3), 229-236 (2001).
- Castellani, A. Further researches on the long viability and growth of many pathogenic fungi and some bacteria in sterile distilled water. Mycopathologia. 20 (1-2), 1-6 (1963).
- Currah, R. S., Zelmer, C. D., Hambleton, S., Richardson, K. A. Fungi from orchid mycorrhizas. Orchid Biology: Reviews and Perspectives, VII. , 117-170 (1997).
- Freitas, E. F. S., et al. Diversity of mycorrhizal Tulasnella associated with epiphytic and rupicolous orchids from the Brazilian Atlantic Forest, including four new species. Scientific Reports. 10 (1), 7069 (2020).
- Sato, M., Inaba, S., Noguchi, M., Nakagiri, A. Vermiculite as a culture substrate greatly improves the viability of frozen cultures of ectomycorrhizal basidiomycetes. Fungal Biology. 124 (8), 742-751 (2020).
- Pereira, O. L., Kasuya, M. C. M., Borges, A. C., Araújo, E. F. D. Morphological and molecular characterization of mycorrhizal fungi isolated from neotropical orchids in Brazil. Canadian Journal of Botany. 83 (1), 54-65 (2005).
- Riddell, R. W. Permanent stained mycological preparations obtained by slide culture. Mycologia. 42 (2), 265-270 (1950).
- Walsh, T. J., Hayden, R. T., Larone, D. H. . Larone's Medically Important Fungi: A Guide to Identification. , (2018).
- Microscopy: Chemical Reagents. British Mycological Society Available from: https://www.britmycolsoc.org.uk/field_mycology/microscopy/reagents (2022)
- Senanayake, I. C., et al. Morphological approaches in studying fungi: Collection, examination, isolation, sporulation and preservation. Mycosphere. 11 (1), 2678-2754 (2020).
- Slifkin, M., Cumbie, R. Congo red as a fluorochrome for the rapid detection of fungi. Journal of Clinical Microbiology. 26 (5), 827-830 (1988).
- Raeder, U., Broda, P. Rapid preparation of DNA from filamentous fungi. Letters in Applied Microbiology. 1 (1), 17-20 (1985).
- Martins, M. K., et al. Molecular characterization of endophytic microorganisms. Endophytic microorganisms: theoretical and practical aspects of isolation and characterization. 1st edition. , 189-211 (2014).
- Rayner, R. W. A Mycological Colour Chart. Commonwealth Mycological Institute. , (1970).
- Kornerup, A., Wanscher, J. H. . Methuen Handbook of Colour. Methuen handbook of colour. , (1967).
- Ridgway, R. . Color Standards and Color Nomenclature. , (1912).
- McGinnis, M. R. . Laboratory Handbook of Medical Mycology. , (2012).
- Webster, J., Weber, R. . Introduction to Fungi. , (2007).
- Sridharan, G., Shankar, A. A. Toluidine blue: A review of its chemistry and clinical utility. Journal of Oral and Maxillofacial Pathology. 16 (2), 251-255 (2012).
- Smith, D., Onions, A. H. S. A comparison of some preservation techniques for fungi. Transactions of the British Mycological Society. 81 (3), 535-540 (1983).
- Ryan, M. J., Smith, D., Jeffries, P. A decision-based key to determine the most appropriate protocol for the preservation of fungi. World Journal of Microbiology and Biotechnology. 16 (2), 183-186 (2000).
- Lalaymia, I., Cranenbrouck, S., Declerck, S. Maintenance and preservation of ectomycorrhizal and arbuscular mycorrhizal fungi. Mycorrhiza. 24 (5), 323-337 (2014).
- Zettler, L. W., Corey, L. L. Orchid mycorrhizal fungi: isolation and identification techniques. Orchid Propagation: From Laboratories to Greenhouses-Methods and Protocols. , 27-59 (2018).
- Yu, S., Wang, Y., Li, X., Yu, F., Li, W. The factors affecting the reproducibility of micro-volume DNA mass quantification in Nanodrop 2000 spectrophotometer. Optik. 145, 555-560 (2017).
- Martos, F., et al. Independent recruitment of saprotrophic fungi as mycorrhizal partners by tropical achlorophyllous orchids. New Phytologist. 184 (3), 668-681 (2009).
- Schoch, C. L., et al. Nuclear ribosomal internal transcribed spacer (ITS) region as a universal DNA barcode marker for Fungi. Proceedings of the National Academy of Sciences. 109 (16), 6241-6246 (2012).
- White, T. J., Bruns, T., Lee, S., Taylor, J. Amplification and direct sequencing of fungal ribosomal RNA genes for phylogenetics. PCR Protocols: A Guide to Methods and Applications. 18 (1), 315-322 (1990).
- Sanger, F., Nicklen, S., Coulson, A. R. DNA sequencing with chain-terminating inhibitors. Proceedings of the National Academy of Sciences. 74 (12), 5463-5467 (1977).
- Ranjard, L., et al. Characterization of bacterial and fungal soil communities by automated ribosomal intergenic spacer analysis fingerprints: biological and methodological variability. Applied and Environmental Microbiology. 67 (10), 4479-4487 (2001).
- Metzker, M. L. Sequencing technologies-the next generation. Nature Reviews Genetics. 11 (1), 31-46 (2010).
Erratum
Formal Correction: Erratum: Isolation, Characterization, and Total DNA Extraction to Identify Endophytic Fungi in Mycoheterotrophic Plants
Posted by JoVE Editors on 1/01/1970. Citeable Link.
An erratum was issued for: Isolation, Characterization, and Total DNA Extraction to Identify Endophytic Fungi in Mycoheterotrophic Plants. The Authors section was updated from:
Juliana Lishcka Sampaio Mayer
to:
Juliana Lischka Sampaio Mayer
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。