Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Isolierung, Charakterisierung und Gesamt-DNA-Extraktion zur Identifizierung endophytischer Pilze in mykoheterotrophen Pflanzen
In diesem Artikel
Erratum Notice
Zusammenfassung
Der vorliegende Artikel zielt darauf ab, detaillierte und adäquate Protokolle für die Isolierung pflanzenassoziierter endophytischer Pilze, die Langzeitkonservierung von Isolaten, die morphologische Charakterisierung und die Gesamt-DNA-Extraktion für die anschließende molekulare Identifizierung und metagenomische Analyse bereitzustellen.
Zusammenfassung
Mykoheterotrophe Pflanzen stellen eine der extremsten Formen der Mykorrhizaabhängigkeit dar, da sie ihre autotrophe Kapazität vollständig verloren haben. Die Pilze, mit denen diese Pflanzen eng verbunden sind, sind für sie so wichtig wie jede andere lebenswichtige Ressource. Daher sind einige der relevantesten Techniken zur Untersuchung mykoheterotropher Arten diejenigen, die die Untersuchung assoziierter Pilze ermöglichen, insbesondere solcher, die Wurzeln und unterirdische Organe bewohnen. In diesem Zusammenhang werden häufig Techniken zur Identifizierung kulturabhängiger und kulturunabhängiger endophytischer Pilze eingesetzt. Die Isolierung von Pilzendophyten bietet die Möglichkeit, sie morphologisch zu identifizieren, ihre Diversität zu analysieren und Inokula für Anwendungen in der symbiotischen Keimung von Orchideensamen zu erhalten. Es ist jedoch bekannt, dass es eine große Vielfalt von nicht kultivierbaren Pilzen gibt, die Pflanzengewebe bewohnen. Kulturunabhängige molekulare Identifizierungstechniken bieten somit eine breitere Abdeckung der Artenvielfalt und -häufigkeit. Ziel dieses Beitrags ist es, die methodische Unterstützung zu geben, die notwendig ist, um zwei Untersuchungsverfahren zu starten: ein kulturabhängiges und ein unabhängiges. In Bezug auf das kulturabhängige Protokoll werden die Prozesse der Entnahme und Pflege von Pflanzenproben von den Entnahmestellen bis hin zu Laboreinrichtungen detailliert beschrieben, zusammen mit der Isolierung filamentöser Pilze aus unterirdischen und luftigen Organen mykoheterotropher Pflanzen, der Aufbewahrung einer Sammlung von Isolaten, der morphologischen Charakterisierung von Hyphen durch Objektträgerkulturmethoden und der molekularen Identifizierung von Pilzen durch vollständige DNA-Extraktion. Zu den detaillierten Verfahren, die kulturunabhängige Methoden umfassen, gehören die Entnahme von Pflanzenproben für metagenomische Analysen und die Extraktion von Gesamt-DNA aus achlorophyllösen Pflanzenorganen mit einem kommerziellen Kit. Schließlich werden auch Kontinuitätsprotokolle (z. B. Polymerase-Kettenreaktion [PCR], Sequenzierung) für Analysen vorgeschlagen und Techniken vorgestellt.
Einleitung
Endophytische Pilze sind per Definition solche, die das Innere von Pflanzenorganen und -geweben in unauffälligen Infektionen besiedeln (d. h. ohne ihrem Wirt Schaden zuzufügen)1,2. Diese Pilze können neutral oder vorteilhaft mit Wirtspflanzen interagieren, können Resistenzen gegen Krankheitserreger und ungünstige Umweltbedingungen verleihen und zur Synthese nützlicher Verbindungen für die Pflanze beitragen (z. B. Wachstumsfaktoren und andere Phytohormone)1,3. Mykorrhiza-Endophyten sind Pilze, die Mykorrhiza-Assoziationen mit der Pflanze eingehen und am Nährstofftransfer beteiligtsind 4. Bei Orchidaceae ist die Interaktion mit Mykorrhiza-Endophyten grundlegend für die Samenkeimung bei der überwiegenden Mehrheit der Arten und die Etablierung von Keimlingen bei allen Pflanzen der Familie5. In solchen Kontexten stellen mykoheterotrophe Orchideen einen Fall von völliger Abhängigkeit von ihren Mykorrhizapartnern dar, da sie während ihres gesamten Lebenszyklus auf die Übertragung von Mineralstoffen und Kohlenstoffverbindungen durch diese Pilze angewiesen sind6. Daher ist die Isolierung und Identifizierung assoziierender Pilze eine grundlegende Grundlage für die Untersuchung mykoheterotropher Lebensstrategien. Darüber hinaus ist wenig über die Rolle von Pilzendophyten in mykoheterotrophen Pflanzen oder gar über die tatsächliche Diversität dieser Pilze bekannt 7,8.
Die Untersuchung endophytischer Pilze kann mit verschiedenen Techniken durchgeführt werden, die traditionell als kulturunabhängig oder -abhängig beschrieben werden, zum Beispiel: (a) direkte Beobachtung, (b) Pilzisolierung und morphologische und/oder molekulare Identifizierung und (c) Gesamt-DNA-Extraktion von Pflanzengeweben und molekulare Identifizierung9. In direkter Beobachtung (a) können endophytische Pilze noch im Inneren von Pflanzenzellen und -geweben mittels Licht- oder Elektronenmikroskopie untersucht werden9, da verschiedene Mikroskopieprotokolle von Pena-Passos et al.10 detailliert beschrieben werden. Mit Hilfe von Isolationsmethoden (b) können pilzliche Endophyten nach ihren Kolonien, Hyphen und der Morphologie der Fortpflanzungs- oder Resistenzstruktur charakterisiert werden. Darüber hinaus ist es über Isolationstechniken möglich, die molekulare Identifizierung von Isolaten durch DNA-Extraktion, Amplifikation molekularer Identifizierungssequenzen (Barcodes oder Fingerabdrücke) und Sequenzierung durchzuführen11. Die letztgenannte Technik (c) ermöglicht die molekulare Identifizierung endophytischer Pilze mittels DNA-Extraktion im Inneren von Pflanzengeweben (Metabarcoding), gefolgt von der Bibliotheksvorbereitung und -sequenzierung12.
Darüber hinaus können Pilzisolate in symbiotischen Keimversuchen unter Verwendung von Samen von autotrophen oder mykoheterotrophen Orchideen eingesetzt werden. Ein Beispiel für eine solche Anwendung ist die von Sisti et al.13 durchgeführte Untersuchung, die die Keimung und die Anfangsstadien der Protokormentwicklung bei Pogoniopsis schenckii, einer mykoheterotrophen Orchidee, in Verbindung mit einigen ihrer Isolate, die nicht-mykorrhiza-endophytische Pilze enthalten, beschreibt. Das angewandte symbiotische Keimungsprotokoll wird detailliert beschrieben und in einem Video von Pena-Passos et al.10 vorgestellt. Die Isolierung von Pilzen in Assoziation mit verschiedenen Pflanzenorganen ermöglicht vielfältige Untersuchungsschwerpunkte hinsichtlich der Natur von Pflanzen-Pilz-Interaktionen (z.B. um entweder ökologische oder physiologische Aspekte der Assoziation zu verstehen, sowie Untersuchungen zum Nährstofftransfer von Pilzen auf die Pflanze)9.
Die in Abschnitt 1 vorgestellten Methoden basieren auf einer Sammlung von unterirdischen Organproben, da diese Organe die größten Schwierigkeiten bei der Entnahme aufweisen und von großem Interesse sind, da sie von Mykorrhiza-Endophyten besiedelt werden. Beide enthaltenen Protokolle (Schritte 1.1 und 1.2) können jedoch auf andere mykoheterotrophe Pflanzenorgane (z. B. Rhizome, Blütenstängel und Früchte) angewendet werden. Die in Schritt 1.1 beschriebene Entnahmemethodik ist für die Isolierung endophytischer Pilze (Abschnitt 2) zur morphologischen Charakterisierung (Abschnitte 4 und 5) und/oder die Gesamt-DNA-Extraktion zur Identifizierung von Isolaten (Abschnitt 6) vorgesehen. Andererseits ist die in Schritt 1.2 beschriebene Entnahmemethodik ausschließlich der Gesamt-DNA-Extraktion von Pflanzengeweben für Metabarcoding-Techniken zugeordnet (Abschnitt 7). In Abschnitt 3 werden vier Methoden zur Lagerung und Konservierung von Fadenpilzen vorgestellt, zwei für die kurzfristige Lagerung (3-6 Monate) und die anderen beiden für die langfristige Lagerung (>1 Jahr). Die morphologische Charakterisierung (Abschnitte 4 und 5) kann mit der molekularen Identifizierung verbunden werden, um sie zu untermauern und wichtige Informationen über die Makro- und Mikromorphologie von Pilzen zu liefern. Abbildung 1 fasst die im Folgenden beschriebenen kollektiven Methoden zusammen.

Abbildung 1: Schematische Zusammenfassung der vorgestellten Methoden. Pflanzensammlung und Pilzisolierung, Konservierung und molekulare Identifizierung durch kulturabhängige und -unabhängige Methoden. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Protokoll
1. Entnahme von Pflanzenproben
- Probenentnahme für kulturabhängige Methoden
- Graben Sie vorsichtig die unterirdischen Organe aus; Dabei kann es sich um Wurzeln, Stängel, Rhizome oder Speicherorgane der zu sammelnden Pflanze handeln. Abgesehen von sehr kompakten Böden sollten Sie diese Proben von Hand sammeln.
HINWEIS: Die Verwendung von Werkzeugen wie Kellen oder Schaufeln in diesem Schritt ist nicht ratsam, da sie die empfindlichen Strukturen mykoheterotropher Pflanzen beschädigen und eine Gewebekontamination durch nicht-endophytische Pilze verursachen können. - Sammle so viele unterirdische Organe wie möglich. Bewahren Sie die Proben in Papiertüten in einem gekühlten Behälter auf (z. B. Styroporschaumbox oder Thermobeutel mit Eis). Wenn auch oberirdische Organe entnommen werden, transportieren Sie diese getrennt von den unterirdischen.
- Graben Sie vorsichtig die unterirdischen Organe aus; Dabei kann es sich um Wurzeln, Stängel, Rhizome oder Speicherorgane der zu sammelnden Pflanze handeln. Abgesehen von sehr kompakten Böden sollten Sie diese Proben von Hand sammeln.
- Probenentnahme für kulturunabhängige Methoden
- Graben Sie vorsichtig die Wurzeln der zu sammelnden Pflanze aus, und zwar gemäß den gleichen Empfehlungen, die in der ANMERKUNG aus Schritt 1.1.1 aufgeführt sind.
- Sammle so viele unterirdische Organe wie möglich. Bewahren Sie die gesammelten Proben in Kryoröhrchen in flüssigem Stickstoff auf (wünschenswerte Option) oder verwenden Sie Zentrifugenröhrchen, die von Trockeneis umgeben sind (alternative Option). Halten Sie die oberirdischen Organe getrennt, wenn sie gesammelt werden.
2. Isolierung endophytischer Pilze, die mit Pflanzenorganen assoziiert sind 14
HINWEIS: Alle Materialien, Lösungen und Reagenzien, die in diesem Abschnitt verwendet werden, müssen steril sein. Diejenigen, die nicht bereits sterilisiert gekauft werden können, sollten bei 121 °C für 20 Minuten autoklaviert werden.
- Oberflächliche Entwesung von Pflanzenorganen
- Waschen Sie die entnommenen Proben (Schritt 1.1.2) unter fließendem Wasser und entfernen Sie so viel Substrat und andere Ablagerungen, wie die Proben enthalten könnten.
- Halten Sie die gewaschenen Proben in einer Laminar-Flow-Haube 1 Minute lang in 70%iges Ethanol getaucht (ein Becherglas oder ein Glasgefäß kann verwendet werden).
- Die Proben werden für 3 Minuten in einen anderen Behälter mit Natriumhypochlorit mit 2% Aktivchlor überführt.
- Wechseln Sie die Proben in einen Behälter mit 70 % Ethanol und lassen Sie sie 1 Minute lang untergetaucht. Anschließend werden die Proben nacheinander in zwei Behältern mit destilliertem Wasser gewaschen.
- Einbau von Pflanzenfragmenten in Nährmedium
HINWEIS: Jeder in Schritt 2.2 beschriebene Schritt muss in einer Laminar-Flow-Haube durchgeführt werden.- Vor der Installation Petrischalen (8-10 cm Durchmesser) mit 19,5 g/L Kartoffel-Dextrose-Agar-Nährmedium (PDA) + 7 g/L bakteriologischem Agar + 3 mL/L Antibiotika (z. B. Streptomycin, Penicillin, Tetracyclin, Ampicillin) vorbereiten.
- Bewahren Sie die Petrischalen mit dem Nährmedium 24 Stunden lang bei 36 °C auf, bevor Sie sie verwenden, um sicherzustellen, dass keine Kontamination vorliegt. Entsorgen Sie Geschirr, das mit Bakterien oder Pilzkolonien kontaminiert ist.
HINWEIS: Die Zugabe von Antibiotika in das Medium ist in diesem Schritt unerlässlich, da die Organe stark von Bakterien infiziert sind, die das Pilzwachstum hemmen können. Überprüfen Sie, ob die Antibiotika während des Autoklavierens erhitzt werden können. einige Antibiotika müssen nach dem Abkühlen des Mediums auf ca. 36 °C (vor dem Einfüllen in die Schalen) zugegeben werden. - Unmittelbar nach der oberflächlichen Entwesung der Proben und noch in einer Laminar-Flow-Haube in PDA werden einige Tropfen des destillierten Wassers aus dem letzten Behälter geimpft, der zum Waschen der Proben verwendet wurde. Dieser Schritt ist wichtig, um die Wirksamkeit der oberflächlichen Entwesung von Proben zu bewerten.
- Geben Sie die Proben in eine leere autoklavierte Schale und verwenden Sie ein geflammtes Skalpell und eine Pinzette, um die Proben auf eine Dicke von etwa 0,2 cm zu schneiden.
- Schneiden Sie zylindrische Proben in Längsrichtung in zwei Hälften, wenn gewünscht, um die Oberfläche in Kontakt mit dem Medium zu vergrößern. Um Früchte und andere kugelförmige Organe besser freizulegen, zerkleinern oder schneiden Sie die Strukturen gründlich in Scheiben. Die Samen können auch wichtige Pilzquellen sein, also stellen Sie sicher, dass die geschnittenen Früchte sie dem Medium aussetzen.
HINWEIS: Bei der Isolierung von Pilzendophyten sollten nur gesunde Organe ohne Gewebeschäden oder Anzeichen für mögliche Krankheiten oder pathogene Infektionen verwendet werden. - Verteilen Sie fünf Fragmente der unterirdischen Organe in den Petrischalen mit PDA + Antibiotikum. Achten Sie darauf, dass die Fragmente so weit wie möglich voneinander entfernt sind und auch die Tellerränder nicht berühren. Ordnen Sie keine Fragmente auf dem Medium an. Bereiten Sie Replikate jedes installierten Organs als Vorsichtsmaßnahme für den Fall einer Kontamination vor.
- Verschließen Sie die Petrischalen mit Frischhaltefolie und lagern Sie sie im Dunkeln bei 25-27 °C (am besten im Inkubator) für 5 Tage.
- Berechnung der Isolationsfrequenz (ZF)11
- Nach 5 Tagen Inkubation der Organfragmente wird der IF entsprechend der Anzahl der inkubierten Fragmente, die wachsende Pilzkolonien aufweisen, dividiert durch die Summe der inkubierten Fragmente berechnet, wie in der folgenden Gleichung dargestellt:
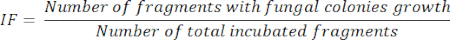
- Nach 5 Tagen Inkubation der Organfragmente wird der IF entsprechend der Anzahl der inkubierten Fragmente, die wachsende Pilzkolonien aufweisen, dividiert durch die Summe der inkubierten Fragmente berechnet, wie in der folgenden Gleichung dargestellt:
- Reinigung von Pilzisolaten durch Streifen und Subkultivieren
HINWEIS: Jeder in Schritt 2.4 beschriebene Schritt muss in einer Laminar-Flow-Haube durchgeführt werden.- Petrischalen (5 cm Durchmesser) mit 5-7 g/L Agar-Agar (AA; nur bakteriologisches Agar) zubereiten. Bewahren Sie das Geschirr 24 Stunden lang bei 36 °C auf, um möglicherweise kontaminierte Gerichte zu entfernen.
- Identifizieren Sie jede gewachsene Pilzkolonie mit einem Code und begrenzen Sie ihre Ränder auf der Unterseite der Schale (kann mit einem Permanentmarker durchgeführt werden). Unterscheiden Sie die Kolonien nach Farbe, Wachstumsmuster, Textur und Randformat.
- Mit einem autoklavierten hölzernen Zahnstocher mit der feinen Spitze eine winzige Menge Myzel aus einer Pilzkolonie gewinnen. Konzentrieren Sie sich vorzugsweise auf die Kolonieränder und wählen Sie ein Gebiet, das so weit wie möglich von einer anderen Kolonie entfernt ist, und vermeiden Sie es, mehr als eine Kolonieart gleichzeitig zu regenerieren.
- Mit demselben Zahnstocher mit Myzel an der Spitze das AA querstreifen, so dass drei Streifen (Rillen) entstehen. Achten Sie darauf, dass jede Striade einen Abstand von 1 cm zu einer anderen und den Tellerrändern hat. Schreiben Sie den entsprechenden Code (mit Aufkleberpapier und Bleistift), verschließen Sie die Schalen und lassen Sie sie 3 Tage lang im Dunkeln bei 25-27 °C inkubieren.
- Petrischalen (8 cm Durchmesser) mit 39 g/L PDA zubereiten. Eine Zugabe von Antibiotika ist in diesem Stadium nicht notwendig.
- Nachdem Sie die AA-Schalen ausgebrütet haben, beobachten Sie sie akribisch gegen Licht (von einer Lampe oder einem Fenster), um feine Hyphen zu identifizieren, die einzelne Kolonien bilden. Begrenzen Sie die Fläche einer einzelnen Kolonie pro Schale mit einem Permanentmarker auf der Unterseite der Petrischale.
- Verwenden Sie in einer Laminar-Flow-Haube einen autoklavierten Zahnstocher, um einen Teil des Mediums zu schneiden, das die Kolonie enthält, und übertragen Sie das geschnittene Volumen in die Mitte einer neuen PDA-Schale.
- Identifizieren Sie die Petrischalen mit den Codes der Isolate, verschließen Sie die Schalen mit Frischhaltefolie und bewahren Sie sie 7-14 Tage lang im Dunkeln bei 25-27 °C auf.
3. Konservierung von gereinigten Pilzisolaten
HINWEIS: Alle Materialien, Lösungen und Reagenzien, die in diesem Abschnitt verwendet werden, müssen steril sein. Diejenigen, die nicht bereits sterilisiert gekauft werden können, sollten bei 121 °C für 20 Minuten autoklaviert werden.
- Konservierung nach Castellani oder in Mineralöl (3-6 Monate)11,15
- Bereiten Sie 2 ml Mikrozentrifugenröhrchen mit 0,5 ml destilliertem Wasser oder 0,5 ml Mineralöl vor (je nach gewählter Methode). Stellen Sie sicher, dass die Röhrchen leer autoklaviert sind und das autoklavierte Wasser und Öl in die Röhrchen in einer Laminar-Flow-Haube gegeben werden.
- Legen Sie die Petrischalen mit gereinigten Isolaten, die bereits in PDA (39 g/L) gezüchtet wurden, für 7-14 Tage in eine Laminar-Flow-Haube. Schneiden Sie mit einem autoklavierten Zahnstocher kleine Quader (0,5 cm x 0,5 cm im oberen Bereich) des Mediums mit den Myzelrändern ab.
- Vier bis sechs Quader in die Mikrozentrifugenröhrchen mit destilliertem Wasser (Castellani-Methode) oder Mineralöl geben. Lagern Sie die Röhrchen so lange wie nötig im Dunkeln bei 25 °C und beachten Sie dabei die zeitlichen Einschränkungen der Methode.
HINWEIS: Vermeiden Sie es, zu viele Quader in die Röhrchen zu geben und sie voll zu machen, da dies die Wahrscheinlichkeit einer Kontamination erhöhen kann. Es ist möglich, die Röhrchen gekühlt zu halten, was die längere Konservierung einiger Isolate begünstigen kann. Currah et al.16 empfehlen, Replikate von Mykorrhizapilz-Isolaten aus tropischen Orchideen sowohl gekühlt als auch bei 25 °C zu lagern, da sie im Kühlhaus verloren gehen können. - Entnehmen Sie einen Quader und stellen Sie ihn in die Mitte einer neuen PDA-Schale, um ein gelagertes Isolat zu züchten.
- Kryokonservierung in ungeschälten Reiskörnern (>1 Jahr)17
- Waschen Sie ungeschälte Reiskörner unter fließendem Wasser und kochen Sie sie, bis sich die Reisschale zu öffnen beginnt. Verteilen Sie die gekochten Körner in Glasreagenzgläsern mit Schraubverschluss und autoklavieren Sie zweimal im Abstand von 24 Stunden.
- Legen Sie die Petrischalen mit gereinigten Isolaten, die bereits in PDA (39 g/L) gezüchtet wurden, für 7-14 Tage in eine Laminar-Flow-Haube. Mit einem autoklavierten Zahnstocher werden fünf kleine Hyphenfragmente aus den Myzelrändern entnommen und in das Röhrchen mit den autoklavierten Reiskörnern geimpft.
HINWEIS: Die Inokulation von Hyphen an verschiedenen Stellen und Tiefen des Röhrchens garantiert, dass das Isolat die ungeschälten Reiskörner in kürzerer Zeit besiedelt. - Inkubieren Sie die Röhrchen 14 Tage lang im Dunkeln bei 25-27 °C. Rühren Sie die Röhrchen, indem Sie sie alle 3 Tage wirbeln, um die Körner individualisiert zu halten.
- Nachdem Sie das Pilzwachstum in den Reiskörnern beobachtet haben, verteilen Sie die Körner in einer autoklavierten Petrischale, die mit Filterpapier ausgekleidet ist, um Feuchtigkeit zu absorbieren, auf dem Papier, damit die Körner trocknen können. Im Dunkeln bei 25-27 °C 2-3 Tage lagern.
- Kryoröhrchen mit 1/3 Kieselgel an der Unterseite und 1/3 Glaswolle über der Kieselsäure vorbereiten. Zum Schluss 1/3 der Reiskörner mit gewachsenem Pilzmyzel verteilen. Bei -20 °C 24 h lagern.
- Lagern Sie die Kryoröhrchen nach 24 h so lange wie nötig bei -80 °C und beachten Sie dabei die zeitlichen Einschränkungen der Methode.
- Kryokonservierung in Vermiculit unter Verwendung eines Kryoprotektivums (>1 Jahr)18
- Bereiten Sie ein flüssiges Nährmedium aus 0,2 % Hefeextrakt und 2 % Glukose in destilliertem Wasser vor. Stellen Sie den pH-Wert auf 5 ein und autoklavieren Sie es.
- 0,2 g Vermiculit (feine Körnung verwenden) in Kryoröhrchen mit Schraubverschluss verteilen und autoklavieren. Geben Sie 0,8 ml autoklaviertes flüssiges Medium zu den Kryoröhrchen, die Vermiculit enthalten.
- Legen Sie die Petrischalen mit gereinigten Isolaten, die bereits in PDA (39 g/L) gezüchtet wurden, für 7-14 Tage in eine Laminar-Flow-Haube. Entnehmen Sie mit einem autoklavierten Zahnstocher drei bis fünf Hyphenfragmente von den Myzelrändern und impfen Sie entlang des Röhrchens mit Vermiculit + flüssigem Kulturmedium. Denken Sie daran, die Kryoröhrchen mit dem entsprechenden Isolatcode zu identifizieren.
HINWEIS: Die Inokulation von Hyphen an verschiedenen Stellen und in verschiedenen Tiefen des Kryoröhrchens garantiert, dass das Isolat den Vermiculit in kürzerer Zeit besiedelt. - Lagern Sie die Kryoröhrchen im Dunkeln bei 25-27 °C, bis eine Besiedlung der meisten Vermiculitkörner beobachtet werden kann, was in der Regel etwa 14 Tage dauert.
- Stellen Sie eine Kryoprotektivlösung aus 5 % Glycerin und 5 % Trehalose in destilliertem Wasser her und autoklavieren Sie sie. Lassen Sie die Lösung abkühlen, bevor Sie sie verwenden.
- Nachdem das Vermiculit in Kryoröhrchen mit dem jeweiligen Pilzisolat besiedelt wurde, verteilen Sie 0,4 ml Kryoprotektivum in jedem Röhrchen und bewahren Sie die Kryoröhrchen 48 Stunden lang bei 4 °C im Kühlschrank auf. Halten Sie die Kryoröhrchen anschließend so lange wie nötig bei -80 °C und beachten Sie dabei die zeitlichen Einschränkungen der Methode.
4. Makromorphologische Charakterisierung filamentöser Pilze (Koloniemorphologie)
- Führen Sie ein fotografisches Register des in jeder Petrischale gezüchteten Myzels mit 39 g/L PDA für 7-14 Tage. Denken Sie daran, beide Seiten der Kolonie, Ober- und Unterseite (umgekehrt), zu registrieren. Wenn das Geschirr in Abschnitt 6 verwendet oder nicht gewartet werden soll, öffnen Sie das Geschirr beim Fotografieren, um bessere Fotos zu erhalten.
- Wenn Sie an quantitativen Daten über die Koloniemorphologie interessiert sind, replizieren Sie die Isolate und halten sie unter den gleichen Wachstumsbedingungen und für einen konstanten Zeitraum, um den Koloniedurchmesser zu registrieren und die Wachstumsrate (normalerweise in mm/h) zu berechnen.
ANMERKUNG: Anspruchsvollere quantitative Ergebnisse können erzielt werden, indem verschiedene Arten von Nährmedien für den Vergleichverwendet werden 19 und die Statistikwerkzeuge zur Behandlung der Daten berücksichtigt werden. - Beobachten Sie die Kolonien auch unter einem Stereomikroskop, um morphologische Merkmale zu identifizieren, und fotografieren Sie sie in Vergrößerung. Bewerten Sie die Kolonien nach ihren makromorphologischen Merkmalen, wie im Abschnitt "Ergebnisse" beschrieben. Konsultieren Sie verschiedene bibliographische Quellen, um die Makromorphologie zu kategorisieren und mit der molekularen und/oder morphologischen Identifizierung in Beziehung zu setzen.
5. Mikromorphologische Charakterisierung filamentöser Pilze (Hyphenmorphologie)
ANMERKUNG: Die mikromorphologischen Techniken werden im Diskussionsteil unter Berücksichtigung ihrer möglichen Verwendungen und Nachteile verglichen.
- Züchten Sie die Isolate auf Petrischalen mit 39 g/L PDA für 7-14 Tage. Zur Beurteilung von mutmaßlichen Orchideen-Mykorrhizapilzen werden die Isolate 3-7 Tage lang auf 17 g/L Maismehl-Agar (CMA; oder einem anderen nährstoffbegrenzenden Medium) gezüchtet19.
- Teaser-Reittier
- Arbeiten Sie in einer Laminar-Flow-Haube. Geben Sie einen Tropfen eines ausgewählten Flecks (Schritt 5.5) auf einen sauberen Objektträger.
- Entferne mit einem autoklavierten Zahnstocher oder einem anderen sterilen Material vorsichtig einige Hyphen aus dem gewachsenen Isolat und lege sie in den Fleckentropfen. Legen Sie ein Deckglas auf (vorzugsweise in einem Anfangswinkel von 45°, um Luftblasen zu vermeiden) und analysieren Sie es unter einem Lichtmikroskop.
- Klebeband-Halterung
HINWEIS: Diese Technik wird in der Regel in Pilzkulturen angewendet, die leicht in Abschnitt 6 verwendet oder nicht gepflegt werden können, da das Klebeband nicht autoklaviert werden kann und nach der Durchführung der Methode Kontaminationen auftreten können.- Geben Sie einen Tropfen eines ausgewählten Flecks (Schritt 5.5) auf einen sauberen Objektträger. Schneiden Sie einen Streifen transparentes Klebeband in einer Größe zu, die gut auf den Objektträger und den zentralen Fleckentropfen passt.
- Richten Sie die klebrige Oberfläche des Streifens auf die Myzeloberfläche. Drücken Sie nicht und versuchen Sie, einige Hyphen zu sammeln, ohne zu viele davon zu kleben.
- Kleben Sie das Klebeband auf den Objektträger und stellen Sie sicher, dass der Fleck mit den gesammelten Hyphen in Kontakt kommt. Geben Sie einen Tropfen Wasser über das Klebeband und legen Sie ein Deckglas auf. Analysieren Sie den Objektträger unter dem Lichtmikroskop.
- Objektträgerkultur von Fadenpilzen20
- In eine große Petrischale (vorzugsweise >9 cm Durchmesser) legen: Filterpapier am unteren Teil, einen U-förmigen Glasstab oder eine Adaption (das Ziel ist es, eine Erhöhung für den Objektträger zu schaffen), einen Objektträger und zwei Deckgläser. Höhere Petrischalen erleichtern die Manipulation. Autoklavieren Sie diese Kits mit Petrischalen, eine für jedes Isolat.
- Bereiten Sie ein kleines Volumen von 39 g/l PDA oder 17 g/l CMA vor, fügen Sie 10 g/l bakteriologischen Agar hinzu und autoklavieren Sie (das Volumen für eine Petrischale reicht für mehr als 30 Isolate). Die Nährmedien, die in der Objektträgerkultur verwendet werden, müssen härter sein als die üblichen Medien. 100 ml destilliertes Wasser autoklavieren.
- Arbeiten Sie in einer Laminar-Flow-Haube. Geben Sie das flüssige Medium in eine Petrischale und bilden Sie eine etwa 0,5 cm hohe Schicht. Lassen Sie das Medium erstarren. Sobald es fest ist, schneiden Sie mit einem sterilen Skalpell Quadrate des Mediums aus, die 1 cm x 1 cm groß sind.
- Öffnen Sie das Petrischalen-Kit in einer Laminar-Flow-Haube und verwenden Sie eine geflammte Pinzette, um das in der Schale autoklavierte Material zu organisieren. Ordnen Sie das Material in der nummerierten Reihenfolge an, wie in Abbildung 2A zu sehen, und legen Sie ein Quadrat des Kulturmediums auf den Objektträger. Bevor Sie das Deckglas über das Medium legen, fahren Sie mit Schritt 5.4.5 fort.
- Verwenden Sie einen autoklavierten Zahnstocher, um einige Hyphen aus einem Isolat zu gewinnen, und reiben Sie vorsichtig die vier seitlichen Flächen des Mediums, das auf dem Objektträger platziert ist. Legen Sie ein autoklaviertes Deckglas mit einer geflammten Pinzette auf das mittlere Quadrat.
- Verwenden Sie eine sterile Pipettenspitze, um autoklaviertes Wasser in das Filterpapier zu geben, um eine feuchte Kammer zu schaffen. Verwenden Sie ein Volumen, um das Papier zu sättigen, ohne überschüssiges Wasser einzufüllen. Verschließen Sie die Petrischale mit Frischhaltefolie und kennzeichnen Sie sie mit dem jeweiligen Isolatcode. Bewahren Sie das Geschirr im Dunkeln bei 25-27 °C für 3-7 Tage auf.
- Beurteilen Sie das Wachstum der Hyphen im Objektträger und im Deckglas. Aus jedem Objektträger-Kultur-Kit werden zwei Objektträger hergestellt, einer mit dem Glasobjektträger mit Hyphen und dem autoklavierten Deckglas, der zweite mit dem Deckglas mit Hyphen und ein weiterer Glasobjektträger.
- Nachdem das Hyphenwachstum beobachtet wurde, lösen Sie vorsichtig das Quadrat des Mediums vom Objektträger (Abbildung 2B), montieren Sie die beiden Objektträger mit einer ausgewählten Färbung (Schritt 5.5) und analysieren Sie sie unter einem Lichtmikroskop. Denken Sie daran, den Isolationscode auf der Folie zu identifizieren. Um semi-permanente Objektträger herzustellen, versiegeln Sie das Deckglas mit klarem Nagellack.
- Um permanente Objektträger herzustellen, waschen Sie den Farbstoff nach dem Färben vorsichtig von anhaftenden Hyphen und lassen Sie den Objektträger trocknen. Montage mit einem Schnelleinbaumedium (Details siehe Pena-Passos et al.10).
HINWEIS: Der Nachteil bei der Herstellung eines permanenten Objektträgers mit dieser Technik besteht darin, dass Sporen und Strukturen, die nicht am Glas haften, gewaschen werden können.
- Färbemethoden und Hyphenbeobachtung
- Lactophenol cotton blue (LPCB)21: 20 ml Milchsäure, 40 ml Glycerin und 20 ml destilliertes Wasser hinzufügen. Lösen Sie 20 g Phenolkristalle in dieser Lösung durch vorsichtiges Erhitzen auf. 0,05 g Methylblau (Baumwollblau oder 2 ml 1%ige wässrige Lösung) werden gelöst. LPCB kann auch einfach bereits vorbereitet erworben werden.
VORSICHT: Phenol ist hochgiftig und flüchtig; Manipulieren Sie es ausschließlich in einem Abzug und tragen Sie Handschuhe. - Toluidinblau O10 (TBO): 0,05 % TBO in 0,1 M Phosphatpuffer (pH 6,8) herstellen.
- Kongorot22,23,24: Eine Lösung von 1%igem Kongorot in destilliertem Wasser zubereiten und filtrieren. 5-10 Minuten inkubieren. Dieser Farbstoff kann auch für die Fluoreszenzmikroskopie25 verwendet werden.
- Wenn Sie Pilzhyphen unter dem Lichtmikroskop beobachten und fotografieren, konsultieren Sie verschiedene bibliographische Quellen, um Strukturen zu identifizieren (siehe Diskussion).
- Lactophenol cotton blue (LPCB)21: 20 ml Milchsäure, 40 ml Glycerin und 20 ml destilliertes Wasser hinzufügen. Lösen Sie 20 g Phenolkristalle in dieser Lösung durch vorsichtiges Erhitzen auf. 0,05 g Methylblau (Baumwollblau oder 2 ml 1%ige wässrige Lösung) werden gelöst. LPCB kann auch einfach bereits vorbereitet erworben werden.

Abbildung 2: Verfahren zur Objektträgerkultur von Fadenpilzen . (A) Schematische Konfiguration eines Objektträger-Kulturkits, wobei die Zahlen die Reihenfolge der Anordnung der Elemente angeben. (B) Das Ablösen des Quadrats des Nährmediums nach dem Hyphenwachstum wird in dem Objektträger und dem Deckglas beobachtet. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
6. Gesamt-DNA-Extraktion aus Pilzisolaten (hausgemachtes Protokoll26 mit Modifikationen 27)
HINWEIS: Alle Materialien, Lösungen und Reagenzien, die in diesem Abschnitt verwendet werden, müssen steril sein. Diejenigen, die nicht bereits sterilisiert gekauft werden können, sollten bei 121 °C für 20 Minuten autoklaviert werden. Tragen Sie während des gesamten Protokolls Handschuhe und führen Sie einige Schritte in einem Abzug durch.
- Bereiten Sie den Extraktionspuffer vor: 1 % Natriumdodecylsulfat (SDS), 250 mM NaCl, 200 mM Tris-HCl (pH 8,0) und 25 mM Ethylendiamintetraessigsäure (EDTA). Züchten Sie die gereinigten Isolate in 39 g/L PDA für 7-14 Tage.
- Kratzen Sie das Myzel vorsichtig mit einem Spatel oder einem Löffel von einem Isolat ab und geben Sie die Fragmente in einen autoklavierten Mörser, wobei Sie vermeiden sollten, das Nährmedium mit dem Hyphenaggregat zu übertragen. Zermahlen Sie das Myzel mit einem Porzellanstößel mit flüssigem Stickstoff zu einem feinen Pulver. Lassen Sie das gemahlene Myzel nicht schmelzen und fügen Sie Stickstoff hinzu, um dies zu vermeiden.
- Geben Sie 1 ml Extraktionspuffer in ein 2-ml-Mikrozentrifugenröhrchen und legen Sie die gemahlene Probe bis zur 1,5-ml-Markierung. Rühren Sie den Inhalt der Röhrchen vorsichtig um, um ihn zu homogenisieren.
- Die Röhrchen werden 5 s in einem Wirbel gerührt und 20 min lang bei 65 °C in einen Thermoblock gelegt. Homogenisieren Sie den Inhalt sorgfältig durch Umkehrung alle 7-10 Minuten.
- Die Röhrchen werden bei 10.000 x g für 10 min bei 4 °C zentrifugiert. 800 μl aus der oberen Phase in ein neues 2-ml-Mikrozentrifugenröhrchen überführen, 800 μl Phenol in die Röhrchen geben und den Inhalt durch Inversion mischen.
VORSICHT: Phenol ist ein hochgiftiges und flüchtiges Reagenz; Es kann Erythem, Gangrän und Gewebenekrose verursachen. Beim Einatmen kann es Dyspnoe und Husten verursachen. Die systemische Resorption kann Leber, Nieren und das zentrale Nervensystem schädigen. Behandeln Sie Phenol ausschließlich in einem Abzug und tragen Sie Handschuhe.
HINWEIS: Tragen Sie ab dem nächsten Schritt Handschuhe und führen Sie das Protokoll in einem Abzug durch. - Die Röhrchen werden bei 10.000 x g für 10 min bei 4 °C zentrifugiert und 800 μl aus der oberen Phase in ein neues 2-ml-Röhrchen überführt, wobei die Übertragung des unteren Phaseninhalts sorgfältig vermieden wird.
- Geben Sie 400 μl Phenol und 400 μl Chloroform in die Röhrchen und mischen Sie den Inhalt durch Inversion. Zentrifugieren Sie sie bei 10.000 x g für 10 min bei 4 °C.
VORSICHT: Chloroform ist ein hochgiftiges und flüchtiges Reagenz; Es kann bei Hautkontakt zu Reizungen und Verletzungen führen und beim Einatmen das zentrale Nerven- und Herz-Kreislauf-System, die Leber und die Nieren beeinträchtigen. Behandeln Sie Chloroform ausschließlich in einem Abzug und tragen Sie Handschuhe. - Rückgewinnung von 800 μl oder weniger aus der oberen Phase in ein neues 2-ml-Röhrchen. Man fügt 800 μl Chloroform hinzu, mischt den Inhalt durch Inversion und zentrifugiert bei 10.000 x g für 10 min bei 4 °C.
HINWEIS: In diesem Schritt dürfen keine Rückstände aus der unteren Phase in neue Röhrchen überführt werden. Seien Sie daher besonders vorsichtig, wenn Sie sich von der oberen Phase erholen. - 600-800 μl von der oberen Phase in ein neues 1,5-ml-Röhrchen überführen und 450 μl Isopropanol hinzufügen. Den Inhalt durch Invertierung mischen und bei 25 °C 5 min inkubieren.
- Zentrifugieren Sie die Röhrchen bei 10.000 x g für 5 min bei 4 °C und entsorgen Sie den Überstand mit einer Mikropipette. Achten Sie darauf, das am Boden des Röhrchens abgelagerte Pellet nicht zu entsorgen.
- Fügen Sie 500 μl 80%iges Ethanol hinzu und zentrifugieren Sie es 5 Minuten lang. Wiederholen Sie den Vorgang zweimal, falls das Pellet nicht geklärt ist.
- Entnehmen Sie das Ethanol mit einer Mikropipette und trocknen Sie das Pellet bei 37 °C für 30-60 min. Fügen Sie 30-50 μl deionisiertes Wasser hinzu und eluieren Sie das Pellet mit einer Mikropipette.
- Halten Sie die Röhrchen über Nacht bei 4 °C, um eine vollständige DNA-Elution zu erreichen, und frieren Sie den Inhalt bei -20 °C ein.
7. Totale DNA-Extraktion aus pflanzlichen Organen für die Metabarcoding-Methodik (kommerzielles Kit)
HINWEIS: Für die folgende Methodik ist es notwendig, das in der Materialtabelle angegebene kommerzielle Kit als Boden-DNA-Extraktionskit zu erwerben. Alle Materialien, Lösungen und Reagenzien, die in diesem Abschnitt verwendet werden, müssen steril sein. Diejenigen, die nicht bereits sterilisiert gekauft werden können, sollten bei 121 °C für 20 Minuten autoklaviert werden. Es wird dringend empfohlen, während des gesamten Protokolls Handschuhe zu tragen, und die Schritte können in einer Laminar-Flow-Haube durchgeführt werden. Das beschriebene Protokoll ist eine Modifikation von De Souza et al.12, ausgehend von dem vom Hersteller detaillierten Protokoll.
- Mit Porzellanstößeln und Mörsern werden die nach Schritt 1.2 gesammelten Wurzeln in flüssigem Stickstoff zerkleinert und die Proben zu einem feinen Pulver zerkleinert. Geben Sie 0,3 g aus den gemahlenen Proben in die PowerBead-Röhrchen und rühren Sie sie vorsichtig um, um sie zu homogenisieren.
- Geben Sie 60 μl Lösung C1 (im Kit enthalten) in die PowerBead-Röhrchen und mischen Sie den Inhalt durch Umkehrung. Binden Sie die Röhrchen mit einem Gewebehomogenisator und einem Zelllyser (Materialtabelle) fest an eine geeignete Stütze, koppeln Sie die Stütze an den Wirbel und starten Sie das Gerät mit maximaler Geschwindigkeit für 10-20 Minuten.
HINWEIS: Falls die C1-Lösung ausgefällt wird, erhitzen Sie sie bei 60 °C, bis sie sich vollständig aufgelöst hat. - Die Röhrchen werden bei 10.000 x g für 30 s bei 25 °C zentrifugiert. 500 μl des Überstandes in 2 mL Mikrozentrifugenröhrchen überführen. Der übertragene Inhalt kann partikulär sein.
- Geben Sie 250 μl Lösung C2 in die Röhrchen und rühren Sie 5 s lang in einem Wirbel. 5 min bei 4 °C inkubieren und dann bei 10.000 x g für 1-2 min bei 25 °C zentrifugieren.
- Füllen Sie mehr als 600 μl des Überstandes in neue 2-ml-Röhrchen um und vermeiden Sie das Pellet. 200 μl Lösung C3 zugeben und 5 s in einem Wirbel rühren.
- 5 min bei 4 °C inkubieren und dann bei 10.000 x g für 1 min bei 25 °C zentrifugieren. Stellen Sie in dieser Phase sicher, dass der Überstand nicht aus Partikeln besteht.
- Übertragen Sie mehr als 750 μl des Überstandes in neue 2-ml-Röhrchen und vermeiden Sie das Pellet. Die Lösung C4 wird gut homogenisiert, 1.100 μl Lösung C4 in den Überstand gegeben und 5 s in einem Wirbel gerührt.
- Laden Sie 675 μl aus dem Inhalt der Röhrchen in den MB-Spin-Säulen über den Filter und zentrifugieren Sie 1 Minute lang bei 10.000 x g bei 25 °C. Entsorgen Sie den flüssigen Inhalt.
- Wiederholen Sie den vorherigen Schritt zweimal, bis der gesamte Inhalt jedes Röhrchens verarbeitet ist. Dann werden 500 μl Lösung C5 in die Mitte des Filters gegeben, der sich an der oberen Säule des Röhrchens befindet, und bei 10.000 x g für 30 s bei 25 °C zentrifugiert.
- Entsorgen Sie den flüssigen Inhalt und zentrifugieren Sie erneut unter den gleichen Bedingungen wie im vorherigen Schritt. Übertragen Sie die überlegene Säule der MB Spin Column vorsichtig in neue 2-ml-Mikrozentrifugenröhrchen und vermeiden Sie, dass flüssiger Inhalt in die Säule tropft.
- 85-100 μl Lösung C6 in die Mitte des Filters geben und 1 Minute warten. Bei 10.000 x g 30 s bei 25 °C zentrifugieren und die MB-Spin-Säule entsorgen. Lagern Sie die Röhrchen bei -80 °C.
8. DNA-Quantifizierung in einem Spektralphotometer (siehe Materialtabelle)
- Öffnen Sie die Software mit dem angegebenen Spektralphotometer. Wählen Sie die Option Nukleinsäure, wählen Sie die Option DNA und stellen Sie die Konzentration auf ng/μL ein.
- Geben Sie 1 μl deionisiertes Wasser in den Spektralphotometer-Detektor und kalibrieren Sie ihn, indem Sie die Option Blank auswählen. Wischen Sie den Detektor nach dem Ablesen vorsichtig mit weichem Seidenpapier ab.
- Benennen Sie die Feldprobe mit dem Isolatcode der zu lesenden Probe und geben Sie 1 μl davon in den Gerätedetektor. Wählen Sie die Option Messen; Es wird ein Diagramm und eine Tabelle mit den Ergebnissen generiert.
- Wiederholen Sie Schritt 8.3, bis alle Proben gelesen wurden. Es wird empfohlen, die Tabelle mit den generierten Ergebnissen für die Aufzeichnung und Analyse zu speichern: Wählen Sie die Option Berichte und den Speicherort, an dem die .xml Datei gespeichert werden soll.
- Geben Sie 5 μl deionisiertes Wasser in den Detektor, warten Sie einige Minuten und wischen Sie ihn vorsichtig mit weichem Seidenpapier ab.
Ergebnisse
In Anbetracht der Tatsache, dass im Isolationsprotokoll eine Kontamination durch das bei der letzten Wäsche verwendete Wasser vorliegt und die Kontamination auch in den Petrischalen mit inokulierten Fragmenten festgestellt wird, können je nach Art der Verunreinigung unterschiedliche Maßnahmen ergriffen werden (Tabelle 1). Dieser Vorgang muss von Anfang an wiederholt werden, wenn es sich um stark sporulierende Pilzverunreinigungen handelt, die auch ein beschleunigtes Wa...
Diskussion
Die oberflächliche Entwesung von Pflanzenproben ist eine der kritischsten Phasen des vorgestellten Protokolls. Keine Kontamination des PDA-Geschirrs mit Tropfen aus dem letzten Waschgang ist sehr wünschenswert. Bakterien werden häufig als Verunreinigungen in den Isolierschalen beobachtet, in der Regel mehr als luftgetragene sporulierende Pilze, wenn man bedenkt, dass endophytische Bakterien auch in Pflanzengeweben häufig vorkommen 3,11. Daher ist die Zugabe v...
Offenlegungen
Die Autoren haben nichts offenzulegen und keinen Interessenkonflikt.
Danksagungen
Wir danken für die Finanzierung durch FAPESP (2015/26479-6) und CNPq (447453/2014-9). JLSM bedankt sich bei CNPq für Produktivitätszuschüsse (303664/2020-7). MPP bedankt sich bei Capes (Master-Stipendium, Prozess 88887.600591/2021-00) und CNPq.
Materialien
| Name | Company | Catalog Number | Comments |
| Adhesive tape | (from any company, for adhesive tape mount in micromorphological analyses) | ||
| Ampicillin | Sigma-Aldrich | A5354 | (for installation of plant fragments; other antibiotics may be used - check step 2.2.1) |
| Autoclave | (from any company, for materials sterilization in many steps) | ||
| Bacteriological agar | Sigma-Aldrich | A1296 | (for many steps) |
| C1, C2, C3, C4, C5, and C6 solutions | Qiagen | 12888-50 | (purchased with DNeasy PowerSoil kit) |
| Centrifuge | Merck/Eppendorf | 5810 G | (for total DNA extraction from fungal isolates) |
| Centrifuge tubes | Merck | CLS430828 | (for samples collection) |
| Chloroform | Sigma-Aldrich | C2432 | (for total DNA extraction from fungal isolates) |
| Congo red | Supelco | 75768 | (for hyphae staining) |
| Cryotubes | Merck | BR114831 | (for many steps) |
| Ethanol | Supelco | 100983 | It will be necessary to carry out the appropriate dilutions (for many steps) |
| Ethylenediaminetetraacetic acid (EDTA) | Sigma-Aldrich | 3609 | (for total DNA extraction from fungal isolates) |
| Filter paper | Merck | WHA10010155 | (for many steps) |
| Glass test tubes | Merck | CLS7082516 | (for cryopreservation in unhulled rice grains) |
| Glass wool | Supelco | 20411 | (for cryopreservation in unhulled rice grains) |
| Glucose | Sigma-Aldrich | G8270 | Or dextrose (for cryopreservation in vermiculite) |
| Glycerol | Sigma-Aldrich | G5516 | Or glycerin (for cryopreservation in vermiculite, for preparing LPCB) |
| Isopropanol | Sigma-Aldrich | 563935 | (for total DNA extraction from fungal isolates) |
| Lactic acid | Sigma-Aldrich | 252476 | (for preparing LPCB - hyphae staining) |
| Lactophenol blue solution (LPCB) | Sigma-Aldrich | 61335 | (for hyphae staining) |
| Laminar flow hood | (class I, from any company, for many steps) | ||
| Light microscope | (from any company, for hyphae observation) | ||
| MB Spin Columns | Qiagen | 12888-50 | (purchased with DNeasy PowerSoil kit) |
| Methyl blue (cotton blue) | Sigma-Aldrich | M5528 | (for preparing LPCB - hyphae staining) |
| Microcentrifuge tube (1.5 mL) | Merck | HS4323 | (for total DNA extraction from fungal isolates) |
| Microcentrifuge tube (2 mL) | Merck | BR780546 | (for many steps) |
| Mineral oil | (for preservation of fungal isolates) | ||
| Paper bags | Average size 150 mm x 200 mm (for samples collection) | ||
| Petri dish (Glass, 120 mm x 20 mm) | Merck/Pyrex | SLW1480/10D | (autoclavable, for fungi slide culture, prefer higher ones) |
| Petri dish (Glass, 50 mm x 17 mm) | Merck/Aldrich | Z740618 | (for purification of fungal isolates); alternatively: polystyrene petri dishes (sterile, γ-irradiated, non-autoclavable) |
| Petri dish (Glass, 80 mm x 15 mm) | Merck/Brand | BR455732 | (for installation of plant fragments); alternatively: polystyrene petri dishes (sterile, γ-irradiated, non-autoclavable) |
| Phenol | Sigma-Aldrich | P1037 | (for total DNA extraction from fungal isolates, for preparing LPCB) |
| Porcelain mortar | Sigma-Aldrich | Z247464 | (for total DNA extraction from fungal isolates) |
| Porcelain pestle | Sigma-Aldrich | Z247502 | (for total DNA extraction from fungal isolates) |
| Potato dextrose agar (PDA) | Millipore | P2182 | (for many steps) |
| PowerBead tubes | Qiagen | 12888-50 | (purchased with DNeasy PowerSoil kit) |
| Rapid mounting medium (Entellan) | Sigma-Aldrich | 1.0796 | (for fungi slide culture) |
| Silica gel | Supelco | 717185 | (for cryopreservation in unhulled rice grains) |
| Sodium chloride (NaCl) | Sigma-Aldrich | S9888 | (for total DNA extraction from fungal isolates) |
| Sodium dodecyl sulfate (SDS) | Sigma-Aldrich | L3771 | Lauryl sulfate sodium salt (for total DNA extraction from fungal isolates) |
| Sodium hypochlorite (w/ 2% active chlorine) | (commercial product, for superficial desinfestation) | ||
| Soil DNA extraction kit (DNeasy PowerSoil kit) | Qiagen | 12888-50 | (for total DNA extraction from plant organs) |
| Spectrophotometer - Nanodrop 2000/2000c | ThermoFisher Scientific | ND2000CLAPTOP | (for total DNA extraction from plant organs) |
| Stereomicroscope | (=dissecting microscope, from any company, for macromorphological analyses) | ||
| Tetracycline | Sigma-Aldrich | T7660 | (for installation of plant fragments) |
| Thermoblock | Merck/Eppendorf | EP5362000035 | (or from other companies) |
| Tissue homogenizer and cell lyzer | SPEX SamplePrep | 2010 Geno/Grinder - Automated Tissue Homogenizer and Cell Lyzer (for total DNA extraction from plant organs) | |
| Toluidine blue O | Sigma-Aldrich/Harleco | 364-M | (for hyphae staining) |
| Trehalose | Sigma-Aldrich | T9531 | (for cryopreservation in vermiculite) |
| Tris Base Solution (Tris) | Sigma-Aldrich | T1699 | (for total DNA extraction from fungal isolates) |
| Unhulled rice grains | (for cryopreservation) | ||
| U-shaped glass rod | (or an adaptation - check step 5.4.1, for fungi slide culture) | ||
| Vermiculite | Fine granulometry (for cryopreservation in vermiculite) | ||
| Vortexer | Sigma-Aldrich/BenchMixer | BMSBV1000 | (for total DNA extraction from fungal isolates) |
| Yeast extract | Sigma-Aldrich | Y1625 | (for cryopreservation in vermiculite) |
Referenzen
- de Azevedo, J. L. Endophytic microorganisms. Ecologia Microbiana. , 117-137 (1998).
- Stone, J. K., Bacon, C. W., White, J. F. An overview of endophytic microbes: endophytism defined. Microbial Endophytes. , 17-44 (2000).
- Schulz, B., Boyle, C. What are Endophytes. Microbial Root Endophytes. , 1-13 (2006).
- Smith, S. E., Read, D. J. . Mycorrhizal Symbiosis. , (2008).
- Rasmussen, H. N., Dixon, K. W., Jersáková, J., Těšitelová, T. Germination and seedling establishment in orchids: a complex of requirements. Annals of Botany. 116 (3), 391-402 (2015).
- Rasmussen, H. N., Rasmussen, F. N. Orchid mycorrhiza: implications of a mycophagous life style. Oikos. 118 (3), 334-345 (2009).
- Ma, X., Kang, J., Nontachaiyapoom, S., Wen, T., Hyde, K. D. Non-mycorrhizal endophytic fungi from orchids. Current Science. 109 (1), 72-87 (2015).
- Favre-Godal, Q., Gourguillon, L., Lordel-Madeleine, S., Gindro, K., Choisy, P. Orchids and their mycorrhizal fungi: an insufficiently explored relationship. Mycorrhiza. 30 (1), 5-22 (2020).
- Sun, X., Guo, L. -. D. Endophytic fungal diversity: review of traditional and molecular techniques. Mycology. 3 (1), 65-76 (2012).
- Pena-Passos, M., Sisti, L. S., Mayer, J. L. S. Microscopy techniques for interpreting fungal colonization in mycoheterotrophic plants tissues and symbiotic germination of seeds. Journal of Visualized Experiments. (183), e63777 (2022).
- Araújo, W. L., et al. . Endophytic microorganisms: Theoretical and Practical Aspects of Isolation and Characterization. 1st ed. 1, 257 (2014).
- de Souza, R. S. C., et al. Unlocking the bacterial and fungal communities assemblages of sugarcane microbiome. Scientific Reports. 6, 28774 (2016).
- Sisti, L. S., et al. The role of non-mycorrhizal fungi in germination of the mycoheterotrophic orchid Pogoniopsis schenckii Cogn. Frontiers in Plant Science. 10, 1589 (2019).
- Araújo, W. L., et al. Variability and interactions between endophytic bacteria and fungi isolated from leaf tissues of citrus rootstocks. Canadian Journal of Microbiology. 47 (3), 229-236 (2001).
- Castellani, A. Further researches on the long viability and growth of many pathogenic fungi and some bacteria in sterile distilled water. Mycopathologia. 20 (1-2), 1-6 (1963).
- Currah, R. S., Zelmer, C. D., Hambleton, S., Richardson, K. A. Fungi from orchid mycorrhizas. Orchid Biology: Reviews and Perspectives, VII. , 117-170 (1997).
- Freitas, E. F. S., et al. Diversity of mycorrhizal Tulasnella associated with epiphytic and rupicolous orchids from the Brazilian Atlantic Forest, including four new species. Scientific Reports. 10 (1), 7069 (2020).
- Sato, M., Inaba, S., Noguchi, M., Nakagiri, A. Vermiculite as a culture substrate greatly improves the viability of frozen cultures of ectomycorrhizal basidiomycetes. Fungal Biology. 124 (8), 742-751 (2020).
- Pereira, O. L., Kasuya, M. C. M., Borges, A. C., Araújo, E. F. D. Morphological and molecular characterization of mycorrhizal fungi isolated from neotropical orchids in Brazil. Canadian Journal of Botany. 83 (1), 54-65 (2005).
- Riddell, R. W. Permanent stained mycological preparations obtained by slide culture. Mycologia. 42 (2), 265-270 (1950).
- Walsh, T. J., Hayden, R. T., Larone, D. H. . Larone's Medically Important Fungi: A Guide to Identification. , (2018).
- Microscopy: Chemical Reagents. British Mycological Society Available from: https://www.britmycolsoc.org.uk/field_mycology/microscopy/reagents (2022)
- Senanayake, I. C., et al. Morphological approaches in studying fungi: Collection, examination, isolation, sporulation and preservation. Mycosphere. 11 (1), 2678-2754 (2020).
- Slifkin, M., Cumbie, R. Congo red as a fluorochrome for the rapid detection of fungi. Journal of Clinical Microbiology. 26 (5), 827-830 (1988).
- Raeder, U., Broda, P. Rapid preparation of DNA from filamentous fungi. Letters in Applied Microbiology. 1 (1), 17-20 (1985).
- Martins, M. K., et al. Molecular characterization of endophytic microorganisms. Endophytic microorganisms: theoretical and practical aspects of isolation and characterization. 1st edition. , 189-211 (2014).
- Rayner, R. W. A Mycological Colour Chart. Commonwealth Mycological Institute. , (1970).
- Kornerup, A., Wanscher, J. H. . Methuen Handbook of Colour. Methuen handbook of colour. , (1967).
- Ridgway, R. . Color Standards and Color Nomenclature. , (1912).
- McGinnis, M. R. . Laboratory Handbook of Medical Mycology. , (2012).
- Webster, J., Weber, R. . Introduction to Fungi. , (2007).
- Sridharan, G., Shankar, A. A. Toluidine blue: A review of its chemistry and clinical utility. Journal of Oral and Maxillofacial Pathology. 16 (2), 251-255 (2012).
- Smith, D., Onions, A. H. S. A comparison of some preservation techniques for fungi. Transactions of the British Mycological Society. 81 (3), 535-540 (1983).
- Ryan, M. J., Smith, D., Jeffries, P. A decision-based key to determine the most appropriate protocol for the preservation of fungi. World Journal of Microbiology and Biotechnology. 16 (2), 183-186 (2000).
- Lalaymia, I., Cranenbrouck, S., Declerck, S. Maintenance and preservation of ectomycorrhizal and arbuscular mycorrhizal fungi. Mycorrhiza. 24 (5), 323-337 (2014).
- Zettler, L. W., Corey, L. L. Orchid mycorrhizal fungi: isolation and identification techniques. Orchid Propagation: From Laboratories to Greenhouses-Methods and Protocols. , 27-59 (2018).
- Yu, S., Wang, Y., Li, X., Yu, F., Li, W. The factors affecting the reproducibility of micro-volume DNA mass quantification in Nanodrop 2000 spectrophotometer. Optik. 145, 555-560 (2017).
- Martos, F., et al. Independent recruitment of saprotrophic fungi as mycorrhizal partners by tropical achlorophyllous orchids. New Phytologist. 184 (3), 668-681 (2009).
- Schoch, C. L., et al. Nuclear ribosomal internal transcribed spacer (ITS) region as a universal DNA barcode marker for Fungi. Proceedings of the National Academy of Sciences. 109 (16), 6241-6246 (2012).
- White, T. J., Bruns, T., Lee, S., Taylor, J. Amplification and direct sequencing of fungal ribosomal RNA genes for phylogenetics. PCR Protocols: A Guide to Methods and Applications. 18 (1), 315-322 (1990).
- Sanger, F., Nicklen, S., Coulson, A. R. DNA sequencing with chain-terminating inhibitors. Proceedings of the National Academy of Sciences. 74 (12), 5463-5467 (1977).
- Ranjard, L., et al. Characterization of bacterial and fungal soil communities by automated ribosomal intergenic spacer analysis fingerprints: biological and methodological variability. Applied and Environmental Microbiology. 67 (10), 4479-4487 (2001).
- Metzker, M. L. Sequencing technologies-the next generation. Nature Reviews Genetics. 11 (1), 31-46 (2010).
Erratum
Formal Correction: Erratum: Isolation, Characterization, and Total DNA Extraction to Identify Endophytic Fungi in Mycoheterotrophic Plants
Posted by JoVE Editors on 1/01/1970. Citeable Link.
An erratum was issued for: Isolation, Characterization, and Total DNA Extraction to Identify Endophytic Fungi in Mycoheterotrophic Plants. The Authors section was updated from:
Juliana Lishcka Sampaio Mayer
to:
Juliana Lischka Sampaio Mayer
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten