このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
菌従属栄養植物における内生菌を同定するための単離、特性評価、および全DNA抽出
Erratum Notice
要約
本稿では、植物関連内生菌の単離、分離株の長期保存、形態学的特性評価、およびその後の分子同定およびメタゲノム解析のための総DNA抽出のための詳細かつ適切なプロトコルを提供することを目的としています。
要約
菌従属栄養植物は、菌根依存の最も極端な形態の1つであり、独立栄養能力を完全に失っています。他の重要な資源と同様に、これらの植物が密接に関連している菌類は、彼らにとって不可欠です。したがって、菌従属栄養種の研究に最も関連性の高い技術のいくつかは、関連する真菌、特に根や地下器官に生息する真菌の調査を可能にするものです。これに関連して、文化依存性および培養非依存性の内生菌を同定する技術が一般的に適用される。真菌エンドファイトを単離することは、それらを形態学的に識別し、それらの多様性を分析し、ランの種子の共生発芽に応用するための接種を維持するための手段を提供します。しかし、植物組織に生息する非培養性真菌は多種多様であることが知られている。このように、培養に依存しない分子同定技術は、種の多様性と存在量をより広くカバーします。本稿は、文化依存型と独立型の2つの調査手順を開始するために必要な方法論的サポートを提供することを目的としています。培養依存性プロトコールに関しては、菌従属栄養植物の地下および空中器官からの糸状菌の単離、分離株の収集、スライド培養法による菌糸の形態学的特徴付け、および全DNA抽出による真菌の分子同定とともに、収集場所から実験室施設までの植物サンプルの収集と維持のプロセスが詳細に説明されています。培養に依存しない方法論を網羅する詳細な手順には、メタゲノム解析のための植物サンプルの収集や、市販のキットを使用したクロロフィル植物器官からの総DNA抽出が含まれます。最後に、連続性プロトコル(ポリメラーゼ連鎖反応[PCR]、シーケンシングなど)も分析に推奨されており、ここではその手法を紹介します。
概要
内生菌は、定義上、目立たない感染症で植物の器官や組織の内部に生息する菌類です(つまり、宿主に害を及ぼすことなく)1,2。これらの真菌は、宿主植物と中立的または有益に相互作用し、病原体や不利な環境条件に対する耐性を与え、植物にとって有益な化合物(成長因子やその他の植物ホルモンなど)の合成に寄与する可能性があります1,3。菌根内生菌は、植物と菌根の関連を確立し、栄養伝達に関与する真菌です4。ラン科では、菌根内生菌との相互作用は、大多数の種で種子の発芽、および科のすべての植物で苗の確立の基本です5。このような文脈では、菌従属栄養性のランは、菌根のパートナーに関する完全な依存の事例を表しており、ライフサイクル全体にわたってこれらの真菌によるミネラル栄養素と炭素化合物の移行に依存しています6。したがって、関連菌類の単離と同定は、菌従属栄養生物の生命戦略を研究する際の基本的な基盤となります。さらに、菌従属栄養植物における真菌エンドファイトの役割や、これらの真菌の実際の多様性についてはほとんど知られていません7,8。
内生菌類の調査は、伝統的に培養非依存性または依存性として説明されているさまざまな手法 を介して 実施できます:たとえば、(a)直接観察、(b)真菌の単離および形態学的および/または分子的同定、および(c)植物組織の全DNA抽出および分子同定9。直接観察(a)では、内生菌は、植物細胞や組織の内部にいる間に、光学顕微鏡または電子顕微鏡法9によって調査される可能性があり、さまざまな顕微鏡プロトコルがPena-Passosらによって詳述されています10。単離法(b)により、真菌エンドファイトは、そのコロニー、菌糸、および生殖または抵抗構造の形態に従って特徴付けることができます。また、単離技術 により 、DNA抽出、分子同定配列(バーコードまたは指紋)の増幅、およびシーケンシングによる単離株の分子同定を行うことが可能です11。後者の技術(c)は、植物組織の内部でDNA抽出(メタバーコーディング)を行い、その後にライブラリの調製とシーケンシングを行う12。
さらに、真菌分離株は、独立栄養または菌従属栄養性のランからの種子を使用して、共生発芽試験に適用できます。このような応用の一例は、Sistiらによって実施された調査であり13、菌従属栄養性のランである Pogoniopsis schenckiiの発芽と原生皮発生の初期段階を、非菌根性内生菌を含むその分離株のいくつかに関連して説明しています。適用された共生発芽プロトコルは、Pena-Passosらによるビデオで詳細に説明されています10。異なる植物器官と関連して真菌を単離することで、植物と真菌の相互作用の性質に関する多様な研究の焦点が可能になります(例えば、関連性の生態学的または生理学的側面のいずれかを理解するため、および真菌から植物への栄養素の移動に関する調査)9。
セクション1で提示された方法論は、地下臓器サンプルの収集に基づいており、これらの臓器は収集が最も困難であり、菌根内生菌がコロニーを形成するため、非常に興味深いものです。ただし、含まれている両方のプロトコル(ステップ1.1および1.2)は、他の菌従属栄養植物器官(例、根茎、花茎、および果実)に適用できます。ステップ1.1で説明した収集方法は、形態学的特性評価(セクション4および5)のための内生菌(セクション2)および/または分離株同定のための全DNA抽出(セクション6)に指定されています。一方、ステップ1.2で説明した収集方法は、メタバーコーディング技術(セクション7)のための植物組織の全DNA抽出にのみ割り当てられます。セクション3では、糸状菌の保存と保存のための4つの方法が提示されており、2つは短期保存(3〜6か月)に、他の2つは長期保存(>1年)に適しています。形態学的特徴付け(セクション4および5)は、それを強化し、真菌のマクロおよびミクロモルフォロジーに関する重要な情報を提供するために、分子同定と関連している可能性があります。 図 1 は、以降に説明する集合的な方法論をまとめたものです。

図1:提示された方法の概略図。植物の収集と真菌の分離、保存、および培養依存および非依存の方法論による分子同定。この図の拡大版をご覧になるには、ここをクリックしてください。
プロトコル
1. 植物サンプル採取
- 培養依存法のサンプル採取
- 地下の臓器を慎重に掘ります。これらは、収集する植物の根、茎、根茎、または貯蔵器官であり得る。非常にコンパクトな土壌とは別に、これらのサンプルを手作業で収集します。
注:このステップでこてやスコップなどのツールを使用することは、菌従属栄養植物の脆弱な構造を損傷し、非内生菌による組織汚染を引き起こす可能性があるため、お勧めできません。 - できるだけ多くの地下臓器を集める。サンプルは、冷却容器(発泡スチロールの箱や氷の入った保温袋など)内の紙袋に入れて保管してください。空中臓器も回収する場合は、地下の臓器とは別に輸送してください。
- 地下の臓器を慎重に掘ります。これらは、収集する植物の根、茎、根茎、または貯蔵器官であり得る。非常にコンパクトな土壌とは別に、これらのサンプルを手作業で収集します。
- 培養に依存しないメソッドのサンプル収集
- ステップ1.1.1のメモで指摘されているのと同じ推奨事項の下で収集する植物の根を慎重に掘ります。
- できるだけ多くの地下臓器を集める。採取したサンプルは、液体窒素内のクライオチューブに保持するか(望ましいオプション)、ドライアイスで囲んだ遠心分離チューブを使用します(代替オプション)。空中器官を集めた場合は、別々に保管してください。
2. 植物器官に付随する内生菌の単離14
注:このセクションで使用するすべての材料、溶液、および試薬は無菌でなければなりません。すでに滅菌済みで購入できないものは、121°Cで20分間オートクレーブ滅菌する必要があります。
- 植物器官の表面的駆除
- 採取したサンプルを流水で洗浄し(ステップ1.1.2)、サンプルに含まれる可能性のある基板やその他の破片をできるだけ多く取り除きます。
- 層流フード内で、洗浄したサンプルを70%エタノールに1分間浸漬した状態を維持します(ビーカーまたはガラス瓶を使用できます)。
- 次亜塩素酸ナトリウムと2%活性塩素の入った別の容器にサンプルを3分間移します。
- サンプルを70%エタノールの入った容器に変更し、1分間浸漬したままにします。その後、蒸留水を入れた2つの容器でサンプルを順次洗浄します。
- 培地への植物片の設置
注意: ステップ2.2で詳述されているすべてのステップは、層流フード内で実施する必要があります。- 設置する前に、19.5 g / Lのジャガイモデキストロース寒天培地(PDA)+ 7 g / Lの細菌寒天薬+ 3 mL / Lの抗生物質(例:ストレプトマイシン、ペニシリン、テトラサイクリン、アンピシリン)でペトリ皿(直径8〜10 cm)を準備します。
- ペトリ皿は、コンタミネーションがないことを確認するために、使用前に培地で36°Cで24時間維持してください。バクテリアや真菌のコロニーで汚染された皿を捨ててください。
注:このステップでは、臓器が真菌の増殖を阻害する可能性のある細菌に強く感染しているため、培地への抗生物質の添加が不可欠です。オートクレーブ中に抗生物質が加熱される可能性があるかどうかを確認します。一部の抗生物質は、培地を約36°Cに冷却した後(皿に入れる前)に添加する必要があります。 - サンプルの表面的な消毒の直後に、まだ層流フード内、PDAで、サンプルの洗浄に使用した最後の容器から蒸留水を数滴接種します。このステップは、サンプルの表面的な消毒の有効性を評価するために重要です。
- 空のオートクレーブ皿にサンプルを入れ、炎を盛ったメスと鉗子を使用してサンプルを約0.2cmの厚さに切片化します。
- 必要に応じて、円筒形サンプルを縦方向に2つに切断し、媒体と接触する表面を増幅する。果物やその他のより球形の臓器をよりよく露出させるには、構造を完全に切り刻むかスライスします。種子も重要な真菌源である可能性があるため、切片化された果実がそれらを培地にさらすことを確認してください。
注:真菌エンドファイトの分離には、組織損傷や病気や病原性感染の兆候がない健康な臓器のみを使用する必要があります。 - 地下臓器の5つの断片をPDA +抗生物質でペトリ皿に配布します。破片が互いにできるだけ離れており、皿の端に触れていないことを確認してください。培地にフラグメントを配列しないでください。汚染の場合の予防措置として、設置された各臓器の複製を準備します。
- ペトリ皿をラップフィルムで密封し、25〜27°Cの暗所(できればインキュベーター内)で5日間保管します。
- 絶縁周波数 (IF) 計算11
- 臓器片のインキュベーションの5日後、次の式で表されるように、真菌コロニーが成長する真菌コロニーを示すインキュベートされたフラグメントの数をインキュベートされたフラグメントの合計で割った値に従ってIFを計算します。
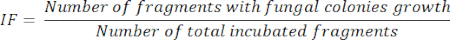
- 臓器片のインキュベーションの5日後、次の式で表されるように、真菌コロニーが成長する真菌コロニーを示すインキュベートされたフラグメントの数をインキュベートされたフラグメントの合計で割った値に従ってIFを計算します。
- 縞模様および継代培養による真菌分離株の精製
注意: ステップ2.4で詳述されているすべてのステップは、層流フード内で実施する必要があります。- ペトリ皿(直径5cm)を5-7 g / L寒天培地(AA;細菌寒天のみ)で準備します。ディッシュを36°Cで24時間維持して、汚染された可能性のあるディッシュを排除します。
- 成長した各真菌コロニーをコードで識別し、皿の下側にその余白を区切ります(油性マーカーで行うことができます)。色、成長パターン、テクスチャ、およびマージンの形式でコロニーを区別します。
- オートクレーブ処理した木製のつまようじで、細い先端を使用して、真菌コロニーから少量の菌糸体を回復します。できればコロニーの縁に集中し、一度に複数の種類のコロニーを回復しないように、別のコロニーからできるだけ遠い領域を選択します。
- 先端に菌糸体を付けた同じ爪楊枝を使用して、AAを縞模様にして3つの線条(溝)を作ります。各線条が別の線条と皿の端から1 cmの距離にあることを確認してください。適切なコード(ステッカー用紙と鉛筆を使用)を書き、皿を密封し、25〜27°Cの暗闇で3日間インキュベートします。
- ペトリ皿(直径8cm)を39g/LのPDAで調製します。この段階で抗生物質を追加する必要はありません。
- AAディッシュをインキュベートした後、光(ランプや窓からのもの)に対して細心の注意を払って観察し、個々のコロニーを形成する微細な菌糸を特定することを目的としています。シャーレの底面にある油性マーカーを使用して、ディッシュごとに個々のコロニーの面積を区切ります。
- 層流フードでは、オートクレーブ滅菌したつまようじを使用して、コロニーを含む培地の一部を切断し、切断した容量を新しいPDAディッシュの中央に移します。
- 分離株のコードでシャーレを識別し、ラップフィルムでディッシュを密封し、25〜27°Cの暗闇で7〜14日間維持します。
3. 精製真菌分離株の保存
注:このセクションで使用するすべての材料、溶液、および試薬は無菌でなければなりません。すでに滅菌済みで購入できないものは、121°Cで20分間オートクレーブ滅菌する必要があります。
- カステラーニ法または鉱油での保存(3〜6か月)11,15
- 0.5 mLの蒸留水または0.5 mLの鉱油を含む2 mLの微量遠心チューブを準備します(選択した方法によって異なります)。チューブが空でオートクレーブされ、オートクレーブされた水とオイルが層流フード内のチューブに添加されていることを確認します。
- 層流フードに、PDA(39 g / L)で7〜14日間培養した精製分離株を入れたペトリ皿を置きます。オートクレーブ滅菌したつまようじを使用して、菌糸体縁を含む培地の小さな直方体(上部0.5 cm x 0.5 cm)を切ります。
- 蒸留水(カステラーニ法)または鉱油を入れた微量遠心チューブに4〜6個の直方体を置きます。チューブは、25°Cの暗所で必要なだけ保管し、メソッドの時間制限を守ってください。
注意: チューブに直方体を追加しすぎていっぱいにすると、汚染の可能性が高まる可能性があります。チューブを冷蔵状態に保つことが可能であり、これにより一部の分離株をより長く保存することができます。Currah et al.16 は、熱帯のランから分離された菌根菌の複製物は、冷蔵保存時に失われる可能性があるため、冷蔵および25°Cで保存することを推奨しています。 - 直方体を回復し、新しいPDAディッシュの中央に置いて、保存された分離株を成長させます。
- 籾摺り米粒の凍結保存(>1年)17
- 籾摺りした米粒を流水で洗い、籾殻が開き始めるまで炊きます。調理した穀物をスクリューキャップ付きのガラス試験管に分配し、オートクレーブを24時間の間隔で2回分配します。
- 層流フードに、PDA(39 g / L)で7〜14日間培養した精製分離株を入れたペトリ皿を置きます。オートクレーブ滅菌したつまようじを使用して、菌糸体縁から5つの小さな菌糸の断片を回収し、オートクレーブ滅菌した米粒の入ったチューブに接種します。
注:チューブのさまざまなポイントと深さに菌糸を接種することで、分離された菌糸がより短い時間で殻をむいた米粒にコロニーを形成することが保証されます。 - チューブを暗所で25〜27°Cで14日間インキュベートします。3日ごとにチューブをボルテックスして攪拌し、粒子を個別化します。
- 米粒の真菌の繁殖を観察した後、湿気を吸収するために濾紙で裏打ちされたオートクレーブ滅菌したペトリ皿に米粒を紙の上に分配し、米粒を乾燥させます。25〜27°Cの暗所で2〜3日間保管してください。
- 底部に1/3のシリカゲル、シリカの上に1/3のグラスウールを敷いたクライオチューブを準備します。最後に、米粒の1/3に成長した真菌菌糸体を分配します。-20°Cで24時間保管してください。
- 24時間後、クライオチューブを-80°Cで必要なだけ保存し、分析法の時間制限を観察します。
- 凍結保護剤を用いたバーミキュライトの凍結保存(>1年)18
- 蒸留水に0.2%酵母抽出物と2%グルコースからなる液体培養培地を調製します。pHを5に調整し、オートクレーブします。
- 0.2 gのバーミキュライト(細かい顆粒測定を使用)をスクリューキャップ付きのクライオチューブに分配し、オートクレーブします。0.8 mLのオートクレーブ処理した液体培地を、バーミキュライトを含むクライオチューブに加えます。
- 層流フードに、PDA(39 g / L)で7〜14日間培養した精製分離株を入れたペトリ皿を置きます。オートクレーブ滅菌したつまようじを使用して、菌糸体縁から3〜5個の菌糸断片を回収し、バーミキュライト+液体培養培地を含むチューブに沿って接種します。クライオチューブをそれぞれのアイソレートコードで識別することを忘れないでください。
注:クライオチューブのさまざまなポイントと深さに菌糸を接種することで、分離物がより短い時間でバーミキュライトにコロニーを形成することが保証されます。 - クライオチューブは、ほとんどのバーミキュライト粒子のコロニー形成が観察されるまで、25〜27°Cの暗所で保管します(通常、約14日かかります)。
- 蒸留水に5%のグリセロールと5%のトレハロースからなる凍結保護剤溶液を調製し、オートクレーブします。使用する前に溶液を冷ましてください。
- クライオチューブ内のバーミキュライトがそれぞれの真菌分離株によってコロニー形成された後、各チューブに0.4 mLの凍結保護剤を分配し、クライオチューブを4°Cで48時間冷蔵保存します。その後、クライオチューブを必要な限り-80°Cに維持し、分析法の時間制限を守ります。
4. 糸状菌類のマクロ形態解析(コロニー形態)
- 各ペトリ皿で成長した菌糸体の写真レジスターを39 g / L PDAで7〜14日間維持します。コロニーの両側、上側と下側(逆)を登録することを忘れないでください。セクション6ですぐに使用できる食器やメンテナンスされていない場合は、写真を撮るときに食器を開けて、より良い写真を撮ってください。
- コロニーの形態に関する定量的データに関心がある場合は、単離株を複製し、同じ増殖条件で一定期間維持して、コロニーの直径を登録し、増殖速度(通常はmm/h)を計算します。
注:比較19 のために異なるタイプの培地を使用し、データを処理するための統計ツールを考慮することにより、より洗練された定量的結果を達成することができる。 - コロニーを実体顕微鏡で観察し、形態学的特徴を特定し、拡大して撮影します。結果のセクションで詳しく説明されているように、コロニーをマクロモルフォロジー特性に従って評価します。多様な書誌資料を参照して、マクロモルフォロジーを分類し、分子および/または形態学的同定に関連付けます。
5. 糸状菌の微細形態学的特性(菌糸形態)
注:微細形態学的手法は、考えられる使用法と欠点を考慮して、ディスカッションセクションで比較されます。
- 39 g / L PDAを使用してペトリ皿で分離株を7〜14日間増殖させます。推定蘭菌根菌を評価するために、17 g / Lコーンミール寒天培地(CMA;または別の栄養制限培地)で3〜7日間分離株を培養します19。
- いじめマウント
- 層流フード内で作業します。選択した染みを一滴垂らします(手順5.5)。
- オートクレーブ滅菌したつまようじまたは別の滅菌材料を使用して、成長した分離物から菌糸を慎重に取り除き、染みの滴に入れます。カバーガラスを置き(気泡を避けるために、できれば初期角度45°)、光学顕微鏡で分析します。
- 粘着テープマウント
注:この技術は、粘着テープをオートクレーブすることができず、メソッドの実行後に汚染が発生する可能性があるため、通常、セクション6で容易に使用できるか、維持されない真菌培養に適用されます。- 選択した染みを一滴垂らします(手順5.5)。透明な粘着テープをスライドガラスに合う大きさにカットし、中央の汚れが落ちます。
- ストリップの粘着性のある表面を菌糸体の表面に向けます。押さず、あまり多くの菌糸を貼り付けずに菌糸を集めてみてください。
- テープをスライドガラスに貼り付け、汚れが採取した菌糸に接触していることを確認します。テープの上に水滴を置き、カバーガラスを置きます。スライドを光学顕微鏡で分析します。
- 糸状菌のスライド培養20
- 大きなシャーレ(直径>9 cmが望ましい)の中に、底部にろ紙、U字型のガラス棒またはアダプテーション(目的はスライドガラスに高さを提供すること)、1つのスライドガラス、および2つのカバーガラスを置きます。より高いペトリ皿は操作を容易にします。これらのペトリ皿のキットを、分離物ごとに1つずつオートクレーブします。
- 39 g/L PDAまたは17 g/L CMAの少量を調製し、10 g/Lの細菌寒天培地とオートクレーブを加えます(ペトリ皿1枚分の容量は30個以上の分離株に十分です)。スライド培養に用いる培地は、通常の培地よりも硬いものでなければなりません。100mLの蒸留水をオートクレーブします。
- 層流フード内で作業します。液体培地をペトリ皿に入れ、高さ約0.5cmの層を作ります。培地を固めます。固まったら、滅菌メスを使用して、寸法が1 cm x 1 cmの培地の正方形を切ります。
- 層流フード内のペトリ皿キットを開き、フレーム付き鉗子を使用して、皿内でオートクレーブ滅菌された材料を整理します。 図2Aに示すように、材料を番号順に並べ、スライドガラスの上に正方形の培地を置きます。カバーガラスを培地の上に置く前に、手順5.4.5に進みます。
- オートクレーブ滅菌したつまようじを使用して、分離物から菌糸を回収し、スライドガラスの上に置かれた培地の4つの側面を注意深くこすります。オートクレーブ処理したカバーガラスを、フレーム鉗子を使用して中型の正方形の上に置きます。
- 滅菌ピペットチップを使用して、オートクレーブした水をろ紙に入れ、湿度の高いチャンバーを作成します。水を過剰に入れずに紙を飽和させるボリュームを使用します。ペトリ皿をラップフィルムで密封し、それぞれの分離コードで識別します。25〜27°Cの暗所で3〜7日間皿を保管します。
- スライドガラスとカバーガラスの菌糸の成長を評価します。各スライド培養キットから2枚のスライドを作製し、1枚目は菌糸入りスライドガラスとオートクレーブカバーガラスを使用し、もう1枚目は菌糸入りカバーガラスともう1枚スライドガラスを使用します。
- 菌糸の成長が観察されたら、正方形の培地をスライドガラスから慎重に取り外し(図2B)、選択した染色剤を使用して2枚のスライドをマウントし(ステップ5.5)、光学顕微鏡で分析します。スライド上の分離コードを忘れずに識別してください。半永久的なスライドを作成するには、カバーガラスを透明なマニキュアで密封します。
- 恒久的なスライドを作るには、染色後に付着した菌糸から染料を注意深く洗い流し、スライドを乾燥させます。迅速な封入剤でマウントします(詳細については、Pena-Passos et al.10 を参照してください)。
注:この技術で恒久的なスライドを製造することの欠点は、ガラスに付着していない胞子や構造を洗浄する可能性があることです。
- 染色法と菌糸の観察
- ラクトフェノールコットンブルー(LPCB)21:乳酸20mL、グリセロール40mL、蒸留水20mLを加えます。20gのフェノール結晶をこの溶液に穏やかに加熱して溶解します。0.05gのメチルブルー(コットンブルー、または2mLの1%水溶液)を溶解します。LPCBは、すでに準備されているものを簡単に購入することもできます。
注意:フェノールは非常に有毒で揮発性です。ドラフト内でのみ操作し、手袋を着用してください。 - トルイジンブルーO10(TBO):0.1 Mリン酸緩衝液(pH 6.8)で0.05%TBOを調製します。
- コンゴレッド22,23,24:蒸留水に1%コンゴレッドの溶液を調製し、ろ過します。5〜10分間インキュベートします。この色素は、蛍光顕微鏡25にも適用することができる。
- 真菌の菌糸を光学顕微鏡で観察・撮影する場合は、構造の同定に役立てるために、さまざまな書誌資料を調べてください(議論を確認してください)。
- ラクトフェノールコットンブルー(LPCB)21:乳酸20mL、グリセロール40mL、蒸留水20mLを加えます。20gのフェノール結晶をこの溶液に穏やかに加熱して溶解します。0.05gのメチルブルー(コットンブルー、または2mLの1%水溶液)を溶解します。LPCBは、すでに準備されているものを簡単に購入することもできます。

図2:糸状菌のスライド培養の手順 。 (A)スライド培養キットの概略構成で、数字は要素の配置順序を示す。(B)スライドガラス及びカバーガラスに菌糸成長後の四角い培地の剥離が観察される。 この図の拡大版をご覧になるには、ここをクリックしてください。
6.真菌分離株からの総DNA抽出(自家製プロトコル26 と修正27)
注:このセクションで使用するすべての材料、溶液、および試薬は無菌でなければなりません。すでに滅菌済みで購入できないものは、121°Cで20分間オートクレーブ滅菌する必要があります。プロトコル全体を通して手袋を着用し、ドラフト内でいくつかの段階を実行します。
- 抽出バッファー(1% ドデシル硫酸ナトリウム(SDS)、250 mM NaCl、200 mM Tris-HCl(pH 8.0)、25 mM エチレンジアミン四酢酸(EDTA)を調製します。精製した分離株を39 g / L PDAで7〜14日間増殖させます。
- へらまたはスプーンを使用して分離物から菌糸体を慎重にこすり落とし、菌糸凝集体と一緒に培地を移さないように、破片をオートクレーブ滅菌した乳鉢に移します。磁器の杵を使用して、液体窒素で菌糸体を微粉末に粉砕します。粉砕される菌糸体を溶かさず、窒素を加えて溶かしてください。
- 2 mLの微量遠心チューブに1 mLの抽出バッファーを加え、粉砕したサンプルを1.5 mLになるまで置きます。チューブの内容物を静かに攪拌して均質化します。
- チューブを渦中で5秒間撹拌し、65°Cのサーモブロックに20分間入れます。7〜10分ごとに反転させて内容物を慎重に均質化します。
- チューブを10,000 x g で4°Cで10分間遠心分離します。 上相から 800 μL を新しい 2 mL 微量遠心チューブに移し、800 μL のフェノールをチューブに添加し、内容物を反転させて混合します。
注意:フェノールは非常に有毒で揮発性の試薬です。紅斑、壊疽、組織壊死を引き起こす可能性があります。吸入すると、呼吸困難や咳を引き起こす可能性があります。全身吸収は、肝臓、腎臓、中枢神経系に損傷を与える可能性があります。ドラフト内でのみフェノールを操作し、手袋を着用してください。
注意: 次のステップ以降は、手袋を使用し、ドラフト内でプロトコルを実施してください。 - チューブを 10,000 x g で 4 °C で 10 分間遠心分離し、下相の内容物の移入を慎重に避けながら、800 μL を上相から新しい 2 mL チューブに移します。
- 400 μLのフェノールと400 μLのクロロホルムをチューブに加え、反転させて内容物を混合します。10,000 x g で4°Cで10分間遠心分離します。
注意:クロロホルムは毒性が高く揮発性の試薬です。皮膚に接触すると刺激や怪我を引き起こす可能性があり、吸入すると中枢神経系、心肺系、肝臓、腎臓に影響を与えます。クロロホルムはドラフト内でのみ操作し、手袋を着用してください。 - 上相から800μL以下を新しい2 mLチューブに再回収します。800μLのクロロホルムを加え、内容物を反転させて混合し、10,000 x g で4°Cで10分間遠心分離します。
注:このステップでは、下相からの残留物を新しいチューブに移してはなりません。したがって、上相を回復するときは特に注意してください。 - 上相から600〜800 μLを新しい1.5 mLチューブに移し、450 μLのイソプロパノールを加えます。内容物を反転させて混合し、25°Cで5分間インキュベートします。
- チューブを10,000 x g で4°Cで5分間遠心分離し、マイクロピペットを使用して上清を廃棄します。チューブの底に堆積したペレットを捨てないように注意してください。
- 500μLの80%エタノールを加え、5分間遠心分離します。ペレットが清澄化されない場合は、2回繰り返します。
- マイクロピペットを使用してエタノールを除去し、ペレットを37°Cで30〜60分間乾燥させます。30〜50μLの脱イオン水を加え、マイクロピペットを使用してペレットを溶出します。
- DNAが完全に溶出するためにチューブを4°Cで一晩維持し、内容物を-20°Cで凍結します。
7. メタバーコーディング法のための植物器官からの総DNA抽出(市販キット)
注:以下の方法論では、土壌DNA抽出キットとして 材料表 に示されている市販のキットを購入する必要があります。このセクションで使用するすべての材料、溶液、および試薬は無菌でなければなりません。すでに滅菌済みで購入できないものは、121°Cで20分間オートクレーブ滅菌する必要があります。プロトコル全体を通して手袋を着用することを強くお勧めし、ステップは層流フード内で行うことができます。記載されたプロトコルは、メーカーによって詳述されたプロトコルから、De Souza et al.12から修正されています。
- 磁器の乳棒と乳鉢を使用して、ステップ1.2に従って収集した根を液体窒素で粉砕し、サンプルを微粉末に還元します。粉砕したサンプルから 0.3 g を PowerBead チューブに添加し、穏やかに撹拌して均質化します。
- 60 μLの溶液C1(キットに付属)をPowerBeadチューブに加え、内容物を反転させて混合します。組織ホモジナイザーとセルライザー(材料表)を使用して、チューブを適切なサポートにしっかりと結び、サポートをボルテックスに結合し、最高速度で10〜20分間装置を始動します。
注意:C1溶液が沈殿した場合は、完全に溶解するまで60°Cで加熱してください。 - チューブを10,000 x g で25°Cで30秒間遠心分離します。 上清 500 μL を 2 mL の微量遠心チューブに移します。転送されたコンテンツは粒子状である可能性があります。
- 250 μL の溶液 C2 をチューブに加え、渦中で 5 秒間撹拌します。4°Cで5分間インキュベートした後、25°Cで10,000 x g で1〜2分間遠心分離します。
- 600 μLを超える上清を新しい2 mLチューブに移し、ペレットを避けます。200 μL の溶液 C3 を加え、ボルテックス上で 5 秒間撹拌します。
- 4°Cで5分間インキュベートした後、25°Cで10,000 x g で1分間遠心分離します。 この段階では、上澄みが微粒子でないことを確認します。
- 750 μLを超える上清を新しい2 mLチューブに移し、ペレットを避けます。溶液C4をよく均質化し、上清に溶液C41,100μLを加え、ボルテックス中で5秒間撹拌する。
- MB スピンカラムのチューブ内容物から 675 μL をフィルターにロードし、10,000 x g で 25 °C で 1 分間遠心分離します。 液体の内容物を廃棄します。
- 各チューブのすべての内容物が処理されるまで、前の手順を2回繰り返します。次に、チューブの上カラムにあるフィルターの中央に500μLの溶液C5を加え、25°Cで10,000 x g で30秒間遠心分離します。
- 液体の内容物を廃棄し、前のステップと同じ条件で再度遠心分離します。MB Spin Columnの優れたカラムを新しい2 mL微量遠心チューブに慎重に移し、カラム内の液体が滴り落ちないようにします。
- 85〜100μLの溶液C6をフィルターの中央に加え、1分間待ちます。10,000 x g で 25 °C で 30 秒間遠心分離し、MB スピンカラムを廃棄します。チューブは-80°Cで保管してください。
8. 分光光度計でのDNA定量(材料表を参照)
- 指定された分光光度計を使用して、ソフトウェアを開きます。 [核酸]オプションを選択し、[ DNA]オプションを選択して、濃度を ng/μL に設定します。
- 分光光度計検出器に 1 μL の脱イオン水を加え、 Blank オプションを選択して校正します。読み取り後、柔らかいティッシュペーパーで検出器をそっと拭きます。
- フィールドサンプルに読み取りたいサンプルのアイソレートコードを付け、1 μLを装置検出器に入れます。[ 測定]オプションを選択します。結果を含むグラフと表が生成されます。
- すべてのサンプルが読み取られるまで、手順8.3を繰り返します。記録と分析のために生成された結果を含むテーブルを保存することをお勧めします: レポート オプションと.xmlファイルを保存する場所を選択します。
- 検出器に5μLの脱イオン水を加え、数分待ってから、ソフトティッシュペーパーでやさしく拭きます。
結果
分離プロトコルでは、最後の洗浄で使用した水からの汚染があり、その汚染が接種された断片を含むペトリ皿でも検出されることを考慮して、汚染物質の種類に応じて異なる措置が講じられる可能性があります(表1)。この手順は、増殖が加速する高度に胞子形成された真菌汚染物質、および選択した抗生物質に耐性のある激しく増殖する細菌の場合には?...
ディスカッション
植物サンプルの表面的な駆除は、提示されたプロトコルの最も重要な段階の1つです。最後の洗浄からの滴でPDA皿に汚染しないことが強く望まれます。内生菌が植物組織内でも一般的であることを考慮すると、細菌は隔離皿の汚染物質として頻繁に観察され、通常は空気中の胞子形成菌よりも多くなります3,11。したがって、臓器片を設置する際の培...
開示事項
著者は開示するものは何もなく、利益相反もありません。
謝辞
FAPESP(2015/26479-6)及びCNPq(447453/2014-9)からの資金提供に感謝します。JLSMは、生産性助成金(303664/2020-7)を提供してくれたCNPqに感謝します。MPPはCapes(修士号奨学金、プロセス88887.600591 / 2021-00)とCNPqに感謝します。
資料
| Name | Company | Catalog Number | Comments |
| Adhesive tape | (from any company, for adhesive tape mount in micromorphological analyses) | ||
| Ampicillin | Sigma-Aldrich | A5354 | (for installation of plant fragments; other antibiotics may be used - check step 2.2.1) |
| Autoclave | (from any company, for materials sterilization in many steps) | ||
| Bacteriological agar | Sigma-Aldrich | A1296 | (for many steps) |
| C1, C2, C3, C4, C5, and C6 solutions | Qiagen | 12888-50 | (purchased with DNeasy PowerSoil kit) |
| Centrifuge | Merck/Eppendorf | 5810 G | (for total DNA extraction from fungal isolates) |
| Centrifuge tubes | Merck | CLS430828 | (for samples collection) |
| Chloroform | Sigma-Aldrich | C2432 | (for total DNA extraction from fungal isolates) |
| Congo red | Supelco | 75768 | (for hyphae staining) |
| Cryotubes | Merck | BR114831 | (for many steps) |
| Ethanol | Supelco | 100983 | It will be necessary to carry out the appropriate dilutions (for many steps) |
| Ethylenediaminetetraacetic acid (EDTA) | Sigma-Aldrich | 3609 | (for total DNA extraction from fungal isolates) |
| Filter paper | Merck | WHA10010155 | (for many steps) |
| Glass test tubes | Merck | CLS7082516 | (for cryopreservation in unhulled rice grains) |
| Glass wool | Supelco | 20411 | (for cryopreservation in unhulled rice grains) |
| Glucose | Sigma-Aldrich | G8270 | Or dextrose (for cryopreservation in vermiculite) |
| Glycerol | Sigma-Aldrich | G5516 | Or glycerin (for cryopreservation in vermiculite, for preparing LPCB) |
| Isopropanol | Sigma-Aldrich | 563935 | (for total DNA extraction from fungal isolates) |
| Lactic acid | Sigma-Aldrich | 252476 | (for preparing LPCB - hyphae staining) |
| Lactophenol blue solution (LPCB) | Sigma-Aldrich | 61335 | (for hyphae staining) |
| Laminar flow hood | (class I, from any company, for many steps) | ||
| Light microscope | (from any company, for hyphae observation) | ||
| MB Spin Columns | Qiagen | 12888-50 | (purchased with DNeasy PowerSoil kit) |
| Methyl blue (cotton blue) | Sigma-Aldrich | M5528 | (for preparing LPCB - hyphae staining) |
| Microcentrifuge tube (1.5 mL) | Merck | HS4323 | (for total DNA extraction from fungal isolates) |
| Microcentrifuge tube (2 mL) | Merck | BR780546 | (for many steps) |
| Mineral oil | (for preservation of fungal isolates) | ||
| Paper bags | Average size 150 mm x 200 mm (for samples collection) | ||
| Petri dish (Glass, 120 mm x 20 mm) | Merck/Pyrex | SLW1480/10D | (autoclavable, for fungi slide culture, prefer higher ones) |
| Petri dish (Glass, 50 mm x 17 mm) | Merck/Aldrich | Z740618 | (for purification of fungal isolates); alternatively: polystyrene petri dishes (sterile, γ-irradiated, non-autoclavable) |
| Petri dish (Glass, 80 mm x 15 mm) | Merck/Brand | BR455732 | (for installation of plant fragments); alternatively: polystyrene petri dishes (sterile, γ-irradiated, non-autoclavable) |
| Phenol | Sigma-Aldrich | P1037 | (for total DNA extraction from fungal isolates, for preparing LPCB) |
| Porcelain mortar | Sigma-Aldrich | Z247464 | (for total DNA extraction from fungal isolates) |
| Porcelain pestle | Sigma-Aldrich | Z247502 | (for total DNA extraction from fungal isolates) |
| Potato dextrose agar (PDA) | Millipore | P2182 | (for many steps) |
| PowerBead tubes | Qiagen | 12888-50 | (purchased with DNeasy PowerSoil kit) |
| Rapid mounting medium (Entellan) | Sigma-Aldrich | 1.0796 | (for fungi slide culture) |
| Silica gel | Supelco | 717185 | (for cryopreservation in unhulled rice grains) |
| Sodium chloride (NaCl) | Sigma-Aldrich | S9888 | (for total DNA extraction from fungal isolates) |
| Sodium dodecyl sulfate (SDS) | Sigma-Aldrich | L3771 | Lauryl sulfate sodium salt (for total DNA extraction from fungal isolates) |
| Sodium hypochlorite (w/ 2% active chlorine) | (commercial product, for superficial desinfestation) | ||
| Soil DNA extraction kit (DNeasy PowerSoil kit) | Qiagen | 12888-50 | (for total DNA extraction from plant organs) |
| Spectrophotometer - Nanodrop 2000/2000c | ThermoFisher Scientific | ND2000CLAPTOP | (for total DNA extraction from plant organs) |
| Stereomicroscope | (=dissecting microscope, from any company, for macromorphological analyses) | ||
| Tetracycline | Sigma-Aldrich | T7660 | (for installation of plant fragments) |
| Thermoblock | Merck/Eppendorf | EP5362000035 | (or from other companies) |
| Tissue homogenizer and cell lyzer | SPEX SamplePrep | 2010 Geno/Grinder - Automated Tissue Homogenizer and Cell Lyzer (for total DNA extraction from plant organs) | |
| Toluidine blue O | Sigma-Aldrich/Harleco | 364-M | (for hyphae staining) |
| Trehalose | Sigma-Aldrich | T9531 | (for cryopreservation in vermiculite) |
| Tris Base Solution (Tris) | Sigma-Aldrich | T1699 | (for total DNA extraction from fungal isolates) |
| Unhulled rice grains | (for cryopreservation) | ||
| U-shaped glass rod | (or an adaptation - check step 5.4.1, for fungi slide culture) | ||
| Vermiculite | Fine granulometry (for cryopreservation in vermiculite) | ||
| Vortexer | Sigma-Aldrich/BenchMixer | BMSBV1000 | (for total DNA extraction from fungal isolates) |
| Yeast extract | Sigma-Aldrich | Y1625 | (for cryopreservation in vermiculite) |
参考文献
- de Azevedo, J. L. Endophytic microorganisms. Ecologia Microbiana. , 117-137 (1998).
- Stone, J. K., Bacon, C. W., White, J. F. An overview of endophytic microbes: endophytism defined. Microbial Endophytes. , 17-44 (2000).
- Schulz, B., Boyle, C. What are Endophytes. Microbial Root Endophytes. , 1-13 (2006).
- Smith, S. E., Read, D. J. . Mycorrhizal Symbiosis. , (2008).
- Rasmussen, H. N., Dixon, K. W., Jersáková, J., Těšitelová, T. Germination and seedling establishment in orchids: a complex of requirements. Annals of Botany. 116 (3), 391-402 (2015).
- Rasmussen, H. N., Rasmussen, F. N. Orchid mycorrhiza: implications of a mycophagous life style. Oikos. 118 (3), 334-345 (2009).
- Ma, X., Kang, J., Nontachaiyapoom, S., Wen, T., Hyde, K. D. Non-mycorrhizal endophytic fungi from orchids. Current Science. 109 (1), 72-87 (2015).
- Favre-Godal, Q., Gourguillon, L., Lordel-Madeleine, S., Gindro, K., Choisy, P. Orchids and their mycorrhizal fungi: an insufficiently explored relationship. Mycorrhiza. 30 (1), 5-22 (2020).
- Sun, X., Guo, L. -. D. Endophytic fungal diversity: review of traditional and molecular techniques. Mycology. 3 (1), 65-76 (2012).
- Pena-Passos, M., Sisti, L. S., Mayer, J. L. S. Microscopy techniques for interpreting fungal colonization in mycoheterotrophic plants tissues and symbiotic germination of seeds. Journal of Visualized Experiments. (183), e63777 (2022).
- Araújo, W. L., et al. . Endophytic microorganisms: Theoretical and Practical Aspects of Isolation and Characterization. 1st ed. 1, 257 (2014).
- de Souza, R. S. C., et al. Unlocking the bacterial and fungal communities assemblages of sugarcane microbiome. Scientific Reports. 6, 28774 (2016).
- Sisti, L. S., et al. The role of non-mycorrhizal fungi in germination of the mycoheterotrophic orchid Pogoniopsis schenckii Cogn. Frontiers in Plant Science. 10, 1589 (2019).
- Araújo, W. L., et al. Variability and interactions between endophytic bacteria and fungi isolated from leaf tissues of citrus rootstocks. Canadian Journal of Microbiology. 47 (3), 229-236 (2001).
- Castellani, A. Further researches on the long viability and growth of many pathogenic fungi and some bacteria in sterile distilled water. Mycopathologia. 20 (1-2), 1-6 (1963).
- Currah, R. S., Zelmer, C. D., Hambleton, S., Richardson, K. A. Fungi from orchid mycorrhizas. Orchid Biology: Reviews and Perspectives, VII. , 117-170 (1997).
- Freitas, E. F. S., et al. Diversity of mycorrhizal Tulasnella associated with epiphytic and rupicolous orchids from the Brazilian Atlantic Forest, including four new species. Scientific Reports. 10 (1), 7069 (2020).
- Sato, M., Inaba, S., Noguchi, M., Nakagiri, A. Vermiculite as a culture substrate greatly improves the viability of frozen cultures of ectomycorrhizal basidiomycetes. Fungal Biology. 124 (8), 742-751 (2020).
- Pereira, O. L., Kasuya, M. C. M., Borges, A. C., Araújo, E. F. D. Morphological and molecular characterization of mycorrhizal fungi isolated from neotropical orchids in Brazil. Canadian Journal of Botany. 83 (1), 54-65 (2005).
- Riddell, R. W. Permanent stained mycological preparations obtained by slide culture. Mycologia. 42 (2), 265-270 (1950).
- Walsh, T. J., Hayden, R. T., Larone, D. H. . Larone's Medically Important Fungi: A Guide to Identification. , (2018).
- Microscopy: Chemical Reagents. British Mycological Society Available from: https://www.britmycolsoc.org.uk/field_mycology/microscopy/reagents (2022)
- Senanayake, I. C., et al. Morphological approaches in studying fungi: Collection, examination, isolation, sporulation and preservation. Mycosphere. 11 (1), 2678-2754 (2020).
- Slifkin, M., Cumbie, R. Congo red as a fluorochrome for the rapid detection of fungi. Journal of Clinical Microbiology. 26 (5), 827-830 (1988).
- Raeder, U., Broda, P. Rapid preparation of DNA from filamentous fungi. Letters in Applied Microbiology. 1 (1), 17-20 (1985).
- Martins, M. K., et al. Molecular characterization of endophytic microorganisms. Endophytic microorganisms: theoretical and practical aspects of isolation and characterization. 1st edition. , 189-211 (2014).
- Rayner, R. W. A Mycological Colour Chart. Commonwealth Mycological Institute. , (1970).
- Kornerup, A., Wanscher, J. H. . Methuen Handbook of Colour. Methuen handbook of colour. , (1967).
- Ridgway, R. . Color Standards and Color Nomenclature. , (1912).
- McGinnis, M. R. . Laboratory Handbook of Medical Mycology. , (2012).
- Webster, J., Weber, R. . Introduction to Fungi. , (2007).
- Sridharan, G., Shankar, A. A. Toluidine blue: A review of its chemistry and clinical utility. Journal of Oral and Maxillofacial Pathology. 16 (2), 251-255 (2012).
- Smith, D., Onions, A. H. S. A comparison of some preservation techniques for fungi. Transactions of the British Mycological Society. 81 (3), 535-540 (1983).
- Ryan, M. J., Smith, D., Jeffries, P. A decision-based key to determine the most appropriate protocol for the preservation of fungi. World Journal of Microbiology and Biotechnology. 16 (2), 183-186 (2000).
- Lalaymia, I., Cranenbrouck, S., Declerck, S. Maintenance and preservation of ectomycorrhizal and arbuscular mycorrhizal fungi. Mycorrhiza. 24 (5), 323-337 (2014).
- Zettler, L. W., Corey, L. L. Orchid mycorrhizal fungi: isolation and identification techniques. Orchid Propagation: From Laboratories to Greenhouses-Methods and Protocols. , 27-59 (2018).
- Yu, S., Wang, Y., Li, X., Yu, F., Li, W. The factors affecting the reproducibility of micro-volume DNA mass quantification in Nanodrop 2000 spectrophotometer. Optik. 145, 555-560 (2017).
- Martos, F., et al. Independent recruitment of saprotrophic fungi as mycorrhizal partners by tropical achlorophyllous orchids. New Phytologist. 184 (3), 668-681 (2009).
- Schoch, C. L., et al. Nuclear ribosomal internal transcribed spacer (ITS) region as a universal DNA barcode marker for Fungi. Proceedings of the National Academy of Sciences. 109 (16), 6241-6246 (2012).
- White, T. J., Bruns, T., Lee, S., Taylor, J. Amplification and direct sequencing of fungal ribosomal RNA genes for phylogenetics. PCR Protocols: A Guide to Methods and Applications. 18 (1), 315-322 (1990).
- Sanger, F., Nicklen, S., Coulson, A. R. DNA sequencing with chain-terminating inhibitors. Proceedings of the National Academy of Sciences. 74 (12), 5463-5467 (1977).
- Ranjard, L., et al. Characterization of bacterial and fungal soil communities by automated ribosomal intergenic spacer analysis fingerprints: biological and methodological variability. Applied and Environmental Microbiology. 67 (10), 4479-4487 (2001).
- Metzker, M. L. Sequencing technologies-the next generation. Nature Reviews Genetics. 11 (1), 31-46 (2010).
Erratum
Formal Correction: Erratum: Isolation, Characterization, and Total DNA Extraction to Identify Endophytic Fungi in Mycoheterotrophic Plants
Posted by JoVE Editors on 1/01/1970. Citeable Link.
An erratum was issued for: Isolation, Characterization, and Total DNA Extraction to Identify Endophytic Fungi in Mycoheterotrophic Plants. The Authors section was updated from:
Juliana Lishcka Sampaio Mayer
to:
Juliana Lischka Sampaio Mayer
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved