Method Article
斑马鱼胚胎中小胶质细胞动力学的实时成像和表征
摘要
我们展示了一种方法,该方法利用快速扫描共聚焦显微镜对发育中的斑马鱼视盖中的小胶质细胞进行实时成像,从而可以在 体内分析这些细胞的动力学。
摘要
小胶质细胞是高度动态的细胞,它们在脑实质中的迁移和定植是大脑正常发育和功能的关键步骤。外部发育的斑马鱼胚胎具有光学透明度,这与荧光标记小胶质细胞的明确表征的转基因报告系一起,使斑马鱼成为此类研究的理想脊椎动物模型。在本文中,我们利用斑马鱼模型的独特功能来可视化 体内和生理 条件下小胶质细胞的动力学。我们使用共聚焦显微镜记录斑马鱼胚胎视盖中小胶质细胞的延时摄影,然后使用 IMARIS 10.0 软件提取跟踪数据,以获得细胞在不同发育阶段的迁移路径、平均速度和在视盖中的分布。该方案可以成为阐明小胶质细胞行为在各种情况下的生理意义的有用工具,有助于更深入地表征这些高度运动的细胞。
引言
作为中枢神经系统 (CNS) 中的常驻巨噬细胞,小胶质细胞代表了一种独特的非神经元群体,占成人大脑中所有神经胶质细胞的 15%。近年来,由于小胶质细胞生物学在发育、生理学和疾病1 中的重要性,研究它们越来越受到关注。在生理条件下,小胶质细胞高度动态,不断研究脑实质 2,3。这种行为允许小胶质细胞在大脑中定植并在其发育中发挥关键作用,例如塑造神经元回路4、突触修剪5 和血管发生6。此外,这种固有的动态性质使小胶质细胞能够持续监测 CNS 是否有感染、损伤或任何偏离体内平衡的迹象7。为了剖析这些错综复杂的细胞动力学,跨空间和时间的小胶质细胞实时成像是必不可少的。幸运的是,外部发育的斑马鱼胚胎的光学透明度,加上荧光标记小胶质细胞的明确表征的转基因报告系的可用性,使斑马鱼成为此类研究的理想脊椎动物模型。斑马鱼胚胎的实时成像提供了一种非侵入性方法,不需要手术或广泛的组织操作,最大限度地减少了对 CNS 状态的潜在干扰。在研究小胶质细胞时,这是一个关键的考虑因素,因为它们对细胞外环境中的细微变化也高度敏感8。
在这里,我们提供了成功跟踪斑马鱼胚胎中 3D 小胶质细胞运动的指南,从而可以前所未有的了解发育中的脑实质完整结构内的小胶质细胞行为(参见 图 1 的协议图形概述)。该分步协议详细介绍了如何在不同的发育阶段设置斑马鱼小胶质细胞并对其进行成像,以及如何提取有关小胶质细胞运动的高分辨率数据,以提供对其迁移模式和对环境线索的反应的宝贵见解。我们还证明该方案可以适应进行实时多色成像,从而将其适用性扩展到研究小胶质细胞与标记邻近细胞的转基因系相结合,包括神经元3、少突胶质细胞9 和内皮细胞10(如图 2 所示)。通过添加到允许人们直接实时和自然环境中观察和表征小胶质细胞行为动态的工具箱中,该协议可能有助于更好地阐明小胶质细胞在生理学和疾病早期发育过程中的功能。
研究方案
根据FELASA42和机构(比利时布鲁塞尔自由大学;ULB) 指南和法规。所有实验程序均已获得 ULB 的 ULB 动物福利伦理委员会 (CEBEA) 的批准。
1. 斑马鱼饲养和胚胎制备
注意:在巨噬细胞(包括小胶质细胞)中表达荧光 eGFP 蛋白的斑马鱼转基因系 Tg(mpeg1:eGFP)gl22 用于生成本协议中所示的跟踪数据。其他巨噬细胞报告基因系可从 ZIRC 获得,也可以使用。在选择转基因系时,重要的是要考虑到高信号强度将促进图像采集和细胞分割。
- 将斑马鱼 (Danio rerio) 保持在 28 °C 的温度下,并按照既定方案管理它们11.
- 在所需胚胎出生日期之前的下午晚些时候,将雌鱼和雄鱼一起放入带有网状底部和隔板的繁殖池中,刺激交配。第二天早上取下分离器,为鱼的交配计时。
- 使用网收集受精卵,并将其转移到含有 E3 培养基(5 mM NaCl、0.17 mM KCl、0.33 mM CaCl2、0.33 mM MgSO4)和 0.01% 亚甲蓝的 100 mm 培养皿中。根据所使用的斑马鱼转基因系的遗传背景,如果要抑制色素形成以促进成像,则从受精后 6 小时 (hpf) 开始,将 0.003% 的 N-苯硫脲 (PTU) 添加到胚胎发育的 E3 缓冲液中,每 2 天刷新一次溶液,直到达到所需的发育阶段。
注:为避免长时间暴露于可能影响胚胎发育的 PTU12,建议在色素缺陷背景上使用转基因系(如果有),例如 Casperw2,这样就可以进行清晰的成像,而无需将胚胎长时间暴露在 PTU中 13。 - 如果要成像的胚胎在受精后 3 天 (dpf) 之前,请在 24 hpf 下对其进行去绒毛膜。使用立体显微镜确认所有胚胎都已去绒毛膜并且未因酶处理而受损(参见步骤 1.4.2)。
- 如果鸡蛋数量有限,请使用精细解剖钳捏住绒毛膜并向外拉,小心地将每个胚胎从绒毛膜中释放出来。
- 如果鸡蛋数量较多,则通过在室温 (RT) 下将鸡蛋在 E3 培养基中的 1 mg/mL 链霉蛋白酶溶液中散装孵育短时间(通常为 5-10 分钟,直到绒毛膜在轻轻漱口下破裂)进行去绒毛膜化。接下来,弃去链霉蛋白酶溶液,用新鲜的 E3 彻底冲洗胚胎 3 次,以去除任何残留的绒毛膜和残留的酶。
2. 斑马鱼安装
- 在 E3 缓冲液中制备 1% 低熔点琼脂糖溶液。在台式加热块中将琼脂糖冷却至 37 °C,并加入新鲜制备的 Tricaine methanesulfonate 溶液 (160 mg/L),这将有助于在成像过程中保持胚胎麻醉。涡旋溶液以使其彻底均质。
- 通过在 E3 缓冲液中将 160 mg/L 甲磺酸三卡因添加到含有胚胎的培养皿中来麻醉胚胎。检查以验证运动是否已停止,并选择具有所需荧光的斑马鱼胚胎。通过使用明场体视显微镜观察胚胎的心跳来检查胚胎的健康状况。使用宽头移液管吸取一个或最多三个选定的胚胎,并将它们转移到底部玻璃成像板上。
- 在立体显微镜下,使用移液管去除尽可能多的培养基,注意不要触摸或损坏胚胎;然后,用先前制备的 1% 低熔点琼脂糖溶液替换培养基。接下来,使用固定在挑逗针末端的塑料细锥形尖端轻轻地将每个胚胎推到成像板的底部,调整其方向,使其背面面向玻璃,以确保良好的视柱状结构,并在胚胎和玻璃之间提供最少量的低熔点琼脂糖。
- 使用具有高质量 Plan Apo 物镜(此处使用的 10x/0.45 Plan Apo 或 20x/0.75 Plan Apo)的倒置共聚焦显微镜立即对安装的胚胎进行成像。如果延时摄影将长时间运行(超过一个小时),请在底部玻璃成像板上加入一滴或两滴含甲磺酸三卡因 (160 mg/L) 的 E3 缓冲液,以防止低熔点琼脂糖过度干燥。
注:在将 E3 缓冲液和甲磺酸三卡因培养基添加到成像盘中之前,等待琼脂糖完全凝固至关重要;否则,鱼可能会缓慢地横向和轴向移动,从而导致延时摄影中的样品漂移。 - 继续设置所需时间的延时采集:将图像分辨率设置为 1,024 x 1,024,像素尺寸为 0.49 μm。以 2-3 μm 的增量收集光学切片,以确保在 z 轴上具有可接受的分辨率。在斑马鱼胚胎中,根据所使用的发育阶段,将视盖封闭在 100-150 μm 的 z 堆栈中。为确保 cell 跟踪成功,请将帧之间的时间间隔保持在 30 秒到 60 秒之间。
3. 跟踪分析和数据导出
- 使用 Imaris 文件转换器软件将延时文件转换为 IMS 格式(参见 材料表)。启动软件后,将文件拖放到输入区域或使用 添加文件... 按钮手动选择它们。在 Output (输出 ) 菜单中,指定转换文件的位置,选择与输入文件相同的文件夹或指定特定文件夹。通过单击“ 设置体素大小 ”按钮确保显示的体素大小的尺寸正确,验证体素大小后,按“ 全部启动 ”按钮开始文件转换(有关所有数据分析的分步指南,请参阅 补充文件 1 )。
注意:如果在两个或多个胚胎上并行执行延时摄影,则文件将自动拆分为单个位置,将每个胚胎分成不同的文件。可以在批处理模式下使用该软件并同时上传和转换多个文件。IMS 文件将被创建并保存在选定的目标文件夹或输入文件所在的文件夹中。 - 启动后,让分析软件在 Arena 中启动;点击 观察文件夹 打开之前转换的文件。将文件加载到软件中后,使用位于视图区域左侧的切片工具栏中的滑块,使用切片视图滚动 z 堆栈,并使用指针绘制所选单元格的直径来测量一些单元格直径。单击并拖动鼠标以绘制跨越单元核心的线段;观察在 Measure 工具栏中绘制的线段的长度,该工具栏位于 View Area 的右侧。
注:使用步骤 2.5 中的成像设置,细胞体直径预计在 6 μm 和 8 μm 之间。 - 要自动检测单元格,请返回 3D 视图并单击对象工具栏中的点图标,在对象列表中添加新的点对象,然后打开自动点创建向导。在向导的第一步中,勾选仅分割感兴趣区域的选项。然后,启用 Track Spots (over time) 选项来计算追踪数据,以及启用 Object-Object Statistics 以允许在点之间进行比较并扩展稍后分析中可用的数据范围。最后,单击向导窗口右下角的 Next 按钮继续向导。
- 在第二步中,继续指定包围胚胎视盖的感兴趣区域 (ROI) 区域。通过单击并拖动 ROI 每个面上的白色小箭头来修改 ROI 的大小。定义 ROI 大小后,通过调整时间间隔将其扩展到所有录制的帧。
- 然后,选择用于检测细胞的通道作为 源通道 ,并将之前在步骤 3.2 中获得的细胞直径测量值设置为 估计的 XY 直径。启用 背景减法 选项,然后单击 下一步.
- 此时,在 Spot 创建向导中,软件包含一个质量过滤步骤,其中根据在每个光斑中心测量的信号强度过滤斑点;在检测到斑点的通道中。通过在阈值下限和阈值上限框的数据字段中输入值,或通过拖动创建向导底部显示的直方图中相应的彩色线条来调整强度阈值,以便检测所有细胞,包括位于组织更深处的细胞,这些细胞通常显示较低的信号强度。所有这些更改在 View Area 中同时可见。
注意:此时可以观察到多个标记单个细胞的点,但可以在稍后的分析中纠正这一点。
在较长的延时摄影中,如果看到一些光漂白,请在设置阈值之前检查多个帧的 View Area (视图区域 ),以确保在最近的帧中也能检测到所有单元格,因此也会检测到较暗的帧。 - 满意后,单击 Next 以验证选定的质量阈值。所有满足质量阈值参数且直径与估计的 XY 直径匹配的光斑都将被标记为光斑。将其可视化为 视图区域中的预览,其中满足这两个参数的点将叠加在源通道上。
- 在下一步中,选择 Autoregressive Motion 作为跟踪算法,以跟踪具有或多或少连续运动的单元格。通过仔细观察两帧之间点的运动来判断单元格在两个时间点之间移动的最长时间,并在 Max Distance 字段中输入估计数字。这是允许一个点偏离预测的未来位置的距离。
- 将 Maximum Gap size (最大间隙 大小) 设置为该点可以在不中断轨道的情况下消失的最小时间点数。如果帧之间的间隔 小于 60 秒,请使用 大小 3。如果帧之间的时间间隔较长,请增加允许的最大间隙大小,以减少轨道碎片。
- 启用 Fill gaps with all detected objects(用所有检测到的对象填充间隙 ),以将检测阈值降低到接近跟踪算法预测的预期位置,并连接因点检测失败而中断的轨迹。最后,点击 Next 并让软件为所有先前生成的点自动生成轨迹。
- 如果获得的轨迹看起来太多或碎片化,则返回 创建向导,单击 Previous 按钮,并调整之前定义的最大距离,增加或减少数量,直到大多数获得的轨迹代表单元运动。
注意:轨迹的数量应与小胶质细胞的数量大致匹配,因此,范围在 30 到 50 之间,具体取决于胚胎的年龄。但是,在完成分析之前,由于错误的轨迹,预计会有更高的数字。 - 计算出轨迹后,该软件会包含一个过滤步骤来过滤掉所有非常简短的轨迹,因为它们通常不准确或信息量不足。通过直接在数据字段中输入轨道持续时间过滤器 下限 的值,或者通过在 创建向导底部显示的直方图中拖动相应的彩色线条,排除短于 3-8 分钟 (min) 的轨道。单击 Next 验证过滤器并继续创建向导。
注意:如前所述,通过修改最大 (max) 和最小 (min) 轨道持续时间阈值,可以预览将被过滤并从分析中排除的轨道。然后,可以将过滤后的轨道复制到新对象中,以便进一步分析。筛选的确切时间阈值可能会因上一个 创建向导 步骤的结果而异。我们建议通过查看 View Area (查看区域 ) 中的预览来微调这些过滤器设置,以获得最佳结果。 - 由于 Tg(mpeg1:eGFP) 转基因系标记所有单核吞噬细胞,因此除小胶质细胞外,皮肤巨噬细胞也会在延时电影中检测到。从分析中手动删除皮肤巨噬细胞生成的轨迹,以专注于脑实质小胶质细胞(图 3)。
- 在轨迹编辑器中手动更正轨迹中的其他潜在错误,其中每个数据点都显示为轨迹中的一个对象。激活 View Area 右侧的圆形选择模式按钮,并高亮显示需要修改或调整的轨道。选择有问题的轨道后,使用 connect 和 disconnect 按钮对其进行编辑以正确表示单元格移动。使用轨道编辑器还可以选择和删除错误地表示同一单元格的重复的单个点或小轨道片段,方法是使用圆形选择模式选择它们,然后按 Canc 或 Delete 键来消除它们。
注意:如果跟踪设置正确,则只有一小部分跟踪的单元格需要此步骤。 - 如果小胶质细胞报告基因系与标记薄壁组织中其他细胞类型的荧光转基因联合使用,则作为新的表面对象或点对象进行分割以可视化它们。为此,请重复上述步骤(从步骤 3.2 到 3.14),修改步骤 3.5 中的 源通道 输入,使其成为用于对第二个细胞群进行成像的输入,并注意调整特定于感兴趣细胞的描述性参数,例如 估计的 XY 直径 (步骤 3.5)、 最大距离 (步骤 3.8)到新的细胞群。其他步骤可以重复进行,无需更改。
注意:在创建新的表面时, 估计的 XY 直径 将替换为基于 背景减去 的阈值选项,其中要给出的输入是 适合对象的最大球体的直径。可以使用 Measure (度量) 工具栏 从 Slice (切片) 视图中估计此值,如步骤 3.2 中所述。 - 对获得的跟踪感到满意后,从 Statistic 选项卡。单击 设置 按钮,选择将为之前创建的每个点对象或表面对象计算的统计数据。如果在同一分析中创建了多个对象,即两个点对象或一个点和一个表面对象,则描述一个对象与另一个对象的相对位置的统计数据 (例如两个对象之间的最短距离) 也将可用于导出。
注意:所有所需的值都可以是特定值或平均值,并且所有原始数据都可以使用 “统计 ”选项卡底部的按钮以逗号分隔值 (CSV) 文件的形式下载。如果分析还包括其他像元类型,则还将提供有关一个对象到另一个对象相对距离的其他统计数据。
结果
表达绿色荧光蛋白 (eGFP) 的小胶质细胞和在 Tg 中表达 DsRed 的内皮细胞(mpeg1:eGFPgl22; kdrl:cres898; actb2:loxP-STOP-loxP-DsRedexpress,sd5) 三重转基因胚胎14 根据所描述的方案在 3 dpf 下成像。将单个斑马鱼胚胎封片在底部玻璃板上的 1% 低熔点琼脂糖中,成像过程在采集期间不会阻碍胚胎的生长。使用配备 10x 0.45 NA 干物镜的商用点扫描共聚焦显微镜系统记录延时摄影,488 nm 和 561 nm 激发激光器分别用于小胶质细胞和内皮细胞成像。此外,时间间隔、图像分辨率、像素大小和 z 步长分别为 30 秒 (s)、1024x1024、0.49 μm 和 2.5 μm。延时摄影持续了 3.5 小时 (h)。
获得的视野覆盖了胚胎的整个头部,但分析特别集中在视盖上,因为在发育的第一周,小胶质细胞主要局限于背中脑该区域的神经元胞体层,使研究人员能够同时可视化整个群体15。如上所述,使用 Imaris 10.0 执行 3D 跟踪分析。如图 4 所示,跟踪成功,产生 25 个轨迹,与 3 dpf16 时视壳中存在的小胶质细胞的预期数量相匹配。需要最少的手动轨道校正。 图 5 显示了可以从成功的跟踪实验中提取的数据示例。巨噬细胞和内皮细胞的同时标记可以量化小胶质细胞相对于后者的位置,使研究人员能够及时可视化每个细胞与最近的内皮细胞的相对距离,并检查潜在相互作用的频率和数量(图 5)。
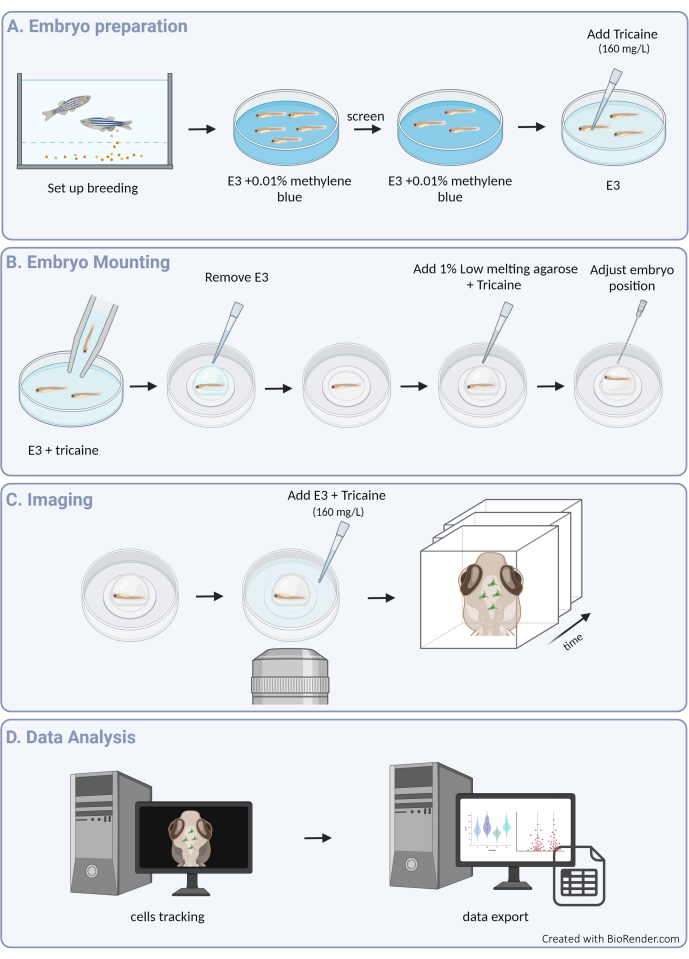
图 1:实验程序概述。 (A) 斑马鱼胚胎制备和麻醉。(B) 样品安装和定位。(C) 图像采集。(D) 图像处理和运动数据的提取。 请单击此处查看此图的较大版本。
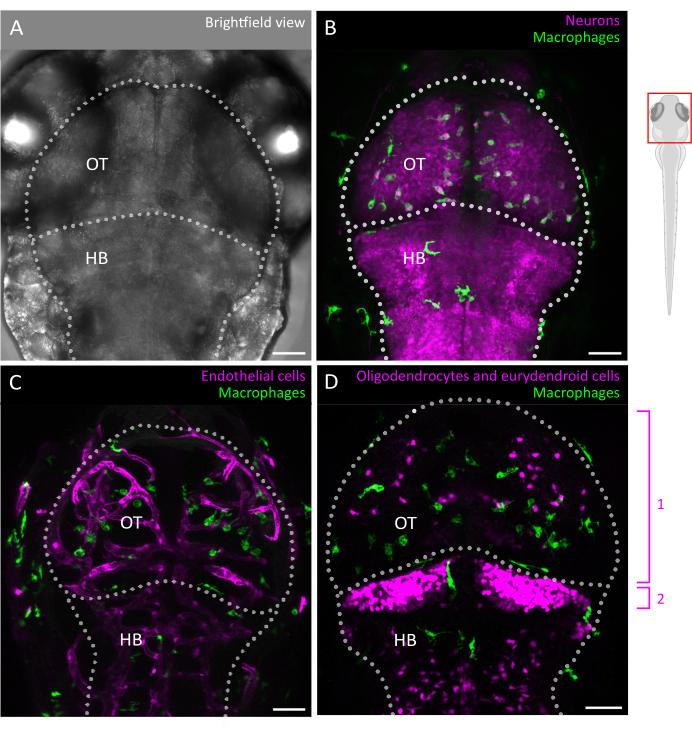
图 2:斑马鱼胚胎中小胶质细胞成像和与脑细胞环境的相互作用。 (A) 3 dpf 胚胎斑马鱼头部和大脑的明场图像(背视图),标记了视盖和后脑。由于斑马鱼的体积小且光学透明,胚胎斑马鱼的大脑在这个阶段可以完整成像。(B - D)小胶质细胞的位置和行为可以通过使用 (B,C) Tg(mpeg1:eGFP)gl22 和 (D) Tg(mpeg1:mcherry)gl23 等转基因品系来可视化,并专注于 OT。(B) 可以使用报告基因系 Tg(XlTubb:D sRed)zf148 识别神经元及其细胞体,并且可以通过将两个通道合并为共表达两个转基因的细胞系来可视化小胶质细胞-神经元相互作用。Seen 是绿色(小胶质细胞)和红色(神经元,品红色)信号的合并。(C,D)报告基因系还可以阐明小胶质细胞(绿色)与内皮细胞的相互作用,此处使用 (C) 双 Tg(kdrl:cres898; actb2:LOXP-STOP-LOXP-Dsredexpress,SD5)转基因,或与神经胶质细胞如少突胶质细胞及其前体一起使用,使用 (D) 双 Tg(olig2:EGFP; MPEG1:mCherry ) 转基因系。1:OT 中的少突胶质细胞和祖细胞,2:小脑中的细突状神经元。比例尺 = 50 μm。缩写:dpf = 受精后天数;OT = 视盖;HB = 后脑。 请单击此处查看此图的较大版本。
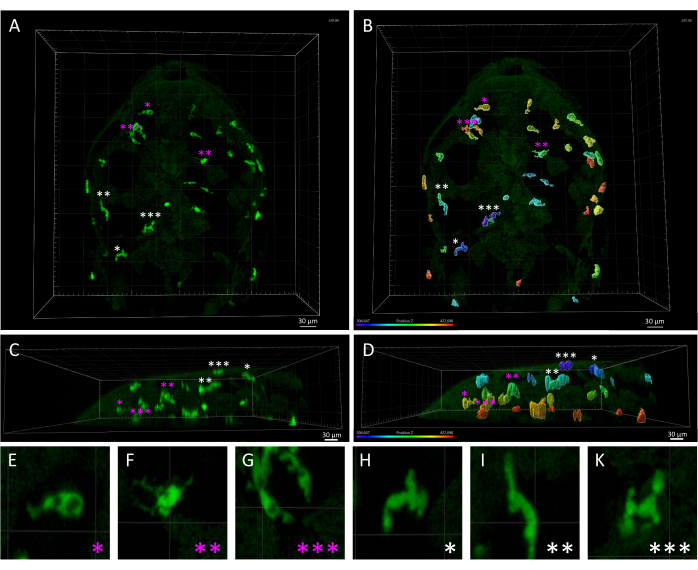
图 3:3 dpf 斑马鱼胚胎中皮肤巨噬细胞和小胶质细胞之间的形态和空间差异。 (A,B) 3 dpf 的 Tg(mpeg1:eGFP)gl22 斑马鱼胚胎的背视图,描绘了 (A) 实质小胶质细胞(以洋红色星号标记)与皮肤巨噬细胞(白色星号),根据它们沿 z 轴的相对位置进行识别,如 (B) 所示),该图像显示使用 z 轴颜色编码渲染的单色 3D 图像。(C,D)(C) A 和 (D) B 的侧视图,展示了胚胎头部内 mpeg1:eGFP+ 细胞的 z 深度,并与小胶质细胞相比突出了皮肤巨噬细胞的浅表定位。(E-K)每个细胞的高放大倍率由 A 和 B 中的星号表示,提供了 (E-G) 变形虫小胶质细胞和 (H-K) 细长皮肤巨噬细胞之间不同形态的详细可视化。比例尺 = 30 μm。缩写:dpf = 受精后天数。 请单击此处查看此图的较大版本。
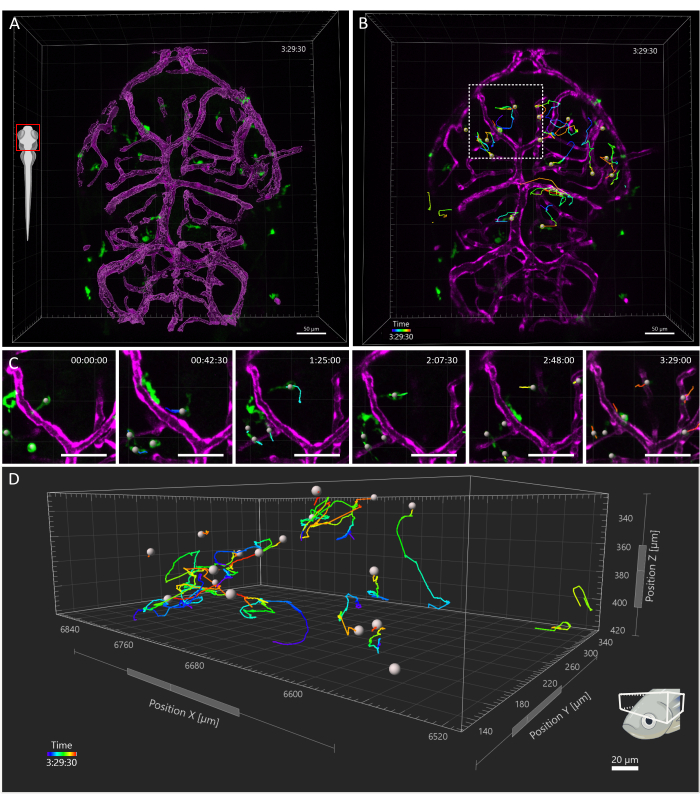
图 4:追踪 3 dpf 斑马鱼胚胎中的小胶质细胞。 (A) 3 dpf Tg(kdrl:cres898; 行为b2:LOXP-STOP-LOXP-Dsredexpress,sd5;MPEG1:eGFPgl22) 三重转基因胚胎,显示小胶质细胞(绿色)和血管表面渲染(品红色)。(B - D)在 (B) 3.5 小时时间窗口内对小胶质细胞运动的代表性跟踪。可以看到单个细胞遵循复杂的轨迹。(C) 延时摄影的细节,显示虚线方块包围的 B 中区域的放大视图。展示了电影中的六帧(间隔 45 分钟),记录了小胶质细胞与其微环境中的内皮细胞建立瞬时接触。(D) 单个小胶质细胞在视盖内迁移的轨迹,其中 X 、 Y 和 Z 轴表示空间维度。比例尺 = 50 μm。缩写:dpf = 受精后天数。 请单击此处查看此图的较大版本。
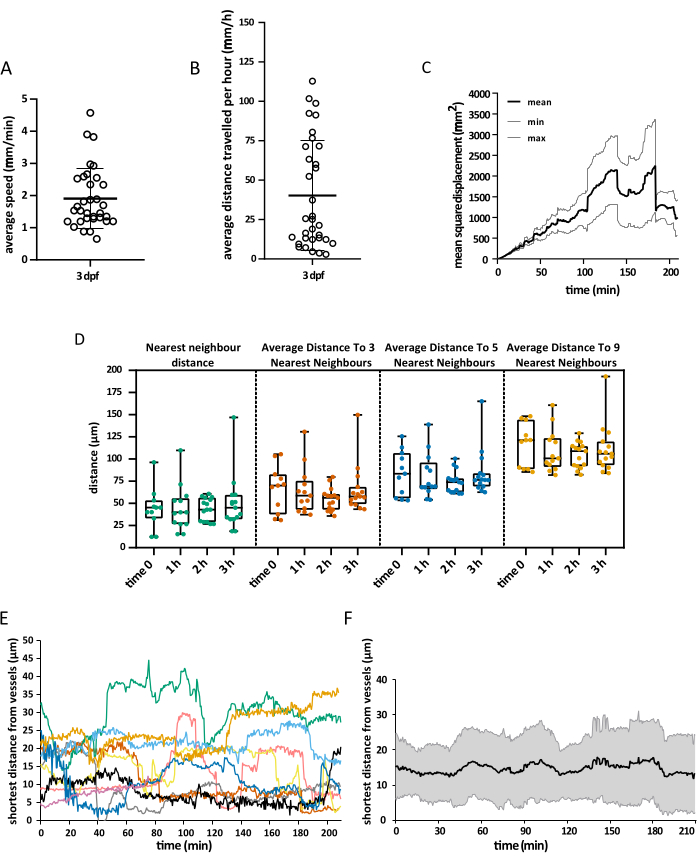
图 5:所获数据的可视化。 可以使用所述协议获取的空间数据示例。这些图表描述了 (A) 小胶质细胞的平均速度,(B) 它们在 1 小时内覆盖的平均距离,(C) 它们的均方位移,以及 (D) 它们在不同时间在空间中的分布。该容器的表面渲染还允许在任何给定时间测量小胶质细胞和内皮细胞之间的最短距离,无论是在 (E) 单细胞水平还是作为 (F) 全球平均值。对于 A、B 和 D,每个点代表一个单独的单元格。N = 一个胚胎。 请单击此处查看此图的较大版本。
补充文件 1:请单击此处下载此文件。
讨论
目前的协议能够对脊椎动物胚胎中的小胶质细胞动力学进行体内成像,并对获得的运动数据进行可视化。发育中的大脑的小胶质细胞定植发生在胚胎发生的早期,并且先于关键事件,例如神经发生高峰、星形胶质细胞生成、少突生成和许多其他细胞过程17。因此,小胶质细胞在塑造大脑发育的特定方面发挥重要作用18 也就不足为奇了,例如,通过调节神经元分化、迁移和存活 19,20,21,以及突触修剪5 和髓鞘形成 22,23,24。
功能失调的小胶质细胞对神经发育障碍的发病机制和/或进展的贡献也越来越得到认可25。事实上,小胶质细胞在形成大脑中的早期存在使这些细胞暴露于不同的生理状态26 和环境变化中。鉴于小胶质细胞在啮齿动物和人类中都是长寿细胞,在生命周期内通过当地祖细胞的自我更新来维持,这可能会产生重大影响 27,28,29。我们相信该协议可以作为一个强大的工具,更好地描述小胶质细胞在这些不同生理状态下的行为,因为它们在大脑形态发生的连续步骤中发育、成熟和建立网络。
使用此处描述的设置,我们已经成功地对年龄为 6 dpf 的斑马鱼幼虫进行成像和获取数据。将分析扩展到后期开发阶段可能会成功,但需要调整成像设置以考虑增加的样本量,尤其是沿 z 轴。在尝试这样做时,我们建议专注于保持低信噪比和快速扫描时间,因为它们是成功分析的关键参数。
我们建议最小成像时间为 1 小时,以便进行小胶质细胞跟踪;使用此协议测试的最长成像窗口为 8 小时。此外,跟踪分析必须使帧之间的时间间隔尽可能短,最好在 30 秒到 60 秒之间。这将允许在下游分析中获得更准确和详细的跟踪数据。因此,特别是在检测多个荧光团时,避免光谱重叠并确保两个荧光团发射光谱之间有足够的分离,以便同时进行采集,而不会发生信号渗漏。
其他用于斑马鱼大脑高质量延时记录的协议可用30,但这是第一个显示如何在胚胎发育过程中长时间成功跟踪所有小胶质细胞运动的协议。尽管此处介绍的工作流程侧重于在生理环境中跟踪小胶质细胞,但它可以很容易地应用于病理学中小胶质细胞的分析。事实上,几种神经发育障碍模型,如自闭症31、癫痫32 和精神分裂症33,以及神经变性34 和癌症35,已经在斑马鱼中建立,为确定疾病条件下的小胶质细胞反应和行为提供了独特的机会。
值得注意的是,这种追踪方案具有高度通用性,也有助于阐明斑马鱼胚胎不同解剖区域的各种细胞类型的迁移模式,从而有可能为本文描述的小胶质细胞研究范围之外的其他应用开辟途径。此外,通过利用组合多个荧光转基因系的能力,我们获得了辨别小胶质细胞与大脑微环境的其他细胞类型之间的空间关系的能力,并有可能以非侵入性的方式在整个延时记录中可视化细胞相互作用和串扰。这可能有助于揭示小胶质细胞行为的生理意义,并有助于更深入地表征这些高度运动的细胞。
披露声明
作者声明没有利益冲突。
致谢
作者衷心感谢 Nicolas Bayens 教授慷慨地提供本研究所必需的共聚焦显微镜。这项工作部分由科学研究基金 (FNRS) 资助,资助编号为 F451218F 和 UG03019F,阿尔茨海默病研究基金会 (SAO-FRA)(至 V.W.),A.M. 得到了 FNRS 研究奖学金的支持。 图 1 是在 biorender.com 创建的。
材料
| Name | Company | Catalog Number | Comments |
| 1 L Breeding tanks | Tecniplast | ZB10BTE | |
| 1-phenyl-2-thiourea (PTU) | Sigma-Aldrich | P7629 | Diluted to 0.2 mM in E3 to prevent embryo pigmentation |
| Bottom glass imaging dish | FluoroDish | FD3510-100 | |
| Disposable Graduated transfer pipette | avantor | 16001-188 | |
| Dry block heater | Novolab | Grant QBD4 | To keep low melting agarose at 37 °C |
| Ethyl 3-aminobenzoate methanesulfonate (Tricaine) | Sigma-Aldrich | E10521-50G | |
| Imaris 10.0 | Oxford Instruments | analysis software | |
| Imaris File Converter | Oxford Instruments | https://imaris.oxinst.com/big-data | |
| Laser-scanning confocal microscope | Nikon | Eclipse Ti2-E | |
| Methylene blue | Sigma-Aldrich | M9140-25G | |
| microloader tips | Eppendorf | 5242956003 | |
| NuSieve GTG Agarose | Lonza | 50081 | |
| Petri dishes (90 mm) | avantor | 391-0559 | |
| Pronase | Sigma-Aldrich | 11459643001 | |
| Stainless Steel Forceps Dumont No. 5 | FineScienceTools | 11254-20 | |
| Stereo microscope | Leica | Leica M80 | To mount the embryos |
| teasing needle | avantor | 76549-024 |
参考文献
- Wolf, S. A., Boddeke, H. W., Kettenmann, H. Microglia in physiology and disease. Annu Rev Physiol. 79, 619-643 (2017).
- Nimmerjahn, A., Kirchhoff, F., Helmchen, F. Resting microglial cells are highly dynamic surveillants of brain parenchyma in vivo. Sci Rep. 308 (5726), 1314-1318 (2005).
- Peri, F., Nusslein-Volhard, C. Live imaging of neuronal degradation by microglia reveals a role for v0-ATPase a1 in phagosomal fusion in vivo. Cell. 133 (5), 916-927 (2008).
- Marın-Teva, J. L., et al. Microglia promote the death of developing Purkinje cells. Neuron. 41 (4), 535-547 (2004).
- Paolicelli, R. C., et al. Synaptic pruning by microglia is necessary for normal brain development. Science. 333 (6048), 1456-1458 (2011).
- Hattori, Y. The microglia-blood vessel interactions in the developing brain. Neurosci Res. 187, 58-66 (2023).
- Colonna, M., Butovsky, O. Microglia function in the central nervous system during health and neurodegeneration. Annu Rev Immunol. 35, 441-468 (2017).
- Xu, H. -T., Pan, F., Yang, G., Gan, W. -B. Choice of cranial window type for in vivo imaging affects dendritic spine turnover in the cortex. Nat Neurosci. 10 (5), 549-551 (2007).
- Shin, J., Park, H. -C., Topczewska, J. M., Mawdsley, D. J., Appel, B. Neural cell fate analysis in zebrafish using olig2 BAC transgenics. Methods Cell Sci. 25 (1), 7-14 (2003).
- Chi, N. C., et al. Foxn4 directly regulates tbx2b expression and atrioventricular canal formation. Genes Dev. 22 (6), 734-739 (2008).
- Avdesh, A., et al. Regular care and maintenance of a zebrafish (Danio rerio) laboratory: An introduction. J Vis Exp. (69), e4196(2012).
- Bohnsack, B. L., Gallina, D., Kahana, A. Phenothiourea sensitizes zebrafish cranial neural crest and extraocular muscle development to changes in retinoic acid and IGF signaling. PLoS ONE. 6 (8), e22991(2011).
- Ariga, J., Walker, S. L., Mumm, J. S. Multicolor time-lapse imaging of transgenic zebrafish: visualizing retinal stem cells activated by targeted neuronal cell ablation. J Vis Exp. (43), e2093(2010).
- Ferrero, G., et al. Embryonic microglia derive from primitive macrophages and are replaced by cmyb-dependent definitive microglia in zebrafish. Cell Rep. 24 (1), 130-141 (2018).
- Xu, J., Wang, T., Wu, Y., Jin, W., Wen, Z. Microglia colonization of developing zebrafish midbrain is promoted by apoptotic neuron and lysophosphatidylcholine. Dev Cell. 38 (2), 214-222 (2016).
- Svahn, A. J., et al. Development of ramified microglia from early macrophages in the zebrafish optic tectum. Dev Neurobiol. 73 (1), 60-71 (2013).
- Thion, M. S., Garel, S. Microglial ontogeny, diversity and neurodevelopmental functions. Curr Opin Genet Dev. 65, 186-194 (2020).
- Bohlen, C. J., Friedman, B. A., Dejanovic, B., Sheng, M. Microglia in brain development, homeostasis, and neurodegeneration. Annu Rev Genet. 53, 263-288 (2019).
- Squarzoni, P., et al. Microglia modulate wiring of the embryonic forebrain. Cell Rep. 8 (5), 1271-1279 (2014).
- Schafer, D. P., et al. Microglia sculpt postnatal neural circuits in an activity and complement-dependent. Neuron. 74 (4), 691-705 (2012).
- Parkhurst, C. N., et al. Microglia promote learning-dependent synapse formation through brain-derived neurotrophic factor. Cell. 155 (7), 1596-1609 (2013).
- Green, L. A., O'Dea, M. R., Hoover, C. A., DeSantis, D. F., Smith, C. J. The embryonic zebrafish brain is seeded by a lymphatic-dependent population of mrc1+ microglia precursors. Nat. Neurosci. 25 (7), 849-864 (2022).
- Hughes, A. N., Appel, B. Microglia phagocytose myelin sheaths to modify developmental myelination. Nat Neurosci. 23 (9), 1055-1066 (2020).
- Yu, Q., et al. C1q is essential for myelination in the central nervous system (CNS). iScience. 26 (12), 108518(2023).
- Zengeler, K. E., Lukens, J. R. Innate immunity at the crossroads of healthy brain maturation and neurodevelopmental disorders. Nat Rev Immunol. 21 (7), 454-468 (2021).
- Paolicelli, R. C., et al. Microglia states and nomenclature: A field at its crossroads. Neuron. 110 (21), 3458-3483 (2022).
- Réu, P., et al. The lifespan and turnover of microglia in the human brain. Cell Rep. 20 (4), 779-784 (2017).
- Ajami, B., Bennett, J. L., Krieger, C., Tetzlaff, W., Rossi, F. M. V. Local self-renewal can sustain CNS microglia maintenance and function throughout adult life. Nat Neurosci. 10 (12), 1538-1543 (2007).
- Mildner, A., et al. Microglia in the adult brain arise from Ly-6ChiCCR2+ monocytes only under defined host conditions. Nat Neurosci. 10 (12), 1544-1553 (2007).
- Cook, Z. T., Brockway, N. L., Weissman, T. A. Visualizing the developing brain in living zebrafish using Brainbow and time-lapse confocal imaging. J Vis Exp. (157), e60593(2020).
- Tayanloo-Beik, A., et al. Zebrafish modeling of autism spectrum disorders, current status and future prospective. Front Psychiatry. 13, 911770(2022).
- D'Amora, M., et al. Zebrafish as an innovative tool for epilepsy modeling: State of the art and potential future directions. Int J Mol Sci. 24 (9), 7702(2023).
- Langova, V., Vales, K., Horacek, J. The role of zebrafish and laboratory rodents in schizophrenia research. Front Psychiatry. 11, 549232(2020).
- Chia, K., Klingseisen, A., Sieger, D., Priller, J. Zebrafish as a model organism for neurodegenerative disease. Front Mol Neurosci. 15, 940484(2022).
- Astell, K. R., Sieger, D. Zebrafish in vivo models of cancer and metastasis. Cold Spring Harb Perspect Med. 10 (8), 037077(2020).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。