Method Article
Imagerie en direct et caractérisation de la dynamique de la microglie chez l’embryon de poisson-zèbre
Dans cet article
Résumé
Nous faisons la démonstration d’une méthode qui tire parti de la microscopie confocale à balayage rapide pour effectuer une imagerie en direct des cellules microgliales dans le tectum optique du poisson-zèbre en développement, permettant ainsi l’analyse de la dynamique de ces cellules in vivo.
Résumé
Les microglies sont des cellules très dynamiques et leur migration et colonisation du parenchyme cérébral est une étape cruciale pour le bon développement et le bon fonctionnement du cerveau. Les embryons de poisson-zèbre en développement externe possèdent une transparence optique, ce qui, avec des lignées signalées transgéniques bien caractérisées qui marquent par fluorescence la microglie, fait du poisson-zèbre un modèle de vertébré idéal pour de telles études. Dans cet article, nous tirons parti des caractéristiques uniques du modèle du poisson-zèbre pour visualiser la dynamique des cellules microgliales in vivo et dans des conditions physiologiques. Nous utilisons la microscopie confocale pour enregistrer un timelapse de cellules microgliales dans le tectum optique de l’embryon de poisson-zèbre, puis nous extrayons des données de suivi à l’aide du logiciel IMARIS 10.0 pour obtenir la voie de migration des cellules, la vitesse moyenne et la distribution dans le tectum optique à différents stades de développement. Ce protocole peut être un outil utile pour élucider la signification physiologique du comportement de la microglie dans divers contextes, contribuant ainsi à une caractérisation plus approfondie de ces cellules très mobiles.
Introduction
En tant que macrophages résidents dans le système nerveux central (SNC), les microglies représentent une population non neuronale distincte qui représente jusqu’à 15 % de toutes les cellules gliales du cerveau adulte. L’étude de la biologie de la microglie a suscité une attention croissante ces dernières années en raison de son importance établie dans le développement, la physiologie et la maladie1. Dans des conditions physiologiques, les cellules microgliales sont très dynamiques, surveillant en permanence le parenchyme cérébral 2,3. Ce comportement permet à la microglie de coloniser le cerveau et de jouer des rôles clés dans son développement, tels que la formation des circuits neuronaux4, l’élagage synaptique5 et la vasculogenèse6. De plus, cette nature dynamique inhérente permet à la microglie de surveiller en permanence le SNC à la recherche de signes d’infection, de blessure ou de tout écart par rapport à l’homéostasie7. Pour disséquer ces dynamiques cellulaires complexes, l’imagerie en direct de la microglie à travers l’espace et le temps est indispensable. Heureusement, la transparence optique des embryons de poisson-zèbre en développement externe, associée à la disponibilité de lignées rapporteures transgéniques bien caractérisées qui marquent par fluorescence la microglie, positionne le poisson-zèbre comme un modèle de vertébré idéal pour de telles recherches. L’imagerie en direct chez les embryons de poisson-zèbre offre une approche non invasive qui ne nécessite pas de chirurgie ou de manipulation tissulaire importante, minimisant ainsi les perturbations potentielles de l’état du SNC. Il s’agit d’une considération essentielle lors de l’étude des cellules microgliales, car elles sont très sensibles aux changements même subtils de l’environnement extracellulaire8.
Ici, nous fournissons une ligne directrice pour suivre avec succès les mouvements des cellules microgliales 3D dans l’embryon de poisson-zèbre, permettant une vue sans précédent du comportement de la microglie dans l’architecture intacte du parenchyme cérébral en développement (voir la figure 1 pour un aperçu graphique du protocole). Ce protocole étape par étape détaille comment configurer et imager la microglie du poisson-zèbre à différents stades de développement et comment extraire des données à haute résolution sur la motilité des cellules microgliales afin de fournir des informations précieuses sur leurs schémas migratoires et leurs réponses aux signaux environnementaux. Nous démontrons également que ce protocole peut être adapté pour effectuer une imagerie multicolore en direct, étendant ainsi son applicabilité à l’étude de la microglie en combinaison avec des lignées transgéniques qui marquent les cellules voisines, y compris les neurones3, les oligodendrocytes9 et les cellules endothéliales10 (comme le montre la figure 2). En s’ajoutant à la boîte à outils qui permet d’observer et de caractériser directement la dynamique du comportement des microglies en temps réel et dans leur environnement naturel, ce protocole contribuera probablement à mieux élucider la fonctionnalité des microglies au cours du développement précoce, tant en physiologie qu’en maladie.
Protocole
Les poissons-zèbres ont été maintenus dans des conditions standard, selon FELASA42 et institutionnels (Université Libre de Bruxelles, Bruxelles, Belgique ; ULB) directives et règlements. Toutes les procédures expérimentales ont été approuvées par le Comité d’éthique pour le bien-être animal de l’ULB (CEBEA) de l’ULB.
1. Élevage du poisson-zèbre et préparation de l’embryon
REMARQUE : La lignée transgénique de poisson-zèbre Tg(mpeg1 :eGFP)gl22 qui exprime la protéine eGFP fluorescente dans les macrophages, y compris la microglie, a été utilisée pour générer les données de suivi présentées dans ce protocole. D’autres lignes de rapporteurs de macrophages sont disponibles auprès de ZIRC et peuvent également être utilisées. Dans le choix de la lignée transgénique, il est important de considérer qu’une intensité de signal élevée facilitera l’acquisition d’images et la segmentation cellulaire.
- Maintenir le poisson-zèbre (Danio rerio) à une température de 28 °C et le gérer conformément aux protocoles établis11.
- Stimulez l’accouplement en plaçant les poissons femelles et mâles ensemble dans un bassin d’élevage, avec un fond grillagé et un séparateur, en fin d’après-midi avant la date de naissance des embryons souhaités. Retirez le séparateur le lendemain matin pour chronométrer l’accouplement du poisson.
- Prélevez les ovules fécondés à l’aide d’un filet et transférez-les dans une boîte de Pétri de 100 mm contenant du milieu E3 (5 mM de NaCl, 0,17 mM de KCl, 0,33 mM de CaCl2, 0,33 mM de MgSO4) avec 0,01 % de bleu de méthylène. En fonction du patrimoine génétique de la lignée transgénique de poisson-zèbre utilisée, si la formation de pigments doit être inhibée pour faciliter l’imagerie, ajouter 0,003 % de N-phénylthiourée (PTU) au tampon E3 dans lequel les embryons se développent, en commençant 6 heures après la fécondation (hpf) et en actualisant la solution tous les 2 jours jusqu’à ce que le stade de développement souhaité soit atteint.
REMARQUE : Pour éviter une longue exposition à la PTU qui pourrait affecter le développement embryonnaire12, il est suggéré d’utiliser, si disponible, une lignée transgénique sur un fond déficient en pigments, comme Casperw2, qui permet une imagerie claire sans avoir besoin d’exposer les embryons à la PTU pendant une période prolongée13. - Si les embryons à imager sont inférieurs à 3 jours après la fécondation (dpf), déchorait-les à 24 hpf. À l’aide d’un stéréomicroscope, on peut confirmer que tous les embryons ont été déchoionnés et qu’ils ne sont pas endommagés par le traitement enzymatique (voir étape 1.4.2).
- Dans le cas d’un nombre limité d’ovules, faites-le manuellement à l’aide d’une pince à dissection fine pour pincer le chorion et tirer vers l’extérieur, en libérant soigneusement chaque embryon de son chorion.
- Dans le cas d’un nombre plus élevé d’œufs, effectuez la déchorionation enzymatique en incubant les œufs en vrac à température ambiante (RT) dans une solution de Pronase à 1 mg/mL dans un milieu E3 pendant une brève période (généralement 5 à 10 minutes, jusqu’à ce que les chorions s’ouvrent en remuant doucement). Ensuite, la solution de Pronase est jetée et les embryons sont rincés abondamment 3 fois avec de l’E3 frais, pour éliminer tout chorion restant et toute enzyme résiduelle.
2. Montage du poisson zèbre
- Préparez une solution d’agarose à faible point de fusion à 1 % dans un tampon E3. Refroidissez l’agarose à 37 °C dans un bloc chauffant de paillasse et ajoutez-y une solution de méthanesulfonate de tricaïne fraîchement préparée (160 mg/L), qui aidera à garder les embryons anesthésiés pendant l’imagerie. Vortex la solution pour l’homogénéiser en profondeur.
- Anesthésier les embryons en ajoutant 160 mg/L de méthanesulfonate de tricaïne dans la boîte de Pétri contenant les embryons dans le tampon E3. Vérifiez que le mouvement s’est arrêté et sélectionnez les embryons de poisson-zèbre avec la fluorescence souhaitée. Vérifiez la santé de l’embryon en visualisant son rythme cardiaque à l’aide d’un stéréomicroscope à fond clair. À l’aide d’une pipette de transfert à pointe large, prélevez un ou jusqu’à trois des embryons sélectionnés et transférez-les sur une plaque d’imagerie en verre inférieure.
- Sous un stéréomicroscope, utilisez une pipette pour prélever le plus de milieu possible, en faisant attention de ne pas toucher ou endommager les embryons ; Ensuite, remplacez le milieu par la solution d’agarose à 1 % à bas point de fusion préalablement préparée. Ensuite, utilisez une pointe mince en plastique fixée à l’extrémité d’une aiguille de taquinerie pour pousser doucement chaque embryon vers le bas de la plaque d’imagerie, en l’orientant de manière à ce que sa face dorsale soit face au verre pour assurer une bonne vue du tectum optique et une quantité minimale d’agarose à faible fusion entre l’embryon et le verre.
- Imagez rapidement l’embryon monté à l’aide d’un microscope confocal inversé doté d’un objectif Plan Apo de haute qualité (10x/0,45 Plan Apo ou 20x/0,75 Plan Apo utilisé ici). Si le timelapse dure une période prolongée (plus d’une heure), ajoutez une ou deux gouttes de tampon E3 avec du méthanesulfonate de tricaïne (160 mg/L) sur la plaque d’imagerie en verre inférieure pour empêcher l’agarose à bas point de fusion de sécher excessivement.
REMARQUE : Il est crucial d’attendre que l’agarose se solidifie complètement avant d’ajouter un tampon E3 et un média de méthanesulfonate de tricaïne au disque d’imagerie ; Sinon, le poisson pourrait se déplacer lentement latéralement et axialement, provoquant ainsi une dérive de l’échantillon dans le timelapse. - Procédez à la configuration de l’acquisition timelapse pour la durée souhaitée : réglez la résolution de l’image sur 1 024 x 1 024, avec une taille de pixel de 0,49 μm. Collectez des coupes optiques par incréments de 2 à 3 μm pour garantir une résolution acceptable sur l’axe z. Dans l’embryon de poisson-zèbre, enfermez le tectum optique dans une pile en Z de 100 à 150 μm, selon le stade de développement utilisé. Pour garantir un suivi réussi des cellules, maintenez l’intervalle de temps entre les images entre 30 s et 60 s.
3. Analyse du suivi et exportation des données
- Convertissez le fichier timelapse au format IMS à l’aide du logiciel de conversion de fichiers Imaris (voir la table des matériaux). Au lancement du logiciel, faites glisser et déposez les fichiers dans la zone de saisie ou utilisez l’option Ajouter des fichiers... pour les sélectionner manuellement. Dans le menu Sortie , désignez l’emplacement des fichiers convertis, en optant pour le même dossier que les fichiers d’entrée ou en spécifiant un dossier particulier. Assurez-vous que les dimensions de la taille du voxel affichées sont correctes en cliquant sur le bouton Définir la taille du voxel et, une fois la taille du voxel vérifiée, appuyez sur le bouton Démarrer tout pour démarrer la conversion du fichier (voir le fichier supplémentaire 1 pour un guide étape par étape de toute l’analyse des données).
REMARQUE : Si le timelapse a été effectué sur deux embryons ou plus en parallèle, le fichier sera automatiquement divisé en positions uniques, séparant chaque embryon en différents fichiers. Il est possible d’utiliser le logiciel en mode batch et de télécharger et convertir plusieurs fichiers en même temps. Les fichiers IMS seront créés et enregistrés dans le dossier de destination sélectionné ou dans le dossier où se trouvent les fichiers d’entrée. - Une fois lancé, laissez le logiciel d’analyse démarrer dans l’Arena ; cliquez sur Observer le dossier pour ouvrir le fichier précédemment converti. Une fois le fichier chargé dans le logiciel, utilisez la vue Slice pour faire défiler la pile z, à l’aide du curseur de la barre d’outils Slice située à gauche de la zone d’affichage, et mesurez certains diamètres de cellules en dessinant le diamètre des cellules sélectionnées à l’aide du pointeur. Cliquez et faites glisser la souris pour dessiner un segment couvrant le noyau de la cellule ; observez la longueur du segment dessiné dans la barre d’outils Mesure, située à droite de la zone d’affichage.
REMARQUE : En utilisant les paramètres d’imagerie à l’étape 2.5, on peut s’attendre à ce que le diamètre du corps cellulaire soit compris entre 6 μm et 8 μm. - Pour détecter automatiquement les cellules, revenez à la vue 3D et cliquez sur l’icône Spot dans la barre d’outils d’objets pour ajouter un nouvel objet Spots dans la liste d’objets et ouvrez l’assistant de création automatique de Spots. Dans la première étape de l’assistant, cochez l’option permettant de segmenter uniquement une région d’intérêt. Ensuite, activez l’option Suivre les points (au fil du temps) pour calculer les données de suivi, ainsi que les statistiques objet-objet pour permettre la comparaison entre les points et élargir la plage de données qui seront disponibles plus tard dans l’analyse. Enfin, procédez dans l’assistant en cliquant sur le bouton Suivant en bas à droite de la fenêtre de l’assistant.
- Dans la deuxième étape, procédez à la spécification d’une zone de région d’intérêt (ROI) qui entoure le tectum optique de l’embryon. Modifiez la taille du ROI en cliquant et en faisant glisser les petites flèches blanches présentes sur chaque face de celui-ci. Une fois la taille du retour sur investissement définie, étendez-la à toutes les images enregistrées en ajustant l’intervalle de temps.
- Ensuite, sélectionnez comme Canal source le canal utilisé pour détecter les cellules, et définissez la mesure du diamètre de la cellule, précédemment obtenue à l’étape 3.2, comme Diamètre XY estimé. Activez l’option Soustraction d’arrière-plan et cliquez sur Suivant.
- À ce stade de l’assistant de création de spots, le logiciel intègre une étape de filtrage de qualité dans laquelle les spots sont filtrés par l’intensité du signal mesurée au centre de chaque spot ; dans le chenal où les taches ont été détectées. Ajustez le seuil d’intensité en saisissant la valeur directement dans le champ de données des cases de seuil inférieur et supérieur ou en faisant glisser les lignes colorées correspondantes dans l’histogramme affiché en bas de l’assistant de création, afin que toutes les cellules soient détectées, y compris celles localisées plus profondément dans le tissu, qui présentent généralement une intensité de signal plus faible. Toutes ces modifications sont visibles simultanément dans la zone d’affichage.
REMARQUE : Il est possible d’observer plusieurs points étiquetant une seule cellule à ce stade, mais cela peut être corrigé plus tard dans l’analyse.
Dans les timelapses plus longs, si un photoblanchiment est observé, vérifiez la zone d’affichage de plusieurs images avant de définir le seuil pour vous assurer que toutes les cellules sont également détectées dans les images les plus récentes et donc les plus faibles. - Une fois satisfait, cliquez sur Suivant pour valider le seuillage de qualité sélectionné. Tous les spots satisfaisant au paramètre de seuillage de qualité et ayant un diamètre correspondant au diamètre XY estimé seront étiquetés comme un spot. Visualisez-le comme un aperçu dans la zone de vue, où les points satisfaisant ces deux paramètres seront superposés au canal source.
- À l’étape suivante, sélectionnez le mouvement autorégressif comme algorithme de suivi pour suivre les cellules qui ont un mouvement plus ou moins continu. Jugez de la plus longue distance parcourue par une cellule entre deux points temporels en observant attentivement le mouvement des points entre deux images et entrez un nombre estimé dans le champ Distance maximale . Il s’agit de la distance à laquelle un point est autorisé à s’écarter de la position future prévue.
- Réglez la taille maximale de l’écart sur le nombre minimum de points temporels pendant lesquels le point peut disparaître sans interrompre la piste. Si l’intervalle entre les images est inférieur à 60 s, utilisez une taille de 3. Si l’intervalle de temps entre les images est plus long, augmentez la taille maximale de l’espace autorisée pour réduire la fragmentation de la piste.
- Activez l’option Remplir les espaces avec tous les objets détectés pour abaisser le seuil de détection près de la position attendue prédite par l’algorithme de suivi et connecter les pistes interrompues par un échec de la détection ponctuelle. Enfin, cliquez sur Suivant et laissez le logiciel générer automatiquement des traces pour tous les spots précédemment générés.
- Si les traces obtenues semblent trop nombreuses ou fragmentées, retournez dans l’assistant de création, en cliquant sur le bouton Précédent , et ajustez la distance maximale précédemment définie, en augmentant ou en diminuant le nombre jusqu’à ce que la plupart des pistes obtenues soient représentatives des mouvements des cellules.
REMARQUE : Le nombre de pistes doit correspondre à peu près au nombre de cellules microgliales, donc compris entre 30 et 50, selon l’âge de l’embryon. Cependant, avant la fin de l’analyse, un nombre plus élevé est attendu en raison de traces erronées. - Une fois les traces calculées, le logiciel intègre une étape de filtrage pour filtrer toutes les traces très brèves car elles sont souvent imprécises ou non informatives. Excluez les pistes de moins de 3 à 8 minutes (min) en saisissant la valeur directement dans le champ de données pour la limite inférieure du filtre de durée de piste, ou en faisant glisser les lignes colorées correspondantes dans l’histogramme affiché en bas de l’assistant de création. Cliquez sur Suivant pour valider le filtre et avancer dans l’assistant de création.
REMARQUE : Comme mentionné précédemment, en modifiant le seuil de durée maximale (max) et minimale (min) des pistes, il est possible de prévisualiser les pistes qui seront filtrées et exclues de l’analyse. Les pistes filtrées peuvent ensuite être dupliquées dans un nouvel objet pour une analyse plus approfondie. Le seuil de temps exact pour le filtrage peut varier en fonction des résultats de l’étape précédente de l’assistant de création. Nous vous recommandons d’affiner ces paramètres de filtre en examinant l’aperçu dans la zone d’affichage pour des résultats optimaux. - Étant donné que la lignée transgénique Tg(mpeg1 :eGFP) marque tous les phagocytes mononucléaires, les macrophages cutanés en plus de la microglie seront également détectés dans les films timelapse. Supprimer manuellement les traces générées à partir des macrophages cutanés de l’analyse pour se concentrer sur la microglie parenchymateuse cérébrale (Figure 3).
- Corrigez manuellement d’autres erreurs potentielles dans les pistes dans l’éditeur de pistes, où chaque point de données est affiché comme un objet dans la piste. Activez le bouton de sélection de cercle à droite de la zone d’affichage et mettez en évidence les pistes qui nécessitent des modifications ou des ajustements. Une fois la piste problématique sélectionnée, à l’aide des boutons de connexion et de déconnexion, modifiez-la pour représenter correctement les mouvements de la cellule. Utilisez l’éditeur de pistes pour sélectionner et supprimer également des points uniques dupliqués ou de petits fragments de piste qui représentent par erreur la même cellule en les sélectionnant avec le mode de sélection circulaire et en appuyant sur Canc ou Suppr pour les éliminer.
REMARQUE : Si les paramètres du suivi sont corrects, cette étape ne doit être requise que pour un petit pourcentage des cellules suivies. - Si la ligne rapporteure de la microglie est utilisée en combinaison avec des transgéniques fluorescents marquant d’autres types de cellules dans le parenchyme, effectuez la segmentation en tant que nouvel objet de surface ou objet ponctuel pour les visualiser. Pour ce faire, répétez les étapes ci-dessus (des étapes 3.2 à 3.14), en modifiant l’entrée du canal source à l’étape 3.5, pour qu’elle soit celle utilisée pour imager la deuxième population de cellules, et en prenant soin d’adapter les paramètres descriptifs spécifiques aux cellules d’intérêt tels que le diamètre XY estimé (étape 3.5), la distance maximale (étape 3.8) à la nouvelle population de cellules. D’autres étapes peuvent être répétées sans modifications.
REMARQUE : Dans le cas d’une nouvelle création de surface, le diamètre XY estimé est remplacé par une option de seuillage basée sur la soustraction d’arrière-plan, où l’entrée à donner est le diamètre de la plus grande sphère qui s’insère dans l’objet. Cette valeur peut être estimée à partir de la vue Tranche à l’aide de la barre d’outils Mesurer , comme expliqué à l’étape 3.2. - Une fois satisfait des traces obtenues, extrayez les statistiques de suivi souhaitées depuis l’onglet Statistiques . En cliquant sur le bouton des paramètres , sélectionnez les statistiques à calculer pour chaque objet ponctuel ou objet de surface précédemment créé. Si plusieurs objets ont été créés dans la même analyse, deux objets ponctuels ou un objet ponctuel et un objet surface, les statistiques décrivant la position relative d’un objet par rapport à l’autre, telles que la distance la plus courte entre les deux, seront également disponibles pour l’exportation.
REMARQUE : Toutes les valeurs souhaitées peuvent être des valeurs spécifiques ou moyennes, et toutes les données brutes sont téléchargeables sous forme de fichier CSV (Comma Separated Values) à l’aide des boutons situés en bas de l’onglet Statistiques . À condition que l’analyse inclue également d’autres types de cellules, des statistiques supplémentaires sur la distance relative d’un objet par rapport à l’autre seront également disponibles.
Résultats
Cellules microgliales exprimant la protéine fluorescente verte (eGFP) et cellules endothéliales exprimant DsRed dans Tg(mpeg1 :eGFPgl22 ; KDRL :CRES898 ; actb2 :loxP-STOP-loxP-DsRedexpress,sd5) embryons transgéniquestriples 14 ont été imagés à 3 dpf, selon le protocole décrit. Un seul embryon de poisson-zèbre a été monté dans de l’agarose à faible point de fusion à 1 % sur une plaque de verre inférieure et le processus d’imagerie n’a pas entravé la croissance de l’embryon pendant la période d’acquisition. Le timelapse a été enregistré à l’aide d’un système commercial de microscopie confocale à balayage ponctuel équipé d’une lentille d’objectif sec 10x 0,45 NA, et des lasers d’excitation de 488 nm et 561 nm ont été utilisés pour l’imagerie des cellules microgliales et endothéliales, respectivement. De plus, l’intervalle de temps, la résolution de l’image, la taille des pixels et l’étape z étaient de 30 secondes (s), 1024x1024, 0,49 μm et 2,5 μm, respectivement. L’enregistrement en accéléré a duré 3,5 heures (h).
Le champ de vision acquis couvrait toute la tête de l’embryon, mais l’analyse s’est concentrée spécifiquement sur le tectum optique, car au cours de la première semaine de développement, les microglies sont principalement limitées à la couche neuronale du soma de cette région du mésencéphale dorsal, ce qui permet aux enquêteurs de visualiser l’ensemble de la population simultanément15. L’analyse de suivi 3D a été réalisée à l’aide d’Imaris 10.0, comme expliqué ci-dessus. Comme le montre la figure 4, le suivi a été couronné de succès, avec 25 traces, ce qui correspond au nombre attendu de cellules microgliales présentes dans le tectum optique à 3 dpf16. Une correction manuelle minimale des traces a été nécessaire. La figure 5 montre un exemple de données qu’il est possible d’extraire d’une expérience de suivi réussie. Le marquage simultané des macrophages et des cellules endothéliales permet de quantifier la position de la microglie par rapport à ces dernières, ce qui permet aux chercheurs de visualiser la distance relative de chaque cellule par rapport à la cellule endothéliale la plus proche dans le temps et d’examiner la fréquence et le nombre d’interactions potentielles (Figure 5).
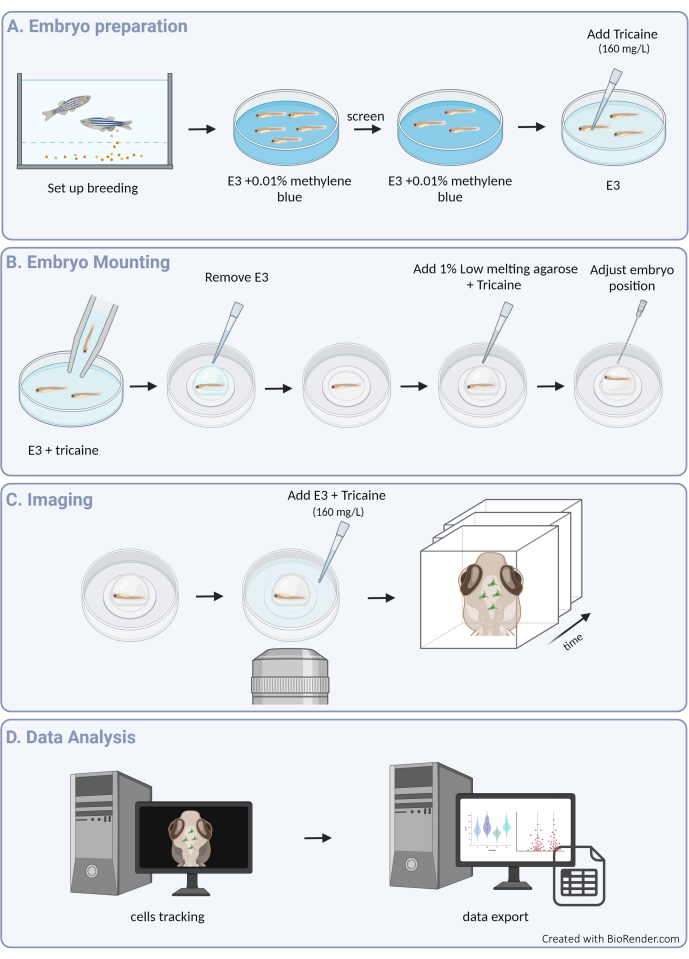
Figure 1 : Vue d’ensemble de la procédure expérimentale. (A) Préparation et anesthésie de l’embryon de poisson-zèbre. (B) Montage et positionnement de l’échantillon. (C) Acquisition d’images. (D) Traitement d’images et extraction de données de motilité. Veuillez cliquer ici pour voir une version agrandie de cette figure.
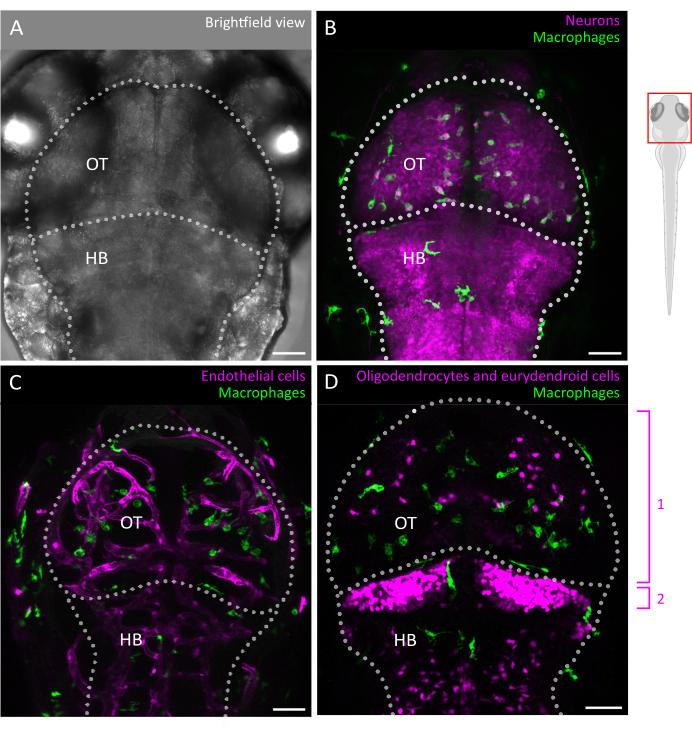
Figure 2 : Imagerie de la microglie et interactions avec l’environnement cellulaire du cerveau chez l’embryon de poisson-zèbre. (A) Image en fond clair de la tête et du cerveau d’un poisson-zèbre embryonnaire de 3 dpf (vue dorsale), marquant le tectum optique et le cerveau postérieur. Le cerveau du poisson-zèbre embryonnaire peut être imagé dans son intégralité à ce stade en raison de sa petite taille et de sa transparence optique. (B-D) L’emplacement et le comportement de la microglie peuvent être visualisés à l’aide de lignées transgéniques telles que (B,C) Tg(mpeg1 :eGFP)gl22 et (D) Tg(mpeg1 :mcherry)gl23, et en se concentrant sur l’OT. (B) Les neurones et leurs corps cellulaires peuvent être identifiés à l’aide de la lignée rapporteuse Tg(XlTubb:D sRed)zf148 et les interactions microglie-neurone peuvent être visualisées en fusionnant les deux canaux dans des lignées qui co-expriment les deux transgènes. On voit une fusion des signaux vert (microglie) et rouge (neurones, en magenta). (C, D) Les lignes rapporteures peuvent également mettre en lumière les interactions de la microglie (en vert) avec les cellules endothéliales, ici étiquetées en rouge à l’aide du (C) double Tg(kdrl :cres898 ; actb2 :LOXP-STOP-LOXP-Dsredexpress, sd5), ou avec des cellules gliales telles que les oligodendrocytes et leurs précurseurs, en utilisant le (D) double Tg(olig2 :EGFP ; mpeg1 :mcherry ) lignée transgénique. 1 : oligodendrocytes et cellules progénitrices dans l’OT, 2 : neurones eurydendroïdes dans le cervelet. Barres d’échelle = 50 μm. Abréviations : dpf = jours après la fécondation ; OT = tectum optique ; HB = cerveau postérieur. Veuillez cliquer ici pour voir une version agrandie de cette figure.
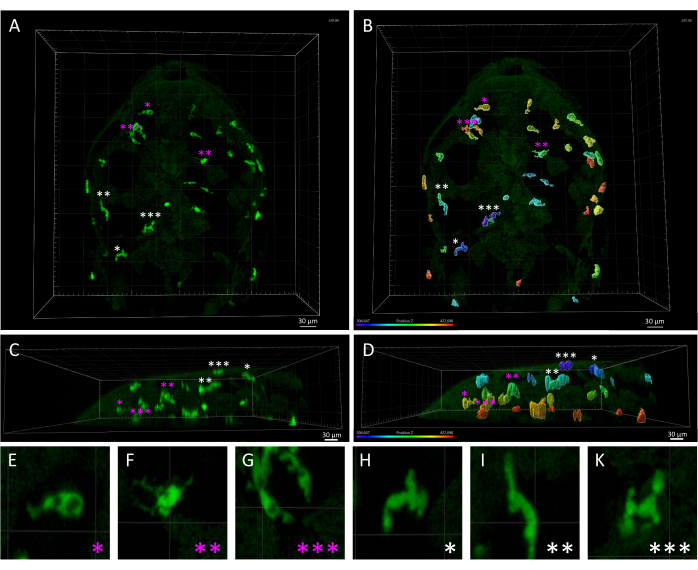
Figure 3 : Disparité morphologique et spatiale entre les macrophages cutanés et la microglie chez un embryon de poisson-zèbre de 3 dpf. (A,B) Vue dorsale d’un embryon de poisson-zèbre Tg(mpeg1 :eGFP)gl22 à 3 dpf, représentant (A) des cellules microgliaires parenchymateuses (marquées par des astérisques magenta) par rapport aux macrophages cutanés (astérisques blancs), identifiés en fonction de leur position relative le long de l’axe z, comme on le voit en (B), qui affiche l’image 3D monochrome rendue avec le codage couleur de l’axe z. (C, D) Vue latérale de (C) A et (D) B, mettant en évidence les profondeurs z des cellules mpeg1 :eGFP+ dans la tête de l’embryon et mettant en évidence la localisation superficielle des macrophages cutanés par rapport aux cellules microgliales. (E-K) Grossissement élevé de chaque cellule indiqué par un astérisque en A et B, fournissant une visualisation détaillée des morphologies distinctes entre la microglie amiboïde (E-G) et les macrophages cutanés allongés (H-K). Barres d’échelle = 30 μm. Abréviation : dpf = jours après la fécondation. Veuillez cliquer ici pour voir une version agrandie de cette figure.
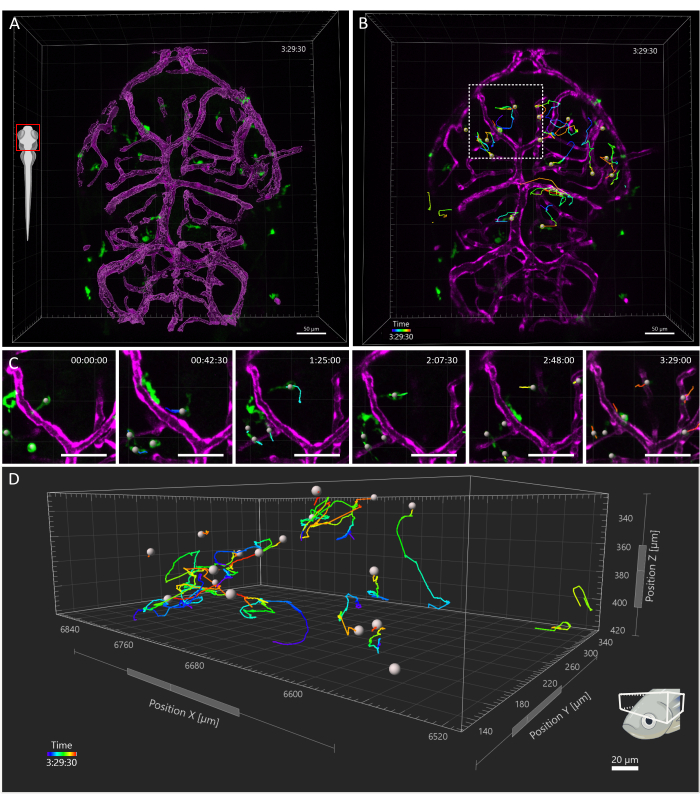
Figure 4 : Suivi des cellules microgliales dans un embryon de poisson-zèbre 3 dpf. (A) Vue dorsale de la tête d’un Tg(kdrl :cres898 ; actb2 :LOXP-STOP-LOXP-Dsredexpress,sd5 ; mpeg1 :eGFPgl22) embryon transgénique triple, montrant la microglie (vert) et le rendu superficiel des vaisseaux (magenta). (B-D) Suivi représentatif des mouvements de la microglie sur une fenêtre de temps de (B) 3,5 heures. On voit des cellules individuelles suivre des trajectoires complexes. (C) Détail du timelapse, montrant une vue agrandie de la région en B délimitée par le carré en pointillés. Six images du film (à 45 minutes d’intervalle) sont présentées, documentant l’établissement de contacts transitoires entre les microglies et les cellules endothéliales dans leur microenvironnement. (D) Trajectoires de migration des cellules microgliales individuelles dans le tectum optique, les axes X, Y et Z représentant les dimensions spatiales. Barre d’échelle = 50 μm. Abréviation : dpf = jours après la fécondation. Veuillez cliquer ici pour voir une version agrandie de cette figure.
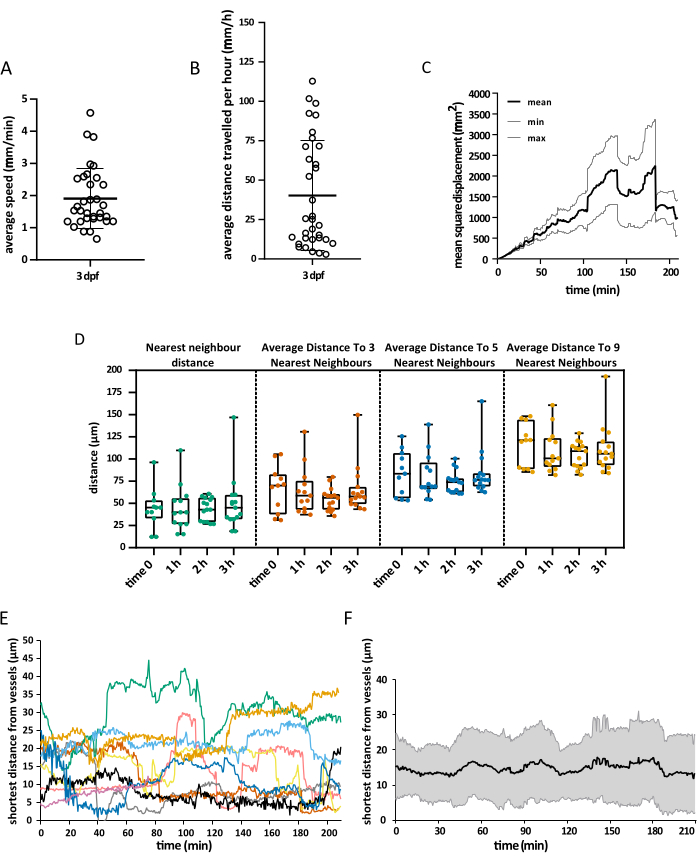
Figure 5 : Visualisation des données obtenues. Exemple de données spatiales pouvant être obtenues à l’aide du protocole décrit. Les graphiques représentent (A) la vitesse moyenne des cellules microgliales, (B) la distance moyenne qu’elles parcourent en 1 h, (C) leur déplacement carré moyen et (D) leur distribution dans l’espace à différents moments. Le rendu de la surface du vaisseau a également permis de mesurer la distance la plus courte entre la microglie et les cellules endothéliales à un moment donné, à la fois au niveau (E) de la cellule unique et en tant que moyenne mondiale (F). Pour A, B et D, chaque point représente une cellule individuelle. N = un embryon. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Fichier supplémentaire 1 : Veuillez cliquer ici pour télécharger ce fichier.
Discussion
Le protocole actuel permet l’imagerie in vivo de la dynamique de la microglie chez un embryon de vertébré et la visualisation des données de motilité acquises. La colonisation microgliale du cerveau en développement se produit très tôt au cours de l’embryogenèse et précède des événements critiques tels que les pics de neurogenèse, l’astrogliogenèse, l’oligodendrogenèse et de nombreux autres processus cellulaires17. Il n’est donc pas surprenant que la microglie joue un rôle important dans la formation d’aspects spécifiques du développement du cerveau18, par exemple, par la régulation de la différenciation neuronale, de la migration et de la survie 19,20,21, ainsi que de l’élagage synaptique 5 et de la myélinisation 22,23,24.
La contribution d’une microglie dysfonctionnelle à la pathogenèse et/ou à la progression des troubles neurodéveloppementaux est également de plus en plus reconnue25. En effet, la présence précoce de microglie dans le cerveau en formation expose ces cellules à des états physiologiques26 et à des changements environnementaux distincts. Cela peut avoir un impact significatif étant donné que les cellules microgliales sont des cellules à longue durée de vie chez les rongeurs et les humains, qui se maintiennent tout au long de la vie grâce à l’auto-renouvellement des progéniteurs locaux 27,28,29. Nous pensons que ce protocole pourrait servir d’outil puissant pour mieux caractériser le comportement des microglies dans ces états physiologiques distincts, à mesure qu’elles se développent, mûrissent et établissent leur réseau au cours des étapes successives de la morphogenèse cérébrale.
En utilisant la configuration décrite ici, nous avons réussi à imager et à acquérir des données sur des larves de poisson-zèbre âgées de 6 dpf. L’extension de l’analyse aux stades ultérieurs du développement sera probablement couronnée de succès, mais nécessitera d’ajuster la configuration de l’imagerie pour tenir compte de l’augmentation de la taille de l’échantillon, en particulier le long de l’axe z. Pour ce faire, nous vous suggérons de vous concentrer sur le maintien d’un faible rapport signal/bruit et d’un temps de balayage rapide, car ce sont des paramètres clés pour une analyse réussie.
Nous suggérons un temps d’imagerie minimum de 1 h pour permettre le suivi de la microglie ; La fenêtre d’imagerie la plus longue qui a été testée avec ce protocole est de 8 h. De plus, il est important pour l’analyse de suivi de maintenir l’intervalle de temps entre les images aussi court que possible, idéalement entre 30 s et 60 s. Cela permettra d’obtenir des données de suivi plus précises et plus détaillées dans les analyses en aval. Par conséquent, en particulier si vous détectez plus d’un fluorophore, il est fondamental d’éviter le chevauchement spectral et d’assurer une séparation suffisante entre les deux spectres d’émission de fluorophore pour permettre une acquisition simultanée, sans fuite du signal.
D’autres protocoles pour l’enregistrement timelapse de haute qualité du cerveau du poisson-zèbre sont disponibles30, mais c’est le premier montrant comment suivre avec succès tous les mouvements de la microglie pendant le développement embryonnaire sur une période prolongée. Bien que le flux de travail présenté ici se concentre sur le suivi de la microglie dans un contexte physiologique, il peut être facilement appliqué à l’analyse de la microglie en pathologie. En effet, plusieurs modèles de troubles neurodéveloppementaux, tels que l’autisme31, l’épilepsie32 et la schizophrénie33, mais aussi la neurodégénérescence34 et le cancer35, ont été établis chez le poisson-zèbre qui offrent des opportunités uniques pour déterminer la réponse microgliale et le comportement dans des conditions pathologiques.
Notamment, ce protocole de suivi est très polyvalent et pourrait également contribuer à faire la lumière sur les modèles de migration de divers types de cellules à travers diverses régions anatomiques de l’embryon de poisson-zèbre, ouvrant ainsi potentiellement des voies pour des applications supplémentaires, au-delà du champ d’investigation microgliale décrit dans cet article. De plus, en exploitant la capacité de combiner plusieurs lignées transgéniques fluorescentes, nous acquérons la capacité de discerner la relation spatiale entre la microglie et d’autres types de cellules du microenvironnement cérébral, avec le potentiel de visualiser les interactions cellulaires et les interactions croisées tout au long des enregistrements en accéléré, de manière non invasive. Cela pourrait contribuer à démêler la signification physiologique du comportement de la microglie et à une caractérisation plus approfondie de ces cellules très mobiles.
Déclarations de divulgation
Les auteurs ne déclarent aucun conflit d’intérêts.
Remerciements
Les auteurs tiennent à exprimer leur sincère gratitude au Professeur Nicolas Bayens pour avoir généreusement fourni l’accès au microscope confocal indispensable à cette étude. Ces travaux ont été financés en partie par le Fonds pour la Recherche Scientifique (FNRS) au titre des subventions n° F451218F et UG03019F, la Fondation pour la Recherche sur l’Alzheimer (SAO-FRA) (à V.W.), A.M. est soutenu par une bourse de recherche du FNRS. La figure 1 a été créée le biorender.com.
matériels
| Name | Company | Catalog Number | Comments |
| 1 L Breeding tanks | Tecniplast | ZB10BTE | |
| 1-phenyl-2-thiourea (PTU) | Sigma-Aldrich | P7629 | Diluted to 0.2 mM in E3 to prevent embryo pigmentation |
| Bottom glass imaging dish | FluoroDish | FD3510-100 | |
| Disposable Graduated transfer pipette | avantor | 16001-188 | |
| Dry block heater | Novolab | Grant QBD4 | To keep low melting agarose at 37 °C |
| Ethyl 3-aminobenzoate methanesulfonate (Tricaine) | Sigma-Aldrich | E10521-50G | |
| Imaris 10.0 | Oxford Instruments | analysis software | |
| Imaris File Converter | Oxford Instruments | https://imaris.oxinst.com/big-data | |
| Laser-scanning confocal microscope | Nikon | Eclipse Ti2-E | |
| Methylene blue | Sigma-Aldrich | M9140-25G | |
| microloader tips | Eppendorf | 5242956003 | |
| NuSieve GTG Agarose | Lonza | 50081 | |
| Petri dishes (90 mm) | avantor | 391-0559 | |
| Pronase | Sigma-Aldrich | 11459643001 | |
| Stainless Steel Forceps Dumont No. 5 | FineScienceTools | 11254-20 | |
| Stereo microscope | Leica | Leica M80 | To mount the embryos |
| teasing needle | avantor | 76549-024 |
Références
- Wolf, S. A., Boddeke, H. W., Kettenmann, H. Microglia in physiology and disease. Annu Rev Physiol. 79, 619-643 (2017).
- Nimmerjahn, A., Kirchhoff, F., Helmchen, F. Resting microglial cells are highly dynamic surveillants of brain parenchyma in vivo. Sci Rep. 308 (5726), 1314-1318 (2005).
- Peri, F., Nusslein-Volhard, C. Live imaging of neuronal degradation by microglia reveals a role for v0-ATPase a1 in phagosomal fusion in vivo. Cell. 133 (5), 916-927 (2008).
- Marın-Teva, J. L., et al. Microglia promote the death of developing Purkinje cells. Neuron. 41 (4), 535-547 (2004).
- Paolicelli, R. C., et al. Synaptic pruning by microglia is necessary for normal brain development. Science. 333 (6048), 1456-1458 (2011).
- Hattori, Y. The microglia-blood vessel interactions in the developing brain. Neurosci Res. 187, 58-66 (2023).
- Colonna, M., Butovsky, O. Microglia function in the central nervous system during health and neurodegeneration. Annu Rev Immunol. 35, 441-468 (2017).
- Xu, H. -T., Pan, F., Yang, G., Gan, W. -B. Choice of cranial window type for in vivo imaging affects dendritic spine turnover in the cortex. Nat Neurosci. 10 (5), 549-551 (2007).
- Shin, J., Park, H. -C., Topczewska, J. M., Mawdsley, D. J., Appel, B. Neural cell fate analysis in zebrafish using olig2 BAC transgenics. Methods Cell Sci. 25 (1), 7-14 (2003).
- Chi, N. C., et al. Foxn4 directly regulates tbx2b expression and atrioventricular canal formation. Genes Dev. 22 (6), 734-739 (2008).
- Avdesh, A., et al. Regular care and maintenance of a zebrafish (Danio rerio) laboratory: An introduction. J Vis Exp. (69), e4196(2012).
- Bohnsack, B. L., Gallina, D., Kahana, A. Phenothiourea sensitizes zebrafish cranial neural crest and extraocular muscle development to changes in retinoic acid and IGF signaling. PLoS ONE. 6 (8), e22991(2011).
- Ariga, J., Walker, S. L., Mumm, J. S. Multicolor time-lapse imaging of transgenic zebrafish: visualizing retinal stem cells activated by targeted neuronal cell ablation. J Vis Exp. (43), e2093(2010).
- Ferrero, G., et al. Embryonic microglia derive from primitive macrophages and are replaced by cmyb-dependent definitive microglia in zebrafish. Cell Rep. 24 (1), 130-141 (2018).
- Xu, J., Wang, T., Wu, Y., Jin, W., Wen, Z. Microglia colonization of developing zebrafish midbrain is promoted by apoptotic neuron and lysophosphatidylcholine. Dev Cell. 38 (2), 214-222 (2016).
- Svahn, A. J., et al. Development of ramified microglia from early macrophages in the zebrafish optic tectum. Dev Neurobiol. 73 (1), 60-71 (2013).
- Thion, M. S., Garel, S. Microglial ontogeny, diversity and neurodevelopmental functions. Curr Opin Genet Dev. 65, 186-194 (2020).
- Bohlen, C. J., Friedman, B. A., Dejanovic, B., Sheng, M. Microglia in brain development, homeostasis, and neurodegeneration. Annu Rev Genet. 53, 263-288 (2019).
- Squarzoni, P., et al. Microglia modulate wiring of the embryonic forebrain. Cell Rep. 8 (5), 1271-1279 (2014).
- Schafer, D. P., et al. Microglia sculpt postnatal neural circuits in an activity and complement-dependent. Neuron. 74 (4), 691-705 (2012).
- Parkhurst, C. N., et al. Microglia promote learning-dependent synapse formation through brain-derived neurotrophic factor. Cell. 155 (7), 1596-1609 (2013).
- Green, L. A., O'Dea, M. R., Hoover, C. A., DeSantis, D. F., Smith, C. J. The embryonic zebrafish brain is seeded by a lymphatic-dependent population of mrc1+ microglia precursors. Nat. Neurosci. 25 (7), 849-864 (2022).
- Hughes, A. N., Appel, B. Microglia phagocytose myelin sheaths to modify developmental myelination. Nat Neurosci. 23 (9), 1055-1066 (2020).
- Yu, Q., et al. C1q is essential for myelination in the central nervous system (CNS). iScience. 26 (12), 108518(2023).
- Zengeler, K. E., Lukens, J. R. Innate immunity at the crossroads of healthy brain maturation and neurodevelopmental disorders. Nat Rev Immunol. 21 (7), 454-468 (2021).
- Paolicelli, R. C., et al. Microglia states and nomenclature: A field at its crossroads. Neuron. 110 (21), 3458-3483 (2022).
- Réu, P., et al. The lifespan and turnover of microglia in the human brain. Cell Rep. 20 (4), 779-784 (2017).
- Ajami, B., Bennett, J. L., Krieger, C., Tetzlaff, W., Rossi, F. M. V. Local self-renewal can sustain CNS microglia maintenance and function throughout adult life. Nat Neurosci. 10 (12), 1538-1543 (2007).
- Mildner, A., et al. Microglia in the adult brain arise from Ly-6ChiCCR2+ monocytes only under defined host conditions. Nat Neurosci. 10 (12), 1544-1553 (2007).
- Cook, Z. T., Brockway, N. L., Weissman, T. A. Visualizing the developing brain in living zebrafish using Brainbow and time-lapse confocal imaging. J Vis Exp. (157), e60593(2020).
- Tayanloo-Beik, A., et al. Zebrafish modeling of autism spectrum disorders, current status and future prospective. Front Psychiatry. 13, 911770(2022).
- D'Amora, M., et al. Zebrafish as an innovative tool for epilepsy modeling: State of the art and potential future directions. Int J Mol Sci. 24 (9), 7702(2023).
- Langova, V., Vales, K., Horacek, J. The role of zebrafish and laboratory rodents in schizophrenia research. Front Psychiatry. 11, 549232(2020).
- Chia, K., Klingseisen, A., Sieger, D., Priller, J. Zebrafish as a model organism for neurodegenerative disease. Front Mol Neurosci. 15, 940484(2022).
- Astell, K. R., Sieger, D. Zebrafish in vivo models of cancer and metastasis. Cold Spring Harb Perspect Med. 10 (8), 037077(2020).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon