Method Article
Imagen en vivo y caracterización de la dinámica de la microglía en el embrión de pez cebra
En este artículo
Resumen
Demostramos un método que aprovecha la microscopía confocal de barrido rápido para realizar imágenes en vivo de las células de la microglía en el tectum óptico del pez cebra en desarrollo, lo que permite el análisis de la dinámica de estas células in vivo.
Resumen
Las microglías son células altamente dinámicas y su migración y colonización del parénquima cerebral es un paso crucial para el desarrollo y la función cerebral adecuados. Los embriones de pez cebra que se desarrollan externamente poseen transparencia óptica, que junto con líneas reporteras transgénicas bien caracterizadas que marcan fluorescentemente la microglía, hacen del pez cebra un modelo de vertebrado ideal para tales estudios. En este artículo, aprovechamos las características únicas del modelo de pez cebra para visualizar la dinámica de las células de la microglía in vivo y en condiciones fisiológicas. Utilizamos microscopía confocal para registrar un lapso de tiempo de las células de la microglía en el tectum óptico del embrión de pez cebra y luego, extraemos los datos de seguimiento utilizando el software IMARIS 10.0 para obtener la ruta de migración de las células, la velocidad media y la distribución en el tectum óptico en diferentes etapas de desarrollo. Este protocolo puede ser una herramienta útil para dilucidar la importancia fisiológica del comportamiento de la microglía en diversos contextos, contribuyendo a una caracterización más profunda de estas células altamente móviles.
Introducción
Como macrófagos residentes en el sistema nervioso central (SNC), la microglía representa una población no neuronal distinta que representa hasta el 15% de todas las células gliales en el cerebro adulto. El estudio de la biología de la microglía ha ganado cada vez más atención en los últimos años debido a su importancia establecida en el desarrollo, la fisiología y la enfermedad1. En condiciones fisiológicas, las células microgliales son altamente dinámicas, inspeccionando continuamente el parénquima cerebral 2,3. Este comportamiento permite que la microglía colonice el cerebro y desempeñe un papel fundamental en su desarrollo, como la conformación de los circuitos neuronales4, la poda sináptica5 y la vasculogénesis6. Además, esta naturaleza dinámica inherente permite a la microglía monitorear constantemente el SNC en busca de signos de infección, lesión o cualquier desviación de la homeostasis7. Para diseccionar estas intrincadas dinámicas celulares, es indispensable obtener imágenes en vivo de la microglía a través del espacio y el tiempo. Afortunadamente, la transparencia óptica de los embriones de pez cebra en desarrollo externo, junto con la disponibilidad de líneas reporteras transgénicas bien caracterizadas que marcan fluorescentemente la microglía, posiciona al pez cebra como un modelo de vertebrado ideal para tales investigaciones. Las imágenes en vivo en embriones de pez cebra ofrecen un enfoque no invasivo que no requiere cirugía ni manipulación extensa del tejido, lo que minimiza las posibles perturbaciones en el estado del SNC. Esta es una consideración crítica cuando se estudian las células microgliales, ya que son muy sensibles incluso a los cambios sutiles en el entorno extracelular8.
Aquí, proporcionamos una guía para rastrear con éxito los movimientos de las células microgliales en 3D en el embrión de pez cebra, lo que permite una vista sin precedentes del comportamiento de la microglía dentro de la arquitectura intacta del parénquima cerebral en desarrollo (consulte la Figura 1 para obtener una descripción gráfica del protocolo). Este protocolo paso a paso detalla cómo configurar y obtener imágenes de la microglía del pez cebra en diferentes etapas de desarrollo y cómo extraer datos de alta resolución sobre la motilidad de las células de la microglía para proporcionar información valiosa sobre sus patrones migratorios y respuestas a las señales ambientales. También demostramos que este protocolo se puede adaptar para realizar imágenes multicolor en vivo, extendiendo así su aplicabilidad al estudio de la microglía en combinación con líneas transgénicas que marcan las células vecinas, incluidas las neuronas3, los oligodendrocitos9 y las células endoteliales10 (como se muestra en la Figura 2). Al añadir a la caja de herramientas que permite observar y caracterizar directamente la dinámica del comportamiento de la microglía en tiempo real y en su entorno natural, este protocolo probablemente contribuirá a dilucidar mejor la funcionalidad de la microglía durante el desarrollo temprano, tanto en fisiología como en enfermedad.
Protocolo
El pez cebra se mantuvo en condiciones estándar, de acuerdo con FELASA42 e instituciones (Université Libre de Bruxelles, Bruselas, Bélgica; ULB) y reglamentos. Todos los procedimientos experimentales fueron aprobados por el Comité Ético para el Bienestar Animal de la ULB (CEBEA) de la ULB.
1. Cría y preparación de embriones de pez cebra
NOTA: La línea transgénica de pez cebra Tg(mpeg1:eGFP)gl22 que expresa la proteína eGFP fluorescente en macrófagos, incluida la microglía, se utilizó para generar los datos de seguimiento que se muestran en este protocolo. Otras líneas reporteras de macrófagos están disponibles en ZIRC y también se pueden utilizar. A la hora de elegir la línea transgénica, es importante tener en cuenta que una alta intensidad de señal facilitará la adquisición de imágenes y la segmentación celular.
- Mantener el pez cebra (Danio rerio) a una temperatura de 28 °C y manejarlo de acuerdo con los protocolos establecidos11.
- Estimule el apareamiento colocando peces hembra y macho juntos en un tanque de cría, con un fondo de malla y un separador, al final de la tarde antes de la fecha de nacimiento de los embriones deseados. Retire el separador a la mañana siguiente para cronometrar el apareamiento de los peces.
- Recoja los óvulos fertilizados con una malla y transfiéralos a una placa de Petri de 100 mm que contenga medio E3 (5 mM de NaCl, 0,17 mM de KCl, 0,33 mM de CaCl2, 0,33 mM de MgSO4) con 0,01% de azul de metileno. Dependiendo de los antecedentes genéticos de la línea transgénica de pez cebra utilizada, si se va a inhibir la formación de pigmento para facilitar la obtención de imágenes, se debe añadir un 0,003% de N-feniltiourea (PTU) al tampón E3 en el que se están desarrollando los embriones, comenzando a las 6 h después de la fertilización (hpf) y refrescando la solución cada 2 días hasta alcanzar la etapa de desarrollo deseada.
NOTA: Para evitar una exposición prolongada a la PTU que podría afectar el desarrollo embrionario12, se sugiere utilizar, si está disponible, una línea transgénica sobre un fondo con deficiencia de pigmento, como Casperw2, que permite obtener imágenes claras sin la necesidad de exponer los embriones a la PTU durante un período prolongado de tiempo13. - Si los embriones a fotografiar son menores de 3 días después de la fertilización (dpf), descorionarlos a 24 hpf. Utilice un microscopio estereoscópico para confirmar que todos los embriones han sido descorionados y no han sido dañados por el tratamiento enzimático (ver paso 1.4.2).
- En el caso de un número limitado de óvulos, hágalo manualmente utilizando pinzas de disección finas para pellizcar el corion y tirar hacia afuera, liberando cuidadosamente cada embrión de su corion.
- En caso de un mayor número de huevos, realice la decorionación enzimática incubando los huevos a granel a temperatura ambiente (RT) en una solución de Pronase de 1 mg/mL en medio E3 durante un breve período (normalmente de 5 a 10 minutos, hasta que los coriones se abran al hacer un suave buche). A continuación, se desecha la solución de Pronase y los embriones se enjuagan a fondo 3 veces con E3 fresco, para eliminar cualquier corion restante y enzima residual.
2. Montaje en pez cebra
- Prepare una solución de agarosa de bajo punto de fusión al 1% en tampón E3. Enfríe la agarosa a 37 °C en un bloque calefactor de sobremesa y agregue una solución de metanosulfonato de tricaína recién hecha (160 mg / L), que ayudará a mantener los embriones anestesiados durante la imagen. Vórtice la solución para homogeneizarla a fondo.
- Anestesiar los embriones añadiendo 160 mg/L de metanosulfonato de tricaína a la placa de Petri que contiene los embriones en tampón E3. Verifique que el movimiento se haya detenido y seleccione los embriones de pez cebra con la fluorescencia deseada. Verifique la salud del embrión visualizando los latidos de su corazón con un microscopio estereoscópico de campo claro. Con una pipeta de transferencia de punta ancha, extraiga uno o hasta tres de los embriones seleccionados y transfiéralos a una placa de imagen de vidrio inferior.
- Bajo un microscopio estereoscópico, use una pipeta para eliminar la mayor cantidad de medio posible, teniendo cuidado de no tocar o dañar los embriones; Luego, reemplace el medio con la solución de agarosa de bajo punto de fusión al 1% previamente preparada. A continuación, utilice una punta delgada de plástico fijada al extremo de una aguja de burla para empujar suavemente cada embrión hacia el fondo de la placa de imagen, orientándola de modo que su lado dorsal quede frente al vidrio para garantizar una buena vista del tectum óptico y una cantidad mínima de agarosa de bajo punto de fusión entre el embrión y el vidrio.
- Visualice el embrión montado rápidamente utilizando un microscopio confocal invertido con un objetivo Plan Apo de alta calidad (10x/0.45 Plan Apo o un Plan Apo 20x/0.75 utilizado aquí). Si el lapso de tiempo se ejecutará durante un período prolongado (más de una hora), agregue una o dos gotas de tampón E3 con metanosulfonato de tricaína (160 mg / L) a la placa de imagen de vidrio inferior para evitar que la agarosa de bajo punto de fusión se seque en exceso.
NOTA: Es crucial esperar a que la agarosa se solidifique por completo antes de agregar tampón E3 y medios de metanosulfonato de tricaína al disco de imagen; De lo contrario, el pez podría moverse lentamente lateral y axialmente, causando así una deriva de la muestra en el lapso de tiempo. - Proceda a configurar la adquisición de lapso de tiempo para el tiempo deseado: establezca la resolución de la imagen en 1.024 x 1.024, con un tamaño de píxel de 0,49 μm. Recoja cortes ópticos en incrementos de 2-3 μm para garantizar una resolución aceptable en el eje z. En el embrión de pez cebra, encierre el tectum óptico en una pila Z de 100-150 μm, dependiendo de la etapa de desarrollo utilizada. Para garantizar el éxito del seguimiento de las células, mantenga el intervalo de tiempo entre fotogramas entre 30 s y 60 s.
3. Seguimiento, análisis y exportación de datos
- Convierta el archivo de lapso de tiempo a formato IMS utilizando el software de conversión de archivos Imaris (consulte la Tabla de materiales). Al iniciar el software, arrastre y suelte los archivos en el área de entrada o utilice el botón Agregar archivos... para seleccionarlos manualmente. En el menú Salida , designe la ubicación de los archivos convertidos, optando por la misma carpeta que los archivos de entrada o especificando una carpeta en particular. Asegúrese de que las dimensiones del tamaño del vóxel que se muestra sean correctas haciendo clic en el botón Establecer tamaño de vóxel y, una vez verificado el tamaño del vóxel, presione el botón Iniciar todo para iniciar la conversión del archivo (consulte el Archivo complementario 1 para obtener una guía paso a paso de todo el análisis de datos).
NOTA: Si el timelapse se realizó en dos o más embriones en paralelo, el archivo se dividirá automáticamente en una sola posición, separando cada embrión en diferentes archivos. Es posible utilizar el software en modo por lotes y cargar y convertir varios archivos al mismo tiempo. Los archivos IMS se crearán y guardarán en la carpeta de destino seleccionada o en la carpeta donde se encuentran los archivos de entrada. - Una vez iniciado, deje que el software de análisis se inicie en la Arena; haga clic en Observar carpeta para abrir el archivo convertido anteriormente. Una vez que el archivo está cargado en el software, use la vista de corte para desplazarse por la pila z, usando el control deslizante en la barra de herramientas de corte situado a la izquierda del área de vista, y mida algunos diámetros de celda dibujando el diámetro de las celdas seleccionadas con el puntero. Haga clic y arrastre el ratón para dibujar un segmento que abarque el núcleo de la célula; observe la longitud del segmento dibujado en la barra de herramientas de medición, situada a la derecha del área de vista.
NOTA: Utilizando la configuración de imagen en el paso 2.5, se puede esperar que el diámetro del cuerpo de la célula esté entre 6 μm y 8 μm. - Para detectar automáticamente las celdas, vuelva a la vista 3D y haga clic en el icono de punto en la barra de herramientas de objetos para agregar un nuevo objeto de manchas en la lista de objetos y abrir el asistente de creación automática de tintas planas. En el primer paso del asistente, marque la opción para segmentar solo una región de interés. A continuación, habilite la opción Rastrear puntos (a lo largo del tiempo) para calcular los datos de seguimiento, así como las Estadísticas de objeto a objeto para permitir la comparación entre puntos y ampliar el rango de datos que estarán disponibles más adelante en el análisis. Finalmente, continúe en el asistente haciendo clic en el botón Siguiente en la parte inferior derecha de la ventana del asistente.
- En el segundo paso, se procede a especificar un área de región de interés (ROI) que encierra el tectum óptico del embrión. Modifique el tamaño del ROI haciendo clic y arrastrando las pequeñas flechas blancas presentes en cada cara del mismo. Una vez definido el tamaño del ROI, extiéndalo a todos los fotogramas grabados ajustando el intervalo de tiempo.
- A continuación, seleccione como Canal de origen el canal utilizado para detectar las células y establezca la medición del diámetro de la célula, obtenida previamente en el paso 3.2, como el Diámetro XY estimado. Habilite la opción Resta de fondo y haga clic en Siguiente.
- En este punto del asistente de creación de spots, el software incorpora un paso de filtrado de calidad en el que los spots se filtran por la intensidad de la señal medida en el centro de cada spot; en el canal donde se detectaron las manchas. Ajuste el umbral de intensidad introduciendo el valor directamente en el campo de datos para los cuadros de umbral inferior y superior o arrastrando las líneas de color correspondientes en el histograma que se muestra en la parte inferior del asistente de creación, de modo que se detecten todas las células, incluidas las localizadas más profundamente en el tejido, que suelen mostrar una intensidad de señal más baja. Todos estos cambios son visibles simultáneamente en el área de visualización.
NOTA: Es posible observar múltiples puntos marcando una sola celda en este punto, pero esto se puede corregir más adelante en el análisis.
En lapsos de tiempo más largos, si se observa algún fotoblanqueo, verifique el área de vista de varios fotogramas antes de establecer el umbral para asegurarse de que todas las celdas también se detecten en los fotogramas más recientes y, por lo tanto, más tenues. - Una vez satisfecho, haga clic en Siguiente para validar el umbral de calidad seleccionado. Todas las manchas que cumplan con el parámetro de umbral de calidad y que tengan un diámetro que coincida con el diámetro XY estimado se etiquetarán como una mancha. Visualice esto como una vista previa en el área de visión, donde los puntos que satisfagan estos dos parámetros se superpondrán en el canal de origen.
- En el siguiente paso, seleccione Movimiento autorregresivo como algoritmo de seguimiento para realizar un seguimiento de las celdas que tienen un movimiento más o menos continuo. Juzgue la distancia más larga que se mueve una celda entre dos puntos de tiempo observando cuidadosamente el movimiento de los puntos entre dos fotogramas e introduzca un número estimado en el campo Distancia máxima . Esta es la distancia a la que se permite que un punto se desvíe de la posición futura predicha.
- Establezca el tamaño del espacio máximo en el menor número de puntos de tiempo en los que el punto pueda desaparecer sin interrumpir la pista. Si el intervalo entre fotogramas es inferior a 60 s, utilice un tamaño de 3. Si el intervalo de tiempo entre fotogramas es más largo, aumente el tamaño máximo del espacio permitido para reducir la fragmentación de la pista.
- Habilite la opción Rellenar huecos con todos los objetos detectados para que el umbral de detección caiga cerca de la posición esperada predicha por el algoritmo de seguimiento y conecte las pistas que se interrumpen por un error de la detección puntual. Finalmente, haga clic en Siguiente y deje que el software genere automáticamente pistas para todos los puntos generados anteriormente.
- Si las pistas obtenidas parecen ser demasiado numerosas o fragmentadas, retroceda en el asistente de creación, haciendo clic en el botón Anterior , y ajuste la distancia máxima previamente definida, ya sea aumentando o disminuyendo el número hasta que la mayoría de las pistas obtenidas sean representativas de los movimientos de las celdas.
NOTA: El número de huellas debe coincidir aproximadamente con el número de células de la microglía, por lo tanto, oscilar entre 30 y 50, dependiendo de la edad del embrión. Sin embargo, antes de completar el análisis, se espera un número mayor debido a pistas erróneas. - Una vez que se han calculado las pistas, el software incorpora un paso de filtrado para filtrar todas las pistas muy breves, ya que a menudo son inexactas o no son informativas. Excluya las pistas de menos de 3-8 minutos (min) introduciendo el valor directamente en el campo de datos para el límite inferior del filtro de duración de la pista, o arrastrando las líneas de color correspondientes en el histograma que se muestra en la parte inferior del asistente de creación. Haga clic en Siguiente para validar el filtro y continuar en el asistente de creación.
NOTA: Como se mencionó anteriormente, al modificar el umbral de duración máxima (máximo) y mínimo (mínimo) de la pista, es posible obtener una vista previa de las pistas que se filtrarán y excluirán del análisis. A continuación, las pistas filtradas se pueden duplicar en un nuevo objeto para su posterior análisis. El umbral de tiempo exacto para el filtrado puede variar en función de los resultados del paso anterior del asistente de creación . Recomendamos ajustar esta configuración de filtro revisando la vista previa en el área de vista para obtener resultados óptimos. - Dado que la línea transgénica Tg (mpeg1: eGFP) marca todos los fagocitos mononucleares, los macrófagos de la piel, además de la microglía, también se detectarán en las películas de lapso de tiempo. Elimine manualmente las huellas generadas por los macrófagos de la piel del análisis para centrarse en la microglía del parénquima cerebral (Figura 3).
- Corrija manualmente otros posibles errores en las pistas en el editor de pistas, donde cada punto de datos se muestra como un objeto en la pista. Active el botón de modo de selección circular a la derecha del área de vista y resalte las pistas que requieran modificaciones o ajustes. Una vez seleccionada la pista problemática, con los botones de conexión y desconexión , edítela para representar correctamente los movimientos de la celda. Utilice el editor de pistas para seleccionar y eliminar también puntos individuales duplicados o fragmentos de pista pequeños que representen erróneamente la misma celda seleccionándolos con el modo de selección de círculo y pulsando Canc o Supr para eliminarlos.
NOTA: Si la configuración del seguimiento es correcta, este paso debería ser necesario solo para un pequeño porcentaje de las celdas rastreadas. - Si la línea reportera de la microglía se utiliza en combinación con transgénicos fluorescentes que marcan otros tipos de células en el parénquima, realice la segmentación como un nuevo objeto de superficie o un objeto puntual para visualizarlos. Para ello, repita los pasos anteriores (de los pasos 3.2 a 3.14), modificando la entrada del Canal de Origen en el paso 3.5, para que sea el que se utilice para obtener la imagen de la segunda población de células, y cuidando de adaptar los parámetros descriptivos específicos de las células de interés, como el Diámetro XY Estimado (paso 3.5), la Distancia Máxima (paso 3.8) a la nueva población de células. Otros pasos se pueden repetir sin cambios.
NOTA: En el caso de la creación de una nueva superficie, el diámetro XY estimado se sustituye por una opción de umbral basada en la sustracción de fondo, donde la entrada que se debe proporcionar es el diámetro de la esfera más grande que cabe en el objeto. Este valor se puede estimar desde la vista de división en zonas utilizando la barra de herramientas Medir, como se explica en el paso 3.2. - Una vez satisfecho con los rastreos obtenidos, extraiga las estadísticas de rastreo deseadas de la pestaña Estadísticas . Al hacer clic en el botón de configuración , seleccione las estadísticas que se calcularán para cada objeto puntual u objeto de superficie creado anteriormente. En caso de que se hayan creado más objetos en el mismo análisis, dos objetos puntuales o un punto y un objeto de superficie, las estadísticas que representan la posición relativa de un objeto con respecto al otro, como la distancia más corta entre los dos, también estarán disponibles para exportar.
NOTA: Todos los valores deseados pueden ser valores específicos o promedio, y todos los datos sin procesar se pueden descargar como un archivo de valores separados por comas (CSV) usando los botones en la parte inferior de la pestaña Estadísticas . Siempre que el análisis también incluya otros tipos de celdas, también se dispondrá de estadísticas adicionales sobre la distancia relativa de un objeto al otro.
Resultados
Células microgliales que expresan proteína verde fluorescente (eGFP) y células endoteliales que expresan DsRed en Tg(mpeg1:eGFPgl22; kdrl:CRES898; actb2:loxP-STOP-loxP-DsRedexpress,sd5) Se obtuvieron imágenes de embriones transgénicos triples14 a 3 dpf, de acuerdo con el protocolo descrito. Un solo embrión de pez cebra se montó en agarosa de bajo punto de fusión al 1% en una placa de vidrio inferior y el proceso de imagen no obstaculizó el crecimiento del embrión durante el tiempo de adquisición. El lapso de tiempo se registró utilizando un sistema comercial de microscopía confocal de barrido puntual equipado con una lente de objetivo seco de 10x 0,45 NA, y se utilizaron láseres de excitación de 488 nm y 561 nm para obtener imágenes de la microglía y las células endoteliales, respectivamente. Además, el intervalo de tiempo, la resolución de la imagen, el tamaño de píxel y el paso z fueron de 30 segundos (s), 1024x1024, 0,49 μm y 2,5 μm, respectivamente. La grabación de timelapse duró 3,5 horas (h).
El campo de visión adquirido abarcó toda la cabeza del embrión, pero el análisis se centró específicamente en el tectum óptico, ya que durante la primera semana de desarrollo, la microglía se restringe principalmente a la capa de soma neuronal de esta región del mesencéfalo dorsal, lo que permite a los investigadores visualizar a toda la población simultáneamente15. El análisis de seguimiento 3D se realizó utilizando Imaris 10.0, como se explicó anteriormente. Como se muestra en la Figura 4, el seguimiento fue exitoso, lo que resultó en 25 pistas, coincidiendo con el número esperado de células de microglía presentes en el tectum óptico a 3 dpf16. Se requirió una corrección manual mínima de las pistas. En la figura 5 se muestra un ejemplo de los datos que se pueden extraer de un experimento de seguimiento exitoso. El marcaje simultáneo de macrófagos y células endoteliales permite cuantificar la posición de la microglía en relación con esta última, lo que permite a los investigadores visualizar la distancia relativa de cada célula a la célula endotelial más cercana en el tiempo y examinar la frecuencia y el número de interacciones potenciales (Figura 5).
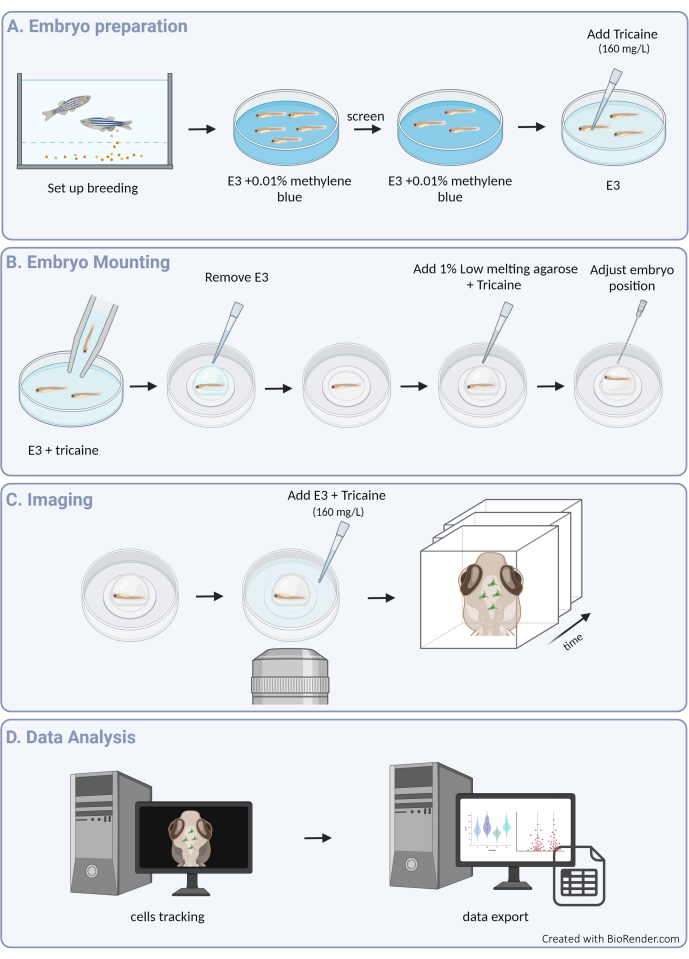
Figura 1: Resumen del procedimiento experimental. (A) Preparación y anestesia del embrión de pez cebra. (B) Montaje y posicionamiento de muestras. (C) Adquisición de imágenes. (D) Procesamiento de imágenes y extracción de datos de motilidad. Haga clic aquí para ver una versión más grande de esta figura.
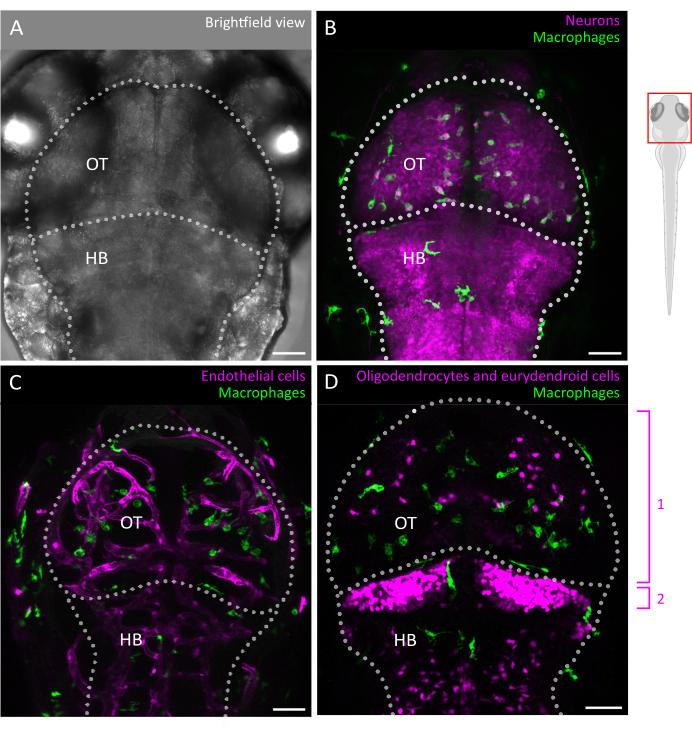
Figura 2: Imágenes de la microglía e interacciones con el entorno celular del cerebro en el embrión de pez cebra. (A) Imagen de campo claro de la cabeza y el cerebro de un pez cebra embrionario de 3 dpf (vista dorsal), marcando el tectum óptico y el cerebro posterior. El cerebro del pez cebra embrionario se puede fotografiar en su totalidad en esta etapa debido a su pequeño tamaño y transparencia óptica. (B-D) La ubicación y el comportamiento de la microglía se pueden visualizar mediante el uso de líneas transgénicas como (B,C) Tg(mpeg1:eGFP)gl22 y (D) Tg(mpeg1:mcherry)gl23, y centrándose en el OT. (B) Las neuronas y sus cuerpos celulares se pueden identificar utilizando la línea reportera Tg(XlTubb:D sRed)zf148 y las interacciones microglía-neurona se pueden visualizar fusionando los dos canales en líneas que co-expresan ambos transgenes. Se ve una fusión de las señales verde (microglía) y roja (neuronas, en magenta). (C,D) Las líneas reporteras también pueden arrojar luz sobre las interacciones de la microglía (en verde) con las células endoteliales, aquí marcadas en rojo usando el (C) doble Tg(kdrl:cres898; actb2:LOXP-STOP-LOXP-Dsredexpress, sd5) transgénicos, o con células gliales como los oligodendrocitos y sus precursores, utilizando el (D) doble Tg(olig2:EGFP; mpeg1:mcherry ) línea transgénica. 1: oligodendrocitos y células progenitoras en el OT, 2: neuronas eurydendroides en el cerebelo. Barras de escala = 50 μm. Abreviaturas: dpf = días después de la fecundación; OT = tectum óptico; HB = cerebro posterior. Haga clic aquí para ver una versión más grande de esta figura.
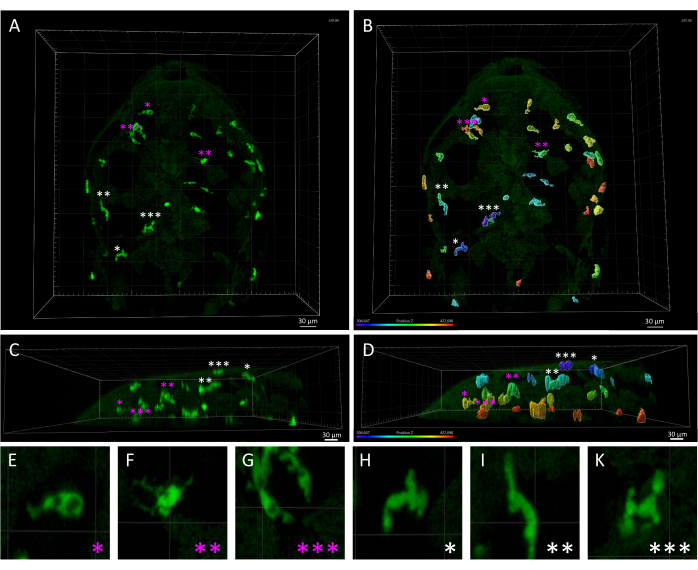
Figura 3: Disparidad morfológica y espacial entre los macrófagos de la piel y la microglía en un embrión de pez cebra de 3 dpf. (A,B) Vista dorsal de un embrión de pez cebra Tg(mpeg1:eGFP)gl22 a 3 dpf, que representa (A) células de microglía parenquimatosas (marcadas con asteriscos magenta) frente a macrófagos de la piel (asteriscos blancos), identificados en función de su posición relativa a lo largo del eje z, como se ve en (B), que muestra la imagen 3D monocromática renderizada con codificación de colores del eje z. (C,D) Vista lateral de (C) A y (D) B, que muestra las profundidades z de las células mpeg1:eGFP+ dentro de la cabeza del embrión y destaca la localización superficial de los macrófagos de la piel en comparación con las células microgliales. (E-K) Gran aumento de cada célula indicado por un asterisco en A y B, proporcionando una visualización detallada de las distintas morfologías entre la microglía ameboide (E-G) y los macrófagos alargados de la piel (H-K). Barras de escala= 30 μm. Abreviatura: dpf = días después de la fecundación. Haga clic aquí para ver una versión más grande de esta figura.
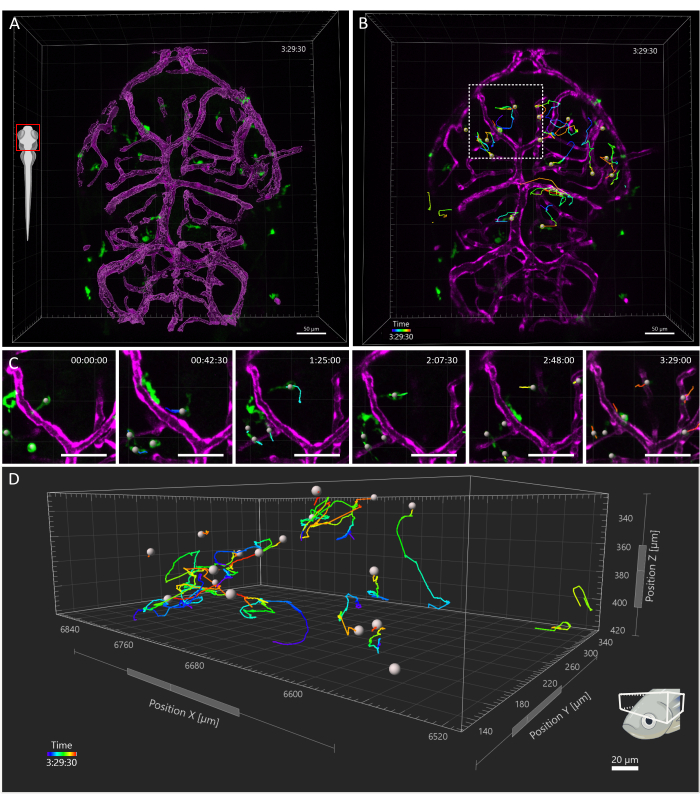
Figura 4: Seguimiento de células microgliales en un embrión de pez cebra de 3 dpf. (A) Vista dorsal de la cabeza de un Tg de 3 dpf(kdrl:cres898; actb2: LOXP-STOP-LOXP-Dsredexpress, sd5; mpeg1:eGFPgl22) embrión transgénico triple, que muestra microglía (verde) y la representación superficial de los vasos (magenta). (B-D) Seguimiento representativo de los movimientos de la microglía durante una ventana de tiempo de (B) 3,5 h. Se observa que las células individuales siguen trayectorias complejas. (C) Detalle de timelapse, que muestra una vista ampliada de la región en B encerrada por el cuadrado discontinuo. Se presentan seis fotogramas de la película (con 45 minutos de diferencia), que documentan el establecimiento de contactos transitorios de la microglía con las células endoteliales en su microambiente. (D) Trayectorias de migración de células microgliales individuales dentro del tectum óptico, con los ejes X, Y y Z que representan dimensiones espaciales. Barra de escala = 50 μm. Abreviatura: dpf = días después de la fecundación. Haga clic aquí para ver una versión más grande de esta figura.
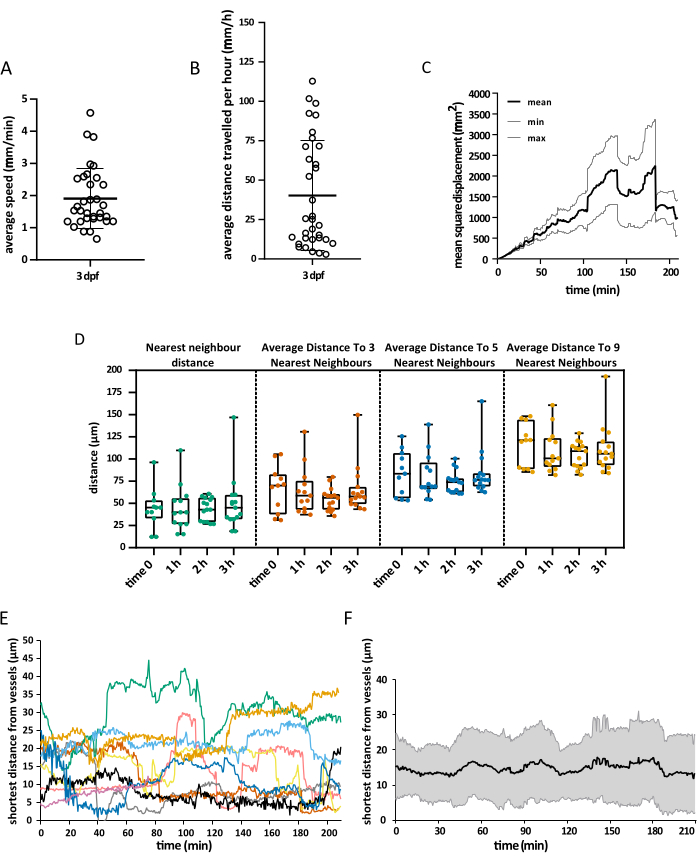
Figura 5: Visualización de los datos obtenidos. Ejemplo de los datos espaciales que se pueden obtener utilizando el protocolo descrito. Los gráficos muestran (A) la velocidad promedio de las células microgliales, (B) la distancia promedio que cubren en 1 h, (C) su desplazamiento cuadrático medio y (D) su distribución en el espacio en diferentes momentos. La representación de la superficie del vaso también permitió la medición de la distancia más corta entre la microglía y las células endoteliales en un momento dado, tanto a nivel de una sola célula (E) como como promedio global (F). Para A, B y D, cada punto representa una celda individual. N = un embrión. Haga clic aquí para ver una versión más grande de esta figura.
Archivo complementario 1: Haga clic aquí para descargar este archivo.
Discusión
El protocolo actual permite la obtención de imágenes in vivo de la dinámica de la microglía en un embrión de vertebrado y la visualización de los datos de motilidad adquiridos. La colonización de la microglía del cerebro en desarrollo ocurre muy temprano durante la embriogénesis y precede a eventos críticos como picos de neurogénesis, astrogliogénesis, oligodendrogénesis y muchos otros procesos celulares17. Por lo tanto, no es sorprendente que la microglía desempeñe funciones importantes en la configuración de aspectos específicos del desarrollo cerebral18, por ejemplo, a través de la regulación de la diferenciación neuronal, la migración y la supervivencia 19,20,21, así como la poda sináptica 5 y la mielinización 22,23,24.
La contribución de la microglía disfuncional a la patogénesis y/o progresión de los trastornos del neurodesarrollo también está siendo cada vez más reconocida25. De hecho, la presencia temprana de microglía en el cerebro en formación expone a estas células a distintos estados fisiológicos26 y cambios ambientales. Esto puede tener un impacto significativo dado que la microglía es una célula longeva tanto en roedores como en humanos, manteniéndose durante toda la vida a través de la autorrenovación de los progenitores locales 27,28,29. Creemos que este protocolo podría servir como una herramienta poderosa para caracterizar mejor el comportamiento de la microglía en estos estados fisiológicos distintos, a medida que se desarrollan, maduran y establecen su red durante los pasos sucesivos de la morfogénesis cerebral.
Usando la configuración descrita aquí, hemos obtenido con éxito imágenes y datos sobre larvas de pez cebra de hasta 6 dpf. Es probable que la extensión del análisis a etapas posteriores del desarrollo tenga éxito, pero requerirá ajustar la configuración de las imágenes para tener en cuenta el mayor tamaño de la muestra, especialmente a lo largo del eje z. Al intentar esto, sugerimos centrarse en mantener una relación señal-ruido baja y un tiempo de escaneo rápido, ya que son parámetros clave para un análisis exitoso.
Sugerimos un tiempo mínimo de imagen de 1 h para permitir el seguimiento de la microglía; La ventana de imagen más larga que se ha probado con este protocolo es de 8 h. Además, es importante que el análisis de seguimiento mantenga el intervalo de tiempo entre fotogramas lo más corto posible, idealmente entre 30 s y 60 s. Esto permitirá obtener datos de seguimiento más precisos y detallados en los análisis posteriores. Por lo tanto, especialmente si se detecta más de un fluoróforo, es fundamental evitar la superposición espectral y garantizar una separación suficiente entre los dos espectros de emisión de fluoróforos para permitir una adquisición simultánea, sin fuga de señal.
Existen otros protocolos para la grabación de alta calidad del cerebro del pez cebra30, pero este es el primero que muestra cómo rastrear con éxito todo el movimiento de la microglía durante el desarrollo embrionario durante un período prolongado. Aunque el flujo de trabajo presentado aquí se centró en el seguimiento de la microglía en un contexto fisiológico, se puede aplicar fácilmente al análisis de la microglía en patología. De hecho, se han establecido varios modelos de trastornos del neurodesarrollo, como el autismo31, la epilepsia32 y la esquizofrenia33, pero también la neurodegeneración34 y el cáncer35, que brindan oportunidades únicas para determinar la respuesta y el comportamiento de la microglía en condiciones de enfermedad.
En particular, este protocolo de seguimiento es muy versátil y también podría ser fundamental para arrojar luz sobre los patrones de migración de varios tipos de células a través de diversas regiones anatómicas del embrión de pez cebra, lo que podría abrir vías para aplicaciones adicionales, más allá del alcance de la investigación microglial descrito en este artículo. Además, al aprovechar la capacidad de combinar múltiples líneas transgénicas fluorescentes, obtenemos la capacidad de discernir la relación espacial entre la microglía y otros tipos de células del microambiente cerebral, con el potencial de visualizar las interacciones celulares y las conversaciones cruzadas a lo largo de las grabaciones de lapso de tiempo, de una manera no invasiva. Esto podría ser fundamental para desentrañar la importancia fisiológica del comportamiento de la microglía y contribuir a una caracterización más profunda de estas células altamente móviles.
Divulgaciones
Los autores declaran no tener conflictos de intereses.
Agradecimientos
Los autores desean expresar su más sincero agradecimiento al profesor Nicolas Bayens por proporcionar generosamente acceso al microscopio confocal esencial para este estudio. Este trabajo fue financiado en parte por los Fondos para la Investigación Científica (FNRS) bajo los números de subvención F451218F y UG03019F, la Fundación para la Investigación del Alzheimer (SAO-FRA) (a V.W.), A.M. cuenta con el apoyo de una beca de investigación del FNRS. La Figura 1 se creó en biorender.com.
Materiales
| Name | Company | Catalog Number | Comments |
| 1 L Breeding tanks | Tecniplast | ZB10BTE | |
| 1-phenyl-2-thiourea (PTU) | Sigma-Aldrich | P7629 | Diluted to 0.2 mM in E3 to prevent embryo pigmentation |
| Bottom glass imaging dish | FluoroDish | FD3510-100 | |
| Disposable Graduated transfer pipette | avantor | 16001-188 | |
| Dry block heater | Novolab | Grant QBD4 | To keep low melting agarose at 37 °C |
| Ethyl 3-aminobenzoate methanesulfonate (Tricaine) | Sigma-Aldrich | E10521-50G | |
| Imaris 10.0 | Oxford Instruments | analysis software | |
| Imaris File Converter | Oxford Instruments | https://imaris.oxinst.com/big-data | |
| Laser-scanning confocal microscope | Nikon | Eclipse Ti2-E | |
| Methylene blue | Sigma-Aldrich | M9140-25G | |
| microloader tips | Eppendorf | 5242956003 | |
| NuSieve GTG Agarose | Lonza | 50081 | |
| Petri dishes (90 mm) | avantor | 391-0559 | |
| Pronase | Sigma-Aldrich | 11459643001 | |
| Stainless Steel Forceps Dumont No. 5 | FineScienceTools | 11254-20 | |
| Stereo microscope | Leica | Leica M80 | To mount the embryos |
| teasing needle | avantor | 76549-024 |
Referencias
- Wolf, S. A., Boddeke, H. W., Kettenmann, H. Microglia in physiology and disease. Annu Rev Physiol. 79, 619-643 (2017).
- Nimmerjahn, A., Kirchhoff, F., Helmchen, F. Resting microglial cells are highly dynamic surveillants of brain parenchyma in vivo. Sci Rep. 308 (5726), 1314-1318 (2005).
- Peri, F., Nusslein-Volhard, C. Live imaging of neuronal degradation by microglia reveals a role for v0-ATPase a1 in phagosomal fusion in vivo. Cell. 133 (5), 916-927 (2008).
- Marın-Teva, J. L., et al. Microglia promote the death of developing Purkinje cells. Neuron. 41 (4), 535-547 (2004).
- Paolicelli, R. C., et al. Synaptic pruning by microglia is necessary for normal brain development. Science. 333 (6048), 1456-1458 (2011).
- Hattori, Y. The microglia-blood vessel interactions in the developing brain. Neurosci Res. 187, 58-66 (2023).
- Colonna, M., Butovsky, O. Microglia function in the central nervous system during health and neurodegeneration. Annu Rev Immunol. 35, 441-468 (2017).
- Xu, H. -T., Pan, F., Yang, G., Gan, W. -B. Choice of cranial window type for in vivo imaging affects dendritic spine turnover in the cortex. Nat Neurosci. 10 (5), 549-551 (2007).
- Shin, J., Park, H. -C., Topczewska, J. M., Mawdsley, D. J., Appel, B. Neural cell fate analysis in zebrafish using olig2 BAC transgenics. Methods Cell Sci. 25 (1), 7-14 (2003).
- Chi, N. C., et al. Foxn4 directly regulates tbx2b expression and atrioventricular canal formation. Genes Dev. 22 (6), 734-739 (2008).
- Avdesh, A., et al. Regular care and maintenance of a zebrafish (Danio rerio) laboratory: An introduction. J Vis Exp. (69), e4196(2012).
- Bohnsack, B. L., Gallina, D., Kahana, A. Phenothiourea sensitizes zebrafish cranial neural crest and extraocular muscle development to changes in retinoic acid and IGF signaling. PLoS ONE. 6 (8), e22991(2011).
- Ariga, J., Walker, S. L., Mumm, J. S. Multicolor time-lapse imaging of transgenic zebrafish: visualizing retinal stem cells activated by targeted neuronal cell ablation. J Vis Exp. (43), e2093(2010).
- Ferrero, G., et al. Embryonic microglia derive from primitive macrophages and are replaced by cmyb-dependent definitive microglia in zebrafish. Cell Rep. 24 (1), 130-141 (2018).
- Xu, J., Wang, T., Wu, Y., Jin, W., Wen, Z. Microglia colonization of developing zebrafish midbrain is promoted by apoptotic neuron and lysophosphatidylcholine. Dev Cell. 38 (2), 214-222 (2016).
- Svahn, A. J., et al. Development of ramified microglia from early macrophages in the zebrafish optic tectum. Dev Neurobiol. 73 (1), 60-71 (2013).
- Thion, M. S., Garel, S. Microglial ontogeny, diversity and neurodevelopmental functions. Curr Opin Genet Dev. 65, 186-194 (2020).
- Bohlen, C. J., Friedman, B. A., Dejanovic, B., Sheng, M. Microglia in brain development, homeostasis, and neurodegeneration. Annu Rev Genet. 53, 263-288 (2019).
- Squarzoni, P., et al. Microglia modulate wiring of the embryonic forebrain. Cell Rep. 8 (5), 1271-1279 (2014).
- Schafer, D. P., et al. Microglia sculpt postnatal neural circuits in an activity and complement-dependent. Neuron. 74 (4), 691-705 (2012).
- Parkhurst, C. N., et al. Microglia promote learning-dependent synapse formation through brain-derived neurotrophic factor. Cell. 155 (7), 1596-1609 (2013).
- Green, L. A., O'Dea, M. R., Hoover, C. A., DeSantis, D. F., Smith, C. J. The embryonic zebrafish brain is seeded by a lymphatic-dependent population of mrc1+ microglia precursors. Nat. Neurosci. 25 (7), 849-864 (2022).
- Hughes, A. N., Appel, B. Microglia phagocytose myelin sheaths to modify developmental myelination. Nat Neurosci. 23 (9), 1055-1066 (2020).
- Yu, Q., et al. C1q is essential for myelination in the central nervous system (CNS). iScience. 26 (12), 108518(2023).
- Zengeler, K. E., Lukens, J. R. Innate immunity at the crossroads of healthy brain maturation and neurodevelopmental disorders. Nat Rev Immunol. 21 (7), 454-468 (2021).
- Paolicelli, R. C., et al. Microglia states and nomenclature: A field at its crossroads. Neuron. 110 (21), 3458-3483 (2022).
- Réu, P., et al. The lifespan and turnover of microglia in the human brain. Cell Rep. 20 (4), 779-784 (2017).
- Ajami, B., Bennett, J. L., Krieger, C., Tetzlaff, W., Rossi, F. M. V. Local self-renewal can sustain CNS microglia maintenance and function throughout adult life. Nat Neurosci. 10 (12), 1538-1543 (2007).
- Mildner, A., et al. Microglia in the adult brain arise from Ly-6ChiCCR2+ monocytes only under defined host conditions. Nat Neurosci. 10 (12), 1544-1553 (2007).
- Cook, Z. T., Brockway, N. L., Weissman, T. A. Visualizing the developing brain in living zebrafish using Brainbow and time-lapse confocal imaging. J Vis Exp. (157), e60593(2020).
- Tayanloo-Beik, A., et al. Zebrafish modeling of autism spectrum disorders, current status and future prospective. Front Psychiatry. 13, 911770(2022).
- D'Amora, M., et al. Zebrafish as an innovative tool for epilepsy modeling: State of the art and potential future directions. Int J Mol Sci. 24 (9), 7702(2023).
- Langova, V., Vales, K., Horacek, J. The role of zebrafish and laboratory rodents in schizophrenia research. Front Psychiatry. 11, 549232(2020).
- Chia, K., Klingseisen, A., Sieger, D., Priller, J. Zebrafish as a model organism for neurodegenerative disease. Front Mol Neurosci. 15, 940484(2022).
- Astell, K. R., Sieger, D. Zebrafish in vivo models of cancer and metastasis. Cold Spring Harb Perspect Med. 10 (8), 037077(2020).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados