Method Article
Live-Bildgebung und Charakterisierung der Mikroglia-Dynamik im Zebrafischembryo
In diesem Artikel
Zusammenfassung
Wir demonstrieren eine Methode, die die Vorteile der schnellen konfokalen Rastermikroskopie nutzt, um Live-Bildgebung von Mikrogliazellen im sich entwickelnden optischen Tectum des Zebrafisches durchzuführen und so die Dynamik dieser Zellen in vivo zu analysieren.
Zusammenfassung
Mikroglia sind hochdynamische Zellen und ihre Wanderung und Besiedlung des Hirnparenchyms ist ein entscheidender Schritt für die richtige Entwicklung und Funktion des Gehirns. Extern entwickelnde Zebrafischembryonen besitzen eine optische Transparenz, die zusammen mit gut charakterisierten transgenen Reporterlinien, die Mikroglia fluoreszierend markieren, den Zebrafisch zu einem idealen Wirbeltiermodell für solche Studien macht. In dieser Arbeit nutzen wir die einzigartigen Eigenschaften des Zebrafischmodells, um die Dynamik von Mikrogliazellen in vivo und unter physiologischen Bedingungen zu visualisieren. Wir verwenden die konfokale Mikroskopie, um einen Zeitraffer von Mikrogliazellen im optischen Tectum des Zebrafischembryos aufzuzeichnen und dann Tracking-Daten mit der IMARIS 10.0-Software zu extrahieren, um den Migrationspfad der Zellen, die mittlere Geschwindigkeit und die Verteilung im optischen Tectum in verschiedenen Entwicklungsstadien zu erhalten. Dieses Protokoll kann ein nützliches Werkzeug sein, um die physiologische Bedeutung des Verhaltens von Mikroglia in verschiedenen Kontexten aufzuklären und zu einer tieferen Charakterisierung dieser hochbeweglichen Zellen beizutragen.
Einleitung
Als residente Makrophagen im Zentralnervensystem (ZNS) stellen Mikroglia eine eigenständige nicht-neuronale Population dar, die bis zu 15 % aller Gliazellen im erwachsenen Gehirn ausmacht. Das Studium der Mikroglia-Biologie hat in den letzten Jahren aufgrund ihrer etablierten Bedeutung für Entwicklung, Physiologie und Krankheit zunehmend an Bedeutung gewonnen1. Unter physiologischen Bedingungen sind Mikrogliazellen hochdynamisch und überwachen kontinuierlich das Hirnparenchym 2,3. Dieses Verhalten ermöglicht es Mikroglia, das Gehirn zu besiedeln und eine entscheidende Rolle in seiner Entwicklung zu spielen, wie z. B. die Gestaltung neuronaler Schaltkreise4, der synaptischen Beschneidung5 und der Vaskulogenese6. Darüber hinaus ermöglicht diese inhärente dynamische Natur den Mikroglia, das ZNS ständig auf Anzeichen von Infektionen, Verletzungen oder Abweichungen von der Homöostase zu überwachen7. Um diese komplizierte Zelldynamik zu entschlüsseln, ist die Live-Bildgebung von Mikroglia über Raum und Zeit hinweg unerlässlich. Glücklicherweise positioniert die optische Transparenz von sich extern entwickelnden Zebrafischembryonen, gepaart mit der Verfügbarkeit gut charakterisierter transgener Reporterlinien, die Mikroglia fluoreszierend markieren, den Zebrafisch als ideales Wirbeltiermodell für solche Untersuchungen. Die Live-Bildgebung in Zebrafischembryonen bietet einen nicht-invasiven Ansatz, der keine Operation oder umfangreiche Gewebemanipulation erfordert, wodurch mögliche Störungen des ZNS-Status minimiert werden. Dies ist ein wichtiger Aspekt bei der Untersuchung von Mikrogliazellen, da sie selbst auf subtile Veränderungen in der extrazellulären Umgebung sehr empfindlich reagieren8.
Hier stellen wir einen Leitfaden zur erfolgreichen Verfolgung von 3D-Mikrogliazellbewegungen im Zebrafischembryo vor, der einen beispiellosen Überblick über das Verhalten von Mikroglia innerhalb der intakten Architektur des sich entwickelnden Hirnparenchyms ermöglicht (siehe Abbildung 1 für einen grafischen Überblick über das Protokoll). Dieses Schritt-für-Schritt-Protokoll beschreibt, wie man Mikroglia von Zebrafischen in verschiedenen Entwicklungsstadien einrichtet und abbildet und wie man hochauflösende Daten über die Beweglichkeit von Mikrogliazellen extrahiert, um wertvolle Einblicke in ihre Migrationsmuster und Reaktionen auf Umwelteinflüsse zu erhalten. Wir zeigen auch, dass dieses Protokoll für die Durchführung von Live-Mehrfarben-Bildgebung angepasst werden kann, wodurch seine Anwendbarkeit auf die Untersuchung von Mikroglia in Kombination mit transgenen Linien erweitert wird, die benachbarte Zellen markieren, einschließlich Neuronen3, Oligodendrozyten9 und Endothelzellen10 (wie in Abbildung 2 gezeigt). Durch die Erweiterung des Werkzeugkastens, der es ermöglicht, die Dynamik des Verhaltens von Mikroglia in Echtzeit und in ihrer natürlichen Umgebung direkt zu beobachten und zu charakterisieren, wird dieses Protokoll wahrscheinlich dazu beitragen, die Funktionalität der Mikroglia während der frühen Entwicklung, sowohl in der Physiologie als auch in der Krankheit, besser aufzuklären.
Protokoll
Die Zebrafische wurden unter Standardbedingungen nach FELASA42 und institutionellen (Université Libre de Bruxelles, Brüssel, Belgien; ULB) Richtlinien und Vorschriften. Alle experimentellen Verfahren wurden von der ULB-Ethikkommission für Tierschutz (CEBEA) der ULB genehmigt.
1. Zebrafischhaltung und Embryonenvorbereitung
HINWEIS: Die transgene Zebrafischlinie Tg(mpeg1:eGFP)gl22 , die das fluoreszierende eGFP-Protein in Makrophagen, einschließlich Mikroglia, exprimiert, wurde verwendet, um die in diesem Protokoll gezeigten Tracking-Daten zu generieren. Andere Makrophagen-Reporterlinien sind über ZIRC erhältlich und können ebenfalls verwendet werden. Bei der Auswahl der transgenen Linie ist zu berücksichtigen, dass eine hohe Signalintensität die Bildaufnahme und Zellsegmentierung erleichtert.
- Halten Sie Zebrafische (Danio rerio) bei einer Temperatur von 28 °C und halten Sie sie gemäß den festgelegten Protokollen11.
- Stimulieren Sie die Paarung, indem Sie weibliche und männliche Fische am späten Nachmittag vor dem Geburtsdatum der gewünschten Embryonen zusammen in ein Zuchtbecken mit Netzboden und Separator setzen. Entfernen Sie den Separator am nächsten Morgen, um die Paarung der Fische zu planen.
- Sammeln Sie die befruchteten Eizellen mit einem Netz und geben Sie sie in eine 100 mm Petrischale, die E3-Medium (5 mM NaCl, 0,17 mM KCl, 0,33 mM CaCl2, 0,33 mM MgSO4) mit 0,01 % Methylenblau enthält. Wenn die Pigmentbildung gehemmt werden soll, um die Bildgebung zu erleichtern, fügen Sie je nach genetischem Hintergrund der verwendeten transgenen Zebrafischlinie 0,003 % N-Phenylthiharnstoff (PTU) zu dem E3-Puffer hinzu, in dem sich die Embryonen entwickeln, beginnend 6 h nach der Befruchtung (hpf) und erfrischen Sie die Lösung alle 2 Tage, bis das gewünschte Entwicklungsstadium erreicht ist.
HINWEIS: Um eine lange Exposition gegenüber PTU zu vermeiden, die die Embryonalentwicklungbeeinträchtigen könnte 12, wird empfohlen, falls verfügbar, eine transgene Linie auf einem pigmentarmen Hintergrund zu verwenden, wie z. B. Casperw2, die eine klare Bildgebung ermöglicht, ohne dass Embryonen über einen längeren Zeitraum PTU ausgesetzt werden müssen13. - Wenn die abzubildenden Embryonen jünger als 3 Tage nach der Befruchtung (dpf) sind, dechorionieren Sie sie bei 24 hpf. Bestätigen Sie mit einem Stereomikroskop, dass alle Embryonen dechorioniert wurden und durch die enzymatische Behandlung nicht geschädigt wurden (siehe Schritt 1.4.2).
- Bei einer begrenzten Anzahl von Eizellen tun Sie dies manuell, indem Sie das Chorion mit einer feinen Präparierzange zusammendrücken und nach außen ziehen, wobei Sie jeden Embryo vorsichtig aus seinem Chorion lösen.
- Bei einer höheren Anzahl von Eiern führen Sie die Dechorionierung enzymatisch durch, indem Sie die Eier in loser Schüttung bei Raumtemperatur (RT) in einer 1 mg/ml-Pronaselösung in E3-Medium für einen kurzen Zeitraum (typischerweise 5-10 Minuten, bis die Chorionen unter leichtem Schwenken aufbrechen) inkubieren. Als nächstes wird die Pronase-Lösung verworfen und die Embryonen werden 3x gründlich mit frischem E3 gespült, um das restliche Chorion und das restliche Enzym zu entfernen.
2. Zebrafisch-Montage
- Bereiten Sie eine 1%ige niedrigschmelzende Agaroselösung in E3-Puffer vor. Kühlen Sie die Agarose in einem Tischheizblock auf 37 °C ab und fügen Sie frisch hergestellte Tricain-Methansulfonat-Lösung (160 mg/l) hinzu, die dazu beiträgt, dass die Embryonen während der Bildgebung betäubt bleiben. Wirbeln Sie die Lösung vor, um sie gründlich zu homogenisieren.
- Betäuben Sie die Embryonen, indem Sie 160 mg/l Tricain-Methansulfonat in die Petrischale geben, die die Embryonen in E3-Puffer enthält. Vergewissern Sie sich, dass die Bewegung gestoppt wurde, und wählen Sie die Zebrafischembryonen mit der gewünschten Fluoreszenz aus. Überprüfen Sie die Gesundheit des Embryos, indem Sie seinen Herzschlag mit einem Hellfeld-Stereomikroskop visualisieren. Entnehmen Sie mit einer Transferpipette mit breiter Spitze einen oder bis zu drei der ausgewählten Embryonen und übertragen Sie sie auf eine Speicherfolie aus Bodenglas.
- Verwenden Sie unter einem Stereomikroskop eine Pipette, um so viel Medium wie möglich zu entfernen, und achten Sie darauf, die Embryonen nicht zu berühren oder zu beschädigen. Ersetzen Sie dann das Medium durch die zuvor vorbereitete 1%ige niedrigschmelzende Agaroselösung. Als nächstes schieben Sie jeden Embryo mit einer dünnen, sich verjüngenden Kunststoffspitze, die am Ende einer Nadel befestigt ist, vorsichtig an den Boden der Speicherfolie und richten Sie sie so aus, dass ihre dorsale Seite zum Glas zeigt, um eine gute Sicht auf das optische Tectum und eine minimale Menge an niedrigschmelzender Agarose zwischen dem Embryo und dem Glas zu gewährleisten.
- Bilden Sie den montierten Embryo umgehend mit einem inversen konfokalen Mikroskop ab, das mit einem hochwertigen Plan Apo Objektiv ausgestattet ist (10x/0,45 Plan Apo oder hier ein 20x/0,75 Plan Apo). Wenn der Zeitraffer über einen längeren Zeitraum (über eine Stunde) läuft, geben Sie ein oder zwei Tropfen E3-Puffer mit Tricain-Methansulfonat (160 mg/L) auf die untere Glas-Speicherfolie, um zu verhindern, dass die niedrigschmelzende Agarose übermäßig austrocknet.
HINWEIS: Es ist wichtig, zu warten, bis sich die Agarose vollständig verfestigt hat, bevor E3-Puffer und Tricain-Methansulfonat-Medien zur Bildgebungsscheibe hinzugefügt werden. Andernfalls könnten sich die Fische langsam seitlich und axial bewegen, was zu einer Probenabdrift im Zeitraffer führt. - Fahren Sie mit der Einrichtung der Zeitrafferaufnahme für die gewünschte Zeit fort: Stellen Sie die Bildauflösung auf 1.024 x 1.024 mit einer Pixelgröße von 0,49 μm ein. Sammeln Sie optische Schichten in Schritten von 2-3 μm, um eine akzeptable Auflösung auf der z-Achse zu gewährleisten. Im Zebrafischembryo wird das optische Tectum je nach verwendetem Entwicklungsstadium in einen 100-150 μm Z-Stack eingeschlossen. Um eine erfolgreiche Zellverfolgung zu gewährleisten, halten Sie das Zeitintervall zwischen den Bildern zwischen 30 s und 60 s.
3. Tracking-Analyse und Datenexport
- Lassen Sie die Analysesoftware nach dem Start in der Arena starten. Klicken Sie auf Ordner beobachten , um die zuvor konvertierte Datei zu öffnen. Nachdem die Datei in die Software geladen wurde, scrollen Sie mit dem Schieberegler in der Segment-Symbolleiste links neben dem Ansichtsbereich in der Segmentansicht durch den Z-Stapel, und messen Sie einige Zellendurchmesser, indem Sie den Durchmesser der ausgewählten Zellen mit dem Mauszeiger zeichnen. Klicken und ziehen Sie die Maus, um ein Segment zu zeichnen, das sich über den Zellkern erstreckt. Beobachten Sie die Länge des Segments, das in der Werkzeugleiste "Messen" auf der rechten Seite des Ansichtsbereichs gezeichnet wird.
HINWEIS: Mit den Bildgebungseinstellungen in Schritt 2.5 kann mit einem Zellkörperdurchmesser zwischen 6 μm und 8 μm gerechnet werden. - Um die Zellen automatisch zu erkennen, gehen Sie zurück zur 3D-Ansicht und klicken Sie auf das Punktsymbol in der Objektsymbolleiste , um ein neues Spots-Objekt in der Objektliste hinzuzufügen und den automatischen Spot-Erstellungsassistenten zu öffnen. Aktivieren Sie im ersten Schritt des Assistenten die Option, nur einen Interessenbereich zu segmentieren. Aktivieren Sie dann die Option Spots verfolgen (im Zeitverlauf), um die Tracking-Daten zu berechnen, sowie die Objekt-Objekt-Statistik , um einen Vergleich zwischen Spots zu ermöglichen und den Bereich der Daten zu erweitern, die später in der Analyse verfügbar sein werden. Fahren Sie abschließend im Assistenten fort, indem Sie auf die Schaltfläche Weiter unten rechts im Assistentenfenster klicken.
- Im zweiten Schritt spezifizieren Sie einen ROI-Bereich (Region of Interest), der das optische Tectum des Embryos umschließt. Ändern Sie die Größe des ROI, indem Sie auf die kleinen weißen Pfeile klicken und diese ziehen, die auf jeder Fläche vorhanden sind. Sobald die ROI-Größe definiert ist, erweitern Sie sie auf alle aufgezeichneten Frames, indem Sie das Zeitintervall anpassen.
- Wählen Sie dann als Quellkanal den Kanal aus, der zur Erkennung der Zellen verwendet wird, und stellen Sie die zuvor in Schritt 3.2 ermittelte Messung des Zelldurchmessers auf den geschätzten XY-Durchmesser ein. Aktivieren Sie die Option Hintergrundsubtraktion und klicken Sie auf Weiter.
- An dieser Stelle des Spot-Erstellungsassistenten enthält die Software einen Qualitätsfilterschritt, bei dem Spots nach der Signalintensität gefiltert werden, die in der Mitte jedes Spots gemessen wird. in dem Kanal, in dem die Flecken entdeckt wurden. Passen Sie den Intensitätsschwellenwert an, indem Sie den Wert direkt in das Datenfeld für die unteren und oberen Schwellenwertfelder eingeben oder indem Sie die entsprechenden farbigen Linien im Histogramm am unteren Rand des Erstellungsassistenten ziehen, so dass alle Zellen erkannt werden, auch diejenigen, die tiefer im Gewebe lokalisiert sind und normalerweise eine geringere Signalintensität aufweisen. Alle diese Änderungen sind gleichzeitig im Ansichtsbereich sichtbar.
HINWEIS: Es ist möglich, an dieser Stelle mehrere Punkte zu beobachten, die eine einzelne Zelle beschriften, aber dies kann später in der Analyse korrigiert werden.
Wenn in längeren Zeitraffern Photobleaching zu sehen ist, überprüfen Sie vor dem Festlegen des Schwellenwerts den Ansichtsbereich mehrerer Frames, um sicherzustellen, dass alle Zellen auch in den neuesten und daher schwächeren Frames erkannt werden. - Wenn Sie zufrieden sind, klicken Sie auf Weiter , um den ausgewählten Qualitätsschwellenwert zu validieren. Alle Punkte, die den Parameter für den Qualitätsschwellenwert erfüllen und deren Durchmesser mit dem geschätzten XY-Durchmesser übereinstimmt, werden als Punkte gekennzeichnet. Visualisieren Sie dies als Vorschau im Ansichtsbereich, in der Spots, die diese beiden Parameter erfüllen, auf dem Quellkanal überlagert werden.
- Wählen Sie im nächsten Schritt die Option Autoregressive Bewegung als Tracking-Algorithmus aus, um Zellen mit einer mehr oder weniger kontinuierlichen Bewegung zu verfolgen. Beurteilen Sie die längste Entfernung, die eine Zelle zwischen zwei Zeitpunkten zurücklegt, indem Sie die Bewegung der Punkte zwischen zwei Frames sorgfältig beobachten, und geben Sie eine geschätzte Zahl in das Feld Maximale Entfernung ein. Dies ist die Entfernung, die ein Spot von der vorhergesagten zukünftigen Position abweichen darf.
- Legen Sie die maximale Lückengröße auf die geringste Anzahl von Zeitpunkten fest, in denen der Spot verschwinden kann, ohne den Track zu unterbrechen. Wenn das Intervall zwischen den Frames unter 60 s liegt, verwenden Sie eine Größe von 3. Wenn das Zeitintervall zwischen den Frames länger ist, erhöhen Sie die maximal zulässige Lückengröße, um die Spurfragmentierung zu verringern.
- Aktivieren Sie die Option Lücken mit allen erkannten Objekten füllen , um den Erkennungsschwellenwert nahe der vom Tracking-Algorithmus vorhergesagten erwarteten Position zu senken und Spuren zu verbinden, die durch einen Fehler der Punkterkennung unterbrochen werden. Klicken Sie abschließend auf Weiter und lassen Sie die Software automatisch Tracks für alle zuvor generierten Spots generieren.
- Wenn die erhaltenen Spuren zu zahlreich oder fragmentiert erscheinen, gehen Sie im Erstellungsassistenten zurück, indem Sie auf die Schaltfläche Zurück klicken, und passen Sie den zuvor definierten maximalen Abstand an, indem Sie die Anzahl entweder erhöhen oder verringern, bis die meisten der erhaltenen Spuren repräsentativ für die Zellenbewegungen sind.
HINWEIS: Die Anzahl der Spuren sollte in etwa der Anzahl der Mikrogliazellen entsprechen und daher je nach Alter des Embryos zwischen 30 und 50 liegen. Vor Abschluss der Analyse ist jedoch mit einer höheren Anzahl aufgrund fehlerhafter Spuren zu rechnen. - Sobald die Tracks berechnet wurden, enthält die Software einen Filterschritt, um alle sehr kurzen Tracks herauszufiltern, da sie oft ungenau oder nicht informativ sind. Schließen Sie Tracks aus, die kürzer als 3-8 Minuten (min) sind, indem Sie den Wert direkt in das Datenfeld für die Untergrenze des Filters für die Trackdauer eingeben oder indem Sie die entsprechenden farbigen Linien im Histogramm ziehen, das am unteren Rand des Erstellungsassistenten angezeigt wird. Klicken Sie auf Weiter , um den Filter zu validieren und im Erstellungsassistenten fortzufahren.
HINWEIS: Wie bereits erwähnt, ist es durch Ändern des Schwellenwerts für die maximale (max) und minimale (min) Trackdauer möglich, eine Vorschau der Tracks anzuzeigen, die gefiltert und von der Analyse ausgeschlossen werden. Die gefilterten Spuren können dann zur weiteren Analyse in einem neuen Objekt dupliziert werden. Der genaue Zeitschwellenwert für die Filterung kann je nach den Ergebnissen des vorherigen Schritts des Erstellungsassistenten variieren. Wir empfehlen, diese Filtereinstellungen zu optimieren, indem Sie die Vorschau im Ansichtsbereich überprüfen, um optimale Ergebnisse zu erzielen. - Da die transgene Linie Tg(mpeg1:eGFP) alle mononukleären Phagozyten markiert, werden neben Mikroglia auch Hautmakrophagen in Zeitrafferfilmen nachgewiesen. Entfernen Sie die von Hautmakrophagen erzeugten Spuren manuell aus der Analyse, um sich auf die Hirnparenchymal-Mikroglia zu konzentrieren (Abbildung 3).
- Korrigieren Sie andere potenzielle Fehler in den Spuren manuell im Spureditor, wo jeder Datenpunkt als Objekt in der Spur angezeigt wird. Aktivieren Sie die Schaltfläche für den Kreisauswahlmodus rechts neben dem Ansichtsbereich und markieren Sie Spuren, die Änderungen oder Anpassungen erfordern. Nachdem Sie die problematische Spur mit den Schaltflächen Verbinden und Trennen ausgewählt haben, bearbeiten Sie sie so, dass die Zellenbewegungen korrekt dargestellt werden. Verwenden Sie den Spureditor , um auch duplizierte einzelne Punkte oder kleine Spurfragmente, die fälschlicherweise dieselbe Zelle darstellen, auszuwählen und zu löschen, indem Sie sie mit dem Kreisauswahlmodus auswählen und die Tastenkombination "Canc" oder "Entf " drücken, um sie zu entfernen.
HINWEIS: Wenn die Einstellungen des Trackings korrekt sind, sollte dieser Schritt nur für einen kleinen Prozentsatz der verfolgten Zellen erforderlich sein. - Wenn die Mikroglia-Reporterlinie in Kombination mit fluoreszierenden Transgenika verwendet wird, die andere Zelltypen im Parenchym markieren, führen Sie die Segmentierung als neues Oberflächenobjekt oder Spot-Objekt durch, um sie zu visualisieren. Wiederholen Sie dazu die obigen Schritte (aus den Schritten 3.2 bis 3.14), indem Sie die Eingabe des Quellkanals in Schritt 3.5 so ändern, dass sie für die Abbildung der zweiten Zellpopulation verwendet wird, und achten Sie darauf, dass die beschreibenden Parameter, die für die interessierenden Zellen spezifisch sind, wie z. B. der geschätzte XY-Durchmesser (Schritt 3.5), die maximale Entfernung (Schritt 3.8) an die neue Zellpopulation angepasst werden. Andere Schritte können ohne Änderungen wiederholt werden.
HINWEIS: Bei der Erstellung einer neuen Fläche wird der geschätzte XY-Durchmesser durch eine Schwellenwertoption ersetzt, die auf der Hintergrundsubtraktion basiert, wobei die einzugebende Eingabe der Durchmesser der größten Kugel ist, die in das Objekt passt. Dieser Wert kann aus der Slice-Ansicht mit der Measure-Symbolleiste geschätzt werden, wie in Schritt 3.2 beschrieben. - Wenn Sie mit den erhaltenen Tracks zufrieden sind, extrahieren Sie die gewünschten Tracking-Statistiken aus der Registerkarte Statistik . Klicken Sie auf die Schaltfläche "Einstellungen " und wählen Sie aus, welche Statistiken für jedes zuvor erstellte Spot- oder Oberflächenobjekt berechnet werden sollen. Falls in derselben Analyse mehrere Objekte erstellt wurden, also zwei Punktobjekte oder ein Punkt- und ein Oberflächenobjekt, können auch Statistiken exportiert werden, die die relative Position eines Objekts zum anderen darstellen, z. B. die kürzeste Entfernung zwischen den beiden.
HINWEIS: Bei allen gewünschten Werten kann es sich entweder um spezifische oder durchschnittliche Werte handeln, und alle Rohdaten können über die Schaltflächen unten auf der Registerkarte "Statistiken " als CSV-Datei (Comma Separated Values) heruntergeladen werden. Unter der Voraussetzung, dass die Analyse auch andere Zelltypen umfasst, werden auch zusätzliche Statistiken über die relative Entfernung eines Objekts zum anderen verfügbar.
Ergebnisse
Mikrogliazellen, die grün fluoreszierendes Protein (eGFP) exprimieren, und Endothelzellen, die DsRed in Tg(mpeg1:eGFPgl22; KDRL:CRES898; actb2:loxP-STOP-loxP-DsRedexpress,sd5) wurden dreifach transgene Embryonen14 gemäß dem beschriebenen Protokoll bei 3 dpf abgebildet. Ein einzelner Zebrafischembryo wurde in 1 % niedrigschmelzender Agarose auf einer unteren Glasplatte montiert, und das bildgebende Verfahren behinderte das Wachstum des Embryos während der Aufnahmezeit nicht. Der Zeitraffer wurde mit einem kommerziellen konfokalen Punktscanning-Mikroskopiesystem aufgenommen, das mit einer 10x 0,45 NA Trockenobjektivlinse ausgestattet war, und 488 nm und 561 nm Anregungslaser wurden für die Abbildung von Mikroglia bzw. Endothelzellen verwendet. Darüber hinaus betrugen das Zeitintervall, die Bildauflösung, die Pixelgröße und der Z-Schritt 30 Sekunden (s), 1024 x 1024, 0,49 μm bzw. 2,5 μm. Die Zeitrafferaufnahme dauerte 3,5 Stunden (h).
Das erworbene Sichtfeld erstreckte sich über den gesamten Kopf des Embryos, aber die Analyse konzentrierte sich speziell auf das optische Tectum, da Mikroglia in der ersten Entwicklungswoche hauptsächlich auf die neuronale Soma-Schicht dieser Region des dorsalen Mittelhirns beschränkt sind, was es den Forschern ermöglicht, die gesamte Population gleichzeitig sichtbar zu machen15. Die 3D-Tracking-Analyse wurde, wie oben erläutert, mit Imaris 10.0 durchgeführt. Wie in Abbildung 4 gezeigt, war das Tracking erfolgreich, was zu 25 Spuren führte, die der erwarteten Anzahl von Mikrogliazellen im optischen Tectum bei 3 dpf16 entsprachen. Eine minimale manuelle Spurkorrektur war erforderlich. Abbildung 5 zeigt ein Beispiel für die Daten, die aus einem erfolgreichen Nachverfolgungsexperiment extrahiert werden können. Die gleichzeitige Markierung von Makrophagen und Endothelzellen ermöglicht die Quantifizierung der Position der Mikroglia relativ zu letzteren, so dass die Forscher den relativen Abstand jeder Zelle zur nächstgelegenen Endothelzelle in der Zeit visualisieren und die Häufigkeit und Anzahl potenzieller Wechselwirkungen untersuchen können (Abbildung 5).
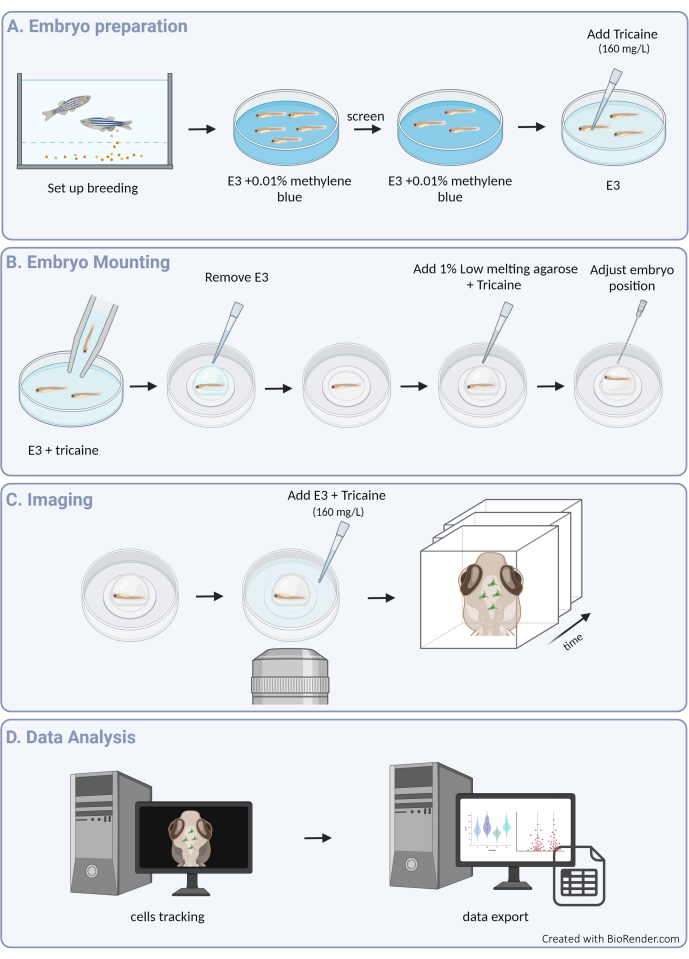
Abbildung 1: Überblick über den experimentellen Ablauf. (A) Vorbereitung des Zebrafischembryos und Anästhesie. (B) Montage und Positionierung der Probe. (C) Bilderstellung. (D) Bildverarbeitung und Extraktion von Motilitätsdaten. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
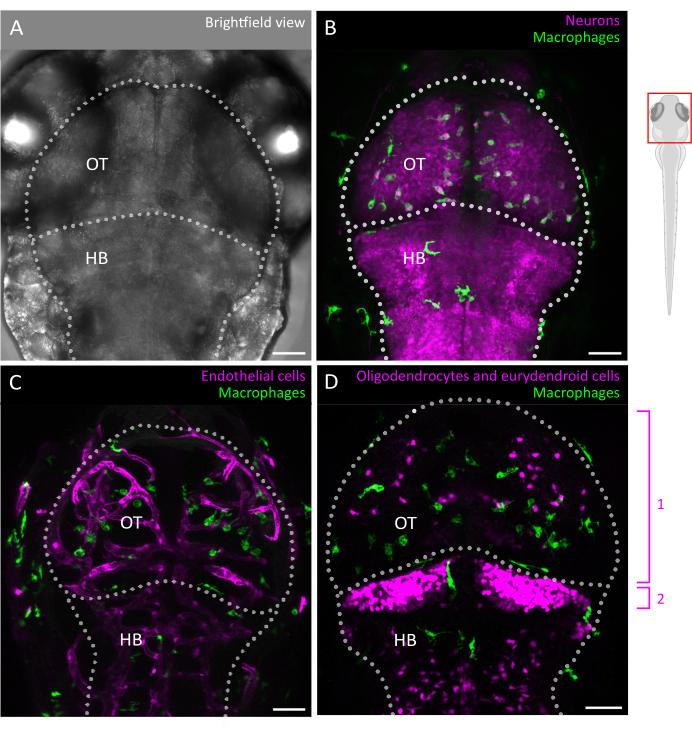
Abbildung 2: Mikroglia-Bildgebung und Wechselwirkungen mit der zellulären Umgebung des Gehirns im Zebrafischembryo. (A) Hellfeldaufnahme eines embryonalen Zebrafischkopfes und -gehirns mit 3 dpf (dorsale Ansicht), mit Beschriftung des optischen Tectums und des Hinterhirns. Das Gehirn des embryonalen Zebrafisches kann in diesem Stadium aufgrund seiner geringen Größe und optischen Transparenz in seiner Gesamtheit abgebildet werden. (B-D) Die Lokalisation und das Verhalten der Mikroglia können durch die Verwendung transgener Linien wie (B,C) Tg(mpeg1:eGFP)gl22 und (D) Tg(mpeg1:mcherry)gl23 und die Fokussierung auf den OT visualisiert werden. (B) Neuronen und ihre Zellkörper können mit Hilfe der Reporterlinie Tg(XlTubb:D sRed)zf148 identifiziert werden, und Mikroglia-Neuronen-Interaktionen können sichtbar gemacht werden, indem die beiden Kanäle in Linien zusammengeführt werden, die beide Transgene koexprimieren. Zu sehen ist eine Verschmelzung der grünen (Mikroglia) und roten (Neuronen, in Magenta) Signale. (C,D) Reporterlinien können auch Aufschluss über die Wechselwirkungen von Mikroglia (in grün) mit Endothelzellen geben, hier rot markiert mit dem (C) Doppel-Tg(kdrl:cres898; actb2:LOXP-STOP-LOXP-Dsredexpress, sd5) Transgene oder mit Gliazellen wie Oligodendrozyten und deren Vorläufern unter Verwendung des (D) doppelten Tg(olig2:EGFP; MPEG1:Mcherry ) transgene Linie. 1: Oligodendrozyten und Vorläuferzellen im OT, 2: Eurydendroiden-Neuronen im Kleinhirn. Maßstabsleisten = 50 μm. Abkürzungen: dpf = Tage nach der Befruchtung; OT = optisches Tectum; HB = Hinterhirn. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
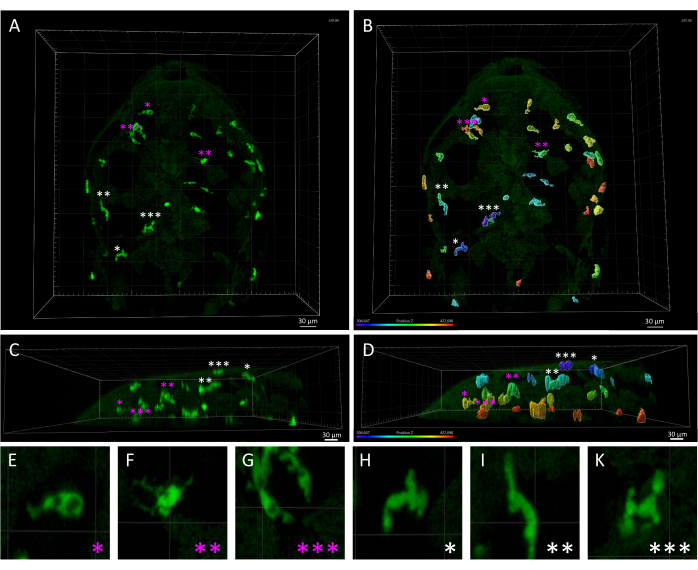
Abbildung 3: Morphologische und räumliche Disparität zwischen Hautmakrophagen und Mikroglia in einem 3 dpf Zebrafischembryo. (A,B) Dorsale Ansicht eines Tg(mpeg1:eGFP)gl22 Zebrafischembryos bei 3 dpf, dargestellt (A) parenchymale Mikrogliazellen (markiert durch magentafarbene Sternchen) im Vergleich zu Hautmakrophagen (weiße Sternchen), identifiziert anhand ihrer relativen Position entlang der z-Achse, wie in (B zu sehen)), in dem das monochrome 3D-Bild angezeigt wird, das mit der Farbcodierung der Z-Achse gerendert wurde. (C,D) Seitenansicht von (C) A und (D) B, die die z-Tiefen von mpeg1:eGFP+ -Zellen im Kopf des Embryos zeigt und die oberflächliche Lokalisation von Hautmakrophagen im Vergleich zu Mikrogliazellen hervorhebt. (E-K) Hohe Vergrößerung jeder Zelle, gekennzeichnet durch ein Sternchen in A und B, bietet eine detaillierte Visualisierung der unterschiedlichen Morphologien zwischen (E-G) Amöben-Mikroglia und (H-K) länglichen Hautmakrophagen. Maßstabsleisten = 30 μm. Abkürzung: dpf = Tage nach der Befruchtung. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
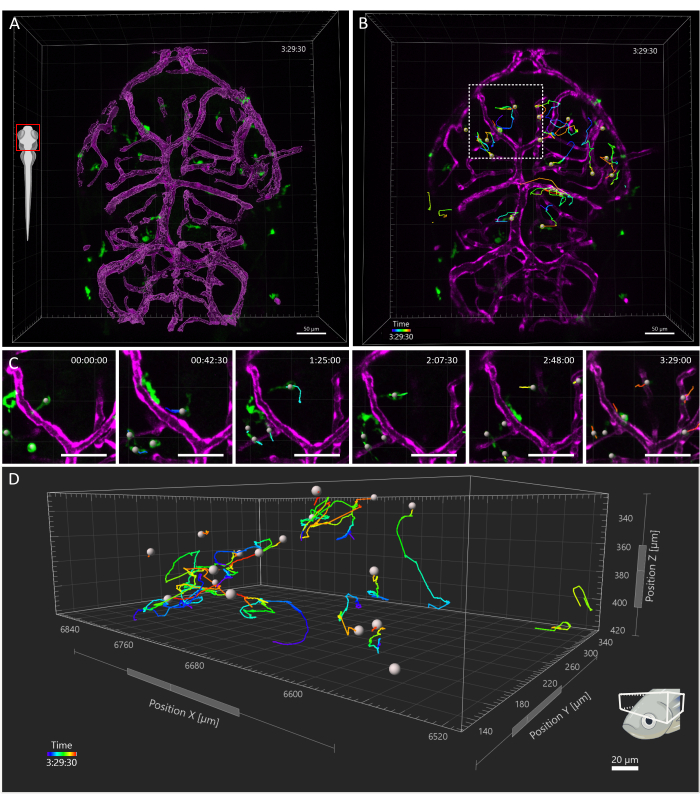
Abbildung 4: Verfolgung von Mikrogliazellen in einem 3 dpf Zebrafischembryo. (A) Kopf-Rückenansicht eines 3 dpf Tg(kdrl:cres898; actb2:LOXP-STOP-LOXP-Dsredexpress,sd5; mpeg1:eGFPgl22) dreifach transgener Embryo mit Mikroglia (grün) und der Oberflächendarstellung der Gefäße (magenta). (B-D) Repräsentative Verfolgung von Mikroglia-Bewegungen über ein (B) 3,5 h Zeitfenster. Einzelne Zellen folgen komplexen Bahnen. (C) Ausschnitt aus dem Zeitraffer, der eine vergrößerte Ansicht des Bereichs in B zeigt, der von dem gestrichelten Quadrat umschlossen ist. Sechs Bilder aus dem Film (im Abstand von 45 Minuten) werden gezeigt, die dokumentieren, wie Mikroglia vorübergehende Kontakte mit Endothelzellen in ihrer Mikroumgebung herstellen. (D) Trajektorien der Migration einzelner Mikrogliazellen innerhalb des optischen Tectums, wobei die X-, Y- und Z-Achsen die räumlichen Dimensionen darstellen. Maßstabsleiste = 50 μm. Abkürzung: dpf = Tage nach der Befruchtung. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
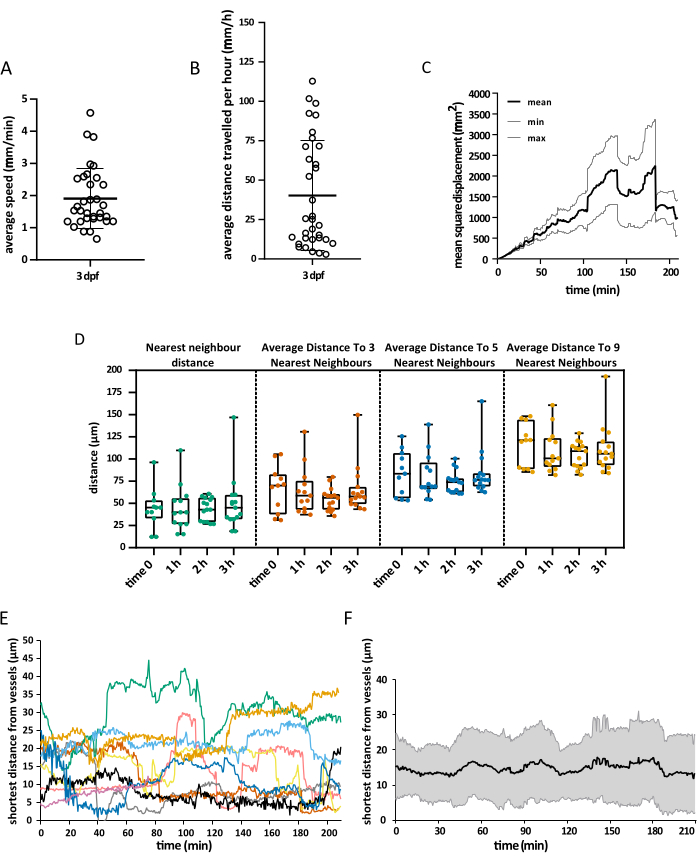
Abbildung 5: Visualisierung der gewonnenen Daten. Beispiel für die Geodaten, die mit dem beschriebenen Protokoll erhalten werden können. Die Diagramme zeigen (A) die durchschnittliche Geschwindigkeit von Mikrogliazellen, (B) die durchschnittliche Entfernung, die sie in 1 h zurücklegen, (C) ihre mittlere quadratische Verschiebung und (D) ihre Verteilung im Raum zu verschiedenen Zeiten. Das Oberflächen-Rendering des Gefäßes ermöglichte auch die Messung des kürzesten Abstands zwischen Mikroglia und Endothelzellen zu einem bestimmten Zeitpunkt, sowohl auf (E) Einzelzellebene als auch als (F) globaler Durchschnitt. Bei A, B und D stellt jeder Punkt eine einzelne Zelle dar. N = ein Embryo. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Ergänzende Datei 1: Bitte klicken Sie hier, um diese Datei herunterzuladen.
Diskussion
Das derzeitige Protokoll ermöglicht die in vivo Bildgebung der Mikroglia-Dynamik in einem Wirbeltierembryo und die Visualisierung der erfassten Motilitätsdaten. Die Mikroglia-Besiedlung des sich entwickelnden Gehirns erfolgt sehr früh während der Embryogenese und geht kritischen Ereignissen wie Spitzen der Neurogenese, Astrogliogenese, Oligodendrogenese und vielen anderen zellulären Prozessen voraus17. Es ist daher nicht verwunderlich, dass Mikroglia wichtige Funktionen bei der Gestaltung spezifischer Aspekte der Gehirnentwicklungspielen 18, z. B. durch die Regulation der neuronalen Differenzierung, Migration und des Überlebens 19,20,21 sowie durch synaptisches Pruning 5 und Myelinisierung 22,23,24.
Auch der Beitrag dysfunktionaler Mikroglia zur Pathogenese und/oder zum Fortschreiten neurologischer Entwicklungsstörungen wird zunehmend anerkannt25. Tatsächlich setzt das frühe Vorhandensein von Mikroglia im sich bildenden Gehirn diese Zellen unterschiedlichen physiologischen Zuständen26 und Umweltveränderungen aus. Dies kann erhebliche Auswirkungen haben, da Mikroglia sowohl bei Nagetieren als auch beim Menschen langlebige Zellen sind, die während der Lebensspanne durch Selbsterneuerung lokaler Vorläuferzellen erhalten bleiben 27,28,29. Wir glauben, dass dieses Protokoll als leistungsfähiges Werkzeug dienen könnte, um das Verhalten von Mikroglia in diesen unterschiedlichen physiologischen Zuständen besser zu charakterisieren, während sie sich entwickeln, reifen und ihr Netzwerk während der aufeinanderfolgenden Schritte der Gehirnmorphogenese etablieren.
Mit dem hier beschriebenen Aufbau ist es uns gelungen, Zebrafischlarven mit einem Alter von bis zu 6 dpf abzubilden und Daten zu erfassen. Die Ausweitung der Analyse auf spätere Entwicklungsstufen wird wahrscheinlich erfolgreich sein, erfordert jedoch eine Anpassung des Bildgebungsaufbaus, um die erhöhte Probengröße zu berücksichtigen, insbesondere entlang der z-Achse. Bei diesem Versuch empfehlen wir, sich auf ein niedriges Signal-Rausch-Verhältnis und eine schnelle Scanzeit zu konzentrieren, da dies Schlüsselparameter für eine erfolgreiche Analyse sind.
Wir empfehlen eine minimale Bildgebungszeit von 1 h, um das Tracking von Mikroglia zu ermöglichen. Das längste Bildgebungsfenster, das mit diesem Protokoll getestet wurde, beträgt 8 h. Darüber hinaus ist es für die Tracking-Analyse wichtig, das Zeitintervall zwischen den Frames so kurz wie möglich zu halten, idealerweise zwischen 30 s und 60 s. Dies ermöglicht genauere und detailliertere Tracking-Daten in nachgelagerten Analysen. Daher ist es insbesondere bei der Detektion von mehr als einem Fluorophor von grundlegender Bedeutung, spektrale Überlappungen zu vermeiden und einen ausreichenden Abstand zwischen den beiden Fluorophor-Emissionsspektren zu gewährleisten, um eine gleichzeitige Erfassung ohne Signaldurchscheinen zu ermöglichen.
Es gibt weitere Protokolle für qualitativ hochwertige Zeitrafferaufnahmen des Zebrafischgehirns30, aber dies ist das erste, das zeigt, wie alle Bewegungen der Mikroglia während der Embryonalentwicklung über einen längeren Zeitraum erfolgreich verfolgt werden können. Obwohl sich der hier vorgestellte Workflow auf die Verfolgung von Mikroglia in einem physiologischen Kontext konzentriert, kann er leicht auf die Analyse von Mikroglia in der Pathologie angewendet werden. In der Tat wurden mehrere Modelle von neurologischen Entwicklungsstörungen, wie Autismus31, Epilepsie32 und Schizophrenie33, aber auch Neurodegeneration34 und Krebs35, im Zebrafisch etabliert, die einzigartige Möglichkeiten bieten, die Reaktion und das Verhalten der Mikroglia bei Krankheitszuständen zu bestimmen.
Insbesondere ist dieses Tracking-Protokoll sehr vielseitig und könnte auch dazu beitragen, die Migrationsmuster verschiedener Zelltypen in verschiedenen anatomischen Regionen des Zebrafischembryos zu beleuchten und so möglicherweise Wege für zusätzliche Anwendungen zu eröffnen, die über den in diesem Artikel beschriebenen Untersuchungsumfang der Mikroglia hinausgehen. Darüber hinaus gewinnen wir durch die Nutzung der Fähigkeit, mehrere fluoreszierende transgene Linien zu kombinieren, die Fähigkeit, die räumliche Beziehung zwischen Mikroglia und anderen Zelltypen der Mikroumgebung des Gehirns zu erkennen, mit dem Potenzial, zelluläre Interaktionen und Wechselwirkungen in Zeitrafferaufnahmen auf nicht-invasive Weise zu visualisieren. Dies könnte dazu beitragen, die physiologische Bedeutung des Verhaltens von Mikroglia zu entschlüsseln und zu einer tieferen Charakterisierung dieser hochbeweglichen Zellen beizutragen.
Offenlegungen
Die Autoren erklären, dass keine Interessenkonflikte bestehen.
Danksagungen
Die Autoren danken Professor Nicolas Bayens herzlich für die großzügige Bereitstellung des für diese Studie unerlässlichen Konfokalmikroskops. Diese Arbeit wurde teilweise von den Funds for Scientific Research (FNRS) unter den Fördernummern F451218F und UG03019F finanziert, die Alzheimer Research Foundation (SAO-FRA) (an V.W.), A.M. wird durch ein Forschungsstipendium der FNRS unterstützt. Abbildung 1 wurde am biorender.com erstellt.
Materialien
| Name | Company | Catalog Number | Comments |
| 1 L Breeding tanks | Tecniplast | ZB10BTE | |
| 1-phenyl-2-thiourea (PTU) | Sigma-Aldrich | P7629 | Diluted to 0.2 mM in E3 to prevent embryo pigmentation |
| Bottom glass imaging dish | FluoroDish | FD3510-100 | |
| Disposable Graduated transfer pipette | avantor | 16001-188 | |
| Dry block heater | Novolab | Grant QBD4 | To keep low melting agarose at 37 °C |
| Ethyl 3-aminobenzoate methanesulfonate (Tricaine) | Sigma-Aldrich | E10521-50G | |
| Imaris 10.0 | Oxford Instruments | analysis software | |
| Imaris File Converter | Oxford Instruments | https://imaris.oxinst.com/big-data | |
| Laser-scanning confocal microscope | Nikon | Eclipse Ti2-E | |
| Methylene blue | Sigma-Aldrich | M9140-25G | |
| microloader tips | Eppendorf | 5242956003 | |
| NuSieve GTG Agarose | Lonza | 50081 | |
| Petri dishes (90 mm) | avantor | 391-0559 | |
| Pronase | Sigma-Aldrich | 11459643001 | |
| Stainless Steel Forceps Dumont No. 5 | FineScienceTools | 11254-20 | |
| Stereo microscope | Leica | Leica M80 | To mount the embryos |
| teasing needle | avantor | 76549-024 |
Referenzen
- Wolf, S. A., Boddeke, H. W., Kettenmann, H. Microglia in physiology and disease. Annu Rev Physiol. 79, 619-643 (2017).
- Nimmerjahn, A., Kirchhoff, F., Helmchen, F. Resting microglial cells are highly dynamic surveillants of brain parenchyma in vivo. Sci Rep. 308 (5726), 1314-1318 (2005).
- Peri, F., Nusslein-Volhard, C. Live imaging of neuronal degradation by microglia reveals a role for v0-ATPase a1 in phagosomal fusion in vivo. Cell. 133 (5), 916-927 (2008).
- Marın-Teva, J. L., et al. Microglia promote the death of developing Purkinje cells. Neuron. 41 (4), 535-547 (2004).
- Paolicelli, R. C., et al. Synaptic pruning by microglia is necessary for normal brain development. Science. 333 (6048), 1456-1458 (2011).
- Hattori, Y. The microglia-blood vessel interactions in the developing brain. Neurosci Res. 187, 58-66 (2023).
- Colonna, M., Butovsky, O. Microglia function in the central nervous system during health and neurodegeneration. Annu Rev Immunol. 35, 441-468 (2017).
- Xu, H. -T., Pan, F., Yang, G., Gan, W. -B. Choice of cranial window type for in vivo imaging affects dendritic spine turnover in the cortex. Nat Neurosci. 10 (5), 549-551 (2007).
- Shin, J., Park, H. -C., Topczewska, J. M., Mawdsley, D. J., Appel, B. Neural cell fate analysis in zebrafish using olig2 BAC transgenics. Methods Cell Sci. 25 (1), 7-14 (2003).
- Chi, N. C., et al. Foxn4 directly regulates tbx2b expression and atrioventricular canal formation. Genes Dev. 22 (6), 734-739 (2008).
- Avdesh, A., et al. Regular care and maintenance of a zebrafish (Danio rerio) laboratory: An introduction. J Vis Exp. (69), e4196(2012).
- Bohnsack, B. L., Gallina, D., Kahana, A. Phenothiourea sensitizes zebrafish cranial neural crest and extraocular muscle development to changes in retinoic acid and IGF signaling. PLoS ONE. 6 (8), e22991(2011).
- Ariga, J., Walker, S. L., Mumm, J. S. Multicolor time-lapse imaging of transgenic zebrafish: visualizing retinal stem cells activated by targeted neuronal cell ablation. J Vis Exp. (43), e2093(2010).
- Ferrero, G., et al. Embryonic microglia derive from primitive macrophages and are replaced by cmyb-dependent definitive microglia in zebrafish. Cell Rep. 24 (1), 130-141 (2018).
- Xu, J., Wang, T., Wu, Y., Jin, W., Wen, Z. Microglia colonization of developing zebrafish midbrain is promoted by apoptotic neuron and lysophosphatidylcholine. Dev Cell. 38 (2), 214-222 (2016).
- Svahn, A. J., et al. Development of ramified microglia from early macrophages in the zebrafish optic tectum. Dev Neurobiol. 73 (1), 60-71 (2013).
- Thion, M. S., Garel, S. Microglial ontogeny, diversity and neurodevelopmental functions. Curr Opin Genet Dev. 65, 186-194 (2020).
- Bohlen, C. J., Friedman, B. A., Dejanovic, B., Sheng, M. Microglia in brain development, homeostasis, and neurodegeneration. Annu Rev Genet. 53, 263-288 (2019).
- Squarzoni, P., et al. Microglia modulate wiring of the embryonic forebrain. Cell Rep. 8 (5), 1271-1279 (2014).
- Schafer, D. P., et al. Microglia sculpt postnatal neural circuits in an activity and complement-dependent. Neuron. 74 (4), 691-705 (2012).
- Parkhurst, C. N., et al. Microglia promote learning-dependent synapse formation through brain-derived neurotrophic factor. Cell. 155 (7), 1596-1609 (2013).
- Green, L. A., O'Dea, M. R., Hoover, C. A., DeSantis, D. F., Smith, C. J. The embryonic zebrafish brain is seeded by a lymphatic-dependent population of mrc1+ microglia precursors. Nat. Neurosci. 25 (7), 849-864 (2022).
- Hughes, A. N., Appel, B. Microglia phagocytose myelin sheaths to modify developmental myelination. Nat Neurosci. 23 (9), 1055-1066 (2020).
- Yu, Q., et al. C1q is essential for myelination in the central nervous system (CNS). iScience. 26 (12), 108518(2023).
- Zengeler, K. E., Lukens, J. R. Innate immunity at the crossroads of healthy brain maturation and neurodevelopmental disorders. Nat Rev Immunol. 21 (7), 454-468 (2021).
- Paolicelli, R. C., et al. Microglia states and nomenclature: A field at its crossroads. Neuron. 110 (21), 3458-3483 (2022).
- Réu, P., et al. The lifespan and turnover of microglia in the human brain. Cell Rep. 20 (4), 779-784 (2017).
- Ajami, B., Bennett, J. L., Krieger, C., Tetzlaff, W., Rossi, F. M. V. Local self-renewal can sustain CNS microglia maintenance and function throughout adult life. Nat Neurosci. 10 (12), 1538-1543 (2007).
- Mildner, A., et al. Microglia in the adult brain arise from Ly-6ChiCCR2+ monocytes only under defined host conditions. Nat Neurosci. 10 (12), 1544-1553 (2007).
- Cook, Z. T., Brockway, N. L., Weissman, T. A. Visualizing the developing brain in living zebrafish using Brainbow and time-lapse confocal imaging. J Vis Exp. (157), e60593(2020).
- Tayanloo-Beik, A., et al. Zebrafish modeling of autism spectrum disorders, current status and future prospective. Front Psychiatry. 13, 911770(2022).
- D'Amora, M., et al. Zebrafish as an innovative tool for epilepsy modeling: State of the art and potential future directions. Int J Mol Sci. 24 (9), 7702(2023).
- Langova, V., Vales, K., Horacek, J. The role of zebrafish and laboratory rodents in schizophrenia research. Front Psychiatry. 11, 549232(2020).
- Chia, K., Klingseisen, A., Sieger, D., Priller, J. Zebrafish as a model organism for neurodegenerative disease. Front Mol Neurosci. 15, 940484(2022).
- Astell, K. R., Sieger, D. Zebrafish in vivo models of cancer and metastasis. Cold Spring Harb Perspect Med. 10 (8), 037077(2020).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten