Method Article
Imaging dal vivo e caratterizzazione della dinamica della microglia nell'embrione di zebrafish
In questo articolo
Riepilogo
Dimostriamo un metodo che sfrutta la microscopia confocale a scansione rapida per eseguire l'imaging dal vivo delle cellule della microglia nel tectum ottico del pesce zebra in via di sviluppo, consentendo l'analisi della dinamica di queste cellule in vivo.
Abstract
Le microglia sono cellule altamente dinamiche e la loro migrazione e colonizzazione del parenchima cerebrale è un passaggio cruciale per il corretto sviluppo e funzione del cervello. Gli embrioni di zebrafish sviluppati esternamente possiedono una trasparenza ottica che, insieme a linee reporter transgeniche ben caratterizzate che marcano in modo fluorescente la microglia, rendono il zebrafish un modello di vertebrato ideale per tali studi. In questo articolo, sfruttiamo le caratteristiche uniche del modello di zebrafish per visualizzare la dinamica delle cellule della microglia in vivo e in condizioni fisiologiche. Utilizziamo la microscopia confocale per registrare un timelapse delle cellule della microglia nel tectum ottico dell'embrione di zebrafish e quindi estrarre i dati di tracciamento utilizzando il software IMARIS 10.0 per ottenere il percorso di migrazione delle cellule, la velocità media e la distribuzione nel tectum ottico in diverse fasi di sviluppo. Questo protocollo può essere uno strumento utile per chiarire il significato fisiologico del comportamento delle microglia in vari contesti, contribuendo a una caratterizzazione più profonda di queste cellule altamente mobili.
Introduzione
Come macrofagi residenti nel sistema nervoso centrale (SNC), le microglia rappresentano una popolazione non neuronale distinta che rappresenta fino al 15% di tutte le cellule gliali nel cervello adulto. Lo studio della biologia della microglia ha guadagnato sempre più attenzione negli ultimi anni a causa della sua consolidata importanza nello sviluppo, nella fisiologia enella malattia. In condizioni fisiologiche, le cellule microgliali sono altamente dinamiche, esaminano continuamente il parenchima cerebrale 2,3. Questo comportamento consente alle microglia di colonizzare il cervello e svolgere ruoli fondamentali nel suo sviluppo, come la modellazione dei circuiti neuronali4, la potatura sinaptica5 e la vasculogenesi6. Inoltre, questa natura dinamica intrinseca consente alla microglia di monitorare costantemente il SNC alla ricerca di segni di infezione, lesioni o eventuali deviazioni dall'omeostasi7. Per analizzare queste intricate dinamiche cellulari, è indispensabile l'imaging dal vivo delle microglia nello spazio e nel tempo. Fortunatamente, la trasparenza ottica degli embrioni di zebrafish in sviluppo esterno, unita alla disponibilità di linee reporter transgeniche ben caratterizzate che marcano in modo fluorescente la microglia, posiziona il zebrafish come un modello di vertebrato ideale per tali indagini. L'imaging dal vivo negli embrioni di zebrafish offre un approccio non invasivo che non richiede interventi chirurgici o un'estesa manipolazione dei tessuti, riducendo al minimo le potenziali perturbazioni dello stato del SNC. Questa è una considerazione fondamentale quando si studiano le cellule microgliali, poiché sono altamente sensibili anche a sottili cambiamenti nell'ambiente extracellulare8.
Qui, forniamo una linea guida per tracciare con successo i movimenti delle cellule microgliali 3D nell'embrione di zebrafish, consentendo una visione senza precedenti del comportamento della microglia all'interno dell'architettura intatta del parenchima cerebrale in via di sviluppo (vedere la Figura 1 per una panoramica grafica del protocollo). Questo protocollo passo-passo descrive in dettaglio come impostare e visualizzare le microglia di pesce zebra in diverse fasi di sviluppo e come estrarre dati ad alta risoluzione sulla motilità delle cellule della microglia per fornire preziose informazioni sui loro modelli migratori e sulle risposte ai segnali ambientali. Dimostriamo anche che questo protocollo può essere adattato per eseguire l'imaging multicolore dal vivo, estendendo così la sua applicabilità allo studio della microglia in combinazione con linee transgeniche che marcano le cellule vicine, inclusi i neuroni3, gli oligodendrociti9 e le cellule endoteliali10 (come mostrato nella Figura 2). Aggiungendo alla cassetta degli attrezzi che consente di osservare e caratterizzare direttamente le dinamiche del comportamento delle microglia in tempo reale e nel loro ambiente naturale, questo protocollo contribuirà probabilmente a chiarire meglio la funzionalità delle microglia durante lo sviluppo precoce, sia in fisiologia che in malattia.
Protocollo
I pesci zebra sono stati mantenuti in condizioni standard, secondo FELASA42 e istituzionali (Université Libre de Bruxelles, Bruxelles, Belgio; ULB) linee guida e regolamenti. Tutte le procedure sperimentali sono state approvate dal Comitato Etico per il Benessere Animale (CEBEA) dell'ULB.
1. Allevamento del pesce zebra e preparazione degli embrioni
NOTA: La linea transgenica di zebrafish Tg(mpeg1:eGFP)gl22 che esprime la proteina fluorescente eGFP nei macrofagi, inclusa la microglia, è stata utilizzata per generare i dati di tracciamento mostrati in questo protocollo. Altre linee di reporter per macrofagi sono disponibili presso ZIRC e possono essere utilizzate. Nella scelta della linea transgenica, è importante considerare che un'elevata intensità del segnale faciliterà l'acquisizione dell'immagine e la segmentazione cellulare.
- Mantenere il pesce zebra (Danio rerio) a una temperatura di 28 °C e gestirlo secondo i protocolli stabiliti11.
- Stimolare l'accoppiamento mettendo insieme pesci femmina e maschio in una vasca di riproduzione, con fondo a rete e separatore, nel tardo pomeriggio prima della data di nascita degli embrioni desiderati. Rimuovere il separatore la mattina seguente per programmare l'accoppiamento dei pesci.
- Raccogliere gli ovuli fecondati utilizzando una rete e trasferirli in una piastra di Petri da 100 mm contenente terreno E3 (5 mM NaCl, 0,17 mM KCl, 0,33 mM CaCl2, 0,33 mM MgSO4) con 0,01% di blu di metilene. A seconda del background genetico della linea transgenica di pesce zebra utilizzata, se la formazione del pigmento deve essere inibita per facilitare l'imaging, aggiungere lo 0,003% di N-feniltiourea (PTU) al tampone E3 in cui si stanno sviluppando gli embrioni, a partire da 6 ore dopo la fecondazione (hpf) e rinfrescando la soluzione ogni 2 giorni fino al raggiungimento dello stadio di sviluppo desiderato.
NOTA: Per evitare una lunga esposizione alla PTU che potrebbe influenzare lo sviluppo embrionale12, si suggerisce di utilizzare, se disponibile, una linea transgenica su uno sfondo carente di pigmento, come Casperw2, che consenta un imaging chiaro senza la necessità di esporre gli embrioni alla PTU per un lungo periodo di tempo13. - Se gli embrioni da sottoporre a imaging sono più giovani di 3 giorni dopo la fecondazione (dpf), decorionarli a 24 hpf. Utilizzare uno stereomicroscopio per confermare che tutti gli embrioni sono stati dechorionati e non sono stati danneggiati dal trattamento enzimatico (vedere passaggio 1.4.2).
- In caso di un numero limitato di ovuli, farlo manualmente utilizzando una pinza da dissezione fine per pizzicare il corion e tirarlo verso l'esterno, rilasciando con cura ogni embrione dal suo chorion.
- In caso di un numero maggiore di uova, eseguire la decorionazione enzimaticamente incubando le uova alla rinfusa a temperatura ambiente (RT) in una soluzione di pronasi da 1 mg/mL in terreno E3 per un breve periodo (tipicamente 5-10 minuti, fino a quando i corioni non si aprono sotto un leggero fruscio). Successivamente, la soluzione di pronasi viene scartata e gli embrioni vengono sciacquati accuratamente 3 volte con E3 fresco, per rimuovere il corion rimanente e l'enzima residuo.
2. Montaggio del pesce zebra
- Preparare una soluzione di agarosio a basso punto di fusione all'1% in tampone E3. Raffreddare l'agarosio a 37 °C in un blocco riscaldante da banco e aggiungere una soluzione di tricaina metansolfonato appena preparata (160 mg/L), che aiuterà a mantenere gli embrioni anestetizzati durante l'imaging. Agitare la soluzione per omogeneizzarla completamente.
- Anestetizzare gli embrioni aggiungendo 160 mg/L di tricaina metansolfonato alla piastra di Petri contenente gli embrioni nel tampone E3. Verificare che il movimento si sia fermato e selezionare gli embrioni di zebrafish con la fluorescenza desiderata. Controlla la salute dell'embrione visualizzando il suo battito cardiaco utilizzando uno stereomicroscopio a campo chiaro. Utilizzando una pipetta di trasferimento a punta larga, prelevare uno o fino a tre degli embrioni selezionati e trasferirli su una piastra di imaging in vetro inferiore.
- Sotto uno stereomicroscopio, utilizzare una pipetta per rimuovere quanto più terreno possibile, facendo attenzione a non toccare o danneggiare gli embrioni; Quindi, sostituire il terreno con la soluzione di agarosio a basso punto di fusione all'1% precedentemente preparata. Quindi, utilizzare una punta di plastica sottile e affusolata fissata all'estremità di un ago per stuzzicare per spingere delicatamente ogni embrione sul fondo della piastra di imaging, orientandolo in modo che il suo lato dorsale sia rivolto verso il vetro per garantire una buona visione del tectum ottico e una quantità minima di agarosio a basso punto di fusione tra l'embrione e il vetro.
- Fotografare prontamente l'embrione montato utilizzando un microscopio confocale invertito dotato di un obiettivo Plan Apo di alta qualità (10x/0,45 Plan Apo o 20x/0,75 Plan Apo utilizzato qui). Se il timelapse durerà per un periodo prolungato (più di un'ora), aggiungere una o due gocce di tampone E3 con tricaina metansolfonato (160 mg/L) alla piastra di imaging in vetro inferiore per evitare che l'agarosio a basso punto di fusione si asciughi eccessivamente.
NOTA: È fondamentale attendere che l'agarosio si solidifichi completamente prima di aggiungere il tampone E3 e il terreno di tricaina metansolfonato al disco di imaging; In caso contrario, il pesce potrebbe muoversi lentamente lateralmente e assialmente, causando così una deriva del campione nel timelapse. - Procedere con l'impostazione dell'acquisizione timelapse per il tempo desiderato: impostare la risoluzione dell'immagine su 1.024 x 1.024, con una dimensione dei pixel di 0,49 μm. Raccogli le fette ottiche con incrementi di 2-3 μm per garantire una risoluzione accettabile sull'asse z. Nell'embrione di zebrafish, racchiudere il tectum ottico in uno z-stack di 100-150 μm, a seconda dello stadio di sviluppo utilizzato. Per garantire il corretto tracciamento delle celle, mantenere l'intervallo di tempo tra i fotogrammi tra 30 s e 60 s.
3. Analisi del tracciamento ed esportazione dei dati
- Converti il file timelapse in formato IMS utilizzando il software di conversione file Imaris (vedi Tabella dei materiali). All'avvio del software, trascinare e rilasciare i file nell'area di input o utilizzare il pulsante Aggiungi file... per selezionarli manualmente. Nel menu Output , specificare la posizione per i file convertiti, scegliendo la stessa cartella dei file di input o specificando una cartella specifica. Assicurati che le dimensioni della dimensione del voxel visualizzata siano corrette facendo clic sul pulsante Imposta dimensione voxel e, una volta verificata la dimensione del voxel, premi il pulsante Avvia tutto per avviare la conversione del file (vedi File supplementare 1 per una guida passo passo di tutta l'analisi dei dati).
NOTA: Se il timelapse è stato eseguito su due o più embrioni in parallelo, il file verrà automaticamente diviso in singole posizioni, separando ogni embrione in file diversi. È possibile utilizzare il software in modalità batch e caricare e convertire più file contemporaneamente. I file IMS verranno creati e salvati nella cartella di destinazione selezionata o nella cartella in cui si trovano i file di input. - Una volta avviato, lascia che il software di analisi si avvii nell'Arena; fare clic su Osserva cartella per aprire il file precedentemente convertito. Una volta caricato il file nel software, utilizzare la vista Slice per scorrere lo z-stack, utilizzando il cursore nella barra degli strumenti Slice situato a sinistra dell'area di visualizzazione, e misurare alcuni diametri delle celle disegnando il diametro delle celle selezionate utilizzando il puntatore. Fare clic e trascinare il mouse per disegnare un segmento che attraversa il nucleo della cella; osservare la lunghezza del segmento disegnato nella barra degli strumenti Misura, situata a destra dell'area di visualizzazione.
NOTA: Utilizzando le impostazioni di imaging al punto 2.5, il diametro del corpo cellulare può essere compreso tra 6 μm e 8 μm. - Per rilevare automaticamente le celle, torna alla vista 3D e fai clic sull'icona spot nella barra degli strumenti degli oggetti per aggiungere un nuovo oggetto Spots nell'elenco degli oggetti e aprire la procedura guidata di creazione automatica delle Spot. Nel primo passaggio della procedura guidata, selezionare l'opzione per segmentare solo una regione di interesse. Quindi, abilita l'opzione Traccia punti (nel tempo) per calcolare i dati di tracciamento, nonché le statistiche oggetto-oggetto per consentire il confronto tra i punti e ampliare la gamma di dati che saranno disponibili più avanti nell'analisi. Infine, procedi nella procedura guidata facendo clic sul pulsante Avanti in basso a destra nella finestra della procedura guidata.
- Nella seconda fase, procedere alla specifica di un'area della regione di interesse (ROI) che racchiude il tectum ottico dell'embrione. Modifica la dimensione del ROI facendo clic e trascinando le piccole frecce bianche presenti su ciascuna faccia di esso. Una volta definita la dimensione del ROI, estendila a tutti i fotogrammi registrati regolando l'intervallo di tempo.
- Quindi, selezionare come Canale sorgente il canale utilizzato per rilevare le celle, e impostare la misura del diametro della cella, precedentemente ottenuta nel passaggio 3.2, come Diametro XY stimato. Abilita l'opzione Sottrazione sfondo e fai clic su Avanti.
- A questo punto della procedura guidata di creazione degli spot, il software incorpora un passaggio di filtraggio di qualità in cui gli spot vengono filtrati in base all'intensità del segnale misurata al centro di ogni spot; nel canale in cui sono state rilevate le macchie. Regolare la soglia di intensità inserendo il valore direttamente nel campo dati per le caselle di soglia inferiore e superiore o trascinando le linee colorate corrispondenti nell'istogramma mostrato nella parte inferiore della procedura guidata di creazione, in modo che vengano rilevate tutte le cellule, comprese quelle localizzate più in profondità nel tessuto, che di solito mostrano un'intensità del segnale inferiore. Tutte queste modifiche sono visibili contemporaneamente nell'Area di visualizzazione.
NOTA: A questo punto è possibile osservare più punti che etichettano una singola cella, ma questo può essere corretto in seguito nell'analisi.
Nei timelapse più lunghi, se si nota un po' di fotosbiancamento, controllare l'area di visualizzazione di più fotogrammi prima di impostare la soglia per assicurarsi che tutte le celle vengano rilevate anche nei fotogrammi più recenti e quindi più deboli. - Una volta soddisfatto, fare clic su Avanti per convalidare la soglia di qualità selezionata. Tutti gli spot che soddisfano il parametro di soglia di qualità e che hanno un diametro corrispondente al diametro XY stimato verranno etichettati come spot. Visualizzatela come un'anteprima nell'Area di Visualizzazione, dove gli spot che soddisfano questi due parametri saranno sovrapposti al Canale Sorgente.
- Nel passaggio successivo, selezionare il Movimento autoregressivo come algoritmo di tracciamento per tracciare le celle che hanno un movimento più o meno continuo. Valuta la distanza massima che una cella percorre tra due punti temporali osservando attentamente il movimento dei punti tra due fotogrammi e inserisci un numero stimato nel campo Distanza massima . Questa è la distanza alla quale un punto può deviare dalla posizione futura prevista.
- Impostare la dimensione massima dello spazio sul numero minimo di punti temporali in cui il punto può scomparire senza interrompere la traccia. Se l'intervallo tra i fotogrammi è inferiore a 60 s, utilizzare una dimensione di 3. Se l'intervallo di tempo tra i fotogrammi è più lungo, aumentate la dimensione massima consentita per ridurre la frammentazione della traccia.
- Abilitare l'opzione Riempi gli spazi vuoti con tutti gli oggetti rilevati per abbassare la soglia di rilevamento vicino alla posizione prevista dall'algoritmo di tracciamento e collegare le tracce interrotte da un errore del rilevamento spot. Infine, fai clic su Avanti e lascia che il software generi automaticamente le tracce per tutti gli spot generati in precedenza.
- Se le tracce ottenute appaiono troppo numerose o frammentate, tornare indietro nella procedura guidata di creazione, cliccando sul pulsante Precedente , e regolare la distanza massima precedentemente definita, aumentando o diminuendo il numero fino a quando la maggior parte delle tracce ottenute non sono rappresentative dei movimenti della cella.
NOTA: Il numero di tracce dovrebbe corrispondere all'incirca al numero di cellule della microglia, quindi variare tra 30 e 50, a seconda dell'età dell'embrione. Tuttavia, prima del completamento dell'analisi, si prevede un numero più elevato a causa di tracce errate. - Una volta calcolate le tracce, il software incorpora un passaggio di filtraggio per filtrare tutte le tracce molto brevi in quanto spesso imprecise o non informative. Escludere le tracce di durata inferiore a 3-8 minuti (min) inserendo il valore direttamente nel campo dati per il limite inferiore del filtro di durata della traccia o trascinando le linee colorate corrispondenti nell'istogramma visualizzato nella parte inferiore della procedura guidata di creazione. Fare clic su Avanti per convalidare il filtro e procedere con la procedura guidata di creazione.
NOTA: Come accennato in precedenza, modificando la soglia di durata massima (max) e minima (min) della traccia, è possibile visualizzare in anteprima le tracce che verranno filtrate ed escluse dall'analisi. Le tracce filtrate possono quindi essere duplicate in un nuovo oggetto per ulteriori analisi. La soglia di tempo esatta per il filtro può variare in base ai risultati del passaggio precedente della procedura guidata di creazione . Si consiglia di ottimizzare queste impostazioni del filtro esaminando l'anteprima nell'area di visualizzazione per ottenere risultati ottimali. - Poiché la linea transgenica Tg(mpeg1:eGFP) marcasce tutti i fagociti mononucleati, i macrofagi cutanei oltre alla microglia saranno rilevati anche nei filmati timelapse. Rimuovere manualmente le tracce generate dai macrofagi cutanei dall'analisi per concentrarsi sulla microglia parenchimale cerebrale (Figura 3).
- Correggi manualmente altri potenziali errori nelle tracce nell'editor delle tracce, in cui ogni punto dati viene visualizzato come un oggetto nella traccia. Attiva il pulsante circolare di selezione della modalità a destra dell'area di visualizzazione ed evidenzia le tracce che richiedono modifiche o regolazioni. Una volta selezionata la traccia problematica, utilizzando i pulsanti di connessione e disconnessione , modificarla per rappresentare correttamente i movimenti della cella. Utilizzare l'editor di tracce per selezionare ed eliminare anche singoli punti duplicati o piccoli frammenti di traccia che rappresentano erroneamente la stessa cella, selezionandoli con la modalità di selezione del cerchio e premendo Canc o Canc per eliminarli.
NOTA: Se le impostazioni del tracciamento sono corrette, questo passaggio dovrebbe essere necessario solo per una piccola percentuale delle celle tracciate. - Se la linea reporter della microglia viene utilizzata in combinazione con transgenici fluorescenti che marcano altri tipi di cellule nel parenchima, eseguire la segmentazione come un nuovo oggetto di superficie o un oggetto spot per visualizzarli. Per fare ciò, ripetere i passaggi precedenti (dai passaggi 3.2 a 3.14), modificando l'input del canale sorgente nel passaggio 3.5, in modo che sia quello utilizzato per visualizzare la seconda popolazione cellulare, e avendo cura di adattare i parametri descrittivi specifici delle celle di interesse come il diametro XY stimato (passaggio 3.5), la distanza massima (passaggio 3.8) alla nuova popolazione cellulare. Altri passaggi possono essere ripetuti senza modifiche.
NOTA: Nel caso di una nuova creazione di superficie, il diametro XY stimato viene sostituito da un'opzione di soglia basata sulla sottrazione dello sfondo, in cui l'input da fornire è il diametro della sfera più grande che si adatta all'oggetto. Questo valore può essere stimato dalla vista Sezione utilizzando la barra degli strumenti Misura , come illustrato nel passaggio 3.2. - Una volta soddisfatti delle tracce ottenute, estrai le statistiche di tracciamento desiderate dalla scheda Statistica . Cliccando sul pulsante delle impostazioni , selezionare quali statistiche verranno calcolate per ogni oggetto punto o oggetto superficie precedentemente creato. Nel caso in cui siano stati creati più oggetti nella stessa analisi, due oggetti punto o un oggetto punto e un oggetto superficie, saranno disponibili per l'esportazione anche le statistiche che descrivono la posizione relativa di un oggetto rispetto all'altro, come la distanza più breve tra i due.
NOTA: Tutti i valori desiderati possono essere valori specifici o medi e tutti i dati grezzi sono scaricabili come file CSV (Comma Separated Values) utilizzando i pulsanti nella parte inferiore della scheda Statistiche . A condizione che l'analisi includa anche altri tipi di celle, saranno disponibili anche ulteriori statistiche sulla distanza relativa di un oggetto dall'altro.
Risultati
Cellule microgliali che esprimono la proteina fluorescente verde (eGFP) e cellule endoteliali che esprimono DsRed in Tg(mpeg1:eGFPgl22; kdrl:cres898; actb2:loxP-STOP-loxP-DsRedexpress,sd5) tripli embrioni transgenici14 sono stati sottoposti a imaging a 3 dpf, secondo il protocollo descritto. Un singolo embrione di zebrafish è stato montato in agarosio a basso punto di fusione all'1% su una lastra di vetro inferiore e il processo di imaging non ha ostacolato la crescita dell'embrione durante il periodo di acquisizione. Il timelapse è stato registrato utilizzando un sistema di microscopia confocale a scansione puntiforme commerciale dotato di una lente dell'obiettivo a secco 10x 0,45 NA e laser di eccitazione da 488 nm e 561 nm sono stati utilizzati rispettivamente per l'imaging della microglia e delle cellule endoteliali. Inoltre, l'intervallo di tempo, la risoluzione dell'immagine, la dimensione dei pixel e il passo z erano rispettivamente di 30 secondi (s), 1024x1024, 0,49 μm e 2,5 μm. La registrazione timelapse è durata 3,5 ore (h).
Il campo visivo acquisito copriva l'intera testa dell'embrione, ma l'analisi si è concentrata specificamente sul tectum ottico, poiché durante la prima settimana di sviluppo, le microglia sono principalmente limitate allo strato soma neuronale di questa regione del mesencefalo dorsale, consentendo agli investigatori di visualizzare l'intera popolazione contemporaneamente15. L'analisi del tracking 3D è stata eseguita utilizzando Imaris 10.0, come spiegato sopra. Come mostrato nella Figura 4, il tracciamento ha avuto successo, risultando in 25 tracce, che corrispondono al numero previsto di cellule microgliali presenti nel tectum ottico a 3 dpf16. È stata richiesta una correzione manuale minima delle tracce. Nella Figura 5 viene illustrato un esempio dei dati che è possibile estrarre da un esperimento di rilevamento riuscito. La marcatura simultanea di macrofagi e cellule endoteliali consente la quantificazione della posizione della microglia rispetto a queste ultime, consentendo ai ricercatori di visualizzare la distanza relativa di ciascuna cellula dalla cellula endoteliale più vicina nel tempo ed esaminare la frequenza e il numero di potenziali interazioni (Figura 5).
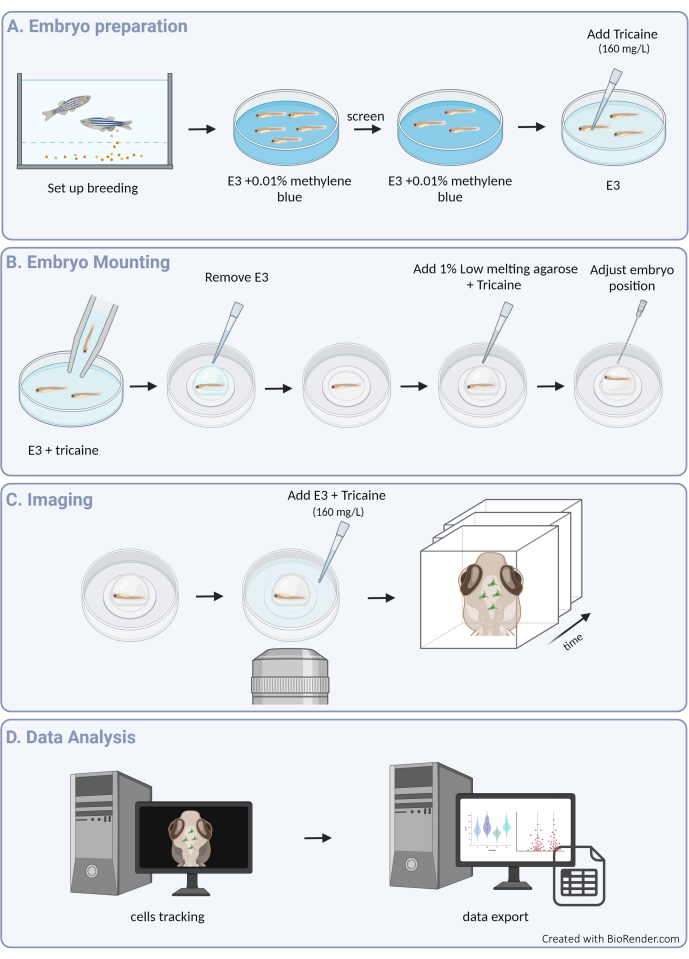
Figura 1: Panoramica della procedura sperimentale. (A) Preparazione dell'embrione di zebrafish e anestesia. (B) Montaggio e posizionamento del campione. (C) Acquisizione dell'immagine. (D) Elaborazione delle immagini ed estrazione dei dati sulla motilità. Clicca qui per visualizzare una versione più grande di questa figura.
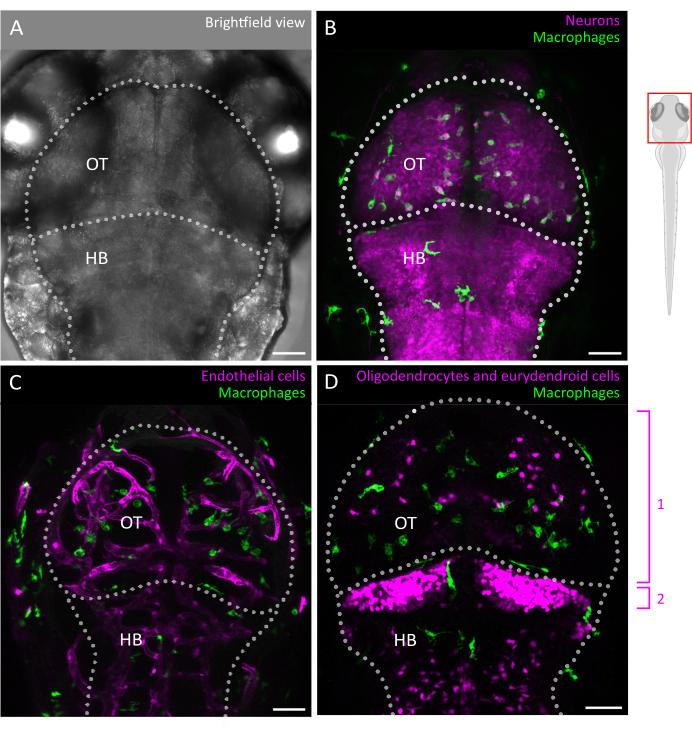
Figura 2: Imaging della microglia e interazioni con l'ambiente cellulare cerebrale nell'embrione di zebrafish. (A) Immagine in campo chiaro della testa e del cervello di un pesce zebra embrionale a 3 dpf (vista dorsale), che etichetta il tectum ottico e il rombencefalo. Il cervello del pesce zebra embrionale può essere ripreso nella sua interezza in questa fase grazie alle sue piccole dimensioni e alla trasparenza ottica. (B-D) La posizione e il comportamento delle microglia possono essere visualizzati mediante l'uso di linee transgeniche come (B,C) Tg(mpeg1:eGFP)gl22 e (D) Tg(mpeg1:mcherry)gl23, e concentrandosi sull'OT. (B) I neuroni e i loro corpi cellulari possono essere identificati utilizzando la linea reporter Tg(XlTubb:D sRed)zf148 e le interazioni microglia-neurone possono essere visualizzate fondendo i due canali in linee che co-esprimono entrambi i transgeni. Si vede una fusione dei segnali verdi (microglia) e rossi (neuroni, in magenta). (C, D) Le linee reporter possono anche far luce sulle interazioni della microglia (in verde) con le cellule endoteliali, qui etichettate in rosso utilizzando la doppia Tg(kdrl:cres898 (C); actb2:LOXP-STOP-LOXP-Dsredexpress, sd5) transgenici, o con cellule gliali come oligodendrociti e i loro precursori, utilizzando il (D) doppio Tg(olig2:EGFP; MPEG1:MCHERRY ) linea transgenica. 1: oligodendrociti e cellule progenitrici nell'OT, 2: neuroni euridendroidi nel cervelletto. Barre di scala = 50 μm. Abbreviazioni: dpf = giorni dopo la fecondazione; OT = tectum ottico; HB = rombencefalo. Clicca qui per visualizzare una versione più grande di questa figura.
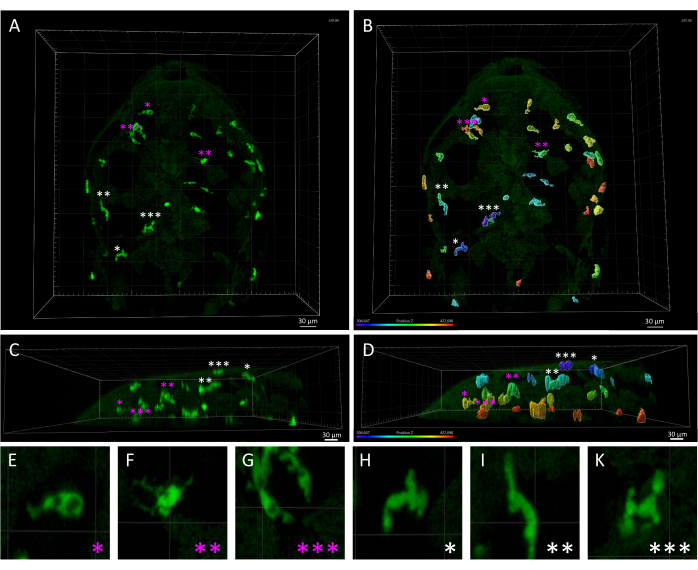
Figura 3: Disparità morfologica e spaziale tra macrofagi cutanei e microglia in un embrione di zebrafish a 3 dpf. (A,B) Vista dorsale di un embrione di zebrafish Tg(mpeg1:eGFP)gl22 a 3 dpf, raffigurante (A) cellule di microglia parenchimale (contrassegnate da asterischi magenta) rispetto a macrofagi cutanei (asterischi bianchi), identificate in base alla loro posizione relativa lungo l'asse z, come visto in (B), che mostra l'immagine 3D monocromatica renderizzata con la codifica a colori dell'asse z. (C, D) Vista laterale di (C) A e (D) B, che mostra le profondità z delle cellule mpeg1:eGFP+ all'interno della testa dell'embrione ed evidenzia la localizzazione superficiale dei macrofagi cutanei rispetto alle cellule microgliali. (E-K) L'elevato ingrandimento di ciascuna cellula è indicato da un asterisco in A e B, fornendo una visualizzazione dettagliata delle distinte morfologie tra le microglia ameboidi (E-G) e i macrofagi cutanei allungati (H-K). Barre di scala= 30 μm. Abbreviazione: dpf = giorni dopo la fecondazione. Clicca qui per visualizzare una versione più grande di questa figura.
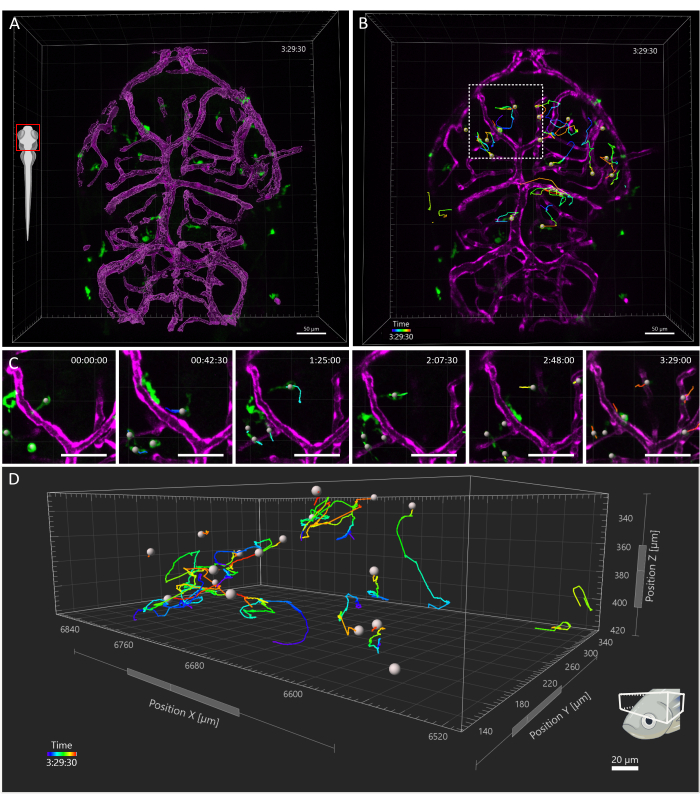
Figura 4: Tracciamento delle cellule microgliali in un embrione di zebrafish da 3 dpf. (A) Vista dorsale della testa di un Tg(kdrl:cres898 da 3 dpf; actb2:LOXP-STOP-LOXP-Dsredespresso,sd5; MPEG1:eGFPgl22) triplo embrione transgenico, che mostra la microglia (verde) e la resa superficiale dei vasi (magenta). (B-D) Tracciamento rappresentativo dei movimenti della microglia in una finestra temporale di (B) 3,5 ore. Si vede che le singole cellule seguono traiettorie complesse. (C) Dettaglio dal timelapse, che mostra una vista ingrandita della regione in B racchiusa dal quadrato tratteggiato. Vengono presentati sei fotogrammi del film (a 45 minuti di distanza) che documentano le microglia che stabiliscono contatti transitori con le cellule endoteliali nel loro microambiente. (D) Traiettorie della migrazione delle singole cellule microgliali all'interno del tectum ottico, con gli assi X, Y e Z che rappresentano le dimensioni spaziali. Barra di scala = 50 μm. Abbreviazione: dpf = giorni dopo la fecondazione. Clicca qui per visualizzare una versione più grande di questa figura.
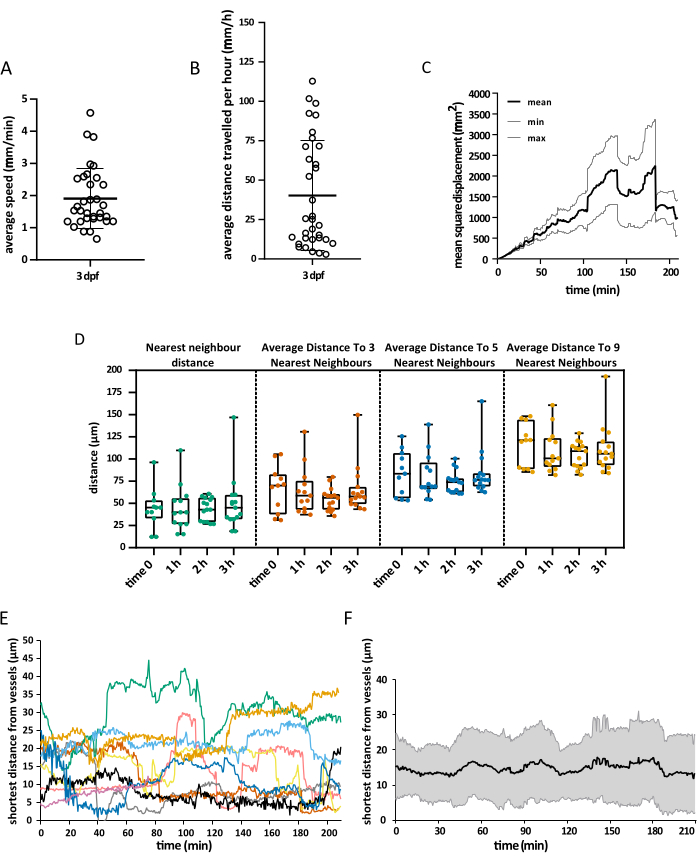
Figura 5: Visualizzazione dei dati ottenuti. Esempio dei dati spaziali che possono essere ottenuti utilizzando il protocollo descritto. I grafici raffigurano (A) la velocità media delle cellule microgliali, (B) la distanza media che percorrono in 1 ora, (C) il loro spostamento quadratico medio e (D) la loro distribuzione nello spazio in tempi diversi. Il rendering della superficie del vaso ha anche permesso di misurare la distanza più breve tra la microglia e le cellule endoteliali in un dato momento, sia a livello di singola cellula (E) che come media globale (F). Per A, B e D, ogni punto rappresenta una singola cella. N = un embrione. Clicca qui per visualizzare una versione più grande di questa figura.
File supplementare 1: Fare clic qui per scaricare questo file.
Discussione
L'attuale protocollo consente l'imaging in vivo delle dinamiche della microglia in un embrione di vertebrato e la visualizzazione dei dati di motilità acquisiti. La colonizzazione della microglia del cervello in via di sviluppo avviene molto presto durante l'embriogenesi e precede eventi critici come picchi di neurogenesi, astrogliogenesi, oligodendrogenesi e molti altri processi cellulari17. Non sorprende quindi che le microglia svolgano funzioni importanti nel plasmare aspetti specifici dello sviluppo cerebrale18, ad esempio, attraverso la regolazione della differenziazione, della migrazione e della sopravvivenza neuronale 19,20,21, nonché la potatura sinaptica 5 e la mielinizzazione 22,23,24.
Anche il contributo della microglia disfunzionale alla patogenesi e/o alla progressione dei disturbi dello sviluppo neurologico è sempre più riconosciuto25. Infatti, la presenza precoce di microglia nel cervello in formazione espone queste cellule a stati fisiologici distinti26 e cambiamenti ambientali. Questo può avere un impatto significativo dato che le microglia sono cellule longeve sia nei roditori che nell'uomo, e vengono mantenute durante la vita attraverso l'autorinnovamento dei progenitori locali 27,28,29. Riteniamo che questo protocollo possa servire come potente strumento per caratterizzare meglio il comportamento delle microglia in questi stati fisiologici distinti, mentre si sviluppano, maturano e stabiliscono la loro rete durante le fasi successive della morfogenesi cerebrale.
Utilizzando la configurazione qui descritta, abbiamo acquisito con successo l'imaging e i dati su larve di zebrafish vecchie fino a 6 dpf. L'estensione dell'analisi alle fasi di sviluppo successive avrà probabilmente successo, ma richiederà la regolazione della configurazione di imaging per prendere in considerazione l'aumento delle dimensioni del campione, in particolare lungo l'asse z. Nel tentativo di farlo, suggeriamo di concentrarsi sul mantenimento di un basso rapporto segnale/rumore e di un tempo di scansione rapido, poiché sono parametri chiave per un'analisi di successo.
Suggeriamo un tempo minimo di imaging di 1 ora per consentire il tracciamento della microglia; La finestra di imaging più lunga che è stata testata con questo protocollo è di 8 ore. Inoltre, è importante che l'analisi del tracking mantenga l'intervallo di tempo tra i fotogrammi il più breve possibile, idealmente tra 30 s e 60 s. Ciò consentirà di ottenere dati di tracciamento più accurati e dettagliati nelle analisi a valle. Pertanto, soprattutto se si rileva più di un fluoroforo, è fondamentale evitare la sovrapposizione spettrale e garantire una separazione sufficiente tra i due spettri di emissione del fluoroforo per consentire un'acquisizione simultanea, senza bleedthrough del segnale.
Altri protocolli per la registrazione timelapse di alta qualità del cervello del pesce zebra sono disponibili30, ma questo è il primo che mostra come tracciare con successo tutti i movimenti della microglia durante lo sviluppo embrionale per un periodo prolungato. Sebbene il flusso di lavoro qui presentato si concentri sul monitoraggio delle microglia in un contesto fisiologico, può essere facilmente applicato all'analisi delle microglia in patologia. Infatti, diversi modelli di disturbi dello sviluppo neurologico, come l'autismo31, l'epilessia32 e la schizofrenia33, ma anche la neurodegenerazione34 e il cancro35, sono stati stabiliti nel pesce zebra che forniscono opportunità uniche per determinare la risposta e il comportamento microgliale in condizioni di malattia.
In particolare, questo protocollo di tracciamento è altamente versatile e potrebbe anche essere strumentale per far luce sui modelli di migrazione di vari tipi di cellule attraverso diverse regioni anatomiche dell'embrione di zebrafish, aprendo così potenzialmente strade per ulteriori applicazioni, oltre all'ambito di indagine microgliale descritto in questo articolo. Inoltre, sfruttando la capacità di combinare più linee transgeniche fluorescenti, acquisiamo la capacità di discernere la relazione spaziale tra la microglia e altri tipi di cellule del microambiente cerebrale, con il potenziale di visualizzare le interazioni cellulari e i cross-talk durante le registrazioni timelapse, in modo non invasivo. Questo potrebbe essere determinante per svelare il significato fisiologico del comportamento delle microglia e contribuire a una caratterizzazione più profonda di queste cellule altamente mobili.
Divulgazioni
Gli autori dichiarano di non avere conflitti di interesse.
Riconoscimenti
Gli autori desiderano esprimere la loro sincera gratitudine al professor Nicolas Bayens per aver generosamente fornito l'accesso al microscopio confocale essenziale per questo studio. Questo lavoro è stato finanziato in parte dai Fondi per la Ricerca Scientifica (FNRS) nell'ambito delle sovvenzioni F451218F e UG03019F, l'Alzheimer Research Foundation (SAO-FRA) (a V.W.), A.M. è supportato da una borsa di ricerca della FNRS. La Figura 1 è stata creata il biorender.com.
Materiali
| Name | Company | Catalog Number | Comments |
| 1 L Breeding tanks | Tecniplast | ZB10BTE | |
| 1-phenyl-2-thiourea (PTU) | Sigma-Aldrich | P7629 | Diluted to 0.2 mM in E3 to prevent embryo pigmentation |
| Bottom glass imaging dish | FluoroDish | FD3510-100 | |
| Disposable Graduated transfer pipette | avantor | 16001-188 | |
| Dry block heater | Novolab | Grant QBD4 | To keep low melting agarose at 37 °C |
| Ethyl 3-aminobenzoate methanesulfonate (Tricaine) | Sigma-Aldrich | E10521-50G | |
| Imaris 10.0 | Oxford Instruments | analysis software | |
| Imaris File Converter | Oxford Instruments | https://imaris.oxinst.com/big-data | |
| Laser-scanning confocal microscope | Nikon | Eclipse Ti2-E | |
| Methylene blue | Sigma-Aldrich | M9140-25G | |
| microloader tips | Eppendorf | 5242956003 | |
| NuSieve GTG Agarose | Lonza | 50081 | |
| Petri dishes (90 mm) | avantor | 391-0559 | |
| Pronase | Sigma-Aldrich | 11459643001 | |
| Stainless Steel Forceps Dumont No. 5 | FineScienceTools | 11254-20 | |
| Stereo microscope | Leica | Leica M80 | To mount the embryos |
| teasing needle | avantor | 76549-024 |
Riferimenti
- Wolf, S. A., Boddeke, H. W., Kettenmann, H. Microglia in physiology and disease. Annu Rev Physiol. 79, 619-643 (2017).
- Nimmerjahn, A., Kirchhoff, F., Helmchen, F. Resting microglial cells are highly dynamic surveillants of brain parenchyma in vivo. Sci Rep. 308 (5726), 1314-1318 (2005).
- Peri, F., Nusslein-Volhard, C. Live imaging of neuronal degradation by microglia reveals a role for v0-ATPase a1 in phagosomal fusion in vivo. Cell. 133 (5), 916-927 (2008).
- Marın-Teva, J. L., et al. Microglia promote the death of developing Purkinje cells. Neuron. 41 (4), 535-547 (2004).
- Paolicelli, R. C., et al. Synaptic pruning by microglia is necessary for normal brain development. Science. 333 (6048), 1456-1458 (2011).
- Hattori, Y. The microglia-blood vessel interactions in the developing brain. Neurosci Res. 187, 58-66 (2023).
- Colonna, M., Butovsky, O. Microglia function in the central nervous system during health and neurodegeneration. Annu Rev Immunol. 35, 441-468 (2017).
- Xu, H. -T., Pan, F., Yang, G., Gan, W. -B. Choice of cranial window type for in vivo imaging affects dendritic spine turnover in the cortex. Nat Neurosci. 10 (5), 549-551 (2007).
- Shin, J., Park, H. -C., Topczewska, J. M., Mawdsley, D. J., Appel, B. Neural cell fate analysis in zebrafish using olig2 BAC transgenics. Methods Cell Sci. 25 (1), 7-14 (2003).
- Chi, N. C., et al. Foxn4 directly regulates tbx2b expression and atrioventricular canal formation. Genes Dev. 22 (6), 734-739 (2008).
- Avdesh, A., et al. Regular care and maintenance of a zebrafish (Danio rerio) laboratory: An introduction. J Vis Exp. (69), e4196(2012).
- Bohnsack, B. L., Gallina, D., Kahana, A. Phenothiourea sensitizes zebrafish cranial neural crest and extraocular muscle development to changes in retinoic acid and IGF signaling. PLoS ONE. 6 (8), e22991(2011).
- Ariga, J., Walker, S. L., Mumm, J. S. Multicolor time-lapse imaging of transgenic zebrafish: visualizing retinal stem cells activated by targeted neuronal cell ablation. J Vis Exp. (43), e2093(2010).
- Ferrero, G., et al. Embryonic microglia derive from primitive macrophages and are replaced by cmyb-dependent definitive microglia in zebrafish. Cell Rep. 24 (1), 130-141 (2018).
- Xu, J., Wang, T., Wu, Y., Jin, W., Wen, Z. Microglia colonization of developing zebrafish midbrain is promoted by apoptotic neuron and lysophosphatidylcholine. Dev Cell. 38 (2), 214-222 (2016).
- Svahn, A. J., et al. Development of ramified microglia from early macrophages in the zebrafish optic tectum. Dev Neurobiol. 73 (1), 60-71 (2013).
- Thion, M. S., Garel, S. Microglial ontogeny, diversity and neurodevelopmental functions. Curr Opin Genet Dev. 65, 186-194 (2020).
- Bohlen, C. J., Friedman, B. A., Dejanovic, B., Sheng, M. Microglia in brain development, homeostasis, and neurodegeneration. Annu Rev Genet. 53, 263-288 (2019).
- Squarzoni, P., et al. Microglia modulate wiring of the embryonic forebrain. Cell Rep. 8 (5), 1271-1279 (2014).
- Schafer, D. P., et al. Microglia sculpt postnatal neural circuits in an activity and complement-dependent. Neuron. 74 (4), 691-705 (2012).
- Parkhurst, C. N., et al. Microglia promote learning-dependent synapse formation through brain-derived neurotrophic factor. Cell. 155 (7), 1596-1609 (2013).
- Green, L. A., O'Dea, M. R., Hoover, C. A., DeSantis, D. F., Smith, C. J. The embryonic zebrafish brain is seeded by a lymphatic-dependent population of mrc1+ microglia precursors. Nat. Neurosci. 25 (7), 849-864 (2022).
- Hughes, A. N., Appel, B. Microglia phagocytose myelin sheaths to modify developmental myelination. Nat Neurosci. 23 (9), 1055-1066 (2020).
- Yu, Q., et al. C1q is essential for myelination in the central nervous system (CNS). iScience. 26 (12), 108518(2023).
- Zengeler, K. E., Lukens, J. R. Innate immunity at the crossroads of healthy brain maturation and neurodevelopmental disorders. Nat Rev Immunol. 21 (7), 454-468 (2021).
- Paolicelli, R. C., et al. Microglia states and nomenclature: A field at its crossroads. Neuron. 110 (21), 3458-3483 (2022).
- Réu, P., et al. The lifespan and turnover of microglia in the human brain. Cell Rep. 20 (4), 779-784 (2017).
- Ajami, B., Bennett, J. L., Krieger, C., Tetzlaff, W., Rossi, F. M. V. Local self-renewal can sustain CNS microglia maintenance and function throughout adult life. Nat Neurosci. 10 (12), 1538-1543 (2007).
- Mildner, A., et al. Microglia in the adult brain arise from Ly-6ChiCCR2+ monocytes only under defined host conditions. Nat Neurosci. 10 (12), 1544-1553 (2007).
- Cook, Z. T., Brockway, N. L., Weissman, T. A. Visualizing the developing brain in living zebrafish using Brainbow and time-lapse confocal imaging. J Vis Exp. (157), e60593(2020).
- Tayanloo-Beik, A., et al. Zebrafish modeling of autism spectrum disorders, current status and future prospective. Front Psychiatry. 13, 911770(2022).
- D'Amora, M., et al. Zebrafish as an innovative tool for epilepsy modeling: State of the art and potential future directions. Int J Mol Sci. 24 (9), 7702(2023).
- Langova, V., Vales, K., Horacek, J. The role of zebrafish and laboratory rodents in schizophrenia research. Front Psychiatry. 11, 549232(2020).
- Chia, K., Klingseisen, A., Sieger, D., Priller, J. Zebrafish as a model organism for neurodegenerative disease. Front Mol Neurosci. 15, 940484(2022).
- Astell, K. R., Sieger, D. Zebrafish in vivo models of cancer and metastasis. Cold Spring Harb Perspect Med. 10 (8), 037077(2020).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon