需要订阅 JoVE 才能查看此. 登录或开始免费试用。
Method Article
通过基于微流控芯片的环介导等温扩增快速检测引起下呼吸道感染的细菌病原体
* 这些作者具有相同的贡献
摘要
各种细菌病原体如果不准确发现和及时治疗,会导致呼吸道感染并导致严重的健康问题。 通过环介导的等温扩增快速准确地检测这些病原体,可在临床环境中有效管理和控制呼吸道感染。
摘要
呼吸道感染 (RTI) 是临床环境中最常见的问题之一。快速准确地鉴定细菌病原体将为管理和治疗RTIs提供实用指南。本研究描述了一种通过多通道环介导的等温扩增 (LAMP) 快速检测引起呼吸道感染的细菌病原体的方法。LAMP是一种灵敏、特异的诊断工具,可快速检测细菌核酸,准确度高,可靠性高。与传统的细菌培养方法相比,所提出的方法具有显着的优势,传统的细菌培养方法非常耗时,并且通常需要更高的灵敏度来检测低水平的细菌核酸。本文介绍了利用LAMP检测下呼吸道样本(痰液、支气管灌洗液和肺泡灌洗液)的肺炎 克雷伯菌 感染及其多种合并感染的代表性结果。综上所述,多通道LAMP方法提供了一种快速有效的方法,用于识别临床样本中的单一和多种细菌病原体,有助于防止细菌性病原体的传播,并有助于RTIs的适当治疗。
引言
由细菌病原体引起的呼吸道感染(RTIs)是全球发病率和死亡率的主要原因1.它被定义为任何上呼吸道或下呼吸道症状,伴有持续 2-3 天的发烧。虽然上呼吸道感染比下呼吸道感染更常见,但慢性和复发性呼吸道感染也是常见的临床疾病,对个人构成巨大风险,并给医疗保健系统带来沉重负担2.RTIs的常见细菌性病原体包括肺炎链球菌3型、流感嗜血杆菌4型、金黄色葡萄球菌、大肠杆菌、肺炎克雷伯菌、麦芽窄食单胞菌等。这些致病菌通常定植在宿主鼻咽和上呼吸道的粘膜表面,引起喉咙痛和支气管炎等 RTI 的典型症状。当它们从上呼吸道扩散到下呼吸道的无菌区域时,它们会引起肺炎,并可能通过呼吸道在人与人之间传播5.在严重的情况下,它们还可能导致侵袭性细菌性疾病,尤其是菌血症性肺炎、脑膜炎和败血症,这些都是全球所有年龄组人群发病和死亡的主要原因。
传统的 RTI 检测包括使用咽拭子和痰液呼吸道样本进行微生物培养6.此外,酶联免疫吸附测定 (ELISA) 等血清学检测可检测血清中的抗体或抗原,而凝集试验可观察抗体和抗原的凝集反应以检测感染7。微生物培养被认为是诊断 RTI 的金标准,但其培养阳性率低、可靠性差和检测周期长限制了诊断效率8。实际上,快速准确地诊断RTIs对于精确根除细菌病原体至关重要。快速有效的检测方法有助于降低病原体的传播率,缩短感染持续时间,并减少不必要的抗生素使用9,10。基于分子生物学的方法大大加快了检测速度,例如聚合酶链反应 (PCR),它通过扩增靶基因的 DNA 序列来检测病原体。然而,传统的PCR需要复杂的温度循环设备,既繁琐又耗时。此外,使用PCR进行的每次DNA扩增(实时荧光定量PCR除外)都以电泳分离产物结束,这也需要时间。产品的可视化需要染料,其中许多染料具有致突变性或致癌性。因此,不断开发诊断RTI细菌病原体的新方法和技术势在必行。
环介导的等温扩增(LAMP)是一种新型的分子技术,最初由Notomi等人于2000年开发11。LAMP可以在稳定的等温条件下扩增DNA,无需复杂的温度循环设备,使其适用于快速检测,降低了设备的复杂性和成本12。LAMP 可以高灵敏度检测低浓度的靶标 DNA13。它使用多种特异性引物来提高靶序列的选择性并减少假阳性的可能性14。LAMP因其易用性、速度和直观的操作而逐渐在临床实验室中得到广泛使用,甚至用于检测RTI。在这项研究中,我们研究了 LAMP 在检测临床样本(痰液、支气管灌洗液和肺泡灌洗液)中较低 RTI 的有效性,如图 1 所示。很明显,与传统测试相比,LAMP在较低的RTI检测方面具有速度、灵敏度和易用性等优势,使其成为一个有前途的应用。
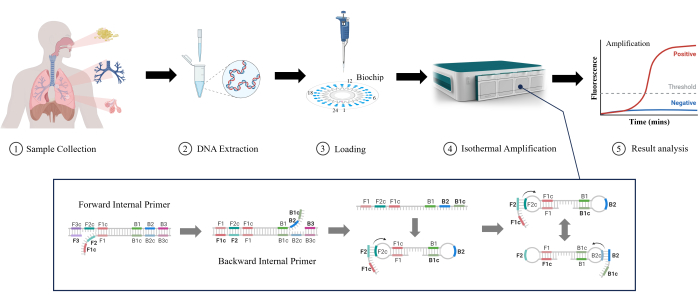
图 1:LAMP 检测方法示意图。 请点击这里查看此图的较大版本.
Access restricted. Please log in or start a trial to view this content.
研究方案
本研究所有样本均经广东省人民医院伦理审查委员会(批准文号:KY2023-1114-01)审定通过。所有参与者在实验前签署了书面知情同意书。用于研究的试剂和设备列在 材料表中。协议中使用的缩写列在 补充表1中。
1.采集下呼吸道临床样本
- 痰液收集
- 用清水清洁口腔和牙齿,如果适用,请确保摘下假牙。用力咳嗽,将深呼吸道痰液排出到痰液容器中(至少 0.6 mL)。
- 避免被唾液或鼻涕污染。如果咳嗽困难,应吸入 45 °C 的 100 g/L NaCl 溶液以促进咳痰。将标本转移到无菌、可密封的容器中。
注意:确保在早晨和使用任何抗菌药物之前收集痰液样本。
- 支气管灌洗液 (BLF)
- 将收集器头从鼻孔或气管切开部位(约 30 厘米深)插入气管。注入 5 mL 生理盐水,建立负压,旋转收集器头,然后慢慢退出。
- 收集提取的粘液,并用取样溶液(生理盐水或注射用无菌水)冲洗收集器一次。或者,将小儿导尿管连接到 50 mL 注射器,作为收集的替代品。
- 肺泡灌洗液 (ALF)
- 通过胸部影像学检查确定病变位置。选择最重要或进展最快的区域进行灌洗。
- 灌洗时通过活检通道将 1-2 mL 2% 利多卡因注射到支气管段,为灌洗肺段15 提供局部麻醉。对于静脉全身麻醉下仍表现出强烈气道反应的患者,注射 1-2 mL 2% 利多卡因。
- 局部麻醉后,将纤维支气管镜插入口腔或鼻子,穿过咽部进入右中叶支气管或左肺舌段。将其尖端放在支气管分支的开口处,慢慢引入无菌生理盐水。
- 每次给药 30-50 mL 生理盐水,体积为 100-250 mL(最大 300 mL)。将提取的粘液收集在密封的无菌容器中。
2. DNA提取
- 根据下呼吸道样品的粘度,加入适量的10%NaOH。对于低粘度的样品,加入液化液样品体积的2-3倍。对于粘度适中的样品,加入液化液样品体积的5-6倍。对于高粘度的样品,加入液化液样品体积的8-10倍。
- 根据样品粘度调节10%NaOH的体积。使用涡旋混合器尽可能均匀地分散样品15秒,并在37°C下孵育30分钟以液化。
注意:对于较厚的样品,增加 NaOH 的体积或延长液化时间。样品的液化质量直接影响后续的提取效率。理想情况下,液化样品应表现出一致的、不粘性的稠度。
- 根据样品粘度调节10%NaOH的体积。使用涡旋混合器尽可能均匀地分散样品15秒,并在37°C下孵育30分钟以液化。
- 将 1 mL 液化样品移液到 1.5 mL 离心管中。在2-6°C下以15,777× g 的速度离心5分钟,然后使用移液管除去并丢弃上清液。
注意: 吸出样品时,避免从管底部吸入杂质;如果杂质较多,可将样品以1,753-2,739 × g 离心1 min,然后再抽吸。 - 向离心管中加入 1 mL 洗涤液并涡旋以从离心管底部提起沉淀物。没有必要完全分散它。
- 将溶液以15,777× g 离心5分钟,弃去上清液,尽量不要接触沉淀物。
注意: 确保彻底去除洗涤液,以避免影响后续扩增。
- 将溶液以15,777× g 离心5分钟,弃去上清液,尽量不要接触沉淀物。
- 向离心管中加入 100 μL 核酸提取溶液。使用移液管吸出并彻底混合沉淀物。将液体和沉淀物一起转移到核酸提取管中。核酸提取溶液的组成如 表1所示。
- 将核酸提取管置于涡旋混合器中,并以中速涡旋至少5分钟。涡旋后,将核酸提取管转移到恒温金属浴中,并在100°C下加热5分钟。
- 在2-6°C下以15,777× g 离心5分钟,然后放在一边。
注意:如果在24小时内进行PCR扩增反应,则核酸可以储存在4°C。 扩增后,将核酸储存在-20°C。 对于长期储存(超过24小时),将核酸储存在-20°C。 当准备再次使用时,自然解冻样品,涡旋混合,在95°C水浴中加热5分钟,以10,956× g 离心1分钟,并使用上清液进行PCR扩增。
| 溶液 | 组件 | 数 | 规范 |
| 洗涤液 | 10毫米EDTA | 1瓶 | 24毫升/瓶 |
| 核酸提取试剂 | 10mM Tris-HCl,1mM EDTA,核酸防腐剂 | 2 管 | 1.2 mL/瓶 |
| 核酸提取管 | 玻璃珠 | 1 袋 | 24瓶/袋 |
表1:核酸提取试剂的组成。
3. 环介导等温放大及微流体芯片
- 试剂和微流控芯片
- 在微流控圆盘形芯片上进行等温放大反应(参见 材料表)。在65°C下进行恒温放大。
- 在恒温放大微流控芯片核酸分析仪上使用荧光染料掺入方法14 进行实时荧光分析。观察使用具有链置换功能的聚合酶的阳性样品的 S 形扩增曲线。
- 环介导的等温扩增
- 使用 4 个特异性引物靶向 6 个序列区域,包括 2 个内引物和 2 个外引物(随 LAMP 试剂盒提供)。
注:DNA 使用具有链置换功能的 DNA 聚合酶在恒定温度下连续复制和扩增。反应过程涉及哑铃形模板合成阶段、循环扩增阶段、伸长阶段和循环阶段,最终形成具有茎环和花椰菜状结构的DNA片段混合物。有关详细信息,请参阅套件说明(补充文件 1)。 - 在反应体系中加入双环引物以提高反应效率,该引物与茎环结构结合,启动链置换合成和循环复制。呼吸道病原体核酸检测试剂盒的组成见 表2。
注:试剂盒采用LAMP方法。该原理基于 DNA 在 65 °C 左右的动态平衡,其中任何通过双链 DNA 互补位点的碱基配对延伸的引物都会导致另一条链解离,形成单链。
- 使用 4 个特异性引物靶向 6 个序列区域,包括 2 个内引物和 2 个外引物(随 LAMP 试剂盒提供)。
- 微流控芯片
注:每个微流控芯片(见 材料表)都配有 24 个逆时针编号的反应孔,孔 1 的入口和出口对应于反应孔 1(图 2)。一组用于扩增和检测特定核酸靶序列的特定引物被嵌入并固定在每个反应孔中。- 将样品 DNA 与扩增试剂混合。将混合物注入芯片中,并将其分配到每个反应孔中,在室温下以 1,000 × g 离心 30 秒。
注意:独立的等温扩增反应和实时荧光检测在芯片上的每个反应孔中同时发生。如果在特定反应孔中检测到 S 形扩增曲线,则该孔的相应检测指数为正。
- 将样品 DNA 与扩增试剂混合。将混合物注入芯片中,并将其分配到每个反应孔中,在室温下以 1,000 × g 离心 30 秒。
| 组件 | 组成 | 数 |
| 芯片 | 引 | 12个地方 |
| 封口膜 | / | 1 张 |
| 等温扩增试剂 | 荧光染料、酵素 | 270 μL/管 |
| 阳性对照 | 大肠杆菌基因组DNA | 160 μL/管 |
表2:呼吸道病原体核酸检测试剂盒的组成。
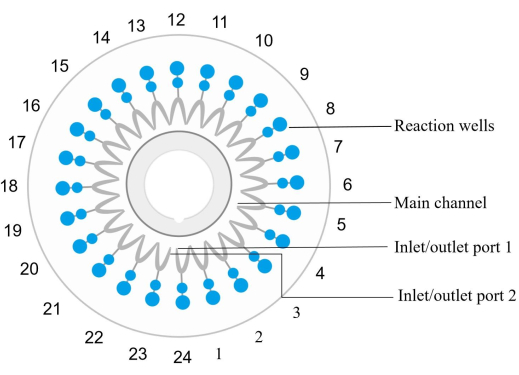
图2:光盘芯片结构图。 反应孔按逆时针顺序编号,其中入口/出口 1 对应于反应孔编号 1。反应孔 1、4、7、10、13、16、19、22 和 24 为阴性对照。反应孔6为阳性对照(大肠杆菌)。反应孔12为内部正对照,反应孔23为外部正对照。反应孔 2 检测到 spn。反应孔 3 检测到 sau。反应孔5检测 MRSA。反应孔8检测kpn。反应孔9检测 pae。反应孔11检测到 aba。反应孔14检测 SMA。反应孔15检测 hin。有关示例详细信息,请参阅 表 5 。 请点击这里查看此图的较大版本.
4. 样品制备和细菌检测
- 制备等温扩增反应体系
- 从试剂盒中取出等温扩增试剂,使其在室温下完全解冻。轻轻摇晃以充分混合,然后短暂离心以收集在管底部。
- 在试剂储存和制备区,将 20 μL 等温扩增试剂移液到准备好的 200 μL 离心管中。盖住试管并将其移动到样品制备区域,每个样品用一个离心管。
- 在标本制备区,加入 34.5 μL 目标核酸样品。轻轻摇晃以充分混合,然后短暂离心以收集在试管底部。每个核酸扩增反应系统的总体积为54.5μL(表3)。
- 切屑装载程序
- 在芯片包装上,标明样品编号。打开微流控芯片,包装标签朝上。
- 放置芯片时,入口和出口口朝上。使用移液管,吸取 50 μL 制备的扩增反应系统,并通过入口将其添加到芯片的主通道中。当主通道充满时停止添加,并迅速用无绒纸巾擦去入口和出口周围的多余液体。
- 用镊子捡起一层密封膜,将其覆盖在入口和出口上。将干净的移液器吸头沿一个方向按在密封膜上。确保紧密密封。
- qPCR反应
- 核酸分析仪的光源完成预热后,点击 打开托盘 按钮。将芯片正面朝上放在托盘上,确保定位小圆柱体从芯片的中心间隙中伸出以固定芯片。将托盘中的中心定位装置与芯片中的大中心孔对齐。
- 单击" 关闭托盘 "按钮,将芯片插入核酸分析仪。
- 在检测界面的 "样品信息 "区域,输入样品的检测信息。样品数量、芯片数量和样品类型是强制性的;其他的则是可选的。
- 单击操作区域中的 "开始检测 "按钮,开始样品检测。仪器将按照预设的程序进行样品测试。
注意: 检测完成后,设备的附带软件将自动进行数据分析。同时,核酸分析仪将启动自动冷却过程。一旦温度降至 37 °C,仪器会自动打开托盘并弹出芯片进行检索。结果将由系统自动解释(表 4)。
- 质量控制标准
- 该试剂盒的每个芯片中都封装了一个阳性质控品和九个阴性质控品。确保阳性质量控制的检测结果为阳性,同时,所有九种阴性质量控制的检测结果均为阴性。
注意: 结果显示在图像的右侧,显示"质量控制结果:实验正常(阳性正常,阴性正常)",表明测试结果有效。如果任何结果不正确,则需要重新测试,并且样本测试结果被视为无效。芯片中封装了一种使用人类特异性引物的阳性内部对照,其结果对临床样本检测为阳性,而如果样本的人类基因组 DNA 含量低,则结果为阴性,表明细胞计数较低。在这种情况下,收集并重新测试样本。
- 该试剂盒的每个芯片中都封装了一个阳性质控品和九个阴性质控品。确保阳性质量控制的检测结果为阳性,同时,所有九种阴性质量控制的检测结果均为阴性。
- 结果分析
- 检测完成后,利用二阶导数最大值方法结合软件中商业保护的算法,计算出S形放大曲线在快速放大阶段的第一拐点。详情见《呼吸道病原体核酸检测试剂盒说明书(补充文件1)》的"检测原理"部分。
注意:TP 值是拐点和原点之间的时间差。结果根据 TP 和正判断值进行解释。如果检测指标的TP值小于或等于该指标的正判断值,则被解释为正。如果 TP 值超过正判断值,则根据说明中"正决策值"的标准,将其解释为负值。"荧光曲线区域"显示归一化曲线,"检测结果"区域显示每个参数的质量控制和检测结果(表5)。
- 检测完成后,利用二阶导数最大值方法结合软件中商业保护的算法,计算出S形放大曲线在快速放大阶段的第一拐点。详情见《呼吸道病原体核酸检测试剂盒说明书(补充文件1)》的"检测原理"部分。
| 反应物 | 体积 (μl) |
| 等温扩增试剂 | 20 |
| 模板 DNA | 34.5 |
表3:等温扩增反应系统。
| 步 | 一 | 二 |
| 温度 (°C) | 37 | 65 |
| 时间(分钟) | 3 | 47 |
表4:核酸扩增反应程序。
| 指标名称 | 阳性对照值 |
| 肺炎链球菌 (sp) | 30 |
| 金黄色葡萄球菌 (sau) | 34 |
| 耐甲氧西林金黄色葡萄球菌 (mrsa) | 22 |
| 肺炎克雷伯菌 (kpn) | 29 |
| 铜绿假单胞菌(pae) | 36 |
| 鲍曼不动杆菌 (aba) | 36 |
| 嗜麦芽窄食单胞菌 (SMA) | 27 |
| 流感嗜血杆菌 (hin) | 36 |
表5:感染指标的阳性对照值。
Access restricted. Please log in or start a trial to view this content.
结果
该实验采用等温放大技术,在微流控圆盘芯片上进行反应。该反应在微流控芯片核酸分析仪上进行,采用荧光染料插入方法。等温反应在65°C的恒温下进行,同时进行实时荧光分析。阳性样品在具有链置换官能团的聚合酶作用下进行扩增,产生S形扩增曲线。这个一步法完成了靶基因的扩增和检测。本研究选取了7个实验结果不同的样品,验证了实验设计的可靠性。大写字母 S 代表样本。S1 是感染单...
Access restricted. Please log in or start a trial to view this content.
讨论
呼吸道感染是普遍存在的医院相关感染,会给患者带来严重后果,并使死亡率不断上升16。及时准确地识别潜在的病原体,然后使用有效的抗生素是成功治疗和改善预后的关键,特别是考虑到传统培养方法固有的局限性17.在这项研究中,我们使用基于 LAMP 的方法来确定单个或多个感染,以快速准确地检测 RTI。该快速检测系统是通过将LAMP技术与微流控技术相结合...
Access restricted. Please log in or start a trial to view this content.
披露声明
作者声明没有利益冲突。
致谢
我们非常感谢广东省基础与应用基础研究基金(批准号:2022A1515220023)和广东省人民医院高级人才研究基金(批准号:2022A1515220023)提供资金支持。KY012023293)。
Access restricted. Please log in or start a trial to view this content.
材料
| Name | Company | Catalog Number | Comments |
| Bath Incubator(MK2000-2) | ALLSHENG | Provide a constant temperature environment | |
| Bronchial lavage fluid collector head | TIANPINGHUACHANG | SEDA 20172081375 | Collecting bronchoalveolar lavage fluid |
| Fiberoptic bronchoscope | OLYMPUS | SEDA 20153062703 | A flexible bronchoscope equipped with a fiberoptic light source and camera, to visually examine the airways and structures within the lungs. Assist in collecting bronchoalveolar lavage |
HR1500- B2 B2 | Haier | SEDA 20183541642 | Biosafety cabinet |
| NAOH | MACKLIN | S817977 | Liquefy viscous lower respiratory tract sample |
| Nucleic acid detection kit for respiratory tract pathogens | Capitalbio Technology | SEDA 20173401346 | Testing for bacteria infection |
| Nucleic acid extraction reagent | Capitalbio Technology | SEDA 20160034 | For DNA extraction |
| RTisochip-W | Capitalbio Technology | SEDA 20193220539 | Loop-mediated Isothermal Amplification |
| THERMO ST16R | Thermo Fisher Scientific | SEDA 20180585 | Centrifuge the residual liquid off the wall of the tube. |
| Vortex mixer VM-5005 | JOANLAB | For mixing reagent |
参考文献
- GBD 2016 Lower Respiratory Infections Collaborators. Estimates of the global, regional, and national morbidity, mortality, and aetiologies of lower respiratory infections in 195 countries, 1990-2016: A systematic analysis for the global burden of disease study 2016. Lancet Infect Dis. 18 (11), 1191-1210 (2018).
- Niederman, M. S., Torres, A. Respiratory infections. Eur Respir Rev. 31 (166), 220150(2022).
- Weiser, J. N., Ferreira, D. M., Paton, J. C. Streptococcus pneumoniae: Transmission, colonization and invasion. Nat Rev Microbiol. 16 (6), 355-367 (2018).
- Watt, J. P., et al. Burden of disease caused by Haemophilus influenzae type b in children younger than 5 years_ global estimates. Lancet. 374 (9693), 903-911 (2009).
- Kadioglu, A., Weiser, J. N., Paton, J. C., Andrew, P. W. The role of streptococcus pneumoniae virulence factors in host respiratory colonization and disease. Nat Rev Microbiol. 6 (4), 288-301 (2008).
- Popova, G., Boskovska, K., Arnaudova-Danevska, I., Smilevska-Spasova, O., Jakovska, T. Sputum quality assessment regarding sputum culture for diagnosing lower respiratory tract infections in children. Open Access Maced J Med Sci. 7 (12), 1926-1930 (2019).
- Nuyttens, H., Cyncynatus, C., Renaudin, H., Pereyre, S., Identification Bébéar, C. expression and serological evaluation of the recombinant ATP synthase beta subunit of mycoplasma pneumoniae. BMC Microbiol. 10 (1), 216(2010).
- Noviello, S., Huang, D. B. The basics and the advancements in diagnosis of bacterial lower respiratory tract infections. Diagnostics (Basel). 9 (2), 37(2019).
- Hanson, K. E., et al. Molecular testing for acute respiratory tract infections: Clinical and diagnostic recommendations from the IDSA's diagnostics committee. Clin Infect Dis. 71 (10), 2744-2751 (2020).
- Daniel Reynolds, J. P. B., et al. The threat of multidrug-resistant/extensively drug-resistant gram-negative respiratory infections: Another pandemic. Eur Respir Rev. 31 (166), 220068(2022).
- T Notomi, H. O., et al. Loop-mediated isothermal amplification of DNA. Nucleic Acids Res. 28 (12), 63(2000).
- Soroka, M., Wasowicz, B., Rymaszewska, A. Loop-mediated isothermal amplification (LAMP): The better sibling of PCR. Cells. 10 (8), 1931(2021).
- Mori, Y., Kitao, M., Tomita, N., Notomi, T. Real-time turbidimetry of lamp reaction for quantifying template DNA. Biophys J. 59 (2), 145-157 (2004).
- Parida, M., Sannarangaiah, S., Dash, P. K., Rao, P. V., Morita, K. Loop mediated isothermal amplification (LAMP): A new generation of innovative gene amplification technique: Perspectives in clinical diagnosis of infectious diseases. Rev Med Virol. 18 (6), 407-421 (2008).
- Chinese Medical Association Respiratory Branch Critical Care Medicine Group, Working Committee on Critical Care Medicine of the Chinese Physicians Association Respiratory Physicians Branch. Standardization of collection, submission, testing, and interpretation of bronchoalveolar lavage fluid in ICU patients. Chin J Tubere Respir Dis. 43 (9), 744-756 (2020).
- Koch, A. M., Nilsen, R. M., Eriksen, H. M., Cox, R. J., Harthug, S. Mortality related to hospital-associated infections in a tertiary hospital: Repeated cross-sectional studies between 2004-2011. Antimicrob Resist Infect Control. 4 (1), 57(2015).
- Seymour, C. W. F. G., et al. Time to treatment and mortality during mandated emergency care for sepsis. N Engl J Med. 376 (23), 2235-2244 (2017).
- Choi, C. W., Hyun, J. W., Hwang, R. Y., Powell, C. A. Loop-mediated isothermal amplification assay for detection of candidatus Liberibacter asiaticus: A causal agent of citrus huanglongbing. Plant Pathol. 34 (6), 499-505 (2018).
- Büscher, P., Njiru, Z. K. Loop-mediated isothermal amplification technology: Towards point of care diagnostics. PLoS Negl Trop Dis. 6 (6), e1572(2012).
- Notomi, T., Mori, Y., Tomita, N., Kanda, H. Loop-mediated isothermal amplification (LAMP): Principle, features, and future prospects. J Microbiol. 53 (1), 1-5 (2015).
- Ajibola, O., Gulumbe, B., Eze, A., Obishakin, E. Tools for detection of schistosomiasis in resource limited settings. Medical Sciences. 6 (2), 39(2018).
- Kumar, Y. S. B., et al. Loop-mediated isothermal amplification (LAMP): A rapid and sensitive tool for quality assessment of meat products. Compr Rev Food Sci Food SAF. 16 (6), 1359-1378 (2017).
- Hongling Ou, Y. W., et al. Rapid detection of multiple pathogens by the combined loop-mediated isothermal amplification technology and microfluidic chip technology. Ann Palliat Med. 10 (10), 11053-11066 (2021).
- Liang Wang, J. -X. L., et al. Quantitative polymerase chain reaction (qPCR)-based rapid diagnosis of helicobacter pylori infection and antibiotic resistance. J Vis Exp. (197), e65689(2023).
- Jing-Wen Lyu, B. X., et al. Rapid prediction of multidrug-resistant klebsiella pneumoniae through deep learning analysis of SERS spectra. Microbiol Spectr. 11 (2), e0412622(2023).
- Wei Liu, J. -W. T., et al. Discrimination between carbapenem-resistant and carbapenem-sensitive klebsiella pneumoniae strains through computational analysis of surface-enhanced Raman spectra a pilot study. Microbiol Spectr. 10 (1), e0240921(2022).
- Zhang, L. Y., et al. Classification and prediction of klebsiella pneumoniae strains with different MLST allelic profiles via SERS spectral analysis. Peer J. 11, e16161(2023).
- Zhi Liu, Q. Z., et al. Rapid and sensitive detection of salmonella in chickens using loop-mediated isothermal amplification combined with a lateral flow dipstick. J Microbiol Biotechnol. 29 (3), 454-464 (2019).
- Chen, F., et al. Fully Integrated microfluidic platform for multiplexed detection of hunov by a dynamic confined-space-implemented one-pot RPA-LAMP system. Adv Sci (Weinh). 21, e2306612(2023).
Access restricted. Please log in or start a trial to view this content.
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。