Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Détection rapide d’agents pathogènes bactériens à l’origine d’infections des voies respiratoires inférieures grâce à une amplification isotherme médiée par boucle à base de puce microfluidique
* Ces auteurs ont contribué à parts égales
Dans cet article
Résumé
Divers agents pathogènes bactériens peuvent provoquer des infections des voies respiratoires et entraîner de graves problèmes de santé s’ils ne sont pas détectés avec précision et traités rapidement. La détection rapide et précise de ces agents pathogènes par amplification isotherme médiée par boucle permet une gestion et un contrôle efficaces des infections des voies respiratoires en milieu clinique.
Résumé
Les infections des voies respiratoires (IVR) sont parmi les problèmes les plus courants en milieu clinique. L’identification rapide et précise des agents pathogènes bactériens fournira des lignes directrices pratiques pour la gestion et le traitement des infections des voies respiratoires. Cette étude décrit une méthode permettant de détecter rapidement les agents pathogènes bactériens qui causent des infections des voies respiratoires par amplification isotherme médiée par boucle multicanal (LAMP). LAMP est un outil de diagnostic sensible et spécifique qui détecte rapidement les acides nucléiques bactériens avec une précision et une fiabilité élevées. La méthode proposée offre un avantage significatif par rapport aux méthodes traditionnelles de culture bactérienne, qui prennent du temps et nécessitent souvent une plus grande sensibilité pour détecter de faibles niveaux d’acides nucléiques bactériens. Cet article présente des résultats représentatifs de l’infection à K. pneumoniae et de ses multiples co-infections à l’aide de la LAMP pour détecter des échantillons (expectorations, liquide de lavage bronchique et liquide de lavage alvéolaire) des voies respiratoires inférieures. En résumé, la méthode LAMP multicanaux fournit un moyen rapide et efficace d’identifier un ou plusieurs agents pathogènes bactériens dans les échantillons cliniques, ce qui peut aider à prévenir la propagation des agents pathogènes bactériens et à faciliter le traitement approprié des infections des voies respiratoires.
Introduction
Les infections des voies respiratoires (IVR) causées par des agents pathogènes bactériens contribuent principalement à la morbidité et à la mortalité dans le monde1. Il est défini comme tout symptôme des voies respiratoires supérieures ou inférieures accompagné d’une fièvre d’une durée de 2 à 3 jours. Bien que les infections des voies respiratoires supérieures soient plus fréquentes que les infections des voies respiratoires inférieures, les infections chroniques et récurrentes des voies respiratoires sont également des affections cliniques courantes, posant de grands risques pour les individus et imposant un fardeau important aux systèmes de santé2. Les agents pathogènes bactériens courants des infections des voies respiratoires comprennent Streptococcus pneumoniae3, Haemophilus influenzae4, Staphylococcus aureus, Escherichia coli, Klebsiella pneumoniae, Stenotrophomonas maltophilia, entre autres. Ces bactéries pathogènes colonisent généralement les surfaces muqueuses du nasopharynx et des voies respiratoires supérieures de l’hôte, provoquant des symptômes typiques des infections des voies respiratoires telles que les maux de gorge et la bronchite. Ils provoquent une pneumonie lorsqu’ils se propagent des voies respiratoires supérieures aux zones stériles des voies respiratoires inférieures et peuvent se propager d’une personne à l’autre par les voies respiratoires5. Dans les cas graves, ils peuvent également entraîner des maladies bactériennes invasives, en particulier la pneumonie bactériémique, la méningite et la septicémie, qui sont les principales causes de morbidité et de mortalité chez les personnes de tous les groupes d’âge dans le monde.
Les tests traditionnels pour les infections respiratoires impliquent une culture microbiologique à l’aide d’écouvillons de gorge et d’échantillons respiratoires d’expectorations6. De plus, les tests sérologiques tels que le test immuno-enzymatique (ELISA) détectent des anticorps ou des antigènes dans le sérum, tandis que les tests d’agglutination observent la réaction d’agglutination des anticorps et des antigènes pour détecter l’infection7. La culture microbienne est considérée comme l’étalon-or pour diagnostiquer les infections des voies respiratoires, mais son faible taux de positivité des cultures, sa faible fiabilité et son long cycle de détection limitent l’efficacité du diagnostic8. En réalité, un diagnostic rapide et précis des infections respiratoires est crucial pour l’éradication précise de l’agent pathogène bactérien. Des méthodes de détection rapides et efficaces peuvent aider à réduire le taux de transmission des agents pathogènes, à raccourcir la durée de l’infection et à diminuer l’utilisation inutile d’antibiotiques 9,10. Les méthodes basées sur la biologie moléculaire accélèrent considérablement la détection, telles que la réaction en chaîne par polymérase (PCR), qui amplifie la séquence d’ADN d’un gène cible pour détecter les agents pathogènes. Cependant, la PCR traditionnelle nécessite un équipement complexe de cyclage de température, ce qui est lourd et chronophage. De plus, chaque amplification d’ADN par PCR (à l’exception de la PCR en temps réel) se termine par une séparation électrophorétique du produit, ce qui prend également du temps. La visualisation du produit nécessite des colorants, dont beaucoup sont mutagènes ou cancérigènes. Par conséquent, il est impératif de développer en permanence de nouvelles méthodes et technologies pour diagnostiquer les agents pathogènes bactériens RTI.
L’amplification isotherme médiée par boucle (LAMP) est une technologie moléculaire nouvelle et émergente initialement développée par Notomi et al. en 200011. LAMP peut amplifier l’ADN dans des conditions isothermes stables sans équipement de cyclage de température complexe, ce qui le rend adapté à une détection rapide et réduit la complexité et le coût de l’équipement12. LAMP peut détecter de faibles concentrations d’ADN cible avec une sensibilité élevée13. Il utilise plusieurs amorces spécifiques pour améliorer la sélectivité des séquences cibles et réduire la possibilité de faux positifs14. LAMP est progressivement largement utilisé dans les laboratoires cliniques en raison de sa facilité, de sa rapidité et de son fonctionnement intuitif, même pour la détection des RTI. Dans cette étude, nous avons examiné l’efficacité de la LAMP dans la détection des IVR plus faibles dans les échantillons cliniques (expectorations, liquide de lavage bronchique et liquide de lavage alvéolaire), comme le montre la figure 1. Il est évident que LAMP offre des avantages tels que la vitesse, la sensibilité et la facilité d’utilisation par rapport aux tests traditionnels en matière de détection à faible RTI, ce qui en fait une application prometteuse.
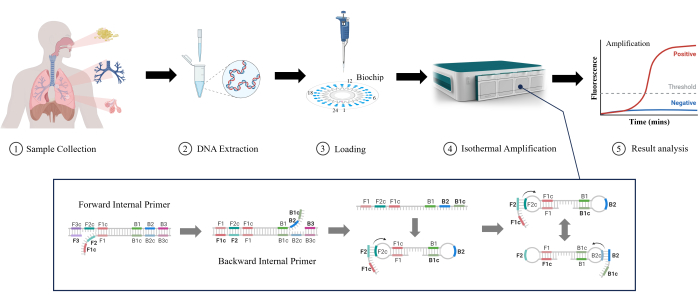
Figure 1 : Illustration schématique de la méthode de détection LAMP. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Access restricted. Please log in or start a trial to view this content.
Protocole
Tous les échantillons de cette étude ont été évalués et approuvés par le comité d’examen de l’éthique de l’hôpital populaire provincial du Guangdong (numéro d’approbation : KY2023-1114-01). Tous les participants ont signé un consentement éclairé écrit avant les expériences. Les réactifs et l’équipement utilisés pour l’étude sont énumérés dans la table des matériaux. Les abréviations utilisées dans le protocole sont énumérées dans le tableau supplémentaire 1.
1. Prélèvement d’échantillons cliniques dans les voies respiratoires inférieures
- Collecte d’expectorations
- Nettoyez la cavité buccale et les dents avec de l’eau claire, en veillant à ce que la prothèse dentaire soit retirée, le cas échéant. Toussez vigoureusement pour expulser les expectorations respiratoires profondes dans un contenant d’expectoration (minimum 0,6 ml).
- Évitez la contamination par la salive ou les écoulements nasaux. Si la toux est difficile, administrer l’inhalation d’une solution de NaCl de 100 g/L à 45 °C pour faciliter l’expectoration. Transférez l’échantillon dans un récipient stérile et refermable.
REMARQUE : Assurez-vous que les échantillons d’expectorations sont prélevés le matin et avant toute utilisation de médicament antibactérien.
- Liquide de lavage bronchique (BLF)
- Insérez la tête collectrice dans la trachée à partir de la narine ou du site de la trachéotomie (environ 30 cm de profondeur). Injectez 5 ml de solution saline, établissez une pression négative, tournez la tête du collecteur et retirez-la lentement.
- Prélever le mucus extrait et rincer une fois le collecteur avec une solution d’échantillonnage (solution saline physiologique ou eau stérile pour injection). Vous pouvez également connecter un cathéter urinaire pédiatrique à une seringue de 50 ml en remplacement du prélèvement.
- Liquide de lavage alvéolaire (ALF)
- Identifier l’emplacement de la lésion grâce à des examens d’imagerie thoracique. Sélectionnez la zone la plus importante ou la plus rapide pour le lavage.
- Injecter 1 à 2 ml de lidocaïne à 2 % dans le segment bronchique par le canal de biopsie pendant le lavage, en fournissant une anesthésie locale pour le segment pulmonaire lavé15. Pour les patients sous anesthésie générale intraveineuse qui présentent toujours de fortes réactions des voies respiratoires, injecter 1 à 2 ml de lidocaïne à 2 %.
- Après une anesthésie locale, insérez un bronchoscope à fibre optique par la bouche ou le nez, en passant par le pharynx dans la bronche du lobe moyen droit ou le segment lingual du poumon gauche. Placez son extrémité à l’ouverture de la branche bronchique et introduisez lentement une solution saline physiologique stérile.
- Administrer 30 à 50 ml de solution saline par séance, avec un volume de 100 à 250 ml (max. 300 ml). Recueillir le mucus extrait dans un récipient stérile scellé.
2. Extraction de l’ADN
- En fonction de la viscosité des échantillons des voies respiratoires inférieures, ajoutez une quantité appropriée de 10 % de NaOH. Pour les échantillons à faible viscosité, ajoutez 2 à 3 fois le volume de l’échantillon de solution de liquéfaction. Pour les échantillons de viscosité modérée, ajoutez 5 à 6 fois le volume de l’échantillon de solution de liquéfaction. Pour les échantillons à viscosité élevée, ajoutez 8 à 10 fois le volume de l’échantillon de solution de liquéfaction.
- Ajustez le volume de 10% de NaOH en fonction de la viscosité de l’échantillon. Répartir les échantillons de la manière la plus uniforme possible à l’aide d’un mélangeur vortex pendant 15 s et incuber à 37 °C pendant 30 min pour la liquéfaction.
REMARQUE : Pour des échantillons plus épais, augmentez le volume de NaOH ou prolongez le temps de liquéfaction. La qualité de liquéfaction des échantillons influence directement l’efficacité de l’extraction ultérieure. Idéalement, les échantillons liquéfiés doivent présenter une consistance constante et non collante.
- Ajustez le volume de 10% de NaOH en fonction de la viscosité de l’échantillon. Répartir les échantillons de la manière la plus uniforme possible à l’aide d’un mélangeur vortex pendant 15 s et incuber à 37 °C pendant 30 min pour la liquéfaction.
- Pipeter 1 mL de l’échantillon liquéfié dans un tube à centrifuger de 1,5 mL. Centrifuger à une vitesse de 15 777 × g pendant 5 min à 2-6 °C, puis utiliser une pipette pour retirer et jeter le surnageant.
REMARQUE : Lors de l’aspiration de l’échantillon, évitez d’aspirer des impuretés du fond du tube ; S’il y a beaucoup d’impuretés, l’échantillon peut être centrifugé à 1 753-2 739 × g pendant 1 min avant l’aspiration. - Ajouter 1 mL de solution de lavage dans le tube de centrifugation et tourbillonner pour soulever le précipité du fond du tube. Il n’est pas nécessaire de le disperser complètement.
- Centrifugez la solution à 15 777 × g pendant 5 min, jetez le surnageant et essayez de ne pas toucher le précipité.
REMARQUE : Assurez-vous d’éliminer complètement la solution de lavage pour éviter d’affecter l’amplification ultérieure.
- Centrifugez la solution à 15 777 × g pendant 5 min, jetez le surnageant et essayez de ne pas toucher le précipité.
- Ajouter 100 μL de solution d’extraction d’acide nucléique dans le tube à centrifuger. À l’aide d’une pipette, aspirez et mélangez bien le précipité. Transférez le liquide et précipitez ensemble dans un tube d’extraction d’acide nucléique. La composition de la solution d’extraction d’acide nucléique est présentée dans le tableau 1.
- Placez les tubes d’extraction d’acide nucléique dans un mélangeur vortex et tourbillonnez à vitesse moyenne pendant au moins 5 min. Après le vortex, transférez les tubes d’extraction d’acides nucléiques dans un bain métallique à température constante et à une chaleur de 100 °C pendant 5 min.
- Centrifuger à 15 777 × g à 2-6 °C pendant 5 min et réserver.
REMARQUE : Si la réaction d’amplification de la PCR est effectuée dans les 24 heures, l’acide nucléique peut être stocké à 4 °C. Après l’amplification, stockez l’acide nucléique à -20 °C. Pour un stockage à long terme (au-delà de 24 h), stocker l’acide nucléique à -20 °C. Lorsque vous êtes à nouveau prêt à l’utiliser, décongelez l’échantillon naturellement, mélangez au vortex, chauffez-le dans un bain-marie à 95 °C pendant 5 min, centrifugez-le à 10 956 × g pendant 1 min et utilisez le surnageant pour l’amplification par PCR.
| Solution | Composants | Nombre | Spécification |
| Solution de lavage | 10 mM EDTA | 1 bouteille | 24 mL/bouteille |
| Réactif d’extraction d’acide nucléique | 10 mM de Tris-HCl, 1 mM d’EDTA, conservateurs d’acides nucléiques | 2 tubes | 1,2 mL/bouteille |
| Tube d’extraction d’acide nucléique | Perles de verre | 1 sac | 24 bouteilles/sac |
Tableau 1 : Composition du réactif d’extraction d’acide nucléique.
3. Amplification isotherme médiée par boucle et puce microfluide
- Réactif et puce microfluidique
- Réaliser des réactions d’amplification isotherme sur une puce microfluidique en forme de disque (voir Tableau des matériaux). Effectuez l’amplification à température constante à 65 °C.
- Effectuez une analyse de fluorescence en temps réel à l’aide d’une méthode d’incorporation de colorant fluorescent14 sur l’analyseur d’acide nucléique à puce microfluidique à amplification à température constante. Observez une courbe d’amplification en forme de S pour les échantillons positifs à l’aide d’une polymérase avec fonctionnalité de déplacement de brin.
- Amplification isotherme médiée par boucle
- Ciblez six régions de séquence avec quatre amorces spécifiques, dont deux amorces internes et deux amorces externes (fournies avec le kit LAMP).
REMARQUE : L’ADN est répliqué et amplifié en continu à une température constante à l’aide d’une ADN polymérase avec fonction de déplacement de brin. Le processus de réaction implique l’étape de synthèse de matrice en forme d’haltère, l’étape d’amplification cyclique, l’étape d’allongement et l’étape de recyclage, formant finalement un mélange de fragments d’ADN avec des structures en forme de tige et de chou-fleur. Reportez-vous aux instructions de la trousse pour obtenir des renseignements détaillés (Fichier supplémentaire 1). - Ajoutez des amorces à deux boucles au système réactionnel pour améliorer l’efficacité de la réaction, qui se lie aux structures de la boucle de tige, initiant la synthèse par déplacement de brin et la réplication cyclique. La composition du kit de détection des acides nucléiques pour les agents pathogènes des voies respiratoires est présentée dans le tableau 2.
REMARQUE : Le kit de réactifs utilise la méthode LAMP. Le principe est basé sur l’équilibre dynamique de l’ADN à environ 65 °C, où toute amorce s’étendant par appariement de bases au site complémentaire de l’ADN double brin provoque la dissociation de l’autre brin, formant un seul brin.
- Ciblez six régions de séquence avec quatre amorces spécifiques, dont deux amorces internes et deux amorces externes (fournies avec le kit LAMP).
- Puce microfluidique
REMARQUE : Chaque puce microfluidique (voir le tableau des matériaux) est équipée de 24 puits de réaction numérotés dans le sens inverse des aiguilles d’une montre, l’entrée et la sortie du puits 1 correspondant au puits de réaction 1 (Figure 2). Un ensemble spécifique d’amorces pour l’amplification et la détection d’une séquence cible d’acide nucléique particulière est intégré et fixé dans chaque puits de réaction.- Mélangez l’ADN de l’échantillon avec le réactif d’amplification. Injecter le mélange dans la puce et le distribuer dans chaque centrifugeuse à 1 000 × g pendant 30 s à température ambiante.
REMARQUE : Des réactions d’amplification isotherme indépendantes et une détection de fluorescence en temps réel coexistent dans chaque puits de réaction sur la puce. Si une courbe d’amplification en forme de S est détectée dans un puits de réaction particulier, l’indice de détection correspondant pour ce puits est positif.
- Mélangez l’ADN de l’échantillon avec le réactif d’amplification. Injecter le mélange dans la puce et le distribuer dans chaque centrifugeuse à 1 000 × g pendant 30 s à température ambiante.
| Composants | Composition | Nombre |
| Puce | Amorces | 12 lieux |
| Film d’étanchéité | / | 1 feuille |
| Réactif d’amplification isotherme | Colorant fluorescent, Enzyme | 270 μL/tube |
| Contrôle positif | ADN génomique d’Escherichia coli | 160 μL/tube |
Tableau 2 : Composition du kit de détection des acides nucléiques pour les agents pathogènes des voies respiratoires.
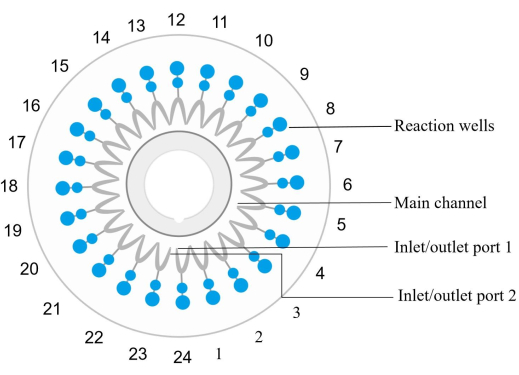
Figure 2 : Schéma de la structure de la puce du disque. Les puits de réaction sont numérotés séquentiellement dans le sens inverse des aiguilles d’une montre, l’orifice d’entrée/sortie 1 correspondant au puits de réaction numéro 1. Les puits de réaction 1, 4, 7, 10, 13, 16, 19, 22 et 24 sont des témoins négatifs. Bien réactionnelle 6 est un témoin positif (E. coli). Le puits de réaction 12 est un contrôle positif interne, et le puits de réaction 23 est un contrôle positif externe. Le puits de réaction 2 détecte spn. Réaction bien 3 détecte sau. Le puits de réaction 5 détecte le SARM. Le puits de réaction 8 détecte kpn. Le puits de réaction 9 détecte l’alcoolémie. La réaction bien 11 détecte aba. La réaction bien 14 détecte la sma. La réaction bien 15 détecte hin. Veuillez consulter le tableau 5 pour obtenir des détails sur les échantillons. Veuillez cliquer ici pour voir une version agrandie de cette figure.
4. Préparation des échantillons et détection des bactéries
- Préparer le système de réaction d’amplification isotherme
- Prenez le réactif d’amplification isotherme du kit et laissez-le décongeler complètement à température ambiante. Secouez doucement pour bien mélanger et centrifugez brièvement pour recueillir au fond du tube.
- Dans la zone de stockage et de préparation du réactif, pipeter 20 μL du réactif d’amplification isotherme dans un tube à centrifuger préparé de 200 μL. Couvrez le tube et déplacez-le vers la zone de préparation de l’échantillon à l’aide d’un tube à centrifuger par échantillon.
- Dans la zone de préparation de l’échantillon, ajoutez 34,5 μL de l’échantillon d’acide nucléique cible. Agitez doucement pour bien mélanger et centrifugez brièvement pour recueillir au fond du tube. Le volume total de chaque système de réaction d’amplification des acides nucléiques est de 54,5 μL (tableau 3).
- Procédure de chargement des copeaux
- Sur l’emballage de la puce, étiquetez le numéro de l’échantillon. Ouvrez la puce microfluidique avec l’étiquette d’emballage vers le haut.
- Placez la puce avec les orifices d’entrée et de sortie vers le haut. À l’aide d’une pipette, prélevez 50 μL du système de réaction d’amplification préparé et ajoutez-le au canal principal de la puce par l’orifice d’entrée. Arrêtez d’ajouter lorsque le canal principal est rempli et essuyez rapidement tout excès de liquide autour des orifices d’entrée et de sortie avec un chiffon non pelucheux.
- À l’aide d’une pince à épiler, prélevez un film d’étanchéité et couvrez-le sur les orifices d’entrée et de sortie. Appuyez une pointe de pipette propre sur le film d’étanchéité dans une direction. Assurez-vous d’une étanchéité parfaite.
- Réaction qPCR
- Une fois que la source lumineuse de l’analyseur d’acides nucléiques a terminé le préchauffage, cliquez sur le bouton Ouvrir le plateau . Placez la puce avec la face avant vers le haut sur le plateau, en veillant à ce que le petit cylindre de localisation émerge de l’espace central de la puce pour la fixer. Alignez le dispositif de positionnement central dans le plateau avec le grand trou central de la puce.
- Cliquez sur le bouton Fermer la barre d’état pour insérer la puce dans l’analyseur d’acides nucléiques.
- Dans la zone Informations sur l’échantillon de l’interface de détection, entrez les informations permettant de tester l’échantillon. Le numéro d’échantillon, le numéro de puce et le type d’échantillon sont obligatoires ; d’autres sont facultatifs.
- Cliquez sur le bouton Démarrer la détection dans la zone d’opération pour lancer la détection de l’échantillon. L’instrument effectuera des tests d’échantillonnage selon le programme prédéfini.
REMARQUE : Une fois la détection terminée, le logiciel qui l’accompagne de l’appareil effectuera automatiquement l’analyse des données. Simultanément, l’analyseur d’acides nucléiques lancera un processus de refroidissement automatique. Une fois que la température descend à 37 °C, l’instrument ouvre automatiquement le plateau et éjecte la puce pour la récupération. Les résultats seront interprétés automatiquement par le système (Tableau 4).
- Normes de contrôle de la qualité
- Un contrôle qualité positif et neuf contrôles qualité négatifs sont inclus dans chaque puce de ce kit de réactifs. Assurez-vous que les résultats de détection du contrôle qualité positif sont positifs et, simultanément, que les résultats des neuf contrôles qualité négatifs sont négatifs.
REMARQUE : Les résultats sont affichés sur le côté droit de l’image indiquant « Résultats du contrôle de la qualité : Expérience normale (normale positive, normale négative) », indiquant que les résultats du test sont valides. Un nouveau test est nécessaire si un résultat est incorrect et que le résultat du test de l’échantillon est jugé invalide. Un contrôle interne positif utilisant des amorces spécifiques à l’homme est enfermé dans la puce, dont le résultat est positif pour les tests d’échantillons cliniques alors qu’il est négatif si l’échantillon a un faible contenu en ADN génomique humain, indiquant un nombre de cellules plus faible. Dans de tels cas, récupérez et analysez à nouveau les échantillons.
- Un contrôle qualité positif et neuf contrôles qualité négatifs sont inclus dans chaque puce de ce kit de réactifs. Assurez-vous que les résultats de détection du contrôle qualité positif sont positifs et, simultanément, que les résultats des neuf contrôles qualité négatifs sont négatifs.
- Analyse des résultats
- Une fois la détection terminée, utilisez la méthode du maximum de dérivée seconde combinée à des algorithmes protégés commercialement dans le logiciel pour calculer le premier point d’inflexion de la courbe d’amplification en forme de S pendant la phase d’amplification rapide. Les détails sont décrits dans la partie « Principes de l’essai » du Guide d’instructions de la trousse de détection des acides nucléiques des agents pathogènes respiratoires (fichier supplémentaire 1).
REMARQUE : La valeur TP est la différence de temps entre le point d’inflexion et l’origine. Le résultat est interprété sur la base du TP et des valeurs de jugement positif. Si la valeur TP d’un indice de détection est inférieure ou égale à la valeur de jugement positif de cet indice, elle est interprétée comme positive. Si la valeur TP dépasse la valeur du jugement positif, elle est interprétée comme négative, selon les critères de « valeur de décision positive » dans les instructions. La « zone de la courbe de fluorescence » affiche la courbe normalisée, et la zone « Résultat de la détection » montre les résultats du contrôle de la qualité et de la détection pour chaque paramètre (Tableau 5).
- Une fois la détection terminée, utilisez la méthode du maximum de dérivée seconde combinée à des algorithmes protégés commercialement dans le logiciel pour calculer le premier point d’inflexion de la courbe d’amplification en forme de S pendant la phase d’amplification rapide. Les détails sont décrits dans la partie « Principes de l’essai » du Guide d’instructions de la trousse de détection des acides nucléiques des agents pathogènes respiratoires (fichier supplémentaire 1).
| Réactif | Volume (μl) |
| Réactif d’amplification isotherme | 20 |
| ADN matrice | 34.5 |
Tableau 3 : Système de réaction d’amplification isotherme.
| Pas | Un | Deux |
| Température (°C) | 37 | 65 |
| Temps (min) | 3 | 47 |
Tableau 4 : Programme de réaction d’amplification des acides nucléiques.
| Nom de l’indicateur | Valeur de contrôle positif |
| Streptococcus pneumoniae (sp) | 30 |
| Staphylococcus aureus (sau) | 34 |
| Staphylococcus aureus résistant à la méthicilline (SARM) | 22 |
| Klebsiella pneumoniae (kpn) | 29 |
| Pseudomonas aeruginosa (pae) | 36 |
| Acinetobacter baumannii (aba) | 36 |
| Stenotrophomonas maltophilia (sma) | 27 |
| Haemophilus influenzae (hin) | 36 |
Tableau 5 : Valeur de contrôle positif pour l’indicateur d’infection.
Access restricted. Please log in or start a trial to view this content.
Résultats
Cette expérience utilise la technologie d’amplification isotherme, en effectuant des réactions sur une puce de disque microfluidique. La réaction se produit sur un analyseur d’acide nucléique à puce microfluidique, en utilisant une méthode d’insertion de colorant de fluorescence. La réaction isotherme est réalisée à une température constante de 65 °C, et l’analyse de fluorescence en temps réel est effectuée simultanément. Les échantillons positifs subissent une amplification sous l’action de la ...
Access restricted. Please log in or start a trial to view this content.
Discussion
Les infections des voies respiratoires sont des infections nosocomiales répandues, qui ont de graves conséquences sur les patients et font grimper les taux de mortalité16. L’identification rapide et précise des agents pathogènes potentiels, suivie d’antibiotiques efficaces, est la clé du succès du traitement et de l’amélioration du pronostic, en particulier compte tenu des limites inhérentes aux méthodes de culture traditionnelles17. Dans cette étude, nous ...
Access restricted. Please log in or start a trial to view this content.
Déclarations de divulgation
Les auteurs ne déclarent aucun conflit d’intérêts.
Remerciements
Nous avons grandement apprécié le soutien financier fourni par la Fondation pour la recherche fondamentale et appliquée du Guangdong (subvention n° 2022A1515220023) et la Fondation de recherche pour les talents avancés de l’hôpital populaire de la province du Guandong (subvention n°. KY012023293).
Access restricted. Please log in or start a trial to view this content.
matériels
| Name | Company | Catalog Number | Comments |
| Bath Incubator(MK2000-2) | ALLSHENG | Provide a constant temperature environment | |
| Bronchial lavage fluid collector head | TIANPINGHUACHANG | SEDA 20172081375 | Collecting bronchoalveolar lavage fluid |
| Fiberoptic bronchoscope | OLYMPUS | SEDA 20153062703 | A flexible bronchoscope equipped with a fiberoptic light source and camera, to visually examine the airways and structures within the lungs. Assist in collecting bronchoalveolar lavage |
HR1500- B2 B2 | Haier | SEDA 20183541642 | Biosafety cabinet |
| NAOH | MACKLIN | S817977 | Liquefy viscous lower respiratory tract sample |
| Nucleic acid detection kit for respiratory tract pathogens | Capitalbio Technology | SEDA 20173401346 | Testing for bacteria infection |
| Nucleic acid extraction reagent | Capitalbio Technology | SEDA 20160034 | For DNA extraction |
| RTisochip-W | Capitalbio Technology | SEDA 20193220539 | Loop-mediated Isothermal Amplification |
| THERMO ST16R | Thermo Fisher Scientific | SEDA 20180585 | Centrifuge the residual liquid off the wall of the tube. |
| Vortex mixer VM-5005 | JOANLAB | For mixing reagent |
Références
- GBD 2016 Lower Respiratory Infections Collaborators. Estimates of the global, regional, and national morbidity, mortality, and aetiologies of lower respiratory infections in 195 countries, 1990-2016: A systematic analysis for the global burden of disease study 2016. Lancet Infect Dis. 18 (11), 1191-1210 (2018).
- Niederman, M. S., Torres, A. Respiratory infections. Eur Respir Rev. 31 (166), 220150(2022).
- Weiser, J. N., Ferreira, D. M., Paton, J. C. Streptococcus pneumoniae: Transmission, colonization and invasion. Nat Rev Microbiol. 16 (6), 355-367 (2018).
- Watt, J. P., et al. Burden of disease caused by Haemophilus influenzae type b in children younger than 5 years_ global estimates. Lancet. 374 (9693), 903-911 (2009).
- Kadioglu, A., Weiser, J. N., Paton, J. C., Andrew, P. W. The role of streptococcus pneumoniae virulence factors in host respiratory colonization and disease. Nat Rev Microbiol. 6 (4), 288-301 (2008).
- Popova, G., Boskovska, K., Arnaudova-Danevska, I., Smilevska-Spasova, O., Jakovska, T. Sputum quality assessment regarding sputum culture for diagnosing lower respiratory tract infections in children. Open Access Maced J Med Sci. 7 (12), 1926-1930 (2019).
- Nuyttens, H., Cyncynatus, C., Renaudin, H., Pereyre, S., Identification Bébéar, C. expression and serological evaluation of the recombinant ATP synthase beta subunit of mycoplasma pneumoniae. BMC Microbiol. 10 (1), 216(2010).
- Noviello, S., Huang, D. B. The basics and the advancements in diagnosis of bacterial lower respiratory tract infections. Diagnostics (Basel). 9 (2), 37(2019).
- Hanson, K. E., et al. Molecular testing for acute respiratory tract infections: Clinical and diagnostic recommendations from the IDSA's diagnostics committee. Clin Infect Dis. 71 (10), 2744-2751 (2020).
- Daniel Reynolds, J. P. B., et al. The threat of multidrug-resistant/extensively drug-resistant gram-negative respiratory infections: Another pandemic. Eur Respir Rev. 31 (166), 220068(2022).
- T Notomi, H. O., et al. Loop-mediated isothermal amplification of DNA. Nucleic Acids Res. 28 (12), 63(2000).
- Soroka, M., Wasowicz, B., Rymaszewska, A. Loop-mediated isothermal amplification (LAMP): The better sibling of PCR. Cells. 10 (8), 1931(2021).
- Mori, Y., Kitao, M., Tomita, N., Notomi, T. Real-time turbidimetry of lamp reaction for quantifying template DNA. Biophys J. 59 (2), 145-157 (2004).
- Parida, M., Sannarangaiah, S., Dash, P. K., Rao, P. V., Morita, K. Loop mediated isothermal amplification (LAMP): A new generation of innovative gene amplification technique: Perspectives in clinical diagnosis of infectious diseases. Rev Med Virol. 18 (6), 407-421 (2008).
- Chinese Medical Association Respiratory Branch Critical Care Medicine Group, Working Committee on Critical Care Medicine of the Chinese Physicians Association Respiratory Physicians Branch. Standardization of collection, submission, testing, and interpretation of bronchoalveolar lavage fluid in ICU patients. Chin J Tubere Respir Dis. 43 (9), 744-756 (2020).
- Koch, A. M., Nilsen, R. M., Eriksen, H. M., Cox, R. J., Harthug, S. Mortality related to hospital-associated infections in a tertiary hospital: Repeated cross-sectional studies between 2004-2011. Antimicrob Resist Infect Control. 4 (1), 57(2015).
- Seymour, C. W. F. G., et al. Time to treatment and mortality during mandated emergency care for sepsis. N Engl J Med. 376 (23), 2235-2244 (2017).
- Choi, C. W., Hyun, J. W., Hwang, R. Y., Powell, C. A. Loop-mediated isothermal amplification assay for detection of candidatus Liberibacter asiaticus: A causal agent of citrus huanglongbing. Plant Pathol. 34 (6), 499-505 (2018).
- Büscher, P., Njiru, Z. K. Loop-mediated isothermal amplification technology: Towards point of care diagnostics. PLoS Negl Trop Dis. 6 (6), e1572(2012).
- Notomi, T., Mori, Y., Tomita, N., Kanda, H. Loop-mediated isothermal amplification (LAMP): Principle, features, and future prospects. J Microbiol. 53 (1), 1-5 (2015).
- Ajibola, O., Gulumbe, B., Eze, A., Obishakin, E. Tools for detection of schistosomiasis in resource limited settings. Medical Sciences. 6 (2), 39(2018).
- Kumar, Y. S. B., et al. Loop-mediated isothermal amplification (LAMP): A rapid and sensitive tool for quality assessment of meat products. Compr Rev Food Sci Food SAF. 16 (6), 1359-1378 (2017).
- Hongling Ou, Y. W., et al. Rapid detection of multiple pathogens by the combined loop-mediated isothermal amplification technology and microfluidic chip technology. Ann Palliat Med. 10 (10), 11053-11066 (2021).
- Liang Wang, J. -X. L., et al. Quantitative polymerase chain reaction (qPCR)-based rapid diagnosis of helicobacter pylori infection and antibiotic resistance. J Vis Exp. (197), e65689(2023).
- Jing-Wen Lyu, B. X., et al. Rapid prediction of multidrug-resistant klebsiella pneumoniae through deep learning analysis of SERS spectra. Microbiol Spectr. 11 (2), e0412622(2023).
- Wei Liu, J. -W. T., et al. Discrimination between carbapenem-resistant and carbapenem-sensitive klebsiella pneumoniae strains through computational analysis of surface-enhanced Raman spectra a pilot study. Microbiol Spectr. 10 (1), e0240921(2022).
- Zhang, L. Y., et al. Classification and prediction of klebsiella pneumoniae strains with different MLST allelic profiles via SERS spectral analysis. Peer J. 11, e16161(2023).
- Zhi Liu, Q. Z., et al. Rapid and sensitive detection of salmonella in chickens using loop-mediated isothermal amplification combined with a lateral flow dipstick. J Microbiol Biotechnol. 29 (3), 454-464 (2019).
- Chen, F., et al. Fully Integrated microfluidic platform for multiplexed detection of hunov by a dynamic confined-space-implemented one-pot RPA-LAMP system. Adv Sci (Weinh). 21, e2306612(2023).
Access restricted. Please log in or start a trial to view this content.
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon