È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Rilevamento rapido di agenti patogeni batterici che causano infezioni del tratto respiratorio inferiore tramite amplificazione isotermica mediata da loop basata su chip microfluidico
* Questi autori hanno contribuito in egual misura
In questo articolo
Riepilogo
Vari agenti patogeni batterici possono causare infezioni del tratto respiratorio e portare a gravi problemi di salute se non rilevati con precisione e trattati tempestivamente. Il rilevamento rapido e accurato di questi patogeni tramite amplificazione isoterma mediata da loop fornisce una gestione e un controllo efficaci delle infezioni del tratto respiratorio in ambito clinico.
Abstract
Le infezioni delle vie respiratorie (RTI) sono tra i problemi più comuni in ambito clinico. L'identificazione rapida e accurata dei patogeni batterici fornirà linee guida pratiche per la gestione e il trattamento delle RTI. Questo studio descrive un metodo per rilevare rapidamente i patogeni batterici che causano infezioni del tratto respiratorio tramite amplificazione isotermica mediata da loop multicanale (LAMP). LAMP è uno strumento diagnostico sensibile e specifico che rileva rapidamente gli acidi nucleici batterici con elevata precisione e affidabilità. Il metodo proposto offre un vantaggio significativo rispetto ai metodi tradizionali di coltura batterica, che richiedono molto tempo e spesso una maggiore sensibilità per rilevare bassi livelli di acidi nucleici batterici. Questo articolo presenta risultati rappresentativi dell'infezione da K. pneumoniae e delle sue molteplici co-infezioni utilizzando LAMP per rilevare campioni (espettorato, liquido di lavaggio bronchiale e liquido di lavaggio alveolare) dal tratto respiratorio inferiore. In sintesi, il metodo LAMP multicanale fornisce un mezzo rapido ed efficiente per identificare patogeni batterici singoli e multipli in campioni clinici, che possono aiutare a prevenire la diffusione di patogeni batterici e aiutare nel trattamento appropriato degli RTI.
Introduzione
Le infezioni del tratto respiratorio (RTI) causate da agenti patogeni batterici contribuiscono principalmente alla morbilità e alla mortalità in tutto il mondo1. È definito come qualsiasi sintomo delle vie respiratorie superiori o inferiori accompagnato da febbre della durata di 2-3 giorni. Sebbene l'infezione delle vie respiratorie superiori sia più comune di quella delle vie respiratorie inferiori, anche le infezioni croniche e ricorrenti del tratto respiratorio sono condizioni cliniche comuni, che pongono grandi rischi agli individui e gravano in modo significativo sui sistemi sanitari2. I patogeni batterici comuni degli RTI includono Streptococcus pneumoniae3, Haemophilus influenzae4, Staphylococcus aureus, Escherichia coli, Klebsiella pneumoniae, Stenotrophomonas maltophilia, tra gli altri. Questi batteri patogeni di solito colonizzano le superfici mucose del rinofaringe e del tratto respiratorio superiore dell'ospite, causando sintomi tipici delle RTI come mal di gola e bronchite. Causano polmonite quando si diffondono dal tratto respiratorio superiore alle aree sterili del tratto respiratorio inferiore e possono diffondersi da persona a persona attraverso il tratto respiratorio5. Nei casi più gravi, possono anche portare a malattie batteriche invasive, in particolare polmonite batteriemica, meningite e sepsi, che sono le principali cause di morbilità e mortalità in persone di tutte le età in tutto il mondo.
I test tradizionali per le RTI prevedono la coltura microbiologica utilizzando tamponi faringei e campioni respiratori di espettorato6. Inoltre, i test sierologici come il test di immunoassorbimento enzimatico (ELISA) rilevano anticorpi o antigeni nel siero, mentre i test di agglutinazione osservano la reazione di agglutinazione di anticorpi e antigeni per rilevare l'infezione7. La coltura microbica è considerata il gold standard per la diagnosi di RTI, ma il suo basso tasso di positività della coltura, la scarsa affidabilità e il lungo ciclo di rilevamento limitano l'efficienza diagnostica8. In realtà, una diagnosi rapida e accurata degli RTI è fondamentale per l'eradicazione precisa del patogeno batterico. Metodi di rilevamento rapidi ed efficaci possono aiutare a ridurre il tasso di trasmissione degli agenti patogeni, abbreviare la durata dell'infezione e diminuire l'uso non necessario di antibiotici 9,10. I metodi basati sulla biologia molecolare accelerano significativamente il rilevamento, come la reazione a catena della polimerasi (PCR), che amplifica la sequenza di DNA di un gene bersaglio per rilevare gli agenti patogeni. Tuttavia, la PCR tradizionale richiede apparecchiature complesse per il ciclo della temperatura, che sono ingombranti e richiedono molto tempo. Inoltre, ogni amplificazione del DNA mediante PCR (ad eccezione della PCR in tempo reale) si conclude con la separazione elettroforetica del prodotto, che richiede anch'essa tempo. La visualizzazione del prodotto richiede coloranti, molti dei quali sono mutageni o cancerogeni. Pertanto, è imperativo sviluppare continuamente nuovi metodi e tecnologie per la diagnosi dei patogeni batterici RTI.
L'amplificazione isotermica mediata da loop (LAMP) è una tecnologia molecolare innovativa ed emergente inizialmente sviluppata da Notomi et al. nel 200011. LAMP è in grado di amplificare il DNA in condizioni isotermiche stabili senza complesse apparecchiature per il ciclo della temperatura, il che lo rende adatto per un rilevamento rapido e riduce la complessità e i costi delle apparecchiature12. LAMP è in grado di rilevare basse concentrazioni di DNA bersaglio con un'elevata sensibilità13. Utilizza più primer specifici per migliorare la selettività per le sequenze target e ridurre la possibilità di falsi positivi14. LAMP sta gradualmente diventando ampiamente utilizzato nei laboratori clinici grazie alla sua facilità, velocità e funzionamento intuitivo, anche per il rilevamento di RTI. In questo studio, abbiamo studiato l'efficacia della LAMP nel rilevare gli RTI più bassi in campioni clinici (espettorato, liquido di lavaggio bronchiale e liquido di lavaggio alveolare), come mostrato nella Figura 1. È evidente che LAMP offre vantaggi come velocità, sensibilità e facilità d'uso rispetto ai test tradizionali con rilevamento RTI inferiore, rendendolo un'applicazione promettente.
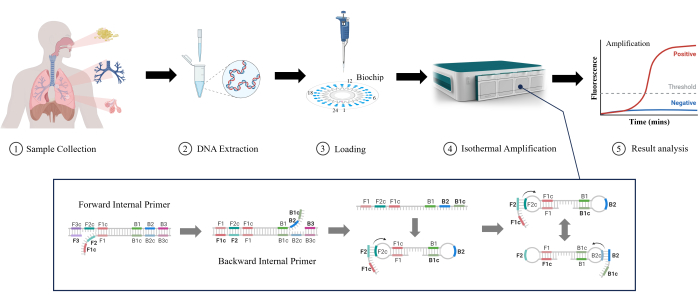
Figura 1: Illustrazione schematica del metodo di rilevamento LAMP. Fare clic qui per visualizzare una versione più grande di questa figura.
Access restricted. Please log in or start a trial to view this content.
Protocollo
Tutti i campioni per questo studio sono stati valutati e approvati dal Comitato di revisione etica dell'Ospedale del popolo provinciale del Guangdong (numero di approvazione: KY2023-1114-01). Tutti i partecipanti hanno firmato il consenso informato scritto prima degli esperimenti. I reagenti e le attrezzature utilizzate per lo studio sono elencati nella tabella dei materiali. Le abbreviazioni utilizzate nel protocollo sono elencate nella Tabella supplementare 1.
1. Raccolta di campioni clinici dalle basse vie respiratorie
- Raccolta dell'espettorato
- Detergere la cavità orale e i denti con acqua pulita, assicurandosi che la protesi sia rimossa se applicabile. Tossire con forza per espellere l'espettorato respiratorio profondo in un contenitore di espettorato (minimo 0,6 ml).
- Evitare la contaminazione con saliva o secrezione nasale. Se la tosse è difficoltosa, somministrare per inalazione una soluzione di NaCl 100 g/L a 45 °C per facilitare l'espettorazione. Trasferire il campione in un contenitore sterile e sigillabile.
NOTA: Assicurarsi che i campioni di espettorato vengano raccolti al mattino e prima dell'uso di qualsiasi farmaco antibatterico.
- Liquido per lavanda bronchiale (BLF)
- Inserire la testa del collettore nella trachea dalla narice o dal sito della tracheostomia (a circa 30 cm di profondità). Iniettare 5 mL di soluzione fisiologica, stabilire una pressione negativa, ruotare la testa del collettore ed estrarre lentamente.
- Raccogliere il muco estratto e sciacquare una volta il collettore con una soluzione di campionamento (soluzione fisiologica salina o acqua sterile per preparazioni iniettabili). In alternativa, collegare un catetere urinario pediatrico a una siringa da 50 ml come sostituto della raccolta.
- Liquido di lavaggio alveolare (ALF)
- Identificare la posizione della lesione attraverso esami di imaging del torace. Selezionare l'area più significativa o in rapida progressione per il lavaggio.
- Iniettare 1-2 ml di lidocaina al 2% nel segmento bronchiale attraverso il canale della biopsia durante il lavaggio, fornendo l'anestesia locale per il segmento polmonare lavato15. Per i pazienti in anestesia generale per via endovenosa che mostrano ancora forti reazioni alle vie aeree, iniettare 1-2 ml di lidocaina al 2%.
- Dopo l'anestesia locale, inserire un broncoscopio a fibre ottiche attraverso la bocca o il naso, passando attraverso la faringe nel bronco del lobo medio destro o nel segmento linguale del polmone sinistro. Posizionare la punta all'apertura del ramo bronchiale e introdurre lentamente soluzione fisiologica sterile.
- Somministrare 30-50 ml di soluzione fisiologica per sessione, con un volume di 100-250 ml (max. 300 ml). Raccogliere il muco estratto in un contenitore sterile sigillato.
2. Estrazione del DNA
- In base alla viscosità dei campioni del tratto respiratorio inferiore, aggiungere una quantità appropriata di NaOH al 10%. Per campioni a bassa viscosità, aggiungere 2-3 volte il volume del campione di soluzione liquefatta. Per campioni con viscosità moderata, aggiungere 5-6 volte il volume del campione di soluzione liquefante. Per campioni con alta viscosità, aggiungere 8-10 volte il volume del campione di soluzione liquefatta.
- Regolare il volume del 10% di NaOH in base alla viscosità del campione. Disperdere i campioni nel modo più uniforme possibile utilizzando un miscelatore a vortice per 15 s e incubare a 37 °C per 30 minuti per la liquefazione.
NOTA: Per campioni più spessi, aumentare il volume di NaOH o prolungare il tempo di liquefazione. La qualità della liquefazione dei campioni influenza direttamente la successiva efficienza di estrazione. Idealmente, i campioni liquefatti dovrebbero mostrare una consistenza costante e non appiccicosa.
- Regolare il volume del 10% di NaOH in base alla viscosità del campione. Disperdere i campioni nel modo più uniforme possibile utilizzando un miscelatore a vortice per 15 s e incubare a 37 °C per 30 minuti per la liquefazione.
- Pipettare 1 mL del campione liquefatto in una provetta da centrifuga da 1,5 mL. Centrifugare a una velocità di 15.777 × g per 5 minuti a 2-6 °C, quindi utilizzare una pipetta per rimuovere ed eliminare il surnatante.
NOTA: Durante l'aspirazione del campione, evitare di aspirare impurità dal fondo della provetta; Se sono presenti molte impurità, il campione può essere centrifugato a 1.753-2.739 × g per 1 minuto prima dell'aspirazione. - Aggiungere 1 mL di soluzione di lavaggio alla provetta da centrifuga e agitare per sollevare il precipitato dal fondo della provetta. Non è necessario disperderlo completamente.
- Centrifugare la soluzione a 15.777 × g per 5 minuti, scartare il surnatante e cercare di non toccare il precipitato.
NOTA: Assicurarsi che la soluzione di lavaggio sia completamente rimossa per evitare di compromettere la successiva amplificazione.
- Centrifugare la soluzione a 15.777 × g per 5 minuti, scartare il surnatante e cercare di non toccare il precipitato.
- Aggiungere 100 μl di soluzione di estrazione dell'acido nucleico alla provetta da centrifuga. Utilizzare una pipetta per aspirare e mescolare accuratamente il precipitato. Trasferire il liquido e precipitare insieme in una provetta per l'estrazione dell'acido nucleico. La composizione della soluzione di estrazione dell'acido nucleico è presentata nella Tabella 1.
- Posizionare le provette di estrazione dell'acido nucleico in un miscelatore a vortice e agitare a velocità media per almeno 5 minuti. Dopo il vortex, trasferire le provette di estrazione dell'acido nucleico in un bagno metallico a temperatura costante e riscaldare a 100 °C per 5 minuti.
- Centrifugare a 15,777 × g a 2-6 °C per 5 min e mettere da parte.
NOTA: Se la reazione di amplificazione della PCR viene eseguita entro 24 ore, l'acido nucleico può essere conservato a 4 °C. Dopo l'amplificazione, conservare l'acido nucleico a -20 °C. Per la conservazione a lungo termine (oltre le 24 ore), conservare l'acido nucleico a -20 °C. Al momento dell'uso, scongelare il campione in modo naturale, agitare per mescolare, scaldare in un bagno d'acqua a 95 °C per 5 minuti, centrifugare a 10.956 × g per 1 minuto e utilizzare il surnatante per l'amplificazione PCR.
| Soluzione | Componenti | Numero | Specificazione |
| Soluzione di lavaggio | 10 mM EDTA | 1 bottiglia | 24 ml/flacone |
| Reagente per l'estrazione dell'acido nucleico | 10mM Tris-HCl, 1mM EDTA, conservanti per acidi nucleici | 2 tubi | 1,2 ml/flacone |
| Provetta per l'estrazione dell'acido nucleico | Perle di vetro | 1 busta | 24 bottiglie/sacchetto |
Tabella 1: Composizione del reagente per l'estrazione dell'acido nucleico.
3. Amplificazione isotermica mediata da loop e chip di microfluidi
- Reagente e chip microfluidico
- Condurre reazioni di amplificazione isoterma su un chip microfluidico a forma di disco (vedi Tabella dei materiali). Eseguire l'amplificazione a temperatura costante a 65 °C.
- Esecuzione di analisi della fluorescenza in tempo reale utilizzando un metodo di incorporazione di coloranti fluorescenti14 sull'analizzatore di acidi nucleici a chip microfluidico con amplificazione a temperatura costante. Osservare una curva di amplificazione a forma di S per campioni positivi utilizzando una polimerasi con funzionalità di spostamento del filamento.
- Amplificazione isoterma mediata da loop
- Mira a sei regioni di sequenza con quattro primer specifici, inclusi due primer interni e due primer esterni (forniti con il kit LAMP).
NOTA: Il DNA viene continuamente replicato e amplificato a temperatura costante utilizzando una DNA polimerasi con funzionalità di spostamento del filamento. Il processo di reazione coinvolge la fase di sintesi del modello a forma di manubrio, la fase di amplificazione ciclica, l'allungamento e la fase di riciclaggio, formando infine una miscela di frammenti di DNA con strutture ad anello e a forma di cavolfiore. Per informazioni dettagliate, consultare le istruzioni del kit (File supplementare 1). - Aggiungi primer a due loop al sistema di reazione per migliorare l'efficienza della reazione, che si lega alle strutture stem-loop, avviando la sintesi dello spostamento del filamento e la replicazione ciclica. La composizione del kit di rilevamento dell'acido nucleico per i patogeni del tratto respiratorio è presentata nella Tabella 2.
NOTA: Il kit di reagenti utilizza il metodo LAMP. Il principio si basa sull'equilibrio dinamico del DNA a circa 65 °C, in cui qualsiasi primer che si estende attraverso l'appaiamento di basi nel sito complementare del DNA a doppio filamento provoca la dissociazione dell'altro filamento, formando un singolo filamento.
- Mira a sei regioni di sequenza con quattro primer specifici, inclusi due primer interni e due primer esterni (forniti con il kit LAMP).
- Chip microfluidico
NOTA: Ogni chip microfluidico (vedi Tabella dei materiali) è dotato di 24 pozzetti di reazione numerati in senso antiorario, con l'ingresso e l'uscita del pozzetto 1 corrispondenti al pozzetto di reazione 1 (Figura 2). Un set specifico di primer per l'amplificazione e la rilevazione di una particolare sequenza target di acido nucleico è incorporato e fissato in ogni pozzetto di reazione.- Miscelare il DNA del campione con il reagente di amplificazione. Iniettare la miscela nel chip e distribuirla ad ogni centrifuga del pozzetto di reazione a 1.000 × g per 30 s a temperatura ambiente.
NOTA: Le reazioni di amplificazione isotermica indipendenti e il rilevamento della fluorescenza in tempo reale si verificano in ogni pozzetto di reazione sul chip. Se viene rilevata una curva di amplificazione a forma di S in un particolare pozzetto di reazione, l'indice di rilevamento corrispondente per quel pozzetto è positivo.
- Miscelare il DNA del campione con il reagente di amplificazione. Iniettare la miscela nel chip e distribuirla ad ogni centrifuga del pozzetto di reazione a 1.000 × g per 30 s a temperatura ambiente.
| Componenti | Composizione | Numero |
| Scheggia | Primer | 12 posti |
| Pellicola sigillante | / | 1 foglio |
| Reagente di amplificazione isotermica | Colorante fluorescente, enzima | 270 μl/provetta |
| Controllo positivo | DNA genomico di Escherichia coli | 160 μl/provetta |
Tabella 2: Composizione del kit di rilevamento dell'acido nucleico per i patogeni del tratto respiratorio.
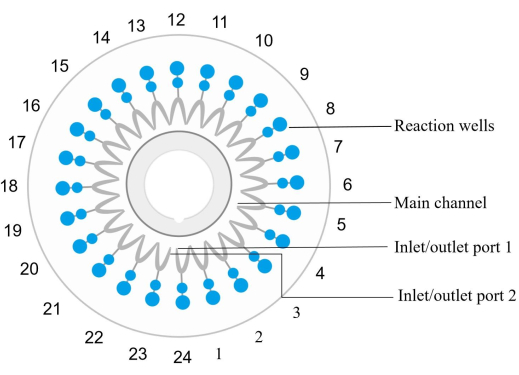
Figura 2: Diagramma della struttura del chip del disco. I pozzetti di reazione sono numerati in sequenza in senso antiorario, dove la porta di ingresso/uscita 1 corrisponde al pozzetto di reazione numero 1. I pozzetti di reazione 1, 4, 7, 10, 13, 16, 19, 22 e 24 sono controlli negativi. Il pozzetto di reazione 6 è un controllo positivo (E. coli). Il pozzetto di reazione 12 è un controllo positivo interno e il pozzetto di reazione 23 è un controllo positivo esterno. Il pozzetto di reazione 2 rileva spn. Reazione bene 3 rileva sau. Il pozzetto di reazione 5 rileva l'MRSA. Il pozzetto di reazione 8 rileva kpn. Il pozzetto di reazione 9 rileva il pae. Il pozzetto di reazione 11 rileva aba. Il pozzetto di reazione 14 rileva sma. Il pozzo di reazione 15 rileva hin. Si prega di consultare la Tabella 5 per i dettagli dell'esempio. Clicca qui per visualizzare una versione più grande di questa figura.
4. Preparazione del campione e rilevamento batterico
- Preparare il sistema di reazione di amplificazione isoterma
- Prendere il reagente di amplificazione isotermico dal kit e lasciarlo scongelare completamente a temperatura ambiente. Agitare delicatamente per mescolare accuratamente e centrifugare brevemente per raccogliere sul fondo della provetta.
- Nell'area di stoccaggio e preparazione dei reagenti, pipettare 20 μl del reagente di amplificazione isotermica in una provetta da centrifuga da 200 μl preparata. Coprire la provetta e spostarla nell'area di preparazione del campione con una provetta da centrifuga per campione.
- Nell'area di preparazione del campione, aggiungere 34,5 μl del campione di acido nucleico target. Agitare delicatamente per mescolare accuratamente e centrifugare brevemente per raccogliere sul fondo della provetta. Il volume totale di ciascun sistema di reazione di amplificazione degli acidi nucleici è di 54,5 μL (Tabella 3).
- Procedura di caricamento trucioli
- Sulla confezione del chip, etichettare il numero del campione. Aprire il chip microfluidico con l'etichetta della confezione rivolta verso l'alto.
- Posizionare il chip con le porte di ingresso e uscita rivolte verso l'alto. Utilizzando una pipetta, aspirare 50 μl del sistema di reazione di amplificazione preparato e aggiungerlo al canale principale del chip attraverso la porta di ingresso. Interrompere l'aggiunta quando il canale principale è pieno e rimuovere rapidamente il liquido in eccesso attorno alle porte di ingresso e uscita con un panno privo di lanugine.
- Usa una pinzetta per raccogliere una pellicola sigillante e coprirla sopra le porte di ingresso e uscita. Premere una punta della pipetta pulita sulla pellicola sigillante in una direzione. Garantire una tenuta ermetica.
- Reazione qPCR
- Dopo che la sorgente luminosa dell'analizzatore di acidi nucleici ha completato il preriscaldamento, fare clic sul pulsante Apri vassoio . Posizionare il chip con il lato anteriore rivolto verso l'alto sul vassoio, assicurandosi che il piccolo cilindro di posizionamento emerga dalla fessura centrale del chip per fissarlo. Allineare il dispositivo di posizionamento centrale nel vassoio con il grande foro centrale nel chip.
- Fare clic sul pulsante Chiudi vassoio per inserire il chip nell'analizzatore di acidi nucleici.
- Nell'area Informazioni campione dell'interfaccia di rilevamento, immettere le informazioni per il test del campione. Il numero di campione, il numero di chip e il tipo di campione sono obbligatori; altri sono facoltativi.
- Fare clic sul pulsante Avvia rilevamento nell'area operativa per avviare il rilevamento del campione. Lo strumento eseguirà test a campione secondo il programma preimpostato.
NOTA: Dopo il completamento del rilevamento, il software fornito in dotazione al dispositivo eseguirà automaticamente l'analisi dei dati. Contemporaneamente, l'analizzatore di acidi nucleici avvierà un processo di raffreddamento automatico. Una volta che la temperatura scende a 37 °C, lo strumento apre automaticamente il vassoio ed espelle il chip per il recupero. I risultati saranno interpretati automaticamente dal sistema (Tabella 4).
- Standard di controllo della qualità
- Un controllo di qualità positivo e nove controlli di qualità negativi sono racchiusi in ogni chip di questo kit di reagenti. Assicurarsi che i risultati del rilevamento per il controllo di qualità positivo siano positivi e, contemporaneamente, che i risultati per tutti e nove i controlli di qualità negativi siano negativi.
NOTA: I risultati vengono visualizzati sul lato destro dell'immagine con l'indicazione "Risultati del controllo di qualità: esperimento normale (normale positivo, normale negativo)", a indicare che i risultati del test sono validi. È necessario ripetere il test se un risultato non è corretto e il risultato del test del campione è ritenuto non valido. Nel chip è racchiuso un controllo interno positivo che utilizza primer specifici per l'uomo, il cui risultato è positivo per i test clinici su campione mentre è negativo se il campione ha un basso contenuto di DNA genomico umano, indicando una conta cellulare inferiore. In questi casi, raccogliere e ripetere l'analisi dei campioni.
- Un controllo di qualità positivo e nove controlli di qualità negativi sono racchiusi in ogni chip di questo kit di reagenti. Assicurarsi che i risultati del rilevamento per il controllo di qualità positivo siano positivi e, contemporaneamente, che i risultati per tutti e nove i controlli di qualità negativi siano negativi.
- Analisi dei risultati
- Dopo il completamento del rilevamento, utilizzare il metodo del massimo della derivata seconda combinato con algoritmi commercialmente protetti nel software per calcolare il primo punto di flesso della curva di amplificazione a forma di S durante la fase di amplificazione rapida. I dettagli sono descritti nella parte "Principi del test" delle Istruzioni del kit per il rilevamento dell'acido nucleico dei patogeni respiratori (File supplementare 1).
NOTA: Il valore TP è la differenza di tempo tra il punto di flesso e l'origine. Il risultato viene interpretato in base al TP e ai valori di giudizio positivo. Se il valore TP per un indice di rilevamento è inferiore o uguale al valore di giudizio positivo per tale indice, viene interpretato come positivo. Se il valore TP supera il valore di giudizio positivo, viene interpretato come negativo, secondo i criteri per il "valore di decisione positivo" nelle istruzioni. L'area "Curva di fluorescenza" visualizza la curva normalizzata e l'area "Risultato rilevamento" mostra il controllo di qualità e i risultati di rilevamento per ciascun parametro (Tabella 5).
- Dopo il completamento del rilevamento, utilizzare il metodo del massimo della derivata seconda combinato con algoritmi commercialmente protetti nel software per calcolare il primo punto di flesso della curva di amplificazione a forma di S durante la fase di amplificazione rapida. I dettagli sono descritti nella parte "Principi del test" delle Istruzioni del kit per il rilevamento dell'acido nucleico dei patogeni respiratori (File supplementare 1).
| Reagente | Volume (μl) |
| Reagente di amplificazione isotermica | 20 |
| DNA modello | 34.5 |
Tabella 3: Sistema di reazione di amplificazione isotermica.
| Passo | Uno | Due |
| Temperatura (°C) | 37 | 65 |
| Tempo (min) | 3 | 47 |
Tabella 4: Programma di reazione di amplificazione degli acidi nucleici.
| Nome indicatore | Valore di controllo positivo |
| Streptococcus pneumoniae (sp) | 30 |
| Staphylococcus aureus (sau) | 34 |
| Staphylococcus aureus meticillino-resistente (mrsa) | 22 |
| Klebsiella pneumoniae (kpn) | 29 |
| Pseudomonas aeruginosa (pae) | 36 |
| Acinetobacter baumannii (aba) | 36 |
| Stenotrophomonas maltofilia (sma) | 27 |
| Haemophilus influenzae (hin) | 36 |
Tabella 5: Valore di controllo positivo per l'indicatore di infezione.
Access restricted. Please log in or start a trial to view this content.
Risultati
Questo esperimento impiega la tecnologia di amplificazione isotermica, conducendo reazioni su un chip di disco microfluidico. La reazione avviene su un analizzatore di acidi nucleici a chip microfluidico, utilizzando un metodo di inserimento di colorante a fluorescenza. La reazione isoterma viene eseguita a una temperatura costante di 65 °C e l'analisi della fluorescenza in tempo reale viene eseguita simultaneamente. I campioni positivi subiscono un'amplificazione sotto l'azione della polimerasi con funzionalità di spo...
Access restricted. Please log in or start a trial to view this content.
Discussione
Le infezioni delle vie respiratorie sono infezioni ospedaliere prevalenti, che impongono gravi conseguenze ai pazienti e aumentano i tassi di mortalità16. L'identificazione tempestiva e accurata dei potenziali agenti patogeni, seguita da antibiotici efficaci, è la chiave per il successo del trattamento e per migliorare la prognosi, in particolare date le limitazioni insite nei metodi di coltura tradizionali17. In questo studio, abbiamo utilizzato un metodo basato su LAMP ...
Access restricted. Please log in or start a trial to view this content.
Divulgazioni
Gli autori dichiarano di non avere conflitti di interesse.
Riconoscimenti
Abbiamo molto apprezzato il sostegno finanziario fornito dalla Guangdong Basic and Applied Basic Research Foundation (sovvenzione n. 2022A1515220023) e dalla Research Foundation for Advanced Talents of Guandong Provincial People's Hospital (sovvenzione n. KY012023293).
Access restricted. Please log in or start a trial to view this content.
Materiali
| Name | Company | Catalog Number | Comments |
| Bath Incubator(MK2000-2) | ALLSHENG | Provide a constant temperature environment | |
| Bronchial lavage fluid collector head | TIANPINGHUACHANG | SEDA 20172081375 | Collecting bronchoalveolar lavage fluid |
| Fiberoptic bronchoscope | OLYMPUS | SEDA 20153062703 | A flexible bronchoscope equipped with a fiberoptic light source and camera, to visually examine the airways and structures within the lungs. Assist in collecting bronchoalveolar lavage |
HR1500- B2 B2 | Haier | SEDA 20183541642 | Biosafety cabinet |
| NAOH | MACKLIN | S817977 | Liquefy viscous lower respiratory tract sample |
| Nucleic acid detection kit for respiratory tract pathogens | Capitalbio Technology | SEDA 20173401346 | Testing for bacteria infection |
| Nucleic acid extraction reagent | Capitalbio Technology | SEDA 20160034 | For DNA extraction |
| RTisochip-W | Capitalbio Technology | SEDA 20193220539 | Loop-mediated Isothermal Amplification |
| THERMO ST16R | Thermo Fisher Scientific | SEDA 20180585 | Centrifuge the residual liquid off the wall of the tube. |
| Vortex mixer VM-5005 | JOANLAB | For mixing reagent |
Riferimenti
- GBD 2016 Lower Respiratory Infections Collaborators. Estimates of the global, regional, and national morbidity, mortality, and aetiologies of lower respiratory infections in 195 countries, 1990-2016: A systematic analysis for the global burden of disease study 2016. Lancet Infect Dis. 18 (11), 1191-1210 (2018).
- Niederman, M. S., Torres, A. Respiratory infections. Eur Respir Rev. 31 (166), 220150(2022).
- Weiser, J. N., Ferreira, D. M., Paton, J. C. Streptococcus pneumoniae: Transmission, colonization and invasion. Nat Rev Microbiol. 16 (6), 355-367 (2018).
- Watt, J. P., et al. Burden of disease caused by Haemophilus influenzae type b in children younger than 5 years_ global estimates. Lancet. 374 (9693), 903-911 (2009).
- Kadioglu, A., Weiser, J. N., Paton, J. C., Andrew, P. W. The role of streptococcus pneumoniae virulence factors in host respiratory colonization and disease. Nat Rev Microbiol. 6 (4), 288-301 (2008).
- Popova, G., Boskovska, K., Arnaudova-Danevska, I., Smilevska-Spasova, O., Jakovska, T. Sputum quality assessment regarding sputum culture for diagnosing lower respiratory tract infections in children. Open Access Maced J Med Sci. 7 (12), 1926-1930 (2019).
- Nuyttens, H., Cyncynatus, C., Renaudin, H., Pereyre, S., Identification Bébéar, C. expression and serological evaluation of the recombinant ATP synthase beta subunit of mycoplasma pneumoniae. BMC Microbiol. 10 (1), 216(2010).
- Noviello, S., Huang, D. B. The basics and the advancements in diagnosis of bacterial lower respiratory tract infections. Diagnostics (Basel). 9 (2), 37(2019).
- Hanson, K. E., et al. Molecular testing for acute respiratory tract infections: Clinical and diagnostic recommendations from the IDSA's diagnostics committee. Clin Infect Dis. 71 (10), 2744-2751 (2020).
- Daniel Reynolds, J. P. B., et al. The threat of multidrug-resistant/extensively drug-resistant gram-negative respiratory infections: Another pandemic. Eur Respir Rev. 31 (166), 220068(2022).
- T Notomi, H. O., et al. Loop-mediated isothermal amplification of DNA. Nucleic Acids Res. 28 (12), 63(2000).
- Soroka, M., Wasowicz, B., Rymaszewska, A. Loop-mediated isothermal amplification (LAMP): The better sibling of PCR. Cells. 10 (8), 1931(2021).
- Mori, Y., Kitao, M., Tomita, N., Notomi, T. Real-time turbidimetry of lamp reaction for quantifying template DNA. Biophys J. 59 (2), 145-157 (2004).
- Parida, M., Sannarangaiah, S., Dash, P. K., Rao, P. V., Morita, K. Loop mediated isothermal amplification (LAMP): A new generation of innovative gene amplification technique: Perspectives in clinical diagnosis of infectious diseases. Rev Med Virol. 18 (6), 407-421 (2008).
- Chinese Medical Association Respiratory Branch Critical Care Medicine Group, Working Committee on Critical Care Medicine of the Chinese Physicians Association Respiratory Physicians Branch. Standardization of collection, submission, testing, and interpretation of bronchoalveolar lavage fluid in ICU patients. Chin J Tubere Respir Dis. 43 (9), 744-756 (2020).
- Koch, A. M., Nilsen, R. M., Eriksen, H. M., Cox, R. J., Harthug, S. Mortality related to hospital-associated infections in a tertiary hospital: Repeated cross-sectional studies between 2004-2011. Antimicrob Resist Infect Control. 4 (1), 57(2015).
- Seymour, C. W. F. G., et al. Time to treatment and mortality during mandated emergency care for sepsis. N Engl J Med. 376 (23), 2235-2244 (2017).
- Choi, C. W., Hyun, J. W., Hwang, R. Y., Powell, C. A. Loop-mediated isothermal amplification assay for detection of candidatus Liberibacter asiaticus: A causal agent of citrus huanglongbing. Plant Pathol. 34 (6), 499-505 (2018).
- Büscher, P., Njiru, Z. K. Loop-mediated isothermal amplification technology: Towards point of care diagnostics. PLoS Negl Trop Dis. 6 (6), e1572(2012).
- Notomi, T., Mori, Y., Tomita, N., Kanda, H. Loop-mediated isothermal amplification (LAMP): Principle, features, and future prospects. J Microbiol. 53 (1), 1-5 (2015).
- Ajibola, O., Gulumbe, B., Eze, A., Obishakin, E. Tools for detection of schistosomiasis in resource limited settings. Medical Sciences. 6 (2), 39(2018).
- Kumar, Y. S. B., et al. Loop-mediated isothermal amplification (LAMP): A rapid and sensitive tool for quality assessment of meat products. Compr Rev Food Sci Food SAF. 16 (6), 1359-1378 (2017).
- Hongling Ou, Y. W., et al. Rapid detection of multiple pathogens by the combined loop-mediated isothermal amplification technology and microfluidic chip technology. Ann Palliat Med. 10 (10), 11053-11066 (2021).
- Liang Wang, J. -X. L., et al. Quantitative polymerase chain reaction (qPCR)-based rapid diagnosis of helicobacter pylori infection and antibiotic resistance. J Vis Exp. (197), e65689(2023).
- Jing-Wen Lyu, B. X., et al. Rapid prediction of multidrug-resistant klebsiella pneumoniae through deep learning analysis of SERS spectra. Microbiol Spectr. 11 (2), e0412622(2023).
- Wei Liu, J. -W. T., et al. Discrimination between carbapenem-resistant and carbapenem-sensitive klebsiella pneumoniae strains through computational analysis of surface-enhanced Raman spectra a pilot study. Microbiol Spectr. 10 (1), e0240921(2022).
- Zhang, L. Y., et al. Classification and prediction of klebsiella pneumoniae strains with different MLST allelic profiles via SERS spectral analysis. Peer J. 11, e16161(2023).
- Zhi Liu, Q. Z., et al. Rapid and sensitive detection of salmonella in chickens using loop-mediated isothermal amplification combined with a lateral flow dipstick. J Microbiol Biotechnol. 29 (3), 454-464 (2019).
- Chen, F., et al. Fully Integrated microfluidic platform for multiplexed detection of hunov by a dynamic confined-space-implemented one-pot RPA-LAMP system. Adv Sci (Weinh). 21, e2306612(2023).
Access restricted. Please log in or start a trial to view this content.
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon