É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Detecção rápida de patógenos bacterianos que causam infecções do trato respiratório inferior por meio de amplificação isotérmica mediada por loop baseada em chip microfluídico
* Estes autores contribuíram igualmente
Neste Artigo
Resumo
Vários patógenos bacterianos podem causar infecções do trato respiratório e levar a sérios problemas de saúde se não forem detectados com precisão e tratados prontamente. A detecção rápida e precisa desses patógenos por meio de amplificação isotérmica mediada por loop fornece gerenciamento e controle eficazes de infecções do trato respiratório em ambientes clínicos.
Resumo
As infecções do trato respiratório (ITRs) estão entre os problemas mais comuns em ambientes clínicos. A identificação rápida e precisa de patógenos bacterianos fornecerá diretrizes práticas para o gerenciamento e tratamento de ITRs. Este estudo descreve um método para detectar rapidamente patógenos bacterianos que causam infecções do trato respiratório por meio de amplificação isotérmica mediada por loop multicanal (LAMP). O LAMP é uma ferramenta de diagnóstico sensível e específica que detecta rapidamente ácidos nucléicos bacterianos com alta precisão e confiabilidade. O método proposto oferece uma vantagem significativa sobre os métodos tradicionais de cultura bacteriana, que são demorados e muitas vezes requerem maior sensibilidade para detectar baixos níveis de ácidos nucléicos bacterianos. Este artigo apresenta resultados representativos da infecção por K. pneumoniae e suas múltiplas coinfecções usando LAMP para detectar amostras (escarro, lavado brônquico e lavado alveolar) do trato respiratório inferior. Em resumo, o método LAMP multicanal fornece um meio rápido e eficiente de identificar patógenos bacterianos únicos e múltiplos em amostras clínicas, o que pode ajudar a prevenir a disseminação de patógenos bacterianos e auxiliar no tratamento adequado de ITRs.
Introdução
As infecções do trato respiratório (ITRs) causadas por patógenos bacterianos contribuem principalmente para a morbidade e mortalidade em todo o mundo1. É definida como qualquer sintoma respiratório superior ou inferior acompanhado de febre com duração de 2-3 dias. Embora a infecção respiratória superior seja mais comum do que a infecção respiratória inferior, as infecções crônicas e recorrentes do trato respiratório também são condições clínicas comuns, apresentando grandes riscos aos indivíduos e sobrecarregando significativamente os sistemas de saúde2. Os patógenos bacterianos comuns de ITRs incluem Streptococcus pneumoniae3, Haemophilus influenzae4, Staphylococcus aureus, Escherichia coli, Klebsiella pneumoniae, Stenotrophomonas maltophilia, entre outros. Essas bactérias patogênicas geralmente colonizam as superfícies mucosas da nasofaringe e do trato respiratório superior do hospedeiro, causando sintomas típicos de ITRs, como dor de garganta e bronquite. Causam pneumonia quando se espalham do trato respiratório superior para áreas estéreis do trato respiratório inferior e podem se espalhar de pessoa para pessoa através do trato respiratório5. Em casos graves, eles também podem levar a doenças bacterianas invasivas, especialmente pneumonia bacterêmica, meningite e sepse, que são as principais causas de morbidade e mortalidade em pessoas de todas as faixas etárias em todo o mundo.
Os testes tradicionais para ITRs envolvem cultura microbiológica usando swabs de garganta e amostras respiratórias de escarro6. Além disso, testes sorológicos, como ensaio imunoenzimático (ELISA), detectam anticorpos ou antígenos no soro, enquanto os testes de aglutinação observam a reação de aglutinação de anticorpos e antígenos para detectar infecção7. A cultura microbiana é considerada o padrão-ouro para o diagnóstico de ITRs, mas sua baixa taxa de positividade da cultura, baixa confiabilidade e longo ciclo de detecção limitam a eficiência diagnóstica8. Na realidade, o diagnóstico rápido e preciso de ITRs é crucial para a erradicação precisa do patógeno bacteriano. Métodos de detecção rápidos e eficazes podem ajudar a reduzir a taxa de transmissão de patógenos, encurtar a duração da infecção e diminuir o uso desnecessário de antibióticos 9,10. Métodos baseados em biologia molecular aceleram significativamente a detecção, como a reação em cadeia da polimerase (PCR), que amplifica a sequência de DNA de um gene-alvo para detectar patógenos. No entanto, a PCR tradicional requer equipamentos complexos de ciclagem de temperatura, que são pesados e demorados. Além disso, toda amplificação de DNA usando PCR (exceto PCR em tempo real) termina com a separação eletroforética do produto, o que também leva tempo. A visualização do produto requer corantes, muitos dos quais são mutagênicos ou cancerígenos. Portanto, é imperativo desenvolver continuamente novos métodos e tecnologias para diagnosticar patógenos bacterianos RTI.
A Amplificação Isotérmica Mediada por Loop (LAMP) é uma tecnologia molecular nova e emergente inicialmente desenvolvida por Notomi et al. em 200011. O LAMP pode amplificar o DNA sob condições isotérmicas estáveis sem equipamentos complexos de ciclagem de temperatura, o que o torna adequado para detecção rápida e reduz a complexidade e o custo do equipamento12. O LAMP pode detectar baixas concentrações de DNA alvo com alta sensibilidade13. Ele usa vários primers específicos para melhorar a seletividade das sequências-alvo e reduzir a possibilidade de falsos positivos14. O LAMP está sendo gradualmente amplamente utilizado em laboratórios clínicos devido à sua facilidade, velocidade e operação intuitiva, mesmo para detectar RTIs. Neste estudo, investigamos a eficácia do LAMP na detecção de ATRs inferiores em amostras clínicas (escarro, lavado brônquico e lavado alveolar), conforme mostrado na Figura 1. É evidente que o LAMP oferece vantagens como velocidade, sensibilidade e facilidade de uso em relação aos testes tradicionais na detecção de RTI mais baixa, tornando-o uma aplicação promissora.
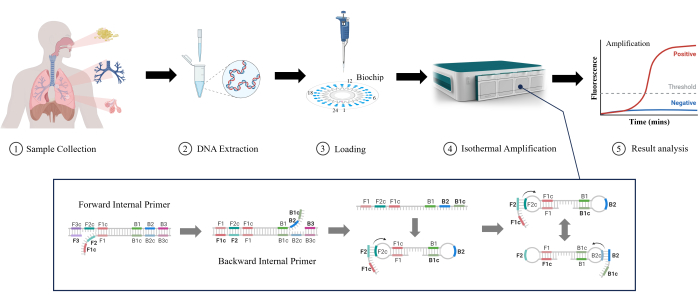
Figura 1: Ilustração esquemática do método de detecção LAMP. Clique aqui para ver uma versão maior desta figura.
Access restricted. Please log in or start a trial to view this content.
Protocolo
Todas as amostras para este estudo foram avaliadas e aprovadas pelo Comitê de Revisão de Ética do Hospital Popular da Província de Guangdong (Número de aprovação: KY2023-1114-01). Todos os participantes assinaram o termo de consentimento livre e esclarecido antes dos experimentos. Os reagentes e equipamentos utilizados para o estudo estão listados na Tabela de Materiais. As abreviaturas usadas no protocolo estão listadas na Tabela Suplementar 1.
1. Coleta de amostras clínicas do trato respiratório inferior
- Coleta de escarro
- Limpe a cavidade oral e os dentes com água limpa, garantindo a remoção da prótese, se aplicável. Tossir com força para expelir o escarro respiratório profundo para um recipiente de escarro (mínimo 0,6 mL).
- Evite a contaminação com saliva ou secreção nasal. Se a tosse for difícil, administre a inalação de uma solução de NaCl a 100 g/L a 45 °C para facilitar a expectoração. Transfira a amostra para um recipiente estéril e lacrável.
NOTA: Certifique-se de que as amostras de escarro sejam coletadas pela manhã e antes do uso de qualquer medicamento antibacteriano.
- Fluido de lavagem brônquica (BLF)
- Insira a cabeça coletora na traqueia a partir da narina ou do local da traqueostomia (aproximadamente 30 cm de profundidade). Injete 5 mL de solução salina, estabeleça pressão negativa, gire a cabeça do coletor e retire lentamente.
- Recolha o muco extraído e lave o colector uma vez com uma solução de amostragem (soro fisiológico ou água estéril para injecção). Como alternativa, conecte um cateter urinário pediátrico a uma seringa de 50 mL como substituto da coleta.
- Fluido de lavagem alveolar (IHA)
- Identificar a localização da lesão por meio de exames de imagem do tórax. Selecione a área mais significativa ou de progresso rápido para lavagem.
- Injetar 1-2 mL de lidocaína a 2% no segmento brônquico através do canal de biópsia durante a lavagem, fornecendo anestesia local para o segmento pulmonar lavado15. Para pacientes sob anestesia geral intravenosa que ainda apresentam fortes reações nas vias aéreas, injete 1-2 mL de lidocaína a 2%.
- Após a anestesia local, insira um broncoscópio de fibra óptica pela boca ou nariz, passando pela faringe para o brônquio do lobo médio direito ou para o segmento lingual do pulmão esquerdo. Coloque sua ponta na abertura do ramo brônquico e introduza lentamente solução salina fisiológica estéril.
- Administre 30-50 mL de solução salina por sessão, com volume de 100-250 mL (máx. 300 mL). Colete o muco extraído em um recipiente estéril lacrado.
2. Extração de DNA
- Com base na viscosidade das amostras do trato respiratório inferior, adicione uma quantidade apropriada de 10% de NaOH. Para amostras com baixa viscosidade, adicione 2-3 vezes o volume da amostra de solução liquefeita. Para amostras com viscosidade moderada, adicione 5-6 vezes o volume da amostra de solução liquefeita. Para amostras com alta viscosidade, adicione 8 a 10 vezes o volume da amostra de solução liquefeita.
- Ajuste o volume de NaOH a 10% de acordo com a viscosidade da amostra. Dispersar as amostras o mais uniformemente possível utilizando um misturador de vórtices durante 15 s e incubar a 37 °C durante 30 min para liquefação.
NOTA: Para amostras mais espessas, aumente o volume de NaOH ou estenda o tempo de liquefação. A qualidade da liquefação das amostras influencia diretamente a eficiência de extração subsequente. Idealmente, as amostras liquefeitas devem exibir uma consistência consistente e não pegajosa.
- Ajuste o volume de NaOH a 10% de acordo com a viscosidade da amostra. Dispersar as amostras o mais uniformemente possível utilizando um misturador de vórtices durante 15 s e incubar a 37 °C durante 30 min para liquefação.
- Pipete 1 mL da amostra liquefeita em um tubo de centrífuga de 1,5 mL. Centrifugar a uma velocidade de 15,777 × g durante 5 min a 2-6 °C e, em seguida, utilizar uma pipeta para remover e eliminar o sobrenadante.
NOTA: Ao aspirar a amostra, evite retirar impurezas do fundo do tubo; Se houver muitas impurezas, a amostra pode ser centrifugada a 1.753-2.739 × g por 1 min antes da aspiração. - Adicione 1 mL de solução de lavagem ao tubo da centrífuga e ao vórtice para levantar o precipitado do fundo do tubo. Não há necessidade de dispersá-lo completamente.
- Centrifugar a solução a 15,777 × g durante 5 min, rejeitar o sobrenadante e tentar não tocar no precipitado.
NOTA: Certifique-se de remover completamente a solução de lavagem para evitar afetar a amplificação subsequente.
- Centrifugar a solução a 15,777 × g durante 5 min, rejeitar o sobrenadante e tentar não tocar no precipitado.
- Adicione 100 μL de solução de extração de ácido nucleico ao tubo da centrífuga. Use uma pipeta para aspirar e misture bem o precipitado. Transfira o líquido e precipite juntos para um tubo de extração de ácido nucleico. A composição da solução de extracção de ácidos nucleicos é apresentada no quadro 1.
- Coloque os tubos de extração de ácido nucleico em um misturador de vórtice e vórtice em velocidade média por pelo menos 5 min. Após o vórtice, transferir os tubos de extracção de ácidos nucleicos para um banho metálico com temperatura constante e aquecer a 100 °C durante 5 min.
- Centrifugar a 15,777 × g a 2-6 °C durante 5 min e retirar a página.
NOTA: Se a reação de amplificação por PCR for realizada dentro de 24 h, o ácido nucleico pode ser armazenado a 4 °C. Após a amplificação, armazenar o ácido nucleico a -20 °C. Para armazenamento de longo prazo (além de 24 h), armazene o ácido nucleico a -20 °C. Quando estiver pronto para usar novamente, descongele a amostra naturalmente, vortex para misturar, aqueça em banho-maria a 95 °C por 5 min, centrifugue a 10.956 × g por 1 min e use o sobrenadante para amplificação por PCR.
| Solução | Componentes | Número | Especificação |
| Solução de lavagem | EDTA de 10 mM | 1 garrafa | 24 mL/frasco |
| Reagente de extração de ácido nucleico | 10mM Tris-HCl, 1mM EDTA, conservantes de ácido nucleico | 2 tubos | 1,2 mL/frasco |
| Tubo de extração de ácido nucleico | Contas de vidro | 1 saco | 24 garrafas/saco |
Tabela 1: Composição do reagente de extração de ácido nucleico.
3. Amplificação isotérmica mediada por loop e chip de microfluido
- Reagente e chip microfluídico
- Conduza reações de amplificação isotérmica em um chip microfluídico em forma de disco (consulte a Tabela de Materiais). Efectuar a amplificação a temperatura constante a 65 °C.
- Realize a análise de fluorescência em tempo real usando um método de incorporação de corante fluorescente14 no analisador de ácido nucleico de chip microfluídico de amplificação de temperatura constante. Observe uma curva de amplificação em forma de S para amostras positivas usando uma polimerase com funcionalidade de deslocamento de fita.
- Amplificação isotérmica mediada por loop
- Segmente seis regiões de sequência com quatro primers específicos, incluindo dois primers internos e dois primers externos (fornecidos com o kit LAMP).
NOTA: O DNA é continuamente replicado e amplificado a uma temperatura constante usando uma DNA polimerase com funcionalidade de deslocamento de fita. O processo de reação envolve o estágio de síntese do modelo em forma de haltere, estágio de amplificação cíclica, alongamento e estágio de reciclagem, formando uma mistura de fragmentos de DNA com estruturas semelhantes a alças e couve-flor. Consulte as instruções do kit para obter informações detalhadas (Arquivo Suplementar 1). - Adicione primers de duas alças ao sistema de reação para aumentar a eficiência da reação, que se liga às estruturas da alça da haste, iniciando a síntese do deslocamento da fita e a replicação cíclica. A composição do kit de detecção de ácidos nucleicos para patógenos do trato respiratório é apresentada na Tabela 2.
NOTA: O kit de reagentes utiliza o método LAMP. O princípio é baseado no equilíbrio dinâmico do DNA em torno de 65 ° C, onde qualquer primer que se estenda através do emparelhamento de bases no local complementar do DNA de fita dupla faz com que a outra fita se dissocie, formando uma fita simples.
- Segmente seis regiões de sequência com quatro primers específicos, incluindo dois primers internos e dois primers externos (fornecidos com o kit LAMP).
- Chip microfluídico
NOTA: Cada chip microfluídico (consulte a Tabela de Materiais) é equipado com 24 poços de reação numerados no sentido anti-horário, com a entrada e saída do poço 1 correspondendo ao poço de reação 1 (Figura 2). Um conjunto específico de primers para amplificação e detecção de uma determinada sequência alvo de ácido nucleico é incorporado e fixado em cada poço de reação.- Misturar o ADN da amostra com o reagente de amplificação. Injete a mistura no chip e distribua-a para cada centrífuga de poço de reação a 1.000 × g por 30 s em temperatura ambiente.
NOTA: Reações independentes de amplificação isotérmica e detecção de fluorescência em tempo real co-ocorrem em cada poço de reação no chip. Se uma curva de amplificação em forma de S for detectada em um determinado poço de reação, o índice de detecção correspondente para esse poço é positivo.
- Misturar o ADN da amostra com o reagente de amplificação. Injete a mistura no chip e distribua-a para cada centrífuga de poço de reação a 1.000 × g por 30 s em temperatura ambiente.
| Componentes | Composição | Número |
| Cavaco | Primers | 12 alojamentos |
| Filme de vedação | / | 1 folha |
| Reagente de amplificação isotérmica | Corante fluorescente, enzima | 270 μL/tubo |
| Controle Positivo | DNA genômico de Escherichia coli | 160 μL/tubo |
Tabela 2: Composição do kit de detecção de ácido nucleico para patógenos do trato respiratório.
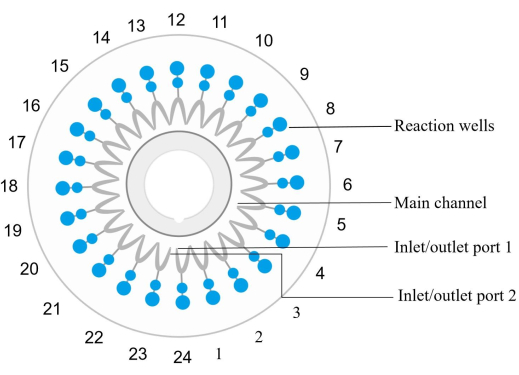
Figura 2: Diagrama da estrutura do chip do disco. Os poços de reação são numerados sequencialmente no sentido anti-horário, onde a porta de entrada/saída 1 corresponde ao poço de reação número 1. Os alvéolos de reação 1, 4, 7, 10, 13, 16, 19, 22 e 24 são controles negativos. O poço de reação 6 é um controle positivo (E. coli). O poço de reação 12 é um controle positivo interno e o poço de reação 23 é um controle positivo externo. O poço de reação 2 detecta spn. O poço de reação 3 detecta sau. O poço de reação 5 detecta MRSA. O poço de reação 8 detecta kpn. O poço de reação 9 detecta pae. O poço de reação 11 detecta aba. O poço de reação 14 detecta sma. A reação bem 15 detecta hin. Consulte a Tabela 5 para obter detalhes da amostra. Clique aqui para ver uma versão maior desta figura.
4. Preparação de amostras e detecção bacteriana
- Preparar o sistema de reação de amplificação isotérmica
- Retire o reagente de amplificação isotérmica do kit e deixe-o descongelar totalmente à temperatura ambiente. Agite suavemente para misturar bem e centrifugue brevemente para coletar no fundo do tubo.
- Na área de armazenamento e preparação do reagente, pipetar 20 μL do reagente de amplificação isotérmica para um tubo de centrífuga preparado de 200 μL. Cubra o tubo e mova-o para a área de preparação da amostra com um tubo de centrífuga por amostra.
- Na área de preparação da amostra, adicione 34,5 μL da amostra de ácido nucleico alvo. Agite suavemente para misturar bem e centrifugue brevemente para coletar no fundo do tubo. O volume total de cada sistema de reação de amplificação de ácido nucleico é de 54,5 μL (Tabela 3).
- Procedimento de carregamento de cavacos
- Na embalagem do chip, rotule o número da amostra. Abra o chip microfluídico com a etiqueta da embalagem voltada para cima.
- Coloque o chip com as portas de entrada e saída voltadas para cima. Usando uma pipeta, retire 50 μL do sistema de reação de amplificação preparado e adicione-o ao canal principal do chip através da porta de entrada. Pare de adicionar quando o canal principal estiver cheio e limpe rapidamente qualquer excesso de líquido ao redor das portas de entrada e saída com um lenço de papel sem fiapos.
- Use uma pinça para pegar um filme de vedação e cobri-lo sobre as portas de entrada e saída. Pressione uma ponta de pipeta limpa no filme de vedação em uma direção. Garanta uma vedação hermética.
- reação de qPCR
- Depois que a fonte de luz do analisador de ácido nucleico concluir o pré-aquecimento, clique no botão Abrir bandeja . Coloque o chip com a frente voltada para cima na bandeja, certificando-se de que o pequeno cilindro de localização saia da abertura central do chip para prendê-lo. Alinhe o dispositivo de posicionamento central na bandeja com o grande orifício central no chip.
- Clique no botão Fechar bandeja para inserir o chip no analisador de ácido nucleico.
- Na área Informações da amostra na interface de detecção, insira as informações para testar a amostra. O número da amostra, o número do chip e o tipo de amostra são obrigatórios; outros são opcionais.
- Clique no botão Iniciar detecção na área de operação para iniciar a detecção da amostra. O instrumento realizará testes de amostra de acordo com o programa predefinido.
NOTA: Após a conclusão da detecção, o software que acompanha o dispositivo conduzirá automaticamente a análise dos dados. Simultaneamente, o analisador de ácido nucleico iniciará um processo de resfriamento automático. Quando a temperatura cai para 37 °C, o instrumento abre automaticamente a bandeja e ejeta o chip para recuperação. Os resultados serão interpretados automaticamente pelo sistema (Tabela 4).
- Padrões de controle de qualidade
- Um controle de qualidade positivo e nove controles de qualidade negativos estão incluídos em cada chip neste kit de reagentes. Certifique-se de que os resultados da detecção para o controle de qualidade positivo sejam positivos e, simultaneamente, os resultados para todos os nove controles de qualidade negativos sejam negativos.
NOTA: Os resultados são exibidos no lado direito da imagem informando "Resultados do controle de qualidade: Experimento normal (normal positivo, normal negativo)", indicando que os resultados do teste são válidos. O novo teste é necessário se algum resultado estiver incorreto e o resultado do teste de amostra for considerado inválido. Um controle interno positivo usando primers específicos para humanos é colocado no chip, cujo resultado é positivo para testes clínicos de amostras e é negativo se a amostra tiver um baixo conteúdo de DNA genômico humano, indicando uma contagem de células mais baixa. Recolha e teste novamente as amostras nesses casos.
- Um controle de qualidade positivo e nove controles de qualidade negativos estão incluídos em cada chip neste kit de reagentes. Certifique-se de que os resultados da detecção para o controle de qualidade positivo sejam positivos e, simultaneamente, os resultados para todos os nove controles de qualidade negativos sejam negativos.
- Análise de resultados
- Após a conclusão da detecção, utilize o método máximo da segunda derivada combinado com algoritmos protegidos comercialmente no software para calcular o primeiro ponto de inflexão da curva de amplificação em forma de S durante a fase de amplificação rápida. Os detalhes são descritos na parte "Princípios do Teste" das Instruções do Kit de Detecção de Ácido Nucleico de Patógenos Respiratórios (Arquivo Suplementar 1).
NOTA: O valor TP é a diferença de tempo entre o ponto de inflexão e a origem. O resultado é interpretado com base nos valores de TP e julgamento positivo. Se o valor de TP para um índice de detecção for menor ou igual ao valor de julgamento positivo para esse índice, ele será interpretado como positivo. Se o valor do TP exceder o valor de julgamento positivo, ele é interpretado como negativo, de acordo com os critérios de "valor de decisão positivo" nas instruções. A "Área da curva de fluorescência" exibe a curva normalizada e a área "Resultado da detecção" mostra o controle de qualidade e os resultados da detecção para cada parâmetro (Tabela 5).
- Após a conclusão da detecção, utilize o método máximo da segunda derivada combinado com algoritmos protegidos comercialmente no software para calcular o primeiro ponto de inflexão da curva de amplificação em forma de S durante a fase de amplificação rápida. Os detalhes são descritos na parte "Princípios do Teste" das Instruções do Kit de Detecção de Ácido Nucleico de Patógenos Respiratórios (Arquivo Suplementar 1).
| Reagente | Volume (μl) |
| Reagente de amplificação isotérmica | 20 |
| DNA do modelo | 34.5 |
Tabela 3: Sistema de reação de amplificação isotérmica.
| Passo | Um | Dois |
| Temperatura (°C) | 37 | 65 |
| Tempo (min) | 3 | 47 |
Tabela 4: Programa de reação de amplificação de ácido nucleico.
| Nome do indicador | Valor de controle positivo |
| Streptococcus pneumoniae (sp) | 30 |
| Staphylococcus aureus (sau) | 34 |
| Staphylococcus aureus resistente à meticilina (mrsa) | 22 |
| Klebsiella pneumoniae (kpn) | 29 |
| Pseudomonas aeruginosa (pae) | 36 |
| Acinetobacter baumannii (aba) | 36 |
| Stenotrophomonas maltophilia (sma) | 27 |
| Haemophilus influenzae (hin) | 36 |
Tabela 5: Valor de controle positivo para indicador de infecção.
Access restricted. Please log in or start a trial to view this content.
Resultados
Este experimento emprega tecnologia de amplificação isotérmica, conduzindo reações em um chip de disco microfluídico. A reação ocorre em um analisador de ácido nucleico de chip microfluídico, empregando um método de inserção de corante de fluorescência. A reação isotérmica é realizada a uma temperatura constante de 65 °C e a análise de fluorescência em tempo real é realizada simultaneamente. As amostras positivas sofrem amplificação sob a ação da polimerase com funcionalidade de deslocamento da ...
Access restricted. Please log in or start a trial to view this content.
Discussão
As infecções do trato respiratório são infecções hospitalares prevalentes, impondo graves consequências aos pacientes e aumentando as taxas de mortalidade16. A identificação oportuna e precisa de patógenos potenciais seguidos de antibióticos eficazes é a chave para o sucesso do tratamento e a melhoria do prognóstico, principalmente devido às limitações inerentes aos métodos tradicionaisde cultura 17. Neste estudo, usamos um método baseado em LAMP para dete...
Access restricted. Please log in or start a trial to view this content.
Divulgações
Os autores declaram não haver conflito de interesses.
Agradecimentos
Agradecemos muito o apoio financeiro fornecido pela Fundação de Pesquisa Básica e Aplicada de Guangdong (Concessão nº 2022A1515220023) e pela Fundação de Pesquisa para Talentos Avançados do Hospital Popular da Província de Guandong (Concessão nº. KY012023293).
Access restricted. Please log in or start a trial to view this content.
Materiais
| Name | Company | Catalog Number | Comments |
| Bath Incubator(MK2000-2) | ALLSHENG | Provide a constant temperature environment | |
| Bronchial lavage fluid collector head | TIANPINGHUACHANG | SEDA 20172081375 | Collecting bronchoalveolar lavage fluid |
| Fiberoptic bronchoscope | OLYMPUS | SEDA 20153062703 | A flexible bronchoscope equipped with a fiberoptic light source and camera, to visually examine the airways and structures within the lungs. Assist in collecting bronchoalveolar lavage |
HR1500- B2 B2 | Haier | SEDA 20183541642 | Biosafety cabinet |
| NAOH | MACKLIN | S817977 | Liquefy viscous lower respiratory tract sample |
| Nucleic acid detection kit for respiratory tract pathogens | Capitalbio Technology | SEDA 20173401346 | Testing for bacteria infection |
| Nucleic acid extraction reagent | Capitalbio Technology | SEDA 20160034 | For DNA extraction |
| RTisochip-W | Capitalbio Technology | SEDA 20193220539 | Loop-mediated Isothermal Amplification |
| THERMO ST16R | Thermo Fisher Scientific | SEDA 20180585 | Centrifuge the residual liquid off the wall of the tube. |
| Vortex mixer VM-5005 | JOANLAB | For mixing reagent |
Referências
- GBD 2016 Lower Respiratory Infections Collaborators. Estimates of the global, regional, and national morbidity, mortality, and aetiologies of lower respiratory infections in 195 countries, 1990-2016: A systematic analysis for the global burden of disease study 2016. Lancet Infect Dis. 18 (11), 1191-1210 (2018).
- Niederman, M. S., Torres, A. Respiratory infections. Eur Respir Rev. 31 (166), 220150(2022).
- Weiser, J. N., Ferreira, D. M., Paton, J. C. Streptococcus pneumoniae: Transmission, colonization and invasion. Nat Rev Microbiol. 16 (6), 355-367 (2018).
- Watt, J. P., et al. Burden of disease caused by Haemophilus influenzae type b in children younger than 5 years_ global estimates. Lancet. 374 (9693), 903-911 (2009).
- Kadioglu, A., Weiser, J. N., Paton, J. C., Andrew, P. W. The role of streptococcus pneumoniae virulence factors in host respiratory colonization and disease. Nat Rev Microbiol. 6 (4), 288-301 (2008).
- Popova, G., Boskovska, K., Arnaudova-Danevska, I., Smilevska-Spasova, O., Jakovska, T. Sputum quality assessment regarding sputum culture for diagnosing lower respiratory tract infections in children. Open Access Maced J Med Sci. 7 (12), 1926-1930 (2019).
- Nuyttens, H., Cyncynatus, C., Renaudin, H., Pereyre, S., Identification Bébéar, C. expression and serological evaluation of the recombinant ATP synthase beta subunit of mycoplasma pneumoniae. BMC Microbiol. 10 (1), 216(2010).
- Noviello, S., Huang, D. B. The basics and the advancements in diagnosis of bacterial lower respiratory tract infections. Diagnostics (Basel). 9 (2), 37(2019).
- Hanson, K. E., et al. Molecular testing for acute respiratory tract infections: Clinical and diagnostic recommendations from the IDSA's diagnostics committee. Clin Infect Dis. 71 (10), 2744-2751 (2020).
- Daniel Reynolds, J. P. B., et al. The threat of multidrug-resistant/extensively drug-resistant gram-negative respiratory infections: Another pandemic. Eur Respir Rev. 31 (166), 220068(2022).
- T Notomi, H. O., et al. Loop-mediated isothermal amplification of DNA. Nucleic Acids Res. 28 (12), 63(2000).
- Soroka, M., Wasowicz, B., Rymaszewska, A. Loop-mediated isothermal amplification (LAMP): The better sibling of PCR. Cells. 10 (8), 1931(2021).
- Mori, Y., Kitao, M., Tomita, N., Notomi, T. Real-time turbidimetry of lamp reaction for quantifying template DNA. Biophys J. 59 (2), 145-157 (2004).
- Parida, M., Sannarangaiah, S., Dash, P. K., Rao, P. V., Morita, K. Loop mediated isothermal amplification (LAMP): A new generation of innovative gene amplification technique: Perspectives in clinical diagnosis of infectious diseases. Rev Med Virol. 18 (6), 407-421 (2008).
- Chinese Medical Association Respiratory Branch Critical Care Medicine Group, Working Committee on Critical Care Medicine of the Chinese Physicians Association Respiratory Physicians Branch. Standardization of collection, submission, testing, and interpretation of bronchoalveolar lavage fluid in ICU patients. Chin J Tubere Respir Dis. 43 (9), 744-756 (2020).
- Koch, A. M., Nilsen, R. M., Eriksen, H. M., Cox, R. J., Harthug, S. Mortality related to hospital-associated infections in a tertiary hospital: Repeated cross-sectional studies between 2004-2011. Antimicrob Resist Infect Control. 4 (1), 57(2015).
- Seymour, C. W. F. G., et al. Time to treatment and mortality during mandated emergency care for sepsis. N Engl J Med. 376 (23), 2235-2244 (2017).
- Choi, C. W., Hyun, J. W., Hwang, R. Y., Powell, C. A. Loop-mediated isothermal amplification assay for detection of candidatus Liberibacter asiaticus: A causal agent of citrus huanglongbing. Plant Pathol. 34 (6), 499-505 (2018).
- Büscher, P., Njiru, Z. K. Loop-mediated isothermal amplification technology: Towards point of care diagnostics. PLoS Negl Trop Dis. 6 (6), e1572(2012).
- Notomi, T., Mori, Y., Tomita, N., Kanda, H. Loop-mediated isothermal amplification (LAMP): Principle, features, and future prospects. J Microbiol. 53 (1), 1-5 (2015).
- Ajibola, O., Gulumbe, B., Eze, A., Obishakin, E. Tools for detection of schistosomiasis in resource limited settings. Medical Sciences. 6 (2), 39(2018).
- Kumar, Y. S. B., et al. Loop-mediated isothermal amplification (LAMP): A rapid and sensitive tool for quality assessment of meat products. Compr Rev Food Sci Food SAF. 16 (6), 1359-1378 (2017).
- Hongling Ou, Y. W., et al. Rapid detection of multiple pathogens by the combined loop-mediated isothermal amplification technology and microfluidic chip technology. Ann Palliat Med. 10 (10), 11053-11066 (2021).
- Liang Wang, J. -X. L., et al. Quantitative polymerase chain reaction (qPCR)-based rapid diagnosis of helicobacter pylori infection and antibiotic resistance. J Vis Exp. (197), e65689(2023).
- Jing-Wen Lyu, B. X., et al. Rapid prediction of multidrug-resistant klebsiella pneumoniae through deep learning analysis of SERS spectra. Microbiol Spectr. 11 (2), e0412622(2023).
- Wei Liu, J. -W. T., et al. Discrimination between carbapenem-resistant and carbapenem-sensitive klebsiella pneumoniae strains through computational analysis of surface-enhanced Raman spectra a pilot study. Microbiol Spectr. 10 (1), e0240921(2022).
- Zhang, L. Y., et al. Classification and prediction of klebsiella pneumoniae strains with different MLST allelic profiles via SERS spectral analysis. Peer J. 11, e16161(2023).
- Zhi Liu, Q. Z., et al. Rapid and sensitive detection of salmonella in chickens using loop-mediated isothermal amplification combined with a lateral flow dipstick. J Microbiol Biotechnol. 29 (3), 454-464 (2019).
- Chen, F., et al. Fully Integrated microfluidic platform for multiplexed detection of hunov by a dynamic confined-space-implemented one-pot RPA-LAMP system. Adv Sci (Weinh). 21, e2306612(2023).
Access restricted. Please log in or start a trial to view this content.
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados