JoVE 비디오를 활용하시려면 도서관을 통한 기관 구독이 필요합니다. 전체 비디오를 보시려면 로그인하거나 무료 트라이얼을 시작하세요.
Method Article
Microfluidic-Chip-based Loop-Mediated Isothermal Amplification을 통해 하기도 감염을 일으키는 세균성 병원체의 신속한 검출
* 이 저자들은 동등하게 기여했습니다
요약
다양한 박테리아 병원체는 호흡기 감염을 일으킬 수 있으며 정확하게 감지하고 신속하게 치료하지 않으면 심각한 건강 문제를 초래할 수 있습니다. 루프 매개 등온 증폭을 통해 이러한 병원체를 빠르고 정확하게 검출하면 임상 환경에서 호흡기 감염을 효과적으로 관리하고 제어할 수 있습니다.
초록
호흡기 감염(RTI)은 임상 환경에서 가장 흔한 문제 중 하나입니다. 세균성 병원체의 신속하고 정확한 식별은 RTI의 관리 및 치료를 위한 실용적인 지침을 제공할 것입니다. 이 연구는 다중 채널 루프 매개 등온 증폭(LAMP)을 통해 호흡기 감염을 일으키는 세균성 병원체를 신속하게 검출하는 방법을 설명합니다. LAMP는 높은 정확도와 신뢰성으로 박테리아 핵산을 신속하게 검출하는 민감하고 특이적인 진단 도구입니다. 제안된 방법은 시간이 많이 걸리고 종종 낮은 수준의 박테리아 핵산을 검출하기 위해 더 높은 감도를 필요로 하는 기존 박테리아 배양 방법에 비해 상당한 이점을 제공합니다. 이 논문은 K. pneumoniae 감염 및 LAMP를 사용하여 하부 호흡기에서 검체(객담, 기관지 세척액 및 폐포 세척액)를 검출하는 다중 동시 감염의 대표적인 결과를 제시합니다. 요약하면, 다중 채널 LAMP 분석법은 임상 샘플에서 단일 및 다중 세균 병원체를 식별하는 빠르고 효율적인 수단을 제공하며, 이는 세균 병원체의 확산을 방지하고 RTI의 적절한 치료를 지원하는 데 도움이 될 수 있습니다.
서문
세균성 병원체에 의한 호흡기 감염(RTI)은 주로 전 세계적으로 이환율과 사망률에 기여합니다1. 2-3일 동안 지속되는 발열을 동반하는 상부 또는 하부 호흡기 증상으로 정의됩니다. 상기도 감염이 하기도 감염보다 더 흔하지만, 만성 및 재발성 호흡기 감염도 흔한 임상 상태로서 개인에게 큰 위험을 초래하고 의료 시스템에 상당한 부담을 준다2. RTI의 일반적인 세균성 병원체로는 폐렴구균3, 헤모필루스 인플루엔자4, 황색포도상구균, 대장균, 폐렴균, 스테노트로포모나스 말토필리아 등이 있습니다. 이러한 병원성 박테리아는 일반적으로 숙주의 비인두와 상부 호흡기의 점막 표면에 서식하여 인후통 및 기관지염과 같은 RTI의 전형적인 증상을 유발합니다. 폐렴은 상부 호흡기에서 하부 호흡기의 무균 영역으로 퍼질 때 폐렴을 일으키며, 호흡기를 통해 사람에서 사람으로 퍼질 수 있다5. 심한 경우에는 침습성 세균성 질환, 특히 세균성 폐렴, 뇌수막염 및 패혈증으로 이어질 수 있으며, 이는 전 세계 모든 연령대의 사람들에서 이환율과 사망률의 주요 원인입니다.
RTI에 대한 전통적인 검사는 인후 면봉과 객담 호흡기 샘플을 사용한 미생물 배양이 포함된다6. 또한 효소 결합 면역 흡착 검사(ELISA)와 같은 혈청학적 검사는 혈청에서 항체 또는 항원을 검출하는 반면, 응집 검사는 항체와 항원의 응집 반응을 관찰하여 감염을 감지합니다7. 미생물 배양은 RTI 진단을 위한 황금 표준으로 간주되지만 배양 양성률이 낮고 신뢰성이 낮으며 검출 주기가 길어 진단 효율성이 제한됩니다8. 실제로 RTI의 신속하고 정확한 진단은 박테리아 병원체의 정확한 박멸에 매우 중요합니다. 빠르고 효과적인 검출 방법은 병원체의 전염 속도를 줄이고 감염 기간을 단축하며 불필요한 항생제 사용을 줄이는 데 도움이 될 수 있습니다 9,10. 분자 생물학 기반 방법은 표적 유전자의 DNA 염기서열을 증폭하여 병원체를 검출하는 중합효소 연쇄 반응(PCR)과 같은 검출 속도를 크게 높입니다. 그러나 기존 PCR은 번거롭고 시간이 많이 소요되는 복잡한 온도 순환 장비를 필요로 합니다. 또한 PCR을 사용한 모든 DNA 증폭(실시간 PCR 제외)은 시간이 걸리는 산물의 전기영동 분리로 마무리됩니다. 제품을 시각화하려면 염료가 필요하며, 그 중 다수는 돌연변이 유발성 또는 발암성입니다. 따라서 RTI 세균성 병원체를 진단하기 위한 새로운 방법과 기술을 지속적으로 개발해야 합니다.
LAMP(Loop-Mediated Isothermal Amplification)는 2000년 Notomi et al.이 처음 개발한 새롭고 새로운 분자 기술입니다11. LAMP는 복잡한 온도 사이클링 장비 없이 안정적인 등온 조건에서 DNA를 증폭할 수 있어 신속한 검출에 적합하고 장비 복잡성과 비용을 줄입니다12. LAMP는 고감도로 낮은 농도의 표적 DNA를 검출할 수 있다13. 여러 특정 프라이머를 사용하여 표적 염기서열에 대한 선택성을 개선하고 위양성(false positive)의 가능성을 줄입니다14. LAMP는 RTI 검출에도 용이성, 속도 및 직관적인 작동으로 인해 임상 실험실에서 점차 널리 사용되고 있습니다. 이 연구에서는 그림 1과 같이 임상 샘플(객담, 기관지 세척액 및 폐포 세척액)에서 더 낮은 RTI를 감지하는 LAMP의 효과를 조사했습니다. LAMP는 낮은 RTI 감지에서 기존 테스트에 비해 속도, 감도 및 사용 편의성과 같은 이점을 제공하여 유망한 응용 분야임이 분명합니다.
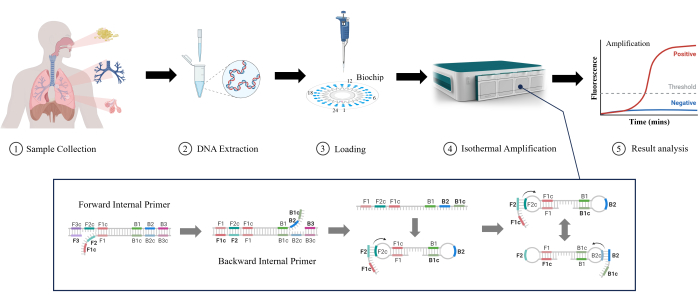
그림 1: LAMP 감지 방법의 개략도. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
Access restricted. Please log in or start a trial to view this content.
프로토콜
본 연구의 모든 검체는 광동성인민병원 윤리심의위원회(승인번호: KY2023-1114-01)의 평가 및 승인을 받았다. 모든 참가자는 실험 전에 정보에 입각한 동의서에 서명했습니다. 연구에 사용된 시약 및 장비는 재료 표에 나열되어 있습니다. 프로토콜에 사용된 약어는 보충 표 1에 나열되어 있습니다.
1. 하부 호흡기에서 임상 검체 채취
- 객담 채취
- 깨끗한 물로 구강과 치아를 세척하고 해당되는 경우 의치를 제거하십시오. 심한 기침을 하여 심부 호흡기 가래를 가래 용기(최소 0.6mL)에 넣습니다.
- 타액이나 콧물로 인한 오염을 피하십시오. 기침이 어려운 경우 45°C에서 100g/L NaCl 용액을 흡입하여 객담을 용이하게 합니다. 검체를 멸균되고 밀봉 가능한 용기에 옮깁니다.
알림: 가래 샘플은 아침과 항균제를 사용하기 전에 수집해야 합니다.
- 기관지 세척액(BLF)
- 수집기 머리를 콧구멍 또는 기관 절개술 부위(약 30cm 깊이)에서 기관으로 삽입합니다. 식염수 5mL를 주입하고 음압을 설정한 후 수집기 헤드를 회전시킨 후 천천히 빼냅니다.
- 추출된 점액을 채취하고 샘플링 용액(생리식염수 또는 주사용 멸균수)으로 수집기를 한 번 헹굽니다. 또는 채취 대신 소아 요로 카테터를 50mL 주사기에 연결합니다.
- 폐포 세척액(ALF)
- 흉부 영상 검사를 통해 병변 위치를 확인합니다. 가장 중요하거나 빠르게 진행되는 세척 영역을 선택합니다.
- 세척 중 생검 채널을 통해 기관지 분절에 2% 리도카인 1-2mL를 주입하여 세척된 폐 분절15에 국소 마취를 제공합니다. 전신 정맥 마취 중인 환자가 여전히 강한 기도 반응을 보이는 경우 2% 리도카인 1-2mL를 주입합니다.
- 국소 마취 후 입이나 코를 통해 광섬유 기관지 내시경을 삽입하여 인두를 통해 오른쪽 중엽의 기관지 또는 왼쪽 폐의 설측 부분으로 통과시킵니다. 기관지 가지의 입구에 팁을 놓고 멸균 생리식염수를 천천히 도입합니다.
- 세션당 30-50mL의 식염수를 100-250mL 용량(최대 300mL)으로 투여합니다. 추출한 점액을 밀봉된 멸균 용기에 모으십시오.
2. DNA 추출
- 하기도 샘플의 점도에 따라 적당량의 10% NaOH를 추가합니다. 점도가 낮은 샘플의 경우 액화 용액 샘플 부피의 2-3배를 추가합니다. 적당한 점도를 가진 샘플의 경우 액화 용액 샘플 부피의 5-6 배를 추가하십시오. 점도가 높은 샘플의 경우 액화 용액 샘플 부피의 8-10배를 추가합니다.
- 샘플 점도에 따라 10% NaOH의 부피를 조정합니다. 와류 믹서를 사용하여 15초 동안 샘플을 가능한 한 균일하게 분산시키고 액화를 위해 37°C에서 30분 동안 배양합니다.
알림: 더 두꺼운 샘플의 경우 NaOH의 부피를 늘리거나 액화 시간을 연장하십시오. 시료의 액화 품질은 후속 추출 효율에 직접적인 영향을 미칩니다. 이상적으로, 액화 샘플은 일관되고 끈적거리지 않는 일관성을 보여야 합니다.
- 샘플 점도에 따라 10% NaOH의 부피를 조정합니다. 와류 믹서를 사용하여 15초 동안 샘플을 가능한 한 균일하게 분산시키고 액화를 위해 37°C에서 30분 동안 배양합니다.
- 액화 시료 1mL를 1.5mL 원심분리 튜브에 피펫팅합니다. 15,777 × g 의 속도로 2-6 °C에서 5분 동안 원심분리한 다음 피펫을 사용하여 상층액을 제거하고 버립니다.
참고: 샘플을 흡입할 때 튜브 바닥에서 불순물을 뽑아내지 마십시오. 불순물이 많은 경우 샘플을 1,753-2,739 × g 에서 흡인 전에 1분 동안 원심분리할 수 있습니다. - 원심분리기 튜브에 세척액 1mL를 추가하고 와류를 넣어 튜브 바닥에서 침전물을 들어 올립니다. 완전히 분산시킬 필요는 없습니다.
- 용액을 15,777× g 에서 5분 동안 원심분리하고 상층액을 버리고 침전물을 만지지 않도록 합니다.
알림: 후속 ampliification에 영향을 미치지 않도록 세척 용액을 철저히 제거하십시오.
- 용액을 15,777× g 에서 5분 동안 원심분리하고 상층액을 버리고 침전물을 만지지 않도록 합니다.
- 원심분리기 튜브에 100μL의 핵산 추출 용액을 추가합니다. 피펫을 사용하여 침전물을 흡입하고 완전히 혼합합니다. 액체를 옮기고 함께 침전시켜 핵산 추출 튜브에 넣습니다. 핵산 추출 용액의 조성은 표 1에 제시되어 있다.
- 핵산 추출 튜브를 와류 믹서에 넣고 최소 5분 동안 중간 속도로 와류합니다. 와류 후 핵산 추출 튜브를 100°C에서 5분 동안 일정한 온도와 가열의 금속 배스로 옮깁니다.
- 15,777 × g 에서 2-6 °C에서 5분 동안 원심분리한 후 따로 보관합니다.
참고: PCR 증폭 반응이 24시간 이내에 수행되면 핵산을 4°C에서 보관할 수 있습니다. 증폭 후 핵산을 -20 °C에서 보관하십시오. 장기 보관(24시간 이상)의 경우 핵산을 -20°C에서 보관하십시오. 다시 사용할 준비가 되면 샘플을 자연적으로 해동하고 와류를 혼합하고 95°C 수조에서 5분 동안 가열하고 10,956× g 에서 1분 동안 원심분리한 다음 PCR 증폭을 위해 상층액을 사용합니다.
| 용액 | 구성 요소 | 수 | 사양 |
| 세척 용액 | 10mM 방전성 가이드 | 1 병 | 24mL/병 |
| 핵산 추출 시약 | 10mM Tris-HCl, 1mM EDTA, 핵산 방부제 | 튜브 2개 | 1.2mL/병 |
| 핵산 추출 튜브 | 유리 구슬 | 1 봉지 | 24 병 / 봉지 |
표 1: 핵산 추출 시약의 조성.
3. 루프 매개 등온 증폭 및 미세유체 칩
- 시약 및 미세유체 칩
- 미세유체 디스크 모양의 칩에서 등온 증폭 반응을 수행합니다( 재료 표 참조). 65°C에서 항온 증폭을 수행합니다.
- 항온 증폭 미세유체 칩 핵산 분석기에서 형광 염료 혼입 방법14 를 사용하여 실시간 형광 분석을 수행합니다. 가닥 치환 기능이 있는 중합효소를 사용하여 양성 시료에 대한 S자형 증폭 곡선을 관찰합니다.
- Loop-mediated isothermal amplification (루프 매개 등온 증폭)
- 2개의 내부 프라이머와 2개의 외부 프라이머(LAMP 키트와 함께 제공)를 포함한 4개의 특정 프라이머로 6개의 염기서열 영역을 표적화합니다.
참고: DNA는 가닥 치환 기능이 있는 DNA 중합효소를 사용하여 일정한 온도에서 지속적으로 복제 및 증폭됩니다. 반응 과정에는 덤벨 모양의 템플릿 합성 단계, 순환 증폭 단계, 신장 및 재활용 단계가 포함되며 궁극적으로 줄기 고리 및 콜리플라워와 같은 구조를 가진 DNA 조각의 혼합물을 형성합니다. 자세한 내용은 키트 지침을 참조하십시오(보충 File 1). - 반응 시스템에 2-loop 프라이머를 추가하여 반응 효율을 높이고, 이는 stem-loop 구조에 결합하여 strand displacement synthesis 및 cyclic replication을 시작합니다. 호흡기 병원체에 대한 핵산 검출 키트의 조성은 표 2에 제시되어 있습니다.
알림: 시약 키트는 LAMP 방법을 사용합니다. 이 원리는 약 65°C에서 DNA의 동적 평형을 기반으로 하며, 이중 가닥 DNA의 상보적 부위에서 염기쌍을 통해 확장되는 모든 프라이머는 다른 가닥이 해리되어 단일 가닥을 형성합니다.
- 2개의 내부 프라이머와 2개의 외부 프라이머(LAMP 키트와 함께 제공)를 포함한 4개의 특정 프라이머로 6개의 염기서열 영역을 표적화합니다.
- 미세유체 칩
참고: 각 미세유체 칩( 재료 표 참조)에는 시계 반대 방향으로 번호가 매겨진 24개의 반응 웰이 장착되어 있으며, 웰 1의 입구와 출구는 반응 웰 1에 해당합니다(그림 2). 특정 핵산 표적 염기서열의 증폭 및 검출을 위한 특정 프라이머 세트가 각 반응 웰에 내장되어 고정됩니다.- 샘플 DNA를 증폭 시약과 혼합합니다. 혼합물을 칩에 주입하고 실온에서 30초 동안 1,000× g 의 원심분리기에 분배합니다.
참고: 독립적인 등온 증폭 반응과 실시간 형광 검출은 칩의 각 반응 웰에서 동시에 발생합니다. 특정 반응 웰에서 S자형 증폭 곡선이 검출되면 해당 웰에 대한 해당 검출 지수는 양수입니다.
- 샘플 DNA를 증폭 시약과 혼합합니다. 혼합물을 칩에 주입하고 실온에서 30초 동안 1,000× g 의 원심분리기에 분배합니다.
| 구성 요소 | 구성 | 수 |
| 칩 | 뇌관 | 12 장소 |
| 씰링 필름 | / | 1 장 |
| 등온 증폭 시약 | 형광 염료, 효소 | 270 μL/튜브 |
| 긍정적 인 제어 | 대장균 게놈 DNA | 160 μL/튜브 |
표 2: 호흡기 병원체에 대한 핵산 검출 키트의 구성.
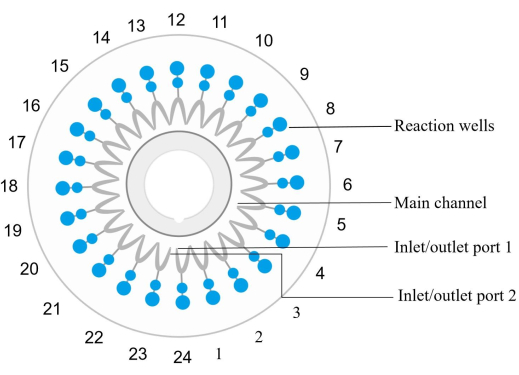
그림 2: 디스크 칩 구조 다이어그램. 반응 웰은 시계 반대 방향으로 순차적으로 번호가 매겨지며, 여기서 입구/출구 포트 1은 반응 웰 번호 1에 해당합니다. 반응웰 1, 4, 7, 10, 13, 16, 19, 22 및 24는 음성 대조군입니다. 반응 웰 6은 양성 대조군(E. coli)이다. 반응웰(12)은 내부 포지티브 대조군이고, 반응웰(23)은 외부 포지티브 대조군이다. 반응 웰 2는 spn을 검출합니다. 반응웰 3은 sau를 검출합니다. 반응웰(5)은 MRSA를 검출한다. 반응웰(8)은 kpn을 검출한다. 반응웰(9)은 pae를 검출한다. 반응웰(11)은 아바를 검출한다. 반응웰(14)은 sma를 검출한다. 반응웰(15)은 힌(hin)을 검출한다. 자세한 내용은 표 5 를 참조하십시오. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
4. 시료 전처리 및 세균 검출
- 등온 증폭 반응 시스템 준비
- 키트에서 등온 증폭 시약을 꺼내 실온에서 완전히 해동하십시오. 부드럽게 흔들어 잘 섞고 잠시 원심분리하여 튜브 바닥에 모입니다.
- 시약 보관 및 준비 영역에서 등온 증폭 시약 20μL를 준비된 200μL 원심분리기 튜브에 피펫팅합니다. 튜브를 덮고 샘플당 하나의 원심분리기 튜브를 사용하여 샘플(specimen) 준비 영역으로 이동합니다.
- 검체 준비 영역에서 표적 핵산 검체 34.5μL를 추가합니다. 부드럽게 흔들어 잘 섞고 잠시 원심분리하여 튜브 바닥에 모입니다. 각 핵산 증폭 반응 시스템의 총 부피는 54.5μL입니다(표 3).
- 칩 로딩 절차
- 칩 포장에 샘플 번호를 표시합니다. 포장 라벨이 위를 향하도록 미세유체 칩을 엽니다.
- 입구와 출구 포트가 위쪽을 향하도록 칩을 놓습니다. 피펫을 사용하여 준비된 증폭 반응 시스템의 50μL를 끌어와 입구 포트를 통해 칩의 주 채널에 추가합니다. 메인 채널이 채워지면 추가를 중지하고 보푸라기가 없는 조직으로 입구와 출구 포트 주변의 과도한 액체를 빠르게 닦아냅니다.
- 핀셋을 사용하여 하나의 밀봉 필름을 집어 입구와 출구 포트에 덮습니다. 깨끗한 피펫 팁을 밀봉 필름에 한 방향으로 누릅니다. 단단히 밀봉하십시오.
- qPCR 반응
- 핵산 분석기의 광원이 예열을 완료한 후 트레이 열기 버튼을 클릭합니다. 앞면이 위로 향하게 하여 칩을 트레이에 놓고 위치에 있는 작은 실린더가 칩의 중앙 틈에서 나와 고정되도록 합니다. 트레이의 중앙 위치 지정 장치를 칩의 큰 중앙 구멍에 맞춥니다.
- Close Tray(트레이 닫기) 버튼을 클릭하여 칩을 핵산 분석기에 삽입합니다.
- 탐지 인터페이스의 Sample Information(샘플 정보 ) 영역에서 샘플을 테스트하기 위한 정보를 입력합니다. 샘플 번호, 칩 번호 및 샘플 유형은 필수입니다. 나머지는 선택 사항입니다.
- 작업 영역에서 Start Detection 버튼을 클릭하여 샘플 검출을 시작합니다. 기기는 사전 설정된 프로그램에 따라 샘플 테스트를 수행합니다.
알림: 감지가 완료되면 장치와 함께 제공되는 소프트웨어가 자동으로 데이터 분석을 수행합니다. 동시에 핵산 분석기는 자동 냉각 프로세스를 시작합니다. 온도가 37°C로 떨어지면 기기가 자동으로 트레이를 열고 칩을 배출하여 회수합니다. 결과는 시스템에 의해 자동으로 해석됩니다(표 4).
- 품질 관리 표준
- 1개의 긍정적인 품질 관리와 9개의 부정적인 품질 관리가 이 시약 키트의 각 칩에 포함되어 있습니다. 긍정적 품질 관리에 대한 탐지 결과가 긍정적인지 확인하고 동시에 9개의 부정적 품질 관리에 대한 결과가 모두 음성인지 확인합니다.
참고: 이미지 오른쪽에 "품질 관리 결과: 실험 정상(Positive Normal, Negative Normal)"이라는 결과가 표시되어 테스트 결과가 유효함을 나타냅니다. 결과가 잘못되고 샘플 테스트 결과가 유효하지 않은 것으로 간주되는 경우 재테스트가 필요합니다. 인간 특이적 프라이머를 사용한 하나의 양성 내부 대조군이 칩에 포함되어 있으며, 그 결과는 임상 검체 검사에서 양성이고 검체의 인간 게놈 DNA 함량이 낮으면 음성으로 세포 수가 적음을 나타냅니다. 이러한 경우 샘플을 회수하고 다시 테스트하십시오.
- 1개의 긍정적인 품질 관리와 9개의 부정적인 품질 관리가 이 시약 키트의 각 칩에 포함되어 있습니다. 긍정적 품질 관리에 대한 탐지 결과가 긍정적인지 확인하고 동시에 9개의 부정적 품질 관리에 대한 결과가 모두 음성인지 확인합니다.
- 결과 분석
- 검출이 완료된 후 소프트웨어에서 상업적으로 보호되는 알고리즘과 결합된 2차 미분 최대 방법을 사용하여 급속 증폭 단계 동안 S자형 증폭 곡선의 첫 번째 변곡점을 계산합니다. 자세한 내용은 Respiratory Pathogens Nucleic Acid Detection Kit Instructions (Supplementary File 1)의 "Principles of the Test" 부분에 설명되어 있습니다.
참고: TP 값은 변곡점과 원점 사이의 시간 차이입니다. 결과는 TP 및 양수 판단 값을 기준으로 해석됩니다. 검출 지수에 대한 TP 값이 해당 지수에 대한 양수 판정 값보다 작거나 같으면 양수로 해석됩니다. TP 값이 양수 판단 값을 초과하면 지침의 "양수 결정 값"에 대한 기준에 따라 음수로 해석됩니다. "Fluorescence Curve Area"에는 정규화된 곡선이 표시되고, "Detection Result" 영역에는 각 파라미터에 대한 품질 관리 및 검출 결과가 표시됩니다(표 5).
- 검출이 완료된 후 소프트웨어에서 상업적으로 보호되는 알고리즘과 결합된 2차 미분 최대 방법을 사용하여 급속 증폭 단계 동안 S자형 증폭 곡선의 첫 번째 변곡점을 계산합니다. 자세한 내용은 Respiratory Pathogens Nucleic Acid Detection Kit Instructions (Supplementary File 1)의 "Principles of the Test" 부분에 설명되어 있습니다.
| 반응물 | 부피 (μl) |
| 등온 증폭 시약 | 20 |
| 템플릿 DNA | 34.5 |
표 3: 등온 증폭 반응 시스템.
| 걸음 | 1 | 2 |
| 온도 (°C) | 37 | 65 |
| 시간(분) | 3 | 47 |
표 4: 핵산 증폭 반응 프로그램.
| 인디케이터 이름 | 포지티브 컨트롤 값 |
| 폐렴 구균 (sp) | 30 |
| 황색포도상구균(sau) | 34 |
| 메티실린 내성 황색포도상구균(MRSA) | 22 |
| 폐렴균(Klebsiella pneumoniae, kpn) | 29 |
| 녹농균(Pseudomonas aeruginosa, pae) | 36 |
| 아시네토박터 바우마니(아바) | 36 |
| Stenotrophomonas maltophilia (sma) | 27 |
| 헤모필루스 인플루엔자(hin) | 36 |
표 5: 감염 지표에 대한 양성 대조 값.
Access restricted. Please log in or start a trial to view this content.
결과
이 실험은 등온 증폭 기술을 사용하여 미세 유체 디스크 칩에서 반응을 수행합니다. 반응은 형광 염료 삽입 방법을 사용하는 미세유체 칩 핵산 분석기에서 발생합니다. 등온 반응은 65°C의 일정한 온도에서 수행되며 실시간 형광 분석이 동시에 수행됩니다. 양성 샘플은 사슬 변위 기능이 있는 중합효소의 작용으로 증폭을 거쳐 S자형 증폭 곡선을 형성합니다. 이 원스텝 과정으로 표적 유전자의 증?...
Access restricted. Please log in or start a trial to view this content.
토론
호흡기 감염은 병원 관련 감염이 만연하여 환자에게 심각한 결과를 초래하고 사망률을 높인다16. 잠재적인 병원체를 시기적절하고 정확하게 식별한 후 효과적인 항생제를 투여하는 것이 성공적인 치료와 예후 개선의 핵심이며, 특히 전통 배양 방법에 내재된 한계를 감안할 때더욱 그렇다 17. 이 연구에서는 RTI를 빠르고 정확하게 탐지하기 위해 LAMP 기반 방법을 ...
Access restricted. Please log in or start a trial to view this content.
공개
저자는 이해 상충이 없음을 선언합니다.
감사의 말
우리는 광둥성 기초 및 응용 기초 연구 재단(보조금 번호 2022A1515220023)과 광동성 인민병원 고급 인재 연구 재단(보조금 번호)이 제공하는 재정적 지원에 크게 감사드립니다. KY012023293).
Access restricted. Please log in or start a trial to view this content.
자료
| Name | Company | Catalog Number | Comments |
| Bath Incubator(MK2000-2) | ALLSHENG | Provide a constant temperature environment | |
| Bronchial lavage fluid collector head | TIANPINGHUACHANG | SEDA 20172081375 | Collecting bronchoalveolar lavage fluid |
| Fiberoptic bronchoscope | OLYMPUS | SEDA 20153062703 | A flexible bronchoscope equipped with a fiberoptic light source and camera, to visually examine the airways and structures within the lungs. Assist in collecting bronchoalveolar lavage |
HR1500- B2 B2 | Haier | SEDA 20183541642 | Biosafety cabinet |
| NAOH | MACKLIN | S817977 | Liquefy viscous lower respiratory tract sample |
| Nucleic acid detection kit for respiratory tract pathogens | Capitalbio Technology | SEDA 20173401346 | Testing for bacteria infection |
| Nucleic acid extraction reagent | Capitalbio Technology | SEDA 20160034 | For DNA extraction |
| RTisochip-W | Capitalbio Technology | SEDA 20193220539 | Loop-mediated Isothermal Amplification |
| THERMO ST16R | Thermo Fisher Scientific | SEDA 20180585 | Centrifuge the residual liquid off the wall of the tube. |
| Vortex mixer VM-5005 | JOANLAB | For mixing reagent |
참고문헌
- GBD 2016 Lower Respiratory Infections Collaborators. Estimates of the global, regional, and national morbidity, mortality, and aetiologies of lower respiratory infections in 195 countries, 1990-2016: A systematic analysis for the global burden of disease study 2016. Lancet Infect Dis. 18 (11), 1191-1210 (2018).
- Niederman, M. S., Torres, A. Respiratory infections. Eur Respir Rev. 31 (166), 220150(2022).
- Weiser, J. N., Ferreira, D. M., Paton, J. C. Streptococcus pneumoniae: Transmission, colonization and invasion. Nat Rev Microbiol. 16 (6), 355-367 (2018).
- Watt, J. P., et al. Burden of disease caused by Haemophilus influenzae type b in children younger than 5 years_ global estimates. Lancet. 374 (9693), 903-911 (2009).
- Kadioglu, A., Weiser, J. N., Paton, J. C., Andrew, P. W. The role of streptococcus pneumoniae virulence factors in host respiratory colonization and disease. Nat Rev Microbiol. 6 (4), 288-301 (2008).
- Popova, G., Boskovska, K., Arnaudova-Danevska, I., Smilevska-Spasova, O., Jakovska, T. Sputum quality assessment regarding sputum culture for diagnosing lower respiratory tract infections in children. Open Access Maced J Med Sci. 7 (12), 1926-1930 (2019).
- Nuyttens, H., Cyncynatus, C., Renaudin, H., Pereyre, S., Identification Bébéar, C. expression and serological evaluation of the recombinant ATP synthase beta subunit of mycoplasma pneumoniae. BMC Microbiol. 10 (1), 216(2010).
- Noviello, S., Huang, D. B. The basics and the advancements in diagnosis of bacterial lower respiratory tract infections. Diagnostics (Basel). 9 (2), 37(2019).
- Hanson, K. E., et al. Molecular testing for acute respiratory tract infections: Clinical and diagnostic recommendations from the IDSA's diagnostics committee. Clin Infect Dis. 71 (10), 2744-2751 (2020).
- Daniel Reynolds, J. P. B., et al. The threat of multidrug-resistant/extensively drug-resistant gram-negative respiratory infections: Another pandemic. Eur Respir Rev. 31 (166), 220068(2022).
- T Notomi, H. O., et al. Loop-mediated isothermal amplification of DNA. Nucleic Acids Res. 28 (12), 63(2000).
- Soroka, M., Wasowicz, B., Rymaszewska, A. Loop-mediated isothermal amplification (LAMP): The better sibling of PCR. Cells. 10 (8), 1931(2021).
- Mori, Y., Kitao, M., Tomita, N., Notomi, T. Real-time turbidimetry of lamp reaction for quantifying template DNA. Biophys J. 59 (2), 145-157 (2004).
- Parida, M., Sannarangaiah, S., Dash, P. K., Rao, P. V., Morita, K. Loop mediated isothermal amplification (LAMP): A new generation of innovative gene amplification technique: Perspectives in clinical diagnosis of infectious diseases. Rev Med Virol. 18 (6), 407-421 (2008).
- Chinese Medical Association Respiratory Branch Critical Care Medicine Group, Working Committee on Critical Care Medicine of the Chinese Physicians Association Respiratory Physicians Branch. Standardization of collection, submission, testing, and interpretation of bronchoalveolar lavage fluid in ICU patients. Chin J Tubere Respir Dis. 43 (9), 744-756 (2020).
- Koch, A. M., Nilsen, R. M., Eriksen, H. M., Cox, R. J., Harthug, S. Mortality related to hospital-associated infections in a tertiary hospital: Repeated cross-sectional studies between 2004-2011. Antimicrob Resist Infect Control. 4 (1), 57(2015).
- Seymour, C. W. F. G., et al. Time to treatment and mortality during mandated emergency care for sepsis. N Engl J Med. 376 (23), 2235-2244 (2017).
- Choi, C. W., Hyun, J. W., Hwang, R. Y., Powell, C. A. Loop-mediated isothermal amplification assay for detection of candidatus Liberibacter asiaticus: A causal agent of citrus huanglongbing. Plant Pathol. 34 (6), 499-505 (2018).
- Büscher, P., Njiru, Z. K. Loop-mediated isothermal amplification technology: Towards point of care diagnostics. PLoS Negl Trop Dis. 6 (6), e1572(2012).
- Notomi, T., Mori, Y., Tomita, N., Kanda, H. Loop-mediated isothermal amplification (LAMP): Principle, features, and future prospects. J Microbiol. 53 (1), 1-5 (2015).
- Ajibola, O., Gulumbe, B., Eze, A., Obishakin, E. Tools for detection of schistosomiasis in resource limited settings. Medical Sciences. 6 (2), 39(2018).
- Kumar, Y. S. B., et al. Loop-mediated isothermal amplification (LAMP): A rapid and sensitive tool for quality assessment of meat products. Compr Rev Food Sci Food SAF. 16 (6), 1359-1378 (2017).
- Hongling Ou, Y. W., et al. Rapid detection of multiple pathogens by the combined loop-mediated isothermal amplification technology and microfluidic chip technology. Ann Palliat Med. 10 (10), 11053-11066 (2021).
- Liang Wang, J. -X. L., et al. Quantitative polymerase chain reaction (qPCR)-based rapid diagnosis of helicobacter pylori infection and antibiotic resistance. J Vis Exp. (197), e65689(2023).
- Jing-Wen Lyu, B. X., et al. Rapid prediction of multidrug-resistant klebsiella pneumoniae through deep learning analysis of SERS spectra. Microbiol Spectr. 11 (2), e0412622(2023).
- Wei Liu, J. -W. T., et al. Discrimination between carbapenem-resistant and carbapenem-sensitive klebsiella pneumoniae strains through computational analysis of surface-enhanced Raman spectra a pilot study. Microbiol Spectr. 10 (1), e0240921(2022).
- Zhang, L. Y., et al. Classification and prediction of klebsiella pneumoniae strains with different MLST allelic profiles via SERS spectral analysis. Peer J. 11, e16161(2023).
- Zhi Liu, Q. Z., et al. Rapid and sensitive detection of salmonella in chickens using loop-mediated isothermal amplification combined with a lateral flow dipstick. J Microbiol Biotechnol. 29 (3), 454-464 (2019).
- Chen, F., et al. Fully Integrated microfluidic platform for multiplexed detection of hunov by a dynamic confined-space-implemented one-pot RPA-LAMP system. Adv Sci (Weinh). 21, e2306612(2023).
Access restricted. Please log in or start a trial to view this content.
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유