Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Schneller Nachweis von bakteriellen Krankheitserregern, die Infektionen der unteren Atemwege verursachen, durch mikrofluidische Chip-basierte schleifenvermittelte isotherme Amplifikation
* Diese Autoren haben gleichermaßen beigetragen
In diesem Artikel
Zusammenfassung
Verschiedene bakterielle Krankheitserreger können Atemwegsinfektionen verursachen und zu ernsthaften Gesundheitsproblemen führen, wenn sie nicht genau erkannt und sofort behandelt werden. Der schnelle und genaue Nachweis dieser Erreger durch schleifenvermittelte isotherme Amplifikation ermöglicht ein effektives Management und eine effektive Kontrolle von Atemwegsinfektionen im klinischen Umfeld.
Zusammenfassung
Atemwegsinfektionen (RTIs) gehören zu den häufigsten Problemen im klinischen Umfeld. Die schnelle und genaue Identifizierung bakterieller Krankheitserreger wird praktische Leitlinien für das Management und die Behandlung von RTI liefern. Diese Studie beschreibt eine Methode zum schnellen Nachweis von bakteriellen Krankheitserregern, die Atemwegsinfektionen verursachen, mittels Mehrkanalschleifen-vermittelter isothermer Amplifikation (LAMP). LAMP ist ein empfindliches und spezifisches Diagnosewerkzeug, das bakterielle Nukleinsäuren schnell und mit hoher Genauigkeit und Zuverlässigkeit nachweist. Die vorgeschlagene Methode bietet einen erheblichen Vorteil gegenüber herkömmlichen bakteriellen Kultivierungsmethoden, die zeitaufwändig sind und oft eine höhere Empfindlichkeit für den Nachweis niedriger Mengen an bakteriellen Nukleinsäuren erfordern. In diesem Artikel werden repräsentative Ergebnisse der K . pneumoniae-Infektion und ihrer multiplen Koinfektionen mit LAMP zum Nachweis von Proben (Sputum, Bronchialspülflüssigkeit und Alveolarspülflüssigkeit) aus den unteren Atemwegen vorgestellt. Zusammenfassend lässt sich sagen, dass die Mehrkanal-LAMP-Methode ein schnelles und effizientes Mittel zur Identifizierung einzelner und mehrerer bakterieller Krankheitserreger in klinischen Proben bietet, was dazu beitragen kann, die Ausbreitung bakterieller Krankheitserreger zu verhindern und die angemessene Behandlung von RTIs zu unterstützen.
Einleitung
Atemwegsinfektionen (RTIs), die durch bakterielle Krankheitserreger verursacht werden, tragen weltweit in erster Linie zur Morbidität und Mortalität bei1. Es ist definiert als alle Symptome der oberen oder unteren Atemwege, die von 2-3 Tagen anhaltendem Fieber begleitet werden. Während Infektionen der oberen Atemwege häufiger sind als Infektionen der unteren Atemwege, sind chronische und wiederkehrende Atemwegsinfektionen auch häufige klinische Erkrankungen, die große Risiken für den Einzelnen darstellen und eine erhebliche Belastung für die Gesundheitssysteme darstellen2. Zu den häufigen bakteriellen Erregern von RTIs gehören unter anderem Streptococcus pneumoniae3, Haemophilus influenzae4, Staphylococcus aureus, Escherichia coli, Klebsiella pneumoniae, Stenotrophomonas maltophilia. Diese pathogenen Bakterien besiedeln in der Regel die Schleimhautoberflächen des Nasopharynx und der oberen Atemwege des Wirts und verursachen typische Symptome von RTIs wie Halsschmerzen und Bronchitis. Sie verursachen eine Lungenentzündung, wenn sie sich von den oberen Atemwegen auf sterile Bereiche der unteren Atemwege ausbreiten und sich über die Atemwege von Mensch zu Mensch ausbreitenkönnen 5. In schweren Fällen können sie auch zu invasiven bakteriellen Erkrankungen führen, insbesondere zu bakteriämischer Lungenentzündung, Meningitis und Sepsis, die weltweit die Hauptursachen für Morbidität und Mortalität bei Menschen aller Altersgruppen sind.
Herkömmliche Tests auf RTI umfassen mikrobiologische Kulturen unter Verwendung von Rachenabstrichen und Sputumproben der Atemwege6. Darüber hinaus weisen serologische Tests wie der Enzyme-Linked Immunosorbent Assay (ELISA) Antikörper oder Antigene im Serum nach, während Agglutinationstests die Agglutinationsreaktion von Antikörpern und Antigenen zum Nachweis einer Infektion beobachten7. Mikrobielle Kulturen gelten als Goldstandard für die Diagnose von RTIs, aber ihre niedrige Positivitätsrate, ihre geringe Zuverlässigkeit und ihr langer Nachweiszyklus schränken die diagnostische Effizienz ein8. In Wirklichkeit ist eine schnelle und genaue Diagnose von RTIs entscheidend für die präzise Ausrottung des bakteriellen Erregers. Schnelle und effektive Nachweismethoden können dazu beitragen, die Übertragungsrate von Krankheitserregern zu verringern, die Infektionsdauer zu verkürzen und den unnötigen Einsatz von Antibiotika zu verringern 9,10. Molekularbiologisch basierte Methoden beschleunigen den Nachweis erheblich, wie z. B. die Polymerase-Kettenreaktion (PCR), bei der die DNA-Sequenz eines Zielgens zum Nachweis von Krankheitserregern amplifiziert wird. Die traditionelle PCR erfordert jedoch komplexe Temperaturzyklusgeräte, die umständlich und zeitaufwändig sind. Darüber hinaus endet jede DNA-Amplifikation mittels PCR (mit Ausnahme der Real-Time-PCR) mit der elektrophoretischen Trennung des Produkts, was ebenfalls Zeit in Anspruch nimmt. Für die Visualisierung des Produkts werden Farbstoffe benötigt, von denen viele mutagen oder krebserregend sind. Daher ist es zwingend erforderlich, kontinuierlich neue Methoden und Technologien für die Diagnose von bakteriellen RTI-Erregern zu entwickeln.
Die schleifenvermittelte isotherme Amplifikation (LAMP) ist eine neuartige und aufstrebende molekulare Technologie, die ursprünglich von Notomi et al. im Jahr 200011 entwickelt wurde. LAMP kann DNA unter stabilen isothermen Bedingungen ohne komplexe Temperaturwechselgeräte amplifizieren, was es für einen schnellen Nachweis geeignet macht und die Komplexität und Kosten der Ausrüstung reduziert12. LAMP kann niedrige Konzentrationen der Ziel-DNA mit hoher Empfindlichkeit nachweisen13. Es werden mehrere spezifische Primer verwendet, um die Selektivität für Zielsequenzen zu verbessern und die Möglichkeit falsch positiver Ergebnisse zu verringern14. LAMP wird aufgrund seiner Leichtigkeit, Geschwindigkeit und intuitiven Bedienung, auch bei der Erkennung von RTIs, nach und nach in klinischen Labors weit verbreitet. In dieser Studie untersuchten wir die Wirksamkeit von LAMP bei der Detektion niedrigerer RTIs in klinischen Proben (Sputum, Bronchialspülflüssigkeit und Alveolarspülflüssigkeit), wie in Abbildung 1 gezeigt. Es ist offensichtlich, dass LAMP Vorteile wie Geschwindigkeit, Empfindlichkeit und Benutzerfreundlichkeit gegenüber herkömmlichen Tests bei der Erkennung von niedrigerem RTI bietet, was es zu einer vielversprechenden Anwendung macht.
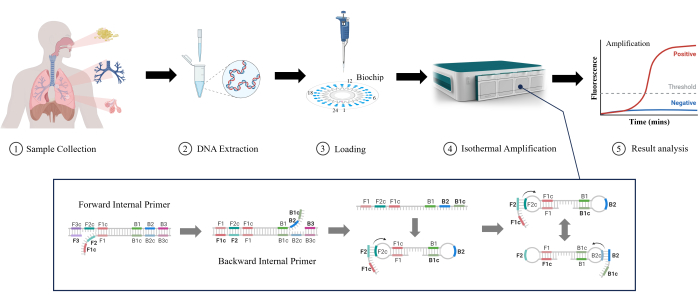
Abbildung 1: Schematische Darstellung der LAMP-Detektionsmethode. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Access restricted. Please log in or start a trial to view this content.
Protokoll
Alle Proben für diese Studie wurden vom Ethikprüfungsausschuss des Volkskrankenhauses der Provinz Guangdong bewertet und genehmigt (Zulassungsnummer: KY2023-1114-01). Alle Teilnehmer unterschrieben vor den Experimenten eine schriftliche Einverständniserklärung. Die für die Studie verwendeten Reagenzien und Geräte sind in der Materialtabelle aufgeführt. Die im Protokoll verwendeten Abkürzungen sind in der ergänzenden Tabelle 1 aufgeführt.
1. Entnahme klinischer Proben aus den unteren Atemwegen
- Sputum-Sammlung
- Reinigen Sie die Mundhöhle und die Zähne mit sauberem Wasser und stellen Sie gegebenenfalls sicher, dass die Prothese entfernt wird. Husten Sie kräftig, um tiefen Atemwegsauswurf in einen Sputumbehälter (mindestens 0,6 ml) auszustoßen.
- Vermeiden Sie eine Kontamination mit Speichel oder Nasenausfluss. Wenn der Husten schwierig ist, verabreichen Sie eine 100 g/l NaCl-Lösung bei 45 °C, um den Abwurf zu erleichtern. Füllen Sie die Probe in einen sterilen, verschließbaren Behälter um.
HINWEIS: Stellen Sie sicher, dass die Sputumproben morgens und vor der Einnahme von antibakteriellen Arzneimitteln entnommen werden.
- Bronchialspülflüssigkeit (BLF)
- Führen Sie den Auffangkopf aus dem Nasenloch oder der Tracheostomiestelle (ca. 30 cm tief) in die Luftröhre ein. Injizieren Sie 5 mL Kochsalzlösung, stellen Sie einen Unterdruck her, drehen Sie den Kollektorkopf und ziehen Sie ihn langsam zurück.
- Sammeln Sie den extrahierten Schleim und spülen Sie den Sammler einmal mit einer Probenahmelösung (physiologische Kochsalzlösung oder steriles Wasser für Injektionszwecke). Alternativ können Sie einen pädiatrischen Harnkatheter an eine 50-ml-Spritze anschließen, um die Entnahme zu ersetzen.
- Alveoläre Lavageflüssigkeit (ALF)
- Identifizieren Sie den Ort der Läsion durch bildgebende Untersuchungen des Brustkorbs. Wählen Sie den wichtigsten oder am schnellsten fortschreitenden Bereich für die Spülung aus.
- Während der Spülung werden 1-2 ml 2 % Lidocain durch den Biopsiekanal in das Bronchialsegment injiziert, wobei das lavierte Lungensegment15 örtlich betäubt wird. Bei Patienten unter intravenöser Vollnarkose, die immer noch starke Atemwegsreaktionen aufweisen, injizieren Sie 1-2 ml 2% Lidocain.
- Nach örtlicher Betäubung führen Sie ein faseroptisches Bronchoskop durch den Mund oder die Nase ein und gelangen über den Pharynx in den Bronchus des rechten Mittellappens oder das linguale Segment der linken Lunge. Platzieren Sie seine Spitze an der Öffnung des Bronchialams und führen Sie langsam sterile physiologische Kochsalzlösung ein.
- Verabreichen Sie 30-50 mL Kochsalzlösung pro Sitzung mit 100-250 mL Volumen (max. 300 mL). Sammeln Sie den extrahierten Schleim in einem verschlossenen sterilen Behälter.
2. DNA-Extraktion
- Basierend auf der Viskosität von Proben der unteren Atemwege eine angemessene Menge von 10 % NaOH hinzufügen. Bei Proben mit niedriger Viskosität das 2-3-fache des Volumens der Probe der Verflüssigungslösung hinzufügen. Bei Proben mit mäßiger Viskosität das 5-6-fache des Volumens der Probe der Verflüssigungslösung hinzufügen. Bei Proben mit hoher Viskosität das 8-10-fache des Volumens der Probe der Verflüssigungslösung hinzufügen.
- Das Volumen von 10 % NaOH wird entsprechend der Viskosität der Probe angepasst. Die Proben werden mit einem Vortex-Mischer 15 s lang so gleichmäßig wie möglich dispergiert und 30 Minuten lang bei 37 °C zur Verflüssigung inkubiert.
HINWEIS: Bei dickeren Proben ist das NaOH-Volumen zu erhöhen oder die Verflüssigungszeit zu verlängern. Die Verflüssigungsqualität der Proben hat direkten Einfluss auf die spätere Extraktionseffizienz. Im Idealfall sollten verflüssigte Proben eine gleichbleibende, nicht klebrige Konsistenz aufweisen.
- Das Volumen von 10 % NaOH wird entsprechend der Viskosität der Probe angepasst. Die Proben werden mit einem Vortex-Mischer 15 s lang so gleichmäßig wie möglich dispergiert und 30 Minuten lang bei 37 °C zur Verflüssigung inkubiert.
- Pipettieren Sie 1 ml der verflüssigten Probe in ein 1,5-ml-Zentrifugenröhrchen. Bei einer Drehzahl von 15.777 × g für 5 min bei 2-6 °C zentrifugieren, dann mit einer Pipette den Überstand entfernen und verwerfen.
HINWEIS: Vermeiden Sie beim Absaugen der Probe das Ziehen von Verunreinigungen vom Boden des Röhrchens. Wenn viele Verunreinigungen vorhanden sind, kann die Probe vor der Aspiration 1 min lang bei 1.753-2.739 × g zentrifugiert werden. - Geben Sie 1 ml Waschlösung in das Zentrifugenröhrchen und wirbeln Sie es ein, um den Niederschlag vom Boden des Röhrchens anzuheben. Es besteht keine Notwendigkeit, es vollständig zu verteilen.
- Zentrifugieren Sie die Lösung bei 15.777 × g für 5 Minuten, entsorgen Sie den Überstand und versuchen Sie, den Niederschlag nicht zu berühren.
HINWEIS: Stellen Sie sicher, dass die Waschlösung gründlich entfernt wird, um eine Beeinträchtigung der nachfolgenden Verstärkung zu vermeiden.
- Zentrifugieren Sie die Lösung bei 15.777 × g für 5 Minuten, entsorgen Sie den Überstand und versuchen Sie, den Niederschlag nicht zu berühren.
- Geben Sie 100 μl Nukleinsäure-Extraktionslösung in das Zentrifugenröhrchen. Verwenden Sie eine Pipette, um den Niederschlag zu aspirieren und gründlich zu mischen. Übertragen Sie die Flüssigkeit und den Niederschlag zusammen in ein Nukleinsäure-Extraktionsröhrchen. Die Zusammensetzung der Nukleinsäure-Extraktionslösung ist in Tabelle 1 dargestellt.
- Legen Sie die Nukleinsäure-Extraktionsröhrchen in einen Vortex-Mischer und wirbeln Sie sie mindestens 5 Minuten lang bei mittlerer Geschwindigkeit ein. Nach dem Vortexen die Nukleinsäure-Extraktionsröhrchen in ein Metallbad mit konstanter Temperatur überführen und 5 min lang auf 100 °C erhitzen.
- Bei 15.777 × g bei 2-6 °C 5 min zentrifugieren und beiseite stellen.
HINWEIS: Wenn die PCR-Amplifikationsreaktion innerhalb von 24 h durchgeführt wird, kann die Nukleinsäure bei 4 °C gelagert werden. Lagern Sie die Nukleinsäure nach der Amplifikation bei -20 °C. Für die Langzeitlagerung (über 24 h hinaus) die Nukleinsäure bei -20 °C lagern. Wenn die Probe wieder verwendet werden kann, tauen Sie sie auf natürliche Weise auf, wirbeln Sie sie zum Mischen ein, erhitzen Sie sie 5 Minuten lang in einem 95 °C heißen Wasserbad, zentrifugieren Sie sie 1 Minute lang bei 10.956 × g und verwenden Sie den Überstand für die PCR-Amplifikation.
| Lösung | Komponenten | Zahl | Spezifikation |
| Waschlösung | 10 mM EDTA | 1 Flasche | 24 mL/Flasche |
| Reagenz für die Nukleinsäure-Extraktion | 10 mM Tris-HCl, 1 mM EDTA, Nukleinsäure-Konservierungsmittel | 2 Röhren | 1,2 mL/Flasche |
| Nukleinsäure-Extraktionsröhrchen | Glasperlen | 1 Beutel | 24 Flaschen/Beutel |
Tabelle 1: Zusammensetzung des Nukleinsäure-Extraktionsreagenzes.
3. Schleifenvermittelte isotherme Amplifikation und Mikrofluid-Chip
- Reagenz und mikrofluidischer Chip
- Führen Sie isotherme Amplifikationsreaktionen auf einem mikrofluidischen scheibenförmigen Chip durch (siehe Materialtabelle). Führen Sie die Verstärkung bei konstanter Temperatur bei 65 °C durch.
- Durchführung einer Echtzeit-Fluoreszenzanalyse unter Verwendung eines Fluoreszenzfarbstoffeinbauverfahrens14 auf dem mikrofluidischen Chip-Nukleinsäureanalysator mit konstanter Temperaturamplifikation. Beobachten Sie eine S-förmige Amplifikationskurve für positive Proben unter Verwendung einer Polymerase mit Strangverdrängungsfunktion.
- Schleifenvermittelte isotherme Amplifikation
- Targeting von sechs Sequenzbereichen mit vier spezifischen Primern, darunter zwei innere Primer und zwei äußere Primer (im Lieferumfang des LAMP-Kits enthalten).
HINWEIS: Die DNA wird kontinuierlich repliziert und bei konstanter Temperatur unter Verwendung einer DNA-Polymerase mit Strangverdrängungsfunktion amplifiziert. Der Reaktionsprozess umfasst die hantelförmige Matrizensynthesephase, die zyklische Amplifikationsphase, die Elongations- und die Recyclingphase, wodurch schließlich eine Mischung aus DNA-Fragmenten mit stängelschleifen- und blumenkohlähnlichen Strukturen gebildet wird. Detaillierte Informationen finden Sie in den Anweisungen des Kits (Ergänzende Datei 1). - Fügen Sie dem Reaktionssystem Zwei-Schleifen-Primer hinzu, um die Reaktionseffizienz zu verbessern, die an die Stammschleifenstrukturen bindet und die Strangverdrängungssynthese und zyklische Replikation initiiert. Die Zusammensetzung des Nukleinsäure-Nachweiskits für Krankheitserreger der Atemwege ist in Tabelle 2 dargestellt.
HINWEIS: Das Reagenzienkit verwendet die LAMP-Methode. Das Prinzip basiert auf dem dynamischen Gleichgewicht der DNA bei etwa 65 °C, wobei jeder Primer, der sich durch die Basenpaarung an der komplementären Stelle der doppelsträngigen DNA erstreckt, dazu führt, dass der andere Strang dissoziiert und ein Einzelstrang gebildet wird.
- Targeting von sechs Sequenzbereichen mit vier spezifischen Primern, darunter zwei innere Primer und zwei äußere Primer (im Lieferumfang des LAMP-Kits enthalten).
- Mikrofluidischer Chip
HINWEIS: Jeder mikrofluidische Chip (siehe Materialtabelle) ist mit 24 Reaktionsvertiefungen ausgestattet, die gegen den Uhrzeigersinn nummeriert sind, wobei der Einlass und Auslass für Vertiefung 1 der Reaktionsvertiefung 1 entspricht (Abbildung 2). Ein spezifischer Satz von Primern für die Amplifikation und den Nachweis einer bestimmten Nukleinsäure-Zielsequenz wird in jede Reaktionsvertiefung eingebettet und fixiert.- Mischen Sie die DNA der Probe mit dem Amplifikationsreagenz. Injizieren Sie die Mischung in den Chip und verteilen Sie sie auf jede Reaktionsvertiefungszentrifuge bei 1.000 × g für 30 s bei Raumtemperatur.
HINWEIS: Unabhängige isotherme Amplifikationsreaktionen und Echtzeit-Fluoreszenzdetektion treten in jeder Reaktionsvertiefung auf dem Chip auf. Wenn eine S-förmige Amplifikationskurve in einer bestimmten Reaktionsvertiefung nachgewiesen wird, ist der entsprechende Nachweisindex für diese Vertiefung positiv.
- Mischen Sie die DNA der Probe mit dem Amplifikationsreagenz. Injizieren Sie die Mischung in den Chip und verteilen Sie sie auf jede Reaktionsvertiefungszentrifuge bei 1.000 × g für 30 s bei Raumtemperatur.
| Komponenten | Zusammensetzung | Zahl |
| Chip | Zündkapseln | 12 Plätze |
| Siegelfolie | / | 1 Blatt |
| Reagenz für die isotherme Amplifikation | Fluoreszierender Farbstoff, Enzym | 270 μL/Röhrchen |
| Positive Regelung | Genomische DNA von Escherichia coli | 160 μL/Röhrchen |
Tabelle 2: Zusammensetzung des Nukleinsäure-Nachweiskits für Krankheitserreger der Atemwege.
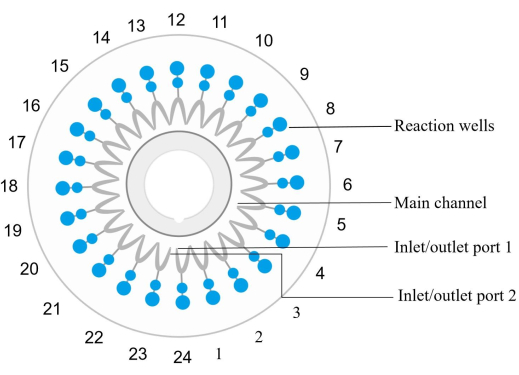
Abbildung 2: Strukturdiagramm des Scheibenchips. Die Reaktionsvertiefungen sind fortlaufend gegen den Uhrzeigersinn nummeriert, wobei der Einlass-/Auslassanschluss 1 der Reaktionsvertiefung Nummer 1 entspricht. Die Reaktionsvertiefungen 1, 4, 7, 10, 13, 16, 19, 22 und 24 sind Negativkontrollen. Die Reaktionsvertiefung 6 ist eine Positivkontrolle (E. coli). Die Reaktionsvertiefung 12 ist eine interne Positivkontrolle, und die Reaktionsvertiefung 23 ist eine externe Positivkontrolle. Die Reaktionsvertiefung 2 weist spn nach. Die Reaktionsvertiefung 3 detektiert sau. Die Reaktionsvertiefung 5 weist MRSA nach. Die Reaktionsvertiefung 8 weist kpn nach. Die Reaktionsvertiefung 9 weist pae nach. Die Reaktionsvertiefung 11 weist aba nach. Die Reaktionsvertiefung 14 detektiert sma. Die Reaktionsvertiefung 15 detektiert hin. Bitte beachten Sie Tabelle 5 für Beispieldetails. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
4. Probenvorbereitung und Bakteriennachweis
- Bereiten Sie das isotherme Amplifikationsreaktionssystem vor
- Nehmen Sie das isotherme Amplifikationsreagenz aus dem Kit und lassen Sie es bei Raumtemperatur vollständig auftauen. Vorsichtig schütteln, um gründlich zu mischen, und kurz zentrifugieren, um sich am Boden des Röhrchens zu sammeln.
- Pipettieren Sie im Bereich der Lagerung und Vorbereitung des Reagenziens 20 μl des isothermen Amplifikationsreagenzes in ein vorbereitetes 200-μl-Zentrifugenröhrchen. Decken Sie das Röhrchen ab und bringen Sie es mit einem Zentrifugenröhrchen pro Probe in den Probenvorbereitungsbereich.
- Geben Sie im Probenvorbereitungsbereich 34,5 μl der Zielnukleinsäureprobe hinzu. Leicht schütteln, um gründlich zu mischen, und kurz zentrifugieren, um sich am Boden des Röhrchens zu sammeln. Das Gesamtvolumen jedes Nukleinsäureamplifikationsreaktionssystems beträgt 54,5 μl (Tabelle 3).
- Verfahren zum Beladen der Späne
- Beschriften Sie auf der Chipverpackung die Probennummer. Öffnen Sie den Mikrofluidik-Chip mit dem Verpackungsetikett nach oben.
- Platzieren Sie den Chip mit den Ein- und Auslassanschlüssen nach oben. Ziehen Sie mit einer Pipette 50 μl des vorbereiteten Amplifikationsreaktionssystems und geben Sie es durch die Einlassöffnung in den Hauptkanal des Chips. Hören Sie auf, nachzufüllen, wenn der Hauptkanal gefüllt ist, und wischen Sie überschüssige Flüssigkeit um die Einlass- und Auslassöffnungen herum schnell mit fusselfreiem Tuch ab.
- Nehmen Sie mit einer Pinzette eine Dichtungsfolie auf und bedecken Sie sie mit den Ein- und Auslassöffnungen. Drücken Sie eine saubere Pipettenspitze in einer Richtung auf die Siegelfolie. Sorgen Sie für eine dichte Abdichtung.
- qPCR-Reaktion
- Nachdem die Lichtquelle des Nukleinsäureanalysators das Vorheizen abgeschlossen hat, klicken Sie auf die Schaltfläche Fach öffnen . Legen Sie den Chip mit der Vorderseite nach oben auf das Fach und stellen Sie sicher, dass der kleine Zylinder aus dem Mittelspalt des Späns herausragt, um ihn zu sichern. Richten Sie die mittlere Positioniervorrichtung im Fach an der großen zentralen Bohrung im Chip aus.
- Klicken Sie auf die Schaltfläche Fach schließen , um den Chip in den Nukleinsäureanalysator einzusetzen.
- Geben Sie im Bereich Probeninformationen auf der Nachweisschnittstelle die Informationen zum Testen der Probe ein. Probennummer, Chipnummer und Probentyp sind obligatorisch; andere sind optional.
- Klicken Sie im Operationsbereich auf die Schaltfläche Detektion starten , um die Probenerkennung zu starten. Das Gerät führt Stichprobentests gemäß dem voreingestellten Programm durch.
HINWEIS: Nach Abschluss der Erkennung führt die zugehörige Software des Geräts automatisch eine Datenanalyse durch. Gleichzeitig leitet der Nukleinsäureanalysator einen automatischen Kühlprozess ein. Sobald die Temperatur auf 37 °C sinkt, öffnet das Gerät automatisch das Fach und wirft den Chip zur Entnahme aus. Die Ergebnisse werden vom System automatisch interpretiert (Tabelle 4).
- Standards für die Qualitätskontrolle
- In diesem Reagenzienkit sind in jedem Chip eine positive und neun negative Qualitätskontrollen enthalten. Stellen Sie sicher, dass die Nachweisergebnisse für die positive Qualitätskontrolle positiv sind und gleichzeitig die Ergebnisse für alle neun negativen Qualitätskontrollen negativ sind.
HINWEIS: Die Ergebnisse werden auf der rechten Seite des Bildes mit dem Hinweis "Quality Control Results: Experiment Normal (Positive Normal, Negative Normal)" angezeigt, was darauf hinweist, dass die Testergebnisse gültig sind. Ein erneuter Test ist erforderlich, wenn ein Ergebnis falsch ist und das Probentestergebnis als ungültig erachtet wird. Eine positive interne Kontrolle mit humanspezifischen Primern ist in dem Chip eingeschlossen, deren Ergebnis für klinische Probentests positiv ist, während es negativ ist, wenn die Probe einen niedrigen Gehalt an humangenomischer DNA aufweist, was auf eine niedrigere Zellzahl hinweist. Sammeln Sie in solchen Fällen Proben erneut und testen Sie sie erneut.
- In diesem Reagenzienkit sind in jedem Chip eine positive und neun negative Qualitätskontrollen enthalten. Stellen Sie sicher, dass die Nachweisergebnisse für die positive Qualitätskontrolle positiv sind und gleichzeitig die Ergebnisse für alle neun negativen Qualitätskontrollen negativ sind.
- Ergebnisermittlung
- Verwenden Sie nach Abschluss der Detektion die Maximalmethode der zweiten Ableitung in Kombination mit kommerziell geschützten Algorithmen in der Software, um den ersten Wendepunkt der S-förmigen Amplifikationskurve während der schnellen Amplifikationsphase zu berechnen. Einzelheiten sind im Abschnitt "Grundsätze des Tests" in den Anweisungen für das Nukleinsäure-Nachweiskit für Atemwegserreger (Ergänzende Datei 1) beschrieben.
HINWEIS: Der TP-Wert ist die Zeitdifferenz zwischen dem Wendepunkt und dem Ursprung. Das Ergebnis wird auf der Grundlage der TP- und positiven Beurteilungswerte interpretiert. Wenn der TP-Wert für einen Erkennungsindex kleiner oder gleich dem positiven Beurteilungswert für diesen Index ist, wird er als positiv interpretiert. Wenn der TP-Wert den positiven Beurteilungswert übersteigt, wird er gemäß den Kriterien für "positiven Entscheidungswert" in der Anleitung als negativ interpretiert. Der "Fluoreszenzkurvenbereich" zeigt die normalisierte Kurve an, und der Bereich "Detektionsergebnis" zeigt die Qualitätskontroll- und Detektionsergebnisse für jeden Parameter an (Tabelle 5).
- Verwenden Sie nach Abschluss der Detektion die Maximalmethode der zweiten Ableitung in Kombination mit kommerziell geschützten Algorithmen in der Software, um den ersten Wendepunkt der S-förmigen Amplifikationskurve während der schnellen Amplifikationsphase zu berechnen. Einzelheiten sind im Abschnitt "Grundsätze des Tests" in den Anweisungen für das Nukleinsäure-Nachweiskit für Atemwegserreger (Ergänzende Datei 1) beschrieben.
| Reaktant | Volumen (μl) |
| Reagenz für die isotherme Amplifikation | 20 |
| Template-DNA | 34.5 |
Tabelle 3: Isothermes Amplifikationsreaktionssystem.
| Schritt | Eins | Zwei |
| Temperatur (°C) | 37 | 65 |
| Zeit (min) | 3 | 47 |
Tabelle 4: Reaktionsprogramm der Nukleinsäureamplifikation.
| Name des Indikators | Positiver Kontrollwert |
| Streptococcus pneumoniae (sp) | 30 |
| Staphylococcus aureus (sau) | 34 |
| Methicillin-resistenter Staphylococcus aureus (mrsa) | 22 |
| Klebsiella pneumoniae (kpn) | 29 |
| Pseudomonas aeruginosa (PAE) | 36 |
| Acinetobacter baumannii (aba) | 36 |
| Stenotrophomonas maltophilia (sma) | 27 |
| Haemophilus influenzae (hin) | 36 |
Tabelle 5: Positiver Kontrollwert für den Infektionsindikator.
Access restricted. Please log in or start a trial to view this content.
Ergebnisse
Bei diesem Experiment kommt eine isotherme Amplifikationstechnologie zum Einsatz, bei der Reaktionen auf einem mikrofluidischen Scheibenchip durchgeführt werden. Die Reaktion erfolgt auf einem mikrofluidischen Chip-Nukleinsäureanalysator unter Verwendung einer Fluoreszenzfarbstoff-Insertionsmethode. Die isotherme Reaktion wird bei einer konstanten Temperatur von 65 °C durchgeführt, gleichzeitig wird eine Echtzeit-Fluoreszenzanalyse durchgeführt. Positive Proben werden unter Einwirkung von Polymerase mit Kettenverdr?...
Access restricted. Please log in or start a trial to view this content.
Diskussion
Atemwegsinfektionen sind weit verbreitete Krankenhausinfektionen, die schwerwiegende Folgen für die Patienten haben und die Sterblichkeitsraten in die Höhe treiben16. Die rechtzeitige und genaue Identifizierung potenzieller Krankheitserreger, gefolgt von wirksamen Antibiotika, ist der Schlüssel zu einer erfolgreichen Behandlung und einer Verbesserung der Prognose, insbesondere angesichts der Einschränkungen traditioneller Kulturmethoden17. In dieser Studie haben wir ein...
Access restricted. Please log in or start a trial to view this content.
Offenlegungen
Die Autoren erklären, dass kein Interessenkonflikt besteht.
Danksagungen
Wir haben die finanzielle Unterstützung durch die Guangdong Basic and Applied Basic Research Foundation (Grant No. 2022A1515220023) und die Research Foundation for Advanced Talents des Guandong Provincial People's Hospital (Grant No. KY012023293).
Access restricted. Please log in or start a trial to view this content.
Materialien
| Name | Company | Catalog Number | Comments |
| Bath Incubator(MK2000-2) | ALLSHENG | Provide a constant temperature environment | |
| Bronchial lavage fluid collector head | TIANPINGHUACHANG | SEDA 20172081375 | Collecting bronchoalveolar lavage fluid |
| Fiberoptic bronchoscope | OLYMPUS | SEDA 20153062703 | A flexible bronchoscope equipped with a fiberoptic light source and camera, to visually examine the airways and structures within the lungs. Assist in collecting bronchoalveolar lavage |
HR1500- B2 B2 | Haier | SEDA 20183541642 | Biosafety cabinet |
| NAOH | MACKLIN | S817977 | Liquefy viscous lower respiratory tract sample |
| Nucleic acid detection kit for respiratory tract pathogens | Capitalbio Technology | SEDA 20173401346 | Testing for bacteria infection |
| Nucleic acid extraction reagent | Capitalbio Technology | SEDA 20160034 | For DNA extraction |
| RTisochip-W | Capitalbio Technology | SEDA 20193220539 | Loop-mediated Isothermal Amplification |
| THERMO ST16R | Thermo Fisher Scientific | SEDA 20180585 | Centrifuge the residual liquid off the wall of the tube. |
| Vortex mixer VM-5005 | JOANLAB | For mixing reagent |
Referenzen
- GBD 2016 Lower Respiratory Infections Collaborators. Estimates of the global, regional, and national morbidity, mortality, and aetiologies of lower respiratory infections in 195 countries, 1990-2016: A systematic analysis for the global burden of disease study 2016. Lancet Infect Dis. 18 (11), 1191-1210 (2018).
- Niederman, M. S., Torres, A. Respiratory infections. Eur Respir Rev. 31 (166), 220150(2022).
- Weiser, J. N., Ferreira, D. M., Paton, J. C. Streptococcus pneumoniae: Transmission, colonization and invasion. Nat Rev Microbiol. 16 (6), 355-367 (2018).
- Watt, J. P., et al. Burden of disease caused by Haemophilus influenzae type b in children younger than 5 years_ global estimates. Lancet. 374 (9693), 903-911 (2009).
- Kadioglu, A., Weiser, J. N., Paton, J. C., Andrew, P. W. The role of streptococcus pneumoniae virulence factors in host respiratory colonization and disease. Nat Rev Microbiol. 6 (4), 288-301 (2008).
- Popova, G., Boskovska, K., Arnaudova-Danevska, I., Smilevska-Spasova, O., Jakovska, T. Sputum quality assessment regarding sputum culture for diagnosing lower respiratory tract infections in children. Open Access Maced J Med Sci. 7 (12), 1926-1930 (2019).
- Nuyttens, H., Cyncynatus, C., Renaudin, H., Pereyre, S., Identification Bébéar, C. expression and serological evaluation of the recombinant ATP synthase beta subunit of mycoplasma pneumoniae. BMC Microbiol. 10 (1), 216(2010).
- Noviello, S., Huang, D. B. The basics and the advancements in diagnosis of bacterial lower respiratory tract infections. Diagnostics (Basel). 9 (2), 37(2019).
- Hanson, K. E., et al. Molecular testing for acute respiratory tract infections: Clinical and diagnostic recommendations from the IDSA's diagnostics committee. Clin Infect Dis. 71 (10), 2744-2751 (2020).
- Daniel Reynolds, J. P. B., et al. The threat of multidrug-resistant/extensively drug-resistant gram-negative respiratory infections: Another pandemic. Eur Respir Rev. 31 (166), 220068(2022).
- T Notomi, H. O., et al. Loop-mediated isothermal amplification of DNA. Nucleic Acids Res. 28 (12), 63(2000).
- Soroka, M., Wasowicz, B., Rymaszewska, A. Loop-mediated isothermal amplification (LAMP): The better sibling of PCR. Cells. 10 (8), 1931(2021).
- Mori, Y., Kitao, M., Tomita, N., Notomi, T. Real-time turbidimetry of lamp reaction for quantifying template DNA. Biophys J. 59 (2), 145-157 (2004).
- Parida, M., Sannarangaiah, S., Dash, P. K., Rao, P. V., Morita, K. Loop mediated isothermal amplification (LAMP): A new generation of innovative gene amplification technique: Perspectives in clinical diagnosis of infectious diseases. Rev Med Virol. 18 (6), 407-421 (2008).
- Chinese Medical Association Respiratory Branch Critical Care Medicine Group, Working Committee on Critical Care Medicine of the Chinese Physicians Association Respiratory Physicians Branch. Standardization of collection, submission, testing, and interpretation of bronchoalveolar lavage fluid in ICU patients. Chin J Tubere Respir Dis. 43 (9), 744-756 (2020).
- Koch, A. M., Nilsen, R. M., Eriksen, H. M., Cox, R. J., Harthug, S. Mortality related to hospital-associated infections in a tertiary hospital: Repeated cross-sectional studies between 2004-2011. Antimicrob Resist Infect Control. 4 (1), 57(2015).
- Seymour, C. W. F. G., et al. Time to treatment and mortality during mandated emergency care for sepsis. N Engl J Med. 376 (23), 2235-2244 (2017).
- Choi, C. W., Hyun, J. W., Hwang, R. Y., Powell, C. A. Loop-mediated isothermal amplification assay for detection of candidatus Liberibacter asiaticus: A causal agent of citrus huanglongbing. Plant Pathol. 34 (6), 499-505 (2018).
- Büscher, P., Njiru, Z. K. Loop-mediated isothermal amplification technology: Towards point of care diagnostics. PLoS Negl Trop Dis. 6 (6), e1572(2012).
- Notomi, T., Mori, Y., Tomita, N., Kanda, H. Loop-mediated isothermal amplification (LAMP): Principle, features, and future prospects. J Microbiol. 53 (1), 1-5 (2015).
- Ajibola, O., Gulumbe, B., Eze, A., Obishakin, E. Tools for detection of schistosomiasis in resource limited settings. Medical Sciences. 6 (2), 39(2018).
- Kumar, Y. S. B., et al. Loop-mediated isothermal amplification (LAMP): A rapid and sensitive tool for quality assessment of meat products. Compr Rev Food Sci Food SAF. 16 (6), 1359-1378 (2017).
- Hongling Ou, Y. W., et al. Rapid detection of multiple pathogens by the combined loop-mediated isothermal amplification technology and microfluidic chip technology. Ann Palliat Med. 10 (10), 11053-11066 (2021).
- Liang Wang, J. -X. L., et al. Quantitative polymerase chain reaction (qPCR)-based rapid diagnosis of helicobacter pylori infection and antibiotic resistance. J Vis Exp. (197), e65689(2023).
- Jing-Wen Lyu, B. X., et al. Rapid prediction of multidrug-resistant klebsiella pneumoniae through deep learning analysis of SERS spectra. Microbiol Spectr. 11 (2), e0412622(2023).
- Wei Liu, J. -W. T., et al. Discrimination between carbapenem-resistant and carbapenem-sensitive klebsiella pneumoniae strains through computational analysis of surface-enhanced Raman spectra a pilot study. Microbiol Spectr. 10 (1), e0240921(2022).
- Zhang, L. Y., et al. Classification and prediction of klebsiella pneumoniae strains with different MLST allelic profiles via SERS spectral analysis. Peer J. 11, e16161(2023).
- Zhi Liu, Q. Z., et al. Rapid and sensitive detection of salmonella in chickens using loop-mediated isothermal amplification combined with a lateral flow dipstick. J Microbiol Biotechnol. 29 (3), 454-464 (2019).
- Chen, F., et al. Fully Integrated microfluidic platform for multiplexed detection of hunov by a dynamic confined-space-implemented one-pot RPA-LAMP system. Adv Sci (Weinh). 21, e2306612(2023).
Access restricted. Please log in or start a trial to view this content.
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten