Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Generation und Reinigung von Menschen INO80 Chromatin Remodeling Komplexe und Subkomplexe
In diesem Artikel
Zusammenfassung
Dieses Protokoll beschreibt ein Verfahren zur Erzeugung und Reinigung von Wildtyp-und Mutantenversionen der menschlichen INO80 Chromatin-Remodeling-Komplexes. Epitop-markierte Versionen der INO80 Untereinheiten stabil in HEK293-Zellen exprimiert, und die Gesamtkomplexe und Komplexe ohne spezifische Sätze von Untereinheiten durch Immunaffinitätschromatographie gereinigt.
Zusammenfassung
INO80 Chromatin-Remodeling-Komplexe regulieren Nukleosomen Dynamik und DNA-Zugänglichkeit durch Katalyse ATP-abhängigen Nukleosomen Umbau. Menschliche INO80 Komplexe aus Protein-Untereinheiten 14 einschließlich INO80 eine SNF2 artigen ATPase, die sowohl als die katalytische Untereinheit und dem Stützgerüst zur Montage der Komplexe dient. Funktionen der anderen Untereinheiten und die Mechanismen, durch die sie den INO80 Komplexes Chromatin-Remodeling-Aktivität beitragen noch schwer, was zum Teil auf die Herausforderung der Erzeugung INO80 Baugruppen in menschlichen Zellen oder heterologen Expressionssystemen. Dieses Protokoll beschreibt JOVE eine Prozedur, die die Reinigung der menschlichen INO80 Chromatin-Remodeling Subkomplexe, die eine Untereinheit fehlt oder eine Teilmenge der Untereinheiten ermöglicht. N-terminal FLAG-Epitop markiert INO80 cDNA stabil in humanen embryonalen Nierenzellen (HEK) 293-Zelllinien mit Flp-vermittelte Rekombination eingeführt. In dem Fall, dass eine Untergruppe von Untereinheiten des Komplexes ist INO80 zu be gelöscht, statt einer Mutante INO80 Proteine, die die Plattform für die Montage dieser Untereinheiten sind, nicht immer zum Ausdruck bringt. Im Falle eine individuelle Untereinheit ist erschöpft sein, eine Transfekte siRNAs dieser Untereinheit in ein HEK 293-Zelllinie, die stabil FLAG getaggt INO80 ATPase. Kernextrakte werden hergestellt und FLAG Immunpräzipitation durchgeführt, um die Proteinfraktionen, die INO80 Derivate bereichern. Die Zusammensetzungen gereinigt INO80 Subkomplexe kann dann unter Verwendung von Verfahren wie beispielsweise Immunoblotting, Silberfärbung und Massenspektrometrie analysiert werden. Die INO80 und INO80 Subkomplexe entsprechend diesem Protokoll erzeugt wird, kann weiter analysiert werden unter Verwendung von verschiedenen biochemischen Tests, welche in den beiliegenden JOVE Protokoll beschrieben sind. Die hier beschriebenen Methoden können für die Untersuchung der strukturellen und funktionellen Eigenschaften einer Säuger mehreren Untereinheiten Chromatin-Remodeling-Komplexe und Modifikation angepasst werden.
Einleitung
Evolutionär konservierte Familie SNF2 Chromatin-Remodeling-Komplexe sind Schlüsselregulatoren des Chromatins Organisation und DNA-Zugänglichkeit 1. Diese Umbau-Komplexe schließen immer eine zentrale SNF2 artigen ATPase Untereinheit, die in einigen Fällen, montiert mit verschiedenen Zubehör Proteine und bildet mit mehreren Untereinheiten makromolekulare Baugruppen. Um die molekularen Details der ATP-abhängigen Chromatin-Remodeling-Prozess zu untersuchen, ist es wichtig, die Beiträge der gegebenen Untermengen von Untereinheiten und / oder den Domain-Strukturen Aktivitäten der Komplexe zu verstehen. Solche Analysen erfordern die Fähigkeit, hoch gereinigten Mutante Komplexe, insbesondere Protein-Untereinheiten oder Domänenstrukturen fehlt erzeugen.
Vorherige Struktur-Funktions-Studien von ATP-abhängigen Chromatin-Remodeling-Komplexe sind allgemein für die Hefe-Modellsystem fokussiert aufgrund der überlegenen Handhabbarkeit des Hefe-Genom (siehe zB Lit. 1-4). Angesichts der Erhaltung derZusammensetzung Einheit und Funktionalität unter ortho-Remodeling-Komplexe, Untersuchungen der Struktur und Funktion der Hefe-Remodeling-Komplexen haben wichtige Einblicke in ihre Gegenstücke in höheren Eukaryoten ist. Dennoch nennenswerte Spezies-spezifische Unterschiede zwischen den Umbau Komplexe existieren, von Gewinn oder Verlust der Spezies-spezifische Untereinheiten, Gewinn oder Verlust der Arten-spezifische Domänen von konservierten Untereinheiten und Sequenzvariabilität innerhalb konservierten Domänen von konservierten Untereinheiten entstehen. Solche Unterschiede können im Prinzip durch die Notwendigkeit für höhere eukaryotische Zellen zu neuen molekularen und zellulären Umgebungen anzupassen angetrieben werden. So verstehen, wie Untereinheiten der höheren eukaryotischen Umbau Komplexe dazu beitragen, die Nukleosomen Umbauprozess ist wertvoll, denn es wirft nicht nur Licht auf die grundlegenden Mechanismen der ATP-abhängigen Chromatin-Remodeling-Prozess, sondern kann auch wertvolle Einblicke in die Mechanismen, durch die Chromatinstruktur und Genexpression in higsie Eukaryonten reguliert.
Bisher gab es nur strukturelle und funktionelle Untersuchungen mit mehreren Untereinheiten Säuger Chromatin-Remodeling-Komplexe, zum Teil aufgrund der Schwierigkeiten bei der biochemisch definiert Chromatin-Remodeling-Komplexe und Unterkomplexe beschränkt. Wir haben teilweise diese Schwierigkeiten mit den unten beschriebenen Verfahren, bei denen Immunaffinitätsreinigung verwendet wird, um intakt INO80 oder INO80 Subkomplexe aus menschlichen Zellen stabil exprimiert N-terminal FLAG-Epitop markierten Wildtyp-oder mutierte Versionen von INO80 5-7 vorzubereiten umgangen (Abbildung 1) . Intakte INO80 Komplexe aus menschlichen Zellen zu erhalten, wird Flp-vermittelte Rekombination verwendet werden, um transgene HEK293-Zelllinien, die stabil FLAG-Epitop markierten cDNAs Untereinheiten des Komplexes INO80 8-10 kodieren, zu erzeugen. Da die Überexpression von INO80 Untereinheiten kann etwas giftig sein, ist es notwendig, zu isolieren und zu pflegen klonalen Zelllinien unter selektiven CoBedingungen ausgebracht, um eine stabile Expression des Transgens in den vielen Passagen für die Erweiterung des Großzellkulturen erforderlich zu gewährleisten. Um kleinere INO80 Subkomplexe, die nur eine Teilmenge der Untereinheiten enthalten, zu erhalten, haben wir erfolgreich verwendet zwei Ansätze (Abbildung 2a, b). In der ersten, erzeugen wir HEK293 Flp-In-Zelllinien, die stabil mutierten Versionen INO80 die Domains für die Wechselwirkung mit spezifischen Untereinheiten 5 erforderlich fehlt. Alternativ wird siRNA-vermittelten Zuschlags verwendet, um die gewünschte Untereinheit von den Zellen eines geeigneten FLAG-markierten INO80 Einheit (unveröffentlichte Daten) Expression führen. Schließlich, um die Menschen INO80 Komplexe zu reinigen, ist FLAG-Agarose-Chromatographie basiert 11 verwendet, um eine INO80 haltigen Fraktion aus Kernextrakten zu bereichern, damit eine effektive Senkung der Anwesenheit von verunreinigenden Cytosolproteine in der letzten Fraktion, die gereinigtes INO80 oder INO80 Subkomplexe.
Protokoll
1. Erzeugung und Kultur der HEK293 Stabile Zelllinien, Ganzkörperansicht oder Mutant Versionen von FLAG-Epitop-markierten INO80 oder Andere INO80 Komplexe Untereinheiten
- Klon-cDNA in voller Länge oder mutierte menschliche INO80 ATPase-Untereinheit oder eine andere INO80 in den Säugetierexpressionsvektor pcDNA5 / FRT mit einer in-frame, N terminalen FLAG-Epitop-Tag kodiert.
- Bestätigung der Sequenz der insertierten cDNAs durch DNA-Sequenzierung, bevor fortgefahren wird.
- Um die Transfektion durchzuführen, wachsen Flp-In HEK293-Zellen in 10 cm-Gewebekulturplatten in einem Medium, das DMEM (Dulbecco Modified Eagle Medium), 5% Glutamin und 10% FBS (fötales Rinderserum).
- Wenn die Zellen ~ 70% Konfluenz in den einzelnen Gewebekulturschale ein Gemisch von 40 ul FuGENE6 Transfektionsreagenz, 0,5 ug der geeigneten pcDNA5 / FRT-Expressionsplasmid, und 9,5 ug pOG44, die Flp-Rekombinase kodiert, in einem Gesamtvolumen 800 ul.
- 48 Stunden später, Trypsinize und split Zellen in einem Verhältnis von 1:30 in 10-cm-Schalen, und sie wachsen in DMEM mit 5% Glutamin, 10% FBS und 100 ug / ml Hygromycin B für 3 - 4 Wochen. Ändern Sie den Kulturmedium, wenn es zu vergilben beginnt (in der Regel alle 3 - 5 Tage).
- Um positive Klone, die die höchste Stufe der FLAG-markierten Protein exprimieren zu identifizieren, wählen Sie einzelne Hygromycin B-resistente Kolonien und sie auf einer einzigen Vertiefung einer 24-Well-Platte.
- Sobald die Zellen Konfluenz erreichen 80%, Ernten der Zellen aus jeder Vertiefung in ~ 1 ml PBS und Pellet durch Zentrifugation bei 1.000 × g für 5 min.
- Nach Entfernen des Überstands, Resuspendieren des Zellpellets in 60 ul SDS-PAGE-Probenpuffer.
- Betreff Hälfte des Zellpellet resuspendiert SDS PAGE und Western-Blot auf die Expression des FLAG überwachen getaggt Köder-Protein; speichern Sie die andere Hälfte für zukünftige Analysen.
- Wenn kein FLAG-markierten menschlichen INO80 Protein in Zelllysaten detektiert, erweitern Sie den ersten klonalen Zell popuweitere nung durch Plattierung Zellen aus einzelnen Vertiefungen einer 24-Well-Platte in 15 cm Gewebekulturschalen.
- Zellen aus 15-cm-Schalen, resuspendieren nahezu konfluenten Zellen in eiskalter PBS und in ein 50 ml konisches Röhrchen geerntet.
- Zu bringen, um ein Endvolumen von 50 ml durch Zugabe von zusätzlichem PBS.
- Pellet-Zellen bei 1000 × g für 5 min entfernt und so viel wie möglich von dem Überstand.
- Zellpellet in 1 ml Lys450-Puffer (20 mM HEPES-NaOH pH 7,9, 450 mM NaCl, 0,5% Triton X-100, 10 mM KCl, 4 mM MgCl 2, 0,2 mM EDTA, 10% Glycerin, 1 mM DTT, 200 uM PMSF und 1: 1.000 Protease Inhibitor Cocktail).
HINWEIS: hier und anderswo, DTT, PMSF und Protease-Inhibitor-Cocktail immer Puffer hinzuzufügen unmittelbar vor Beginn eines Experiments. - Immuno-Niederschlag FLAG-markierten Proteine aus der resultierenden Ganzzelllysat mit 20 ul Anti-FLAG-Agarose-Gel, und zu analysieren, die die Flagge Eluate mittels Western Blot. [Für Details immunoprecipitation Verfahren siehe Schritt 5]
- Bereiten gefrorenen Bestände der gewünschten klonalen Zelllinien 12 und speichern sie in flüssigem Stickstoff bis zur Verwendung.
2. Wachsende HEK293 Zelllinien in Rollerflaschen
Für große Vorbereitung INO80 Komplexe, Kulturzellen in 10 - 20 Rollerflaschen; eine typische Ausbeute aus jeder Rolle Flasche ist ~ 1 ml gepackte Zellen.
- Zu jedem Rollflasche, 200 ml DMEM, 5% Glutamin und 10% Kälberserum, ohne Hygromycin B.
- Übertragen Sie alle Zellen aus einer nahezu konfluenten 15 cm Schüssel in jeder Rolle Flasche
- Ort Rollerflaschen in einem 37 ° C Inkubator Rollflasche und drehen sich mit 0,2 Umdrehungen pro Minute.
- Sobald Zellen zu erreichen ~ 70% Konfluenz, abgießen und entsorgen Sie die Medium.
- Fügen ~ 50 ml eiskaltem PBS zu jeder Flasche. Halten Flaschen horizontal, leicht schwenken, um die Zellschicht zu lösen.
- Übertragen Sie die resuspendierten Zellen auf 250 ml plastic konischen Flaschen und auf Eis.
- Spülen Rollerflaschen mit zusätzlichen PBS, sequentiell übertragen sie von Flasche zu Flasche. Wenn Spüllösung nicht mehr klar ist (in der Regel nach Gebrauch auf etwa 5 Flaschen spülen), fügen Sie ihn der Zellsuspension.
- Pellet-Zellen durch Zentrifugation bei 400 × g für 10 min.
- Vorsichtig resuspendieren Zellen in PBS, kombinieren sie zu einem einzigen 250-ml-Erlen Flasche, und auf Eis zu halten, bis die Weiterverarbeitung.
3. siRNA-vermittelten Knockdown von INO80 Untereinheiten in Zellen, Another FLAG-markierten INO80 Subunit
Um INO80 Subkomplexe fehlt eine einzige Untereinheit zu erhalten, zu nutzen FLAG-Immunkomplexe aus INO80 siRNA behandelten Zellen oder Zellen, die stabil shRNA reinigen. Die "umgekehrte" siRNA (small interfering RNA) Transfektion hier beschriebene Protokoll ist für HEK293 Zellen, die in 15 cm-Schalen optimiert. Das Protokoll ist für eine einzelne 15-cm-Schale von Zellen und shoULD sich dementsprechend abhängig von der Anzahl der Zellen benötigt skaliert werden. Biochemisch nützlich Mengen INO80-Komplex aus siRNA-behandelten Zellen herzustellen, sollte man Skalierung auf bis zu 40 Kulturen in 15 cm-Schalen gewachsen; 4 ml gepacktes Zellpellet - diese werden ca. 2 ergeben.
- HEK293-Zellen wachsen die gewünschte Untereinheit des Komplexes INO80 stabil exprimieren, um in der Nähe von Konfluenz in 15 cm-Schalen.
- Hinzufügen eines Volumens von siRNA Resuspensionspuffer (20 mM KCl, 6 mM HEPES, pH 7,5, und 0,2 mM MgCl 2) ausreicht, um eine 50 uM Stammlösung von siRNA in ein Röhrchen lyophilisiert siRNA herzustellen. Pipettieren die Lösung auf und ab ein paar Mal und Inkubation auf einem Kolbenpendel für 30 min bei RT, um sicherzustellen, dass die siRNA vollständig aufgelöst.
- Bereiten Sie eine Transfektionscocktail enthält siRNAs und Transfektionsreagenz. Mix 10 ul der 50 uM siRNA-Stammlösung mit 32 ul Lipofectamine RNAiMAX und zu 4 ml Opti-MEM hinzufügen Reduzierte Serum Medium mit sehr schonende Durchmischung; alNieder alle Reagenzien auf RT vor Gebrauch ins Gleichgewicht.
- Inkubieren der Mischung bei RT für 30 min.
- Bereiten Flp-In HEK293-Zellen die gewünschte INO80 Untereinheit für die Transfektion stabil exprimieren.
- Während der Inkubation von Schritt 3.4, waschen Zellen in den 15-cm-Schalen einmal mit PBS RT.
- Nach dem Entfernen PBS, Behandlung Zellen mit 1 ml Trypsin nur, bis sie zum Abheben der Platte beginnen.
- Sofort 10 ml Vollmedium (DMEM + 5% Glutamin + 10% FBS), um Zellen mit Trypsin behandelt und vorsichtig mischen. Sammle die Zellen durch Zentrifugation bei 1.000 xg für 5 Minuten bei RT.
- Zellpellet in ~ 4 ml Vollmedium, und zählen Sie die Zelle mit einer Aufwirbelung Hämocytometers.
- Verdünnte Zellen mit komplettem Medium auf eine Konzentration von ~ 5,4 × 10 6 / ml.
- Zu jeder 15 cm Schüssel, fügen Sie 15 ml komplettem Kulturmedium, gefolgt von 4 ml Transfektion Cocktail. Vorsichtig schwenken, um die mittel-und Transfektionscocktail gewährleisten thoroughly gemischt. Schließlich, fügen Sie einem ml Zellsuspension und wieder vorsichtig schwenken, um Zellen gleichmäßig zu verteilen.
- Nach 60 h der Kultur in einem 37 ° C, 5% CO 2-Inkubator, entfernen Sie vorsichtig Medium, Zellen in eiskaltem PBS, und fahren Sie sofort auf Kernextrakte herzustellen.
4. Herstellung von Kernextrakten
Dieses Verfahren ist aus dem Protokoll von Dignam 13 modifiziert und kann nach oben oder unten in Abhängigkeit von der Größe des Ausgangszellpellets skaliert werden. Typischerweise 1 ml gepacktes Zellpellet ergibt 1 ml des endgültigen Kernextrakt. Alle Puffer sollten eiskalt sein, und alle Schritte sollten in einem Kühlraum oder auf Eis durchgeführt, wenn ein geeignetes Kühlraum nicht zur Verfügung steht.
- Isolierung der Kerne:
- Sanft Zellen übertragen zu einem geeignet bemessenen (15 oder 50 ml) abgestuft konischen Rohr und Schleudern bei 1000 × g für 10 min bei 4 ° C ist.
- Entfernen Sie den Überstand, und messen die Lautstärke des gepackten Zell pellassen. 1 ml gepackte Zellen entspricht ~ 3 x 10 8 HEK293-Zellen.
- Hinzufügen 5 gepackten Zellvolumina Puffer A (10 mM HEPES, pH 7,9, 1,5 mM MgCl 2, 10 mM KCl und frisch zuge 1 mM DTT, 200 uM PMSF und 1: 1.000 Protease Inhibitor Cocktail). Zellpellet durch vorsichtiges Pipettieren.
- Inkubieren auf Eis für genau 10 min.
- Pellet die Zellen bei 1.000 g für 10 min bei 4 ° C, und entfernen Sie den Überstand.
- Die Grße des Zellpellets sollte während der Inkubation in Puffer A bis zu 2-fach erhöht
- Die Zellen in zwei gepackte Zellvolumen Puffer A, und übertragen Sie die Zellsuspension auf einem passenden Dounce Gewebehomogenisator. Wenn die mit weniger als 2 ml gepackter Zellen, mit einem 7 ml Homogenisator; für 2 - 4 ml gepackte Zellen, verwenden Sie ein 15 ml-Homogenisator; und für 10 oder mehr ml gepackter Zellen, verwenden Sie ein 40 ml-Homogenisator.
- Homogenisierung der Zellsuspension mit dem losen Glaspistill der Dounce Homogenisator until 90% der Zellen färben sich positiv mit 1% Trypanblau.
- Die Suspension in einem 45-ml-Hochgeschwindigkeitszentrifugenröhrchen und Schleudern bei 25.000 xg für 20 min bei 4 ° C in einem JA-17-Rotor oder ähnliches (siehe Tabelle der Werkstoffe / Zubehör).
- Von der Kernpellet, den Überstand, der cytosolischen Proteine oder Proteine, die bei der Fraktionierung aus dem Kern entweichen enthält.
- Extrahieren Kerne mit Salz:
- Fügen Puffer C (20 mM HEPES, pH 7,9, 25% Glycerin, 1,5 mM MgCl 2, 0,2 mM EDTA und frisch zuge 1 mM DTT, 200 uM PMSF und 1: 1.000 Protease Inhibitor Cocktail) in den Kernpellet; verwenden 2,5 ml Puffer C für jeweils 3 ml Ausgangszellvolumen (~ 1 x 10 9 Zellen).
- Mit einem Glasstab oder einer Pipette zu verdrängen das Kernpellet von der Wand des Rohres und übertragen die gesamte Mischung auf einem Dounce-Homogenisator mit einer geeigneten Größe.
- Resuspendieren der Kerne durch Homogenisieren the Mischung mit zwei Schlägen aus einer losen Stößel.
- Übertragen Sie die Kernfraktion resuspendiert in ein gekühltes Becherglas. Wähle einen Becher derart, daß die Suspension wird das Becherglas auf mindestens 0,5 cm tief füllen.
- Die Kernproteine aus Chromatin oder andere unlösliche Strukturen zu extrahieren, schrittweise Erhöhung der Salzkonzentration der Suspension auf 0,42 M NaCl durch tropfenweise Zugabe von 5 M NaCl unter leichtem Rühren der Suspension mit einer Pipette oder Glasstab auf Eis; Wenn alle 5 M NaCl wurde zugegeben, sollte die Lösung sehr viskos oder gelartigen. Berechnung des Volumens von 5 M NaCl nötig, um die Lösung auf eine Endkonzentration von 0,42 M NaCl nach der folgenden Formel zu bringen:
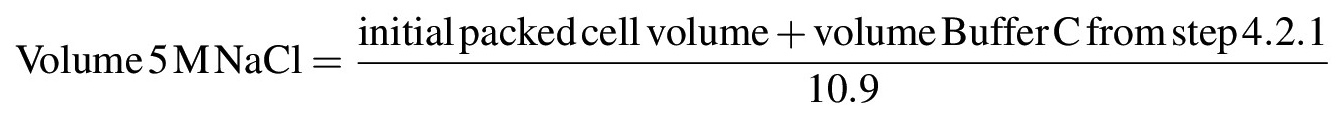
- Die viskose Suspension sorgfältig Transfer in 10 ml Polycarbonat Rohre für einen Typ 70.1 Ti-Rotor (oder gleichwertig) oder 70 ml Polycarbonat-Flaschen für einen Typ 45 Ti or ähnlich Rotor (siehe Tabelle der Materialien / Ausrüstung). Dicht schließen mit Parafilm bei Verwendung von 10-ml-Röhrchen mit Kappe oder Montage, wenn unter Verwendung von 70-ml-Flaschen.
- Langsam rocken die verschlossenen Röhrchen bei 4 ° C für 30 min mit einem Kolbenpendel.
- Dreh die Proben in einem 45 Ti-Typ oder 70.1 Ti-Rotor für 30 Minuten bei 120.000 × g bei 4 ° C ist.
- Den Überstand in einem einzigen Kunststoffrohr oder Flasche. Dieser Überstand ist die Kernextrakt und das Pellet enthält Chromatin und andere Kerntrümmer.
- Teilen Sie die Kernextrakt in handliche Teilmengen, einfrieren in flüssigem Stickstoff, und speichern Sie sie bei -80 ° C.
5. Immunoaffinitätsreinigung des Menschen INO80 oder INO80 Subkomplexe
- Zum Auftauen gefrorenen Kernextrakt, Ort Röhrchen, die den Extrakt auf den Tisch-oder Walzenrohre zwischen den Händen, bis die gefrorene Material wird zu einem Brei. Dann legen Sie die Röhrchen auf Eis oder im Kühlraum, bis der Extrakt ist komplett thawed.
- Übertragen der aufgetauten Zellkernextrakt 10 ml Ultrazentrifugenröhrchen aus Polycarbonat und Spin bei 100.000 × g für 20 min bei 4 ° C in einem Typ 70.1 Ti-Rotor oder gleichwertig jede Niederschlag, die während des Gefrier-Auftau-Zyklus gebildet haben können.
- Den Überstand in einem 15 ml konischen Röhrchen. Fügen Sie frisches DTT, PMSF und Protease Inhibitor Cocktail, um Endkonzentrationen von 1 mM DTT, 200 uM PMSF und 1: 1.000 Protease Inhibitor Cocktail.
- Anti-Flag Agarose zur Immun vorzubereiten, Transfer 200 ul 50% Gülle von Anti-Flag Agarose-Kügelchen in ein 1,5 ml Mikrozentrifugenröhrchen mit einem P200 oder ähnliche Pipette mit einer Spitze, aus dem das Ende wurde mit einem sauberen Skalpell oder Rasierklinge.
- Pellet die Perlen durch Zentrifugation in einer Tischmikrozentrifuge bei 8.000 g für 30 sek. Entfernen Sie den Überstand und Waschen der Kügelchen durch Resuspendieren der Beads in 1 ml Lys450 Puffer und Pellet die Perlen bei 8.000 g für 30 sek. Waschen Sie die bEADS zwei weitere Male.
- Resuspendieren gewaschen Anti-FLAG-Agarose-Kügelchen in etwa 100 ul des Kernextrakt mit einem P200 oder ähnliches Volumen einstellbare Pipette mit einer Spitze, aus dem das Ende wurde mit einem sauberen Skalpell oder einer Rasierklinge und, mit der gleichen Spitze, Übertragung geschnitten die resuspendierten Perlen an den 15 ml konischen Röhrchen, die den Extrakt. Ein paar Mal wiederholen, bis alle Perlen haben an den 15-ml-Tube überführt.
- Inkubieren des Extrakts / Perlen-Mischung für 4 h bei 4 ° C unter langsamer Rotation auf einer Labor-Rotator. OPTIONAL: Fügen Sie in einer Konzentration von 25 Einheiten / ml Benzonase zu entfernen verunreinigende DNA.
- Sammeln Sie die FLAG-Agarose durch Zentrifugation bei 1.000 × g für 5 min bei 4 ° C ist.
- Resuspendieren in 10 ml Lys450 zu waschen, Inkubation 5 min bei 4 ° C unter leichtem Schütteln auf einem Kolbenpendel. Pellet die Kügelchen bei 1.000 × g für 5 min bei 4 ° C ist.
- Resuspendieren in 100 bis 150 ul Lys450 und Transfer Perlen in ein 1,5 ml Mikrozentrifugenröhrchen. Co150 ul Lys450, bis alle die Perlen auf die Mikrozentrifugenröhrchen überführt worden - ntinue um die 15 ml konischen Röhrchen mit 100 zu spülen.
- Spin-down die Perlen bei 8.000 xg für 30 sec bei 4 ° C in einer Mikrozentrifuge. Waschen dreimal mit 1 ml Lys450 und einmal mit 1 ml EB100-Puffer (10 mM HEPES pH 7,9, 10% Glycerin, 100 mM NaCl, 1,5 mM MgCl 2, 0,05% Triton X-100, und frisch zuge 1 mM DTT, 200 uM PMSF , und 1: 1.000 Protease Inhibitor Cocktail).
- Zur Elution gebundener Proteine, 200 ul EB100-Puffer mit 0,25 mg / ml 1x FLAG-Peptid. Inkubieren 30 min bei jeder 4 ° C auf einem Kolbenpendel.
- Pellet die Kügelchen bei 8.000 xg für 30 sec bei 4 ° C in einer Mikrozentrifuge. Den Überstand, der den eluierten INO80 Komplex enthält, in ein frisches Mikrozentrifugenröhrchen.
- Wiederholen Sie die Elution vier weitere Male, und bündeln alle Überstände in einem einzigen Rohr.
- Um restliche FLAG-Agarose-Perlen aus der eluierten Proteinfraktion zu entfernen,passieren die Eluat durch eine leere Spin-Säule.
- Konzentrieren die eluierte Proteinfraktion ~ 10-fach unter Verwendung einer Ultrafilterzentrifuge Gerät (50.000 Molekulargewichtsgrenze).
- Um das FLAG-Peptid zu entfernen, übergeben Sie die konzentrierte Proteinfraktion nacheinander durch zwei Entsalzungssäulen.
- Teilen das gereinigte entsalzte Proteinfraktion in 20 ul-Aliquots in flüssigem Stickstoff eingefroren und bei -80 ° C gelagert.
- Analysieren Sie die Zusammensetzung der Untereinheiten INO80 oder INO80 Subkomplexe auf Silber-gefärbten Gelen oder durch Western Blotting, und schätzen ihre Konzentrationen von semi-quantitativen Western-Blot unter Verwendung von rekombinanten Präparaten INO80 Untereinheiten bekannter Konzentration als Standards.
Ergebnisse
Figur 1 zeigt ein Flussdiagramm eine Zusammenfassung der zur Erzeugung, Reinigung und Charakterisierung menschlicher INO80 ATP-abhängigen Chromatin-Remodeling-Komplexen Verfahren.
Wie in den 2 und 3 dargestellt, diese Verfahren ermöglichen die Erzeugung von beiden Wildtyp INO80 und INO80 Subkomplexe, die verschiedenen Untereinheiten fehlen, wodurch die nachfolgende biochemische Analysen der Beitrag dieser fehlenden Untereinheiten an enzyma...
Diskussion
Strukturelle und funktionelle Untersuchungen mit mehreren Untereinheiten Säuger Chromatin-Remodeling-Komplexe aus höheren Eukaryoten durch die Schwierigkeit der Herstellung von biochemisch nützlichen Mengen solcher Komplexe, die mutierte oder fehlende bestimmte Untereinheiten Untereinheiten insgesamt behindert. Es gibt eine Reihe von technischen Hürden: Erstens hat die genetische Manipulation in Säugerzellen technisch anspruchsvoll und zeitaufwendig gewesen. Anders als Hefezellen, deren Genom kann leicht bearbeitet...
Offenlegungen
Die Autoren erklären, dass sie keine finanziellen Interessen konkurrieren.
Danksagungen
Arbeit in der Autoren Labor wird durch einen Zuschuss aus dem National Institute of General Medical Sciences (GM41628) und durch einen Zuschuss an die Stowers Institut für medizinische Forschung von der Helen Nelson Medical Research Fund in der Greater Kansas City Community Foundation unterstützt.
Materialien
| Name | Company | Catalog Number | Comments |
| Dulbecco's Modified Eagle Medium | Cellgro | 10-013-CV | |
| Glutamax-I (stablized glutamine) | Life Technologies | 35050-079 | |
| Fetal Bovine Serum | SAFC | 12176C | |
| FuGENE6 transfection reagent | Promega | E2312 | |
| Hygromycin B, sterile in PBS | AG Scientific | H-1012-PBS | |
| pcDNA5/FRT vector | Life Technologies | V6010-20 | |
| Flp-In HEK293 cells | Life Technologies | R780-07 | |
| pOG44 Flp-Recombinase Expression Vector | Life Technologies | V600520 | |
| EZview Red ANTI-FLAG M2 Affinity Gel | Sigma | F2426 | |
| calf serum | SAFC | 12138C | |
| TARGETplus SMARTsiRNA pool | Dharmacon / Thermo Scientific | various | |
| 5x siRNA resuspension buffer | Dharmacon / Thermo Scientific | #B-002000-UB-100 | |
| Lipofectamine RNAiMAX | Life Technologies | 13778 | |
| Opti-MEM Reduced Serum Medium | Life Technologies | 51985-091 | |
| PBS | Cellgro | 45000 | VWR |
| TrypLE (trypsin) | Life Technologies | 12604 | |
| 1x FLAG Peptide | Sigma | F3290 | |
| Micro Bio-Spin Chromatography Column | Bio-Rad | 737-5021 | |
| Amicon Ultra Centrifugal Filter Device (50k MWCO) | Amicon | UFC805024 | Fisher Scientific |
| Zeba Desalting Columns | Thermo Scientific | 89882 | |
| Anti-FLAG M2 antibody, mouse | Sigma | F3165 | |
| Anti-FLAG M2 antibody, rabbit | Sigma | F7425 | |
| Protease Inhibitor Cocktail | Sigma | P8340 | |
| benzonase | Novagen | 70664 | |
| Equipment | |||
| JS-4.2 rotor in a J6 centrifuge | Beckman-Coulter | 339080 | |
| JA-17 rotor | Beckman-Coulter | 369691 | |
| 10 ml polycarbonate tubes | Beckman-Coulter | 355630 | |
| 70 ml polycarbonate bottles | Beckman-Coulter | 355655 | |
| Type 45 Ti rotor | Beckman-Coulter | 339160 | |
| Type 70.1 Ti rotor | Beckman-Coulter | 342184 | |
| BD Clay Adams Nutator Mixer | BD Diagnostics | 15172-203 | VWR |
| Glas-Col Tube/Vial Rotator | Glas-Col | 099A RD4512 | |
| PCR thermal cycler PTC 200 | MJ Research | PTC 200 | |
| roller bottle incubator | Bellco biotechnology | 353348 | |
| Immobilon-FL Transfer Membrane 7 x 8.4 | Millipore | IPFL07810 | |
| lubricated 1.5 ml microcentrifuge tubes | Costar | 3207 | |
Referenzen
- Clapier, C. R., Cairns, B. R. The biology of chromatin remodeling complexes, Annual Review of Biochemistry. 78, 273-304 (2009).
- Szerlong, H., Hinada, K., Viswanathan, R., Erdjument-Bromage, H., Tempst, P., Cairns, B. R. The HSA domain binds nuclear actin-related proteins to regulate chromatin-remodeling ATPases. Nature Struct. Mol. Biol. 15 (5), 469-476 (2008).
- Shen, X., Mizuguchi, G., Hamiche, A., Wu, C. A chromatin remodelling complex involved in transcription and DNA processing. Nature. 406 (6795), 541-544 (2000).
- Shen, X., Ranallo, R., Choi, E., Wu, C. Involvement of actin-related proteins in ATP-dependent chromatin remodelling. Mol. Cell. 12, 147-155 (2003).
- Chen, L., et al. Subunit organization of the human INO80 chromatin remodeling complex: an evolutionarily conserved core complex catalyzes ATP-dependent nucleosome remodeling. Journal of Biological Chemistry. 286 (13), 11283-11289 (2011).
- Sauer, B. Site-specific recombination: developments and applications. 5 (5), 521-527 (1994).
- Gorman, S., Fox, D. T., Wahl, G. M. Recombinase-mediated gene activation and site-specific integration in mammalian cells. Science. 251 (4999), 1351-1355 (1991).
- Jin, J., et al. A mammalian chromatin remodeling complex with similarities to the yeast INO80 complex. Journal of Biological Chemistry. 280 (50), 41207-41212 (2005).
- Cai, Y., et al. YY1 functions with INO80 to activate transcription. Nature Structural and Molecular Biology. 14 (9), 872-874 (2007).
- Yao, T., et al. Distinct Modes of Regulation of the Uch37 Deubiquitinating Enzyme in the Proteasome and in the Ino80 Chromatin-Remodeling Complex. Mol.Cell. 31 (6), 909-917 (2008).
- Brizzard, B. L., Chubet, R. G., Vizard, D. L. Immunoaffinity purification of FLAG epitope-tagged bacterial alkaline phosphatase using a novel monoclonal antibody and peptide elution, Biotechniques. 16 (4), 730-735 (1994).
- Barker, K. . At the Bench: A Laboratory Navigator. 10, 207-245 (2005).
- Dignam, J. D., Lebovitz, R. M., Roeder, R. G. Accurate transcription initiation by RNA polymerase II in a soluble extract from isolated mammalian cell nuclei. Nucleic. Acids. Res. 11 (5), 1475-1489 (1983).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten