È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Generazione e Purificazione di umani INO80 rimodellamento della cromatina Complessi e Sottocomplessi
In questo articolo
Riepilogo
Questo protocollo descrive una procedura per la generazione e purificare wild type e mutanti versioni del complesso rimodellamento della cromatina INO80 umana. Epitopi tagged versioni di subunità INO80 sono stabilmente espressi in cellule HEK293 e complessi completi e complessi privi di specifici gruppi di subunità sono purificati mediante cromatografia di immunoaffinità.
Abstract
INO80 complessi di rimodellamento della cromatina regolano le dinamiche nucleosoma e l'accessibilità del DNA catalizzando ATP-dipendente rimodellamento nucleosoma. Complessi INO80 umani sono composti da 14 subunità proteiche tra cui INO80, un SNF2-come ATPasi, che serve sia come la subunità catalitica e il patibolo per l'assemblaggio dei complessi. Funzioni delle altre subunità ei meccanismi attraverso i quali essi contribuiscono alla cromatina attività rimodellamento del complesso del INO80 rimangono poco conosciuti, in parte a causa della sfida di generare sottoinsiemi INO80 nelle cellule umane o sistemi di espressione eterologhi. Questo protocollo JOVE descrive una procedura che permette la purificazione di INO80 cromatina Sottocomplessi rimodellamento umani che mancano di una subunità o un sottoinsieme di subunità. N-terminale epitopo FLAG tagged INO80 cDNA vengono stabilmente introdotte nel rene embrionale umano (HEK) 293 linee cellulari con ricombinazione Flp-mediata. Nel caso in cui un sottoinsieme di subunità del complesso INO80 è abe cancellato, si esprime invece le proteine mutanti INO80 che non hanno la piattaforma necessaria per il montaggio di queste subunità. Nel caso in cui un individuo subunità deve essere impoverito, uno transfects di targeting siRNA questa subunità in una linea cellulare HEK 293 che esprimono stabilmente FLAG taggati INO80 ATPasi. Estratti nucleari sono preparate, e FLAG immunoprecipitazione viene effettuata per arricchire frazioni proteiche contenenti derivati INO80. Le composizioni di Sottocomplessi INO80 purificati possono poi essere analizzati utilizzando metodi quali immunoblotting, colorazione argento, e spettrometria di massa. I Sottocomplessi INO80 e INO80 generati in base a questo protocollo possono essere ulteriormente analizzati utilizzando vari saggi biochimici, che sono descritte nel protocollo JOVE accompagnamento. I metodi qui descritti possono essere adattati per lo studio delle proprietà strutturali e funzionali di ogni mammifero multi-subunità rimodellamento della cromatina e complessi modificano.
Introduzione
Evolutivamente conservata di famiglia SNF2 complessi di rimodellamento della cromatina sono regolatori chiave di organizzazione della cromatina e l'accessibilità del DNA 1. Questi complessi di rimodellamento comprendono sempre una centrale SNF2-come ATPasi subunità, che, in alcuni casi, assembla con diverse proteine accessorie e forma macro-molecolare assiemi multi-subunità. Per studiare i dettagli molecolari del processo di rimodellamento della cromatina ATP-dipendente, è importante comprendere i contributi di determinati sottoinsiemi di subunità e / o strutture di dominio per le attività dei complessi. Tali analisi richiedono la capacità di generare complessi mutanti altamente purificate che non hanno particolari subunità proteiche o strutture di dominio.
Precedenti studi struttura-funzione di ATP-dipendenti complessi di rimodellamento della cromatina sono ampiamente concentrata sul sistema modello di lievito a causa della manipolabilità superiore del genoma del lievito (vedi, per esempio, arbitri 1-4). Data la conservazione deglicomposizione subunità e funzionalità tra complessi di rimodellamento ortologhi, studi sulla struttura e la funzione dei lieviti complessi di rimodellamento hanno fornito importanti informazioni nelle loro controparti in eucarioti superiori. Tuttavia, esistono specie-specifici apprezzabili differenze tra i complessi di rimodellamento, derivante dalla plusvalenza o la perdita di specie-specifica subunità, il guadagno o la perdita di domini specie-specifici della subunità conservati, e la variabilità di sequenza all'interno di domini conservati di subunità conservati. Tali differenze possono in linea di principio essere guidati dalla necessità di cellule eucariotiche superiori di adattarsi a nuovi ambienti molecolari e cellulari. Quindi, capire come subunità di maggiori complessi di rimodellamento eucarioti contribuiscono al processo di rimodellamento nucleosoma è prezioso, perché getta luce non solo sui meccanismi di base del processo di rimodellamento della cromatina ATP-dipendente, ma anche in grado di fornire preziose informazioni sui meccanismi attraverso cui la struttura della cromatina e l'espressione genica in higsuoi eucarioti sono regolati.
Finora, ci sono stati solo limitati studi strutturali e funzionali di multi-subunità mammiferi complessi di rimodellamento della cromatina, in parte a causa delle difficoltà nell'ottenere biochimicamente definiti complessi di rimodellamento della cromatina e Sottocomplessi. Abbiamo parzialmente aggirato queste difficoltà con le procedure descritte qui di seguito, in cui la purificazione per immunoaffinità viene utilizzato per preparare intatti INO80 o INO80 Sottocomplessi da cellule umane che esprimono stabilmente N-terminale epitopo FLAG tag wild type o mutanti versioni di INO80 5-7 (Figura 1) . Per ottenere complessi INO80 intatte da cellule umane, ricombinazione Flp-mediata viene utilizzato per generare linee cellulari transgeniche HEK293 stabilmente esprimenti epitopi FLAG cDNA taggati codificano per le subunità del complesso INO80 8-10. Poiché sovra-espressione di subunità INO80 può essere alquanto tossico, è necessario isolare e mantenere linee cellulari clonali sotto co selettivalli operativi per garantire l'espressione stabile del transgene durante i numerosi passaggi necessari per l'espansione delle colture cellulari su larga scala. Per ottenere Sottocomplessi INO80 più piccoli che contengono solo un sottoinsieme di subunità, abbiamo utilizzato con successo due approcci (Figura 2A, B). Nel primo, generiamo linee cellulari HEK293 Flp-In esprimono stabilmente versioni mutanti di INO80 che mancano i domini necessari per l'interazione con le subunità specifiche 5. In alternativa, knockdown siRNA-mediata è utilizzato per esaurire la subunità desiderato da cellule che esprimono un adeguato INO80 subunità FLAG-tag (dati non pubblicati). Infine, per purificare i complessi INO80 umani, FLAG cromatografia a base di agarosio 11 viene utilizzato per arricchire una frazione-INO80 contenente da estratti nucleari, riducendo efficacemente la presenza di contaminanti proteine citosoliche nella frazione finale contenente depurati INO80 o INO80 Sottocomplessi.
Protocollo
1 Generazione e Cultura HEK293 linee cellulari stabili che esprimono versioni mutate di FLAG Figura intera o epitopi-tagged INO80 o altre subunità INO80 Complex
- Clone cDNA codificante intera lunghezza o mutante umano INO80 ATPasi o un'altra subunità INO80 nel vettore di espressione di mammifero pcDNA5 / FRT con un morsetto N tag epitopo FLAG in-frame.
- Confermare la sequenza dei cDNA inserito dal sequenziamento del DNA prima di procedere.
- Per eseguire la trasfezione, crescere Flp-In cellule HEK293 in 10 centimetri piastre di coltura dei tessuti in un mezzo contenente DMEM (Dulbecco Modified Eagle Medium), 5% glutammina, e il 10% FBS (siero fetale bovino).
- Quando le cellule raggiungono ~ 70% di confluenza, aggiungere a ciascun piatto di coltura tissutale da una miscela di 40 ml di reagente di trasfezione FuGENE6, 0,5 mcg della appropriata pcDNA5 / FRT plasmide di espressione, e 9,5 ug di pOG44, che codifica ricombinasi Flp, in un volume totale di 800 microlitri.
- 48 ore più tardi, trypsinize e scellule Plit in un rapporto di 1:30 in 10 piatti cm, e farle crescere in DMEM con il 5% glutammina, 10% FBS, e 100 mcg / ml igromicina B per 3 - 4 settimane. Modificare il terreno di coltura ogni volta che comincia a ingiallire (in genere ogni 3 - 5 giorni).
- Per identificare i cloni positivi che esprimono il più alto livello di proteine FLAG-tag, selezionare colonie igromicina individuale B-resistente e trasferirli su un unico pozzetto di una piastra da 24 pozzetti.
- Una volta che le cellule raggiungono l'80% di confluenza, le cellule raccolto da ciascun pozzetto in ~ 1 ml di PBS e pellet per centrifugazione a 1000 xg per 5 min.
- Dopo aver rimosso il surnatante, risospendere il pellet cellulare in 60 ml di tampone campione SDS-PAGE.
- Oggetto metà del pellet cellulare risospeso alla pagina SDS e western blotting per monitorare espressione della proteina FLAG tag esca; salvare l'altra metà per le analisi future.
- Se nessuna proteina INO80 umana FLAG-tag viene rilevato in lisati cellulari, espandere la popo iniziale cellulare clonalemento ulteriormente placcatura cellule provenienti da singoli pozzetti di una piastra da 24 pozzetti in 15 centimetri piastre di coltura tissutale.
- Per raccogliere le cellule da 15 piatti cm, risospendere nei pressi di cellule confluenti nel freddo ghiaccio PBS e trasferire in una provetta da 50 ml.
- Portare a volume finale di 50 ml con l'aggiunta di ulteriori PBS.
- Cellule pellet a 1.000 xg per 5 minuti, e rimuovere quanto più possibile del surnatante.
- Risospendere il pellet di cellule in 1 ml di tampone Lys450 (20 mM HEPES-NaOH pH 7,9, 450 mM NaCl, 0.5% TritonX-100, 10 mM KCl, 4 mM MgCl 2, 0.2 mM EDTA, 10% glicerolo, 1 mM DTT, 200 mM PMSF, e 1: 1.000 inibitore della proteasi Cocktail).
NOTA: qui e altrove, aggiungere sempre DTT, PMSF, e inibitore della proteasi cocktail di buffer immediatamente prima di iniziare un esperimento. - Immuno-precipitato proteine dalla risultante lisato cellulare intero FLAG-tagged utilizzando 20 l di anti-FLAG gel di agarosio, e analizzare gli eluati FLAG di western blotting. [Per i dettagli di immunoprecipitation procedura, vedere il passo 5]
- Preparare scorte congelate desiderato linee cellulari clonali 12 e conservarli in azoto liquido fino al momento dell'uso.
2. Growing HEK293 linee cellulari in bottiglie Roller
Per la preparazione di grandi dimensioni di complessi INO80, le cellule di coltura in 10-20 bottiglie rulli; una resa tipica da ogni bottiglia rullo è ~ 1 ml di emazie concentrate.
- Per ogni bottiglia rullo, aggiungere 200 ml di DMEM, 5% glutammina, e siero di vitello al 10%, senza che igromicina B.
- Trasferire tutte le celle da un'unica quasi confluenti piatto 15 centimetri in ciascun flacone rullo
- Bottiglie Posizionare rulli in un 37 ° rullo C bottiglia incubatore e ruotano a 0,2 rpm.
- Una volta che le cellule raggiungono ~ 70% di confluenza, versare fuori e scartare il medium.
- Aggiungere ~ 50 ml di ghiaccio freddo PBS per ogni bottiglia. Tenendo bottiglie in orizzontale, miscelare delicatamente per allentare il monostrato cellulare.
- Trasferire le cellule risospese a 250 ml plastbottiglie coniche ic e posto sul ghiaccio.
- Sciacquare bottiglie a rulli con l'aggiunta di PBS, trasferendolo in sequenza da bottiglia a bottiglia. Quando soluzione di risciacquo non è più chiaro (in genere dopo essere stato utilizzato per sciacquare circa 5 bottiglie), aggiungere alla sospensione cellulare.
- Cellule pellet per centrifugazione a 400 xg per 10 min.
- Risospendere delicatamente le cellule in PBS, combinarle in un unico flacone da 250 ml conica, e tenere in ghiaccio fino al procedimento.
3. siRNA-mediata Knockdown di INO80 Subunità nelle cellule che esprimono altro FLAG-tagged INO80 subunità
Per ottenere Sottocomplessi INO80 privi di una singola subunità, utilizzare il flag-immunopurification per purificare complessi INO80 dalle cellule trattate con siRNA o cellule che esprimono shRNA in modo stabile. Il siRNA "reverse" (breve RNA interferente) protocollo di trasfezione qui descritto è ottimizzato per HEK293 cellule in crescita in 15 piatti cm. Il protocollo è un piatto unico a 15 cm da cellule e shoULD rettificati di conseguenza a seconda del numero di cellule necessarie. Per preparare quantità biochimicamente utili di complesso INO80 dalle cellule trattate con siRNA, si dovrebbe scalare fino a colture coltivate in 40 15 piatti cm; questi produrranno circa 2 - 4 ml di pellet di ematocrito.
- Crescere cellule HEK293 che esprimono stabilmente la subunità desiderata del complesso INO80 vicino a confluenza in 15 piatti cm.
- Aggiungere un volume di tampone di risospensione siRNA (20 mM KCl, 6 mM HEPES-pH 7.5, e 0.2 mM MgCl 2) sufficiente per preparare una soluzione stock 50 mM di siRNA in una provetta contenente liofilizzato siRNA. Pipettare la soluzione su e giù un paio di volte e incubare su un nutator per 30 minuti a temperatura ambiente per garantire il siRNA è completamente sciolto.
- Preparare un cocktail trasfezione contenente siRNA e reagente di trasfezione. Miscelare 10 ml di soluzione madre 50 mM siRNA con 32 microlitri Lipofectamine RNAiMAX e aggiungerlo a 4 ml di Opti-MEM ridotto Siero medio con molto delicata miscelazione; albassi tutti i reagenti raggiungano la RT prima dell'uso.
- Incubare la miscela a temperatura ambiente per 30 min.
- Preparare le cellule Flp-In HEK293 che esprimono stabilmente la subunità INO80 desiderato per trasfezione.
- Durante l'incubazione di Fase 3.4, lavare le cellule nei piatti cm 15 una volta con PBS RT.
- Dopo aver rimosso PBS, trattare le cellule con 1 ml di tripsina solo fino a quando non iniziano a sollevare la piastra.
- Aggiungere immediatamente 10 ml di mezzo completo (DMEM + 5% glutammina + 10% FBS) per tripsinizzati cellule e mescolare delicatamente. Raccogliere le cellule per centrifugazione a 1000 xg per 5 minuti a temperatura ambiente.
- Risospendere il pellet di cellule in ~ 4 ml di terreno completo, e contare il risospensione delle cellule utilizzando un emocitometro.
- Diluire le cellule con mezzo completo ad una concentrazione di ~ 5,4 x 10 6 / ml.
- Per ogni piatto 15 centimetri, aggiungere 15 ml di terreno di coltura completo seguito da 4 ml trasfezione cocktail. Agitare delicatamente per garantire il cocktail medio e trasfezione sono Thoroughly misto. Infine, aggiungere un ml di sospensione cellulare e di nuovo agitare delicatamente per disperdere le cellule in modo uniforme.
- Dopo 60 ore di coltura a 37 ° C, 5% CO 2 incubatore, rimuovere delicatamente medio, risospendere le cellule in PBS freddo ghiaccio, e procedere immediatamente a preparare estratti nucleari.
4 Preparazione di estratti nucleari
Questa procedura è stata modificata dal protocollo di Dignam 13 e può essere scalata verso l'alto o verso il basso a seconda delle dimensioni di iniziare pellet cellulari. Tipicamente, 1 ml di imballaggio rendimenti pellet cellulare 1 ml di estratto nucleare finale. Tutti i tamponi devono essere ghiacciata, e tutte le misure devono essere eseguite in una stanza fredda o ghiaccio, se una stanza fredda appropriato non sia disponibile.
- Isolamento dei nuclei:
- Trasferire delicatamente le cellule ad una di dimensioni adeguate (15 o 50 ml) si è laureato tubo conico e far girare a 1000 xg per 10 min a 4 ° C.
- Rimuovere il surnatante, e misurare il volume della pel ematocritolasciare. 1 ml di emazie concentrate corrisponde a ~ 3 x 10 8 HEK293 cellule.
- Aggiungere 5 volumi imballati cellulari di tampone A (HEPES 10 mM, pH 7,9, 1,5 mM MgCl 2, KCl 10 mM e appena aggiunto 1 mM DTT, 200 mM PMSF, e 1: 1.000 inibitore della proteasi Cocktail). Risospendere il pellet di cellule delicatamente pipettaggio.
- Incubare in ghiaccio per esattamente 10 minuti.
- Agglomerare le cellule a 1.000 xg per 10 min a 4 ° C, e rimuovere il surnatante.
- Le dimensioni del pellet cellulare dovrebbe aumentare fino a 2 volte durante l'incubazione in tampone A.
- Risospendere le cellule in due volumi di ematocrito di tampone A, e trasferire la sospensione cellulare a una di dimensioni adeguate omogeneizzatore tessuto Dounce. Se inizia con meno di 2 ml di emazie concentrate, utilizzare un omogeneizzatore 7 ml; per 2 - 4 ml cellule imballato, utilizzare un omogeneizzatore 15 ml; e per 10 o più ml di emazie concentrate, utilizzare un omogeneizzatore 40 ml.
- Omogeneizzare la sospensione cellulare con il pestello di vetro libera del omogeneizzatore ONU Douncetil 90% delle cellule macchiare positivamente con 1% trypan blu.
- Trasferire la sospensione di un 45 ml ad alta velocità provetta da centrifuga e centrifugare a 25.000 xg per 20 min a 4 ° C in un rotore JA-17 o simili (vedi Tabella dei materiali / attrezzature).
- Dal pellet nucleare, rimuovere il surnatante, che contiene proteine citosoliche o proteine che fuoriescono dal nucleo durante il frazionamento.
- Estrazione nuclei con sale:
- Aggiungi Buffer C (20 mM HEPES, pH 7,9, 25% glicerolo, 1,5 mM MgCl 2, 0.2 mM EDTA, e appena aggiunto 1 mM DTT, 200 mM PMSF, e 1: 1.000 inibitore della proteasi Cocktail) al pellet nucleare; utilizzare 2,5 ml di tampone C per ogni 3 ml di iniziare ematocrito (~ 1 x 10 9 cellule).
- Utilizzando una bacchetta di vetro o una pipetta, rimuovere il pellet nucleare dalla parete del tubo e trasferire l'intera miscela di un omogeneizzatore Dounce di dimensioni adeguate.
- Risospendere i nuclei omogeneizzando the miscela con due colpi di un pestello LOOSE.
- Trasferire la frazione nucleare risospeso in un bicchiere ghiacciato. Scegli un bicchiere in modo che la sospensione si riempirà il bicchiere di almeno 0,5 cm di profondità.
- Per estrarre proteine nucleari da cromatina o altre strutture insolubili, aumentare gradualmente la concentrazione salina della sospensione di 0,42 M di NaCl da gocciolamento aggiunta di 5 M NaCl agitando moderatamente la sospensione con una bacchetta di vetro pipetta o su ghiaccio; una volta tutte le 5 M NaCl è stato aggiunto, la soluzione dovrebbe diventare molto viscoso o gel-like. Calcolare il volume di 5 M NaCl necessaria per portare la soluzione ad una concentrazione finale di 0,42 M NaCl secondo la seguente formula:
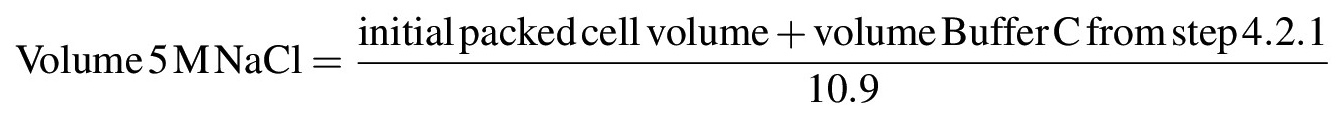
- Trasferire accuratamente la sospensione viscoso in 10 ml tubi in policarbonato per un tipo 70.1 Ti rotore (o equivalente) bottiglie in policarbonato o 70 ml per una Ti o Tipo 45r rotore simile (vedi Tabella dei materiali / attrezzature). Sigillare ermeticamente con parafilm se utilizzando 10 ml provette o con l'assemblea del tappo se si utilizzano 70 ml bottiglie.
- Lentamente ruotare i tubi sigillati a 4 ° C per 30 minuti usando un nutator.
- Spin i campioni in un tipo 45 Ti o 70,1 Ti rotore per 30 min a 120.000 xg a 4 ° C.
- Trasferire il surnatante in un unico tubo di plastica o una bottiglia. Questo surnatante è l'estratto nucleare, e il pellet contiene cromatina e altri detriti nucleare.
- Dividere l'estratto nucleare in aliquote di dimensioni convenientemente, congelare in azoto liquido, e conservarlo a -80 ° C.
5. Immunoaffinity Purificazione della INO80 Umano o INO80 Sottocomplessi
- Per scongelare estratto nucleare congelato, tubi luogo contenenti l'estratto sui tubi da banco o rotolo tra le mani fino a quando il materiale ghiacciato diventa un impasto. Poi posto le provette in ghiaccio o in camera fredda fino a quando l'estratto è completamente thawed.
- Trasferire l'estratto nucleare scongelati per tubi in policarbonato ultracentrifuga 10 ml, e far girare a 100.000 xg per 20 min a 4 ° C in un rotore di Ti o equivalente Tipo 70,1 per rimuovere l'eventuale precipitato che possono essersi formate durante il ciclo di congelamento-scongelamento.
- Trasferire il surnatante in una provetta da 15 ml. Aggiungere fresco DTT, PMSF, e inibitore della proteasi Cocktail a concentrazioni finali di 1 mM DTT, 200 mM PMSF, e 1: 1.000 inibitore della proteasi Cocktail.
- Per preparare anti-FLAG agarosio per la immunopurification, trasferire 200 ml di 50% impasto di anti-FLAG agarosio perline in una provetta da 1,5 ml con una pipetta P200 o simile con una punta da cui il termine è stato tagliato con un bisturi pulito o lama di rasoio.
- Pellet le perle di centrifugazione in una microcentrifuga da banco a 8.000 xg per 30 sec. Rimuovere il surnatante e lavare i beads di risospendere le sfere in 1 ml di tampone Lys450, pellet e le perline a 8.000 xg per 30 sec. Lavare il bEADS altre due volte.
- Risospendere le anti-FLAG perline agarosio lavati in circa 100 ml di estratto nucleare utilizzando un P200 o simili pipetta volume regolabile con una punta da cui il termine è stato tagliato con un bisturi pulito o lama di rasoio e, utilizzando la stessa punta, il trasferimento le microsfere in sospensione al tubo da 15 ml contenente l'estratto. Ripetere un paio di volte fino a quando tutte le perline sono stati trasferiti al tubo 15 ml.
- Incubare la miscela di estratto / tallone per 4 ore a 4 ° C con rotazione lenta su un rotatore laboratorio. OPTIONAL: Includi benzonasi ad una concentrazione di 25 unità / ml per rimuovere contaminazione del DNA.
- Raccogliere il FLAG agarosio mediante centrifugazione a 1000 xg per 5 minuti a 4 ° C.
- Risospendere in 10 ml Lys450 per lavare, incubare 5 minuti a 4 ° C con dolce dondolo su un nutator. Pellet le perline a 1.000 xg per 5 minuti a 4 ° C.
- Risospendere in 100 - 150 ml di Lys450 e perle trasferimento in una provetta da 1,5 ml microcentrifuga. Continue per sciacquare il tubo da 15 ml con 100 - 150 ml di Lys450 fino a quando tutte le perle sono stati trasferiti alla provetta.
- Centrifugare le perline a 8.000 xg per 30 sec a 4 ° C in una microcentrifuga. Lavare tre volte con 1 ml di Lys450 e una volta con tampone 1ml EB100 (10 mM HEPES pH 7,9, 10% glicerolo, NaCl 100 mM, 1,5 mM MgCl 2, 0.05% TritonX-100, e appena aggiunto 1 mM DTT, 200 mM PMSF , e 1: 1.000 inibitore della proteasi Cocktail).
- Per eluire le proteine legate, aggiungere tampone EB100 200 ml contenente 0,25 mg / ml 1x peptide FLAG. Incubare 30 min a 4 ° C ciascuno su una nutator.
- Agglomerare le perline a 8.000 xg per 30 sec a 4 ° C in una microcentrifuga. Trasferire il surnatante, che contiene il complesso INO80 eluito, in una provetta da microcentrifuga fresca.
- Ripetere l'eluizione altre quattro volte, e riunire tutti i surnatanti in un unico tubo.
- Per rimuovere eventuali residui di perline FLAG-agarosio dalla frazione proteica eluita,passare l'eluato attraverso una colonna di spin vuoto.
- Concentrare la frazione proteica eluita ~ 10 volte utilizzando un dispositivo di filtro centrifugo ultra (50.000 molecolare cutoff peso).
- Per rimuovere il peptide FLAG, passare il concentrato frazione proteica in sequenza attraverso due colonne dissalazione.
- Dividere la frazione proteica purificata, dissalato in 20 microlitri aliquote, congelati in azoto liquido e conservati a -80 ° C.
- Analizzare la composizione subunità di INO80 o Sottocomplessi INO80 su gel-argento macchiato o mediante western blotting, e stimare la loro concentrazione mediante western blotting semi-quantitativa utilizzando preparazioni di subunità INO80 ricombinanti di concentrazione nota come standard.
Risultati
La figura 1 mostra un diagramma di flusso che riassume le procedure utilizzate per generare, purificarsi, e caratterizzare INO80 ATP-dipendenti complessi di rimodellamento della cromatina umani.
Secondo quanto illustrato nelle figure 2 e 3, tali procedure consentono la generazione di entrambe le tipo INO80 e INO80 Sottocomplessi selvatici che mancano varie subunità, consentendo così successive analisi biochimiche del contributo di tali sub...
Discussione
Studi strutturali e funzionali di multi-subunità complessi di rimodellamento della cromatina mammiferi da eucarioti superiori sono stati ostacolati dalla difficoltà di preparare biochimicamente quantità utili di tali complessi contenenti subunità mutante o privi di alcune subunità del tutto. Ci sono una serie di ostacoli tecnici: In primo luogo, la manipolazione genetica in cellule di mammifero è stato tecnicamente impegnativo e richiede tempo. A differenza delle cellule di lievito, il cui genoma può essere facil...
Divulgazioni
Gli autori dichiarano di non avere interessi finanziari in competizione.
Riconoscimenti
Lavoro in laboratorio degli autori è supportata da una sovvenzione da parte del National Institute of scienze mediche generali (GM41628) e da una sovvenzione all'Istituto Stowers per la ricerca medica da Helen Nelson Medical Research Fund presso la Greater Kansas City Community Foundation.
Materiali
| Name | Company | Catalog Number | Comments |
| Name of Reagent/Material | Company | Catalog Number | Comments |
| Dulbecco's Modified Eagle Medium | Cellgro | 10-013-CV | |
| Glutamax-I (stablized glutamine) | Life Technologies | 35050-079 | |
| Fetal Bovine Serum | SAFC | 12176C | |
| FuGENE6 transfection reagent | Promega | E2312 | |
| Hygromycin B, sterile in PBS | AG Scientific | H-1012-PBS | |
| pcDNA5/FRT vector | Life Technologies | V6010-20 | |
| Flp-In HEK293 cells | Life Technologies | R780-07 | |
| pOG44 Flp-Recombinase Expression Vector | Life Technologies | V600520 | |
| EZview Red ANTI-FLAG M2 Affinity Gel | Sigma | F2426 | |
| calf serum | SAFC | 12138C | |
| TARGETplus SMARTsiRNA pool | Dharmacon / Thermo Scientific | various | |
| 5x siRNA resuspension buffer | Dharmacon / Thermo Scientific | #B-002000-UB-100 | |
| Lipofectamine RNAiMAX | Life Technologies | 13778 | |
| Opti-MEM Reduced Serum Medium | Life Technologies | 51985-091 | |
| PBS | Cellgro | 45000 | VWR |
| TrypLE (trypsin) | Life Technologies | 12604 | |
| 1x FLAG Peptide | Sigma | F3290 | |
| Micro Bio-Spin Chromatography Column | Bio-Rad | 737-5021 | |
| Amicon Ultra Centrifugal Filter Device (50k MWCO) | Amicon | UFC805024 | Fisher Scientific |
| Zeba Desalting Columns | Thermo Scientific | 89882 | |
| Anti-FLAG M2 antibody, mouse | Sigma | F3165 | |
| Anti-FLAG M2 antibody, rabbit | Sigma | F7425 | |
| Protease Inhibitor Cocktail | Sigma | P8340 | |
| benzonase | Novagen | 70664 | |
| Equipment | Company | ||
| Wheaton Dounce Tissue Grinders | Wheaton | 06-435C | |
| JS-4.2 rotor in a J6 centrifuge | Beckman-Coulter | 339080 | |
| JA-17 rotor | Beckman-Coulter | 369691 | |
| 10 ml polycarbonate tubes | Beckman-Coulter | 355630 | |
| 70 ml polycarbonate bottles | Beckman-Coulter | 355655 | |
| Type 45 Ti rotor | Beckman-Coulter | 339160 | |
| Type 70.1 Ti rotor | Beckman-Coulter | 342184 | |
| BD Clay Adams Nutator Mixer | BD Diagnostics | 15172-203 | VWR |
| Glas-Col Tube/Vial Rotator | Glas-Col | 099A RD4512 | |
| PCR thermal cycler PTC 200 | MJ Research | PTC 200 | |
| roller bottle incubator | Bellco biotechnology | 353348 | |
| Immobilon-FL Transfer Membrane 7 x 8.4 | Millipore | IPFL07810 | |
| lubricated 1.5ml microcentrifuge tubes | Costar | 3207 |
Riferimenti
- Clapier, C. R., Cairns, B. R. The biology of chromatin remodeling complexes, Annual Review of Biochemistry. 78, 273-304 (2009).
- Szerlong, H., Hinada, K., Viswanathan, R., Erdjument-Bromage, H., Tempst, P., Cairns, B. R. The HSA domain binds nuclear actin-related proteins to regulate chromatin-remodeling ATPases. Nature Struct. Mol. Biol. 15 (5), 469-476 (2008).
- Shen, X., Mizuguchi, G., Hamiche, A., Wu, C. A chromatin remodelling complex involved in transcription and DNA processing. Nature. 406 (6795), 541-544 (2000).
- Shen, X., Ranallo, R., Choi, E., Wu, C. Involvement of actin-related proteins in ATP-dependent chromatin remodelling. Mol. Cell. 12, 147-155 (2003).
- Chen, L., et al. Subunit organization of the human INO80 chromatin remodeling complex: an evolutionarily conserved core complex catalyzes ATP-dependent nucleosome remodeling. Journal of Biological Chemistry. 286 (13), 11283-11289 (2011).
- Sauer, B. Site-specific recombination: developments and applications. 5 (5), 521-527 (1994).
- Gorman, S., Fox, D. T., Wahl, G. M. Recombinase-mediated gene activation and site-specific integration in mammalian cells. Science. 251 (4999), 1351-1355 (1991).
- Jin, J., et al. A mammalian chromatin remodeling complex with similarities to the yeast INO80 complex. Journal of Biological Chemistry. 280 (50), 41207-41212 (2005).
- Cai, Y., et al. YY1 functions with INO80 to activate transcription. Nature Structural and Molecular Biology. 14 (9), 872-874 (2007).
- Yao, T., et al. Distinct Modes of Regulation of the Uch37 Deubiquitinating Enzyme in the Proteasome and in the Ino80 Chromatin-Remodeling Complex. Mol.Cell. 31 (6), 909-917 (2008).
- Brizzard, B. L., Chubet, R. G., Vizard, D. L. Immunoaffinity purification of FLAG epitope-tagged bacterial alkaline phosphatase using a novel monoclonal antibody and peptide elution, Biotechniques. 16 (4), 730-735 (1994).
- Barker, K. . At the Bench: A Laboratory Navigator. 10, 207-245 (2005).
- Dignam, J. D., Lebovitz, R. M., Roeder, R. G. Accurate transcription initiation by RNA polymerase II in a soluble extract from isolated mammalian cell nuclei. Nucleic. Acids. Res. 11 (5), 1475-1489 (1983).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon