Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Production et purification des droits de INO80 remodelage de la chromatine Complexes et Subcomplexes
Dans cet article
Résumé
Ce protocole décrit un procédé pour la production et la purification de type sauvage et les versions mutantes du complexe de remodelage de la chromatine humaine INO80. Marquage épitopique versions de sous-unités INO80 sont exprimés de manière stable dans des cellules HEK293, et les complexes et les complexes dépourvus complets des ensembles spécifiques de sous-unités sont purifiés par chromatographie d'immuno-affinité.
Résumé
INO80 complexes de remodelage réguler la dynamique du nucléosome et l'accessibilité de l'ADN en catalysant ATP-dépendant nucléosome remodelage. Complexes INO80 humaines se composent de 14 sous-unités protéiques dont Ino80, une ATPase SNF2 comme, qui sert à la fois de la sous-unité catalytique et l'échafaud pour l'assemblage des complexes. Fonctions des autres sous-unités et les mécanismes par lesquels ils contribuent à l'activité de remodelage de la chromatine du complexe INO80 restent mal connus, en partie en raison de la difficulté de générer des sous-ensembles de INO80 dans les cellules humaines ou systèmes d'expression hétérologues. Ce protocole de JOVE décrit une procédure qui permet la purification de INO80 chromatine Subcomplexes de remodelage humaines qui font défaut une sous-unité ou un sous-ensemble de sous-unités. N-terminale de l'épitope FLAG Ino80 ADNc marqués de manière stable sont introduits dans le rein embryonnaire humain (HEK) 293 lignes de cellules en utilisant la recombinaison médiée par Flp. Dans le cas où un sous-ensemble des sous-unités du complexe à b est INO80e supprimé, on exprime des protéines mutantes INO80 place qui n'ont pas la plate-forme nécessaire pour l'assemblage de ces sous-unités. Dans le cas d'une sous-unité individuelle doit être appauvri, un transfecte siRNA ciblant cette sous-unité dans une ligne HEK 293 cellules exprimant de façon stable FLAG marqués Ino80 ATPase. Des extraits nucléaires sont préparés, et FLAG immumoprécipitées d'enrichir fractions protéiques contenant des dérivés de INO80. Les compositions de sous-complexes de INO80 purifiés peuvent ensuite être analysées en utilisant des méthodes telles que immunoblot, coloration à l'argent, et la spectrométrie de masse. Les Subcomplexes INO80 et INO80 générés selon ce protocole peuvent être analysées en utilisant différents dosages biochimiques, qui sont décrites dans le protocole de JOVE d'accompagnement. Les méthodes décrites ici peuvent être adaptées pour l'étude des propriétés structurales et fonctionnelles de tout sous-unités multiples remodelage de la chromatine chez les mammifères et les complexes de modification.
Introduction
Complexes de remodelage de la famille SNF2 évolutives conservées sont des régulateurs clés de l'organisation de la chromatine et de l'ADN accessibilité 1. Ces complexes de remodelage comprennent toujours une sous-unité ATPase de SNF2 central en forme, ce qui, dans certains cas, les assemble avec différents accessoires et des protéines formant des ensembles de macro-moléculaire sous-unités multiples. Pour étudier les détails moléculaires des processus de remodelage de la chromatine dépendant de l'ATP, il est important de comprendre les contributions des sous-ensembles de données de sous-unités et / ou des structures de domaine pour les activités des complexes. Ces analyses doivent être capables de générer des complexes mutants hautement purifiés qui manquent particulier sous-unités protéiques ou des structures de domaine.
Des études antérieures structure-fonction de complexes de remodelage de la chromatine dépendant de l'ATP ont largement centrée sur le système de levure modèle en raison de la manipulabilité supérieure du génome de la levure (voir, par exemple, 1-4) refs. Compte tenu de la conservation decomposition de la sous-unité et la fonctionnalité entre complexes de remodelage orthologues, des études de la structure et la fonction des complexes de remodelage de levure ont fourni des informations importantes sur leurs homologues chez les eucaryotes supérieurs. Néanmoins, il n'existe spécifiques aux espèces sensibles différences entre les complexes de remodelage, résultant de gain ou de perte de sous-unités spécifiques de l'espèce, le gain ou la perte de domaines spécifiques à l'espèce de sous-unités conservées, et la variabilité de séquence dans des domaines conservés de sous-unités conservées. Ces différences ne peuvent en principe être motivées par la nécessité pour les cellules eucaryotes supérieures à s'adapter à de nouveaux environnements moléculaires et cellulaires. Ainsi, comprendre comment les sous-unités de complexes de remodelage eucaryotes supérieurs contribuent au processus de remodelage du nucléosome est précieux, non seulement parce qu'il met en lumière les mécanismes de base du processus de remodelage de la chromatine dépendant de l'ATP, mais peut également fournir de précieuses informations sur les mécanismes par lesquels la structure de la chromatine et l'expression des gènes dans higses eucaryotes sont réglementés.
Jusqu'à présent, il ya eu seulement limité études structurales et fonctionnelles de plusieurs sous-unités de mammifères complexes de remodelage, en partie en raison des difficultés à obtenir des complexes de remodelage de la chromatine biochimiquement définies et Subcomplexes. Nous avons partiellement contourné ces difficultés avec les procédures décrites ci-dessous, dans lequel la purification par immuno-affinité est utilisé pour préparer intactes INO80 ou INO80 Subcomplexes de cellules humaines exprimant de façon stable N-terminale épitope FLAG étiqueté de type sauvage ou des versions mutantes de Ino80 5-7 (Figure 1) . Pour obtenir des complexes de INO80 intactes à partir de cellules humaines, la recombinaison médiée par Flp est utilisée pour générer des lignées de cellules HEK293 exprimant de manière stable transgéniques épitope FLAG ADNc codant pour des sous-unités de la catégorie du complexe INO80 8-10. Étant donné que la surexpression de sous-unités de INO80 peut être quelque peu toxique, il est nécessaire d'isoler et de conserver les lignées cellulaires clonales de moins de co sélectifnditions pour assurer l'expression stable du transgène au cours des nombreux passages nécessaires à l'expansion de cultures cellulaires à grande échelle. Pour obtenir Subcomplexes de INO80 petits qui contiennent un sous-ensemble de sous-unités, nous avons utilisé avec succès deux approches (Figure 2A, B). Dans la première, on génère des lignées cellulaires HEK293 Flp-In exprimant de manière stable les versions mutantes de Ino80 domaines qui n'ont pas requis pour l'interaction avec les sous-unités spécifiques 5. Sinon, knockdown siARN médiation est utilisée pour épuiser la sous-unité souhaitée à partir de cellules exprimant une sous-unité de INO80 étiquetée FLAG approprié (données non publiées). Enfin, pour purifier les complexes INO80 humaines, FLAG chromatographie à base d'agarose 11 est utilisé pour enrichir une fraction contenant INO80-à partir d'extraits nucléaires, réduisant ainsi efficacement la présence de protéines contaminantes cytosoliques dans la fraction finale contenant INO80 ou INO80 Subcomplexes purifiés.
Protocole
1. génération et de la culture de lignées cellulaires HEK293 stable exprimant Cadrage en pied ou versions mutantes de épitope FLAG-étiqueté Ino80 ou autres sous-unités INO80 complexes
- Clone ADNc codant pleine longueur ou mutant humain Ino80 ATPase ou d'une autre sous-unité INO80 dans le vecteur d'expression mammifère pcDNA5 / FRT avec un, N-terminal épitope FLAG tag dans le cadre.
- Confirmer la séquence des ADNc insérés par séquençage de l'ADN avant de poursuivre.
- Pour effectuer la transfection, Flp-In croître des cellules HEK293 dans des plats de 10 cm pour culture de tissus dans un milieu contenant du DMEM (milieu de Eagle modifié par Dulbecco), 5% de glutamine, et 10% de FBS (sérum fœtal bovin).
- Lorsque les cellules atteignent environ 70% de confluence, ajouter à chaque boîte de culture de tissu d'un mélange de 40 ul de FuGENE6 réactif de transfection, 0,5 pg du pcDNA5 / FRT plasmide d'expression approprié, et 9,5 pg de pOG44, qui code pour une recombinase Flp, dans un volume total de 800 ul.
- 48 h plus tard, et s trypsiniserPLIT cellules dans un rapport de 1:30 en boîtes de 10 cm, et les cultiver dans du DMEM avec 5% de glutamine, 10% de FBS, et 100 pg / ml d'hygromycine B pour 3 - 4 semaines. Changer le milieu de culture quand il commence à jaunir (généralement tous les 3 - 5 jours).
- Pour identifier les clones positifs qui expriment le plus haut niveau de la protéine étiquetée FLAG, sélectionnez colonies hygromycine individu résistant B-et les transférer sur un seul puits d'une plaque de 24 puits.
- Une fois que les cellules atteignent 80% de confluence, les cellules de prélèvement de chaque puits à ~ 1 ml de PBS et culot par centrifugation à 1000 xg pendant 5 min.
- Après élimination du surnageant, remettre en suspension le culot cellulaire dans 60 pi de tampon d'échantillon SDS-PAGE.
- Sous réserve de la moitié du culot cellulaire remis en suspension à la page de SDS et transfert de Western pour surveiller l'expression de la protéine étiquetée FLAG d'appât; sauver l'autre moitié pour les analyses futures.
- Si aucune protéine Ino80 humaine étiquetée FLAG est détectée dans des lysats cellulaires, étendre la première popu cellulaire clonalelation de plus par étalement des cellules provenant de puits individuels d'une plaque de 24 puits dans 15 cm des boîtes de culture de tissus.
- Pour récolter des cellules de 15 cm plats, remettre en suspension près de cellules confluentes dans le froid de la glace PBS et transférer dans un tube conique de 50 ml.
- Porter à un volume final de 50 ml par addition supplémentaire de PBS.
- Cellules à granules à 1000 g pendant 5 min, et supprimer autant que possible de le surnageant.
- Remettre en suspension le culot cellulaire dans 1 ml de tampon Lys450 (20 mM de HEPES-NaOH à pH 7,9, NaCl 450, 0,5% de Triton X-100, 10 mM de KCl, 4 mM de MgCl2, EDTA 0,2 mM, glycerol à 10%, DTT 1 mM, 200 uM PMSF et 1: 1000 Inhibiteur de la protéase Cocktail).
NOTE: ici et d'ailleurs, toujours ajouter TNT, PMSF, et inhibiteur de la protéase cocktail de tampons immédiatement avant de commencer une expérience. - Protéines du lysat de cellules entières résultant immuno-précipité FLAG-balisé au moyen de 20 pi de gel d'agarose anti-FLAG, et d'analyser les éluats FLAG par western blot. [Pour plus de détails immunprocédure de oprecipitation, voir l'étape 5]
- Préparer stocks congelés de lignées de cellules clonales souhaités 12 et rangez-les dans de l'azote liquide jusqu'à utilisation.
2. croissance des lignées cellulaires HEK293 dans des flacons roulants
Pour une préparation à grande échelle de complexes INO80, dans des cellules de culture de 10 à 20 flacons roulants; un rendement typique de chaque bouteille de rouleau est ~ 1 ml de cellules tassées.
- Pour chaque bouteille à rouleaux, ajouter 200 ml de DMEM, 5% de glutamine, et 10% de sérum de veau, sans hygromycine B.
- Transférer toutes les cellules d'un seul près de confluence 15 cm plat dans chaque bouteille de rouleau
- Placer les bouteilles de rouleaux dans un 37 ° C rouleau bouteille incubateur et tournent à 0,2 rpm.
- Une fois que les cellules atteignent ~ 70% de confluence, versez-la et jetez le milieu.
- Ajouter ~ 50 ml de PBS glacé à chaque bouteille. Soutenir des bouteilles horizontalement, agiter doucement pour desserrer la monocouche cellulaire.
- Transférer les cellules remises en suspension à 250 ml plastbouteilles coniques ic et placer sur la glace.
- Rincer les flacons roulants avec plus PBS, transférer séquentiellement d'une bouteille à. Lorsque la solution de rinçage est plus claire (généralement après avoir été utilisée pour le rinçage environ 5 bouteilles), ajouter à la suspension de cellules.
- Cellules granulés par centrifugation à 400 g pendant 10 min.
- Resuspendre doucement les cellules dans du PBS, les combiner en une seule bouteille de 250 ml conique, et garder sur la glace jusqu'à ce que le traitement ultérieur.
3 siRNA médiation knockdown de INO80 sous-unités dans les cellules exprimant autre étiquetée FLAG INO80 sous-unité
Pour obtenir Subcomplexes INO80 qui n'ont pas une seule unité, l'utilisation FLAG-immunopurification pour purifier des complexes INO80 à partir de cellules ou de cellules siRNA traités exprimant de façon stable shRNA. Le siRNA "reverse" (petits ARN interférents) protocole de transfection décrit ici est optimisé pour les cellules HEK293 qui poussent dans 15 cm plats. Le protocole est pour un seul 15 cm plat de cellules et shoULD être mis à l'échelle en conséquence, selon le nombre de cellules nécessaires. Pour préparer des quantités utiles de biochimiquement complexe INO80 à partir de cellules de siARN traité, il faut intensifier les cultures cultivées dans 40 15 cm plats; ceux-ci donneront environ 2 - 4 ml de culot globulaire.
- Cultiver les cellules HEK293 exprimant de façon stable le sous-unité du complexe désiré INO80 à proximité de confluence dans des boîtes de 15 cm.
- Ajouter un volume de tampon de remise en suspension de siRNA (20 mM de KCl, 6 mM HEPES, pH 7,5, et 0,2 mM de MgCl2), suffisante pour préparer une solution mère de 50 pM de siRNA à un tube lyophilisé contenant des siRNA. Introduire à la pipette la solution de haut en bas plusieurs fois et incuber sur un nutator pendant 30 min à température ambiante pour assurer le siRNA est entièrement dissous.
- Préparer un cocktail de transfection contenant siRNA et réactif de transfection. Mélanger 10 ul de la solution mère de 50 uM de siRNA avec 32 ul Lipofectamine RNAiMAX et ajouter 4 ml d'Opti-MEM milieu sérique réduit avec un mélange très doux; albas tous les réactifs s'équilibrent à la température ambiante avant de l'utiliser.
- Incuber le mélange à température ambiante pendant 30 min.
- Préparation des cellules Flp-In HEK293 exprimant de façon stable le sous-unité INO80 souhaitée pour la transfection.
- Au cours de l'incubation de l'étape 3.4, laver les cellules dans les plats 15 cm une fois avec RT PBS.
- Après avoir retiré PBS, traiter les cellules avec 1 ml de trypsine juste jusqu'à ce qu'ils commencent à se décoller de la plaque.
- Ajouter immédiatement 10 ml de milieu complet (DMEM + 5% de glutamine + 10% de FBS) à cellules trypsinisées et mélanger doucement. Recueillir les cellules par centrifugation à 1000 g pendant 5 min à température ambiante.
- Remettre en suspension le culot cellulaire dans ~ 4 ml de milieu complet, et compter la remise en suspension des cellules en utilisant un hématimètre.
- Diluer les cellules avec du milieu complet à une concentration de 5,4 x 10 ~ 6 / ml.
- Pour chaque 15 cm plat, ajouter 15 ml de milieu de culture complet suivi par 4 ml de cocktail de transfection. Agiter doucement pour assurer le support et la transfection cocktail sont thoroughly mélangé. Enfin, ajouter un ml de suspension cellulaire et de nouveau agiter doucement pour disperser uniformément les cellules.
- Après 60 h de culture dans un 37 ° C, 5% de CO 2 incubateur, retirez délicatement moyen, remettre les cellules en PBS glacé, et procéder immédiatement à préparer des extraits nucléaires.
4 Préparation des extraits nucléaires
Cette procédure a été modifiée par le protocole de Dignam 13 et peut être mise à l'échelle vers le haut ou vers le bas selon la taille de départ culots cellulaires. Typiquement, 1 ml de rendements en emballage de pastilles de cellule 1 ml d'extrait nucléaire final. Tous les tampons doivent être froid glace, et toutes les mesures doivent être effectuées dans une chambre froide ou sur de la glace d'une chambre froide approprié n'est pas disponible.
- Isolement des noyaux:
- Transférer en douceur les cellules à une taille appropriée (15 ou 50 ml) tube gradué et de spin conique à 1000 g pendant 10 min à 4 ° C.
- Enlever le surnageant et mesurer le volume du pixel de l'hématocritelaisser. 1 ml de cellules tassées correspond à ~ 3 x 10 8 cellules HEK293.
- Ajouter 5 volumes conditionnés de cellules de tampon A (10 mM d'HEPES, pH 7,9, 1,5 mM de MgCl2, 10 mM de KCl et fraîchement ajouté 1 mM de DTT, 200 uM de PMSF et 1: 1000 cocktail inhibiteur de la protéase). Remettre en suspension le culot de cellules par pipetage doux.
- Incuber sur de la glace pendant exactement 10 minutes.
- Pellet les cellules à 1000 g pendant 10 min à 4 ° C, et retirer le surnageant.
- La taille du culot cellulaire devrait augmenter jusqu'à deux fois au cours de l'incubation dans le tampon A.
- Remettre en suspension les cellules dans deux volumes de cellules tassées de tampon A, et transférer la suspension cellulaire à un homogénéisateur de taille appropriée du tissu Dounce. Si à partir de moins de 2 ml de cellules sont emballées, un homogénéisateur 7 ml; pour 2 - 4 ml cellules sont emballées, 15 ml d'homogénéisation; et pour plus de 10 ml de cellules sont emballées, 40 ml d'homogénéisation.
- Homogénéiser la suspension cellulaire avec le pilon en verre LOOSE de l'homogénéisation non Douncejusqu'à 90% des cellules colorer positivement avec 1% de bleu de trypan.
- Transférer la suspension dans un tube de centrifugeuse à grande vitesse 45 ml et centrifuger à 25 000 g pendant 20 min à 4 ° C dans un rotor JA-17 ou similaire (voir le tableau des Matériaux / Équipement).
- Du culot nucléaire, éliminer le surnageant, qui contient des protéines ou des protéines qui s'échappent du noyau lors du fractionnement du cytosol.
- Extraction des noyaux de sel:
- Ajouter du tampon C (20 mM de HEPES, pH 7,9, 25% de glycerol, 1,5 mM de MgCl2, 0,2 mM d'EDTA, et fraîchement ajouté 1 mM de DTT, 200 uM de PMSF et 1: 1000 cocktail inhibiteur de protéase) pour le culot nucléaire; utiliser 2,5 ml de tampon C pour 3 ml de volume de départ de l'hématocrite (~ 1 x 10 9 cellules).
- L'utilisation d'une tige de verre ou d'une pipette, déloger le culot nucléaire à partir de la paroi du tube et de transférer l'ensemble du mélange dans un homogénéisateur Dounce d'une taille appropriée.
- Remettre en suspension les noyaux en homogénéisant emélange de e avec deux coups de pilon LOOSE.
- Transférer la fraction nucléaire remis en suspension dans un bécher refroidi. Choisir un bécher de telle sorte que la suspension va remplir le récipient à au moins 0,5 cm de profondeur.
- Pour extraire les protéines de la chromatine nucléaire ou d'autres structures insolubles, augmenter progressivement la concentration en sel de la suspension à 0,42 M de NaCl par addition goutte à goutte de 5 M de NaCl, tout en agitant doucement la suspension avec une pipette ou une tige de verre sur de la glace; une fois la totalité du NaCl 5 M ont été ajoutées, la solution doit devenir très visqueux ou de type gel. Calculer le volume de NaCl 5 M nécessaire pour porter la solution à une concentration finale de 0,42 M de NaCl selon la formule suivante:
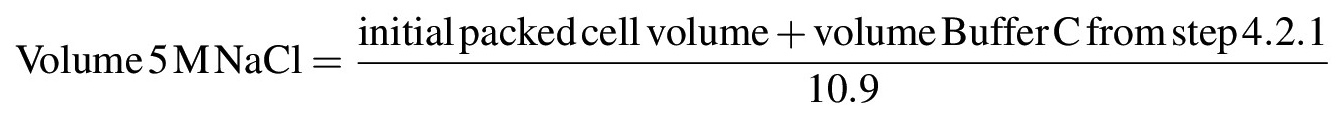
- Soigneusement transférer la suspension visqueuse dans 10 ml tubes en polycarbonate pour un type 70,1 Ti rotor (ou l'équivalent) des bouteilles de polycarbonate ou 70 ml pour un type 45 Ti or rotor similaire (voir le tableau des Matériaux / Équipement). Fermer hermétiquement avec du parafilm si vous utilisez tubes de 10 ml ou avec l'ensemble de bouchon si vous utilisez 70 ml bouteilles.
- Basculer lentement les tubes scellés à 4 ° C pendant 30 min en utilisant un nutator.
- Faites tourner les échantillons dans un type de 45 ou 70,1 Ti Ti rotor pendant 30 min à 120 000 xg à 4 ° C.
- Transférer le surnageant dans un tube en matière plastique unique ou d'une bouteille. Ce surnageant est l'extrait nucléaire, et le culot contenant la chromatine nucléaire et d'autres débris.
- Diviser l'extrait nucléaire en aliquotes de taille conventionnelle, le congeler dans l'azote liquide, et de le stocker à -80 ° C.
5. immunoaffinité Purification de la INO80 humaine ou INO80 Subcomplexes
- Pour décongeler extrait nucléaire congelés, placez les tubes contenant l'extrait sur la paillasse ou faire rouler les tubes entre les mains jusqu'à ce que la matière congelée devient une bouillie. Ensuite, placer les tubes sur de la glace ou dans la chambre froide jusqu'à ce que l'extrait est totalement thawed.
- Transférer l'extrait nucléaire décongelé à tubes de 10 ml en polycarbonate d'ultracentrifugation, et essorage à 100 000 g pendant 20 min à 4 ° C dans un rotor de type Ti 70.1 ou équivalent pour éliminer tout précipité qui s'est formée au cours du cycle de gel-dégel.
- Transférer le surnageant dans un tube conique de 15 ml. Ajouter frais Cocktail TNT, PMSF, et l'inhibiteur de protéase à des concentrations finales de 1 mM de DTT, 200 uM PMSF et 1: 1000 Inhibiteur de la protéase Cocktail.
- Pour préparer anti-FLAG agarose pour la immunopurification, transférer 200 pl de 50% suspension de billes d'agarose anti-FLAG à un tube de 1,5 ml à l'aide d'un P200 ou d'une pipette similaire avec une pointe dont l'extrémité a été coupée avec un scalpel propre ou une lame de rasoir.
- Pellet les billes par centrifugation dans une microcentrifugeuse de paillasse à 8000 xg pendant 30 sec. Enlever le surnageant et laver les billes en remettant en suspension les billes dans 1 ml de tampon Lys450, et sédimenter les billes à 8000 g pendant 30 sec. Laver le bEADS deux fois de plus.
- Remettre en suspension les billes d'agarose anti-FLAG lavées dans environ 100 ul de l'extrait nucléaire au moyen d'un P200 ou similaire pipette de volume variable avec une pointe à partir de laquelle l'extrémité a été coupée avec un scalpel propre ou une lame de rasoir et, en utilisant la même extrémité, le transfert les billes en suspension au tube de 15 ml conique contenant l'extrait. Répétez plusieurs fois jusqu'à ce que toutes les perles ont été transférés dans le tube de 15 ml.
- Incuber le mélange extrait / perle pendant 4 heures à 4 ° C avec une rotation lente sur un agitateur de laboratoire. OPTION: Inclure benzonase à une concentration de 25 unités / ml pour éliminer l'ADN contaminant.
- Recueillir l'agarose FLAG par centrifugation à 1 000 g pendant 5 min à 4 ° C.
- Remettre en suspension dans 10 ml Lys450 à laver, incuber 5 min à 4 ° C avec un doux balancement sur nutator. Pellet les perles à 1000 g pendant 5 min à 4 ° C.
- Remettre en suspension dans 100 - 150 pi de Lys450 et perles de transfert à un tube de 1,5 ml. Continue pour rincer le tube de 15 ml conique avec 100 - 150 pi de Lys450 jusqu'à ce que toutes les perles ont été transférés dans le tube à centrifuger.
- Isoler les perles à 8000 g pendant 30 sec à 4 ° C dans une microcentrifugeuse. Laver trois fois de plus avec 1 ml Lys450 et une fois avec un tampon de 1 ml d'EB100 (10 mM HEPES pH 7,9, 10% de glycerol, 100 mM de NaCl, 1,5 mM de MgCl2, 0,05% de Triton X-100, et fraîchement ajouté mM DTT 1, 200 uM de PMSF , et 1: 1000 Inhibiteur de la protéase Cocktail).
- Pour éluer les protéines liées, ajouter du tampon de EB100 200 pi contenant 0,25 mg / ml de peptide FLAG 1x. Incuber 30 min à 4 ° C sur une nutator chacun.
- Pellet les perles à 8000 g pendant 30 sec à 4 ° C dans une microcentrifugeuse. Transférer le surnageant, qui contient le complexe de INO80 élue, à un microtube frais.
- Répétez l'élution quatre fois, et mettre en commun toutes les surnageants dans un seul tube.
- Pour éliminer les billes d'agarose-FLAG résiduel à partir de la fraction protéique éluée,passer l'éluat à travers une colonne de centrifugation vide.
- Concentrer la fraction de protéine éluée ~ 10 fois en utilisant un dispositif de filtre centrifuge ultra (50000 moléculaire de coupure de poids).
- Pour éliminer le peptide FLAG, passer le concentré fraction protéique séquentiellement par deux colonnes de dessalage.
- Diviser la fraction protéique purifiée, débarrassée des sels dans 20 aliquotes, congelés dans l'azote liquide et stockés à -80 ° C.
- Analyser la composition de sous-unité de INO80 ou Subcomplexes de INO80 sur des gels colorés à l'argent ou par western blot, et estimer leurs concentrations en semi-quantitative western blot en utilisant des préparations de sous-unités de INO80 recombinantes de concentration connue en tant que normes.
Résultats
La figure 1 montre un diagramme résumant les procédures utilisées pour produire, purifier et de caractériser des complexes humains de remodelage de la chromatine INO80 dépendant de l'ATP.
Comme illustré dans les figures 2 et 3, ces procédures permettent la génération de deux types INO80 et INO80 Subcomplexes sauvages qui manquent diverses sous-unités, permettant ainsi des analyses biochimiques ultérieures de la contribution de...
Discussion
Études structurales et fonctionnelles de plusieurs sous-unités des complexes de mammifères de remodelage du eucaryotes supérieurs ont été entravés par la difficulté de préparer biochimiquement des quantités utiles de ces complexes contenant des sous-unités mutantes ou manquant certaines sous-unités au total. Il ya un certain nombre d'obstacles techniques: d'abord, la manipulation génétique dans les cellules de mammifères a été techniquement difficile et de longue haleine. A la différence des ce...
Déclarations de divulgation
Les auteurs déclarent qu'ils n'ont aucun intérêt financier concurrents.
Remerciements
Travail dans le laboratoire des auteurs est financé par une subvention de l'Institut national des sciences médicales générales (GM41628) et par une subvention de l'Institut Stowers pour la recherche médicale de Fonds de recherche médicale Nelson Helen à la Fondation communautaire du Grand Kansas City.
matériels
| Name | Company | Catalog Number | Comments |
| Dulbecco's Modified Eagle Medium | Cellgro | 10-013-CV | |
| Glutamax-I (stablized glutamine) | Life Technologies | 35050-079 | |
| Fetal Bovine Serum | SAFC | 12176C | |
| FuGENE6 transfection reagent | Promega | E2312 | |
| Hygromycin B, sterile in PBS | AG Scientific | H-1012-PBS | |
| pcDNA5/FRT vector | Life Technologies | V6010-20 | |
| Flp-In HEK293 cells | Life Technologies | R780-07 | |
| pOG44 Flp-Recombinase Expression Vector | Life Technologies | V600520 | |
| EZview Red ANTI-FLAG M2 Affinity Gel | Sigma | F2426 | |
| calf serum | SAFC | 12138C | |
| TARGETplus SMARTsiRNA pool | Dharmacon / Thermo Scientific | various | |
| 5x siRNA resuspension buffer | Dharmacon / Thermo Scientific | #B-002000-UB-100 | |
| Lipofectamine RNAiMAX | Life Technologies | 13778 | |
| Opti-MEM Reduced Serum Medium | Life Technologies | 51985-091 | |
| PBS | Cellgro | 45000 | VWR |
| TrypLE (trypsin) | Life Technologies | 12604 | |
| 1x FLAG Peptide | Sigma | F3290 | |
| Micro Bio-Spin Chromatography Column | Bio-Rad | 737-5021 | |
| Amicon Ultra Centrifugal Filter Device (50k MWCO) | Amicon | UFC805024 | Fisher Scientific |
| Zeba Desalting Columns | Thermo Scientific | 89882 | |
| Anti-FLAG M2 antibody, mouse | Sigma | F3165 | |
| Anti-FLAG M2 antibody, rabbit | Sigma | F7425 | |
| Protease Inhibitor Cocktail | Sigma | P8340 | |
| benzonase | Novagen | 70664 | |
| Equipment | |||
| JS-4.2 rotor in a J6 centrifuge | Beckman-Coulter | 339080 | |
| JA-17 rotor | Beckman-Coulter | 369691 | |
| 10 ml polycarbonate tubes | Beckman-Coulter | 355630 | |
| 70 ml polycarbonate bottles | Beckman-Coulter | 355655 | |
| Type 45 Ti rotor | Beckman-Coulter | 339160 | |
| Type 70.1 Ti rotor | Beckman-Coulter | 342184 | |
| BD Clay Adams Nutator Mixer | BD Diagnostics | 15172-203 | VWR |
| Glas-Col Tube/Vial Rotator | Glas-Col | 099A RD4512 | |
| PCR thermal cycler PTC 200 | MJ Research | PTC 200 | |
| roller bottle incubator | Bellco biotechnology | 353348 | |
| Immobilon-FL Transfer Membrane 7 x 8.4 | Millipore | IPFL07810 | |
| lubricated 1.5 ml microcentrifuge tubes | Costar | 3207 | |
Références
- Clapier, C. R., Cairns, B. R. The biology of chromatin remodeling complexes, Annual Review of Biochemistry. 78, 273-304 (2009).
- Szerlong, H., Hinada, K., Viswanathan, R., Erdjument-Bromage, H., Tempst, P., Cairns, B. R. The HSA domain binds nuclear actin-related proteins to regulate chromatin-remodeling ATPases. Nature Struct. Mol. Biol. 15 (5), 469-476 (2008).
- Shen, X., Mizuguchi, G., Hamiche, A., Wu, C. A chromatin remodelling complex involved in transcription and DNA processing. Nature. 406 (6795), 541-544 (2000).
- Shen, X., Ranallo, R., Choi, E., Wu, C. Involvement of actin-related proteins in ATP-dependent chromatin remodelling. Mol. Cell. 12, 147-155 (2003).
- Chen, L., et al. Subunit organization of the human INO80 chromatin remodeling complex: an evolutionarily conserved core complex catalyzes ATP-dependent nucleosome remodeling. Journal of Biological Chemistry. 286 (13), 11283-11289 (2011).
- Sauer, B. Site-specific recombination: developments and applications. 5 (5), 521-527 (1994).
- Gorman, S., Fox, D. T., Wahl, G. M. Recombinase-mediated gene activation and site-specific integration in mammalian cells. Science. 251 (4999), 1351-1355 (1991).
- Jin, J., et al. A mammalian chromatin remodeling complex with similarities to the yeast INO80 complex. Journal of Biological Chemistry. 280 (50), 41207-41212 (2005).
- Cai, Y., et al. YY1 functions with INO80 to activate transcription. Nature Structural and Molecular Biology. 14 (9), 872-874 (2007).
- Yao, T., et al. Distinct Modes of Regulation of the Uch37 Deubiquitinating Enzyme in the Proteasome and in the Ino80 Chromatin-Remodeling Complex. Mol.Cell. 31 (6), 909-917 (2008).
- Brizzard, B. L., Chubet, R. G., Vizard, D. L. Immunoaffinity purification of FLAG epitope-tagged bacterial alkaline phosphatase using a novel monoclonal antibody and peptide elution, Biotechniques. 16 (4), 730-735 (1994).
- Barker, K. . At the Bench: A Laboratory Navigator. 10, 207-245 (2005).
- Dignam, J. D., Lebovitz, R. M., Roeder, R. G. Accurate transcription initiation by RNA polymerase II in a soluble extract from isolated mammalian cell nuclei. Nucleic. Acids. Res. 11 (5), 1475-1489 (1983).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon