このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
発生と人間INO80クロマチンリモデリング複合体とSubcomplexesの精製
要約
このプロトコルは、野生型と人間INO80クロマチンリモデリング複合体の変異型を生成し、精製するための手順を説明します。エピトープはINO80サブユニットのバージョンが安定してHEK293細胞で発現されタグ付けされ、サブユニットの特定のセットを欠いている完全な複合体と複合体を、免疫親和性クロマトグラフィーによって精製される。
要約
INO80クロマチンリモデリング複合体は、ATP依存性ヌクレオソームのリモデリングを触媒することによってヌクレオソームのダイナミクスとDNAのアクセシビリティを調節する。人間INO80複合体は、触媒サブユニットとの複合体のアセンブリのための足場とを兼ねINO80、SNF2様ATPアーゼを含む14のタンパク質サブユニットから構成されています。他のサブユニットと、彼らはINO80複合体のクロマチンリモデリング活性に寄与するメカニズムの機能は、人間の細胞または異種発現系にINO80サブアセンブリを生成するという課題に一部、よくわかっていないままである。このJOVEプロトコルは、サブユニットまたはサブユニットのサブセットを欠いている人間INO80クロマチンリモデリングsubcomplexesの精製を可能にする手順について説明します。 N末端のFLAGエピトープはINO80するcDNAを安定的にタグ付けされたヒト胚腎臓(HEK)のFlp介在組換えを用いて293細胞株に導入される。 INO80複合体のサブユニットのサブセットがb側にある場合でeは削除された、人はこれらのサブユニットの組み立てに必要なプラットフォームを欠いている代わりに、変異体INO80タンパク質を発現。イベントでは、個別のサブユニットが枯渇する場合には、安定的にFLAGを発現するHEK 293細胞株に、このサブユニットをターゲットに1トランスフェクトするのsiRNAはINO80 ATPアーゼは、タグ付き。核抽出物を調製し、FLAG免疫沈降はINO80誘導体を含有するタンパク質画分を濃縮するために行われる。精製INO80のsubcomplexesの組成物は、その後、免疫ブロッティング、銀染色、および質量分析などの方法を用いて分析することができる。このプロトコルに従って生成INO80およびINO80 subcomplexesは、添付JOVEプロトコルに記載されているさまざまな生化学的アッセイを用いて分析することができる。ここに記載される方法は、任意の哺乳動物マルチサブユニットクロマチンリモデリングの構造的および機能的特性の研究及び改変する複合体に適合させることができる。
概要
進化的に保存されたSNF2ファミリークロマチンリモデリング複合体はクロマチン組織とDNAのアクセシビリティ1の重要な調節因子である。これらのリモデリング複合体は常にいくつかのケースでは、さまざまなアクセサリータンパク質で組み立て、マルチサブユニットの巨大分子集合体を形成し、中央SNF2様ATPアーゼサブユニットを含む。 ATP依存性クロマチンリモデリングプロセスの分子的詳細を研究するためには、複合体の活動サブユニットおよび/またはドメイン構造の所定のサブセットの寄与を理解することが重要である。このような分析は、特定のタンパク質サブユニットまたはドメイン構造を欠いている高度に精製された変異体の複合体を生成する能力が必要です。
ATP依存性クロマチンリモデリング複合体の以前の構造-機能研究が広く(参考文献1-4等参照)により、酵母ゲノムの優れた操作性に酵母モデル系に焦点を当てている。の保全を考えるオーソロガスモデリング複合体のうち、サブユニット組成と機能性、酵母リモデリング複合体の構造と機能の研究は、高等真核生物における対応に重要な洞察を提供してきました。それにもかかわらず、リモデリング複合体の中でかなりの種特異的な違いは、保存されたサブユニットの保存されたドメイン内の利得または種特異的サブユニットの喪失、保存されたサブユニットの種特異的なドメインの損益、および配列変動に起因する、存在します。このような差異は、原理的には高等真核細胞が新たな分子および細胞環境に適応する必要性によって駆動することができる。それはATP依存性クロマチンリモデリングプロセスの基本的なメカニズムに光を投げかけて、だけでなく、そのクロマチン構造メカニズムに貴重な洞察を提供することができませんという理由だけでこのように、高等真核生物のリモデリング複合体のサブユニットはヌクレオソームのリモデリングプロセスに寄与するかを理解することは、価値があるHIG内および遺伝子発現彼女の真核生物が規制されている。
これまでのところ、生化学的に定義されたクロマチンリモデリング複合体とsubcomplexesを得ることが困難に一部起因マルチサブ哺乳類のクロマチンリモデリング複合体の限られた構造的および機能的研究が行われている。私たちは、野生型またはINO80 5-7の変異型( 図1)タグ付き免疫親和性精製を安定的にN末端FLAGエピトープを発現するヒト細胞から無傷INO80またはINO80 subcomplexesを調製するために使用される下記の手順を用いてこれらの問題を部分的に回避している。ヒト細胞から無傷INO80複合体を得るために、のFlp介在組換えを安定INO80複合体8-10のサブユニットをコードするFLAGエピトープタグ付きcDNAを発現するトランスジェニックHEK293細胞株を生成するために使用される。 INO80サブユニットの過剰発現がいくらか毒性であり得るため、単離および選択的共同下クローン細胞系を維持するために必要であるnditions大規模な細胞培養の増殖のために必要な多くの通路の間に安定した導入遺伝子発現を保証する。サブユニットのサブセットのみが含まれている小さなINO80のsubcomplexesを得るために、正常に2つのアプローチを使用した( 図2A、B)。まず、当社は安定的に特定のサブユニット5との相互作用に必要なドメインを欠いているINO80の変異型を発現するHEK293 FLP-Inの細胞株を作製。または、siRNAによるノックダウンは、適切なFLAGタグINO80サブユニット(未発表データ)を発現する細胞から目的のサブユニットを枯渇させるために使用されます。最後に、人間INO80複合体を精製するために、FLAGアガロースベースのクロマトグラフィー11は、効果的に精製されたINO80またはINO80 subcomplexesを含む最終画分に細胞質ゾル汚染タンパク質の存在を減らし、核抽出物からINO80を含む画分を濃縮するために使用されます。
プロトコル
全身またはFLAGエピトープタグINO80またはその他INO80複合体のサブユニットの変異型を発現するHEK293安定細胞株の1世代と文化
- 全長または変異ヒトINO80 ATPアーゼまたはインフレーム、N末端FLAGエピトープタグを持つ哺乳動物発現ベクターpcDNA5 / FRTに別INO80サブユニットをコードするcDNAクローン。
- 先に進む前に、DNA配列決定により挿入されたcDNAの配列を確認。
- トランスフェクションを行うために、DMEM(ダルベッコ改変イーグル培地)、5%のグルタミンを含有する培地中で10cmの組織培養皿中で、HEK293細胞でのFlpし、成長し、10%FBS(ウシ胎児血清)。
- 細胞は、〜70%のコンフルエンシーに達したときに、総容積は、各組織培養皿に40適切なpcDNA5 / FRT発現プラスミドのFuGENE6トランスフェクション試薬μlの、0.5μgの、およびFlpリコンビナーゼリコンビナーゼをコードするpOG44を、9.5μgのの混合物を添加800μlに。
- 48時間後、トリプシン処理し、split 10cmの皿に1:30の比率で細胞、および3 5%グルタミン、10%FBS、および100μg/ mlのハイグロマイシンBを含むDMEM中でそれらを成長 - 4週間。 ( - 5日間、通常、3日毎)が黄色に回転し始めるたびに、培地を変更します。
- FLAG標識タンパク質の最高レベルを表す陽性クローンを同定するために、個別のハイグロマイシンB耐性コロニーを選択し、24ウェルプレートの単一のウェルに移す。
- 細胞を5分間、1,000×gでの遠心分離により〜1mlのPBS、ペレットに各ウェルから80%の密集度、収穫細胞に達すると。
- 上清を除去した後、SDS-PAGE試料緩衝液60μlの中で細胞ペレットを再懸濁する。
- SDSページおよびFLAGの発現をモニターするためのウェスタンブロッティングに再懸濁細胞ペレットの件名半分はベイトタンパク質をタグ付け。今後の分析のために、残りの半分を保存します。
- 何FLAGタグ付きヒトINO80タンパク質は、細胞溶解物において検出されない場合、最初のクローン細胞POPUを展開15cmの組織培養皿に24ウェルプレートの単一のウェルから細胞をプレーティングすることによって、更に設置と。
- 15cmの皿、50mlの円錐チューブに氷冷PBS中で、コンフルエント細胞に近い再懸濁し、転送から細胞を採取する。
- 追加のPBSを添加することによって最終体積50mlにもたらす。
- ペレットを5分間、1,000×gで細胞および上清をできるだけ多く除去する。
- Lys450緩衝液(20mM HEPES-NaOHでpHを7.9、450 mMの塩化ナトリウム、0.5%トリトンX-100、10mMのKCl、4mMのMgCl 2を 、0.2mMのEDTA、10%グリセロール、1mMのDTT、1mlの細胞ペレットを再懸濁200μMのPMSF、および1:1,000プロテアーゼ阻害剤カクテル)。
注:ここでは、他の場所は、必ずすぐに実験を開始する前に、バッファに、DTT、PMSF、およびプロテアーゼ阻害剤カクテルを追加します。 - 抗FLAGアガロースゲル20μlのを使用して、結果として全細胞溶解物から免疫沈殿物FLAGタグタンパク質、およびウェスタンブロッティングによってFLAG溶出液を分析します。 [IMMUNの詳細については、oprecipitation手順は、手順5を参照してください]
- 希望クローン細胞株12の凍結ストックを準備し、使用するまで液体窒素中で保管してください。
ローラーボトル中で2成長するHEK293細胞株
INO80複合体の大規模調製のために、培養細胞を10 - 20ローラーボトル;各ローラーボトルからの典型的な収量は、パックされた細胞の約1 mlである。
- 各ローラーボトルに、ハイグロマイシンBをせずに200ミリリットルDMEM、5%のグルタミンおよび10%ウシ血清を追加
- 各ローラーボトルにシングルほぼコンフルエント15cmの皿からの細胞のすべてを転送する
- 37℃ローラーボトルインキュベーターにローラーボトルを配置し、0.2 rpmで回転する。
- 細胞は〜70%の集密度に達すると、捨てる、中を捨てる。
- 各ボトルに〜氷50mlの冷PBSを追加します。水平にボトルを持ち、静かに細胞単層を緩め旋回。
- 250ミリリットルプラストに再懸濁した細胞を移しIC円錐ボトルや氷の上の場所。
- ボトルにボトルから順次に転送、追加のPBSでローラーボトルをすすぐ。リンス液はもはや明確な(典型的には約5瓶をリンスするために使用された後に)しない場合には、細胞懸濁液に加える。
- 10分間400×gでの遠心分離によって細胞をペレット化。
- 静かに、PBS中で細胞を再懸濁シングル250ミリリットルコニカル瓶にそれらを組み合わせて、さらに処理するまで氷上に保つ。
もう一つのFLAGタグINO80のサブユニットを発現する細胞においてINO80のサブユニットの3 siRNA介在ノックダウン
単一のサブユニットを欠いINO80 subcomplexesを得るためには、安定的にshRNAを発現するsiRNA処理した細胞または細胞からINO80複合体を精製するために、FLAG-免疫精製を使用しています。ここでいう「逆」のsiRNA(低分子干渉RNA)のトランスフェクションプロトコルは、15cmの皿で成長するHEK293細胞のために最適化される。プロトコルは、細胞や笙のシングル15cmのディッシュ用です必要な細胞の数に応じてそれに応じてスケールアップすることがULD。 siRNAで処理した細胞からINO80複合体の生化学的に有用な量を用意するためには、40 15cmの皿で増殖させた培養物まで拡張する必要があり;パックされた細胞ペレットを4ml - これらは、およそ2が得られる。
- 安定的に15cmの皿で近く密集するまでINO80複合体の所望のサブユニットを発現するHEK293細胞を成長させる。
- siRNAの再懸濁緩衝液の容積の追加(20ミリモルのKCl、6mMのHEPES、pHが7.5、および0.2mMのMgCl 2)で十分に凍結乾燥したsiRNAを含むチューブに対するsiRNAの50μMのストック溶液を調製した。ソリューションを上にピペットで数回上下およびsiRNAが完全に溶解されていることを確認するために室温で30分間ニューテーター上でインキュベートする。
- siRNAおよびトランスフェクション試薬を含むトランスフェクションカクテルを準備します。非常に穏やかに混合しながら低血清培地32μlのリポフェクタミンRNAiMAXトランスと、50μMsiRNAストック溶液10μlを混合し、のOpti-MEM 4ミリリットルに追加。アル低すべての試薬は、使用前に室温に平衡化する。
- 室温で30分間、混合物をインキュベートする。
- FLP-でHEK293細胞を安定にトランスフェクションのために必要なINO80サブユニットを発現して準備します。
- ステップ3.4のインキュベーションの間、RT PBSで一度15cmの皿で細胞を洗浄する。
- PBSを除去した後、彼らはプレートを持ち上げ始めるちょうどまで、1ミリリットルのトリプシンで細胞を処理。
- すぐに細胞をトリプシン処理し、穏やかに混合するために完全培地(DMEM + 5%グルタミン+ 10%FBS)の10ミリリットルを追加します。室温で5分間1,000×gでの遠心分離によって細胞を回収します。
- 〜4ミリリットル完全培地中に細胞ペレットを再懸濁し、血球計数器を用いて細胞を再懸濁カウント。
- 〜5.4×10 6 / mlの濃度に完全培地で細胞を希釈する。
- 各15cmの皿に、4ミリリットルのトランスフェクションカクテルに続いて15ミリリットル完全培地を追加します。培地およびトランスフェクションカクテルを確保するために、静かに旋回されthoroughly混合。最後に、細胞懸濁液1ミリリットルを追加し、再度均一に細胞を分散させるために穏やかに旋回。
- 37℃、5%CO 2インキュベーターで培養し、60時間後、静かに、氷冷PBS中で再懸濁細胞を培地を除去し、核抽出物を調製するためにすぐに進みます。
核抽出物4。準備
この手順は、ディグナム13のプロトコルから変更されて、細胞ペレットを開始するのサイズに応じて拡大または縮小することができる。典型的には、パックされた細胞ペレット収率1mlの最終核抽出物を1ml。全ての緩衝液は氷冷でなければならず、適切なコールドルームが利用できない場合、すべてのステップは低温室で、または氷上で行うべきである。
- 核の単離:
- 静かに、適切な大きさ(15または50ミリリットル)に細胞を移す4℃で10分間千×gで円錐管とスピンを卒業しました。
- 上清を除去し、充填細胞ペルの体積を測定てみましょう。パックされた細胞の1mlを〜3×10 8個のHEK293細胞に相当する。
- 5充填細胞緩衝液Aのボリューム(1,000プロテアーゼインヒビターカクテル10のHEPES、pHは7.9、1.5のMgCl 2、10mMのKClおよびたて1のDTT、200μMのPMSFを加え、1)を追加します。穏やかにピペッティングすることにより細胞ペレットを再懸濁する。
- 正確に10分間氷上でインキュベートする。
- 4℃で10分間、1,000×gで細胞をペレット、上清を除去します。
- 細胞ペレットのサイズは、緩衝液A中でのインキュベーションの間に2倍まで増加するはず
- 緩衝液Aの2つの充填細胞体積の細胞を再懸濁し、適切なサイズのDounce組織ホモジナイザーに細胞懸濁液を移す。パックされた細胞の2ml未満で開始した場合、7ミリリットルホモジナイザーを使用します。 2のために - 4mlの詰まった細胞、15ミリリットルホモジナイザーを使用します。と詰まった細胞の10以上ミリリットルのために、40ミリリットルホモジナイザーを使用しています。
- ダウンスホモジナイザー国連のLOOSEガラス乳棒で細胞懸濁液を均質化ゴマ細胞の90%が1%トリパンブルーで陽性に染色。
- ( 材料/機器の表を参照)、JA-17または類似のローターで、4℃で20分間、25000×gで45ミリリットルの高速遠心管とスピンにサスペンションを転送します。
- 核ペレットから、サイトゾルタンパク質または分画中に核から漏れタンパク質を含む上清を取り除く。
- 塩で核を抽出。
- 緩衝液C(20mMのHEPES、pHが7.9、25%グリセロール、1.5のMgCl 2、0.2mMのEDTA、および新たに添加1のDTT、200μMのPMSF、および1:1,000プロテアーゼインヒビターカクテル)が追加核ペレットに。血球容積(〜1×10 9個 )を開始するごとに3ミリリットル、2.5 mlのBuffer Cを使う。
- ガラス棒またはピペットを用いて、チューブの壁から核ペレットを取り除くし、適切なサイズのダウンスホモジナイザーに混合物全体を移す。
- 目を均質化することによって核を再懸濁LOOSE乳棒2打された電子の混合物である。
- チルドビーカーに再懸濁核分画を転送します。サスペンションが深い少なくとも0.5 cmのビーカーいっぱいになりますようにビーカーを選択してください。
- 穏やかに氷上ピペットまたはガラス棒で懸濁液を攪拌しながら徐々5 M NaClを滴下することによって0.42 M NaClの懸濁液の塩濃度を増加させ、クロマチンまたは他の不溶性構造から核タンパク質を抽出する;一度すべての5M NaClが添加された、溶液は非常に粘性またはゲル状になるべきである。 M NaClを以下の式に従って0.42 MのNaClの最終濃度に溶液をもたらすために必要とされる5の体積を計算する。
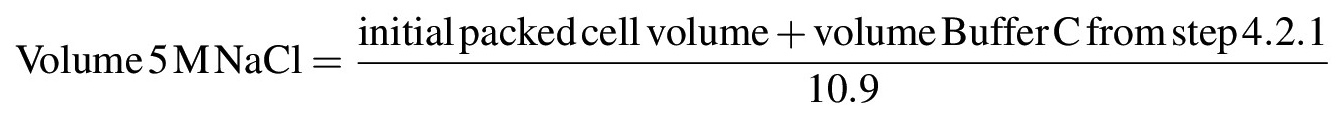
- 慎重にタイプ45のTi Oのためにタイプ70.1 Tiローターのために10ミリリットルのポリカーボネートチューブに、粘性の懸濁液を転送(または同等)、または70ミリリットルのポリカーボネートボトル同じようなローター(材料/機器の表を参照してください)R。 70ミリリットルのボトルを使用している場合は10ミリリットルチューブを使用する場合はキャップアセンブリをパラフィルムでしっかりとシール。
- ゆっくりと旋回装置を用いて30分間、4℃で密封されたチューブを揺する。
- 45型Ti又は4℃で12万×gで30分間、70.1 Tiローターでサンプルをスピン。
- シングルプラスチックチューブやボトルに上清を移す。この上清を核抽出物であり、ペレットは、クロマチンと他の核の破片が含まれています。
- 、使いやすい量に核抽出物を分割し、液体窒素中で凍結し、-80℃で保管してください。
人間INO80またはINO80 Subcomplexes 5。免疫親和性精製
- 凍結した核抽出、凍結した材料は、スラリーになるまで手の間ベンチやロールチューブに抽出物を含有する場所管を解凍します。抽出物が完全にトンになるまで、氷の上または低温室で試験管を置くhawed。
- 10ミリリットルのポリカーボネート超遠心管に解凍した核抽出物を移し、凍結 - 解凍サイクルの間に形成した沈殿物を除去するタイプ70.1 Tiローターまたは同等の4℃で20分間、100,000×gでスピン。
- 15ミリリットルコニカルチューブに上清を移す。千プロテアーゼ阻害剤カクテル:1のDTT、200μMのPMSF、および1の最終濃度に新鮮DTT、PMSF、およびプロテアーゼ阻害剤カクテルを追加します。
- 、免疫精製のための抗FLAGアガロースを準備端は清潔なメスで切断した後のチップを用いてP200または類似のピペットを用いて、1.5mlのマイクロチューブに抗FLAGアガロースビーズの50%スラリー200μlのを転送するにはまたはかみそりの刃。
- 30秒間8,000×gで卓上微量遠心分離によりビーズをペレット。上清を除去し、Lys450緩衝液1ml中にビーズを再懸濁することによりビーズを洗浄し、30秒間8,000×gでビーズをペレット化。 Bを洗うさらに2回EADS。
- 最後はきれいなメスまたはカミソリの刃と、同じチップを用いて、トランスファーで切断した後のチップを備えたP200または同様の調整可能なボリュームピペットを用いて、核抽出物を約100μlにおける洗浄し、抗FLAGアガロースビーズを再懸濁抽出物を含有する15ミリリットルコニカルチューブに再懸濁ビーズ。ビーズの全てまで数回繰り返す15mlチューブに移してきた。
- 実験室での回転装置上でゆっくりと回転させながら、4℃で4時間抽出/ビーズ混合物をインキュベートする。オプション:混入DNAを除去するために25単位/ mlの濃度でベンゾナーゼ含めます。
- 4℃で5分間1,000×gでの遠心分離によってアガロースFLAGを収集します。
- 10ミリリットルLys450に再懸濁穏やかに旋回装置に揺らしながら、4℃で5分間インキュベート、洗浄する。 4℃で5分間千×gでビーズをペレット化。
- 1.5mlのマイクロチューブにLys450および転送ビーズ150μlの - 100に再懸濁。株式会社すべてのビーズをマイクロ遠心管に移しされるまで、Lys450150μlの - ntinueは100と15ミリリットルコニカルチューブを洗浄します。
- 微量4℃で30秒間、8,000×gでビーズをスピンダウン。 1ミリリットルEB100緩衝液(10mM HEPES pHは7.9、10%グリセロール、100mMのNaCl、1.5mMのMgCl 2、0.05%トリトンX-100、および新たに添加1mMのDTT、200μMのPMSFで一回1ミリリットルLys450で三回以上洗浄し、1:1,000プロテアーゼ阻害剤カクテル)。
- 結合したタンパク質を溶出するために、0.25 mg / mlの1×FLAGペプチドを含む200μlのEB100バッファーを追加します。ニューテーター上で4℃でそれぞれ30分間インキュベートする。
- 微量4℃で30秒間、8,000×gでビーズをペレット化。新しいマイクロチューブに溶出INO80複合体を含む、上清を移し。
- 溶出を4回繰り返し、一つのチューブにすべての上清をプールする。
- 溶出されたタンパク質画分から残留FLAG-アガロースビーズを除去するために、空のスピンカラムを通して溶出液を通過させる。
- 溶出したタンパク質画分を濃縮〜10倍超遠心フィルター装置(50,000分子量カットオフ)を使用して。
- FLAGペプチドを削除するには、2脱塩カラムを通して濃縮されたタンパク質画分を順次渡します。
- 液体窒素中で凍結し、-80℃で保存し、20μlのアリコートに精製され、脱塩タンパク質画分を分割する。
- 銀染色ゲル上またはウェスタンブロッティングによりINO80またはINO80のsubcomplexesのサブユニット組成を分析し、標準として既知濃度の組換えINO80サブユニットの調製物を用いて半定量的ウェスタンブロッティングによってそれらの濃度を推定する。
結果
図1は 、生成、精製、およびヒトINO80 ATP依存性クロマチン再構築複合体を特徴付けるために使用される手順を要約したフローチャートを示す。
図2および図3に示すように、これらの手順は、それによってINO80の酵素活性にこれらの不足しているサブユニットの寄与のその後の生化学的な分析を可能にする、さまざまなサブユニットを...
ディスカッション
高等真核生物から多サブ哺乳類のクロマチンリモデリング複合体の構造と機能の研究は、変異型サブユニットを含むか、完全に特定のサブユニットを欠いているような複合体の生化学的に有用な量を準備することの難しさによって妨げられてきた。技術的なハードルがいくつかあります:最初に、哺乳動物細胞における遺伝子操作は技術的に困難で時間がかかるしています。そのゲノムが容?...
開示事項
著者は、彼らが競合する金融利害がないことを宣言します。
謝辞
著者らの研究室での作業は一般的な医学研究所(GM41628)からの助成金によってグレーターカンザスシティコミュニティ財団のヘレン·ネルソン医学研究基金からの医学研究のためのStowers研究所への補助金によってサポートされています。
資料
| Name | Company | Catalog Number | Comments |
| Dulbecco's Modified Eagle Medium | Cellgro | 10-013-CV | |
| Glutamax-I (stablized glutamine) | Life Technologies | 35050-079 | |
| Fetal Bovine Serum | SAFC | 12176C | |
| FuGENE6 transfection reagent | Promega | E2312 | |
| Hygromycin B, sterile in PBS | AG Scientific | H-1012-PBS | |
| pcDNA5/FRT vector | Life Technologies | V6010-20 | |
| Flp-In HEK293 cells | Life Technologies | R780-07 | |
| pOG44 Flp-Recombinase Expression Vector | Life Technologies | V600520 | |
| EZview Red ANTI-FLAG M2 Affinity Gel | Sigma | F2426 | |
| calf serum | SAFC | 12138C | |
| TARGETplus SMARTsiRNA pool | Dharmacon / Thermo Scientific | various | |
| 5x siRNA resuspension buffer | Dharmacon / Thermo Scientific | #B-002000-UB-100 | |
| Lipofectamine RNAiMAX | Life Technologies | 13778 | |
| Opti-MEM Reduced Serum Medium | Life Technologies | 51985-091 | |
| PBS | Cellgro | 45000 | VWR |
| TrypLE (trypsin) | Life Technologies | 12604 | |
| 1x FLAG Peptide | Sigma | F3290 | |
| Micro Bio-Spin Chromatography Column | Bio-Rad | 737-5021 | |
| Amicon Ultra Centrifugal Filter Device (50k MWCO) | Amicon | UFC805024 | Fisher Scientific |
| Zeba Desalting Columns | Thermo Scientific | 89882 | |
| Anti-FLAG M2 antibody, mouse | Sigma | F3165 | |
| Anti-FLAG M2 antibody, rabbit | Sigma | F7425 | |
| Protease Inhibitor Cocktail | Sigma | P8340 | |
| benzonase | Novagen | 70664 | |
| Equipment | |||
| JS-4.2 rotor in a J6 centrifuge | Beckman-Coulter | 339080 | |
| JA-17 rotor | Beckman-Coulter | 369691 | |
| 10 ml polycarbonate tubes | Beckman-Coulter | 355630 | |
| 70 ml polycarbonate bottles | Beckman-Coulter | 355655 | |
| Type 45 Ti rotor | Beckman-Coulter | 339160 | |
| Type 70.1 Ti rotor | Beckman-Coulter | 342184 | |
| BD Clay Adams Nutator Mixer | BD Diagnostics | 15172-203 | VWR |
| Glas-Col Tube/Vial Rotator | Glas-Col | 099A RD4512 | |
| PCR thermal cycler PTC 200 | MJ Research | PTC 200 | |
| roller bottle incubator | Bellco biotechnology | 353348 | |
| Immobilon-FL Transfer Membrane 7 x 8.4 | Millipore | IPFL07810 | |
| lubricated 1.5 ml microcentrifuge tubes | Costar | 3207 | |
参考文献
- Clapier, C. R., Cairns, B. R. The biology of chromatin remodeling complexes, Annual Review of Biochemistry. 78, 273-304 (2009).
- Szerlong, H., Hinada, K., Viswanathan, R., Erdjument-Bromage, H., Tempst, P., Cairns, B. R. The HSA domain binds nuclear actin-related proteins to regulate chromatin-remodeling ATPases. Nature Struct. Mol. Biol. 15 (5), 469-476 (2008).
- Shen, X., Mizuguchi, G., Hamiche, A., Wu, C. A chromatin remodelling complex involved in transcription and DNA processing. Nature. 406 (6795), 541-544 (2000).
- Shen, X., Ranallo, R., Choi, E., Wu, C. Involvement of actin-related proteins in ATP-dependent chromatin remodelling. Mol. Cell. 12, 147-155 (2003).
- Chen, L., et al. Subunit organization of the human INO80 chromatin remodeling complex: an evolutionarily conserved core complex catalyzes ATP-dependent nucleosome remodeling. Journal of Biological Chemistry. 286 (13), 11283-11289 (2011).
- Sauer, B. Site-specific recombination: developments and applications. 5 (5), 521-527 (1994).
- Gorman, S., Fox, D. T., Wahl, G. M. Recombinase-mediated gene activation and site-specific integration in mammalian cells. Science. 251 (4999), 1351-1355 (1991).
- Jin, J., et al. A mammalian chromatin remodeling complex with similarities to the yeast INO80 complex. Journal of Biological Chemistry. 280 (50), 41207-41212 (2005).
- Cai, Y., et al. YY1 functions with INO80 to activate transcription. Nature Structural and Molecular Biology. 14 (9), 872-874 (2007).
- Yao, T., et al. Distinct Modes of Regulation of the Uch37 Deubiquitinating Enzyme in the Proteasome and in the Ino80 Chromatin-Remodeling Complex. Mol.Cell. 31 (6), 909-917 (2008).
- Brizzard, B. L., Chubet, R. G., Vizard, D. L. Immunoaffinity purification of FLAG epitope-tagged bacterial alkaline phosphatase using a novel monoclonal antibody and peptide elution, Biotechniques. 16 (4), 730-735 (1994).
- Barker, K. . At the Bench: A Laboratory Navigator. 10, 207-245 (2005).
- Dignam, J. D., Lebovitz, R. M., Roeder, R. G. Accurate transcription initiation by RNA polymerase II in a soluble extract from isolated mammalian cell nuclei. Nucleic. Acids. Res. 11 (5), 1475-1489 (1983).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved