É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Geração e Purificação de INO80 cromatina remodelação Complexos Humanos e subcomplexos
Neste Artigo
Resumo
Este protocolo descreve um procedimento para gerar e purificação de tipo selvagem e as versões mutantes do complexo remodelação INO80 cromatina humana. Marcada com epitopo versões de subunidades INO80 são estavelmente expressos em células HEK293, e complexos completos e complexos que faltam conjuntos específicos de subunidades são purificados por cromatografia de imunoafinidade.
Resumo
INO80 complexos de remodelação da cromatina regular a dinâmica nucleossomos e acessibilidade DNA, catalisando ATP-dependente remodelação nucleosome. Complexos INO80 humanos consistem de 14 subunidades de proteína incluindo INO80, um SnF2-como ATPase, que serve tanto como a subunidade catalítica e a armação para a montagem dos complexos. Funções das outras subunidades e os mecanismos pelos quais eles contribuem para a remodelação da cromatina actividade do complexo INO80 permanecem pouco compreendidos, em parte devido à dificuldade de gerar subconjuntos INO80 em células humanas ou de sistemas de expressão heterólogos. Este protocolo JOVE descreve um processo que permite a purificação de subcomplexos INO80 remodelação da cromatina humanos que carecem de uma subunidade ou um subconjunto de subunidades. FLAG no terminal-N marcada com epitopo INO80 cDNA são estavelmente introduzido no rim embrionário humano (HEK) 293 linhas de células que utilizam recombinação mediada por Flp. No caso em que um subconjunto de subunidades do complexo INO80 é abe excluídos, uma vez expressa proteínas INO80 mutantes que não possuem a plataforma necessária para a montagem dessas subunidades. No caso de um indivíduo é a subunidade a ser esvaziada, uma transfects siRNAs voltadas para esta subunidade em uma linha de células HEK 293 que expressam estavelmente FLAG etiquetado INO80 ATPase. Extratos nucleares são preparados, e imunoprecipitação FLAG é realizada para enriquecer frações de proteínas contendo derivados INO80. As composições de subcomplexos INO80 purificadas podem então ser analisados por meio de métodos tais como imunoblot, coloração com prata, e espectrometria de massa. Os INO80 e INO80 subcomplexos gerados de acordo com este protocolo pode ser ainda analisada utilizando vários ensaios bioquímicos, que são descritos no protocolo JOVE acompanha. Os métodos aqui descritos podem ser adaptados para o estudo das propriedades estruturais e funcionais de qualquer mamífero de subunidades múltiplas de remodelação da cromatina e de complexos de modificadores.
Introdução
Família SnF2 complexos de remodelação da cromatina evolutivamente conservadas são reguladores chave da organização da cromatina e acessibilidade DNA 1. Estes complexos de remodelação sempre incluir uma subunidade ATPase centro SnF2 semelhante, o qual, em alguns casos, monta com várias proteínas acessórias e forma de multi-subunidades macro-molecular conjuntos. Para estudar os detalhes moleculares do processo de remodelação da cromatina dependentes de ATP, é importante compreender as contribuições de determinados subconjuntos de subunidades e / ou estruturas de domínio para as atividades dos complexos. Tais análises exigem a capacidade de gerar complexos mutantes puras que não possuem determinadas subunidades de proteínas ou estruturas de domínio.
Anteriores estudos de estrutura-função de complexos de remodelação da cromatina dependentes de ATP foram amplamente centrado no sistema modelo de levedura devido à manipulabilidade superior do genoma de uma levedura (ver, por exemplo, refs 1-4). Dada a conservação dacomposição de subunidades e funcionalidade entre os complexos de remodelação ortólogas, estudos da estrutura e função dos complexos de remodelação levedura ter fornecido informações importantes sobre os seus homólogos em eucariotos superiores. No entanto, existem espécies específicas apreciáveis diferenças entre os complexos de remodelação, decorrente de ganho ou perda de espécies específicas subunidades, ganho ou perda de domínios específicos de espécies de subunidades conservadas, ea variabilidade de sequência dentro de domínios conservados de subunidades conservadas. Estas diferenças podem, em princípio, ser conduzido pela necessidade de células eucarióticas superiores a adaptar-se a novos ambientes moleculares e celulares. Assim, a compreensão de como subunidades maiores complexos de remodelação eucarióticas contribuir para o processo de remodelação nucleosome é valioso, porque não só lança luz sobre os mecanismos básicos do processo de remodelação da cromatina ATP-dependente, mas também pode fornecer informações valiosas sobre os mecanismos pelos quais a estrutura da cromatina e expressão gênica em higos eucariontes são regulados.
Até o momento, não foram apenas limitado estudos estruturais e funcionais de várias subunidades de mamíferos complexos de remodelação da cromatina, em parte devido às dificuldades na obtenção de complexos de remodelação da cromatina bioquimicamente definidos e subcomplexos. Temos contornada parcialmente estas dificuldades com os procedimentos descritos abaixo, em que a purificação de imunoafinidade é utilizada para preparar INO80 ou INO80 subcomplexos intactas a partir de células humanas que expressam estavelmente FLAG no terminal-N marcada com epitopo do tipo selvagem ou mutantes de versões INO80 5-7 (Figura 1) . Para obter os complexos INO80 intactas de células humanas, a recombinação mediada por Flp é usado para gerar linhas de células HEK293 que expressam estavelmente transgénicos epitopo FLAG tag cDNAs que codificam as subunidades do complexo INO80 8-10. Porque o excesso de expressão de subunidades INO80 pode ser um pouco tóxica, é necessário isolar e manter linhas de células clonais sob co selectivonditions para assegurar a expressão do transgene estável durante as muitas passagens necessárias para a expansão de culturas de células em larga escala. Para obter subcomplexos INO80 menores que contenham apenas um subconjunto de subunidades, temos utilizado com sucesso duas abordagens (Figura 2A, B). No primeiro, podemos gerar linhagens de células HEK293 Flp-In expressando estavelmente versões mutantes de INO80 que não possuem domínios necessários para a interação com subunidades específicas 5. Alternativamente, knockdown siRNA mediada é utilizada para esgotar a subunidade desejada a partir de células que expressam uma subunidade INO80 marcado com FLAG apropriado (dados não publicados). Finalmente, para purificar os complexos INO80 humanos, FLAG cromatografia de agarose com base 11 é utilizada para enriquecer uma fracção contendo INO80 de extractos nucleares, reduzindo de forma eficaz a presença de proteínas contaminantes na fracção citosólica final contendo purificados INO80 ou INO80 subcomplexos.
Protocolo
1 Geração e Cultura de HEK293 Lines Estável célula que expressa Corpo Inteiro ou versões mutantes FLAG marcadas com epítopos INO80 ou outras subunidades INO80 complexos
- O clone de cDNA que codifica para o comprimento completo ou mutante humano INO80 ATPase ou outra subunidade INO80 no vector de expressão de mamífero pcDNA5 / FRT com um, o terminal N epitopo marcador FLAG em grelha.
- Confirme a sequência dos cDNAs inseridos por sequenciamento de DNA antes de prosseguir.
- Para realizar a transfecção, crescer Flp-Em células HEK293 10 centímetros em pratos de cultura de tecidos em meio contendo DMEM (Dulbecco Modified Eagle Médium), 5% de glutamina, e 10% de FBS (soro fetal bovino).
- Quando as células atingem a ~ 70% de confluência, adicionar a cada placa de cultura de tecido de uma mistura de 40 ul de reagente de transfecção FuGENE6, 0,5 ug do pcDNA5 / FRT do plasmídeo de expressão apropriado, e 9,5 ug de pOG44, que codifica a recombinase Flp, em um volume total de 800 ul.
- 48 horas mais tarde, e s trypsinizeplit células a uma razão de 1:30 em placas de 10 cm, e crescê-las em DMEM com 5% de glutamina, FBS a 10%, e 100 ug / ml de higromicina B para a 3 - 4 semanas. Mude o meio de cultura, sempre que começa a virar amarelo (normalmente a cada 3-5 dias).
- Para identificar os clones positivos que expressam o nível mais elevado de proteína etiquetada com FLAG, seleccionar colónias individuais higromicina B-resistentes e transferi-los para um único poço de uma placa de 24 poços.
- Uma vez que as células atingirem 80% de confluência, as células de cada poço de colheita em ~ 1 ml de PBS e sedimento por centrifugação a 1000 xg durante 5 min.
- Depois de remover o sobrenadante, ressuspender o sedimento celular em 60 ul de tampão de amostra de SDS-PAGE.
- Assunto metade do pellet celular foi novamente suspensa a SDS página e western blot para monitorar a expressão da bandeira com a tag proteína isca; salvar a outra metade para análises futuras.
- Se a proteína INO80 humano marcado com FLAG é detectada em lisados celulares, expanda a popu celular clonal inicialmento adicional por plaqueamento de células a partir de poços individuais de uma placa de 24 poços em placas de cultura de 15 centímetros de tecido.
- Para colher as células a partir de 15 centímetros pratos, ressuspender perto de células confluentes em PBS gelado e transferir para um tubo de 50 ml.
- Levar o volume final de 50 ml por adição de PBS adicional.
- Células de pellets em 1000 xg durante 5 min, e remover o máximo possível de sobrenadante.
- Ressuspender o sedimento de células em 1 ml de tampão de Lys450 (20 mM de HEPES-NaOH pH 7,9, NaCl 450 mM, 0,5% Triton X-100, 10 mM de KCl, 4 mM de MgCl2, 0,2 mM EDTA, 10% glicerol, 1 mM de DTT, 200 uM de PMSF, e 1: 1000 Protease Inhibitor Cocktail).
NOTA: aqui e em outros lugares, sempre adicionar DTT, PMSF, e inibidor de protease cocktail para buffers imediatamente antes de iniciar um experimento. - Proteínas a partir do lisado celular total resultante imuno-precipitado FLAG-etiquetado usando 20 ul de gel de agarose de anti-FLAG, e analisar os eluatos FLAG por Western blotting. [Para mais detalhes sobre immunprocedimento oprecipitation, consulte o passo 5]
- Prepare os estoques congelados de desejadas linhas de células clonais 12 e armazená-los em nitrogênio líquido até o uso.
2. Crescer linhas celulares HEK293 em frascos cilíndricos
Para preparação em grande escala de complexos INO80, células de cultura em cada 10 - 20 garrafas de rolo; um rendimento típico de cada frasco rolante é ~ 1 ml de células embaladas.
- Para cada garrafa cilíndrica, adicionar 200 ml de DMEM, 5% de glutamina, e 10% de soro de vitelo, sem higromicina B.
- Transferir todas as células a partir de um único quase confluentes 15 centímetros prato em cada frasco rolante
- Garrafas lugar de rolo em um 37 ° C rolo garrafa incubadora e rodam a 0,2 rpm.
- Uma vez que as células chegar a 70% de confluência, deitar fora e descartar o meio.
- Adicionar ~ 50 ml de PBS gelado a cada frasco. Segurando garrafas na horizontal, agite bem para soltar a monocamada de células.
- Transferir as células ressuspensas para 250 ml plastgarrafas cônicas ic e colocar no gelo.
- Lavar frascos cilíndricos com PBS adicional, transferindo-o sequencialmente de garrafa a garrafa. Quando a solução de lavagem não é mais clara (tipicamente depois de ter sido usada para lavar garrafas a cerca de 5), adicionar-se à suspensão de células.
- As células por centrifugação a 400 xg durante 10 min.
- Delicadamente ressuspender as células em PBS, combiná-los em um único frasco cónico de 250 ml, e manter em gelo até ulterior processamento.
3 mediada por siRNA knockdown de INO80 subunidades em células que expressam Outra marcado com FLAG INO80 Subunidade
Para obter subcomplexos INO80 falta uma única subunidade, utilização FLAG-imunopurificação para purificar complexos INO80 de células ou células tratadas de siRNA que expressam estavelmente shRNA. O siRNA "reverso" (pequeno RNA de interferência) protocolo de transfecção descrito aqui é otimizado para HEK293 células que crescem em 15 centímetros pratos. O protocolo é de um único prato de 15 centímetros de células e shoUld ser aumentadas proporcionalmente, dependendo do número de células necessárias. Para preparar quantidades úteis do complexo bioquimicamente INO80 de células tratadas com siRNA, deve dimensionar-se a culturas cultivadas em pratos de 40 15 centímetros; estes irão originar aproximadamente 2-4 ml de sedimento globular.
- Cultivar células HEK293 que expressam estavelmente a subunidade desejada do complexo INO80 para próximo da confluência em pratos de 15 centímetros.
- Adicionar um volume de tampão de ressuspensão siRNA (KCl 20 mM, 6 mM de HEPES pH 7,5, e 0,2 mM de MgCl 2), suficiente para preparar uma solução de estoque 50 mM de siRNA para um tubo contendo liofilizado siRNA. Pipetar a solução para cima e para baixo algumas vezes e incuba-se em um nutator durante 30 min à temperatura ambiente para garantir que o siRNA é totalmente dissolvido.
- Preparar um cocktail de transfecção contendo siRNAs e reagente de transfecção. Misturar 10 ml de solução-mãe de 50 uM siRNA com 32 ul de Lipofectamina RNAiMAX e adicioná-lo aos 4 ml de Opti-MEM Meio de Soro Reduzido com agitação muito suave; albaixos todos os reagentes atinjam a temperatura ambiente antes do uso.
- Incubar a mistura à temperatura ambiente durante 30 min.
- Preparar células Flp-In HEK293 expressando estavelmente a subunidade INO80 desejado para a transfecção.
- Durante a incubação do Passo 3.4, lave as células nos pratos de 15 centímetros uma vez com PBS temperatura ambiente.
- Depois de retirar PBS, tratar as células com 1 ml de tripsina apenas até que eles começam a levantar a placa.
- Imediatamente adicionar 10 ml de meio completo (DMEM + 5% de glutamina + 10% de FBS) para as células tratadas com tripsina e misturar suavemente. Recolher as células por centrifugação a 1000 xg durante 5 min à TA.
- Ressuspender o sedimento de células em ~ 4 ml de meio completo, e a contagem de células utilizando um ressuspensão hemocitómetro.
- Dilui-se as células com meio completo a uma concentração de ~ 5,4 x 10 6 / ml.
- Para cada 15 centímetros prato, adicione 15 ml de meio de cultura completo seguido de 4 ml de transfecção cocktail. Mexa cuidadosamente para garantir a médio e transfecção cocktail são thoroughly misturado. Finalmente, adicionar um ml de suspensão de células e novamente agitar suavemente para dispersar as células uniformemente.
- Após 60 horas de cultura em um 37 ° C, 5% incubadora de CO 2, remover suavemente médio, Ressuspender as células em PBS gelado, e proceder imediatamente a preparar os extractos nucleares.
4 Preparação de Extractos Nucleares
Este procedimento foi modificado a partir do protocolo de Dignam 13 e podem ser ajustados para cima ou para baixo, dependendo do tamanho dos agregados de células de partida. Tipicamente, 1 mL de sedimento de células empacotadas rendimento de 1 ml de extracto nuclear final. Todos os tampões devem ser arrefecida com gelo, e todos os passos devem ser realizados numa câmara fria ou em gelo, se um quarto frio adequado não estiver disponível.
- Isolamento de núcleos:
- Suavemente transferir células de um tamanho adequado (de 15 ou 50 ml) e graduado de tubo cónico de centrifugação a 1000 xg durante 10 min a 4 ° C.
- Retirar o sobrenadante, e medir o volume da célula embalada peldeixar. 1 ml de células embaladas corresponde a 3 x 10 ~ 8 de células HEK293.
- Adicionam-se 5 volumes de células embaladas de tampão A (10 mM de HEPES, pH 7,9, 1,5 mM MgCl2, 10 mM de KCl e recentemente adicionado 1 mM de DTT, 200 uM de PMSF, e 1: 1000 Protease Inhibitor Cocktail). Ressuspender o sedimento de células por pipetagem suave.
- Incubar no gelo por exatamente 10 min.
- Agregar as células a 1.000 xg durante 10 min a 4 ° C, e remover o sobrenadante.
- O tamanho do agregado de células deve aumentar até 2 vezes durante a incubação em tampão A.
- Ressuspender as células em dois volumes de células embaladas de tampão A, e transferir a suspensão de células para um homogeneizador de tamanho apropriado do tecido de Dounce. Se começar com menos de 2 ml de hemácias, usar um homogeneizador de 7 ml; para 2-4 ml hemácias, use um homogeneizador de 15 ml; e para 10 ou mais ml de hemácias, usar um homogeneizador de 40 ml.
- Homogeneizar a suspensão de células com o pilão de vidro solta do homogeneizador un Dounceaté 90% das células coram positivamente com 1% de azul de tripano.
- Transferir a suspensão para um tubo de centrífuga de 45 ml de alta velocidade e de rotação a 25000 xg durante 20 min a 4 ° C num rotor JA-17 ou semelhante (ver Tabela de Materiais / Equipamento).
- A partir do sedimento nuclear, remover o sobrenadante, que contém as proteínas ou as proteínas que vazam para fora do núcleo durante o fraccionamento citosólicas.
- Extraindo núcleos com sal:
- Adicionar tampão C (HEPES 20 mM, pH 7,9, 25% de glicerol, MgCl2 1,5 mM, EDTA 0,2 mM, e adicionada de fresco DTT 1 mM, 200 uM de PMSF, e 1: 1000 Protease Inhibitor Cocktail) para o sedimento nuclear; utilizar 2,5 ml Tampão C para cada 3 ml de começar volume globular (~ 1 x 10 9 células).
- Usando uma vareta de vidro ou uma pipeta, desalojar o sedimento nuclear a partir da parede do tubo e transferir toda a mistura para um homogeneizador de Dounce de um tamanho apropriado.
- Ressuspender os núcleos por homogeneização thmistura e com dois golpes de um pilão solto.
- Transferir a fracção nuclear ressuspendido para uma proveta fria. Escolha uma taça de modo a que a suspensão irá encher o copo de pelo menos 0,5 cm de profundidade.
- Para extrair as proteínas nucleares de cromatina ou outras estruturas insolúveis, gradualmente, aumentar a concentração de sal da suspensão de 0,42 M de NaCl por adição gota a gota de 5 M de NaCl, com agitação suave da suspensão com uma pipeta ou vareta de vidro em gelo; uma vez que todos os 5 M NaCl foi adicionado, a solução deve tornar-se muito viscoso ou semelhante a gel. Calcula-se o volume de 5 M de NaCl necessária para trazer a solução para uma concentração final de 0,42 M de NaCl de acordo com a seguinte fórmula:
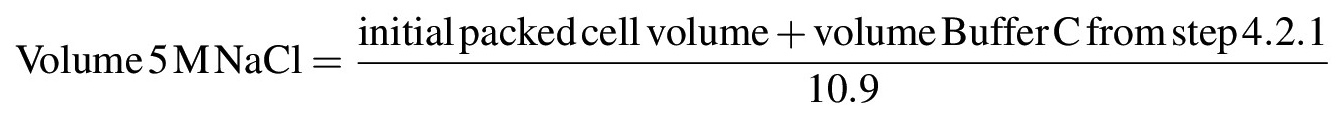
- Transferir cuidadosamente a suspensão viscosa em 10 ml tubos de policarbonato para um tipo de 70,1 Ti rotor (ou equivalente) ou 70 ml garrafas de policarbonato para um Tipo 45 Ti or rotor semelhante (ver Tabela de Materiais / Equipamentos). Seal firmemente com parafilme se usar 10 ml ou tubos com conjunto de tampa se estiver usando 70 ml.
- Lentamente balançar os tubos selados a 4 ° C durante 30 min, utilizando um nutator.
- Rodar as amostras em um Tipo 45 Ti ou rotor Ti 70.1 durante 30 minutos a 120.000 x g a 4 ° C.
- Transferir o sobrenadante para um tubo de plástico simples ou garrafa. Este sobrenadante é o extracto nuclear, e o pellet contém cromatina e outros detritos nucleares.
- Dividir o extracto nuclear em alíquotas convenientemente tamanho, congelá-lo em nitrogênio líquido, e armazená-lo a -80 ° C.
5. imunoafinidade purificação do INO80 humano ou INO80 subcomplexos
- Para descongelar extrato nuclear congelado, tubos lugar contendo o extrato na bancada ou rolo tubos entre as mãos até que o material congelado torna-se uma pasta. Em seguida, colocar os tubos em gelo ou no ambiente frio até que o extracto é completamente thawed.
- Transferir o extracto nuclear descongeladas a 10 ml de tubos de policarbonato de ultracentrífuga, e centrifugação a 100.000 xg durante 20 min a 4 ° C num rotor Ti tipo 70,1 ou equivalente para remover qualquer precipitado que se possa ter formado durante o ciclo de congelação-descongelação.
- Transferir o sobrenadante para um tubo cónico de 15 ml. Adicionar DTT fresco cocktail, PMSF, e Inibidor de Protease para concentrações finais de 1 mM de DTT, 200 uM de PMSF, e 1: 1000 Cocktail de Inibidor de Protease.
- Para preparar a agarose anti-FLAG para a imunopurificação, transferir 200 ul de 50% de pasta de contas de agarose anti-FLAG para um tubo de microcentrífuga de 1,5 ml usando uma pipeta de P200 ou semelhante, com uma ponta da qual a extremidade foi cortada com um bisturi limpa ou lâmina de barbear.
- Agregar as esferas por centrifugação numa microcentrífuga de bancada a 8000 xg durante 30 seg. Remover o sobrenadante e lavar as pérolas por ressuspensão das pérolas em 1 ml de tampão de Lys450 e sedimentar as esferas a 8000 xg durante 30 seg. Lave a beads mais duas vezes.
- Ressuspender as pérolas lavadas de agarose de anti-FLAG em cerca de 100 ul de extracto nuclear, utilizando uma pipeta de P200 ou semelhante de volume ajustável, com uma ponta da qual a extremidade foi cortada com um bisturi limpa ou lâmina de barbear e, usando a mesma ponta, transferência os grânulos em suspensão para o tubo de 15 ml contendo o extrato. Repita algumas vezes até que todos os grânulos foram transferidos para o tubo de 15 ml.
- Incubar a mistura de extracto / pérola durante 4 horas a 4 ° C com rotação lenta em um rotor de laboratório. OPCIONAL: Incluir benzonase a uma concentração de 25 unidades / ml para remover contaminantes de DNA.
- Recolhe-se o FLAG agarose por centrifugação a 1000 xg durante 5 min a 4 ° C.
- Ressuspender em 10 ml de Lys450 para lavar, incube 5 minutos a 4 ° C com agitação suave num nutator. Agregar as esferas a 1000 xg durante 5 min a 4 ° C.
- Ressuspender em 100-150 ul de Lys450 e contas de transferência para um tubo de microcentrífuga de 1,5 ml. Continue para enxaguar o tubo de 15 ml com 100-150 ul de Lys450 até que todos os grânulos foram transferidos para o tubo de microcentrífuga.
- Girar as esferas a 8000 xg durante 30 seg a 4 ° C numa microcentrífuga. Lavar três vezes mais com 1 ml de Lys450 e uma vez com 1 ml de tampão EB100 (10 mM de HEPES pH 7,9, 10% de glicerol, 100 mM de NaCl, 1,5 mM de MgCl2, 0,05% de Triton X-100, e recentemente adicionado DTT a 1 mM, PMSF 200 uM , e 1: 1000 Inibidor de Protease Cocktail).
- Para eluir proteínas ligadas, adicionar tampão EB100 200 mL contendo 0,25 mg / ml peptídeo FLAG 1x. Incubar 30 min a 4 ° C em cada uma nutator.
- Agregar as esferas a 8000 xg durante 30 seg a 4 ° C numa microcentrífuga. Transferir o sobrenadante, que contém o complexo INO80 eluido, para um tubo de microcentrifugação fresco.
- Repete-se a eluição mais quatro vezes, e reunir todos os sobrenadantes para um único tubo.
- Para remover todas as pérolas residual FLAG-agarose a partir da fracção de proteína eluída,passar o eluato através de uma coluna de spin vazio.
- Concentra-se a fracção de proteína eluída ~ 10 vezes utilizando um dispositivo de filtro de ultra-centrífuga (corte de peso molecular de 50.000).
- Para remover o peptídeo FLAG, passar a fracção de proteína concentrada sequencialmente através de duas colunas de dessalinização.
- Dividir a fracção de proteína purificada, dessalinizada em 20 mL alíquotas, congelada em azoto líquido, e armazenado a -80 ° C.
- Analisar a composição de subunidades de INO80 ou subcomplexos INO80 em géis corados com prata ou por transferência de western, e estimar as suas concentrações por western blotting semi-quantitativa usando preparações de subunidades INO80 recombinantes de concentração conhecida como padrões.
Resultados
A Figura 1 mostra um fluxograma que resume os procedimentos usados para gerar, purificar e caracterizar INO80 dependentes de ATP complexos de remodelação da cromatina humanos.
Como ilustrado nas Figuras 2 e 3, estes procedimentos permitem a geração de ambos os tipo INO80 e INO80 subcomplexos selvagens que faltam várias subunidades, permitindo assim análises bioquímicas subsequentes da contribuição destas subunidades que faltam...
Discussão
Estudos estruturais e funcionais de várias subunidades complexos mamíferos remodelação da cromatina de eucariotos superiores foram prejudicados pela dificuldade de preparar bioquimicamente quantidades úteis de tais complexos contendo subunidades mutantes ou falta certas subunidades completamente. Há um certo número de dificuldades técnicas: Em primeiro lugar, a manipulação genética em células de mamíferos tem sido tecnicamente difícil e demorado. Ao contrário das células de levedura, cujo genoma pode ser...
Divulgações
Os autores declaram que não têm interesses financeiros concorrentes.
Agradecimentos
Trabalho no laboratório dos autores é apoiado por uma bolsa do Instituto Nacional de Ciências Médicas Gerais (GM41628) e por um subsídio ao Instituto Stowers de Pesquisa Médica do Nelson Fundo de Investigação Médica Helen na Fundação Comunidade Maior Kansas City.
Materiais
| Name | Company | Catalog Number | Comments |
| Dulbecco's Modified Eagle Medium | Cellgro | 10-013-CV | |
| Glutamax-I (stablized glutamine) | Life Technologies | 35050-079 | |
| Fetal Bovine Serum | SAFC | 12176C | |
| FuGENE6 transfection reagent | Promega | E2312 | |
| Hygromycin B, sterile in PBS | AG Scientific | H-1012-PBS | |
| pcDNA5/FRT vector | Life Technologies | V6010-20 | |
| Flp-In HEK293 cells | Life Technologies | R780-07 | |
| pOG44 Flp-Recombinase Expression Vector | Life Technologies | V600520 | |
| EZview Red ANTI-FLAG M2 Affinity Gel | Sigma | F2426 | |
| calf serum | SAFC | 12138C | |
| TARGETplus SMARTsiRNA pool | Dharmacon / Thermo Scientific | various | |
| 5x siRNA resuspension buffer | Dharmacon / Thermo Scientific | #B-002000-UB-100 | |
| Lipofectamine RNAiMAX | Life Technologies | 13778 | |
| Opti-MEM Reduced Serum Medium | Life Technologies | 51985-091 | |
| PBS | Cellgro | 45000 | VWR |
| TrypLE (trypsin) | Life Technologies | 12604 | |
| 1x FLAG Peptide | Sigma | F3290 | |
| Micro Bio-Spin Chromatography Column | Bio-Rad | 737-5021 | |
| Amicon Ultra Centrifugal Filter Device (50k MWCO) | Amicon | UFC805024 | Fisher Scientific |
| Zeba Desalting Columns | Thermo Scientific | 89882 | |
| Anti-FLAG M2 antibody, mouse | Sigma | F3165 | |
| Anti-FLAG M2 antibody, rabbit | Sigma | F7425 | |
| Protease Inhibitor Cocktail | Sigma | P8340 | |
| benzonase | Novagen | 70664 | |
| Equipment | |||
| JS-4.2 rotor in a J6 centrifuge | Beckman-Coulter | 339080 | |
| JA-17 rotor | Beckman-Coulter | 369691 | |
| 10 ml polycarbonate tubes | Beckman-Coulter | 355630 | |
| 70 ml polycarbonate bottles | Beckman-Coulter | 355655 | |
| Type 45 Ti rotor | Beckman-Coulter | 339160 | |
| Type 70.1 Ti rotor | Beckman-Coulter | 342184 | |
| BD Clay Adams Nutator Mixer | BD Diagnostics | 15172-203 | VWR |
| Glas-Col Tube/Vial Rotator | Glas-Col | 099A RD4512 | |
| PCR thermal cycler PTC 200 | MJ Research | PTC 200 | |
| roller bottle incubator | Bellco biotechnology | 353348 | |
| Immobilon-FL Transfer Membrane 7 x 8.4 | Millipore | IPFL07810 | |
| lubricated 1.5 ml microcentrifuge tubes | Costar | 3207 | |
Referências
- Clapier, C. R., Cairns, B. R. The biology of chromatin remodeling complexes, Annual Review of Biochemistry. 78, 273-304 (2009).
- Szerlong, H., Hinada, K., Viswanathan, R., Erdjument-Bromage, H., Tempst, P., Cairns, B. R. The HSA domain binds nuclear actin-related proteins to regulate chromatin-remodeling ATPases. Nature Struct. Mol. Biol. 15 (5), 469-476 (2008).
- Shen, X., Mizuguchi, G., Hamiche, A., Wu, C. A chromatin remodelling complex involved in transcription and DNA processing. Nature. 406 (6795), 541-544 (2000).
- Shen, X., Ranallo, R., Choi, E., Wu, C. Involvement of actin-related proteins in ATP-dependent chromatin remodelling. Mol. Cell. 12, 147-155 (2003).
- Chen, L., et al. Subunit organization of the human INO80 chromatin remodeling complex: an evolutionarily conserved core complex catalyzes ATP-dependent nucleosome remodeling. Journal of Biological Chemistry. 286 (13), 11283-11289 (2011).
- Sauer, B. Site-specific recombination: developments and applications. 5 (5), 521-527 (1994).
- Gorman, S., Fox, D. T., Wahl, G. M. Recombinase-mediated gene activation and site-specific integration in mammalian cells. Science. 251 (4999), 1351-1355 (1991).
- Jin, J., et al. A mammalian chromatin remodeling complex with similarities to the yeast INO80 complex. Journal of Biological Chemistry. 280 (50), 41207-41212 (2005).
- Cai, Y., et al. YY1 functions with INO80 to activate transcription. Nature Structural and Molecular Biology. 14 (9), 872-874 (2007).
- Yao, T., et al. Distinct Modes of Regulation of the Uch37 Deubiquitinating Enzyme in the Proteasome and in the Ino80 Chromatin-Remodeling Complex. Mol.Cell. 31 (6), 909-917 (2008).
- Brizzard, B. L., Chubet, R. G., Vizard, D. L. Immunoaffinity purification of FLAG epitope-tagged bacterial alkaline phosphatase using a novel monoclonal antibody and peptide elution, Biotechniques. 16 (4), 730-735 (1994).
- Barker, K. . At the Bench: A Laboratory Navigator. 10, 207-245 (2005).
- Dignam, J. D., Lebovitz, R. M., Roeder, R. G. Accurate transcription initiation by RNA polymerase II in a soluble extract from isolated mammalian cell nuclei. Nucleic. Acids. Res. 11 (5), 1475-1489 (1983).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados