Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Methodologien für das Studium
In diesem Artikel
Zusammenfassung
This study presents the development of reproducible methodologies to study biofilm inhibitors and their effects on Bacillus subtilis multicellularity.
Zusammenfassung
This work assesses different methodologies to study the impact of small molecule biofilm inhibitors, such as D-amino acids, on the development and resilience of Bacillus subtilis biofilms. First, methods are presented that select for small molecule inhibitors with biofilm-specific targets in order to separate the effect of the small molecule inhibitors on planktonic growth from their effect on biofilm formation. Next, we focus on how inoculation conditions affect the sensitivity of multicellular, floating B. subtilis cultures to small molecule inhibitors. The results suggest that discrepancies in the reported effects of such inhibitors such as D-amino acids are due to inconsistent pre-culture conditions. Furthermore, a recently developed protocol is described for evaluating the contribution of small molecule treatments towards biofilm resistance to antibacterial substances. Lastly, scanning electron microscopy (SEM) techniques are presented to analyze the three-dimensional spatial arrangement of cells and their surrounding extracellular matrix in a B. subtilis biofilm. SEM facilitates insight into the three-dimensional biofilm architecture and the matrix texture. A combination of the methods described here can greatly assist the study of biofilm development in the presence and absence of biofilm inhibitors, and shed light on the mechanism of action of these inhibitors.
Einleitung
Mehrzelligen Bakteriengemeinschaften spielen eine bedeutende Rolle in natürlichen und anthropogenen Umgebungen und kann vorteilhaft oder sehr schädlich sein. Diese mehrzelligen Kolonien werden als Biofilm bekannt ist, wobei die einzelnen Zellen in einer selbst hergestellten extrazellulären polymeren Substanzen (EPS) Matrix eingebettet sind. Die EPS haften stark die Zellen an der Oberfläche sie besiedeln. Sie dienen als Schutzschild gegen mechanische und chemische Kräfte und schaffen einen engen Kontakt zwischen benachbarten Zellen, die Erleichterung zellulären Kommunikation 1. Ein Biofilm kann als differenzierte Gemeinschaft betrachtet werden, in denen Zellen stark regulierten verwenden, Prozesse orchestriert ihre Aktivitäten innerhalb der Gemeinschaft zu koordinieren, sowie über Artgrenzen 2-5. Der Übergang von einer planktonischen, wird freilebenden Modus des Wachstums eines Biofilms Zustand oft mit Entwicklungsprozessen verbunden. Ein gutes Beispiel ist die Gram-positiven Bodenbakteriums Bacillus subtilis, und daher ein undomesticated Stamm dient als robuster Organismus Modell die Entwicklungsstadien, die zu Biofilmbildung zu untersuchen. In diesem Bakterium, sich in auffälliger vielzelligen Strukturen bewegliche Zellen organisieren , die spezielle Aufgaben 4 auszuführen. Eine Gruppe von Zellen, die matrix-Erzeuger sekretieren Exopolysaccharide 6, das Amyloid - Protein tasa 7,8, und die Oberfläche Hydrophobizität Protein BSLA 9,10; von denen alle mit bei der Montage der EPS 11-13.
Angesichts der Fülle von Biofilmen in natürlichen und anthropogenen Nischen und die vermeintliche tödlichen Schaden, den sie verursachen können, gibt es eine dringende Notwendigkeit, Wege zu finden, ihre Bildung zu verhindern. Kleine Molekül-Inhibitoren können in der Entdeckung neuer Regulationswege unterstützen, Enzyme und Struktur der Biofilmbildung beteiligten Proteine und damit fördern Einblicke in die komplexen Prozesse der vielzelligen Gemeindeversammlung. Als B. subtilis ist ein gut untersuchtes Modell für BioFilmbildung 14,15, kann es verwendet werden , um die Wirkungen verschiedener Biofilm Inhibitoren zu bewerten. Diese Studie befasst sich vier grundlegende Methoden, die für die Bewertung der Reaktion von Biofilmen zu kleinen Molekül-Inhibitoren sind der Schlüssel. Erstens, um sicherzustellen, dass diese Inhibitoren haben einen Biofilm-spezifischen Ziel, die Trennung der planktonischen Wirkung auf das Wachstum von der Wirkung auf die Bildung von Biofilmen ist kritisch. Die meisten antibakterielle Mittel Zielzellen in ihre planktonischen Wachstumsphase, sondern Moleküle, die die Biofilm Lebensstil Ziel sind selten. Zusätzlich kann , wie Moleküle , die nicht toxisch nicht planktonischen Wachstum beeinflussen, können sie den Selektionsdruck begünstigt antibiotische resistente Mutanten 16 reduzieren. Wenn zum Beispiel Biofilme mit D-Aminosäuren oder bestimmten anderen Zellwand-störenden Molekülen behandelt werden, werden sie entweder gestört oder zerlegt werden , aber diese Inhibitoren leicht nur planktonischen Wachstums 12,17 beeinflussen. Im Gegensatz dazu beeinträchtigen viele Antibiotika dramatisch Planktonwachstum, mit lenig oder keine Wirkung auf die Bildung von Biofilmen 17.
Zweitens, eine konsistente und robuste experimentellen Rahmen zur Gründung der Wirkung der kleinen Moleküle zu untersuchen ist von entscheidender Bedeutung. Wir haben beobachtet, dass die aktive Konzentrationsbereich von kleinen Molekül-Inhibitoren war empfindlich auf die Vorkultur Bedingungen und zu den experimentellen Aufbau die Wirkung dieser kleinen Molekül-Inhibitoren zu untersuchen verwendet. Verschiedene Berichte, insbesondere solche B. das Studium subtilis zeigten Variationen in dem Konzentrationsbereich , bei der D-Aminosäuren , die Bildung von Häutchen hemmen - die schwimmenden bakteriellen Biofilmen 12,17-19. Die hier vorgestellten Ergebnisse deuten darauf hin , dass die folgenden Faktoren für Unterschiede in der aktiven Konzentrationsbereich ausmachen: Die Vorkultur Bedingungen (logarithmische 12,17 im Vergleich zu späten stationären Wachstumsphase 20), das Wachstumsmedium in der Vorkultur Zustand verwendet (reich, undefined [Luria Broth, LB] gegen definiert [Natriumglutamatglycerin, MSgg]), das Verhältnis der Inokulation und insbesondere die Entfernung des Vorkulturmediums vor der Inokulation. Die Temperatur des statischen Pellicle Wachstum zeigten eine weniger wichtige Rolle bei der Aktivitätsbereich des kleinen Molekül-Hemmstoff D-Leucin, einer repräsentativen D-Aminosäure in dieser Studie verwendet.
Schließlich, nachdem die Biofilme mit spezifischen Biofilm-Inhibitoren behandelt, robust und informative Verfahren erforderlich sind, um die Auswirkungen dieser Inhibitoren auf Biofilm Fitness zu charakterisieren. Hier sind zwei Methoden, unabhängig die Wirkung von niedermolekularen Inhibitoren kennzeichnen, sind im einzelnen beschrieben: (1) Die Wirkung auf die einzelnen Zellen innerhalb eines Biofilms Kolonie und ihre Resistenz gegen antimikrobielle Mittel. Die Zellen in Biofilmen sind in der Regel resistent gegen Antibiotika im Vergleich zu den Bakterien freilebenden 21-23. Während dieses Phänomen multifaktoriell ist, wurde die Fähigkeit der EPS - Antibiotikum Penetration zu reduzieren oft als attraktiver Erklärung 24 betrachtet . Diese Methode bewertet das Überleben der vorher festgelegten Biofilm-Zellen nach der Exposition gegenüber antibakterielle Substanzen. (2) Die Wirkung auf die Biofilm-Kolonie-Architektur von der großen zur kleinen Maßstab. Biofilm-Kolonien werden durch ihre dreidimensionale Struktur und die Anwesenheit der EPS gekennzeichnet. Verwendung von Rasterelektronenmikroskopie, Veränderungen in der Zellmorphologie, Biofilm Koloniestruktur und die Architektur und die Abundanz der EPS kann aus der großen (mm) sichtbar gemacht werden, um die kleinen Maßstab (um).
Protokoll
1. Die Beurteilung der Wirkung von niedermolekularen Inhibitoren auf Pellicle und Biofilm Koloniebildung
- Bereiten Sie eine 2x Lösung definierter Biofilm-induzierenden MSgg Medium 25 ohne Calciumchlorid und Eisen (III) -chlorid - Hexahydrat. Nach Filtersterilisation, das Calciumchlorid hinzu. Das Medium bereit ist, direkt zu verwenden, oder es kann in der Dunkelheit bei 4 ° C gelagert werden.
- Bereiten Sie die 1x MSgg Verdünnungs am Tag des Experiments.
- Verdünne die 2x MSgg Medium mit sterilem destilliertem Wasser (Häutchen) oder sterile 3% heißem (80 ° C) Agar (Biofilme) und fügen Eisen (III) -chlorid-Hexahydrat auf eine Endkonzentration von 50 uM (Häutchen) oder 250 & mgr; M bis 1 x ( Biofilme). Fügen Sie Antibiotika oder kleine Molekül-Inhibitoren auf die gewünschte Konzentration und gut mischen. Um beispielsweise eine Endkonzentration von 0,5 mM D-leucin in 30 ml zu erhalten, Häutchen oder Biofilme zu etablieren, fügen 196,6 ul einer 76,3 mM (10 mg / ml) D-Leucin-Stammlösung.
NEINTE:. Die endgültige 1x MSgg Zusammensetzung in Tabelle 1 beschrieben , ist im Vergleich zu dem Originalrezept 25 enthielt das Medium 50 ug / ml Threonin und die Eisenkonzentration Biofilm - Kolonien auf festem Medium wachsen MSgg wurde 2,5 - fach erhöht , um die faltigen Koloniemorphologie zu optimieren .
- Verdünne die 2x MSgg Medium mit sterilem destilliertem Wasser (Häutchen) oder sterile 3% heißem (80 ° C) Agar (Biofilme) und fügen Eisen (III) -chlorid-Hexahydrat auf eine Endkonzentration von 50 uM (Häutchen) oder 250 & mgr; M bis 1 x ( Biofilme). Fügen Sie Antibiotika oder kleine Molekül-Inhibitoren auf die gewünschte Konzentration und gut mischen. Um beispielsweise eine Endkonzentration von 0,5 mM D-leucin in 30 ml zu erhalten, Häutchen oder Biofilme zu etablieren, fügen 196,6 ul einer 76,3 mM (10 mg / ml) D-Leucin-Stammlösung.
- Nach dem Erstarren des Agars, trockne die festen MSgg Platten in einer biologischen Haube für 30-45 min vor der Inokulation.
- Um spezifische Inhibitoren auszuwählen , die mit den Mechanismen der Pellikelbildung (Abbildung 1) interferieren, auszuschließen , dass die verwendeten Konzentrationen beeinflussen planktonischen und statische Wachstum.
- Bestimmen planktonischen Wachstums (Zunahme der optischen Dichte über die Zeit in Flüssigkultur) in einem einfachen Wachstumskurve, die durch die optische Dichte bei 600 nm, bis die stationäre Wachstumsphase jede Stunde gemessen wird.
- Um zu bestätigen, daß die gemessene Trübung Kultur lebende Zellzahlen darstellt, bestimmen die Anzahl der koloniebildenden Einheiten (CFU)von Zellen in der planktonischen Wachstumsphase aus einer Schüttelkultur nach mehreren Zeitpunkten.
- Um die Wirkung von kleinen Molekül-Inhibitoren auf die statischen Häutchen Wachstum, Ernte Zellen am Ende eines 3-tägigen Inkubation bei 23 ° C von einer 24-Well-Zellkultur zu bewerten und unter den gleichen Bedingungen beimpft wie in den Abschnitten 1.7-1.9 beschrieben und bestimmen die CFU. Für diese Steuerung verwenden , um ein Häutchen-defizienten Stamm, der die Operons für die extrazelluläre Matrixkomponenten (dh B. subtilis Δ epsH, Δ Tasa) fehlt.
HINWEIS: Dieser Stamm ist in der Lage unter statischen Bedingungen wachsen, aber im Gegensatz zu einem Pellicle-bildenden Wildtyps ist es einen Mangel an der Fähigkeit, die Flüssigkeit-Luft - Grenzfläche zu schwimmen, wo das Wachstum durch erhöhte Sauerstoffgehalt 26 begünstigt wird. Somit ist diese extrazelluläre Matrix- und Häutchen-defizienten Stamm ist eine empfohlene Referenzstamm Wachstum unter statischen Bedingungen zu beurteilen.
HINWEIS: Für die spezifische Exausreichend der nicht-kanonische D-Aminosäure D-Leucin unten beschrieben, eine Wirkung auf planktonischen und statische Wachstum bei Konzentrationen , die mit Pellikelbildung gestört wurde 12,17 ausgeschlossen. Die Methoden planktonischen und statische Wachstum zu bestimmen , sind im Detail 17 beschrieben.
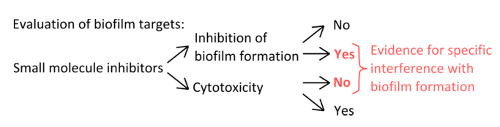
Abbildung 1. Konzeptübersicht für die Identifizierung eines robusten Versuchsaufbau die spezifische Hemmung der Biofilmbildung zu bewerten. Die Auswahlkriterien für kleine Molekül - Inhibitoren , die spezifische Interferenz mit Biofilmbildung zeigen , ohne ausgeprägte Wirkung auf Planktonwachstum. Bitte klicken Sie hier , um eine größere Version zu sehen dieser Figur.
- Streak aus B. subtilis von einem -80 ° C Lager (LB culture von 10 9 Zellen / ml in 20% Glycerin eingefroren) einzelne Kolonien auf einem LB-1,5% Agar - Platte mit einer sterilen Spitze oder Applikatorstäbchen zu isolieren.
- Wachsen über Nacht bei 30 ° C.
- Kritischste Schritt: Für eine robuste Pellicle Hemmung durch die nicht-kanonische D-Aminosäuren wie D-Leucin, zu wachsen nahm eine einzelne Kolonie von der LB-1,5% Agar - Platte in 3 ml LB - Brühe bei 37 ° C für 4 h in einem Schüttelinkubator (Schüttelgeschwindigkeit 200 Umdrehungen pro Minute). Ersetzen die LB-Brühe mit Biofilm-induzierenden MSgg Medium vor der Beimpfung durch Zentrifugieren 1,5 ml Starterkultur für 4 min bei 6000 xg, sorgfältig den Überstand zu entfernen und Resuspendieren des Pellets in 1,5 ml MSgg Medium. Der Rest der Kultur kann verworfen werden.
Wichtig: Um die Robustheit des Systems zu gewährleisten, die optische Dichte bei 600 nm (OD 600) der gewaschenen Starterkultur sollte zwischen 0,6 und 1 liegen. - Während des Wachstums der Starterkultur, bereiten eine 12-Well-Zellkultur Multidish Platte mit deg 3 ml MSgg Medium ohne oder mit einem Konzentrationsbereich des kleinen Molekül - Hemmstoff (beispielsweise 0,3, 0,5, 1 mM D-Leucin - 17). Um Randeffekte auszuschließen, verteilen sich die Position der verschiedenen Konzentrationen über die Multidish Platte. Alternativ können Sie auch 24-Well-Zellkulturplatten Multidish 1,5 ml MSgg Medium enthält.
- Beimpfen die Vertiefungen der 12-Well-Zellkultur Multidish-Platte mit 3 ul der gewaschenen Starterkultur (1: 1000 Verdünnung).
HINWEIS: Eine niedrigere Verdünnungsverhältnis, das heißt, 1: 500 verwendet werden. Dies verringert die Entwicklungszeit der Häutchen. - Wachsen die Häutchen bei 23 ° C unter statischen Bedingungen für drei Tage. Nicht die Häutchen in dieser Zeit bewegen, wie es die endgültige Oberflächenmorphologie der Pellicle beeinflussen können.
- Erwerben Sie Bilder mit einem Binokular und homogene Belichtung von Blitz. Alternativ können Sie auch ein Bild von den Häutchen mit einer hochauflösenden Kamera. Um zu vermeiden, durch inconsis verursachte ArtefakteZelt Licht Winkel und Schatten, nehmen Top-down-Bilder mit der Kamera auf einem Stativ befestigt ist und eine weiche und große Lichtquelle bei 45 ° von beiden Seiten nutzen.
ANMERKUNG: Eine alternative Methode B. zu studieren subtilis Vielzelligkeit ist der Biofilm - Kolonie - Assay auf festem, Biofilm-induzierenden MSgg Medium. Wie Häutchen, ermöglicht dieser Test die Untersuchung von Raum-Zeit-Prozesse. Sobald der aktive Bereich von niedermolekularen Inhibitoren bestimmt wird, deren Wirkung auf die Koloniebildung Biofilm untersucht werden. - Zur Biofilm-Kolonien wachsen, punktsymmetrisch 1,5 ul der ungewaschenen Vorkultur (Schritt 1.7) auf der getrockneten MSgg 1,5% Agar-Platte mit Hilfe einer Schablone, - 4 Tropfen pro Petrischale von 8,5 cm Durchmesser. Lassen Sie die Tropfen auf die Platte adsorbieren, bevor sie in Bewegung.
HINWEIS: Die Vorlage hilft, eine gleichmäßige Verteilung der Biofilm Kolonien innerhalb des Gebiets zu erhalten, wo die Zellen gezüchtet werden. Um die Vorlage zu bereiten, die Gesamtfläche des Wachstums an der ursprünglichen Skalen ziehen, teilen sie gleich Sekteors und die Mitte markieren. Für eine Runde Petrischale von 8,5 cm Durchmesser, weist diese 14 cm 2 zu einem Biofilm Kolonie. - Inkubiere die Platten bei 30 ° C für drei Tage. Während dieser Zeit Biofilm Kolonien entwickeln und bilden eine dreidimensionale, faltige Struktur.
- Nehmen Sie Bilder wie in Schritt 1.11.
2. Ethanol-Resistenztest
- Wachsen Biofilme wie in den Schritten 1,1-1,7 und 1,12-1,14 beschrieben.
- Nach 68 h Wachstum bei 30 ° C, schneiden die Biofilm-Kolonien in zwei gleiche Teile mit Hilfe einer Rasierklinge und der Schablone.

Abbildung 2. Beispiel experimentelle Design der Beständigkeit von Biofilm Kolonie Zellen zu Sterilisationsmitteln. (A) Vorlage zur Beurteilung für die gleichmäßige Verteilung der Biofilm Kolonien in einer Petrischale verwendet und zum Schneiden. (B)Top-down-Bilder von unbehandelten Wildtyp-Biofilm für 68 Stunden auf einer soliden, definiert Biofilm-induzierenden MSgg Medium bei 30 ° C gezüchtet. Die Erweiterung zeigt, wie ein Biofilm Kolonie kann in zwei gleiche Hälften geschnitten werden. (C) Die zwei gleiche Hälften Biofilm gleich behandelt (Kontrolle, PBS) oder mit entweder PBS oder Sterilisationsmittel und wie beschrieben verarbeitet. Maßstabsbalken:. 1 cm Bitte hier klicken um eine größere Version dieser Figur zu sehen.
- Heben Sie jede Hälfte des Biofilms Kolonie von der Agarplatte mit einem kleinen Spachtel und es in ein 1,5-ml-Mikroröhrchen mit 500 ul phosphatgepufferter Salzlösung (PBS) bewegen. Falls erforderlich, kratzen die verbleibenden Zellen von der Platte und übertragen sie auch auf das Reaktionsgefäß.
HINWEIS: Die zweite Hälfte des Biofilms Kolonie unterschiedlich behandelt wird, je nachdem, ob es die Steuerung oder den Widerstand gegen Steri zu testenlizing Mittel. - Für die Steuerung, wie in Schritt 2.3 die zweite Hälfte des Biofilms Kolonie in 500 & mgr; l PBS inkubiert. Zur Beurteilung der Beständigkeit gegen Sterilantien, übertragen die zweite Hälfte des Biofilms Kolonie zu 500 ul 50% (v / v) Ethanol.
HINWEIS: Alternative Mittel wie Natriumhypochlorit Sterilisieren verwendet werden. Für alle Sterilisationsmittel verwendet werden, die aktive Konzentration und Inkubationszeit in einem Vorversuch bestimmen. - Inkubieren der Biofilm-Kolonien für 10 min auf der Tisch bei Raumtemperatur.
- Zentrifugieren Sie die Biofilm Kolonien für 5 min bei 18.000 xg und entfernen Sie den Überstand vorsichtig mit einer Pipette. In 300 ul PBS.
- Beschallen die Zellen leicht (Amplitude 10%, Puls 5 sec) mit der Mikrospitze eines Beschallungsgerät.
HINWEIS: Die Beschallungsenergie auf separaten Biofilm Aggregate ausreichend sein. Aber auch harte Beschallung kann die Zellen lysieren. Bestätigen Sie im Voraus durch Lichtmikroskopie, dass die Beschallungsenergieverwendet, um die Zellen nicht lysieren, und dass alle Aggregate gelöst sind. - Hinzufügen, 700 & mgr; l PBS zu einem Endvolumen von 1 ml. Führen Sie eine serielle Verdünnung (bis 10 -7) in PBS und verteilt 100 ul 3 Verdünnungen auf einem LB-1,5% Agar - Platte unter Verwendung von sterilen Glasperlen.
HINWEIS: Die optimalen Verdünnungen plattiert werden sollten in einem Vorversuch bestimmt werden, wie dies hängt von der Menge von Zellen im Biofilm Kolonie von Interesse und die Überlebensrate der Zellen in Reaktion auf das Sterilisationsmittel. - Inkubieren Sie die Platten über Nacht bei 30 ° C, die CFU zählen und KBE / ml zu bestimmen. Von der letzten KBE / ml jeweils die Hälfte Biofilm Kolonien, berechnen den Prozentsatz der Überlebenden.
HINWEIS: Bei durchgeführt und analysiert, wie beschrieben, die beiden Hälften des Steuer Biofilm Kolonie und der unbehandelte im Vergleich zu mit Ethanol behandelten Hälfte der unbehandelten Biofilm Kolonie sollten Unterschiede von unter 10% in Lebendzellanzahl ergeben, die Symmetrie oder den Widerstand der Kolonie Verifizieren , welche nach Süctively. Alternativ können die Ergebnisse insgesamt CFU dargestellt werden. Die Zellzahlen der Kontrolle und unbehandeltem Biofilm Kolonie sollte in der gleichen Größenordnung bleiben. Im Gegensatz dazu sind die Zellzahlen der mit Ethanol behandelten Hälfte eines kleinen Moleküls behandelten Biofilms Kolonie erwartet um mindestens zwei Größenordnungen fallen für eine erhöhte Empfindlichkeit gegenüber dem Sterilisationsmittel zu beanspruchen.
3. Biofilm Colony Probenvorbereitung für die Rasterelektronenmikroskopie
- Biofilm wachsen Kolonien wie in den Schritten 1,1-1,7 und 1,12 bis 1,14 beschrieben.
- Eine frische Charge von 2% (v / v) Glutaraldehyd, 3% (v / v) Paraformaldehyd-Lösung in 100 mM Natriumcacodylat, 5 mM Calciumchlorid-Puffer, pH 7,3. Bereiten Sie 5 ml Fixiermittel für jede Petrischale von 8,5 cm Durchmesser.
ACHTUNG: Glutaraldehyd und Paraformaldehyd sind gefährlich. Behandeln Sie sie mit Sicherheitsausrüstung innerhalb einer chemischen Haube. Entsorgen Sie die Lösungen und die kontaminierten Materialien zur hazardous Abfälle. - Vorsichtig die Fixiermittel zu den Biofilm Kolonien, ohne direkt auf der Biofilme zu verzichten.
HINWEIS: Aufgrund des hydrophoben Charakters des Biofilms Kolonie, langsam die Kolonien aus dem Agar zu lösen und zu schweben beginnen. - Dichtung sorgfältig die Platten mit einem Streifen Parafilm. Inkubieren auf einem Rotationsschüttler für 2 Stunden bei Raumtemperatur und anschließend die Platten auf 4ºC übertragen über Nacht.
- Am nächsten Tag, entfernen Sie vorsichtig die Flüssigkeit mit einem mit einer Vakuumpumpe verbunden Glaspipette Pasteur.
- Vorsichtig 10 ml 100 mM hinzufügen Natriumcacodylat, 5 mM Calciumchlorid-Puffer des Biofilms und Inkubation für 5 min zu waschen. Entfernen Sie vorsichtig die Flüssigkeit mit dem Pasteur Glaspipette aus der Ecke der Platte durch vorsichtiges Pipettieren Beschädigung des Biofilms und fügen Sie frische Waschlösung zu vermeiden. Wiederholen Sie einmal diesen Schritt.
- Für die Entwässerung der Biofilm Kolonien, gehen Sie mit den folgenden Schritten: 2x 5 min in ddH 2 O; 2x 20 min in 30% Ethanol; 2x 20 min in 50% Ethanol; 2x 20 min in 70% Ethanol; 2x 20 min in 96% Ethanol; 2x 30 min in 100% -igem Ethanol.
- 15 ml Flüssigkeit für jede Petrischale von 8,5 cm Durchmesser in jedem Schritt und entfernen Sie die Flüssigkeit sorgfältig nach jeder Inkubation.
- Verwenden eines von zwei unterschiedlichen Verfahren zum Trocknen der Proben aus Ethanol.
- Für Lufttrocknung von Ethanol:
- Schneiden Sie ein Zellulosefilterpapier (Durchmesser von 9 cm) in den Quartalen. Kurz ein Viertel in 100% Ethanol versenken und dann auf sie sorgfältig zu übertragen, eine Floating-Biofilm-Kolonie. Setzen Sie den nassen Filterpapier in einer Petrischale mit einem Filterpapier ausgekleidet. Decken Sie die Petrischale und lassen Sie den Biofilm Kolonien über Nacht trocknen in einer chemischen Haube.
- Zum kritischen Punkt (CP) -TROCKENTUNNEL Kohlendioxid (CO 2) als Übergangsflüssigkeit unter Verwendung:
- Füllen Sie 75% des kritischen Punktes Trockenmaschine Kammer mit 100% Ethanol. Übertragen Sie die Proben in einen Halter, um jede Probe in die eigene chamber. Wenn nötig, schneiden Sie den Biofilm mit einer Schere in kleinere Stücke. Lassen Sie die Unterwasserproben in Ethanol während der gesamten Behandlung. Dann übertragen Sie den Halter in die Kammer und die Kammer fest schließen.
- Kühlen der Kammer auf 7 ° C und Rühren beginnen. Füllen Sie die Kammer vollständig mit flüssigem CO 2. Während einer 7 min Inkubationszeit, lassen die Ethanolmischung mit dem CO 2. Dann entlädt 25% der Lösung.
HINWEIS: Die Kammer unter dem Niveau der Probe nicht leer. - Wiederholen Sie Schritt 3.8.2.2 viermal.
- Wiederholen Sie Schritt 3.8.2.2 fünfmal mit einer Inkubationszeit von 5 min nur. Schließlich sollte das Ethanol vollständig durch CO 2 ersetzt werden.
- Während der letzten Runde leer nur 5% der Kammer. Drehen Sie das Rühren und die Kühlung ab. Starten Sie die Kammer auf 42 ° C erhitzt wird. Bei einer Temperatur von 31,1 ° C und einem Druck von 73,9 bar, die Flüssigkeit CO 2 erreicht seinen kritischen Punkt, den Zustand , in dem der gasförmige phase hat die gleiche Dichte wie die Flüssigphase des Lösungsmittels 27. Sobald die Temperatur 42 ° C erreicht, für 10 min inkubieren. Bei 42 ° C besteht das CO 2 in der Kammer als überkritisches Gas.
HINWEIS: Ständig den Druck der Kammer überprüfen. Der Druck sollte nicht mehr als 120 bar bei 42 ° C. - Beginnen Sie langsam das Gas freizusetzen mit kontinuierlichen Erwärmung. Dies hält die Proben in der CO 2 -Gas - Phase und verhindert die Verformung der Probenmorphologie durch die Flüssigkeitsoberflächenspannung. Stellen Sie den Durchflussmesser bis 5 l / h durch eine Feinabstimmung das Dosierventil steuert das Messgerät. Warten, bis der gesamte Druck in der Kammer freigesetzt wird. Jetzt die Kammer geöffnet und die Proben vorsichtig aus der Halterung entfernen.
- Für Lufttrocknung von Ethanol:
- Mantel ein Elektronenmikroskopie Stummel mit Kohlenstoffband. Mit Hilfe einer Pinzette, übertragen sorgfältig die Biofilm Kolonien auf den Stummel. Schließen Sie jede Kolonie mit dem Stummel durch eine dünne Brücke aus dem Auto Hinzufügenbon Band, die zur Ladungsableitung unter dem Elektronenstrahl von entscheidender Bedeutung ist. In diesem Stadium behandeln die Biofilm Kolonien mit Vorsicht, da sie sehr zerbrechlich sind. Speichern der Proben in einem Exsikkator für mindestens 24 h oder bis zur Untersuchung.
- Am Tag der Untersuchung mit dem Rasterelektronenmikroskop, Sputter-Beschichtung der Biofilm-Kolonien für 2 min in einem Winkel von 60º in einer Gold-Palladium-Sputter-Beschichtungsvorrichtung. Wiederholen Sie diesen Schritt zweimal und drehen Sie die Proben von 120 ° dazwischen. Am Ende, Sputter-coat die Proben einmal für 3 min von oben. Die 20 nm dünnen Schicht Gold-Palladium verbessert die Leitfähigkeit und erhöht den Kontrast der Probe für die Bildgebung in der SEM.
- Speichern der Proben in einem Exsikkator 28 die Rehydratisierung der Probe zu vermeiden , bis die Abbildung mit einem Rasterelektronenmikroskop 29,30.
Ergebnisse
Das Häutchen - Test ist eine Methode , die stark reguliert und dynamische Prozesse von B. zu studieren subtilis Vielzelligkeit. Außerdem wird der Pellicle-Assay eine Reihe von entweder pre-starter Bedingungen oder kleine Molekülkonzentrationen in einer einzigen Zellkultur Multidish Platte in einem Experiment zum Testen geeignet. Jedoch B. subtilis Pellikelbildung ist empfindlich für die Vorkultur Bedingungen (zB Wachstumsmedium der Vorkultur und der Wachstumsphase), der Inokulatio...
Diskussion
Bacillus subtilis Formen robust und stark strukturierte Biofilmen sowohl in flüssiger (Häutchen) und auf festem Medium (Kolonien). Daher dient sie als ideale Modellorganismus der Wirkungsweise von spezifischen Inhibitoren Biofilm zu charakterisieren. Auf festen Medien bilden Zellen mehrzelliger Strukturen mit charakteristischen Eigenschaften, die nicht offensichtlich in Häutchen sind, wie Falten von der Mitte zum Rand hin ausstrahlen. So sind Häutchen und Kolonien ergänzende Systeme zu studieren B. sub...
Offenlegungen
The authors have nothing to disclose.
Danksagungen
Electron microscope imaging was conducted at the Electron Microscopy Unit of the Weizmann Institute of Science, supported in part by the Irving and Cherna Moskowitz Center for Nano and Bio-Nano Imaging. This research was also supported by the ISF I-CORE grant 152/1, Mr. and Mrs. Dan Kane, Ms. Lois Rosen, by a Yeda-Sela research grant, by the Larson Charitable Foundation, by Ruth and Herman Albert Scholars Program for New Scientists, by the Ilse Katz Institute for Materials Sciences and Magnetic Resonance Research grant, by the Ministry of Health grant for alternative research methods, and by the France-Israel Cooperation - Maimonide-Israel Research Program. IKG is a recipient of the Rowland and Sylvia Career Development Chair.
Materialien
| Name | Company | Catalog Number | Comments |
| Luria Broth, Lennox | Difco | 240230 | |
| Bacto Agar | Difco | 214010 | |
| potassium phosphate monobasic | Sigma, 136.09 g/mol | P0662-500G | |
| potassium phosphate dibasic | Fisher Scientific, 174.18 g/mol | BP363-1 | |
| 3-(N-morpholino)propanesulfonic acid | Fisher Scientific, 209.27 g/mol | BP308-500 | |
| magnesium chloride hexahydrate | Merck, 203.30 g/mol | 1.05833.0250 | |
| calcium chloride anhydrous | J.T. Baker, 110.98 g/mol | 1311-01 | |
| manganese(II) chloride tetrahydrate | Sigma, 197.91 g/mol | 31422-250G-R | |
| iron(III) chloride hexahydrate | Sigma, 270.30 g/mo) | F2877-500G | |
| zinc chloride anhydrous | Acros Organics, 136.29 g/mol | 424592500 | |
| thiamine hydrochloride | Sigma, 337.27 g/mol | T1270-100G | |
| L-tryptophan | Fisher Scientific, 204.1 g/mol | BP395-100 | |
| L-phenylalanine | Sigma, 165.19 g/mol | P5482-100G | |
| L-threonine | Sigma, 119.12 g/mol | T8625-100G | |
| glycerol anhydrous | Bio-Lab Itd | 712022300 | |
| L-glutamic acid monosodium salts hydrate | Sigma, 169.11 g/mol | G1626-1KG | |
| D-leucine | Sigma, 169.11 g/mol | 855448-10G | |
| ethanol anhydrous | Gadot | 830000054 | |
| razor blade | Eddison | NA | |
| circular cellulose filter papers | Whatman, 90 mm | 1001-090 | |
| glutaraldehyde | EMS (Electron Micoscopy Science), 25% in water | 16220 | |
| paraformaldehyde | EMS, 16% in water | 15710 | |
| sodium cacodylate | Merck, 214.05 g/mol | 8.2067 | |
| calcium chloride 2-hydrate | Merck, 147.02 g/mol | 1172113 | |
| stub-aluminium mount | EMS, sloted head | 75230 | |
| carbon adhesive tape | EMS | 77825-12 | |
| Shaker 37 °C | New Brunswick Scientific Innowa42 | NA | |
| Centrifuge | Eppendorf table top centrifuge 5424 | NA | |
| Digital Sonifier, Model 250, used with Double Step Microtip | Branson | NA | |
| Incubator 30 °C | Binder | NA | |
| Incubator 23 °C | Binder | NA | |
| Filter System, 500 ml, polystyrene | Cornig Incorporated | NA | |
| Rotary Shaker - Orbitron Rotatory II | Boekel | NA | |
| S150 Sputter Coater | Edwards | NA | |
| CPD 030 Critical Point Dryer | BAL-TEC | NA | |
| Environmental Scanning Electron Microscope | XL30 ESEM FEG Philips (FEI) | NA |
Referenzen
- Branda, S. S., Vik, S., Friedman, L., Kolter, R. Biofilms: the matrix revisited. Trends Microbiol. 13, 20-26 (2005).
- Stoodley, P., Sauer, K., Davies, D. G., Costerton, J. W. Biofilms as complex differentiated communities. Annu Rev Microbiol. 56, 187-209 (2002).
- Miller, M. B., Bassler, B. L. Quorum sensing in bacteria. Annu Rev Microbiol. 55, 165-199 (2001).
- Aguilar, C., Vlamakis, H., Losick, R., Kolter, R. Thinking about Bacillus subtilis as a multicellular organism. Curr Opin Microbiol. 10, 638-643 (2007).
- Kolter, R., Greenberg, E. P. Microbial sciences: the superficial life of microbes. Nature. 441, 300-302 (2006).
- Kearns, D. B., Chu, F., Branda, S. S., Kolter, R., Losick, R. A master regulator for biofilm formation by Bacillus subtilis. Mol Microbiol. 55, 739-749 (2005).
- Branda, S. S., Chu, F., Kearns, D. B., Losick, R., Kolter, R. A major protein component of the Bacillus subtilis biofilm matrix. Mol Microbiol. 59, 1229-1238 (2006).
- Romero, D., Aguilar, C., Losick, R., Kolter, R. Amyloid fibers provide structural integrity to Bacillus subtilis biofilms. Proc Natl Acad Sci USA. 107, 2230-2234 (2010).
- Kobayashi, K., Iwano, M. BslA(YuaB) forms a hydrophobic layer on the surface of Bacillus subtilis biofilms. Mol Microbiol. 85, 51-66 (2012).
- Hobley, L., et al. BslA is a self-assembling bacterial hydrophobin that coats the Bacillus subtilis biofilm. Proc Natl Acad Sci USA. 110, 13600-13605 (2013).
- Romero, D., Vlamakis, H., Losick, R., Kolter, R. An accessory protein required for anchoring and assembly of amyloid fibres in B. subtilis biofilms. Mol Microbiol. 80, 1155-1168 (2011).
- Kolodkin-Gal, I., et al. D-amino acids trigger biofilm disassembly. Science. 328, 627-629 (2010).
- Chan, Y. G., Kim, H. K., Schneewind, O., Missiakas, D. The capsular polysaccharide of Staphylococcus aureus is attached to peptidoglycan by the LytR-CpsA-Psr (LCP) family of enzymes. J Biol Chem. 289, 15680-15690 (2014).
- Mielich-Suss, B., Lopez, D. Molecular mechanisms involved in Bacillus subtilis biofilm formation. Environ Microbiol. 17, 555-565 (2014).
- Cairns, L. S., Hobley, L., Stanley-Wall, N. R. Biofilm formation by Bacillus subtilis: new insights into regulatory strategies and assembly mechanisms. Mol Microbiol. 93, 587-598 (2014).
- Chen, M., Yu, Q., Sun, H. Novel strategies for the prevention and treatment of biofilm related infections. Int J Mol Sci. 14, 18488-18501 (2013).
- Bucher, T., Oppenheimer-Shaanan, Y., Savidor, A., Bloom-Ackermann, Z., Kolodkin-Gal, I. Disturbance of the bacterial cell wall specifically interferes with biofilm formation. Environ Microbiol Rep. 7, 990-1004 (2015).
- Sarkar, S., Pires, M. M. D-Amino acids do not inhibit biofilm formation in Staphylococcus aureus. PLoS One. 10, e0117613 (2015).
- Wei, W., Bing, W., Ren, J., Qu, X. Near infrared-caged D-amino acids multifunctional assembly for simultaneously eradicating biofilms and bacteria. Chem Commun (Camb). 51, 12677-12679 (2015).
- Leiman, S. A., et al. D-amino acids indirectly inhibit biofilm formation in Bacillus subtilis by interfering with protein synthesis. J Bacteriol. 195, 5391-5395 (2013).
- Costerton, J. W., Stewart, P. S., Greenberg, E. P. Bacterial biofilms: a common cause of persistent infections. Science. 284, 1318-1322 (1999).
- Davies, D. Understanding biofilm resistance to antibacterial agents. Nat Rev Drug Discov. 2, 114-122 (2003).
- Olsen, I. Biofilm-specific antibiotic tolerance and resistance. Eur J Clin Microbiol Infect Dis. 34, 877-886 (2015).
- Tseng, B. S., et al. The extracellular matrix protects Pseudomonas aeruginosa biofilms by limiting the penetration of tobramycin. Environ Microbiol. 15, 2865-2878 (2013).
- Branda, S. S., Gonzalez-Pastor, J. E., Ben-Yehuda, S., Losick, R., Kolter, R. Fruiting body formation by Bacillus subtilis. Proc Natl Acad Sci USA. 98, 11621-11626 (2001).
- Holscher, T., et al. Motility, Chemotaxis and Aerotaxis Contribute to Competitiveness during Bacterial Pellicle Biofilm Development. J Mol Biol. 427, 3695-3708 (2015).
- Bray, D. . Methods in Biotechnology. 13, 235-243 (2000).
- Ensikat, H. J., Ditsche-Kuru, P., Barthlott, W. . Scanning electron microscopy of plant surfaces: simple but sophisticated methods for preparation and examination. 1, 248-255 (2010).
- Hayat, M. A. . Principles and techniques of scanning electron microscopy: Biological applications. 2, (1976).
- Schatten, H. . Scanning Electron Microscopy for the Life Sciences. , (2013).
- Bridier, A., Meylheuc, T., Briandet, R. Realistic representation of Bacillus subtilis biofilms architecture using combined microscopy (CLSM, ESEM and FESEM). Micron. 48, 65-69 (2013).
- Boyde, A., MacOnnachie, E. Volume changes during preparation of mouse embryonic tissue for scanning electron microscopy. SCANNING. 2, 149-163 (1979).
- Yao, Z., Kahne, D., Kishony, R. Distinct single-cell morphological dynamics under beta-lactam antibiotics. Mol Cell. 48, 705-712 (2012).
- Epstein, A. K., Pokroy, B., Seminara, A., Aizenberg, J. Bacterial biofilm shows persistent resistance to liquid wetting and gas penetration. Proc Natl Acad Sci USA. 108, 995-1000 (2011).
- Vlamakis, H., Chai, Y., Beauregard, P., Losick, R., Kolter, R. Sticking together: building a biofilm the Bacillus subtilis way. Nat Rev Microbiol. 11, 157-168 (2013).
- Shemesh, M., Chai, Y. A combination of glycerol and manganese promotes biofilm formation in Bacillus subtilis via histidine kinase KinD signaling. J Bacteriol. 195, 2747-2754 (2013).
- Kolodkin-Gal, I., et al. Respiration control of multicellularity in Bacillus subtilis by a complex of the cytochrome chain with a membrane-embedded histidine kinase. Genes Dev. 27, 887-899 (2013).
- Oppenheimer-Shaanan, Y., et al. Spatio-temporal assembly of functional mineral scaffolds within microbial biofilms. npj Biofilms and Microbiomes. 2, 15031 (2016).
- Garcia-Betancur, J. C., Yepes, A., Schneider, J., Lopez, D. Single-cell analysis of Bacillus subtilis biofilms using fluorescence microscopy and flow cytometry. J Vis Exp. , e3796 (2012).
- Bogino, P. C., Oliva Mde, L., Sorroche, F. G., Giordano, W. The role of bacterial biofilms and surface components in plant-bacterial associations. Int J Mol Sci. 14, 15838-15859 (2013).
- Fratamico, P. M., Annous, B. A., Guenther, N. W. . Biofilms in the Food and Beverage Industires. 1, (2009).
- Gao, G., et al. Effect of biocontrol agent Pseudomonas fluorescens 2P24 on soil fungal community in cucumber rhizosphere using T-RFLP and DGGE. PLoS One. 7, e31806 (2012).
- Chen, Y., et al. Biocontrol of tomato wilt disease by Bacillus subtilis isolates from natural environments depends on conserved genes mediating biofilm formation. Environ Microbiol. 15, 848-864 (2013).
- Bryers, J. D. Medical biofilms. Biotechnol Bioeng. 100, 1-18 (2008).
- Logan, B. E. Exoelectrogenic bacteria that power microbial fuel cells. Nat Rev Microbiol. 7, 375-381 (2009).
- Nevin, K. P., Woodard, T. L., Franks, A. E., Summers, Z. M., Lovley, D. R. Microbial electrosynthesis: feeding microbes electricity to convert carbon dioxide and water to multicarbon extracellular organic compounds. MBio. 1, (2010).
- Torres, C. I., et al. A kinetic perspective on extracellular electron transfer by anode-respiring bacteria. FEMS Microbiol Rev. 34, 3-17 (2010).
- Li, J., Wang, N. Foliar application of biofilm formation-inhibiting compounds enhances control of citrus canker caused by Xanthomonas citri subsp. citri. Phytopathology. 104, 134-142 (2014).
- Okegbe, C., Price-Whelan, A., Dietrich, L. E. Redox-driven regulation of microbial community morphogenesis. Curr Opin Microbiol. 18, 39-45 (2014).
- Mann, E. E., Wozniak, D. J. Pseudomonas biofilm matrix composition and niche biology. FEMS Microbiol Rev. 36, 893-916 (2012).
- Bouffartigues, E., et al. Sucrose favors Pseudomonas aeruginosa pellicle production through the extracytoplasmic function sigma factor SigX. FEMS Microbiol Lett. 356, 193-200 (2014).
- Wu, C., Lim, J. Y., Fuller, G. G., Cegelski, L. Quantitative analysis of amyloid-integrated biofilms formed by uropathogenic Escherichia coli at the air-liquid interface. Biophys J. 103, 464-471 (2012).
- Serra, D. O., Richter, A. M., Hengge, R. Cellulose as an Architectural Element in Spatially Structured Escherichia coli Biofilms. J Bacteriol. 195, 5540-5554 (2013).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten