Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Méthodologies pour l'étude
Dans cet article
Résumé
This study presents the development of reproducible methodologies to study biofilm inhibitors and their effects on Bacillus subtilis multicellularity.
Résumé
This work assesses different methodologies to study the impact of small molecule biofilm inhibitors, such as D-amino acids, on the development and resilience of Bacillus subtilis biofilms. First, methods are presented that select for small molecule inhibitors with biofilm-specific targets in order to separate the effect of the small molecule inhibitors on planktonic growth from their effect on biofilm formation. Next, we focus on how inoculation conditions affect the sensitivity of multicellular, floating B. subtilis cultures to small molecule inhibitors. The results suggest that discrepancies in the reported effects of such inhibitors such as D-amino acids are due to inconsistent pre-culture conditions. Furthermore, a recently developed protocol is described for evaluating the contribution of small molecule treatments towards biofilm resistance to antibacterial substances. Lastly, scanning electron microscopy (SEM) techniques are presented to analyze the three-dimensional spatial arrangement of cells and their surrounding extracellular matrix in a B. subtilis biofilm. SEM facilitates insight into the three-dimensional biofilm architecture and the matrix texture. A combination of the methods described here can greatly assist the study of biofilm development in the presence and absence of biofilm inhibitors, and shed light on the mechanism of action of these inhibitors.
Introduction
communautés bactériennes multi-cellulaires jouent un rôle important dans les milieux naturels et anthropiques, et peuvent être bénéfiques ou très néfaste. Ces colonies multicellulaires sont appelées biofilms, dans lequel les cellules individuelles sont noyées dans une (EPS) de la matrice extracellulaire des substances polymères auto-produites. Les EPS adhèrent fortement les cellules à la surface qu'ils colonisent. Ils servent de bouclier contre les forces mécaniques et chimiques et de créer un contact étroit entre les cellules voisines, ce qui facilite la communication cellulaire 1. Un biofilm peut être considérée comme une communauté différenciée, où les cellules utilisent très réglementées, orchestré des processus pour coordonner leurs activités au sein de la communauté, ainsi que toutes les espèces 2-5. La transition d'un planctoniques, mode libre-vie de la croissance à un état de biofilm est souvent associée à des processus de développement. Un bon exemple est la bactérie du sol Bacillus subtilis Gram positif, et donc un undómësouche sticated sert organisme modèle robuste pour étudier les étapes du développement menant à la formation de biofilm. Dans cette bactérie, cellules mobiles s'organisent en structures multicellulaires visibles qui effectuent des tâches spécialisées 4. Un groupe de cellules, la matrice producteurs sécrètent exopolysaccharides 6, la protéine amyloïde Tasa 7,8, et la protéine de surface hydrophobe BSLA 9,10; qui tous participent à l'assemblage des EPS 11-13.
Compte tenu de l'abondance des biofilms dans des niches naturelles et anthropiques et les dommages mortels putative ils peuvent causer, il y a un besoin urgent de trouver des moyens de prévenir leur formation. Les inhibiteurs de petites molécules peuvent aider à la découverte de nouvelles voies de régulation, des enzymes et des protéines structurales impliquées dans la formation de biofilm, et promouvoir ainsi un aperçu des processus complexes de l'ensemble de la communauté multicellulaire. Comme B. subtilis est un modèle bien étudié pour la bioformation d' un film 14,15, il peut être utilisé pour évaluer les effets de divers inhibiteurs de biofilm. Cette étude aborde quatre méthodes fondamentales qui sont essentielles pour évaluer la réponse des biofilms aux inhibiteurs de petites molécules. Tout d'abord, pour s'assurer que ces inhibiteurs ont une cible spécifique de biofilm, la séparation de l'effet sur la croissance planctonique de l'effet sur la formation de biofilm est critique. La plupart des agents antibactériens cellules cibles dans leur phase de croissance planctonique, mais des molécules qui ciblent le mode de vie de biofilm sont rares. En outre, en tant que molécules qui ne touchent pas la croissance planctonique ne sont pas toxiques, ils peuvent réduire la pression sélective favorisant des mutants résistants aux antibiotiques 16. Par exemple, lorsque les biofilms sont traités avec des acides aminés D ou de certaines autres molécules de paroi interférant cellule, ils sont soit perturbés ou démontées, mais ces inhibiteurs affectent seulement légèrement 12,17 croissance planctonique. En revanche, de nombreux antibiotiques altèrent considérablement la croissance planctonique, avec little ou pas d' effet sur la formation de biofilm 17.
Deuxièmement, l'établissement d'un cadre expérimental cohérente et solide pour étudier l'effet des petites molécules est cruciale. Nous avons observé que la plage de concentration active d'inhibiteurs de petites molécules est sensible aux conditions de préculture et le dispositif expérimental utilisé pour étudier l'effet de ces inhibiteurs à petites molécules. Divers rapports, en particulier ceux qui étudient B. subtilis, a révélé des variations dans la plage de concentration à laquelle les acides D-aminés inhibent la formation de pellicules - les biofilms bactériens flottants 12,17-19. Les résultats présentés ici suggèrent que les facteurs suivants expliquent les différences dans la gamme de concentration active: les conditions de pré-culture (logarithmique 12,17 par rapport à la phase tardive stationnaire 20 croissance), le milieu de croissance utilisé dans l'état pré-culture (riche, undefined [Luria Broth, LB] par rapport défini [glutamate monosodique-glycérol, MSgg]), le rapport d'inoculation et en particulier l'élimination du milieu de pré-culture avant l'inoculation. La température de croissance pelliculaire statique a montré un rôle moins important dans le domaine d'activité de l'inhibiteur de petite molécule de D-leucine, représentant un acide D-aminé utilisé dans la présente étude.
Enfin, une fois que les biofilms sont traités avec des inhibiteurs de biofilm spécifiques, des méthodes robustes et informatives sont nécessaires pour caractériser les effets de ces inhibiteurs sur biofilm fitness. Ici, deux méthodes pour caractériser de façon indépendante l'effet des inhibiteurs de petites molécules sont décrits en détail: (1) L'effet sur des cellules individuelles au sein d'une colonie du biofilm et leur résistance aux agents antimicrobiens. Les cellules dans les biofilms sont généralement plus résistantes aux antibiotiques par rapport à des bactéries vivant librement 21-23. Bien que ce phénomène est multifactorielle, la capacité des EPS pour réduire la pénétration des antibiotiques a souvent été considérée comme une explication attrayante 24 . Cette méthode évalue la survie des cellules pré-établies biofilm après exposition à des substances antibactériennes. (2) L'effet sur l'architecture biofilm de la colonie, de la grande à la petite échelle. les colonies biofilm se caractérisent par leur structure tridimensionnelle et la présence de l'EPS. En utilisant la microscopie électronique à balayage, des changements dans la morphologie cellulaire, la structure de la colonie du biofilm et l'architecture et l'abondance de l'EPS peut être visualisée à partir de la grande taille (mm) à la petite échelle (um).
Protocole
1. Évaluation de l'effet des inhibiteurs de petites molécules sur Pellicle et formation de biofilms Colony
- Préparer une solution de 2 x milieu défini inducteur MSgg biofilm 25 sans que le chlorure de calcium et de fer (III) hexahydraté. Après stérilisation du filtre, ajouter le chlorure de calcium. Le milieu est prêt à être utilisé directement ou il peut être conservé à 4 ° C dans l'obscurité.
- Préparer la dilution 1x MSgg le jour de l'expérience.
- Diluer le moyen 2x MSgg à 1x avec de l'eau distillée stérile (des pellicules) ou 3% à chaud (80 ° C) agar stérile (biofilms) et ajouter du fer (III) hexahydraté à une concentration finale de 50 uM (pellicules) ou 250 uM ( biofilms). Ajouter des antibiotiques ou des inhibiteurs de petites molécules à la concentration souhaitée et bien mélanger. Par exemple, pour obtenir une concentration finale mM de D-leucine 0,5 dans 30 ml d'établir ou de pellicules biofilms, ajouter 196,6 76,3 ul d'une mM (10 mg / ml) Solution mère de D-leucine.
NONTE. La composition finale 1x MSgg est décrite dans le tableau 1 par rapport à la recette initiale 25, le milieu contenait 50 pg / ml de thréonine et de la concentration en fer à cultiver les colonies de biofilm sur un milieu de MSgg solide a été augmentée 2.5x pour optimiser la morphologie des colonies ridées .
- Diluer le moyen 2x MSgg à 1x avec de l'eau distillée stérile (des pellicules) ou 3% à chaud (80 ° C) agar stérile (biofilms) et ajouter du fer (III) hexahydraté à une concentration finale de 50 uM (pellicules) ou 250 uM ( biofilms). Ajouter des antibiotiques ou des inhibiteurs de petites molécules à la concentration souhaitée et bien mélanger. Par exemple, pour obtenir une concentration finale mM de D-leucine 0,5 dans 30 ml d'établir ou de pellicules biofilms, ajouter 196,6 76,3 ul d'une mM (10 mg / ml) Solution mère de D-leucine.
- Après solidification de la gélose, on sèche les plaques de MSgg solides dans une hotte biologique de 30 à 45 minutes avant l'inoculation.
- Pour sélectionner des inhibiteurs spécifiques qui interfèrent avec les mécanismes de formation de la pellicule (figure 1), excluent que les concentrations utilisées affectent planctoniques et la croissance statique.
- Déterminer la croissance planctoniques (augmentation de la densité optique au cours du temps en milieu liquide) dans une courbe de croissance simple en mesurant la densité optique à 600 nm toutes les heures jusqu'à la phase de croissance stationnaire.
- Pour confirmer que la turbidité de la culture mesurée représente le nombre de cellules vivantes, déterminer le nombre de unités formant colonie (UFC)des cellules dans la phase de croissance planctonique à partir d'une culture sous agitation au bout de plusieurs points dans le temps.
- Pour évaluer l'effet des inhibiteurs de petites molécules sur la croissance de la pellicule statique, les cellules de récolte à la fin d'une incubation de 3 jours à 23 ° C à partir d'une culture cellulaire de 24 puits et inoculés dans les mêmes conditions que celles décrites dans les sections 1,7-1,9 et déterminer la CFU. Pour ce contrôle, utilisez une souche pelliculaire déficiente qui manque les opérons codant pour les composants de la matrice extracellulaire (ie, B. subtilis Δ EPSH, Δ TASA).
Remarque: Cette souche est capable de croître dans des conditions statiques, mais contrairement à un type sauvage pelliculaire formant, il est déficient dans la capacité de flotter à l'interface liquide-air, où la croissance est favorisée en raison des niveaux accrus d'oxygène 26. Ainsi, cette matriciel extracellulaire et la souche pelliculaire déficiente est une souche de référence recommandée pour évaluer la croissance dans des conditions statiques.
NOTE: Pour l'ex spécifiquesuffisante de la D-amino-acide D-leucine non canonique décrit ci - après, un effet sur la croissance statique planctoniques et à des concentrations qui interfèrent avec la formation pelliculaire a été écartée 12,17. Les méthodes pour déterminer planctoniques et la croissance statique sont décrites en détail 17.
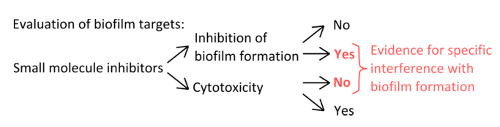
Figure 1. Vue d'ensemble conceptuel pour l'identification d'un dispositif expérimental robuste pour évaluer l'inhibition spécifique de la formation de biofilm. Les critères de sélection pour les inhibiteurs de petites molécules qui indiquent l' interférence spécifique avec la formation de biofilm sans effet prononcé sur la croissance planctonique. S'il vous plaît cliquer ici pour voir une version plus grande de ce chiffre.
- Streak sur B. subtilis à partir d' un -80 ° C stock (LB culture de 10 9 cellules / ml congelés dans 20% de glycérol) pour isoler des colonies isolées sur une plaque de gélose%-1,5 LB avec une pointe stérile ou bâton applicateur.
- Cultivez une nuit à 30 ° C.
- ÉTAPE CRITIQUE: Pour une pellicule solide inhibition par les acides non-canonique D-amino tels que D-leucine, grandir une seule colonie prélevée du% plaque de gélose-1,5 LB dans 3 ml de bouillon LB à 37 ° C pendant 4 heures dans un secouant incubateur (vitesse d'agitation de 200 tours par minute). Remplacer le bouillon LB avec du milieu MSgg biofilm induisant avant l'inoculation par centrifugation culture 1,5 ml de démarrage pendant 4 min à 6000 xg, enlevant soigneusement le surnageant et remise en suspension du culot dans 1,5 ml de milieu MSgg. Le reste de la culture peut être mis au rebut.
Important: Afin d' assurer la robustesse du système, la densité optique à 600 nm (DO 600) de la culture de départ lavé doit être compris entre 0,6 et 1. - Au cours de la croissance de la culture starter, préparer un 12 puits de culture cellulaire plaque multiple à containing 3 ml de milieu MSgg sans ou avec une gamme de concentrations de l'inhibiteur à petite molécule (par exemple, 0,3, 0,5, 1 mM de D-leucine , 17). Pour exclure les effets de bord, distribuer l'emplacement des différentes concentrations sur la plaque multiple à. Vous pouvez également utiliser de culture cellulaire 24 puits plaques Multidish contenant 1,5 ml de milieu MSgg.
- Inoculer des puits de la plaque de culture cellulaire à 12 puits boîte multiple avec 3 ul de la culture de départ lavée (1: 1000 dilution).
REMARQUE: un taux de dilution plus faible, à savoir 1: 500 peut être utilisé. Ceci diminue le temps de développement des pellicules. - Cultiver les pellicules à 23 ° C dans des conditions statiques pendant trois jours. Ne déplacez pas les pellicules pendant ce temps, car il peut affecter la morphologie de la surface finale de la pellicule.
- Acquérir des photos avec une exposition binoculaire et homogène de la foudre. Vous pouvez également prendre une photo des pellicules avec une caméra à haute résolution. Pour éviter les artefacts causés par inconsisles angles et les ombres de lumière tente, de prendre des photos de haut en bas avec la caméra fixée sur un trépied et d'utiliser une source de lumière douce et grande à 45 ° des deux côtés.
NOTE: Une autre méthode pour étudier B. subtilis multicellularité est le biofilm colonie test sur solide, moyenne MSgg biofilm induisant. Comme pellicules, ce test permet l'étude des processus spatio-temporels. Une fois que la plage active d'inhibiteurs de petites molécules est déterminée, leur effet sur la formation de biofilm de colonies peut être étudiée. - Pour faire pousser des colonies de biofilms, repérer symétriquement 1,5 pl de la pré-culture non lavés (étape 1.7) sur le MSgg 1,5% plaque de gélose séchée à l'aide d'un modèle - 4 gouttes par boîte de Pétri de 8,5 cm de diamètre. Laissez les gouttes adsorber à la plaque avant de les déplacer.
NOTE: Le modèle permet d'obtenir une répartition égale des colonies de biofilms dans la zone où les cellules sont cultivées. Pour préparer le modèle, dessiner la superficie totale de la croissance à l'échelle d'origine, diviser à l'égalité de secteeurs et marquer le centre. Pour un plat rond Petri de 8,5 cm de diamètre, ce affecte 14 cm 2 à une colonie de biofilm. - Incuber les plaques à 30 ° C pendant trois jours. Pendant ce temps, les colonies se développent biofilm et forment trois dimensions, la structure plissée.
- Prenez des photos comme à l'étape 1.11.
2. Ethanol Résistance Assay
- Cultivez biofilms comme décrit dans les étapes 1,1-1,7 et 1.12-1.14.
- Après 68 h de croissance à 30 ° C, couper les colonies de biofilms en deux parties égales avec l'aide d'une lame de rasoir et le modèle.

Figure 2. Exemple de conception expérimentale pour évaluer la résistance des cellules biofilm de colonies aux agents stérilisants. (A) Modèle utilisé pour la distribution égale des colonies de biofilms dans une boîte de Pétri et pour la coupe. (B)images Top-down de non traitées de type sauvage biofilm cultivées pendant 68 h sur solide, un milieu défini biofilm induisant MSgg à 30 ° C. L'élargissement montre comment une colonie de biofilm peut être coupé en deux moitiés égales. (C) Les deux moitiés de biofilm égales sont traitées de manière égale (contrôle, PBS) ou soit avec PBS ou agent stérilisant et traité comme décrit. Barre d'échelle:. 1 cm S'il vous plaît cliquer ici pour voir une version plus grande de cette figure.
- Soulevez délicatement chaque moitié de la colonie de biofilm de la plaque de gélose avec une petite spatule et le déplacer vers un tube de 1,5 ml contenant 500 ul de solution saline tamponnée au phosphate (PBS). Si nécessaire, gratter les cellules restantes de la plaque et de les transférer dans le tube à centrifuger ainsi.
Remarque: la seconde moitié de la colonie du biofilm sont traitées différemment, selon que ce soit la commande ou pour tester la résistance à stériagents Lizing. - Pour le contrôle, incuber la seconde moitié de la colonie de biofilm dans 500 ul de PBS comme dans l'étape 2.3. Pour évaluer la résistance aux agents stérilisants, transférer la deuxième moitié de la colonie du biofilm de 500 ul de 50% (v / v) d'éthanol.
NOTE: Variante de stérilisation des agents tels que l'hypochlorite de sodium peuvent être utilisés. Pour tous les agents stérilisants utilisés, déterminer la concentration et la durée d'incubation active dans un essai préliminaire. - Incuber les colonies de biofilms pendant 10 minutes sur la paillasse à température ambiante.
- Centrifuger les colonies de biofilms pendant 5 min à 18000 xg et retirer délicatement le surnageant avec une pipette. Ajouter 300 ul de PBS.
- Soniquer les cellules légèrement (amplitude de 10%, pouls 5 sec) avec la micropointe d'un sonicateur.
NOTE: L'énergie de sonication doit être suffisante pour les agrégats de biofilm séparés. Cependant, sonication trop sévère peut lyser les cellules. Confirmer à l'avance par microscopie optique que l'énergie de sonicationutilisés ne lyser pas les cellules et que tous les agrégats sont dissous. - Ajouter 700 ul de PBS dans un volume final de 1 ml. Effectuer une dilution en série (à 10 -7) dans du PBS et 100 ul de s'étendre 3 dilutions% sur une plaque de gélose LB-1,5 en utilisant des billes de verre stériles.
REMARQUE: Les dilutions optimales à plaquer doit être déterminée dans un essai préliminaire, car cela dépend de la quantité de cellules dans la colonie du biofilm d'intérêt et le taux de survie des cellules en réponse à l'agent de stérilisation. - Incuber les plaques pendant une nuit à 30 ° C, comptez le CFU et déterminer CFU / ml. De la finale UFC / ml de chaque demi-colonies de biofilms, calculer le pourcentage de survivants.
REMARQUE: Quand elle est réalisée et analysé comme décrit, les deux moitiés de la colonie de contrôle du biofilm et non traitée par rapport à la moitié de l'éthanol traité par de la colonie du biofilm non traitée devrait donner des différences inférieures à 10% du nombre de cellules viables, en vérifiant la symétrie ou la résistance de la colonie , respectively. Sinon, les résultats peuvent être représentés au total CFU. Les comptages de cellules de contrôle et des colonies de biofilm non traitées doivent rester dans le même ordre de grandeur. En revanche, le nombre de cellules de la moitié de l'éthanol-traitée d'une petite colonie biofilm molécule traités devraient baisser par un minimum de deux ordres de grandeur à la revendication d'une sensibilité accrue à l'agent de stérilisation.
3. biofilm Colony Préparation d'échantillons pour microscopie électronique à balayage
- Cultivez colonies biofilm comme décrit dans les étapes 1,1-1,7 et 1.12-1.14.
- Préparer un nouveau lot de 2% (v / v) de glutaraldéhyde, 3% (v / v) d'une solution de paraformaldehyde dans du cacodylate de sodium 100 mM, un tampon de chlorure de calcium 5 mM, pH 7,3. Préparer 5 ml de fixateur pour chaque boîte de Pétri de 8,5 cm de diamètre.
ATTENTION: Glutaraldéhyde et paraformaldéhyde sont dangereux. Manipulez-les avec l'équipement de sécurité à l'intérieur d'une hotte chimique. Jeter les solutions et les matériaux contaminés à l'hazdéchets ardous. - Ajouter délicatement le fixateur dans les colonies de biofilms, sans distribuer directement sur les biofilms.
NOTE: En raison du caractère hydrophobe de la colonie du biofilm, les colonies se détachent lentement de l'agar-agar et commencent à flotter. - Sceller soigneusement les plaques avec une bande de Parafilm. Incuber sur un agitateur rotatif pendant 2 heures à température ambiante, et transférer ensuite les plaques à 4 ° C pendant une nuit.
- Le lendemain, retirez avec précaution le liquide avec une pipette en verre Pasteur relié à une pompe à vide.
- ajouter avec précaution 10 ml de 100 mM de cacodylate de sodium, tampon de chlorure de calcium 5 mM à laver le biofilm et laisser incuber pendant 5 min. Retirez délicatement le liquide avec la pipette en verre Pasteur du coin de la plaque pour éviter d'endommager le biofilm et ajouter la solution de lavage frais par pipetage doux. Répétez cette étape une fois.
- Pour la déshydratation des colonies de biofilms, procéder aux étapes suivantes: 2x 5 min dans ddH 2 O; 2x 20 min à 3d'éthanol de 0%; 2 x 20 minutes dans 50% d'éthanol; 2 x 20 minutes dans 70% d'éthanol; 2 x 20 min dans 96% d'éthanol; 2x 30 min à 100% d'éthanol.
- Ajouter 15 ml de liquide pour chaque boîte de Pétri de 8,5 cm de diamètre dans chaque étape et enlever le liquide soigneusement après chaque incubation.
- Utilisez l'une des deux méthodes différentes pour sécher les échantillons à partir d'éthanol.
- Pour séchage à l'air à partir d'éthanol:
- Coupez un papier filtre de cellulose (diamètre de 9 cm) en quarts. immergez brièvement un quart dans 100% d'éthanol, puis transférer soigneusement une colonie flottante biofilm sur elle. Placez le papier filtre humide dans une boîte de Pétri garni d'un papier filtre. Couvrir la boîte de Pétri et laisser les colonies de biofilms sécher pendant la nuit dans une hotte chimique.
- Pour le point critique (CP) -Séchage en utilisant du dioxyde de carbone (CO 2) comme fluide de transition:
- Remplir 75% de la chambre de la machine de séchage au point critique avec 100% d'éthanol. Transférer les échantillons dans un support, chaque échantillon dans son propre chambre. Si nécessaire, couper le biofilm avec des ciseaux en petits morceaux. Laissez les échantillons immergés dans de l'éthanol pendant toute la manipulation. Ensuite, transférer le support dans la chambre et fermer la chambre hermétiquement.
- Refroidir la chambre à 7 ° C et commencer l'agitation. Remplir la chambre complètement avec CO 2 liquide. Au cours d' une période d'incubation de 7 minutes, laisser le mélange d'éthanol avec du CO 2. Ensuite, décharger 25% de la solution.
REMARQUE: Ne pas vider la chambre au-dessous du niveau de l'échantillon. - Répétez l'étape 3.8.2.2 quatre fois.
- Répétez l'étape 3.8.2.2 cinq fois avec un temps d'incubation de 5 minutes seulement. Enfin, l'éthanol doit être complètement remplacé par CO 2.
- Au cours de la dernière ronde, vide seulement 5% de la chambre. Éteignez l'agitation et le refroidissement. Commencer à chauffer la chambre à 42 ° C. A une température de 31,1 ° C et une pression de 73,9 bars, le CO 2 liquide atteint son point critique, l'état où le ph gazeuxase a la même densité que la phase liquide du solvant 27. Une fois que la température atteint 42 ° C, incuber pendant 10 min. À 42 ° C, le CO 2 dans la chambre existe en tant que gaz supercritique.
REMARQUE: vérifier en permanence la pression de la chambre. La pression ne doit pas dépasser 120 bar à 42 ° C. - Commencer à libérer lentement le gaz avec chauffage continu. Cela permet de maintenir les échantillons dans la phase de CO 2 gazeux et empêche la déformation de l'échantillon à travers la morphologie de la tension superficielle du liquide. Réglez le débitmètre à 5 L / h par un réglage fin de la valve doseuse contrôlant le débitmètre. Attendez que la toute la pression dans la chambre est libérée. Maintenant, ouvrez la chambre et retirer soigneusement les échantillons du support.
- Pour séchage à l'air à partir d'éthanol:
- Manteau d'un microscope électronique stub avec du ruban de carbone. Avec l'aide de pinces, transférer soigneusement les colonies de biofilm sur le moignon. Connectez chaque colonie à la souche en ajoutant un mince pont de la voiturebande bon, ce qui est crucial pour l'élimination de la charge sous le faisceau d'électrons. A ce stade, gérer les colonies de biofilms avec précaution car ils sont très fragiles. Conserver les échantillons dans un dessiccateur pendant au moins 24 heures ou jusqu'à ce que l'examen.
- Le jour de l'examen au microscope électronique à balayage, par pulvérisation cathodique enrober les colonies de biofilm pendant 2 min dans un angle de 60 ° dans un or-palladium par pulvérisation cathodique coucheuse. Répéter cette étape deux fois et faire tourner les échantillons de 120 ° entre les deux. A la fin, par pulvérisation enrober les échantillons une fois pendant 3 minutes à partir du haut. 20 nm, la couche mince d'or-palladium permet d'améliorer la conductivité et améliore le contraste de l'échantillon pour l'imagerie dans la MEB.
- Stocker les échantillons dans un dessiccateur afin d' éviter la réhydratation de l'échantillon 28 jusqu'à formation d' image avec un microscope électronique à balayage , 29,30.
Résultats
Le dosage de la pellicule est une méthode pour étudier les processus hautement réglementés et dynamiques de B. multicellularité subtilis. En outre, le dosage de la pellicule est adapté pour tester une gamme de conditions soit pré-démarrage ou des concentrations de petites molécules dans une plaque multiple à la culture cellulaire unique dans une expérience. Cependant, B. la formation pelliculaire subtilis est sensible aux conditions de pré-culture (par exemple, le...
Discussion
Biofilms de Bacillus subtilis formes robustes et très structurés dans les deux liquides (pellicules) et sur un milieu solide (colonies). Par conséquent, il sert d'organisme modèle idéal pour caractériser le mode d'action des inhibiteurs de biofilm spécifiques. Sur un support solide, les cellules forment une structure multicellulaire présentant des caractéristiques distinctives qui ne sont pas évidents dans des pellicules, telles que les rides rayonnant à partir du centre vers le bord. Ainsi, ...
Déclarations de divulgation
The authors have nothing to disclose.
Remerciements
Electron microscope imaging was conducted at the Electron Microscopy Unit of the Weizmann Institute of Science, supported in part by the Irving and Cherna Moskowitz Center for Nano and Bio-Nano Imaging. This research was also supported by the ISF I-CORE grant 152/1, Mr. and Mrs. Dan Kane, Ms. Lois Rosen, by a Yeda-Sela research grant, by the Larson Charitable Foundation, by Ruth and Herman Albert Scholars Program for New Scientists, by the Ilse Katz Institute for Materials Sciences and Magnetic Resonance Research grant, by the Ministry of Health grant for alternative research methods, and by the France-Israel Cooperation - Maimonide-Israel Research Program. IKG is a recipient of the Rowland and Sylvia Career Development Chair.
matériels
| Name | Company | Catalog Number | Comments |
| Luria Broth, Lennox | Difco | 240230 | |
| Bacto Agar | Difco | 214010 | |
| potassium phosphate monobasic | Sigma, 136.09 g/mol | P0662-500G | |
| potassium phosphate dibasic | Fisher Scientific, 174.18 g/mol | BP363-1 | |
| 3-(N-morpholino)propanesulfonic acid | Fisher Scientific, 209.27 g/mol | BP308-500 | |
| magnesium chloride hexahydrate | Merck, 203.30 g/mol | 1.05833.0250 | |
| calcium chloride anhydrous | J.T. Baker, 110.98 g/mol | 1311-01 | |
| manganese(II) chloride tetrahydrate | Sigma, 197.91 g/mol | 31422-250G-R | |
| iron(III) chloride hexahydrate | Sigma, 270.30 g/mo) | F2877-500G | |
| zinc chloride anhydrous | Acros Organics, 136.29 g/mol | 424592500 | |
| thiamine hydrochloride | Sigma, 337.27 g/mol | T1270-100G | |
| L-tryptophan | Fisher Scientific, 204.1 g/mol | BP395-100 | |
| L-phenylalanine | Sigma, 165.19 g/mol | P5482-100G | |
| L-threonine | Sigma, 119.12 g/mol | T8625-100G | |
| glycerol anhydrous | Bio-Lab Itd | 712022300 | |
| L-glutamic acid monosodium salts hydrate | Sigma, 169.11 g/mol | G1626-1KG | |
| D-leucine | Sigma, 169.11 g/mol | 855448-10G | |
| ethanol anhydrous | Gadot | 830000054 | |
| razor blade | Eddison | NA | |
| circular cellulose filter papers | Whatman, 90 mm | 1001-090 | |
| glutaraldehyde | EMS (Electron Micoscopy Science), 25% in water | 16220 | |
| paraformaldehyde | EMS, 16% in water | 15710 | |
| sodium cacodylate | Merck, 214.05 g/mol | 8.2067 | |
| calcium chloride 2-hydrate | Merck, 147.02 g/mol | 1172113 | |
| stub-aluminium mount | EMS, sloted head | 75230 | |
| carbon adhesive tape | EMS | 77825-12 | |
| Shaker 37 °C | New Brunswick Scientific Innowa42 | NA | |
| Centrifuge | Eppendorf table top centrifuge 5424 | NA | |
| Digital Sonifier, Model 250, used with Double Step Microtip | Branson | NA | |
| Incubator 30 °C | Binder | NA | |
| Incubator 23 °C | Binder | NA | |
| Filter System, 500 ml, polystyrene | Cornig Incorporated | NA | |
| Rotary Shaker - Orbitron Rotatory II | Boekel | NA | |
| S150 Sputter Coater | Edwards | NA | |
| CPD 030 Critical Point Dryer | BAL-TEC | NA | |
| Environmental Scanning Electron Microscope | XL30 ESEM FEG Philips (FEI) | NA |
Références
- Branda, S. S., Vik, S., Friedman, L., Kolter, R. Biofilms: the matrix revisited. Trends Microbiol. 13, 20-26 (2005).
- Stoodley, P., Sauer, K., Davies, D. G., Costerton, J. W. Biofilms as complex differentiated communities. Annu Rev Microbiol. 56, 187-209 (2002).
- Miller, M. B., Bassler, B. L. Quorum sensing in bacteria. Annu Rev Microbiol. 55, 165-199 (2001).
- Aguilar, C., Vlamakis, H., Losick, R., Kolter, R. Thinking about Bacillus subtilis as a multicellular organism. Curr Opin Microbiol. 10, 638-643 (2007).
- Kolter, R., Greenberg, E. P. Microbial sciences: the superficial life of microbes. Nature. 441, 300-302 (2006).
- Kearns, D. B., Chu, F., Branda, S. S., Kolter, R., Losick, R. A master regulator for biofilm formation by Bacillus subtilis. Mol Microbiol. 55, 739-749 (2005).
- Branda, S. S., Chu, F., Kearns, D. B., Losick, R., Kolter, R. A major protein component of the Bacillus subtilis biofilm matrix. Mol Microbiol. 59, 1229-1238 (2006).
- Romero, D., Aguilar, C., Losick, R., Kolter, R. Amyloid fibers provide structural integrity to Bacillus subtilis biofilms. Proc Natl Acad Sci USA. 107, 2230-2234 (2010).
- Kobayashi, K., Iwano, M. BslA(YuaB) forms a hydrophobic layer on the surface of Bacillus subtilis biofilms. Mol Microbiol. 85, 51-66 (2012).
- Hobley, L., et al. BslA is a self-assembling bacterial hydrophobin that coats the Bacillus subtilis biofilm. Proc Natl Acad Sci USA. 110, 13600-13605 (2013).
- Romero, D., Vlamakis, H., Losick, R., Kolter, R. An accessory protein required for anchoring and assembly of amyloid fibres in B. subtilis biofilms. Mol Microbiol. 80, 1155-1168 (2011).
- Kolodkin-Gal, I., et al. D-amino acids trigger biofilm disassembly. Science. 328, 627-629 (2010).
- Chan, Y. G., Kim, H. K., Schneewind, O., Missiakas, D. The capsular polysaccharide of Staphylococcus aureus is attached to peptidoglycan by the LytR-CpsA-Psr (LCP) family of enzymes. J Biol Chem. 289, 15680-15690 (2014).
- Mielich-Suss, B., Lopez, D. Molecular mechanisms involved in Bacillus subtilis biofilm formation. Environ Microbiol. 17, 555-565 (2014).
- Cairns, L. S., Hobley, L., Stanley-Wall, N. R. Biofilm formation by Bacillus subtilis: new insights into regulatory strategies and assembly mechanisms. Mol Microbiol. 93, 587-598 (2014).
- Chen, M., Yu, Q., Sun, H. Novel strategies for the prevention and treatment of biofilm related infections. Int J Mol Sci. 14, 18488-18501 (2013).
- Bucher, T., Oppenheimer-Shaanan, Y., Savidor, A., Bloom-Ackermann, Z., Kolodkin-Gal, I. Disturbance of the bacterial cell wall specifically interferes with biofilm formation. Environ Microbiol Rep. 7, 990-1004 (2015).
- Sarkar, S., Pires, M. M. D-Amino acids do not inhibit biofilm formation in Staphylococcus aureus. PLoS One. 10, e0117613 (2015).
- Wei, W., Bing, W., Ren, J., Qu, X. Near infrared-caged D-amino acids multifunctional assembly for simultaneously eradicating biofilms and bacteria. Chem Commun (Camb). 51, 12677-12679 (2015).
- Leiman, S. A., et al. D-amino acids indirectly inhibit biofilm formation in Bacillus subtilis by interfering with protein synthesis. J Bacteriol. 195, 5391-5395 (2013).
- Costerton, J. W., Stewart, P. S., Greenberg, E. P. Bacterial biofilms: a common cause of persistent infections. Science. 284, 1318-1322 (1999).
- Davies, D. Understanding biofilm resistance to antibacterial agents. Nat Rev Drug Discov. 2, 114-122 (2003).
- Olsen, I. Biofilm-specific antibiotic tolerance and resistance. Eur J Clin Microbiol Infect Dis. 34, 877-886 (2015).
- Tseng, B. S., et al. The extracellular matrix protects Pseudomonas aeruginosa biofilms by limiting the penetration of tobramycin. Environ Microbiol. 15, 2865-2878 (2013).
- Branda, S. S., Gonzalez-Pastor, J. E., Ben-Yehuda, S., Losick, R., Kolter, R. Fruiting body formation by Bacillus subtilis. Proc Natl Acad Sci USA. 98, 11621-11626 (2001).
- Holscher, T., et al. Motility, Chemotaxis and Aerotaxis Contribute to Competitiveness during Bacterial Pellicle Biofilm Development. J Mol Biol. 427, 3695-3708 (2015).
- Bray, D. . Methods in Biotechnology. 13, 235-243 (2000).
- Ensikat, H. J., Ditsche-Kuru, P., Barthlott, W. . Scanning electron microscopy of plant surfaces: simple but sophisticated methods for preparation and examination. 1, 248-255 (2010).
- Hayat, M. A. . Principles and techniques of scanning electron microscopy: Biological applications. 2, (1976).
- Schatten, H. . Scanning Electron Microscopy for the Life Sciences. , (2013).
- Bridier, A., Meylheuc, T., Briandet, R. Realistic representation of Bacillus subtilis biofilms architecture using combined microscopy (CLSM, ESEM and FESEM). Micron. 48, 65-69 (2013).
- Boyde, A., MacOnnachie, E. Volume changes during preparation of mouse embryonic tissue for scanning electron microscopy. SCANNING. 2, 149-163 (1979).
- Yao, Z., Kahne, D., Kishony, R. Distinct single-cell morphological dynamics under beta-lactam antibiotics. Mol Cell. 48, 705-712 (2012).
- Epstein, A. K., Pokroy, B., Seminara, A., Aizenberg, J. Bacterial biofilm shows persistent resistance to liquid wetting and gas penetration. Proc Natl Acad Sci USA. 108, 995-1000 (2011).
- Vlamakis, H., Chai, Y., Beauregard, P., Losick, R., Kolter, R. Sticking together: building a biofilm the Bacillus subtilis way. Nat Rev Microbiol. 11, 157-168 (2013).
- Shemesh, M., Chai, Y. A combination of glycerol and manganese promotes biofilm formation in Bacillus subtilis via histidine kinase KinD signaling. J Bacteriol. 195, 2747-2754 (2013).
- Kolodkin-Gal, I., et al. Respiration control of multicellularity in Bacillus subtilis by a complex of the cytochrome chain with a membrane-embedded histidine kinase. Genes Dev. 27, 887-899 (2013).
- Oppenheimer-Shaanan, Y., et al. Spatio-temporal assembly of functional mineral scaffolds within microbial biofilms. npj Biofilms and Microbiomes. 2, 15031 (2016).
- Garcia-Betancur, J. C., Yepes, A., Schneider, J., Lopez, D. Single-cell analysis of Bacillus subtilis biofilms using fluorescence microscopy and flow cytometry. J Vis Exp. , e3796 (2012).
- Bogino, P. C., Oliva Mde, L., Sorroche, F. G., Giordano, W. The role of bacterial biofilms and surface components in plant-bacterial associations. Int J Mol Sci. 14, 15838-15859 (2013).
- Fratamico, P. M., Annous, B. A., Guenther, N. W. . Biofilms in the Food and Beverage Industires. 1, (2009).
- Gao, G., et al. Effect of biocontrol agent Pseudomonas fluorescens 2P24 on soil fungal community in cucumber rhizosphere using T-RFLP and DGGE. PLoS One. 7, e31806 (2012).
- Chen, Y., et al. Biocontrol of tomato wilt disease by Bacillus subtilis isolates from natural environments depends on conserved genes mediating biofilm formation. Environ Microbiol. 15, 848-864 (2013).
- Bryers, J. D. Medical biofilms. Biotechnol Bioeng. 100, 1-18 (2008).
- Logan, B. E. Exoelectrogenic bacteria that power microbial fuel cells. Nat Rev Microbiol. 7, 375-381 (2009).
- Nevin, K. P., Woodard, T. L., Franks, A. E., Summers, Z. M., Lovley, D. R. Microbial electrosynthesis: feeding microbes electricity to convert carbon dioxide and water to multicarbon extracellular organic compounds. MBio. 1, (2010).
- Torres, C. I., et al. A kinetic perspective on extracellular electron transfer by anode-respiring bacteria. FEMS Microbiol Rev. 34, 3-17 (2010).
- Li, J., Wang, N. Foliar application of biofilm formation-inhibiting compounds enhances control of citrus canker caused by Xanthomonas citri subsp. citri. Phytopathology. 104, 134-142 (2014).
- Okegbe, C., Price-Whelan, A., Dietrich, L. E. Redox-driven regulation of microbial community morphogenesis. Curr Opin Microbiol. 18, 39-45 (2014).
- Mann, E. E., Wozniak, D. J. Pseudomonas biofilm matrix composition and niche biology. FEMS Microbiol Rev. 36, 893-916 (2012).
- Bouffartigues, E., et al. Sucrose favors Pseudomonas aeruginosa pellicle production through the extracytoplasmic function sigma factor SigX. FEMS Microbiol Lett. 356, 193-200 (2014).
- Wu, C., Lim, J. Y., Fuller, G. G., Cegelski, L. Quantitative analysis of amyloid-integrated biofilms formed by uropathogenic Escherichia coli at the air-liquid interface. Biophys J. 103, 464-471 (2012).
- Serra, D. O., Richter, A. M., Hengge, R. Cellulose as an Architectural Element in Spatially Structured Escherichia coli Biofilms. J Bacteriol. 195, 5540-5554 (2013).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon