Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Untersuchung der Dynamik der zellulären Adhäsion und Ausbreitung von Epithelzellen auf Fibronectin während oxidativen Stresses
In diesem Artikel
Zusammenfassung
Diese Methode ist nützlich, um die frühe Dynamik der zellulären Adhäsion und die Ausbreitung von verankerungsabhängigen Zellen auf das Fibronectin zu quantifizieren. Darüber hinaus kann dieser Assay verwendet werden, um die Auswirkungen einer veränderten Redoxhomöostase auf die Zellausbreitung und/oder zellspezifische intrazelluläre Signalwege zu untersuchen.
Zusammenfassung
Die Adhäsion und Ausbreitung von Zellen auf die extrazelluläre Matrix (ECM) sind wesentliche zelluläre Prozesse während der Entwicklung des Organismus und für die Homöostase von erwachsenen Geweben. Interessanterweise kann oxidativer Stress diese Prozesse verändern und so zur Pathophysiologie von Krankheiten wie metastasierendem Krebs beitragen. Daher kann das Verständnis der Mechanismen, wie Zellen sich während Störungen im Redoxstatus an das ECM anheften und verbreiten, einen Einblick in Normal- und Krankheitszustände geben. Im Folgenden beschrieben ist ein stufenweisen Protokoll, das einen immunfluoreszenzbasierten Assay nutzt, um die Zelladhäsion und die Verbreitung von verewigten Fibroblastenzellen auf Fibronectin (FN) in vitro gezielt zu quantifizieren. Kurz gesagt, verankerungsabhängige Zellen werden in Suspension gehalten und dem ATM-Kinase-Inhibitor Ku55933 ausgesetzt, um oxidativen Stress zu induzieren. Die Zellen werden dann auf FN-beschichtete Oberfläche plattiert und dürfen für vorgegebene Zeiträume befestigt werden. Zellen, die noch angebracht sind, sind fixiert und mit fluoreszenzbasierten Antikörpermarkern der Adhäsion (z. B. Paxillin) und der Ausbreitung (z. B. F-Actin) gekennzeichnet. Die Datenerfassung und -analyse erfolgt mit allgemein verfügbaren Laborgeräten, einschließlich eines Epifluoreszenzmikroskops und einer frei verfügbaren Fidschi-Software. Dieses Verfahren ist sehr vielseitig und kann für eine Vielzahl von Zelllinien, ECM-Proteinen oder Inhibitoren modifiziert werden, um ein breites Spektrum biologischer Fragen zu untersuchen.
Einleitung
Zellmatrix-Adhäsionen (d.h. fokale Verklebungen) sind große und dynamische multimolekulare Proteinkomplexe, die zelladisieren und sich ausbreiten. Diese Prozesse sind entscheidend für die Gewebeentwicklung, Wartung und physiologische Funktion. Fokale Adhäsionen bestehen aus membrangebundenen Rezeptoren, wie Integrinen, sowie Gerüstproteinen, die Cytoskelett-Aktin mit der extrazellulären Matrix (ECM)1verbinden. Diese Komplexe sind in der Lage, auf physiochemische Hinweise in der extrazellulären Umgebung durch die Aktivierung verschiedener Signaltransduktionswege zu reagieren. Als solche dienen fokale Verklebungen als Signalzentren, um extrazelluläre mechanische Hinweise in eine Reihe von zellulären Prozessen zu verbreiten, einschließlich gerichteter Migration, Zellzyklusregulation, Differenzierung und Überleben1,2. Eine Gruppe von Signalmolekülen, die regulieren und mit fokalen Adhäsionen interagieren gehören Mitglieder der Rho-Familie von kleinen GTPases. Rho GTPases sind Schlüsselproteine, die die Zellmigration und Haftdynamik durch ihre spezifische raumzeitliche Aktivierung regulieren3. Es überrascht nicht, dass die Dysregulation der Rho-Proteinfunktion in eine Reihe von menschlichen Pathologien wie Metastasierung, Angiogenese und andere involviert ist. Von besonderem Interesse, zelluläre Redox-Status spielt eine vorherrschende Rolle bei der Modulation der Zellmigration und Adhäsion. Veränderungen in der Redoxhomöostase, wie z. B. die Zunahme der reaktiven Sauerstoffspezies (ROS), wurden nachgewiesen, um die Rho-Proteinaktivität sowie die Adhäsion bei einer Reihe von Zelltypen und menschlichen Krankheiten zu regulieren4,5,6 ,7,8. Zum Beispiel haben Personen, die an der neurologischen Erkrankung Ataxia-Teangiektasien (A-T) leiden, die durch eine Mutation in der DNA-Schädigung verursacht wird, eine Serin-/Threoninkinase A-T-mutiert (ATM), ein erhöhtes Risiko für metastasierenden Krebs9, 10. Der Verlust der ATM-Kinase-Aktivität bei diesen Patienten und Zelllinien, entweder durch genetische Mutation oder chemische Hemmung, führt zu einem hohen Gehalt an oxidativem Stress aufgrund einer Dysfunktion des Pentosephosphatwegs7,11, 12. Darüber hinaus haben neuere Studien aus dem Labor eine pathophysiologische Rolle für ROS in A-T hervorgehoben, indem die Zytoskelettdynamik (d. h. Haftung und Ausbreitung) als direktes Ergebnis der Aktivierung von GTPases der Rho-Familie in vitro5verändertwurde. Letztlich können diese Veränderungen in der zytoskelettalen Dynamik, die durch die Aktivierung der Rho-Familie verursacht werden, zu dem erhöhten Risiko für metastasierenden Krebs führen, das bei A-T-Patienten5,13festgestellt wird. Daher kann das Verständnis des Zusammenspiels zwischen Zell-Matrix-Wechselwirkungen während oxidativen Stresses Einblicke in die Regulation von Haftung und Ausbreitung liefern. Diese Studien können auch die Voraussetzungen für weitere Untersuchungen über eine mögliche Rolle der Rho-Familie GTPases in diesen Signalprozessen bereiten.
Beschrieben hierin ist ein Protokoll, um die frühe zelluläre Dynamik der Adhäsionsmontage zu studieren und sich während oxidativen Stresses zu verbreiten, der durch hemmung der ATM-Kinase-Aktivität verursacht wird. Dieser Test basiert auf dem gut charakterisierten Mechanismus der verankerungsabhängigen Zellen, der an dem ECM-Protein Fibronectin (FN) adhäsioniert ist. Wenn Zellen, die in Suspension gehalten werden, auf FN plattiert werden, koordinieren mehrere Rho GTPases die Steuerung des Aktin-Zytoskelett-Remodells3,14. Morphologische Veränderungen werden beobachtet, wenn sich Zellen von rundem und kreisförmigem Aussehen zu abgeflachten und erweiterten Veränderungen verschieben. Gleichzeitig mit diesen Beobachtungen ist die Entwicklung zahlreicher Matrixverklebungen mit dem ECM. Diese Veränderungen werden der biphasischen Aktivierung von RhoA mit Rac1 während der ersten Stunde zugeschrieben, da Zellen 15,16haften und verbreiten.
Eine Vielzahl von Methoden wurden verwendet, um Adhäsionmorphologie und Dynamik sowie Zellausbreitung zu untersuchen. Diese Methoden stützen sich jedoch auf ausgeklügelte langfristige, live-imaging totale interne Reflexionsfluoreszenz (TIRF) oder konfokale Mikroskopiesysteme. Daher müssen Benutzer Zugang zu spezialisierter Ausrüstung und Software haben. Darüber hinaus macht die für diese Bio-Imaging-Systeme erforderliche Einrichtungszeit die Erfassung früher Haftungsereignisse schwierig, insbesondere bei der gleichzeitigen Prüfung mehrerer Inhibitoren oder Behandlungsbedingungen.
Die hierin beschriebenen Methoden bieten eine einfache, wirtschaftliche und dennoch quantitative Möglichkeit, Parameter zu bewerten, die die Haftungsmontage und -verteilung in vitro steuern. Das Protokoll wird mit allgemein verfügbaren Laborgeräten wie einem Epifluoreszenzmikroskop und einer CCD-Kamera durchgeführt. Dieser Test beinhaltet die Anwendung von verankerungsabhängigen Zellen auf eine FN-beschichtete Oberfläche nach einer Periode oxidativer Belastung, die durch chemische Hemmung der ATM-Kinase-Aktivität verursacht wurde, die zuvor5nachgewiesen wurde. Nach der Beschichtung dürfen Zellen für bestimmte Zeiträume anhaften und haften. Nicht angeschlossene Zellen werden weggespült, während angehängte Zellen fixiert und mit fluoreszenzbasierten Antikörpern gegen Haftungsmarker (z. B. Paxillin) und Ausbreitung (z. B. F-Actin)2,5gekennzeichnet sind. Diese Proteine werden dann mit einem Epifluoreszenzmikroskop visualisiert und aufgezeichnet. Die anschließende Datenanalyse erfolgt mit frei verfügbarer Fidschi-Software. Darüber hinaus kann diese Methode angepasst werden, um die Haftdynamik unter einer Vielzahl von Bedingungen zu untersuchen, einschließlich verschiedener ECM-Proteine, der Behandlung mit verschiedenen Oxidationsmitteln/Zellkulturbedingungen oder einer Vielzahl von verankerungsabhängigen Zelllinien, um eine breite Palette von biologische Fragen.
Access restricted. Please log in or start a trial to view this content.
Protokoll
1. Vorbereitungen
HINWEIS: Das unten beschriebene Protokoll wurde für die Verwendung mit REF52-Zellen und ATM+/+ oder ATM-/- menschlichen Fibroblasten optimiert. Andere Zelltypen erfordern möglicherweise eine weitere Optimierung, wie in den abschnitts Abschnitten "Hinweise" und "Fehlerbehebung" unten beschrieben.
- Machen Sie 500 ml komplette zellkulturmittel für REF52-Zellen. Zu 500 ml hochglukoshaltigem Dulbecco-modifiziertem Eagle-Medium (DMEM) werden 10% FBS, 2 mM L-Glutamin und 100 Einheiten/ml Penicillin-Streptomycin hinzugefügt.
- Bereiten Sie eine 25-g/ml-Lösung von Fibronectin (FN) vor, indem Sie 300 l von 1 mg/ml FN-Lösung zu 12 ml steriler 1x Phosphatgepufferter Saline (PBS), pH 7,4 hinzufügen. Gut mischen.
- Bereiten Sie eine 0,5% (w/v) delipidierte (d.h. fettsäurefreie) Rinderserumalbumin (dlBSA) Lösung im serumfreien DMEM-Zellkulturmedium vor. Fügen Sie 0,5 g dlBSA auf 100 ml Serumfreies DMEM Medium hinzu. Mischen Sie die Lösung gut, aber nicht Wirbel. Steriles Filtern der Lösung in einen neuen sterilen Behälter, mit einem 0,22 M Spritzenfilter vor dem Gebrauch. Bei 4 °C lagern.
- Machen Sie 3,7% Paraformaldehydlösung, indem Sie 3,7 g Paraformaldehyd in 100 ml 1x PBS auflösen. Verwenden Sie sanfte Hitze und Rühren, um das Paraformaldehyd in Lösung zu bringen.
HINWEIS: Die Paraformaldehydlösung ist lichtempfindlich und sollte vor Licht geschützt werden. Es ist gut für bis zu einer Woche, wenn bei 4 °C gelagert.
VORSICHT: Paraformaldehyd ist giftig, entzündlich, ätzend und gesundheitsgefährdend. Überprüfen Sie das Materialsicherheitsdatenblatt auf Paraformaldehyd vor der Verwendung. Verwenden Sie die geeignete persönliche Schutzausrüstung bei der Handhabung, einschließlich Augenschild, Gesichtsschutz, Vollgesichts-Partikel-Atemschutzgerät, Handschuhe und Labormantel. - Bereiten Sie die Permeabilisationslösung vor, die 0,2 % nichtionisches Tensid in 1x PBS (v/v)enthält. Für 100 ml langsam 0,2 ml Triton X-100 bis 100 ml 1x PBS unter Rühren hinzufügen.
- Den Immunfluoreszenzblockigungspuffer mit 2,5% BSA, 5% Ziegenserum und 0,05% nichtionischem Tensid (w/v/v) in 1x PBS-Lösung lösen. Für 100 ml 5 ml Ziegenserum, 2,5 g BSA und 0,05 ml Triton X-100 in ca. 95 ml 1x PBS unter Rühren hinzufügen.
- Grow REF52 Zellen in DMEM komplettes Kulturmedium in einer 10 cm2 (oder jeder anderen Gefäßgröße) Zellkultur behandeltplatte in einem Zellkultur-Inkubator bei 37 °C und 5%CO2.
2. Beschichtung von Zellkulturplatten mit dem extrazellulären Matrixprotein Fibronectin
HINWEIS: Führen Sie diesen Abschnitt mit aseptischer Technik und sterilen Reagenzien in einer BSL-2-zertifizierten laminaren Durchflusshaube durch. Eine Übersicht über die wichtigsten Schritte vor beginn finden Sie in Abbildung 1A.
- Mit einer Gewebekultur zertifiziert 24-Well-Platte, legen Sie einen Glasdeckel (12-Cir-1) in jedem Brunnen. Beschriften Sie die Platte gemäß Abbildung 1B.
- Pipette 500 l der 25-g/ml-FN-Lösung für jeden Brunnen einer 24-Well-Platte.
- Pipette die Lösung über jede Abdeckungrutschen ein paar Mal, um eine gleichmäßige Beschichtung und vollständige sernweiter. Legen Sie den Deckel wieder auf die Platte.
- Inkubieren Sie die Platte in einem Zellkultur-Inkubator bei 37 °C und 5%CO2 für 1 h.
HINWEIS: Alternativ über Nacht bei 4 °C inkubieren. - Nach 1 h die Platte aus dem Inkubator entfernen und die FN-Lösung aus den Brunnen absaugen.
- Waschen Sie Brunnen dreimal mit 500 l 1x PBS. Aspirieren Sie die Endwäsche von 1x PBS.
- Blockbrunnen mit 500 l 0,5% dlBSA Lösung für mindestens 15 min bei 37 °C und 5%CO2.
- Aspirieren Sie die dlBSA-Lösung vor der Beschichtung von Zellen in Schritt 3 unten.
HINWEIS: Wenn Sie Platten lagern, fügen Sie jedem Deckelnachschlag nach dem Absetzen der dlBSA-Lösung 500 l 1x PBS hinzu. Die Platten können dann bis zu einer Woche bei 4 °C aufbewahrt werden.
3. Vorbereitung von ankerabhängigen Zellen für den Adhäsionstest
HINWEIS: Führen Sie diesen Abschnitt mit aseptischer Technik und sterilen Reagenzien in einer BSL-2-zertifizierten laminaren Durchflusshaube durch.
- Mindestens 30 min vor der Zellbeschichtung, vorwärmen Sie die folgenden Lösungen: DMEM Komplettmedium, dlBSA-Lösung, 1x PBS, 0,5% Trypsin-EDTA-Lösung und Trypsin-Neutralisierungsserum (TNS) in einem 37°C Wasserbad.
- Beginnend mit einer konfluenten Monoschicht von REF52-Zellen in einer 10 cm2 Schale, waschen Zellen zweimal mit 6 ml warmem 1x PBS. Serum verhungern die Zellen für mindestens 1 h (je nach Zelltyp) in 6 ml warmer dlBSA-Lösung bei 37 °C und 5%CO2.
- Waschen Sie Zellen mit 6 ml erwärmtem 1x PBS, aspirieren PBS und fügen Sie 1,5 ml warme 0,5% Trypsin-EDTA-Lösung hinzu.
- Legen Sie Zellen in einen Zellkultur-Inkubator bei 37 °C und 5%CO2 für 2 min.
- Beobachten Sie Zellen unter einem Lichtmikroskop, um sicherzustellen, dass die Ablösung abgeschlossen ist. Wenn Zellen nach dem Tippen auf die Platte auf der Bankplatte noch haften, kehren Sie für weitere 2 min zum 37 °C-Inkubator zurück. Versuchen Sie die Zellen so wenig Zeit wie nötig.
- Pipette 1,5 ml warme Trypsin-Neutralisationslösung (TNS) an die Schale, um die Trypsinisierung zu stoppen und abgetrennte Zellen zu sammeln. Pipette die Lösung nach oben und unten über den Boden der Platte mehrmals, um alle verbleibenden haftenden Zellen zu entfernen. Wenn Zellen klumpig erscheinen, trituieren Sie die Zellsuspension weiter, indem Sie sanft über der Rückseite der Schale nach oben und unten pfeifen.
- Zählen Sie Zellen mit Trypanblau-Ausschluss und einem Hämozytometer unter einem Lichtmikroskop. Alternativ können Sie einen automatisierten Zellenzähler verwenden.
- Entfernen Sie eine geeignete Menge an Zellen, um eine 1,0 - 3,0 x 104 Zellen/ml Zellsuspension in 5 ml dlBSA in einem 15 ml konischen Rohr zu erzeugen.
- Zentrifugenzellen bei 300 x g für 5 min mit einem festen Winkelrotor in einer Tischzentrifuge.
- Aspirieren Sie den Überstand aus dem Zellpellet und setzen Sie Zellen in insgesamt 7 ml warmer dlBSA-Lösung wieder aus. Lassen Sie nicht zu, dass die Zellen bei der Deckslipbeschichtung übermäßig konfluent sind, sondern gleichmäßig verteilt, wobei sich nur wenige Zellen berühren.
- Teilen Sie die Zellsuspension gleichmäßig in zwei 15 ml konische Röhren auf, eine für die Fahrzeugsteuerung (DMSO) und eine für Ku55933 (ATM-Kinase-Inhibitor, Oxidationsmittel)5. Stellen Sie sicher, dass jedes Rohr 3,5 ml der Zellsuspension enthält.
- Drehen Sie die Rohre mit einem Rohrrotator bei 37 °C für 90-120 min in einem Zellkultur-Inkubator.
- 30 min vor der Beschichtung, fügen Sie eine Endkonzentration von 10 M Ku55933 und DMSO (1:1.000) zu jedem jeweiligen Rohr hinzu. Legen Sie die Zellaufhängung für die verbleibende Zeit wieder auf den Rotator.
- Unmittelbar vor der Beschichtung der Zellen die 24-Well-Platte aus dem Inkubator holen und die dlBSA-Lösung aspirieren.
- Nach dem Drehen der Zellsuspension für 90-120 min, entfernen Sie 500 l Zellsuspension aus jeder Behandlungsgruppe und fügen Sie zu einem FN beschichteten Deckelin in der 24-Well-Platte aus Schritt 2, wie dargestellt(Abbildung 1B). Bringen Sie die Platte auf den 37 °C und 5% CO2-Zellkultur-Inkubator und die Zellsuspension zurück in die Rotation.
- Nach dem Beschichten der Zellsuspension auf den FN-Bedecktenlipslips lassen Sie die Zellen für die gewünschte Zeit daueren (z. B. 10 min, 15 min, 20 min, 30 min) und fahren Sie dann sofort mit Schritt 4 fort.
4. Zellfixierung und Antikörperfärbung zur Immunfluoreszenz
HINWEIS: Die folgenden Schritte werden unter nicht-sterilen Bedingungen und bei Raumtemperatur durchgeführt, sofern nicht anders angegeben.
- Nachdem die gewünschte Haftzeit verstrichen ist, die Zelllösung aus jedem Deckelinrutsch in der Platte aspirieren.
- Mit den Seiten des Brunnens, geben Sie vorsichtig 500 l 3,7% Paraformaldehydlösung auf jeden Deckelschlupf und warten Sie 10-15 min.
- Entfernen Sie die Paraformaldehydlösung und waschen Sie jeden Deckelschlupf mit 500 l 1x PBS insgesamt zweimal.
HINWEIS: Paraformaldehydabfälle entsprechend dem Umweltgesundheits- und Sicherheitsplan der Institution verantwortungsvoll entsorgen. - Aspirieren Sie die PBS, und permeabilisieren Zellen auf jedem Deckelschlupf mit 500 l von 0,2% Triton X-100 in 1x PBS (v/v) für 10-15 min bei Raumtemperatur.
- Waschen Sie jeden Deckelrutsch mit 500 l 1x PBS dreimal.
- Blockzellen auf jedem Coverslip mit 500 l Immunfluoreszenz-Blockierungspuffer, der 5% Ziegenserum, 2,5% BSA und 0,05% Triton X-100 enthält, gelöst in einer 1 x PBS-Lösung für 30-60 Minuten.
- Den primären Anti-Paxillin-Antikörper (1:250) im Sperrpuffer verdünnen. Mischen Sie gut und fügen Sie 200 l der Antikörperlösung zu jedem Coverslip hinzu. Bei Raumtemperatur mindestens 1 h inkubieren.
HINWEIS: Alternativ kann die primäre Antikörperlösung über Nacht bei 4 °C inkubiert werden. Es gibt viele häufige fokale Adhäsionsmarker, die für die Färbung von Haftkomplexen und nachfolgende FA-Analysen verwendet werden könnten. Dazu gehören Antikörper gegen die folgenden Proteine: Integrin-Untereinheiten (1, 5 oder V), Talin oder Vinculin2. - Die Antikörperlösung ansaugen und jeden Deckelschlupf mit 500 l 1x PBS dreimal für jeweils 10 min waschen. Schützen Sie die Proben von diesem Punkt aus vor Licht.
- Verdünnen Sie die Phalloidin F-Actin Sonde konjugiert mit dem roten fluoreszierenden Alexa 594 Farbstoff (1:1000) und Ziegenanti-Maus 488 fluoreszierenden Sekundärantikörper (1:400) in der gleichen Blockierpufferlösung. Mischen Sie gut und fügen Sie 200 l der Antikörperlösung zu jedem Coverslip für 30 min.
HINWEIS: Fluoreszierend konjugierte Sekundärantikörper anderer Arten können ebenfalls verwendet werden. Die Verwendung von Antikörpern anderer Arten erfordert jedoch eine Änderung des Sperrpufferserums. - Die Antikörperlösung ansaugen und jeden Deckelschlupf mit 500 l 1x PBS dreimal für jeweils 10 min waschen.
- Aspirieren Sie die 1x PBS und spülen Sie einmal mit 500 l dIH2O.
- Montieren Sie Abdeckungen auf Mikroskopschlitten mit Anti-Fade-Montagemedium, das DAPI enthält.
- Lassen Sie Mikroskop-Dias über Nacht im Dunkeln bei Raumtemperatur einstellen.
- Mikroskopdias im Dunkeln bei 4 °C für die Langzeitlagerung und bis zur Bildgebung lagern.
HINWEIS: Bild mit Standard-Immunfluoreszenztechniken. Es wird empfohlen, eine leistungsstarke Öl-Tauchlinse zu verwenden, um eine ausreichende Auflösung zu gewährleisten, um die Fokaladesionen und peripheren Rüschen an den Zellkanten zu beachten. Erfassen Sie Bilder von 20-30 Zellen in mehreren Sichtfeldern für jeden Deckschein unter jeder Behandlungsbedingung und -zeit. Aus kombinierten Replikationen sollte dies mindestens 60 Zellen ergeben, um statistische Analysen durchzuführen. Speichern und exportieren Sie Fluoreszenzbilder als . TIFF-Datei mit einer Auflösung von mindestens 300 dpi.
5. Quantifizierung von Spannungsfasern, Zellzirkularität und fokaler Adhäsionsbildung
HINWEIS: Die folgenden Bildanalysen werden mit der neuesten Version des Open-Source-Bildverarbeitungspakets Fiji Is Just Image J (Fiji) durchgeführt, das kostenlos unter (http://fiji.sc/) heruntergeladen werden kann.
- Allgemeine Bildverarbeitung
HINWEIS: Alle Bilder müssen für Rechenanalysen vorbereitet werden, indem die Schritte 5.1.1-5.1.5 unten ausgeführt werden (Abbildung 2). Danach können alle oder alle nachfolgenden Quantifizierungsverfahren ausgewählt werden.- Öffnen Sie die . TIFF Fluoreszenzbild mit Fidschi. Stellen Sie sicher, dass die Bilder 8-Bit- und Graustufen sind.
- Wählen Sie Image-Adjust-Window/level und wählen Auto (Abbildung 2A).
- Wählen Sie Prozesssubtrahieren Hintergrund aus, um die Hintergrundfluoreszenz zu subtrahieren. Aktivieren Sie Sliding Paraboloid und wählen Sie die Option eines Rolling Ball Radius von 50 Pixeln(Abbildung 2B).
HINWEIS: Um die richtige Größe für den rollenden Kugelradius zu überprüfen, wählen Sie das Linienwerkzeug aus und zeichnen Sie einen Radius auf der größten Haftung im Bild. Wählen Sie Messen aus, um die Länge der gezeichneten Linie zu überprüfen. Wenn der Wert des Radius zu groß ist, gehen Features, einschließlich Verklebungen, im Bild verloren. Wenn der Radius zu klein ist, entstehen im verarbeiteten Bild aufgrund von Hintergrundgeräuschen Artefakte. - Wählen Sie Bild-Anpassung- Helligkeit/Kontrast, um die Intensität der Haftung über dem Hintergrund zu überprüfen. Passen Sie ggf. an.
ANMERKUNG: Um die Helligkeit/kontrastieren und eine Sättigung des Signals zu vermeiden, verwenden Sie das Suchwerkzeug des Bildes, um das Histogramm zu untersuchen, um die Helligkeit/den Kontrast anzupassen. - Wählen Sie unter Analyse-Set-Messungendie folgenden Parameter aus: Fläche, mittlerer Grauwert, Formbeschreibungen und integrierte Dichte.
- Stressfaserbildungsanalyse
HINWEIS: Spannungsfasern können je nach Phänotyp mehrfach quantifiziert werden.- Zählen Sie die Anzahl der Zellen mit Spannungsfasern als Prozentsatz über die Gesamtzahl der Zellen. Diese Analyse ist am besten, wenn es visuelle Unterschiede in der Anzahl der Spannungsfasern gibt, die unter verschiedenen experimentellen Bedingungen gebildet werden.
- Zählen Sie die Anzahl der Spannungsfasern, die die Zelle transverqueren. Diese Analyse ermöglicht den Vergleich der Anzahl der pro Zelle gebildeten Spannungsfasern.
- Messen Sie die Gesamtfluoreszenzintensität des Phalloidins (z.B. F-Actin) Färbung pro Zelle17,18. Diese Methode wird drastische Erhöhungen/Abnahmen der Fluoreszenzintensität aufgrund von F-Actin-Färbung hervorheben.
- Stellen Sie die Messparameter in Schritt 5.1.5 oben ein.
- Wählen Sie das Freihandwerkzeug in der Fidschi-Symbolleiste aus und verfolgen Sie manuell die gewünschten Zellen. Wählen Sie Analyze-Measure. Es wird ein neues Fenster mit den ausgewählten Messparametern angezeigt.
- Wählen Sie das Freihandwerkzeug in der Fidschi-Symbolleiste aus, und verfolgen Sie manuell einen leeren Raum ohne Zellen. Wählen Sie Analyze-Measure. Diese Messung dient als Hintergrundfluoreszenz.
- Verwenden Sie die folgende Gleichung, um die gesamte F-Actin-Fluoreszenz pro Zelle zu bestimmen:
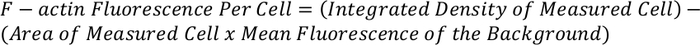
HINWEIS: Die resultierende Messung kann normalisiert und mit anderen Zellen verglichen werden, um F-Actin-Fluoreszenz pro Zelle zu geben.
- Zellzirkularitätsanalyse
ANMERKUNG: Informationen über den Zellbereich (ein Indikator für die Zellausbreitung im Laufe der Zeit) sowie die Zirkularität können ebenfalls aufgezeichnet werden. Diese Messung wird als Verhältnis zwischen 0 und 1 angegeben, um Zellen zu quantifizieren, die zu runden, bzw.- Wählen Sie das Freihandwerkzeug in der Fidschi-Symbolleiste aus, und verfolgen Sie eine einzelne Zelle. Wählen Sie Bild-Messen aus, und zeichnen Sie die Zellenflächen- und Umfangsmessungen für jede Zelle auf. Wiederholen Sie diesen Vorgang für jede Zelle.
HINWEIS: Unter der Funktion Messungen festlegen wird die Zirkularität als Messung der Formdeskriptoren (Schritt 5.1.5) bereitgestellt. - Zählen Sie manuell actin-angereicherte Rüschen oder Vorsprünge pro Zelle, wie in Abbildung 3 und Abbildung 4dargestellt.
- Wählen Sie das Freihandwerkzeug in der Fidschi-Symbolleiste aus, und verfolgen Sie eine einzelne Zelle. Wählen Sie Bild-Messen aus, und zeichnen Sie die Zellenflächen- und Umfangsmessungen für jede Zelle auf. Wiederholen Sie diesen Vorgang für jede Zelle.
- Fokale Haftungsanalyse
HINWEIS: Bevor Sie eine Fokushaftungsanalyse durchführen, installieren Sie das Mexikanische Hutfilter-Plugin auf der neuesten Version von Fidschi. Das folgende Protokoll wurde aus früheren Studien19,20,21geändert.- Wählen Sie Process-Enhance Local Contrast (Clahe) mit einer Blockgröße von 19, Histogramm-Bins 256 und einer maximalen Neigung von 6, ohne Maske und nicht schnell. (Abbildung 2C)
- Wählen Sie Prozess-Filter- Gaußsche Unschärfe mit einem Sigma (Radius) von 2.0, um das Bild zu filtern (Abbildung 2D).
- Wählen Sie Plugins-Mexikanische Hutfilter (Mhf) mit einem Radius von 2.0 (Abbildung 2E).
- Führen Sie Threshold aus, und wählen Sie Dunklen Hintergrund und Über/Unter aus, indem Sie entweder Huang oder Isodata als Schwellenwertmethode verwenden. Wählen Sie Auto-Threshold.
HINWEIS: Dieser Schritt stellt sicher, dass Adhäsionen hervorgehoben werden, sich aber auch voneinander unterscheiden. - Wählen Sie Partikel analysieren mit den folgenden ausgewählten Parametern aus: Größe=20, Pixel-Unendlichkeit und Zirkularität=0,00-0,99. Überprüfen Sie die Umrisse, um die ordnungsgemäße Erkennung und Trennung von Fokaladesionen zu gewährleisten.
HINWEIS: Diese Ergebnisse ergeben die Anzahl, Fläche und Formbeschreibung einzelner Fokaladesionen.
Access restricted. Please log in or start a trial to view this content.
Ergebnisse
Ein allgemeines Schema des Versuchsaufbaus
Abbildung 1 stellt das allgemeine Schema für das Zellhaftungs- und Ausbreitungsprotokoll dar, das mit dem Serumhunger von REF52-Zellen beginnt und mit einer rechnerischen Analyse der erfassten Fluoreszenzbilder endet. Die wichtigsten Schritte im Protokoll sind in der Zeitleiste dargestellt. Bemerkenswert ist, dass Schritt 2 des Protokolls die Herstellung der FN-beschichteten Abdeckungen beschreibt, die gleichzeitig mit Sch...
Access restricted. Please log in or start a trial to view this content.
Diskussion
Das hier beschriebene Protokoll ist eine vielseitige und wirtschaftliche Möglichkeit, eine Reihe von verankerungsabhängigen Zelltypen für die dynamische Zytoskelett-Remodellierung während der Zellstreuung schnell abzuschirmen. Insbesondere untersucht diese Methode quantitativ die Bildung von Spannungsfasern und fokalen Adhäsionwährend während des oxidativen Stresses, wenn Zellen an FN haften (Abbildung 1A). Darüber hinaus können diese zellulären Phänotypen eine regulatorische Roll...
Access restricted. Please log in or start a trial to view this content.
Offenlegungen
Die Autoren erklären, dass sie keine konkurrierenden finanziellen Interessen haben.
Danksagungen
Die Autoren danken Drs. Scott R. Hutton und Meghan S. Blackledge für die kritische Überprüfung des Manuskripts. Diese Arbeit wurde von den Forschungs- und Förderprogrammen der High Point University (MCS) und dem Biotechnology Program der North Carolina State University (MCS) finanziert.
Access restricted. Please log in or start a trial to view this content.
Materialien
| Name | Company | Catalog Number | Comments |
| 0.05% Trypsin-EDTA (1x) | Gibco by Life Technologies | 25300-054 | cell dissociation |
| 10 cm2 dishes | Cell Treat | 229620 | sterile, tissue culture treated |
| 15 mL conical tubes | Fisher Scientific | 05-539-5 | sterile |
| 1X Phosphate Buffered Saline | Corning Cellgro | 21-031-CV | PBS, sterile, free of Mg2+ and Ca2+ |
| 24-well cell culture treated plates | Fisher Scientific | 07-200-740 | sterile, tissue culture treated |
| 4°C refrigerator | Fisher Scientific | ||
| Mouse IgG anti-paxillin primary antibody (clone 165) | BD Transduction Laboratories | 610620 | marker of focal adhesions |
| Aspirator | Argos | EV310 | |
| Biosafety cabinet | Nuair | NU-477-400 | Class II, Type A, series 5 |
| Delipidated Bovine Serum Albumin (Fatty Acid Free) Powder | Fisher Scientific | BP9704-100 | dlBSA |
| Dimethyl Sulfoxide | Fisher Scientific | BP231-100 | organic solvent to dissolve Ku55933 |
| Dulbecco's Modified Eagle Media, High Glucose | Fisher Scientific | 11965092 | REF52 base cell culture medium |
| Fetal bovine serum | Fisher Scientific | 16000044 | certified, cell culture medium supplement |
| Fiji | National Institutes of Health | http://fiji.sc/ | image analysis program |
| Filter syringe | Fisher Scientific | 6900-2502 | 0.2 µM, sterile |
| Glass coverslips (12-Cir-1.5) | Fisher Scientific | 12-545-81 | autoclave in foil to sterilize |
| Goat anti-mouse IgG secondary antibody Alexa Fluor 488 | Invitrogen | A11001 | fluorescent secondary antibody, light sensitive |
| Goat Serum | Gibco by Life Technologies | 16210-064 | component of blocking solution for immunofluorescence |
| Hemocytometer | Fisher Scientific | 22-600-107 | for cell counting |
| Human Plasma Fibronectin | Gibco by Life Technologies | 33016-015 | FN |
| IX73 Fluorescence Inverted Microscope | Olympus | microscope to visualize fluorescence, cell morphology, counting and dissociation | |
| Ku55933 | Sigma-Aldrich | SML1109-25MG | ATM kinase inhibitor, inducer of reactive oxygen species |
| L-glutamine | Fisher Scientific | 25-030-081 | cell culture medium supplement |
| Monochrome CMOS 16 bit camera | Optimos | ||
| Paraformaldehyde | Sigma-Aldrich | P6148-500G | PFA, fixative for immunofluorescence |
| Penicillin-streptomycin | Fisher Scientific | 15-140-122 | P/S, antibiotic solution for culture medium |
| Alexa Fluor 594 phalloidin (F-actin probe) | Invitrogen | A12381 | marker of F-actin, light sensitive |
| ProLong Gold Anti-fade reagent with DAPI | Invitrogen | P36941 | cover slip mounting media including nuclear dye DAPI, light sensitive |
| REF52 cells | Graham, D.M. et. al. Journal of Cell Biology 2018 | ||
| Stir plate with heat control | Corning Incorporated | PC-420D | |
| Syringe | BD Biosciences | 309653 | 60 mL syringe |
| Tissue culture incubator | Nuair | ||
| Triton X-100 | Fisher Scientific | BP151-500 | detergent used to permeabilize cell membranes |
| Trypan Blue Solution | Fisher Scientific | 15-250-061 | for cell counting |
| Trypsin Neutralizing Solution (1x) | Gibco by Life Technologies | R-002-100 | TNS, neutralizes trypsin instead of fetal bovine serum |
| tube rotator | Fisher Scientific | 11-676-341 | |
| water bath | Fisher Scientific | FSGPD02 |
Referenzen
- Geiger, B., Bershadsky, A., Pankov, R., Yamada, K. M. Transmembrane crosstalk between the extracellular matrix--cytoskeleton crosstalk. Nature Reviews: Molecular Cell Biology. 2 (11), 793-805 (2001).
- Geiger, B., Yamada, K. M. Molecular architecture and function of matrix adhesions. Cold Spring Harbor Perspectives in Biology. 3 (5), (2011).
- Lawson, C. D., Burridge, K. The on-off relationship of Rho and Rac during integrin-mediated adhesion and cell migration. Small GTPases. 5, e27958(2014).
- Heo, J., Campbell, S. L. Mechanism of redox-mediated guanine nucleotide exchange on redox-active Rho GTPases. Journal of Biological Chemistry. 280 (35), 31003-31010 (2005).
- Tolbert, C. E., Beck, M. V., Kilmer, C. E., Srougi, M. C. Loss of ATM positively regulates Rac1 activity and cellular migration through oxidative stress. Biochemical and Biophysical Research Communications. 508 (4), 1155-1161 (2019).
- Hobbs, G. A., et al. Redox regulation of Rac1 by thiol oxidation. Free Radical Biology and Medicine. 79, 237-250 (2015).
- Zhang, Y., et al. Mitochondrial redox sensing by the kinase ATM maintains cellular antioxidant capacity. Science Signaling. 11 (538), (2018).
- Hobbs, G. A., Zhou, B., Cox, A. D., Campbell, S. L. Rho GTPases, oxidation, and cell redox control. Small GTPases. 5, e28579(2014).
- Shiloh, Y., Ziv, Y. The ATM protein kinase: regulating the cellular response to genotoxic stress, and more. Nature Reviews: Molecular Cell Biology. 14 (4), 197-210 (2013).
- Lang, L., et al. ATM-Mediated Phosphorylation of Cortactin Involved in Actin Polymerization Promotes Breast Cancer Cells Migration and Invasion. Cellular Physiology and Biochemistry. 51 (6), 2972-2988 (2018).
- Peter, Y., et al. Elevated Cu/Zn-SOD exacerbates radiation sensitivity and hematopoietic abnormalities of Atm-deficient mice. European Molecular Biology Organization Journal. 20 (7), 1538-1546 (2001).
- Takao, N., Li, Y., Yamamoto, K. Protective roles for ATM in cellular response to oxidative stress. Federation of European Biochemical Societies Letters. 472 (1), 133-136 (2000).
- Jansen, S., Gosens, R., Wieland, T., Schmidt, M. Paving the Rho in cancer metastasis: Rho GTPases and beyond. Pharmacology & Therapeutics. 183, 1-21 (2018).
- Berrier, A. L., Martinez, R., Bokoch, G. M., LaFlamme, S. E. The integrin beta tail is required and sufficient to regulate adhesion signaling to Rac1. Journal of Cell Science. 115 (Pt 22), 4285-4291 (2002).
- Arthur, W. T., Petch, L. A., Burridge, K. Integrin engagement suppresses RhoA activity via a c-Src-dependent mechanism. Current Biology. 10 (12), 719-722 (2000).
- Arthur, W. T., Burridge, K. RhoA inactivation by p190RhoGAP regulates cell spreading and migration by promoting membrane protrusion and polarity. Molecular Biology of the Cell. 12 (9), 2711-2720 (2001).
- Chandra, S., Kalaivani, R., Kumar, M., Srinivasan, N., Sarkar, D. P. Sendai virus recruits cellular villin to remodel actin cytoskeleton during fusion with hepatocytes. Molecular Biology of the Cell. 28 (26), 3801-3814 (2017).
- Fitzpatrick, M. Measuring Cell Fluorescence Using ImageJ. , https://theolb.readthedocs.io/en/latest/imaging/measuring-cell-fluorescence-using-imagej.html (2014).
- Berginski, M. E., Vitriol, E. A., Hahn, K. M., Gomez, S. M. High-resolution quantification of focal adhesion spatiotemporal dynamics in living cells. PLoS One. 6 (7), e22025(2011).
- Horzum, U., Ozdil, B., Pesen-Okvur, D. Step-by-step quantitative analysis of focal adhesions. MethodsX. 1, 56-59 (2014).
- Elosegui-Artola, A., et al. Image analysis for the quantitative comparison of stress fibers and focal adhesions. PLoS One. 9 (9), e107393(2014).
- Meller, J., Vidali, L., Schwartz, M. A. Endogenous RhoG is dispensable for integrin-mediated cell spreading but contributes to Rac-independent migration. Journal of Cell Science. 121 (Pt 12), 1981-1989 (2008).
- Donaldson, J. G. Immunofluorescence Staining. Current Protocols in Cell Biology. 69 (43), 1-7 (2015).
- Burry, R. W. Controls for immunocytochemistry: an update. Journal of Histochemistry and Cytochemistry. 59 (1), 6-12 (2011).
- Grashoff, C., et al. Measuring mechanical tension across vinculin reveals regulation of focal adhesion dynamics. Nature. 466 (7303), 263-266 (2010).
- Kumar, A., et al. Correction: Talin tension sensor reveals novel features of focal adhesion force transmission and mechanosensitivity. Journal of Cell Biology. 214 (2), 231(2016).
- Kumar, A., et al. Talin tension sensor reveals novel features of focal adhesion force transmission and mechanosensitivity. Journal of Cell Biology. 213 (3), 371-383 (2016).
- Friedrichs, J., Helenius, J., Muller, D. J. Quantifying cellular adhesion to extracellular matrix components by single-cell force spectroscopy. Nature Protocols. 5 (7), 1353-1361 (2010).
- Brown, M. A., et al. The use of mild trypsinization conditions in the detachment of endothelial cells to promote subsequent endothelialization on synthetic surfaces. Biomaterials. 28 (27), 3928-3935 (2007).
Access restricted. Please log in or start a trial to view this content.
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten