É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Examinando a dinâmica da adesão celular e disseminação de células epiteliais em fibronectina durante o estresse oxidativo
Neste Artigo
Resumo
Este método é útil para quantificar a dinâmica precoce da adesão celular e disseminação de células dependentes de ancoragem para a fibronectina. Além disso, este ensaio pode ser usado para investigar os efeitos da homeostase redox alterada na propagação da pilha e/ou nas vias de sinalização intracelular adesão-relacionadas da pilha.
Resumo
A adesão e disseminação de células para a matriz extracelular (ECM) são processos celulares essenciais durante o desenvolvimento organizador e para a homeostase de tecidos adultos. Curiosamente, o estresse oxidativo pode alterar esses processos, contribuindo assim para a fisiopatologia de doenças como o câncer metastático. Conseqüentemente, compreender o mecanismo (s) de como as pilhas anexam e espalham no ECM durante as perturbações no status redox pode fornecer a introspecção em Estados normais e da doença. É descrito abaixo um protocolo passo-sábio que utilize um ensaio immunofluorescence-baseado para quantificar especificamente a adesão e a propagação da pilha de pilhas imortalizado do fibroblasto no fibronectina (FN) in vitro. Brevemente, as células dependentes de ancoragem são mantidas em suspensão e expostas ao inibidor da quinase ATM Ku55933 para induzir o estresse oxidativo. As pilhas são chapeadas então na superfície FN-revestida e permitidas anexar por períodos de tempo predeterminados. As células que permanecem ligadas são fixas e rotuladas com marcadores de anticorpos de adesão à base de fluorescência (por exemplo, paxillin) e espalhamento (por exemplo, F-Actina). A aquisição e a análise dos dados são executadas usando o equipamento de laboratório geralmente disponível, incluindo um microscópio da epifluorescência e software livremente disponível de Fiji. Este procedimento é altamente versátil e pode ser modificado para uma variedade de linhas celulares, proteínas de ECM, ou nervos inibidores a fim examinar uma escala larga de perguntas biológicas.
Introdução
Aderências de matriz celular (ou seja, aderências focais) são grandes e dinâmicos complexos proteicos multimoleculares que mediam a adesão e disseminação celular. Estes processos são críticos para o desenvolvimento do tecido, a manutenção, e a função physiological. As aderências focais são compostas de receptores ligados à membrana, como as integrinas, bem como as proteínas de andaimes que ligam a actina citoesquelética à matriz extracelular (ECM)1. Esses complexos são capazes de responder a pistas fisioquímicas presentes no ambiente extracelular através da ativação de várias vias de transdução de sinalização. Como tal, as aderências focais servem como centros de sinalização para propagar pistas mecânicas extracelulares em vários processos celulares, incluindo migraçãodirecionada, regulaçãodo ciclo celular, diferenciação e sobrevida1,2. Um grupo de moléculas de sinalização que regulam e interagem com aderências focais inclui membros da família Rho de pequenos GTPases. As GTPases Rho são proteínas-chave que regulam a migração celular e a dinâmica de aderência através de sua ativação espaciotemporal específica3. Não surpreendentemente, a desregulação da função da proteína Rho tem sido implicada em um número de patologias humanas, tais como metástases, angiogênese, e outros. De particular interesse, o status redox celular desempenha um papel predominante na modulação da migração celular e adesão. Alterações na homeostase redox, como aumentos de espécies reativas de oxigênio (ROS), têm sido demonstradas para regular a atividade protéica de Rho, bem como adesão, em um número de tipos de células e doenças humanas4,5,6 ,7,8. Por exemplo, indivíduos que sofrem da desordem neurológica ataxia-Telangiectasia (A-T), que é causada por uma mutação no reparo de danos de DNA serina/treonina quinase A-T-Mutated (ATM), têm um risco aumentado de câncer metastático9, a 10. A perda da atividade da quinase ATM nesses pacientes e linhagens celulares, seja por meio da mutação genética ou da inibição química, resulta em altos níveis de estresse oxidativo devido à disfunção da via de fosfato pentose7,11, doze anos. Além disso, os estudos recentes do laboratório destacaram um papel patofisiológico para ROS em A-T alterando a dinâmica do cytoesquelético (isto é. adesão e espalhar) como um resultado direto de ativar GTPases da família de Rho in vitro5. Em última análise, essas alterações na dinâmica citoesquelética causadas pela ativação da família Rho podem levar ao aumento do risco de câncer metastático observado em pacientes com A-T5,13. Portanto, compreender a interação entre as interações célula-matriz durante o estresse oxidativo pode fornecer insights sobre a regulação da adesão e disseminação. Estes estudos podem igualmente ajustar o estágio para umas investigações mais adicionais em um papel possível para GTPases da família de Rho nestes processos de sinalização.
Descrito aqui é um protocolo para estudar a dinâmica celular precoce do conjunto de aderência e disseminação durante o estresse oxidativo causado pela inibição da atividade da quinase ATM. Este ensaio baseia-se no mecanismo bem caracterizado de adesão de células dependentes de ancoragem à fibronectina de proteína ECM (FN). Quando as células mantidas em suspensão são chapeadas na FN, vários Rho GTPases coordenam o controledo remodelamentocitoesquelético de actina14,15. Alterações morfológicas são observadas à medida que as células mudam de volta e circulares na aparência para achatado e expandido. Concomitante com estas observações é o desenvolvimento de aderências numerosas da matriz com o ECM. Essas alterações são atribuídas à ativação bifásica de Rhoa com Rac1 durante a primeira hora em que as células aderem e se espalham 15,16.
Uma variedade de métodos tem sido utilizada para examinar a morfologia da adesão e dinâmica, bem como a propagação celular. No entanto, esses métodos dependem de sistemas sofisticados de fluorescência de reflexão interna (TIRF) ou de microscopia confocal de longo prazo e de imagem viva. Assim, os usuários devem ter acesso a equipamentos e softwares especializados. Além disso, o tempo de set-up exigido por esses sistemas de bioimagem torna a captura de eventos de adesão precoce desafiador, especialmente quando o teste de múltiplos inibidores ou condições de tratamento simultaneamente.
Os métodos detalhados, aqui, fornecem uma maneira direta, econômica, contudo quantitativa de avaliar os parâmetros que governam o conjunto da adesão e que espalham in vitro. O protocolo é realizado usando equipamentos laboratoriais comumente disponíveis, como um microscópio de epifluorescência e câmera CCD. Este ensaio envolve a aplicação de células dependentes de ancoragem a uma superfície revestida com FN após um período de estresse oxidativo causado pela inibição química da atividade da quinase ATM, que tem sido demonstrada anteriormente5. Após o chapeamento, as células são permitidas para anexar e aderir para comprimentos especificados de tempo. As células não anexadas são lavadas, enquanto as células anexadas são fixas e marcadas com anticorpos à base de fluorescência para marcadores de aderência (por exemplo, paxillin) e espalhamento (por exemplo, F-Actina)2,5. Essas proteínas são então visualizadas e gravadas usando um microscópio de epifluorescência. A análise de dados subseqüente é executada usando o software livremente disponível de Fiji. Além disso, este método pode ser adaptado para examinar a dinâmica de adesão uma ampla gama de condições, incluindo diferentes proteínas de ECM, tratamento com vários oxidantes/condições de cultura celular ou uma variedade de linhas celulares dependentes de ancoragem para abordar uma ampla gama de questões biológicas.
Access restricted. Please log in or start a trial to view this content.
Protocolo
1. os preparativos
Nota: o protocolo descrito abaixo foi otimizado para o uso com células REF52 e ATM+/+ ou ATM-/- fibroblastos humanos. Outros tipos de célula podem exigir mais otimização conforme descrito nas seções de anotações e solução de problemas abaixo.
- Faça 500 mL de meio de cultura celular completo para células REF52. Para 500 mL de alta-glicose contendo Dulbecco ' s modificado Eagle ' s médio (DMEM) adicionar 10% FBS, 2 mM L-glutamina, e 100 unidades/mL penicilina-estreptomicina.
- Prepare uma solução de 25 μg/mL de fibronectina (FN) adicionando 300 μL de 1 mg/mL de solução FN a 12 mL de soro fisiológico tamponado de fosfato 1x estéril (PBS), pH 7,4. Misture bem.
- Prepare uma solução de albumina sérica bovina (dlBSA) de 0,5% (p/v) delipidada (ou seja, livre de ácidos graxos) em meio de cultura de células DMEM livre de soro. Adicionar 0,5 g de dlBSA a 100 mL de meio livre de soro DMEM. Misture bem a solução, mas não vórtice. Filtro estéril a solução em um novo recipiente estéril, usando um filtro de seringa de 0,22 μM antes de usar. Conservar a 4 ° c.
- Fazer 3,7% paraformaldeído solução dissolvendo 3,7 g de paraformaldeído em 100 mL de 1X PBS. Use calor suave e agitação para obter o paraformaldeído em solução.
Nota: a solução paraformaldeído é sensível à luz e deve ser protegida da luz. É bom para até uma semana quando armazenado em 4 ° c.
Cuidado: o paraformaldeído é tóxico, inflamável, corrosivo e um perigo para a saúde. Reveja a ficha de dados de segurança do material para o paraformaldeído antes da utilização. Use o equipamento de proteção pessoal apropriado ao manusear, incluindo protetor ocular, protetor facial, respirador de partículas de rosto completo, luvas e jaleco. - Prepare a solução de permeabilização contendo 0,2% de surfactante não iônico em 1X PBS (v/v). Para 100 mL, adicione lentamente 0,2 mL de Triton X-100 a 100 mL de 1X PBS, enquanto mexendo.
- Faça o tampão de obstrução da imunofluorescência que contem 2,5% BSA, soro de cabra de 5%, e 0, 5% surfactante non-ionic (w/v/v) dissolvido na solução de 1X PBS. Para 100 mL, adicione 5 mL de soro de cabra, 2,5 g de BSA e 0, 5 mL de Triton X-100 em ~ 95 mL 1X PBS, enquanto mexendo.
- Cresça REF52 pilhas no meio de cultura completo de DMEM em uma placa cultura-tratada da pilha de 10 cm2 (ou qualquer outro tamanho da embarcação) em uma incubadora da cultura de pilha em 37 ° c e em 5% co2.
2. placas da cultura da pilha do revestimento com o fibronectina da proteína da matriz extracelular
Nota: Realize esta seção usando a técnica asséptica e os reagentes estéreis em uma capa de fluxo laminar certificada BSL-2. Refira a Figura 1a para uma vista geral das etapas chaves antes do começo.
- Usando uma cultura de tecido certificada 24-placa bem, coloque uma lamínula de vidro (12-CIR-1) em cada poço. Rotule a chapa de acordo com a Figura 1b.
- Pipete 500 μL da solução de 25 μg/mL FN para cada poço de uma placa de 24 poços.
- Pipeta a solução sobre cada lamela algumas vezes para assegurar mesmo o revestimento e a submersão completa. Coloque a tampa de volta na placa.
- Incubar a placa em uma incubadora da cultura de pilha em 37 ° c e em 5% CO2 para 1 h.
Nota: Alternativamente, incubar durante a noite a 4 ° c. - Após 1 h, retire a placa da incubadora e aspirar a solução FN dos poços.
- Lave os poços três vezes com 500 μL de 1X PBS. Aspirar a lavagem final de 1X PBS.
- Bloqueie os poços com 500 μL de solução de dlBSA 0,5% por um mínimo de 15 min a 37 ° c e 5% CO2.
- Aspirar a solução de dlBSA antes de células de chapeamento na etapa 3 abaixo.
Nota: se armazenar placas, adicione 500 μL de 1X PBS a cada lamínula após aspiração da solução de dlBSA. As placas podem então ser mantidas em 4 ° c por até uma semana.
3. preparação de células dependentes de ancoragem para o ensaio de adesão
Nota: Realize esta seção usando a técnica asséptica e os reagentes estéreis em uma capa de fluxo laminar certificada BSL-2.
- Pelo menos 30 minutos antes do chapeamento de pilha, Pre-aqueça as seguintes soluções: meio completo de DMEM, solução de dlbsa, 1X PBS, solução do tripsina-EDTA de 0,5%, e soro neutralizando do tripsina (TNS) em um banho de água de 37 ° c.
- Começando com uma monocamada confluente de células REF52 em um prato de 10 cm2 , lave as células duas vezes com 6 ml de PBS 1x quente. O soro passa fome nas células por pelo menos 1 h (dependendo do tipo de célula) em 6 mL de solução de dlBSA morna a 37 ° c e 5% CO2.
- As células de lavagem com 6 mL de PBS aquecido 1x, aspiram PBS e adicionam 1,5 mL de solução morna de Trypsin-EDTA a 0,5%.
- Coloc pilhas em uma incubadora da cultura de pilha em 37 ° c e em 5% CO2 para ~ 2 min.
- Observe as células um microscópio de luz para garantir que o descolamento esteja completo. Se as pilhas são ainda aderentes após ter escutados a placa na parte superior de banco, retorne à incubadora do ° c 37 por uns 2 minutos adicionais Trypsinize as pilhas para tão pouco tempo como é necessário.
- Pipetar 1,5 ml de solução de neutralização de tripsina quente (TNS) para o prato para parar a tripsinização e coletar células destacadas. Pipeta a solução para cima e para baixo sobre a parte inferior da placa inúmeras vezes para remover todas as restantes células aderentes. Se as células pareceram desajeitando, triturar ainda mais a suspensão da célula gentilmente pipetando para cima e para baixo sobre a parte de trás do prato.
- Contagem de células usando Tripan exclusão azul e um hemocitômetro um microscópio de luz. Alternativamente, use um contador de células automatizado.
- Remova uma quantidade apropriada de células para criar uma suspensão de célula 1,0-3,0 x 104 Cells/ml em 5 ml de dlbsa em um tubo cônico de 15 ml.
- Centrifugue pilhas em ~ 300 x g por 5 minutos usando um rotor de ângulo fixo em uma centrífuga Table-Top.
- Aspirar o sobrenadante do pellet celular, e suspender as células em um total de 7 mL de solução de dlBSA quente. Não permita que as pilhas sejam excessivamente confluentes em cima do chapeamento do lamela, mas distribuído uniformente com poucas pilhas que tocam-se um outro.
- Divida uniformemente a suspensão celular em 2 15 mL de tubos cônicos, um para o controle do veículo sozinho (DMSO) e um para Ku55933 (inibidor da quinase ATM, oxidante)5. Assegure-se de que cada tubo contenha 3,5 mL da suspensão celular.
- Usando um rotator do tubo, giram em torno os tubos em 37 ° c por 90-120 minutos em uma incubadora da cultura de pilha.
- 30 min antes do revestimento, adicione uma concentração final de 10 μM Ku55933 e DMSO (1:1000) a cada tubo respectivo. Coloque a suspensão da célula de volta no rotor para o tempo restante.
- Imediatamente antes de galvanizar as células, recupere a placa de 24 poços da incubadora e aspire a solução de dlBSA.
- Depois de girar a suspensão celular por 90-120 min, remova 500 μL de suspensão celular de cada grupo de tratamento e acrescente a uma cobertura de FN revestida na placa de 24 poços da etapa 2 como ilustrado (Figura 1b). Retorne a placa ao ° c 37 e à incubadora da cultura da pilha de 5% CO2 e à suspensão da pilha de volta à rotação.
- Depois de chapeamento da suspensão da célula no FN cobertas-COVERSLIP, permitir que as células aderir para o período de tempo desejado (por exemplo, 10 min, 15 min, 20 min, 30 min) e, em seguida, prossiga imediatamente para o passo 4.
4. fixação celular e coloração de anticorpos para imunofluorescência
Nota: as seguintes etapas são executadas circunstâncias não estéreis e na temperatura ambiente a menos que indicado de outra maneira.
- Após o tempo desejado para a adesão passou, aspirar a solução celular de cada lamínula na placa.
- Usando os lados do poço, dispense suavemente 500 μL de solução de paraformaldeído 3,7% em cada lamínula e aguarde 10-15 min.
- Retire a solução paraformaldeído e lave cada lamínula com 500 μL de 1X PBS para um total de duas vezes.
Nota: descarte o resíduo de paraformaldeído de forma responsável, de acordo com o plano de saúde e segurança ambiental da instituição. - Aspirar a PBS, e permeabilizar as células em cada lamínula com 500 μL de 0,2% Triton X-100 em 1X PBS (v/v) para 10-15 min à temperatura ambiente.
- Lave cada lamínula com 500 μL de 1X PBS três vezes.
- Bloqueie as células em cada lamínula com 500 μL de tampão de bloqueio de imunofluorescência contendo 5% de soro de cabra, 2,5% BSA e 0, 5% Triton X-100 dissolvidos em uma solução de 1 X PBS por 30-60 minutos.
- Diluir o anticorpo primário anti-paxilina (1:250) no tampão de bloqueio. Misture bem e adicione 200 μL da solução de anticorpos a cada lamínula. Incubar à temperatura ambiente durante pelo menos 1 h.
Nota: Alternativamente, a solução de anticorpos primários pode ser incubada durante a noite a 4 ° c. Há muitos marcadores de adesão focal comuns que podem ser usados para a coloração de complexos de adesão e posterior análise de FA. Estes incluem anticorpos contra as seguintes proteínas: subunidades de integrina (β1, α5, ou αV), Talin, ou vinculin2. - Aspirar a solução de anticorpos e lavar cada lamínula com 500 μL de 1X PBS três vezes por 10 min cada. Proteja as amostras da luz deste ponto para a frente.
- Diluir a sonda de faloidina F-Actin conjugada ao corante fluorescente vermelho Alexa 594 (1:1000) e o anticorpo secundário fluorescente de cabra-anti-rato 488 (1:400) na mesma solução tampão de bloqueio. Misture bem e adicione 200 μL da solução de anticorpos a cada lamínula durante 30 min.
Nota: os anticorpos secundários conjugados fluorescently de outras espécies podem ser usados também. No entanto, o uso de anticorpos de outras espécies exigirá a modificação do soro tampão de bloqueio. - Aspirar a solução de anticorpos e lavar cada lamínula com 500 μL de 1X PBS três vezes por 10 min cada.
- Aspirar o 1X PBS e enxaguar uma vez com 500 μL de dIH2O.
- Monte as coberturas em lâminas de microscópio usando o meio de montagem antirdesvanecimento contendo DAPI.
- Deixe slides de microscópio para definir durante a noite no escuro à temperatura ambiente.
- Armazene as lâminas do microscópio no escuro em 4 ° c para o armazenamento a longo prazo e até a imagem latente.
Nota: imagem utilizando técnicas de imunofluorescência padrão. Recomenda-se usar uma lente objetiva de 60 x de imersão de óleo de alta potência para garantir uma resolução suficiente para observar as aderências focais e os babados periféricos nas bordas das células. Adquira imagens de 20-30 células em vários campos de visão para cada lamínula cada condição de tratamento e tempo. A partir de repetições combinadas, isso deve render pelo menos 60 células, a fim de realizar a análise estatística. Salvar e exportar imagens de fluorescência como um. TIFF com um mínimo de resolução de 300 dpi.
5. quantificando as fibras de estresse, circularidade celular, e formação de adesão focal
Nota: as análises de imagem a seguir são executadas usando a versão mais recente do pacote de processamento de imagens de código aberto Fiji is just Image J (Fiji), que pode ser baixado gratuitamente em (http://fiji.sc/).
- Processamento de imagem geral
Nota: todas as imagens precisarão ser preparadas para análises computacionais realizando as etapas 5.1.1-5.1.5 abaixo (Figura 2). Posteriormente, qualquer ou todos os procedimentos de quantificação subsequentes podem ser selecionados.- Abra o. Imagem de fluorescência TIFF usando Fiji. Verifique se as imagens são de 8 bits e escala de cinza.
- Selecione imagem-ajustar-janela/nível e selecione auto (Figura 2a).
- Selecione processo-subtrair fundo para subtrair a fluorescência de fundo. Verifique deslizando Paraboloid e selecione a opção de um raio de bola rolando de 50 pixels (Figura 2b).
Nota: para verificar o tamanho adequado para o raio da bola de rolamento, selecione a ferramenta de linha e desenhe um raio na maior aderência da imagem. Selecione Measure para verificar o comprimento da linha desenhada. Se o valor do raio for demasiado grande, as características que incluem aderências serão perdidas na imagem. Se o raio for muito pequeno, ele dará origem a artefatos na imagem processada devido ao ruído de fundo. - Selecione imagem-ajuste-brilho/contraste para verificar a intensidade da aderência sobre o fundo. Ajuste se necessário.
Observação: para otimizar o brilho/contraste e evitar saturar o sinal, use a ferramenta de pesquisa da imagem para examinar seu histograma para ajustar o brilho/contraste. - Selecione os seguintes parâmetros em analisar-definir medições: área, valor médio cinza, descritores de forma e densidade integrada.
- Análise de formação de fibras de stress
Nota: as fibras de stress podem ser quantificadas várias formas, dependendo do fenótipo.- Conte o número de células com fibras de tensão como uma porcentagem sobre o número total de células. Essa análise é melhor se houver diferenças visuais no número de fibras de estresse formadas diferentes condições experimentais.
- Conte o número de fibras de estresse que transvertem a célula. Esta análise permite a comparação do número de fibras de estresse formadas por célula.
- Medir a intensidade de fluorescência total dada pela coloração de faloidina (por exemplo, F-Actina) por célula17,18. Este método destacará aumentos/diminuições drásticas na intensidade da fluorescência devido à mancha de F-Actin.
- Defina os parâmetros de medição na etapa 5.1.5 acima.
- Selecione a ferramenta mão livre na barra de ferramentas de Fiji e Trace manualmente as células de interesse. Selecione analisar-medida. Uma nova janela aparecerá mostrando os parâmetros de medição selecionados.
- Selecione a ferramenta mão livre na barra de ferramentas de Fiji e Trace manualmente um espaço vazio sem células presentes. Selecione analisar-medida. Esta medida servirá como a fluorescência de fundo.
- Use a equação abaixo para determinar a fluorescência total de F-Actina por célula:
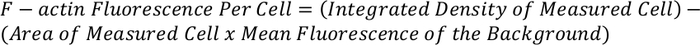
Nota: a medição resultante pode ser normalizada e comparada com outras células para dar fluorescência de F-Actina por célula.
- Análise de circularidade celular
Nota: informações sobre a área celular (um indicador de propagação celular ao longo do tempo), bem como, a circularidade também pode ser gravado. Esta medida é dada como uma relação entre 0 a 1 como uma maneira de quantificar as células que são alongadas para arredondar, respectivamente.- Selecione a ferramenta mão livre na barra de ferramentas Fiji e Trace uma célula individual. Selecione imagem-medida e registre a área da célula e as medições de perímetro para cada célula. Repita este procedimento para cada célula.
Nota: a função set medições , circularidade é fornecida como a medida descritores de forma (passo 5.1.5). - Conte manualmente as ruinas ou saliências enriquecidas com actina por célula, conforme representado na Figura 3 e na Figura 4.
- Selecione a ferramenta mão livre na barra de ferramentas Fiji e Trace uma célula individual. Selecione imagem-medida e registre a área da célula e as medições de perímetro para cada célula. Repita este procedimento para cada célula.
- Análise de aderência focal
Nota: antes de realizar a análise de aderência focal, instale o plugin mexicano Hat Filter na última versão do Fiji. O seguinte protocolo foi modificado a partir de estudos anteriores19,20,21.- Selecione processo-realce o contraste local (Clahe) usando um tamanho de bloco de 19, escaninhos do histograma 256, e uma inclinação máxima de 6, sem a máscara e não rápido. (Figura 2C)
- Selecione processo-filtros-Gaussian Blur com um Sigma (RADIUS) de 2,0 para filtrar a imagem (Figura 2D).
- Selecione plugins-Mexican Hat Filter (MHF) com um raio de 2,0 (Figura 2e).
- Execute o limiar e selecione fundo escuro e acima/abaixo usando Huang ou isodata como o método limiarização. Selecione limite automático.
Nota: esta etapa assegura-se de que as aderências estejam destacadas, mas igualmente distintas de uma outra. - Selecione analisar-analisar partículas com os seguintes parâmetros selecionados: Size = 20, pixels-Infinity e circularity = 0.00-0.99. Verific os contornos para assegurar a deteção e a separação apropriadas de aderências focais.
Nota: estes resultados produzem o número, a área, e a descrição da forma de aderências focais individuais.
Access restricted. Please log in or start a trial to view this content.
Resultados
Um esquema geral da set-up experimental
A Figura 1 representa o esquema geral para a adesão da célula e o protocolo de espalhamento começando com a inanição sérica de células REF52 e terminando com a análise computacional de imagens de fluorescência adquiridas. As etapas principais no protocolo são ilustradas na linha do tempo. Da nota, a etapa 2 do protocolo descreve a preparação dos COVERSLIP FN-revestidos, que devem ser executados simultaneamente com ...
Access restricted. Please log in or start a trial to view this content.
Discussão
O protocolo descrito aqui é uma maneira versátil e econômica de tela ràpida uma série de tipos ancoragem-dependentes da pilha para o citoesqueleto dinâmico que remodela durante espalhar da pilha. Em particular, este método examina quantitativamente a fibra de estresse e a formação de adesão focal durante o estresse oxidativo quando as células aderem à FN (Figura 1a). Além disso, estes fenótipos celulares podem sugerir um papel regulamentar para membros da família de Rho de GTP...
Access restricted. Please log in or start a trial to view this content.
Divulgações
Os autores declaram que não têm interesses financeiros concorrentes.
Agradecimentos
Os autores agradecem aos Drs. Scott R. Hutton e Meghan S. Blackledge pela revisão crítica do manuscrito. Este trabalho foi financiado pela High Point University ' s Research and patrocinado Programs (MCS) e o programa de biotecnologia da North Carolina State University (MCS).
Access restricted. Please log in or start a trial to view this content.
Materiais
| Name | Company | Catalog Number | Comments |
| 0.05% Trypsin-EDTA (1x) | Gibco by Life Technologies | 25300-054 | cell dissociation |
| 10 cm2 dishes | Cell Treat | 229620 | sterile, tissue culture treated |
| 15 mL conical tubes | Fisher Scientific | 05-539-5 | sterile |
| 1X Phosphate Buffered Saline | Corning Cellgro | 21-031-CV | PBS, sterile, free of Mg2+ and Ca2+ |
| 24-well cell culture treated plates | Fisher Scientific | 07-200-740 | sterile, tissue culture treated |
| 4°C refrigerator | Fisher Scientific | ||
| Mouse IgG anti-paxillin primary antibody (clone 165) | BD Transduction Laboratories | 610620 | marker of focal adhesions |
| Aspirator | Argos | EV310 | |
| Biosafety cabinet | Nuair | NU-477-400 | Class II, Type A, series 5 |
| Delipidated Bovine Serum Albumin (Fatty Acid Free) Powder | Fisher Scientific | BP9704-100 | dlBSA |
| Dimethyl Sulfoxide | Fisher Scientific | BP231-100 | organic solvent to dissolve Ku55933 |
| Dulbecco's Modified Eagle Media, High Glucose | Fisher Scientific | 11965092 | REF52 base cell culture medium |
| Fetal bovine serum | Fisher Scientific | 16000044 | certified, cell culture medium supplement |
| Fiji | National Institutes of Health | http://fiji.sc/ | image analysis program |
| Filter syringe | Fisher Scientific | 6900-2502 | 0.2 µM, sterile |
| Glass coverslips (12-Cir-1.5) | Fisher Scientific | 12-545-81 | autoclave in foil to sterilize |
| Goat anti-mouse IgG secondary antibody Alexa Fluor 488 | Invitrogen | A11001 | fluorescent secondary antibody, light sensitive |
| Goat Serum | Gibco by Life Technologies | 16210-064 | component of blocking solution for immunofluorescence |
| Hemocytometer | Fisher Scientific | 22-600-107 | for cell counting |
| Human Plasma Fibronectin | Gibco by Life Technologies | 33016-015 | FN |
| IX73 Fluorescence Inverted Microscope | Olympus | microscope to visualize fluorescence, cell morphology, counting and dissociation | |
| Ku55933 | Sigma-Aldrich | SML1109-25MG | ATM kinase inhibitor, inducer of reactive oxygen species |
| L-glutamine | Fisher Scientific | 25-030-081 | cell culture medium supplement |
| Monochrome CMOS 16 bit camera | Optimos | ||
| Paraformaldehyde | Sigma-Aldrich | P6148-500G | PFA, fixative for immunofluorescence |
| Penicillin-streptomycin | Fisher Scientific | 15-140-122 | P/S, antibiotic solution for culture medium |
| Alexa Fluor 594 phalloidin (F-actin probe) | Invitrogen | A12381 | marker of F-actin, light sensitive |
| ProLong Gold Anti-fade reagent with DAPI | Invitrogen | P36941 | cover slip mounting media including nuclear dye DAPI, light sensitive |
| REF52 cells | Graham, D.M. et. al. Journal of Cell Biology 2018 | ||
| Stir plate with heat control | Corning Incorporated | PC-420D | |
| Syringe | BD Biosciences | 309653 | 60 mL syringe |
| Tissue culture incubator | Nuair | ||
| Triton X-100 | Fisher Scientific | BP151-500 | detergent used to permeabilize cell membranes |
| Trypan Blue Solution | Fisher Scientific | 15-250-061 | for cell counting |
| Trypsin Neutralizing Solution (1x) | Gibco by Life Technologies | R-002-100 | TNS, neutralizes trypsin instead of fetal bovine serum |
| tube rotator | Fisher Scientific | 11-676-341 | |
| water bath | Fisher Scientific | FSGPD02 |
Referências
- Geiger, B., Bershadsky, A., Pankov, R., Yamada, K. M. Transmembrane crosstalk between the extracellular matrix--cytoskeleton crosstalk. Nature Reviews: Molecular Cell Biology. 2 (11), 793-805 (2001).
- Geiger, B., Yamada, K. M. Molecular architecture and function of matrix adhesions. Cold Spring Harbor Perspectives in Biology. 3 (5), (2011).
- Lawson, C. D., Burridge, K. The on-off relationship of Rho and Rac during integrin-mediated adhesion and cell migration. Small GTPases. 5, e27958(2014).
- Heo, J., Campbell, S. L. Mechanism of redox-mediated guanine nucleotide exchange on redox-active Rho GTPases. Journal of Biological Chemistry. 280 (35), 31003-31010 (2005).
- Tolbert, C. E., Beck, M. V., Kilmer, C. E., Srougi, M. C. Loss of ATM positively regulates Rac1 activity and cellular migration through oxidative stress. Biochemical and Biophysical Research Communications. 508 (4), 1155-1161 (2019).
- Hobbs, G. A., et al. Redox regulation of Rac1 by thiol oxidation. Free Radical Biology and Medicine. 79, 237-250 (2015).
- Zhang, Y., et al. Mitochondrial redox sensing by the kinase ATM maintains cellular antioxidant capacity. Science Signaling. 11 (538), (2018).
- Hobbs, G. A., Zhou, B., Cox, A. D., Campbell, S. L. Rho GTPases, oxidation, and cell redox control. Small GTPases. 5, e28579(2014).
- Shiloh, Y., Ziv, Y. The ATM protein kinase: regulating the cellular response to genotoxic stress, and more. Nature Reviews: Molecular Cell Biology. 14 (4), 197-210 (2013).
- Lang, L., et al. ATM-Mediated Phosphorylation of Cortactin Involved in Actin Polymerization Promotes Breast Cancer Cells Migration and Invasion. Cellular Physiology and Biochemistry. 51 (6), 2972-2988 (2018).
- Peter, Y., et al. Elevated Cu/Zn-SOD exacerbates radiation sensitivity and hematopoietic abnormalities of Atm-deficient mice. European Molecular Biology Organization Journal. 20 (7), 1538-1546 (2001).
- Takao, N., Li, Y., Yamamoto, K. Protective roles for ATM in cellular response to oxidative stress. Federation of European Biochemical Societies Letters. 472 (1), 133-136 (2000).
- Jansen, S., Gosens, R., Wieland, T., Schmidt, M. Paving the Rho in cancer metastasis: Rho GTPases and beyond. Pharmacology & Therapeutics. 183, 1-21 (2018).
- Berrier, A. L., Martinez, R., Bokoch, G. M., LaFlamme, S. E. The integrin beta tail is required and sufficient to regulate adhesion signaling to Rac1. Journal of Cell Science. 115 (Pt 22), 4285-4291 (2002).
- Arthur, W. T., Petch, L. A., Burridge, K. Integrin engagement suppresses RhoA activity via a c-Src-dependent mechanism. Current Biology. 10 (12), 719-722 (2000).
- Arthur, W. T., Burridge, K. RhoA inactivation by p190RhoGAP regulates cell spreading and migration by promoting membrane protrusion and polarity. Molecular Biology of the Cell. 12 (9), 2711-2720 (2001).
- Chandra, S., Kalaivani, R., Kumar, M., Srinivasan, N., Sarkar, D. P. Sendai virus recruits cellular villin to remodel actin cytoskeleton during fusion with hepatocytes. Molecular Biology of the Cell. 28 (26), 3801-3814 (2017).
- Fitzpatrick, M. Measuring Cell Fluorescence Using ImageJ. , https://theolb.readthedocs.io/en/latest/imaging/measuring-cell-fluorescence-using-imagej.html (2014).
- Berginski, M. E., Vitriol, E. A., Hahn, K. M., Gomez, S. M. High-resolution quantification of focal adhesion spatiotemporal dynamics in living cells. PLoS One. 6 (7), e22025(2011).
- Horzum, U., Ozdil, B., Pesen-Okvur, D. Step-by-step quantitative analysis of focal adhesions. MethodsX. 1, 56-59 (2014).
- Elosegui-Artola, A., et al. Image analysis for the quantitative comparison of stress fibers and focal adhesions. PLoS One. 9 (9), e107393(2014).
- Meller, J., Vidali, L., Schwartz, M. A. Endogenous RhoG is dispensable for integrin-mediated cell spreading but contributes to Rac-independent migration. Journal of Cell Science. 121 (Pt 12), 1981-1989 (2008).
- Donaldson, J. G. Immunofluorescence Staining. Current Protocols in Cell Biology. 69 (43), 1-7 (2015).
- Burry, R. W. Controls for immunocytochemistry: an update. Journal of Histochemistry and Cytochemistry. 59 (1), 6-12 (2011).
- Grashoff, C., et al. Measuring mechanical tension across vinculin reveals regulation of focal adhesion dynamics. Nature. 466 (7303), 263-266 (2010).
- Kumar, A., et al. Correction: Talin tension sensor reveals novel features of focal adhesion force transmission and mechanosensitivity. Journal of Cell Biology. 214 (2), 231(2016).
- Kumar, A., et al. Talin tension sensor reveals novel features of focal adhesion force transmission and mechanosensitivity. Journal of Cell Biology. 213 (3), 371-383 (2016).
- Friedrichs, J., Helenius, J., Muller, D. J. Quantifying cellular adhesion to extracellular matrix components by single-cell force spectroscopy. Nature Protocols. 5 (7), 1353-1361 (2010).
- Brown, M. A., et al. The use of mild trypsinization conditions in the detachment of endothelial cells to promote subsequent endothelialization on synthetic surfaces. Biomaterials. 28 (27), 3928-3935 (2007).
Access restricted. Please log in or start a trial to view this content.
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados