È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Esame della dinamica dell'adesione cellulare e della diffusione delle cellule epiteliali su fibronectina durante lo stress ossidativo
In questo articolo
Riepilogo
Questo metodo è utile per quantificare la dinamica precoce dell'adesione cellulare e la diffusione delle cellule dipendenti dall'ancoraggio sulla fibronectina. Inoltre, questo saggio può essere utilizzato per studiare gli effetti dell'omeostasi redox alterata sulla diffusione cellulare e/o sulle vie di segnalazione intracellulare correlate all'adesione alle cellule.
Abstract
L'adesione e la diffusione delle cellule sulla matrice extracellulare (ECM) sono processi cellulari essenziali durante lo sviluppo dell'organismo e per l'omeostasi dei tessuti adulti. È interessante notare che lo stress ossidativo può alterare questi processi, contribuendo così alla fisiopatologia di malattie come il cancro metastatico. Pertanto, comprendere i meccanismi di come le cellule si attaccano e si diffondono sull'ECM durante le perturbazioni nello stato di redox può fornire informazioni sugli stati normali e della malattia. Descritto di seguito è un protocollo graduale che utilizza un saggio basato sull'immunofluorescenza per quantificare specificamente l'adesione cellulare e la diffusione di cellule fibroblaste immortalate sulla fibronectina (FN) in vitro. In breve, le cellule dipendenti dall'ancoraggio sono tenute in sospensione ed esposte all'inibitore della chinasi ATM Ku55933 per indurre lo stress ossidativo. Le celle vengono quindi placcate su superficie rivestita FN e possono essere attaccate per periodi di tempo predeterminati. Le cellule che rimangono attaccate sono fisse ed etichettate con marcatori anticorpi basati sulla fluorescenza di adesione (ad esempio, paxillin) e diffusione (ad esempio, F-actin). L'acquisizione e l'analisi dei dati vengono eseguite utilizzando apparecchiature di laboratorio comunemente disponibili, tra cui un microscopio a epifluorescenza e un software Fiji liberamente disponibile. Questa procedura è altamente versatile e può essere modificata per una varietà di linee cellulari, proteine ECM, o inibitori al fine di esaminare una vasta gamma di domande biologiche.
Introduzione
Le adeguarzioni cellula-matrice (ad esempio, le adeguarzioni focali) sono grandi e dinamiche complessi proteici multimolecolari che mediano l'adesione e la diffusione cellulare. Questi processi sono fondamentali per lo sviluppo dei tessuti, la manutenzione e la funzione fisiologica. Le adere focali sono composte da recettori legati alla membrana, come le integrine, così come le proteine dell'impalcatura che collegano l'actina citoscheletrica alla matrice extracellulare (ECM)1. Questi complessi sono in grado di rispondere ai segnali fisiochimici presenti nell'ambiente extracellulare attraverso l'attivazione di varie vie di trasduzione di segnalazione. Come tale, le adesioni focali servono come centri di segnalazione per propagare segnali meccanici extracellulari in una serie di processi cellulari tra cui la migrazione diretta, la regolazione del ciclo cellulare, la differenziazione e la sopravvivenza1,2. Un gruppo di molecole di segnalazione che regolano e interagiscono con le ademioni focali includono membri della famiglia Rho di piccole GTPases. Rho GTPases sono proteine chiave che regolano la migrazione cellulare e la dinamica di adesione attraverso la loro specifica attivazione spatiotemporale3. Non sorprendentemente, la disregolazione della funzione della proteina Rho è stata implicata in una serie di patologie umane come metastasi, angiogenesi e altri. Di particolare interesse, lo stato di redox cellulare svolge un ruolo predominante nella modulazione della migrazione cellulare e dell'adesione. Sono state dimostrate alterazioni nell'omeostasi redox, come l'aumento delle specie reattive dell'ossigeno (ROS), per regolare l'attività della proteina Rho, così come l'adesione, in un certo numero di tipi di cellule e malattie umane4,5,6 ,7,8. Ad esempio, gli individui affetti dal disturbo neurologico ataxia-telangiectasia (A-T), che è causato da una mutazione del danno del DNA riparare serina / trenina chinasi A-T-mutato (ATM), hanno un aumentato rischio di cancro metastatico9, 10. La perdita di attività della chinasi ATM in questi pazienti e linee cellulari, sia per mutazione genetica che per inibizione chimica, si traduce in alti livelli di stress ossidativo a causa della disfunzione del percorso del fosfato pentozoico7,11, 12. Inoltre, recenti studi del laboratorio hanno evidenziato un ruolo patofisiologico per ROS in A-T alterando la dinamica citoscheletrica (ad esempio adesione e diffusione) come risultato diretto dell'attivazione di Rho family GTPases in vitro5. In definitiva, queste alterazioni nelle dinamiche citoscheletriche causate dall'attivazione della famiglia Rho possono portare all'aumento del rischio di cancro metastatico osservato nei pazienti A-T5,13. Pertanto, comprendere l'interazione tra le interazioni cellula-matrice durante lo stress ossidativo può fornire informazioni sulla regolazione dell'adesione e della diffusione. Questi studi possono anche mettere in stadio per ulteriori indagini su un possibile ruolo per la famiglia Rho GTPases in questi processi di segnalazione.
Descritto qui è un protocollo per studiare la dinamica cellulare precoce dell'assemblaggio dell'adesione e della diffusione durante lo stress ossidativo causato dall'inibizione dell'attività della chinasi ATM. Questo test si basa sul meccanismo ben caratterizzato dell'adesione delle cellule dipendenti dall'ancoraggio alla fibroctina della proteina ECM (FN). Quando le cellule mantenute in sospensione sono placcate su FN, diversi Rho GTPases coordinano il controllo dell'actin citoskeletal rimodellamento3,14. I cambiamenti morfologici si osservano quando le cellule si spostano da rotonde e circolari nell'aspetto a appiattite ed espanse. Concomitanti con queste osservazioni è lo sviluppo di numerose adeguarsi matrice con l'ECM. Questi cambiamenti sono attribuiti all'attivazione bifasica di RhoA con Rac1 durante la prima ora man mano che le cellule aderiscono e si diffondono 15,16.
Una varietà di metodi sono stati utilizzati per esaminare la morfologia e la dinamica dell'adesione, nonché la diffusione delle cellule. Tuttavia, questi metodi si basano su sofisticati sistemi di microscopia a lunga e lunga durata, a immagine reale di riflessione interna (TIRF) o microscopia confocale. Pertanto, gli utenti devono avere accesso a apparecchiature e software specializzati. Inoltre, il tempo di creazione richiesto da questi sistemi di bio-imaging rende difficile l'acquisizione di eventi di adesione precoce, soprattutto quando si testano più inibitori o condizioni di trattamento contemporaneamente.
I metodi descritti, nel presente documento, forniscono un modo semplice, economico, ma quantitativo per valutare i parametri che regolano l'assemblaggio dell'adesione e la diffusione in vitro. Il protocollo viene eseguito utilizzando apparecchiature di laboratorio comunemente disponibili, come un microscopio a epifluorescenza e una telecamera CCD. Questo saggio prevede l'applicazione di cellule dipendenti dall'ancoraggio a una superficie rivestita con FN dopo un periodo di stress ossidativo causato dall'inibizione chimica dell'attività della chinasi ATM, che è stato dimostrato in precedenza5. Dopo la placcatura, le cellule possono attaccare e aderire per periodi di tempo specificati. Le cellule non attaccate vengono lavate via, mentre le cellule attaccate sono fisse ed etichettate con anticorpi basati sulla fluorescenza ai marcatori di adesione (ad esempio, paxillin) e diffusione (ad esempio, F-actin)2,5. Queste proteine vengono quindi visualizzate e registrate utilizzando un microscopio a epifluorescenza. L'analisi dei dati successiva viene eseguita utilizzando il software Fiji liberamente disponibile. Inoltre, questo metodo può essere adattato per esaminare le dinamiche di adesione in una vasta gamma di condizioni, tra cui diverse proteine ECM, il trattamento con vari ossidanti / condizioni di coltura cellulare o una varietà di linee cellulari dipendenti dall'ancoraggio per affrontare un'ampia gamma di domande biologiche.
Access restricted. Please log in or start a trial to view this content.
Protocollo
1. Preparativi
NOTA: Il protocollo descritto di seguito è stato ottimizzato per l'uso con le cellule REF52 e ATMo ATM-/-- fibroblasti umani. Altri tipi di celle possono richiedere un'ulteriore ottimizzazione, come descritto nelle note e nelle sezioni di risoluzione dei problemi riportate di seguito.
- Crea 500 mL di supporto di coltura cellulare completo per le celle REF52. A 500 mL di alto glucosio contenente il mezzo dell'Aquila modificato di Dulbecco (DMEM) aggiungere 10% FBS, 2 mM L-glutamine, e 100 unità/mL penicillina-streptomiacina.
- Preparare una soluzione di fibronectina (FN) a 25 g/mL aggiungendo 300 -L di 1 mg/mL di soluzione FN a 12 mL di salina tampina tampina bufferdi (PBS), pH 7.4. Mescolare bene.
- Preparare una soluzione di scimo (dlBSA) dello 0,5% (w/v) delipidata (cioè priva di acidi grassi) in un mezzo di coltura cellulare DMEM privo di siero. Aggiungere 0,5 g di dlBSA a 100 mL di supporto DMEM senza siero. Mescolare bene la soluzione, ma non vortice. Sterile filtrare la soluzione in un nuovo contenitore sterile, utilizzando un filtro di siringa 0,22 m prima dell'uso. Conservare a 4 gradi centigradi.
- Rendere 3.7% soluzione paraformaldeide sciogliendo 3.7 g di paraformaldeide in 100 mL di 1x PBS. Utilizzare calore delicato e mescolando per ottenere la paraformaldeide in soluzione.
NOTA: La soluzione di paraformaldeide è sensibile alla luce e deve essere protetta dalla luce. E 'bene per un massimo di una settimana quando conservato a 4 gradi centigradi.
AVVISO: la paraformaldeide è tossica, infiammabile, corrosiva e un pericolo per la salute. Esaminare la scheda dati di sicurezza dei materiali per la paraformaldeia prima dell'uso. Utilizzare l'attrezzatura di protezione personale appropriata durante la manipolazione, tra cui scudo oculare, scudo facciale, respiratore di particelle integrale, guanti e camice da laboratorio. - Preparare la soluzione di permeabilizzazione contenente 0,2% di surfactant non ionico in 1x PBS (v/v). Per 100 mL, aggiungere lentamente 0,2 mL di Triton X-100 a 100 mL di 1x PBS, mescolando.
- Rendere il buffer di blocco dell'immunoorescenza contenente il 2,5% di BSA, il siero di capra del 5% e lo 0,05% di surfactant non ionico (w/v/v) sciolto nella soluzione PBS 1x. Per 100 mL, aggiungere 5 mL di siero di capra, 2,5 g di BSA e 0,05 mL di Triton X-100 in 95 mL 1x PBS, mescolando.
- Coltivare le cellule REF52 in DMEM mezzo di coltura completa in una piastra di 10 cm2 (o qualsiasi altra dimensione del vaso) trattata con coltura cellulare in un'incubatrice a coltura cellulare a 37 gradi centigradi e 5% di CO2.
2. Rivestimento delle piastre di coltura cellulare con la fibronectina della proteina della matrice extracellulare
NOTA: Eseguire questa sezione utilizzando la tecnica asettica e reagenti sterili in una cappa a flusso laminare certificata BSL-2. Fare riferimento alla Figura 1A per una panoramica dei passaggi chiave prima di iniziare.
- Utilizzando una piastra di 24 pozzetti certificata per la coltura dei tessuti, inserire un coperchio di vetro (12-Cir-1) in ogni pozzo. Etichettare la piastra in base alla figura 1B.
- Pipetta 500 l della soluzione FN da 25 g/mL per ogni pozzetto di una piastra di 24 pozze.
- Pipette la soluzione su ogni coverslip un paio di volte per garantire anche il rivestimento e l'ingorgire completo. Posizionare il coperchio sul piatto.
- Incubare la piastra in un'incubatrice a coltura cellulare a 37 gradi centigradi e il 5% di CO2 per 1 h.
NOTA: In alternativa, incubare durante la notte a 4 gradi centigradi. - Dopo 1 h, togliere la piastra dall'incubatrice e aspirare la soluzione FN dai pozzetti.
- Lavare i pozzi tre volte con 500 l- l di 1x PBS. Aspirate il lavaggio finale di 1x PBS.
- Pozze a blocchi con 500 gradi centigradi di 0,5% dlBSA per un minimo di 15 min a 37 e 5% di CO2.
- Aspirare la soluzione dlBSA prima di placcare le cellule nel passaggio 3 di seguito.
NOTA: Se si memorizzano le piastre, aggiungere 500 luna di 1x PBS a ogni coverslip dopo l'aspirazione della soluzione dlBSA. Le piastre possono quindi essere tenute a 4 gradi centigradi per un massimo di una settimana.
3. Preparazione delle cellule dipendenti dall'ancoraggio per il saggio di adesione
NOTA: Eseguire questa sezione utilizzando la tecnica asettica e reagenti sterili in una cappa a flusso laminare certificata BSL-2.
- Almeno 30 minuti prima della placcatura cellulare, pre-warm le seguenti soluzioni: DMEM completo medio, soluzione dlBSA, 1x PBS, 0.5% trypsin-EDTA soluzione, e trypsin siero neutralizzante (TNS) in un bagno d'acqua a 37 gradi centigradi.
- A partire da un monostrato confluente di cellule REF52 in un piatto di 10 cm2, lavare le cellule due volte con 6 mL di caldo 1x PBS. Il siero affama le cellule per almeno 1 h (a seconda del tipo di cellula) in 6 mL di soluzione dlBSA calda a 37 e 5% di CO2.
- Lavare le cellule con 6 mL di PBS 1x riscaldato, PBS aspirato e aggiungere 1,5 mL di calda soluzione 0,5% Trypsin-EDTA.
- Collocare le cellule in un'incubatrice a coltura cellulare a 37 e 5% di CO2 per 2 min.
- Osservare le cellule al microscopio leggero per assicurarsi che il distacco sia completo. Se le cellule sono ancora aderenti dopo aver toccato la piastra sul piano della panca, tornare all'incubatrice di 37 gradi centigradi per altri 2 min.
- Pipetta 1,5 mL di soluzione di neutralizzazione calda della prova (TNS) al piatto per fermare la prova e raccogliere le cellule staccate. Pipette la soluzione su e giù sul fondo della piastra numerose volte per rimuovere tutte le cellule rimanenti aderenti. Se le cellule appaiono goffe, triturare ulteriormente la sospensione cellulare pipetting su e giù sopra la parte posteriore del piatto.
- Contare le cellule utilizzando l'esclusione blu trypan e un emocitometro al microscopio luminoso. In alternativa, utilizzare un contatore cellulare automatizzato.
- Rimuovere una quantità appropriata di celle per creare un 1.0 - 3.0 x 104 celle / sospensione cellulare mL in 5 mL di dlBSA in un tubo conico da 15 mL.
- Centrifuga residui a 300 x g per 5 min utilizzando un rotore ad angolo fisso in una centrifuga da tavolo.
- Aspirare il supernatante dal pellet cellulare, e respendere le cellule in un totale di 7 mL di soluzione dlBSA calda. Non permettere che le cellule siano eccessivamente confluenti su placcatura coverslip, ma distribuite uniformemente con poche cellule che si toccano l'un l'altro.
- Dividere uniformemente le sospensioni cellulari in due tubi conici da 15 mL, uno per il controllo da solo del veicolo (DMSO) e uno per Ku55933 (inibitore della chinasi ATM, ossidante)5. Assicurarsi che ogni tubo contenga 3,5 mL della sospensione cellulare.
- Utilizzando un rotatore a tubo, ruotare i tubi a 37 gradi centigradi per 90-120 min in un incubatore di coltura cellulare.
- 30 min prima della placcatura, aggiungere una concentrazione finale di 10 : Ku55933 e DMSO (1:1,000) per ogni rispettivo tubo. Posizionare nuovamente la sospensione della cella sul rotatore per il tempo rimanente.
- Immediatamente prima di placcare le cellule, recuperare la piastra di 24 pozzetti dall'incubatrice e aspirare la soluzione dlBSA.
- Dopo aver ruotato la sospensione cellulare per 90-120 min, rimuovere 500 l-L di sospensione cellulare da ogni gruppo di trattamento e aggiungere a un coperchio rivestito FN nella piastra di 24 pozzetti dal punto 2 come illustrato (Figura 1B). Riportare la piastra all'incubatore di coltura cellulare 37 e 5% CO2 e la sospensione cellulare alla rotazione.
- Dopo aver placcato la sospensione cellulare sul FN coperto-coverslips, consentire alle cellule di aderire per il periodo di tempo desiderato (ad esempio, 10 min, 15 min, 20 min, 30 min) e quindi procedere immediatamente al passaggio 4.
4. Fissazione cellulare e colorazione degli anticorpi per l'immunofluorescenza
NOTA: i seguenti passaggi vengono eseguiti in condizioni non sterili e a temperatura ambiente, se non diversamente specificato.
- Dopo che il tempo desiderato per l'adesione è passato, aspirare la soluzione cellulare da ogni coverslip nella piastra.
- Utilizzando i lati del pozzo, erogare delicatamente 500 luna di 3,7% di soluzione di paraformaldeide su ogni coperchio e attendere 10-15 min.
- Rimuovere la soluzione di paraformaldeide e lavare ogni coverslip con 500 .
NOTA: Smaltire i rifiuti di paraformaldeide in modo responsabile, secondo il piano di salute e sicurezza ambientale dell'istituzione. - Aspirati le celle PBS e permeabilizzano su ogni coverslip con 500 litri di 0,2% Triton X-100 in 1x PBS (v/v) per 10-15 min a temperatura ambiente.
- Lavare ogni coperchio con 500 , luna di 1x PBS tre volte.
- Blocca le cellule su ogni coverslip con 500 l l di immunofluorescenza bloccante buffer contenenti 5% siero di capra, 2,5% BSA e 0,05% Triton X-100 disciolto in una soluzione 1 x PBS per 30-60 minuti.
- Diluire l'anticorpo anti-paxillin primario (1:250) nel buffer di blocco. Mescolare bene e aggiungere 200 l della soluzione anticorpale ad ogni coperchio. Incubare a temperatura ambiente per almeno 1 h.
NOTA: In alternativa, la soluzione anticorpale primaria può essere incubata durante la notte a 4 gradi centigradi. Ci sono molti marcatori di adesione focale comuni che potrebbero essere utilizzati per i complessi di adesione colorazione e la successiva analisi FA. Questi includono anticorpi contro le seguenti proteine: sottounità integrina (n. 1, 5 o V), talin o vinculin2. - Aspirare la soluzione anticorpale e lavare ogni coperchio con 500 luna di 1x PBS tre volte per 10 min ciascuno. Proteggere i campioni dalla luce da questo punto in avanti.
- Diluire la sonda Phalloidin F-actin coniugata al colorante fluorescente rosso Alexa 594 (1:1000) e effetto secondario fluorescente 488 (1:400) nella stessa soluzione di blocco del buffer. Mescolare bene e aggiungere 200 l della soluzione anticorpale ad ogni coverslip per 30 min.
NOTA: possono essere utilizzati anche anticorpi secondari coniugati fluorescentmente di altre specie. Tuttavia, l'uso di anticorpi di altre specie richiederà la modifica del siero tampone di blocco. - Aspirare la soluzione anticorpale e lavare ogni coperchio con 500 luna di 1x PBS tre volte per 10 min ciascuno.
- Aspirati il 1x PBS e risciacquare una volta con 500 L di dIH2O.
- Montare i coperchi sui vetrini al microscopio utilizzando un supporto di montaggio anti-dissolvenza contenente DAPI.
- Lasciare i vetrini al microscopio da impostare durante la notte al buio a temperatura ambiente.
- Conservare i vetrini al microscopio al buio a 4 gradi centigradi per la conservazione a lungo termine e fino all'imaging.
NOTA: Immagine che utilizza tecniche di immunofluorescenza standard. Si consiglia di utilizzare una lente obiettivo 60x ad immersione ad olio ad alta potenza per garantire una risoluzione sufficiente a notare le adere focali e le ruches periferiche ai bordi delle cellule. Acquisire immagini di 20-30 cellule in più campi di vista per ogni coverslip sotto ogni condizione di trattamento e il tempo. Da repliche combinate, questo dovrebbe produrre almeno 60 cellule al fine di eseguire analisi statistiche. Salvare ed esportare le immagini a fluorescenza come file . TIFF con una risoluzione minima di 300 dpi.
5. Quantificare le fibre da stress, la circolarità cellulare e la formazione di adesione focale
NOTA: le seguenti analisi delle immagini vengono eseguite utilizzando l'ultima versione del pacchetto di elaborazione di imaging open source Fiji Is Just Image J (Fiji), che può essere scaricata gratuitamente all'indirizzo (http://fiji.sc/).
- Elaborazione generale delle immagini
NOTA: tutte le immagini dovranno essere preparate per le analisi computazionali eseguendo i passaggi 5.1.1-5.1.5 riportati di seguito (Figura 2). Successivamente, è possibile selezionare una o tutte le procedure di quantificazione successive.- Aprire il file . Immagine di fluorescenza TIFF utilizzando Fiji. Assicurarsi che le immagini siano a 8 bit e in scala di grigi.
- Selezionare Image-Adjust-Window/level e Scegliere Auto (Figura 2A).
- Selezionare Sottrai processo-Sottrai sfondo per sottrarre la fluorescenza di sfondo. Selezionare Paraboloide scorrevole e selezionare l'opzione di un Raggio della palla rotante di 50 pixel (Figura 2B).
NOTA: per verificare le dimensioni corrette per il raggio della palla rotante, selezionare lo strumento Linea e disegnare un raggio sulla maggiore adesione nell'immagine. Selezionare Misura per verificare la lunghezza della linea disegnata. Se il valore del raggio è troppo grande, le funzioni incluse le ademe saranno perse nell'immagine. Se il raggio è troppo piccolo, darà origine a artefatti nell'immagine elaborata a causa del rumore di fondo. - Selezionare Regola immagine - Luminosità/Contrasto per verificare l'intensità dell'adesione sullo sfondo. Regolare se necessario.
NOTA: Per ottimizzare la luminosità/contrasto ed evitare di saturare il segnale, utilizzare lo strumento di ricerca dell'immagine per esaminarne l'istogramma per regolare la luminosità/contrasto. - Selezionare i seguenti parametri in Analizza misurazioni -Imposta:Area, Valore grigio medio, Descrittori forma e Densità integrata.
- Analisi della formazione delle fibre di stress
NOTA: Le fibre di stress possono essere quantificate in più modi a seconda del fenotipo.- Contare il numero di cellule con fibre di sollecitazione come percentuale sul numero totale di cellule. Questa analisi è migliore se ci sono differenze visive nel numero di fibre di stress formate in diverse condizioni sperimentali.
- Contare il numero di fibre di stress che trasversano la cellula. Questa analisi consente il confronto del numero di fibre di stress formate per cellula.
- Misurare l'intensità totale di fluorescenza data dalla colorazione phalloidin (ad esempio, F-actin) per cella17,18. Questo metodo evidenzierà drastici aumenti/diminuzioni dell'intensità della fluorescenza dovute alla colorazione F-actin.
- Impostare i parametri di misura nel passaggio 5.1.5 precedente.
- Selezionare lo strumento Mano libera nella barra degli strumenti Figi e tracciare manualmente le celle di interesse. Selezionare Analizza-Misura. Apparirà una nuova finestra che mostra i parametri di misura selezionati.
- Selezionare lo strumento Mano libera nella barra degli strumenti Figi e tracciare manualmente uno spazio vuoto senza celle presenti. Selezionare Analizza-Misura. Questa misurazione servirà come fluorescenza di fondo.
- Utilizzare l'equazione seguente per determinare la fluorescenza Totale F-actin per cella:
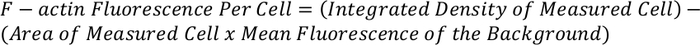
NOTA: La misura risultante può essere normalizzata e confrontata con altre cellule per dare la fluorescenza F-actin per cella.
- Analisi della circolarità cellulare
NOTA: è possibile registrare anche informazioni sull'area della cella (un indicatore di diffusione delle cellule nel tempo), nonché la circolarità. Questa misurazione viene fornita come rapporto tra 0 e 1 come un modo per quantificare le cellule che sono allungate per arrotondare, rispettivamente.- Selezionare lo strumento Mano libera nella barra degli strumenti Figi e tracciare una singola cella. Selezionare Image-Measure e registrare l'area della cella e le misure perimetrali per ogni cella. Ripetere questa procedura per ogni cella.
NOTA: nella funzione Imposta misurazioni, la circolarità viene fornita come misura dei descrittori di forma (passaggio 5.1.5). - Contare manualmente le volantine arricchite di actin o le sporgere per cella come illustrato nella Figura 3 e Figura 4.
- Selezionare lo strumento Mano libera nella barra degli strumenti Figi e tracciare una singola cella. Selezionare Image-Measure e registrare l'area della cella e le misure perimetrali per ogni cella. Ripetere questa procedura per ogni cella.
- Analisi dell'adesione focale
NOTA: Prima di eseguire l'analisi dell'adesione focale, installare il plug-in Mexican Hat Filter sull'ultima versione di Fiji. Il seguente protocollo è stato modificato da studi precedenti19,20,21.- Selezionare Contrasto locale di miglioramento del processo (Clahe) utilizzando una dimensione del blocco di 19, i contenitori dell'istogramma 256 e una pendenza massima di 6, senza maschera e non veloce. (Figura 2C)
- Selezionate Filtri di processo - Sfocatura gaussiana con un Sigma (Radius) di 2.0 per filtrare l'immagine ( Figura2D).
- Selezionare Plugins-Mexican Hat Filter (Mhf) con un Raggio di 2.0 (Figura 2E).
- Soglia di esecuzione e selezionare Sfondo scuro e Sopra/sotto utilizzando Huang o Isodata come metodo di soglia. Selezionare Soglia automatica.
NOTA: questo passaggio assicura che le ademi siano evidenziate, ma anche distinte l'una dall'altra. - Selezionare Analizza-Analizza particelle con i seguenti parametri selezionati: size: 20, pixel-infinity e circularity -0.00-0.99. Controllare i contorni per garantire il rilevamento e la separazione corretta delle adeguarzioni focali.
NOTA: questi risultati producono il numero, l'area e la descrizione della forma delle singole adeguarzioni focali.
Access restricted. Please log in or start a trial to view this content.
Risultati
Uno schema generale dell'allestito sperimentale
La figura 1 rappresenta lo schema generale per l'adesione cellulare e il protocollo di diffusione che iniziano con la fame del siero delle cellule REF52 e terminano con l'analisi computazionale delle immagini di fluorescenza acquisite. I passaggi chiave del protocollo sono illustrati nella sequenza temporale. Va notato, il passaggio 2 del protocollo descrive la preparazione dei coprilabbra rivestiti FN, che devono esse...
Access restricted. Please log in or start a trial to view this content.
Discussione
Il protocollo qui descritto è un modo versatile ed economico per vagliare rapidamente una serie di tipi di cellule dipendenti dall'ancoraggio per il rimodellamento dinamico del citoscheletro durante la diffusione delle cellule. In particolare, questo metodo esamina quantitativamente la fibra di stress e la formazione di adesione focale durante lo stress ossidativo quando le cellule aderiscono al FN (Figura 1A). Inoltre, questi fenotipi cellulari possono suggerire un ruolo normativo per i me...
Access restricted. Please log in or start a trial to view this content.
Divulgazioni
Gli autori dichiarano di non avere interessi finanziari concorrenti.
Riconoscimenti
Gli autori ringraziano il dottor Scott R. Hutton e Meghan S. Blackledge per la revisione critica del manoscritto. Questo lavoro è stato finanziato dai programmi di ricerca e sponsorizzazione (MCS) della High Point University e dal Programma biotecnologico presso la North Carolina State University (MCS).
Access restricted. Please log in or start a trial to view this content.
Materiali
| Name | Company | Catalog Number | Comments |
| 0.05% Trypsin-EDTA (1x) | Gibco by Life Technologies | 25300-054 | cell dissociation |
| 10 cm2 dishes | Cell Treat | 229620 | sterile, tissue culture treated |
| 15 mL conical tubes | Fisher Scientific | 05-539-5 | sterile |
| 1X Phosphate Buffered Saline | Corning Cellgro | 21-031-CV | PBS, sterile, free of Mg2+ and Ca2+ |
| 24-well cell culture treated plates | Fisher Scientific | 07-200-740 | sterile, tissue culture treated |
| 4°C refrigerator | Fisher Scientific | ||
| Mouse IgG anti-paxillin primary antibody (clone 165) | BD Transduction Laboratories | 610620 | marker of focal adhesions |
| Aspirator | Argos | EV310 | |
| Biosafety cabinet | Nuair | NU-477-400 | Class II, Type A, series 5 |
| Delipidated Bovine Serum Albumin (Fatty Acid Free) Powder | Fisher Scientific | BP9704-100 | dlBSA |
| Dimethyl Sulfoxide | Fisher Scientific | BP231-100 | organic solvent to dissolve Ku55933 |
| Dulbecco's Modified Eagle Media, High Glucose | Fisher Scientific | 11965092 | REF52 base cell culture medium |
| Fetal bovine serum | Fisher Scientific | 16000044 | certified, cell culture medium supplement |
| Fiji | National Institutes of Health | http://fiji.sc/ | image analysis program |
| Filter syringe | Fisher Scientific | 6900-2502 | 0.2 µM, sterile |
| Glass coverslips (12-Cir-1.5) | Fisher Scientific | 12-545-81 | autoclave in foil to sterilize |
| Goat anti-mouse IgG secondary antibody Alexa Fluor 488 | Invitrogen | A11001 | fluorescent secondary antibody, light sensitive |
| Goat Serum | Gibco by Life Technologies | 16210-064 | component of blocking solution for immunofluorescence |
| Hemocytometer | Fisher Scientific | 22-600-107 | for cell counting |
| Human Plasma Fibronectin | Gibco by Life Technologies | 33016-015 | FN |
| IX73 Fluorescence Inverted Microscope | Olympus | microscope to visualize fluorescence, cell morphology, counting and dissociation | |
| Ku55933 | Sigma-Aldrich | SML1109-25MG | ATM kinase inhibitor, inducer of reactive oxygen species |
| L-glutamine | Fisher Scientific | 25-030-081 | cell culture medium supplement |
| Monochrome CMOS 16 bit camera | Optimos | ||
| Paraformaldehyde | Sigma-Aldrich | P6148-500G | PFA, fixative for immunofluorescence |
| Penicillin-streptomycin | Fisher Scientific | 15-140-122 | P/S, antibiotic solution for culture medium |
| Alexa Fluor 594 phalloidin (F-actin probe) | Invitrogen | A12381 | marker of F-actin, light sensitive |
| ProLong Gold Anti-fade reagent with DAPI | Invitrogen | P36941 | cover slip mounting media including nuclear dye DAPI, light sensitive |
| REF52 cells | Graham, D.M. et. al. Journal of Cell Biology 2018 | ||
| Stir plate with heat control | Corning Incorporated | PC-420D | |
| Syringe | BD Biosciences | 309653 | 60 mL syringe |
| Tissue culture incubator | Nuair | ||
| Triton X-100 | Fisher Scientific | BP151-500 | detergent used to permeabilize cell membranes |
| Trypan Blue Solution | Fisher Scientific | 15-250-061 | for cell counting |
| Trypsin Neutralizing Solution (1x) | Gibco by Life Technologies | R-002-100 | TNS, neutralizes trypsin instead of fetal bovine serum |
| tube rotator | Fisher Scientific | 11-676-341 | |
| water bath | Fisher Scientific | FSGPD02 |
Riferimenti
- Geiger, B., Bershadsky, A., Pankov, R., Yamada, K. M. Transmembrane crosstalk between the extracellular matrix--cytoskeleton crosstalk. Nature Reviews: Molecular Cell Biology. 2 (11), 793-805 (2001).
- Geiger, B., Yamada, K. M. Molecular architecture and function of matrix adhesions. Cold Spring Harbor Perspectives in Biology. 3 (5), (2011).
- Lawson, C. D., Burridge, K. The on-off relationship of Rho and Rac during integrin-mediated adhesion and cell migration. Small GTPases. 5, e27958(2014).
- Heo, J., Campbell, S. L. Mechanism of redox-mediated guanine nucleotide exchange on redox-active Rho GTPases. Journal of Biological Chemistry. 280 (35), 31003-31010 (2005).
- Tolbert, C. E., Beck, M. V., Kilmer, C. E., Srougi, M. C. Loss of ATM positively regulates Rac1 activity and cellular migration through oxidative stress. Biochemical and Biophysical Research Communications. 508 (4), 1155-1161 (2019).
- Hobbs, G. A., et al. Redox regulation of Rac1 by thiol oxidation. Free Radical Biology and Medicine. 79, 237-250 (2015).
- Zhang, Y., et al. Mitochondrial redox sensing by the kinase ATM maintains cellular antioxidant capacity. Science Signaling. 11 (538), (2018).
- Hobbs, G. A., Zhou, B., Cox, A. D., Campbell, S. L. Rho GTPases, oxidation, and cell redox control. Small GTPases. 5, e28579(2014).
- Shiloh, Y., Ziv, Y. The ATM protein kinase: regulating the cellular response to genotoxic stress, and more. Nature Reviews: Molecular Cell Biology. 14 (4), 197-210 (2013).
- Lang, L., et al. ATM-Mediated Phosphorylation of Cortactin Involved in Actin Polymerization Promotes Breast Cancer Cells Migration and Invasion. Cellular Physiology and Biochemistry. 51 (6), 2972-2988 (2018).
- Peter, Y., et al. Elevated Cu/Zn-SOD exacerbates radiation sensitivity and hematopoietic abnormalities of Atm-deficient mice. European Molecular Biology Organization Journal. 20 (7), 1538-1546 (2001).
- Takao, N., Li, Y., Yamamoto, K. Protective roles for ATM in cellular response to oxidative stress. Federation of European Biochemical Societies Letters. 472 (1), 133-136 (2000).
- Jansen, S., Gosens, R., Wieland, T., Schmidt, M. Paving the Rho in cancer metastasis: Rho GTPases and beyond. Pharmacology & Therapeutics. 183, 1-21 (2018).
- Berrier, A. L., Martinez, R., Bokoch, G. M., LaFlamme, S. E. The integrin beta tail is required and sufficient to regulate adhesion signaling to Rac1. Journal of Cell Science. 115 (Pt 22), 4285-4291 (2002).
- Arthur, W. T., Petch, L. A., Burridge, K. Integrin engagement suppresses RhoA activity via a c-Src-dependent mechanism. Current Biology. 10 (12), 719-722 (2000).
- Arthur, W. T., Burridge, K. RhoA inactivation by p190RhoGAP regulates cell spreading and migration by promoting membrane protrusion and polarity. Molecular Biology of the Cell. 12 (9), 2711-2720 (2001).
- Chandra, S., Kalaivani, R., Kumar, M., Srinivasan, N., Sarkar, D. P. Sendai virus recruits cellular villin to remodel actin cytoskeleton during fusion with hepatocytes. Molecular Biology of the Cell. 28 (26), 3801-3814 (2017).
- Fitzpatrick, M. Measuring Cell Fluorescence Using ImageJ. , https://theolb.readthedocs.io/en/latest/imaging/measuring-cell-fluorescence-using-imagej.html (2014).
- Berginski, M. E., Vitriol, E. A., Hahn, K. M., Gomez, S. M. High-resolution quantification of focal adhesion spatiotemporal dynamics in living cells. PLoS One. 6 (7), e22025(2011).
- Horzum, U., Ozdil, B., Pesen-Okvur, D. Step-by-step quantitative analysis of focal adhesions. MethodsX. 1, 56-59 (2014).
- Elosegui-Artola, A., et al. Image analysis for the quantitative comparison of stress fibers and focal adhesions. PLoS One. 9 (9), e107393(2014).
- Meller, J., Vidali, L., Schwartz, M. A. Endogenous RhoG is dispensable for integrin-mediated cell spreading but contributes to Rac-independent migration. Journal of Cell Science. 121 (Pt 12), 1981-1989 (2008).
- Donaldson, J. G. Immunofluorescence Staining. Current Protocols in Cell Biology. 69 (43), 1-7 (2015).
- Burry, R. W. Controls for immunocytochemistry: an update. Journal of Histochemistry and Cytochemistry. 59 (1), 6-12 (2011).
- Grashoff, C., et al. Measuring mechanical tension across vinculin reveals regulation of focal adhesion dynamics. Nature. 466 (7303), 263-266 (2010).
- Kumar, A., et al. Correction: Talin tension sensor reveals novel features of focal adhesion force transmission and mechanosensitivity. Journal of Cell Biology. 214 (2), 231(2016).
- Kumar, A., et al. Talin tension sensor reveals novel features of focal adhesion force transmission and mechanosensitivity. Journal of Cell Biology. 213 (3), 371-383 (2016).
- Friedrichs, J., Helenius, J., Muller, D. J. Quantifying cellular adhesion to extracellular matrix components by single-cell force spectroscopy. Nature Protocols. 5 (7), 1353-1361 (2010).
- Brown, M. A., et al. The use of mild trypsinization conditions in the detachment of endothelial cells to promote subsequent endothelialization on synthetic surfaces. Biomaterials. 28 (27), 3928-3935 (2007).
Access restricted. Please log in or start a trial to view this content.
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon