Method Article
Hämogene Reprogrammierung menschlicher Fibroblasten durch erzwungene Expression von Transkriptionsfaktoren
In diesem Artikel
Erratum Notice
Zusammenfassung
Dieses Protokoll demonstriert die Induktion eines hämogen Programms in humanen dermalen Fibroblasten durch erzwungene Expression der Transkriptionsfaktoren GATA2, GFI1B und FOS zur Erzeugung hämatopoetischer Stamm- und Vorläuferzellen.
Zusammenfassung
Die zellulären und molekularen Mechanismen, die der Spezifikation menschlicher hämatopoetischer Stammzellen (HSCs) zugrunde liegen, bleiben schwer fassbar. Strategien zur Rekapitulation menschlicher HSC-Emergence in vitro sind erforderlich, um Einschränkungen bei der Untersuchung dieses komplexen Entwicklungsprozesses zu überwinden. Hier beschreiben wir ein Protokoll zur Erzeugung hämatopoetischer Stamm- und Vorläuferzellen aus menschlichen dermalen Fibroblasten mit einem direkten Zell-Reprogrammierungsansatz. Diese Zellen durchlaufen einen hämogen Zwischenzelltyp, der dem für die HSC-Spezifikation charakteristischen endotheliaal-hematopoetischen Übergang (EHT) ähnelt. Fibroblasten wurden über Transduktion mit GATA2-, GFI1B- und FOS-Transkriptionsfaktoren auf hämogene Zellen umprogrammiert. Diese Kombination von drei Faktoren induzierte morphologische Veränderungen, Expression hämogener und hämatopoetischer Marker und dynamische EHT-Transkriptionsprogramme. Umprogrammierte Zellen erzeugen hämatopoetische Nachkommenschaft und bevölkern drei Monate lang immundefizienten Mäusen. Dieses Protokoll kann an die mechanistische Zerlegung des menschlichen EHT-Prozesses angepasst werden, wie hier durch die Definition von GATA2-Zielen in den frühen Phasen der Umprogrammierung veranschaulicht wird. Daher bietet die humane hämogene Reprogrammierung einen einfachen und transportablen Ansatz, um neuartige Marker und Regulatoren der menschlichen HSC-Entstehung zu identifizieren. In Zukunft kann eine getreue Induktion hämogener Schicksale bei Fibroblasten zur Erzeugung patientenspezifischer HSCs für die Transplantation führen.
Einleitung
Definitive hämatopoetische Stamm- und Vorläuferzellen (HSPCs) entstehen in der Aorta-Gonad-Mesonephros-Region und Plazenta aus endothelialer Vorläufern mit hämogener Kapazität durch einen endothelial-hematopoetischen Übergang (EHT)1, 2. Hämogene Vorläufer (HPs) exprimiert sowohl endotheliale als auch hämatopoetische Marker, aber ihre genaue Identifizierung bleibt schwer fassbar, insbesondere im menschlichen System. Obwohl es sich um einen relativ konservierten Prozess bei Säugetieren handelt, unterscheidet sich die entwicklung der hämatopoetischen Stammzell (HSC) immer noch signifikant zwischen Menschen und Mausmodellen3,4. Daher sind In-vitro-Ansätze zur Rekapitulation der menschlichen HSC-Entwicklung erforderlich.
Die Differenzierung von pluripotenten Stammzellen (PSCs) zu HSCs, obwohl vielversprechend, hat in den letzten 20 Jahren nur begrenzten Erfolg erzielt, vor allem aufgrund der verfügbaren Differenzierungsprotokolle, die zu primitiven hämatopoetischen Vorläufern mit schlechter Engraftmentierung führen. Fähigkeit5,6,7. Alternativ wurden direkte Methoden zur Zellneuprogrammierung angewendet, um HSPC-ähnliche Zellen aus mehreren Zelltypen zu generieren, indem Transkriptionsfaktoren (TFs)8,9verwendet werden. Insbesondere die Überexpression von drei TFs, Gata2, Gfi1b und cFos, wandelte embryonale Fibroblasten von Maus durch ein HP-Zwischenprodukt mit einem definierten Phänotyp (Prom1+Sca-1+CD34+CD45-)10in HSPCs um. Dieser Prozess ähnelte dem EHT, das im Embryo und in der Plazenta während der Spezifikation der endgültigen Hämatopoese auftritt. Dieser Phänotyp ermöglichte die Identifizierung und Isolierung einer Population von HPs in der Maus Plazenta, die nach kurzfristiger Kultur und Notch-Aktivierung seriell transplantierbare HSCs11generierte.
Bisher wurde kein Phänotyp nachgewiesen, der menschliche HSCs von ihren Vorläufern unterscheidet, aber einige Moleküle sind dafür bekannt, in aufkommenden HSCs exprimiert zu werden. Integrin alpha 6 (ITGA6 oder CD49f) ist stark in langfristig wiederbevölkernden HSCs exprimiert, die unreifsten Zellen im HSC-Fach12und Angiotensin-converting Enzym (ACE oder CD143) ist in CD34 negativen hämatopoetischen Vorläufern in embryonalen blutbildenden Geweben13vorhanden.
Kürzlich haben wir gezeigt, dass menschliche Versionen der drei TFs, GATA2, FOS und GFI1B menschliche dermale Fibroblasten (HDFs) zu HPs mit kurzfristiger Transplantationskapazität14umprogrammieren. In den Anfangsphasen der Neuprogrammierung greift GATA2 offenes Chromatin ein und rekrutiert GFI1B und FOS, um Fibroblasten gene zu unterdrücken und endotheliale und hämatopoetische Gene zu aktivieren. Induzierte Zellen hochexprimiert CD49f und ACE, und enthielt einen kleinen Prozentsatz der Zellen, die den HSPC-Marker CD34 exprimieren. Das CD9-Gen, das in HSCs15 exprimiert wird und für HSC homing16wichtig ist, erwies sich als direktes Ziel von GATA2 und zu den am stärksten hochregulierten Genen in umprogrammierten Zellen14. CD9 kann daher einen zusätzlichen Marker für PS menschlicher endgültiger Hämatopoese darstellen.
In diesem Protokoll beschreiben wir die Erzeugung von HSPC-ähnlichen Zellen aus menschlichen Fibroblasten durch erzwungene Expression von GATA2, GFI1B und FOS sowie eine angepasste Methode zur Chromatin-Immunpräzipation (ChIP)-Sequenzierung (seq) Neuprogrammierung. TFs wurden in einem Doxycyclin (DOX)-induzierbaren lentiviralen Vektor (pFUW-tetO) kodiert, der ein Tetracyclin-Antwortelement (TRE) und einen minimalen CMV-Promotor enthält, und wurden zusammen mit einem konstitutiven Vektor transduziert, der das umgekehrte Tetracyclin enthält. Transaktivatorprotein (pFUW-M2rtTA). Wenn DOX (analog von Tetracyclin) nach der Transduktion hinzugefügt wird, bindet es an das rtTA-Protein, das mit dem TRE interagiert und TF-Transkription (Tet-On-System) ermöglicht. Das Verfahren dauert 25 Tage. Für ChIP-seq-Experimente wurden HDFs mit markierten Versionen von GATA2 (pFUW-tetO-3xFLAG-GATA2) und GFI1B (pLV-tetO-HA-GFI1B) sowie pFUW-tetO-FOS- und TF-Bindungsstellen zwei Tage nach der DOX-Supplementierung analysiert.
Letztlich bietet die hämogene Reprogrammierung menschlicher Fibroblasten ein in vitro-traktierbares System zur Untersuchung der Mechanismen, die der humanen Entwicklungshämatopoese zugrunde liegen, und eine potenzielle Quelle patientenspezifischer HSPCs für die zukünftige klinische Anwendung.
Protokoll
Dieses Protokoll wurde gemäß den Richtlinien der Ethikkommission für Humanforschung der Universität durchgeführt und sollte in Übereinstimmung mit den individuellen institutionellen Richtlinien durchgeführt werden.
1. Reagenz-Vorbereitung
- Für Dulbeccos modifiziertes Eagle-Medium (DMEM)/20% fetales Rinderserum (FBS), hohe Glukose DMEM mit Natriumpyruvat mit 20% FBS, 1% Penicillin-Streptomycin (Pen/Strep), 1% L-Glutamin, 1% nicht-essentielle Aminosäuren und 10-4-M 2- mischen Mercaptoethanol.
- Für vollständiges DMEM mit Natriumpyruvat mit 10% FBS, 1% Pen/Strep und 1% L-Glutamin mit natriumpyruvat mischen.
- Für hämatopoetisches Medium hämatopoetisches Medium (Materialtabelle) mit 10-6 M Hydrocortison und 1% Pen/Strep mischen.
- Verwenden Sie Phosphat-gepufferte Saline (PBS) ohne Kalzium oder Magnesium.
2. Menschliche Dermale Fibroblasten-Isolierung
HINWEIS: HDFs können bei zertifizierten Lieferanten(Materialtabelle)erworben werden. In diesem Fall können Fibroblasten erweitert und direkt in Umprogrammierungsexperimenten verwendet werden (Abschnitt 4). Alternativ können HDFs von Spendern isoliert werden. Wenn Fibroblasten von verschiedenen Spendern isoliert sind, halten Sie die Proben bei allen Schritten des Protokolls voneinander getrennt. Etikettenschilder/Brunnen und Sammelrohre mit der Identifikationsnummer jedes Spenders.
- Erhalten Sie HDFs von 3 mm runden Hautpunschbiopsien, die von qualifizierten Ärzten durchgeführt werden.
- Beschichten Sie drei Brunnen einer gewebekulturbehandelten 6-Well-Platte mit 500 l 0,1% Gelatine und inkubieren Sie 20 min bei 37 °C.
- Die verbleibende Gelatinelösung aspirieren und 750 L DMEM/20% FBS zu jedem Brunnen hinzufügen. Die gesamte Oberfläche des Brunnens sollte mit Medium bedeckt werden.
- 1,5 ml DMEM/20% FBS auf die Innenseite eines sterilen 100 mm Petrischalendeckels geben und den Tropfen mit Hilfe einer 5 ml serologischen Pipette verteilen.
- Legen Sie die Hautbiopsie im Medium mit sterilisierter Zange auf den Deckel.
- Sezieren Sie die Hautbiopsie in neun identische Abschnitte, mit einem sterilisierten Skalpell, um die Biopsie an Ort und Stelle zu halten und ein zweites Skalpell zu schneiden.
- Legen Sie drei Biopsiestücke pro Brunnen mit spitzen Zangen. Stellen Sie sicher, dass die Stücke an der Unterseite des Brunnens befestigt werden.
- Legen Sie einen 22 mm Deckelschlupf auf die Stücke und drücken Sie etwas Unterdruck.
- Inkubieren Sie die Platte bei 37 °C, 5%CO2, für eine Woche. Überprüfen Sie die Zellen täglich und fügen Sie alle 2 Tage 200 L DMEM/20 % FBS hinzu, um das verdampfte Medium zu ersetzen.
- Nach einer Woche bis zu 2 ml DMEM/20% FBS addieren und Medium alle 2 bis 3 Tage ersetzen.
-
Durchgangszellen im Verhältnis 1:4, wenn Brunnen konfluent sind (ca. 4-8 Wochen).
- Bereiten Sie 0,1% gelatinebeschichtete Gewebekultur-behandelte 6-Well-Platten vor.
- Absaugmedium aus Brunnen bei 80% Koninfluenza und einmal mit 1 ml PBS waschen.
- Entfernen Sie den Deckelrutsch mit steriler Zange und legen Sie den Deckelinin in einen neuen Brunnen einer 6-Well-Platte, mit der Gewebeseite nach oben.
HINWEIS: Auch Zellen, die am Deckschein befestigt waren, werden geerntet. - Fügen Sie 500 l Dissoziationslösung(Materialtabelle) pro Brunnen (einschließlich Brunnen mit den Abdeckungen) und inkubieren bei 37 °C, 5%CO2 für 5 x 10 min. Überprüfen Sie, wann Zellen beginnen, von der Unterseite des Brunnens zu steigen und inaktivieren Die Dissoziationslösung durch Zugabe von 500 l DMEM/20% FBS zu jedem Brunnen.
- Sammeln Sie Fibroblasten aus allen Brunnen in einem 15 ml konischen Rohr. Fügen Sie den Brunnen zusätzliches Medium hinzu, um die verbleibenden Zellen zu sammeln. Zentrifugieren Sie das Rohr bei 350 x g für 5 min.
- In der Zwischenzeit fügen Sie jedem Brunnen von zuvor gelatinebeschichteten Platten 500 l DMEM/20% FBS hinzu.
- Aspirieren Sie Medium und resuspendieren Sie Fibroblasten in 6 ml DMEM/20% FBS.
- Fügen Sie jedem Brunnen 500 l Fibroblastsuspension hinzu (insgesamt zwei 6-Well-Platten pro Probe/Spender). Inkubieren Sie Zellen über Nacht bei 37 °C, 5%CO2.
- Am nächsten Tag 1 ml DMEM/20% FBS zu jedem Brunnen hinzufügen. Ersetzen Sie Medium durch 2 ml DMEM/20% FBS alle 2 bis 3 Tage, bis die Brunnen zu 80 % konfluent sind.
- Wiederholen Sie Abschnitt 2.11 für drei konfluente Brunnen, bis der dritte Durchgang erreicht ist.
-
Einfrieren von Fibroblasten aus Konfluentbrunnen (Passagen 1 und 3).
- Medium aus den Brunnen aussaugen und einmal mit 1 ml PBS waschen.
- Dissoziieren und Sammeln von Fibroblasten, wie in den Schritten 2.11.4 und 2.11.5 beschrieben.
- Zählzellen mit einem Hämozytometer und Zentrifugieren sie das Rohr bei 350 x g für 5 min.
- Nach zentrifugieren, Aspirieren Medium und Resuspend Fibroblasten in FBS mit 10% DMSO bei einer Dichte von 5 x 105 Zellen/ml.
- 1 ml der Zellsuspension pro Kryovial und Gefrierzellen über Nacht bei -80 °C mit einem Gefrierbehälter hinzufügen. Bewegen Sie Fläschchen auf -150 °C (flüssiger Stickstoff) für die langfristige Lagerung.
3. Lentivirale Produktion
- Wachsen Sie HEK293T-Zellen in einer 100 mm Gewebekultur behandelten Schale mit 10 ml komplettem DMEM, bei 37 °C, 5%CO2, bis die Konfluenz erreicht ist.
- Am Tag vor der Transfektion, aspirieren Medium und waschen Sie die Schale sorgfältig mit 5 ml PBS.
- Nach dem Entfernen von PBS 1,5 ml Dissoziationslösung hinzufügen und bei 37 °C, 5%CO2 für 5 x 10 min inkubieren, um Zellen von der Schale zu trennen.
HINWEIS: Es wird empfohlen, sowohl PBS als auch Dissoziationslösung vor der Verwendung zu erwärmen, damit die Zellen keinen thermischen Schock erleiden. - Inaktivieren Sie die Dissoziationslösung mit 3 ml komplettem DMEM und übertragen Sie die Zellsuspension in ein 15 ml konisches Rohr. Waschen Sie die Schale mit 5 ml komplettem DMEM, um die verbleibenden angeschlossenen Zellen zu entfernen und dieses Volumen auf das 15 ml konische Rohr zu übertragen.
- Zentrifugenzellsuspension bei 350 x g für 5 min.
- Aspirieren Sie Überstand und teilen Sie das Zellpellet gleichmäßig zwischen sechs 100 mm Gewebekultur behandelten Gerichten in einem Endvolumen von 10 ml komplettem DMEM pro Schale. Die Zellen sollten zum Zeitpunkt der Transfektion etwa 60 % konfluent sein.
- Am nächsten Tag vermischen sich die transfekten Zellen mit dem Plasmid wie folgt:
HINWEIS: Dieser Teil des Protokolls beschreibt die Produktion von Lentiviren in einer 100 mm Gewebekultur-behandelten Schale pro Plasmidmischung. Um höhere Mengen an lentiviralem Überstand für die Konzentration zu erhalten, verwenden Sie mindestens vier 100 mm HEK293T Zellkulturschalen pro Mischung.- In einem 15 ml konischen Röhrchen fügen Sie 10 g der drei Transferplasmide zusammen: 3,33 g pFUW-tetO-GATA2 (Addgene Plasmid #125028)14, 3,33 g pFUW-tetO-GFI1B (Addgene #125597)14 und 3,33 g pFUW-tetO-FOS (Addgene #125598)14, plus 10 g der2. Generation psPAX2 Verpackungsvektor codierung die Gag, Pol, Tat und Rev Gene (Addgene #12260) und 5 'g pMD2.G Hüllkurvenvektor Kodierung der VSV-G Gen (Addgene #12259). Fügen Sie Wasser bis zu 500 l hinzu.
- In zwei neuen 15 ml konischen Röhrchen fügen Sie 10 g FUW-M2rtTA-Plasmid (Addgene #20342)17, 10 g psPAX2-Verpackungsvektor und 5 g pMD2.G Hüllkurvenvektor zu jeder Röhre hinzu. Fügen Sie Wasser bis zu 500 l hinzu. Ein Rohr wird als Steuerung verwendet werden.
- Zu jedem Rohr 62,5 l von 2 M CaCl2hinzufügen. Lassen Sie als Nächstes Blasen in jede Mischung mit einer Pasteur Pipette los, die in einen Pipettenregler eingesetzt wird. Während sich Blasen bilden, pufferte Pipette 500 l N,N-Bis(2-Hydroxyethyl)-2-Aminoethanesulfonsäure (BES) salin (pH 7,1, 25 °C), mit einer P1000 Pipette, tropfenweise gegen die Pasteurpipetundundund und auf das Gemisch.
- Röhren bei Raumtemperatur für mindestens 15 min inkubieren. Die Mischungen werden nach einiger Zeit leicht bewölkt erscheinen.
- In der Zwischenzeit, Aspirieren Medium aus HEK293T Zellschalen (am Vortag durchgelassen) und fügen Sie 10 ml komplette DMEM ohne Antibiotika. Seien Sie vorsichtig und fügen Sie Medium langsam hinzu, da HEK293T-Zellen semi-adherent sind.
- Jede einzelne Mischung (ca. 1 ml) gleichmäßig und tropfenweise in getrennte Gerichte verteilen und über Nacht bei 37 °C, 5%CO2inkubieren.
- Medium durch 4 ml komplette DMEM, 24 h nach inkubationszeit. Über Nacht bei 37 °C, 5%CO2inkubieren. Wenn verfügbar, inkubieren Sie stattdessen bei 32 °C, 5%CO2, da die reduzierte Temperatur die Halbwertszeit der lentiviralen Partikel erhöht.
- Sammeln Sie Überstand mit lentiviralen Partikeln dreimal zu einem 50 ml konischen Rohr. Mischen Sie an dieser Stelle keine verschiedenen lentiviralen Partikel. Jedes Gericht führt zu 12 ml Lentiviralüberstand. Vier Gerichte der gleichen viralen Zubereitung passen in eine 50 ml konische Röhre.
VORSICHT: Führen Sie die lentivirale Sammlung in einem Labor der Biosicherheitsstufe 2 in einer laminaren Durchflusshaube für lentivirale Arbeit durch und legen Sie viral kontaminierte Abfälle (Rohre, Spitzen, Geschirr) in einen geeigneten Behälter für biogefährliche Materialien.- Führen Sie die erste Sammlung 16 h nach der letzten Inkubation durch und fügen Sie 4 ml vollständiges DMEM hinzu. Inkubieren bei 37 °C, 5%CO2.
- Nehmen Sie die zweite Sammlung 8 h nach der ersten auf das gleiche Rohr, fügen Sie 4 ml komplette DMEM und inkubieren bei 37 °C, 5%CO2.
- Machen Sie die dritte Sammlung 16 h nach der zweiten auf die gleiche Röhre und entsorgen Sie die Gerichte.
HINWEIS: Lentivirale Überstoffe nach jeder Entnahme bei 4 °C lagern.
- Filtern Sie jeden lentiviralen Überstand mit einem 0,45 m-Protein-Bindungsfilter mit einer Celluloseacetatmembran (Materialtabelle) zu einem sauberen Rohr.
- Fügen Sie maximal 15 ml gefilterten Überstand zu einer Zentrifugalfiltereinheit mit einer regenerierten Zellulosemembran (Materialtabelle)hinzu und drehen Sie bei 4.000 x g für 25 min bei 4 °C. Entsorgen Sie den Durchfluss. Eine viskose Flüssigkeit, die Lentiviren enthält, verbleibt in der Filtereinheit.
- Wiederholen Sie Schritt 3.13, indem Sie 15 ml Überstand auf der Oberseite der Filtereinheit hinzufügen, bis kein lentiviraler Überstand mehr vorhanden ist.
HINWEIS: Wenn es nur wenige Milliliter Überstand zu konzentrieren, verringern Sie die Spinnzeit auf 10 min. Wenn sich noch zusätzliche Flüssigkeit (nicht viskos) am Filter befindet, zentrifugieren Sie für weitere 10 min. - Machen Sie Aliquots (50 bis 200 L je nach anfänglichem Überstandvolumen) jeder Art von konzentrierten Lentiviren und lagern Sie bei -80 °C für die Langzeitlagerung (1 bis 2 Jahre) oder bei 4 °C für die kurzfristige Lagerung (1 bis 2 Wochen).
HINWEIS: Konzentrierte oder nicht konzentrierte Lentiviren können auch frisch eingesetzt werden. Nicht wieder einfrieren und auftauen, da dies zu einem reduzierten Titer führt.
4. Hämogene Neuprogrammierung
HINWEIS: Verwenden Sie HDFs mit einer Durchgangszahl von drei (P3) oder höher (bis P10), um Neuprogrammierungsexperimente durchzuführen.
- Eine 100 mm Gewebekultur behandelte Schale mit 5 ml 0,1% Gelatine beschichten und 20 min bei 37 °C inkubieren. Die verbleibende Gelatinelösung ansaugen.
- Eine Fibroblasten-Durchstechflasche und Plattenzellen in der 0,1% gelatinebeschichteten Schale auftauen. Über Nacht bei 37 °C, 5%CO2inkubieren. Bei Bedarf Fibroblasten über einen längeren Zeitraum ausdehnen, bis der gewünschte Durchgang und die Einmündung erreicht sind.
- Beschichten Sie eine 6-Well-Gewebekultur-behandelte Platte mit 500 l 0,1% Gelatinelösung und inkubieren Sie bei 37 °C für 20 min. Entfernen Sie zusätzliche Gelatine.
- Platten-HDFs mit einer Dichte von 150.000 Zellen pro Platte (25.000 Zellen pro Bohrkörper) in 2 ml vollständiger DMEM pro Brunnen. Über Nacht bei 37 °C, 5%CO2inkubieren, um eine Zellanhaftung zu ermöglichen.
- Ersetzen Sie Medium durch 2 ml komplettes DMEM plus 8 g/ml Polybren. Bereiten Sie einen 1:1-Verhältnismix aus poolproduzierten TF-Lentiviren und M2rtTA in einem neuen Mikrozentrifugenrohr vor.
HINWEIS: In diesem Protokoll wird die Pool-Produktion von Lentiviren für die drei TFs durchgeführt, was in den Händen der Autoren zu einer höheren Reprogrammierungseffizienz führt. Alternativ wird vorgeschlagen, eine Titration der einzelnen lentiviralen Partikel durch qPCR18an einer Standardzelllinie durchzuführen. Dies wird verwendet, um das Volumen der einzelnen Viren zu definieren, die notwendig sind, um eine Vielzahl von Infektionen (MOI) zu erfüllen, die für die Kotransduktion und hämogene Reprogrammierung optimal sind. - Verteilen Sie 10 bis 100 L Lentiviral-Mischung pro Brunnen, um HDFs zu transducen. Dies ist Tag -2 der Neuprogrammierung.
HINWEIS: Die Definition des optimalen Volumens des lentiviralen Mixes für eine effiziente Neuprogrammierung, ohne die Zelllebensfähigkeit zu beeinträchtigen, erfordert eine Optimierung (weitere Einzelheiten siehe Ergänzende Abbildung 1). HDFs mit mehr als 7 Durchgängen können höhere Virenmengen erfordern als Zellen mit niedrigeren Durchgängen. - Entfernen Sie nach 16 h Inkubation Viren und fügen Sie vollständiges DMEM hinzu. Lassen Sie Zellen für 6 x 8 h wiederherstellen.
- Nach der Wiederherstellung, Aspirieren Medium und fügen Sie 2 ml komplette DMEM mit 8 g/ml Polybren.
- Führen Sie eine zweite Transduktion, wie in Schritt 4.6 beschrieben, und brüten Sie bei 37 °C, 5%CO2 für 16 h. Dies ist Tag -1 der Neuprogrammierung. Die lentivirale Mischung kann am Tag -2 für beide Transduktionen vorbereitet und bei 4 °C gehalten werden.
- Entfernen Sie am nächsten Tag die Viren und fügen Sie komplettes DMEM hinzu, das mit 1 g/ml DOX ergänzt wird. Dies ist Tag 0 der Neuprogrammierung. Bei 37 °C inkubieren, 5%CO2 für 48 h.
-
Am Tag 2 der Neuprogrammierung, teilen Sie jeden Brunnen bei 1:2 Verhältnis.
- Aspirieren von Medium- und Waschzellen mit 1 ml PBS.
- Aspirieren Sie PBS und dissoziieren Sie Zellen mit 500 L Dissoziationslösung. Inkubieren Sie 5 bis 10 min bei 37 °C, 5%CO2.
- Inaktivieren Sie die Dissoziationslösung mit 1 ml komplettem DMEM und sammeln Sie Zellen in einem konischen Rohr. Zentrifuge bei 350 x g für 5 min.
- Setzen Sie das Pellet in hämatopoetischem Medium (siehe Schritt 1.3), ergänzt durch 1 g/ml DOX, und Plattenzellen in neue gewebekulturbehandelte 6-Well-Platten, die mit 0,1% Gelatine mit einem Endvolumen von 2 ml pro Brunnen beschichtet sind.
- Wechselmedium (hämatopoetisches Medium plus DOX) zweimal pro Woche für die Dauer der Umprogrammierungskulturen (25 Tage).
- Analysieren Sie die resultierenden neu programmierten Zellen zu unterschiedlichen Zeitpunkten durch Hellfeld- oder Fluoreszenzmikroskopie (siehe Ergänzende Abbildung 2), Durchflusszytometrie, Massen- und einzellige RNA-Sequenzierung und Transplantationstests zur Erfassung von hämatopoetische Morphologie, Vorhandensein von endothelialeund hematopoetischen Markern, Erfassung des endotheliaalen/hämatopoetischen Genexpressionsprofils und Regenerationsfähigkeit14.
5. Optimierung der Fibroblastenerweiterung für die ChIP-seq-Analyse beim Beginn der hämogenEn Reprogrammierung
- Platte 300.000 HDFs (CO2inkubieren.
- Ersetzen Sie am nächsten Tag Medium durch vollständiges DMEM, ergänzt durch 8 g/ml Polybren.
- Transduce Zellen mit individuellen Faktoren: pFUW-tetO-FOS14, pLV-tetO-HA-GFI1B (Addgene #125599)14 und pFUW-tetO-3xFLAG-GATA2 (Addgene #125600)14 oder mit einem Pool der drei Faktoren, plus FUW-M2rtTA bei 1:1 Verhältnis. Verwenden Sie 10 x 20 L Gesamtvirus (individuelle TF + M2rtTA oder drei TFs + M2rtTA). Inkubieren Sie Zellen über Nacht bei 37 °C, 5%CO2.
HINWEIS: Es wird empfohlen, zwölf 6-Well-Platten pro Zustand zu verwenden (für jeden einzelnen TF und die drei TFs kombiniert). - Entfernen Sie lentiviren und fügen Sie komplette DMEM 16 h nach der ersten Transduktion hinzu. Lassen Sie Zellen für 6 x 8 h wiederherstellen.
- Transduce Zellen ein zweites Mal mit der gleichen Menge an Virus pro Zustand und inkubieren bei 37 °C, 5%CO2.
- Entfernen Sie am nächsten Tag Viren und fügen Sie vollständiges DMEM hinzu. Bei 37 °C inkubieren, 5% CO2 für 24 h.
- Proplatte jeweils gut in eine 0,1% gelatinebeschichtete, mit Gewebekultur behandelte 100-mm-Schale mit vollständiger DMEM bis zu einem Endvolumen von 10 ml pro Schale. Dies entspricht ungefähr einer 1:6-Passage.
- Lassen Sie Zellen 6 Tage bei 37 °C, 5%CO2wachsen.
- Am 6. Tag nach dem Erneutbeschichten, Aspirieren Medium und fügen Sie komplette DMEM mit 1 g/mL DOX. Inkubieren Sie Zellen bei 37 °C, 5%CO2 für 2 Tage.
- Sammeln Sie Fibroblasten und analysieren Sie genomische Bindungsstellen der drei TFs, die einzeln oder in Kombination durch ChIP-seq 2 Tage nach DOX-Supplementierung14transduziert wurden.
HINWEIS: Die letzten zweiundsiebzig 100 mm Schalen enthalten zwischen 20 x 50 x 106 Zellen, ausreichend, um ChIP-seq-Experimente durchzuführen und zu replizieren.
Ergebnisse
Eine schematische Darstellung des Reprogrammierungsansatzes mit HDFs ist in Abbildung 1Adargestellt. Fibroblasten werden aus kommerziellen Quellen gewonnen oder von menschlichen Spendern gesammelt und vor der Umprogrammierung in vitro erweitert. Nach der Beschichtung werden die Zellen zweimal mit GATA2-, GFI1B- und FOS-Lentiviren (und M2rtTA) transduziert, und Doxycyclin wird am Tag 0 der Neuprogrammierung zugegeben. Am 2. Tag werden die Zellen geteilt und in hämatopoetischem Medium bis zum 25. Tag der Kultur plattiert. Umprogrammierte Zellen können zu verschiedenen Zeitpunkten für mehrere Anwendungen erzeugt werden, einschließlich Transplantation bei immungeschwächten Mäusen, einzellige RNA-Sequenzierung (scRNA-seq) von gereinigten Zellpopulationen (Tag 2 unsortiert, Tag 15 CD49f+ CD34 und Tag 25 CD49f+CD34+ Zellen) sowie Mikroskopie- und Durchflusszytometrieanalyse für die Zelloberflächenmarker CD49f, CD34, CD9 und CD143. Repräsentative Zytometrie-Plots zeigen nach 25 Tagen Neuprogrammierung 17 % der umprogrammierten Zellen, die sowohl CD49f als auch CD9 (Abbildung 1B, linkes Panel)exdrücken. Die Mehrheit der doppelten positiven Zellen drückt CD143 (86 %) aus, und eine kleine Population drückt CD34 (0,9%) aus, was auf eine dynamische hämogene Schicksalsinduktion hindeutet. Diese Marker werden in M2rtTA transduced HDFs, die 25 Tage lang kultiviert wurden, nicht aktiviert(Abbildung 1B, rechtes Panel). Immunfluoreszenzbilder bestätigen die Expression von CD9 und CD143 in anhaftenden und runden Zellen, die sich morphologisch von Fibroblasten unterscheiden, die für diese Marker negativ sind (Abbildung 1C). Menschliche hämogene Kolonien drücken auch CD49f und CD3414aus. Die ScRNA-seq-Analyse von HDFs, unsortierten Zellen des Tages 2 und gereinigten neu programmierten Zellen an Tag 15 (CD49f+CD34- )und Tag 25 (CD49f+CD34+) zeigen eine schrittweise Zunahme der CD49f-, CD9- und CD143-Expression vom 2. tag bis zum 25. Tag. CD49f- und CD9-positive Zellen erscheinen zuerst während des Reprogrammierungsprozesses, zwischen Tag 2 und 15, was darauf hindeutet, dass diese Moleküle Marker der frühen menschlichen Hämogenese darstellen können. Die CD143-Expression beginnt am Tag 15 zu erkennen und CD34-Expressionszellen werden erst zu späteren Zeitpunkten (Tag 25) erkannt. CD34+ Nabelschnurblutzellen (UCB) wurden als Referenz verwendet (Abbildung 1D).
Abbildung 2A beschreibt ein modifiziertes Protokoll zur Generierung einer ausreichenden Anzahl von Zellen für die ChIP-seq-Analyse in den Anfangsstadien der hämogenen Reprogrammierung (Tag 2). Erstens werden HDFs mit einer Dichte plattiert, die um das Zweifache höher ist als im Standardprotokoll (300.000 Zellen gegenüber 150.000 Zellen pro Platte). Nach der Transduktion wird jeder Brunnen in eine 100-mm-Schale umgetaust, so dass sich die Zellen 6 Tage lang ausdehnen können, bevor sie das Medium mit DOX ergänzen. Zellen werden 2 Tage nach dem Hinzufügen von DOX und dem daraus resultierenden TF-Ausdruck analysiert. Abbildung 2B zeigt Genom-Browserprofile von GATA2, die an genomische Regulierungsregionen von ITGA6 und ACE binden, wenn Zellen mit den drei Faktoren (3TFs) oder GATA2 einzeln kodukiert werden. GATA2 bindet auch an offene Chromatinregionen der CD9- und CD34-Gene 14.
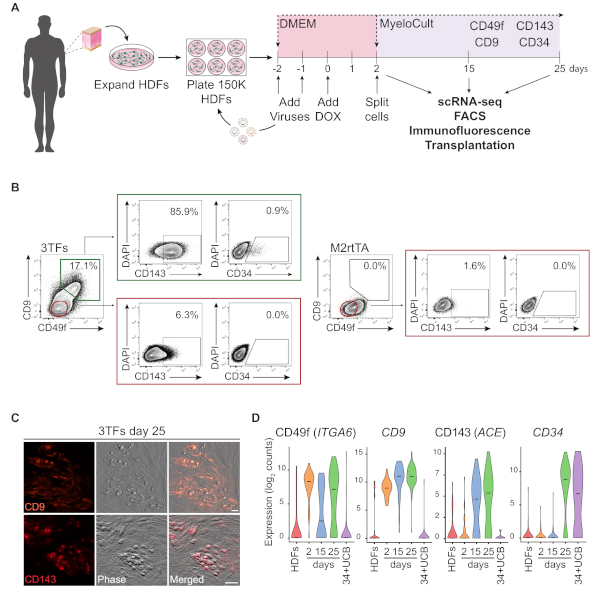
Abbildung 1: Induktion hämogenen Schicksals in menschlichen dermalen Fibroblasten. (A) Experimentelle Strategie zur hämogenen Reprogrammierung menschlicher dermaler Fibroblasten (HDFs). Fibroblasten aus Hautpunschbiopsien werden von Spendern gesammelt, erweitert und mit GATA2-, GFI1B-, FOS- und M2rtTA-Lentiviren transduziert. Doxycyclin (DOX) wird der Kultur an Tag 0 der Umprogrammierung hinzugefügt und Zellen werden zu mehreren Zeitpunkten bis zum 25. Tag analysiert. scRNA-seq, einzellige RNA-Sequenzierung. FACS, Fluoreszenz-aktivierte Zellsortierung. (B) Gating-Strategie zur Bewertung der Expression hämogen/hämatopoetischer Marker durch Durchflusszytometrie an Tag 25 nach Transduktion mit den drei Transkriptionsfaktoren (3TFs). Zytometrie-Plots zeigen einen Prozentsatz doppelt positiver Zellen für CD49f und CD9, die in der Lebendzellenpopulation (DAPI-negativ) eingezäutt sind. Innerhalb der doppelt positiven Population wird der Ausdruck von CD143 und CD34 angezeigt. HDFs, die nur mit dem M2rtTA-Virus unter den gleichen Kulturbedingungen transduziert werden, werden als Kontrolle verwendet. (C) Immunfluoreszenzbilder von Tag 25 umprogrammierten Kolonien, die den Ausdruck von CD9 (Oberpanel) und CD143 (unteres Panel) bestätigen. Die Zellen wurden mit Antikörpern(Materialtabelle) verdünnt 1:100 in PBS/2% FBS mit Mausserum, 20 min bei 37 °C, 5%CO2,dreimal gewaschen und in PBS/2% FBS abgebildet. Phasen-Gradientenkontrast. Skalenstäbe = 50 m. (D) ScRNA-seq-Analyse von 253 Zellen zu verschiedenen Zeitpunkten. Expression von ITGA6, CD9, ACE und CD34 wird während der Neuprogrammierung aktiviert. Die Zellen werden an Tag 2 (unsortiert), Tag 15 (CD49f+CD34- )und Tag 25 (CD49f+CD34+) gesammelt. HDFs und CD34+ Nabelschnurblut (34+UCB) Zellen werden als Referenzen verwendet. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
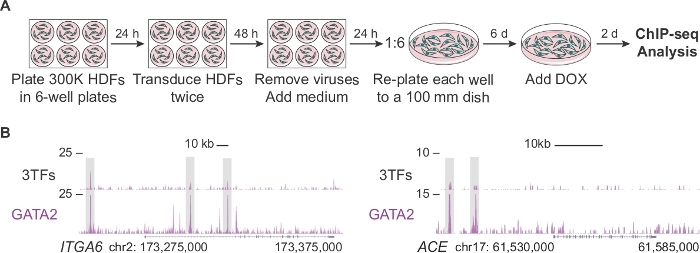
Abbildung 2: Erweiterung der menschlichen dermalen Fibroblasten für die ChIP-seq-Analyse. (A) Experimentelle Strategie, die ein modifiziertes Protokoll darstellt, um eine hohe Anzahl von transduzierten menschlichen Dermale-Fibroblasten (HDFs) für ChIP-seq am 2. Tag der Neuprogrammierung zu erzeugen. 300.000 Zellen werden in 6-Well-Platten plattiert und zweimal mit Einzelfaktoren (pFUW-tetO-FOS, pLV-tetO-HA-GFI1B oder pFUW-tetO-3xFLAG-GATA2) oder einer Kombination der drei Faktoren (plus M2rtTA) transduziert. Nach dem Entfernen von Viren werden Fibroblasten für sechs Tage in 100 mm Geschirr erweitert. Doxycyclin (DOX) wird an Tag 0 zugegeben und Zellen werden zwei Tage nach DOX-Zugabe gesammelt. (B) Genom-Browserprofile, die GATA2-bindungsstellen (graue Felder) bei ITGA6 und ACE loci zwei Tage nach der Transduktion mit den drei Transkriptionsfaktoren (3TFs) oder nur mit GATA2 hervorheben. Die Gesamtanzahl der zugeordneten Lesevorgänge wird auf der y-Achse dargestellt. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
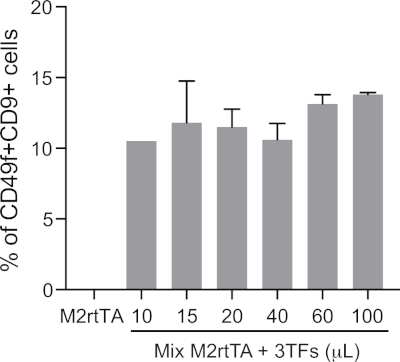
Ergänzende Abbildung 1: Definition eines optimierten lentiviralen Volumens für eine effiziente hämogene Reprogrammierung. Zunehmende Mengen konzentrierter (10 bis 100 l) im Pool produzierter lentiviraler Partikel (3TFs: GATA2, GFI1B und FOS) werden verwendet, um menschliche dermale Fibroblasten (HDFs) zusammen mit M2rtTA im Verhältnis 1:1 zu transdutieren, wobei die Schritte 4.5-4.12 des Protokolls folgen. Umprogrammierte Zellen werden an Tag 25 analysiert, um ein optimales Volumen der Transduktion für hämogene Reprogrammierung zu definieren, angegeben durch den Prozentsatz von CD49f+CD9+ Zellen, die in Lebendenzellen abgeziert sind (DAPI-negativ). Die Zelllebensfähigkeit kann durch Quantifizierung der absoluten Anzahl lebender Zellen an Tag 25 beurteilt werden. ALS Negativkontrolle werden HDFs verwendet, die mit M2rtTA (100 l) transduziert wurden. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Ergänzende Abbildung 2: Morphologieveränderungen während der hämogenen Reprogrammierung menschlicher dermaler Fibroblasten. Humane Dermal-Fibroblasten -Kulturen (HDF) werden am Tag der ersten Transduktion (Tag -2), wenn DOX den Kulturen (Tag 0), zwei Tagen (Tag 2) und fünfzehn Tagen (Tag 15) nach der DOX-Supplementierung und am Ende des Experiments (Tag 25) zugesetzt wird, abgebildet. Hämogene Kolonien an den Tagen 15 und 25 werden hervorgehoben. Skalenbalken = 100 m. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Diskussion
In diesem Artikel wird eine Methode beschrieben, um hämatopoetische Vorläuferzellen direkt aus menschlichen Fibroblasten zu erzeugen, die durch ein HP-Zell-Zwischenprodukt gehen, ähnlich wie definitive HSCs14.
Die Poolproduktion von lentiviralen Partikeln, die GATA2, GFI1B und FOS kodieren, wurde der individuellen Produktion vorgezogen, da sie in unseren Händen zu höheren Reprogrammierungseffizienzen (unveröffentlichte Daten) führt. Lentiviren enthalten als Mitglieder der Retroviridae-Familie normalerweise zwei Kopien positiver einsträngiger RNA19. Die erhöhte Reprogrammierungseffizienz kann auf die Verpackung von zwei verschiedenen Transgenen in demselben lentiviralen Teilchen zurückzuführen sein, was zu einer erhöhten Anzahl von Zellen führt, die mit den drei Transkriptionsfaktoren kodukiert werden. Um den Erfolg dieses Protokolls zu gewährleisten, ist es notwendig, HDFs mit einer ausreichenden Virusmenge abhängig von der Zellpassage zu transduzieren, um ein optimales Gleichgewicht zwischen Derprogrammierungseffizienz und Zelllebensfähigkeit zu erreichen, wie in Schritt 4.6 empfohlen. Darüber hinaus können frische nicht konzentrierte Viren verwendet werden. Es wird empfohlen, Zellen mit 0,5-3 ml 3TFs Pool und M2rtTA zu transduzieren. Außerdem sollte die Zelldichte entsprechend der Anwendung angepasst werden. 150 000 HDFs pro 6-Well-Platte (Schritt 4.4) lieferten die optimale Dichte für die FACS-, Transplantations- und Durchflusszytometrie-Analyse von umprogrammierten Zellen. Für ChIP-seq-Experimente waren von Anfang an mehr Zellen erforderlich (Schritt 5.1). Es ist wichtig, Zellen regelmäßig auf morphologische Veränderungen zu überprüfen und zweimal pro Woche das hämatopoetische Medium zu ersetzen, um die Entstehung induzierter hämatopoetischer Zellen zu unterstützen. Die Zugabe von hämatopoetischen Zytokinen oder Co-Kultur in Feederschichten kann die Effizienz der Neuprogrammierung erhöhen.
Mit dieser Methode können wir neue hämatopoetische Marker identifizieren, die bei der hämogenen Reprogrammierung dynamisch exprimiert werden. CD9, das sich in neu programmierten Zellen auf der Transkriptionsebene14als up-reguliert zeigte, wird in den Anfangsphasen der Neuprogrammierung zusammen mit CD49f und CD143 schnell an der Zelloberfläche exprimiert und dient als neuartiger Marker menschlicher HSC-Vorläufer. Wir zeigen auch, dass ITGA6 und ACE direkte Ziele von GATA2 in den Anfangsphasen der hämogenEn Reprogrammierung sind, zusätzlich zu CD9 und CD3414, die eine direkte mechanistische Verbindung zwischen humanen hämogen Vorläufer-Phänotyp und GATA2.
Ein Vorteil dieses Systems liegt in der Verwendung relativ homogener Fibroblastenkulturen. Während PSCs leicht erweitert und in vitrogepflegt werden können, erzeugen Differenzierungsprotokolle heterogene Populationen, die hämatopoetische Vorläufer enthalten, die schlecht5,6,7engrafieren. Darüber hinaus besteht bei der Transplantation von PSC-abgeleiteten HSPCs die Gefahr einer Tumorgenese, da undifferenzierte PSCs auch nach Verwendung von Differenzierungsprotokollen in der Kultur bleiben können. Alternativ zu Fibroblasten wurde die direkte Umprogrammierung auf HSCs auf blutverströmte Vorläufer20 und Endothelzellen21angewendet. Jedoch, beginnend mit blutbeschränkten Vorläuferzellen behindert die therapeutische Anwendung der resultierenden HSCs, wenn der Patient Mutationen trägt, die den Stamm/ Vorläufer hämatopoetische Population beeinflussen22. Bei Endothelzellen sind diese im Vergleich zu Fibroblasten schwieriger zu erhalten und stellen eine sehr heterogene Zellpopulation in Bezug auf Phänotyp, Funktion und Struktur dar, die organabhängig sind23. Andere Studien haben es geschafft, Maus-Fibroblasten in engrafierbare hämatopoetische Vorläufer24,25 umzuprogrammieren, aber bisher beschreibt kein anderes Protokoll die Erzeugung von HSPC-ähnlichen Zellen aus menschlichen Fibroblasten.
Dieser Ansatz, gekoppelt mit pharmakologischen Hemmungen, Gen-Knock-out oder Knock-down ermöglicht es, individuelle oder Kombination von Faktoren zu definieren, die erforderlich sind, um menschliche HSCs direkt induzieren. CRISPR-Cas9-Technologien in HDFs vor der Neuprogrammierung stellen ein spannendes Unterfangen dar, um neue Regulatoren der menschlichen endgültigen Hämatopoese zu definieren. In Zukunft wird die Neuprogrammierung nicht blutbezogener menschlicher Zelltypen wie Fibroblasten als Plattform dienen, um gesunde, auf Patienten zugeschnittene hämatopoetische Vorläuferzellen für klinische Anwendungen zu generieren.
Offenlegungen
Die Autoren haben nichts zu verraten.
Danksagungen
Die Knut und Alice Wallenberg Stiftung, die Medizinische Fakultät der Universität Lund und der Region Skéne sind für großzügige finanzielle Unterstützung ausgezeichnet. Diese Arbeit wurde durch ein Stipendium von Olle Engkvists Stiftelse (194-0694 an Filipe Pereira) und Doktorandenstipendien von Fundaéo para a Ciéncia e Tecnologia (PTDC/BIM-MED/0075/2014 an Filipe Pereira, und SFRH/BD/135725/2018 und SFRH/BD/51968/2012 an Rita Alves und Andreia Gomes). Diese Studie wurde auch durch Mittel von NIH und NYSTEM (1R01HL119404 und C32597GG an Kateri A. Moore) unterstützt.
Materialien
| Name | Company | Catalog Number | Comments |
| 0.45 μm low-protein binding filter, 150 mL Bottle Top Vacuum Filter | Corning | #430625 | |
| 2-Mercaptoethanol | Sigma-Aldrich | #M6250 | |
| Alexa Fluor 488 anti-human CD34 clone 581 | BioLegend | #343518 | |
| BD Pharmingen APC Mouse Anti-Human Angiotensin Converting Enzyme (CD143) clone BB9 | BD Biosciences | #557929 | |
| BES buffered saline | Sigma-Aldrich | #14280 | |
| Calcium chloride (CaCl2) | Sigma-Aldrich | #449709 | |
| Centrifugal filter unit, Amicon Ultra-15 Centrifugal Filter Unit | Sigma-Aldrich | #UFC903096 | |
| Dissociation solution, TrypLE Express Enzyme (1x) no phenol red | Gibco | #12604-021 | |
| Doxycycline hyclate (DOX) | Sigma-Aldrich | #D9891 | |
| eBioscience CD49f (Integrin alpha 6) Monoclonal Antibody (eBioGoH3 (GoH3)), PE-Cyanine7 | Invitrogen | #25-0495-82 | |
| FUW-M2rtTA | Addgene | #20342 | |
| Gelatin from Porcine Skin Type A | Sigma-Aldrich | #G1890 | |
| Gibco L-Glutamine (200 mM) | ThermoFisher Scientific | #25030-024 | |
| Gibco MEM Non-Essential Amino Acids Solution (100x) | ThermoFisher Scientific | #11140-035 | |
| Hematopoietic medium, MyeloCult H5100 | STEMCELL Technologies | #05150 | |
| Hexadimethrine bromide (polybrene) | Sigma-Aldrich | #H9268 | |
| Human Dermal Fibroblasts (HDFs) | ScienCell | #2320 | |
| HyClone Dulbecco's Modified Eagles Medium (DMEM) | GE Healthcare | #SH30243.01 | |
| HyClone Fetal Bovine Serum (FBS) | GE Healthcare | #SV30160.03 | |
| HyClone Penicillin Streptomycin 100x Solution (Pen/Strep) | GE Healthcare | #SV30010 | |
| HyClone Phosphate Buffered Saline solution (PBS) | GE Healthcare | #SH30256.01 | |
| Hydrocortisone | STEMCELL Technologies | #7904 | |
| Mouse serum | Sigma-Aldrich | #M5905 | |
| PE anti-human CD9 Antibody clone HI9a | BioLegend | #312105 | |
| pFUW-tetO-3xFLAG-GATA2 | Addgene | #125600 | |
| pFUW-tetO-FOS | Addgene | #125598 | |
| pFUW-tetO-GATA2 | Addgene | #125028 | |
| pFUW-tetO-GFI1B | Addgene | #125597 | |
| pLV-tetO-HA-GFI1B | Addgene | #125599 | |
| pMD2.G | Addgene | #12259 | |
| psPAX2 | Addgene | #12260 |
Referenzen
- Ivanovs, A., et al. Highly potent human hematopoietic stem cells first emerge in the intraembryonic aorta-gonad-mesonephros region. Journal of Experimental Medicine. 208, 2417-2427 (2011).
- Tavian, M., Biasch, K., Sinka, L., Vallet, J., Péault, B. Embryonic origin of human hematopoiesis. International Journal of Developmental Biology. 1065, 1061-1065 (2010).
- Medvinsky, A., Rybtsov, S., Taoudi, S. Embryonic origin of the adult hematopoietic system: advances and questions. Development. 138, 1017-1031 (2011).
- Ivanovs, A., et al. Human haematopoietic stem cell development: from the embryo to the dish. Development. 144, 2323-2337 (2017).
- Daniel, M. G., Pereira, C. F., Lemischka, I. R., Moore, K. A. Making a Hematopoietic Stem Cell. Trends in Cell Biology. 26, 202-214 (2016).
- Vo, L., Daley, G. De novo generation of HSCs from somatic and pluripotent stem cell sources. Blood. 125, 2641-2648 (2015).
- Rafii, S., et al. Human ESC-derived hemogenic endothelial cells undergo distinct waves of endothelial to hematopoietic transition. Blood. 121, 770-781 (2013).
- Ebina, W., Rossi, D. J. Transcription factor-mediated reprogramming toward hematopoietic stem cells. EMBO Journal. 34, 694-709 (2015).
- Sugimura, R., et al. Haematopoietic stem and progenitor cells from human pluripotent stem cells. Nature. 545, 432-438 (2017).
- Pereira, C. F., et al. Induction of a Hemogenic Program in Mouse Fibroblasts. Cell Stem Cell. 13, 205-218 (2013).
- Pereira, C. F., et al. Hematopoietic Reprogramming In vitro Informs In Vivo Identification of Hemogenic Precursors to Definitive Hematopoietic Stem Cells. Developmental Cell. 36, 525-539 (2016).
- Notta, F., et al. Isolation of Single Human Hematopoietic Stem Cells Capable of Long-Term Multilineage Engraftment. Science. 333, 218-221 (2011).
- Sinka, L., Biasch, K., Khazaal, I., Péault, B., Tavian, M. Angiotensin-converting enzyme (CD143) specifies emerging lympho-hematopoietic progenitors in the human embryo. Blood. 119, 3712-3724 (2012).
- Gomes, A. M., et al. Cooperative Transcription Factor Induction Mediates Hemogenic Reprogramming. Cell Reports. 25, 2821-2835 (2018).
- Karlsson, G., et al. Report The Tetraspanin CD9 Affords High-Purity Capture of All Murine Hematopoietic Stem Cells. Cell Reports. 4, 642-648 (2013).
- Leung, K. T., et al. The tetraspanin CD9 regulates migration, adhesion, and homing of human cord blood CD34+ hematopoietic stem and progenitor cells. Blood. 117, 1840-1851 (2011).
- Hockemeyer, D., et al. A drug-inducible system for direct reprogramming of human somatic cells to pluripotency. Cell Stem Cell. 3, 346-353 (2008).
- Kutner, R. H., Zhang, X., Reiser, J. Production concentration and titration of pseudotyped HIV-1-based lentiviral vectors. Nature Protocols. 4, 495-505 (2009).
- Suzuki, Y. S., Suzuki, Y., Ke, X. Gene Regulatable Lentiviral Vector System. Viral Gene Therapy. , (2011).
- Riddell, J., et al. Reprogramming committed murine blood cells to induced hematopoietic stem cells with defined factors. Cell. 157, 549-564 (2014).
- Lis, R., et al. Conversion of adult endothelium to immunocompetent haematopoietic stem cells. Nature. 545, 439-445 (2017).
- Pereira, C., Lemischka, I. R., Moore, K. From blood to blood’: de-differentiation of hematopoietic progenitors to stem cells. EMBO Journal. 33, 1511-1513 (2014).
- Nolan, D. J., et al. Molecular Signatures of Tissue-Specific Microvascular Endothelial Cell Heterogeneity in Organ Maintenance and Regeneration. Developmental Cell. 26, 204-219 (2013).
- Batta, K., Florkowska, M., Kouskoff, V., Lacaud, G. Direct Reprogramming of Murine Fibroblasts to Hematopoietic Progenitor Cells. Cell Reports. 9, 1871 (2014).
- Cheng, H., et al. Reprogramming mouse fibroblasts into engraftable myeloerythroid and lymphoid progenitors. Nature Communications. 7, 1-15 (2016).
Erratum
Formal Correction: Erratum: Hemogenic Reprogramming of Human Fibroblasts by Enforced Expression of Transcription Factors
Posted by JoVE Editors on 12/03/2019. Citeable Link.
An erratum was issued for: Hemogenic Reprogramming of Human Fibroblasts by Enforced Expression of Transcription Factors. The author affiliations were updated.
The second author affiliation was updated from:
2Wallenberg Center for Molecular, Lund University
to:
2Wallenberg Center for Molecular Medicine, Lund University
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten