Method Article
Reprogrammation hémogénique des fibroblastes humains par expression forcée des facteurs de transcription
Dans cet article
Erratum Notice
Résumé
Ce protocole démontre l'induction d'un programme hémogène dans les fibroblastes dermiques humains par l'expression forcée des facteurs de transcription GATA2, GFI1B et FOS pour produire des cellules hématopoïétiques de tige et d'ancêtre.
Résumé
Les mécanismes cellulaires et moléculaires sous-jacents à la spécification des cellules souches hématopoïétiques humaines (HSC) restent insaisissables. Des stratégies pour récapituler l'émergence humaine de HSC in vitro sont nécessaires pour surmonter des limitations en étudiant ce processus développemental complexe. Ici, nous décrivons un protocole pour produire la tige hématopoïétique et les cellules progénitrice-comme des fibroblastes dermiques humains employant une approche directe de reprogrammation de cellules. Ces cellules transitent par un type de cellule intermédiaire hémogène, ressemblant à la transition endothéliale-hématopoietic (EHT) caractéristique de la spécification de HSC. Les fibroblastes ont été reprogrammés en cellules hémogéniques par transduction avec des facteurs de transcription GATA2, GFI1B et FOS. Cette combinaison de trois facteurs a induit des changements morphologiques, l'expression des marqueurs hémogènes et hématopoïétiques et des programmes transcriptionnels dynamiques d'EHT. Les cellules reprogrammées génèrent la progéniture hématopoïétique et repeuplent les souris immunodéficientes pendant trois mois. Ce protocole peut être adapté à la dissection mécaniste du processus d'EHT humain, comme l'illustre ici la définition des cibles GATA2 au cours des premières phases de la reprogrammation. Ainsi, la reprogrammation hémogène humaine fournit une approche simple et traitable pour identifier de nouveaux marqueurs et régulateurs de l'émergence humaine de HSC. À l'avenir, l'induction fidèle du destin hémogène dans les fibroblastes peut mener à la génération des HSC patient-spécifiques pour la transplantation.
Introduction
Des cellules hématopoïétiques définitives de tige et d'ancêtre (HSPc) émergent dans la région aorte-gonad-mesonephros (AGM) et le placenta des précurseurs endothéliaux avec la capacité hémogène, par une transition endothéliale-à-hématopoietic (EHT)1, , 2. Les précurseurs hémogènes (PS) expriment des marqueurs endothéliales et hématopoïétiques, mais leur identification précise reste insaisissable, en particulier dans le système humain. Bien qu'il s'en soit un processus relativement conservé chez les mammifères, le développement des cellules souches hématopoïétiques (HSC) diffère encore considérablement entre les modèles humains et muraux3,4. Par conséquent, des approches in vitro pour récapituler le développement humain de HSC sont nécessaires.
La différenciation des cellules souches pluripotentes (CSP) aux HSC, bien que prometteuse, a rencontré un succès limité au cours des 20 dernières années, principalement en raison des protocoles de différenciation disponibles, qui ont comme conséquence les progéniteurs hématopoïétiques primitifs avec l'engraftment pauvre capacité5,6,7. Alternativement, des méthodologies directes de reprogrammation de cellules ont été appliquées pour produire des cellules HSPC-comme de plusieurs types de cellules, utilisant des facteurs de transcription (TFs)8,9. En particulier, la surexpression de trois TF, Gata2, Gfi1b et cFos, a converti les fibroblastes embryonnaires de souris en HSPC par le biais d'un intermédiaire HP avec un phénotype défini (Prom1-Sca-1-CD34-CD45-)10. Ce processus a ressemblé à l'EHT qui se produit dans l'embryon et le placenta, pendant la spécification de l'hématopoiesis définitif. Ce phénotype a permis l'identification et l'isolement d'une population de HPs dans le placenta de souris qui après la culture à court terme et l'activation de Notch ont généré des HSC transplantables en série11.
Jusqu'à présent, aucun phénotype n'a été établi qui distingue les HSC humains de leurs précurseurs, mais certaines molécules sont connues pour être exprimées dans les HSC émergents. Integrin alpha 6 (ITGA6 ou CD49f) est fortement exprimé dans les HSC repeuplement à long terme, les plus immatures les cellules dans le compartimentHSC 12,et l'enzyme angiotensin-conversion (ACE ou CD143) est présente dans les précurseurs hématopoïétiques négatifs cD34 dans les tissus sanguins embryonnaires13.
Récemment, nous avons démontré que les versions humaines des trois TF, GATA2, FOS et GFI1B reprogramment les fibroblastes dermiques humains (HDF) en HPs avec une capacité d'engraftment à court terme14. Dans les phases initiales de la reprogrammation, GATA2 engage la chromatine ouverte et recrute GFI1B et FOS pour réprimer les gènes du fibroblaste et activer les gènes endothélials et hématopoïétiques. Les cellules induites ont fortement exprimé CD49f et ACE, et ont contenu un petit pourcentage de cellules exprimant le marqueur CD34 de HSPC. Le gène CD9, qui est exprimé dans les HSC15 et est important pour HSC homing16, s'est avéré être une cible directe de GATA2 et parmi les gènes les plus régulés dans les cellules reprogrammées14. Le CD9 peut donc constituer un marqueur supplémentaire pour les PS de l'hématopoiesis définitif humain.
Dans ce protocole, nous décrivons la génération de cellules de type HSPC à partir de fibroblastes humains par l'expression forcée de GATA2, GFI1B et FOS, ainsi qu'une méthode adaptée pour l'immunoprécipitation de chromatine (ChIP)-séquençage (seq) au début de Reprogrammation. Les TF ont été codés dans un vecteur lentiviral inductible de doxycycline (DOX) (pFUW-tetO) qui contient un élément de réponse de tétracycline (TRE) et un promoteur minimal de CMV, et ont été transducés avec un vecteur constitutif contenant la tétracycline inverse protéine transactivatrice (pFUW-M2rtTA). Lorsque DOX (analogue de la tétracycline) est ajouté après la transduction, il se lie à la protéine rtTA qui interagit avec la transcription TF TRE permettant (Tet-On système). La procédure nécessite 25 jours pour terminer. Pour les expériences ChIP-seq, les HDF ont été transdus avec des versions étiquetées de GATA2 (pFUW-tetO-3xFLAG-GATA2) et GFI1B (pLV-tetO-HA-GFI1B), ainsi que des sites de liaison pFUW-tetO-FOS et TF ont été analysés deux jours après la supplémentation DOX.
En fin de compte, la reprogrammation hémogénique des fibroblastes humains fournit un système in vitro tractable pour étudier les mécanismes sous-jacents à l'hématopoiesis développemental humain et une source potentielle de HSPC spécifiques aux patients pour une application clinique future.
Protocole
Ce protocole a été exécuté conformément aux lignes directrices du comité d'éthique de la recherche humaine de l'Université de Lund et devrait être fait conformément aux lignes directrices institutionnelles individuelles.
1. Préparation de réactif
- Pour le milieu d'aigle modifié (DMEM) modifié de Dulbecco (DMEM)/20% sérum bovin foetal (FBS), mélangez le DMEM à haute teneur en glucose contenant du pyruvate de sodium avec 20% de FBS, 1% pénicilline-streptomycine (pen/strep), 1% L-glutamine, 1% d'acides aminés non essentiels et 10-4-M 2- Mercaptoéthanol.
- Pour le DMEM complet, mélangez le DMEM à haute teneur en glucose contenant du pyruvate de sodium avec 10 % de FBS, 1 % de stylo/streptocoque et 1 % de L-glutamine.
- Pour le milieu hématopoïétique, mélanger le milieu hématopoïétique(tableau des matériaux) avec 10-6 M d'hydrocortisone et 1% de stylo/streptocoque.
- Utilisez de la saline tamponnée en phosphate (PBS) sans calcium ni magnésium.
2. Isolement de fibroblaste dermique humain
REMARQUE: Les HDF peuvent être achetés auprès de fournisseurs certifiés(Tableau des matériaux). Dans ce cas, dissiez les fibroblastes et utilisez-les directement dans les expériences de reprogrammation (section 4). Alternativement, les HDF peuvent être isolés des donateurs. Si les fibroblastes sont isolés de différents donneurs, gardez les échantillons séparés les uns des autres à toutes les étapes du protocole. Étiquette des plaques/puits et des tubes de collecte avec le numéro d'identification de chaque donneur.
- Obtenir des HDF à partir de biopsies rondes de poinçon de peau de 3 mm effectuées par des médecins qualifiés.
- Enrober trois puits d'une plaque de 6 puits traitée par la culture des tissus avec 500 l de gélatine de 0,1 % et incuber pendant 20 min à 37 oC.
- Aspirez la solution de gélatine restante et ajoutez 750 L de DMEM/20% FBS à chaque puits. Toute la surface du puits doit être recouverte de milieu.
- Ajouter 1,5 ml de DMEM/20% FBS à la surface intérieure d'un couvercle stérile de 100 mm de pétri et étaler la goutte à l'aide d'une pipette sérologique de 5 ml.
- Placer la biopsie de la peau dans le milieu sur le couvercle avec des forceps stérilisés.
- Disséquer la biopsie de la peau en neuf sections identiques, en utilisant un scalpel stérilisé pour maintenir la biopsie en place et un deuxième scalpel à couper.
- Placer trois morceaux de biopsie par puits à l'aide de forceps pointus. Assurez-vous que les pièces se fixent au fond du puits.
- Déposer un couvercle de 22 mm sur le dessus des morceaux et appliquer un peu de pression.
- Incuber la plaque à 37 oC, 5 % de CO2,pendant une semaine. Vérifier les cellules tous les jours et ajouter 200 L de DMEM/20% FBS tous les 2 jours pour remplacer le milieu évaporé.
- Après une semaine, ajouter jusqu'à 2 ml de DMEM/20% FBS et remplacer le milieu tous les 2/3 jours.
-
Cellules de passage à un rapport de 1:4 lorsque les puits sont confluents (environ 4 à 8 semaines).
- Préparer 0,1 % de gélatine enduite de plaques de 6 puits traitées par la culture des tissus.
- Aspirer le milieu des puits à 80% de consistance et laver une fois avec 1 ml de PBS.
- Enlever la feuille de couverture avec des forceps stériles et placer la couverture dans un nouveau puits d'une plaque de 6 puits, avec le côté tissulaire vers le haut.
REMARQUE: Les cellules qui sont restées attachées à la couverture seront également récoltées. - Ajouter 500 l de solution de dissociation (Tableau des matériaux) par puits (y compris les puits avec les couvercles) et incuber à 37 oC, 5 % de CO2 pendant 5 à 10 min. Vérifier quand les cellules commencent à s'élever du fond du puits et à inactiver la solution de dissociation en l'ajout de 500 L de DMEM/20% FBS à chaque puits.
- Recueillir les fibroblastes de tous les puits dans un tube conique de 15 ml. Ajouter un milieu supplémentaire aux puits pour recueillir les cellules restantes. Centrifuger le tube à 350 x g pendant 5 min.
- Entre-temps, ajouter 500 L de DMEM/20% FBS à chaque puits de plaques enduites de gélatine.
- Aspirer le milieu et resuspendre des fibroblastes dans 6 ml de DMEM/20% FBS.
- Ajouter 500 ll de suspension de fibroblaste à chaque puits (deux plaques de 6 puits par échantillon/donneur au total). Incuber les cellules pendant la nuit à 37 oC, 5 % de CO2.
- Le lendemain, ajouter 1 mL de DMEM/20% FBS à chaque puits. Remplacer le milieu par 2 ml de DMEM/20% DE FBS tous les 2/3 jours jusqu'à ce que les puits soient confluents à 80%.
- Répétez la section 2.11 pour trois puits de confluents jusqu'à ce que le troisième passage soit atteint.
-
Congeler les fibroblastes des puits de confluents (passages 1 et 3).
- Aspirer le milieu des puits et laver une fois avec 1 ml de PBS.
- Dissocier et recueillir des fibroblastes tels que décrits dans les étapes 2.11.4 et 2.11.5.
- Compter les cellules avec un hémocytomètre et centrifuger le tube à 350 x g pendant 5 min.
- Après centrifugation, aspirez le milieu et resuspendez des fibroblastes dans FBS avec 10% de DMSO à une densité de 5 x 105 cellules/mL.
- Ajouter 1 ml de suspension cellulaire par cryovial et congeler les cellules pendant la nuit à -80 oC à l'aide d'un contenant de congélation. Déplacer les flacons à -150 oC (azote liquide) pour le stockage à long terme.
3. Production lentivirale
- Cultivez des cellules HEK293T dans un plat traité par culture tissulaire de 100 mm avec 10 ml de DMEM complet, à 37 oC, 5 % de CO2,jusqu'à ce que la confluence soit atteinte.
- Le jour précédant la transfection, aspirez à mi-mesure et lavez soigneusement le plat avec 5 ml de PBS.
- Après avoir enlevé le PBS, ajouter 1,5 mL de solution de dissociation et incuber à 37 oC, 5 % de CO2 pendant 5 à 10 min pour dissocier les cellules du plat.
REMARQUE: Il est recommandé de réchauffer à la fois le PBS et la solution de dissociation avant de l'utiliser, afin que les cellules ne subissent pas de choc thermique. - Inactiver la solution de dissociation avec 3 mL de DMEM complet et transférer la suspension cellulaire à un tube conique de 15 ml. Laver le plat avec 5 ml de DMEM complet pour enlever les cellules attachées restantes et transférer ce volume dans le tube conique de 15 ml.
- Suspension de cellule centrifugeuse à 350 x g pendant 5 min.
- Aspirez le supernatant et divisez uniformément le granule cellulaire entre six plats traités par culture tissulaire de 100 mm dans un volume final de 10 ml de DMEM complet par plat. Les cellules doivent être environ 60% confluents au moment de la transfection.
- Le lendemain, les cellules transfect avec le plasmide se mélangecommesuit :
REMARQUE : Cette partie du protocole décrit la production des lentivirus dans un plat de culture de tissu de 100 mm par mélange de plasmide. Pour obtenir des volumes plus élevés de supernatant lentiviral pour la concentration, utilisez au moins quatre plats de culture cellulaire HEK293T de 100 mm par mélange.- Dans un tube conique de 15 ml, ajouter 10 g des trois plasmides de transfert ensemble : 3,33 g de pFUW-tetO-GATA2 (Addgene plasmid #125028)14, 3,33 g de pFUW-tetO-GFI1B (Addgene #125597)14 et 3,33 g de pFUW-tetO-FOS (Addgene #125598)14plus 10 g du vecteur d'emballage psPAX2 de 2nd de génération codant les gènes Gag, Pol, Tat et Rev (Addgene #12260) et 5 g de vecteur d'enveloppe pMD2.G codant le gène VSV-G (Addgene #12259). Ajouter de l'eau jusqu'à 500 l.
- Dans deux nouveaux tubes coniques de 15 ml, ajoutez 10 g de plasmide FUW-M2rtTA (Addgene #20342)17, 10 g de vecteur d'emballage psPAX2 et 5 g de vecteur d'enveloppe de pMD2.G à chaque tube. Ajouter de l'eau jusqu'à 500 l. Un tube va être utilisé comme un contrôle.
- À chaque tube ajouter 62,5 'L de 2 M CaCl2. Ensuite, relâchez des bulles dans chaque mélange à l'aide d'une pipet Pasteur insérée dans un contrôleur de pipet. Pendant que des bulles se forment, pipette 500 'L de N,N-bis(2-hydroxyethyl)-2-aminoethanesulfonic acide (BES) tamponné saline (pH 7,1, 25 oC), avec une pipette P1000, goutte-sage contre la pipet Pasteur et sur le mélange.
- Incuber les tubes à température ambiante pendant au moins 15 min. Les mélanges apparaîtront légèrement nuageux après un certain temps.
- Pendant ce temps, aspirer à partir de la vaisselle cellulaire HEK293T (passage la veille) et ajouter 10 ml de DMEM complet sans antibiotiques. Soyez prudent et ajoutez moyen lentement car les cellules HEK293T sont semi-adhérentes.
- Distribuer chaque mélange individuel (environ 1 ml) uniformément et déposer dans des plats séparés et incuber toute la nuit à 37 oC, 5 % CO2.
- Remplacer le milieu par 4 ml de DMEM complet, 24 h après l'incubation. Incuber toute la nuit à 37 oC, 5 % de CO2. Si disponible, incuber à la place à 32 oC, 5% CO2, car la température réduite augmentera la demi-vie des particules lentivirales.
- Recueillir le supernatant avec des particules lentivirales trois fois à un tube conique de 50 ml. Ne mélangez pas différentes particules lentivirales à ce stade. Chaque plat se traduira par 12 ml de supernatant lentiviral. Quatre plats de la même préparation virale s'insèrent dans un tube conique de 50 ml.
CAUTION : Effectuez la collecte de lentilles dans un laboratoire de niveau 2 de biosécurité dans une hotte à débit laminaire dédiée au travail lentiviral et placez les déchets contaminés viraux (tubes, pointes, vaisselle) dans un contenant approprié pour les matières biodangereuses.- Faites la première collecte 16 h après la dernière incubation et ajoutez 4 ml de DMEM complet. Incuber à 37 oC, 5 % DE CO2.
- Faire la deuxième collecte 8 h après la première au même tube, ajouter 4 ml de DMEM complet et incuber à 37 oC, 5% CO2.
- Faire la troisième collection 16 h après la seconde au même tube et jeter la vaisselle.
REMARQUE: Conserver les supernatants lentiviraux à 4 oC après chaque collecte.
- Filtrer chaque supernatant lentiviral à l'aide d'un filtre de liaison à faible teneur en protéines de 0,45 m avec une membrane d'acétate de cellulose (Tableau des matériaux) à un tube propre.
- Ajouter un maximum de 15 ml de supernatant filtré à une unité de filtre centrifuge avec une membrane de cellulose régénérée(tableau des matériaux)et tourner à 4 000 x g pendant 25 min, à 4 oC. Jeter le flux à travers. Un liquide visqueux contenant des lentivirus restera dans l'unité de filtration.
- Répétez l'étape 3.13 en ajoutant 15 ml de supernatant sur le dessus de l'unité de filtre, jusqu'à ce qu'il n'y ait plus de supernatant lentiviral à gauche.
REMARQUE: Quand il n'y a que quelques millilitres de supernatant pour se concentrer, diminuer le temps de rotation à 10 min. S'il y a encore du liquide supplémentaire (non visqueux) sur le filtre, centrifugeuser pendant 10 min supplémentaires. - Faire des aliquots (50 à 200 l selon le volume initial de supernatant) de chaque type de lentivirus concentrés et stocker à -80 oC pour le stockage à long terme (1 à 2 ans) ou à 4 oC pour le stockage à court terme (1 à 2 semaines).
REMARQUE: Les lentivirus concentrés ou non concentrés peuvent également être utilisés frais. Ne pas recongeler et décongeler car cela entraîne une réduction de la titer.
4. Reprogrammation hémogène
REMARQUE: Utilisez hDFs avec un nombre de passage de trois (P3) ou plus (jusqu'à P10) pour effectuer des expériences de reprogrammation.
- Enrober un plat traité par culture tissulaire de 100 mm de 5 ml de gélatine de 0,1 % et incuber à 37 oC pendant 20 min. Aspirez la solution de gélatine restante.
- Décongeler une fiole de fibroblaste et des cellules de plaque dans le plat enrobé de gélatine de 0,1 %. Incuber toute la nuit à 37 oC, 5 % de CO2. Si nécessaire, dilater les fibroblastes pendant une plus longue période jusqu'à ce que le passage et la confluence souhaités soient atteints.
- Enrober une plaque traitée à la culture de 6 puits de tissus avec 500 l de solution de gélatine de 0,1 % et couver à 37 oC pendant 20 min. Enlever la gélatine supplémentaire.
- Plaques HDF à une densité de 150.000 cellules par plaque (25.000 cellules par puits) dans 2 ml de DMEM complet par puits. Incuber toute la nuit à 37 oC, 5 % de CO2,pour permettre l'attachement cellulaire.
- Remplacer le milieu par 2 ml de DMEM complet plus 8 polybrenes de g/mL. Préparer un mélange de ratio 1:1 de lentivirus TF produits en piscine et de M2rtTA dans un nouveau tube de microcentrifuge.
REMARQUE: Dans ce protocole, la production en piscine de lentivirus pour les trois TF est effectuée, ce qui, dans les mains des auteurs, se traduit par une plus grande efficacité de reprogrammation. Alternativement, il est suggéré d'effectuer une titration des particules lentivirales individuelles par qPCR18, sur une ligne cellulaire standard. Ceci sera utilisé pour définir le volume de virus individuels nécessaires pour répondre à une multiplicité d'infection (MOI) optimale pour la co-transduction et la reprogrammation hémogène. - Distribuer de 10 à 100 L de mélange lentiviral par puits, pour transduire les HDF. C'est le jour -2 de la reprogrammation.
REMARQUE: Définir le volume optimal de mélange de lentiviraux pour une reprogrammation efficace, sans compromettre la viabilité cellulaire, nécessite une optimisation (voir la figure supplémentaire 1 pour plus de détails). Les HDF de plus de 7 passages peuvent nécessiter des volumes plus élevés de virus que les cellules ayant des passages inférieurs. - Après 16 h d'incubation, retirez les virus et ajoutez du DMEM complet. Laisser les cellules récupérer pendant 6 à 8 h.
- Après la récupération, aspirer à mi-mesure et ajouter 2 ml de DMEM complet avec 8 polybrenes g/mL.
- Faites une deuxième transduction telle que décrite à l'étape 4.6 et incuber à 37 oC, 5 % de CO2 pendant 16 h. C'est le jour -1 de la reprogrammation. Le mélange lentiviral peut être préparé le jour -2 pour les deux transductions et conservé à 4 oC.
- Le lendemain, retirez les virus et ajoutez du DMEM complet complété par un DOX de 1 g/mL. C'est le jour 0 de la reprogrammation. Incuber à 37 oC, 5 % de CO2 pour 48 h.
-
Au jour 2 de la reprogrammation, divisez chaque puits à 1:2 ratio.
- Aspirer les cellules moyennes et laver avec 1 ml de PBS.
- Aspirez PBS et dissociez les cellules avec 500 L de solution de dissociation. Incuber 5 à 10 min à 37 oC, 5 % DE CO2.
- Inactiver la solution de dissociation avec 1 ml de DMEM complet et collecter les cellules dans un tube conique. Centrifugeuse à 350 x g pendant 5 min.
- Resuspendre la pastille dans le milieu hématopoïétique (voir l'étape 1.3), complétée par 1 'g/mL DOX, et les cellules de plaque dans de nouvelles plaques de 6 puits traitées par la culture des tissus enduites de 0,1% de gélatine à un volume final de 2 ml par puits.
- Changer de milieu (moyen hématopoïétique plus DOX) deux fois par semaine pour la durée des cultures de reprogrammation (25 jours).
- Analyser les cellules reprogrammées résultantes à différents moments par microscopie de champ lumineux ou de fluorescence (voir la figure supplémentaire 2), cytométrie du débit, séquençage de l'ARN en vrac et à cellule unique, et essais de transplantation pour l'acquisition de morphologie hématopoïétique, présence de marqueurs endothéliales et hématopoïétiques, acquisition du profil d'expression génique/hématopoïétique et capacité de régénération14.
5. Optimisation de l'expansion du fibroblaste pour l'analyse ChIP-seq au début de la reprogrammation hémogène
- Plaque 300 000 HDF (lt;P8) dans 0,1 % de gélatine enduite de plaques de 6 puits traitées par culture de tissus avec un DMEM complet à un volume final de 2 ml par puits. Incuber toute la nuit à 37 oC, 5 % de CO2.
- Le lendemain, remplacer le milieu par du DMEM complet complété par du polybrene de 8 g/mL.
- Transduce cellules avec des facteurs individuels: pFUW-tetO-FOS14, pLV-tetO-HA-GFI1B (Addgene #125599)14 et pFUW-tetO-3xFLAG-GATA2 (Addgene #125600)14 ou avec un pool des trois facteurs, plus FUW-M2rtTA à 1:1 ratio. Utilisez un virus total de 10 à 20 L (TF individuel et M2rtTA ou trois TF et M2rtTA). Incuber les cellules pendant la nuit à 37 oC, 5 % de CO2.
REMARQUE: Il est recommandé d'utiliser douze plaques de 6 puits par condition (pour chaque TF individuel et les trois TF combinés). - Enlever les lentivirus et ajouter le DMEM complet 16 h après la première transduction. Laisser les cellules récupérer pendant 6 à 8 h.
- Transduce les cellules une deuxième fois avec la même quantité de virus par condition et incuber à 37 oC, 5% CO2.
- Le lendemain, supprimez les virus et ajoutez du DMEM complet. Incuber à 37 oC, 5 % de CO2 pour 24 h.
- Re-plaquer chacun bien dans un plat de 100 mm traité par la culture de tissu enduit de 100 mm de gélatine de 0,1 % avec un dMEM complet jusqu'à un volume final de 10 ml par plat. Cela représente environ un passage de 1:6.
- Laisser les cellules se développer pendant 6 jours à 37 oC, 5 % de CO2.
- Le jour 6 après le re-plaquage, aspirez à moyen et ajoutez d'un DMEM complet avec 1 DOX de 1 g/mL. Incuber les cellules à 37 oC, 5 % de CO2 pendant 2 jours.
- Recueillir les fibroblastes et analyser les sites de liaison génomique des trois TF transduisés individuellement ou en combinaison, par ChIP-seq 2 jours après la supplémentation DOX14.
REMARQUE: Les 72 derniers plats de 100 mm contiendront entre 20 et 50 x 106 cellules, suffisantes pour effectuer des expériences et des répliques ChIP-seq.
Résultats
Une représentation schématique de l'approche de reprogrammation à l'aide de HDF est illustrée dans la figure 1A. Les fibroblastes sont acquis à partir de sources commerciales ou recueillis auprès de donneurs humains et étendus in vitro avant la reprogrammation. Après le placage, les cellules sont transduisantes deux fois avec gatA2, GFI1B et FOS (et M2rtTA) lentivirus, et la doxycycline est ajoutée au jour 0 de la reprogrammation. Le jour 2, les cellules sont divisées et plaquées dans le milieu hématopoïétique jusqu'au jour 25 de la culture. Les cellules reprogrammées peuvent être générées à différents moments pour de multiples applications, y compris la transplantation chez des souris immunodéprimées, le séquençage de l'ARN unicellulaire (scRNA-seq) des populations de cellules purifiées (jour 2 non triés, jour 15 CD49f- CD34 et jour 25 CD49fetCD34, ainsi que la microscopie et l'analyse de cytométrie de flux pour les marqueurs de surface cellulaire CD49f, CD34, CD9 et CD143. Les parcelles cytométries représentatives montrent 17 % des cellules reprogrammées exprimant à la fois cD49f et CD9(figure 1B, panneau gauche),après 25 jours de reprogrammation. La majorité des cellules doublement positives expriment CD143 (-86%), et une petite population exprime CD34 (0.9%), suggérant une induction hémogène dynamique de destin. Ces marqueurs ne sont pas activés dans les HDF transducrés M2rtTA cultivés pendant 25 jours(figure 1B, panneau droit). Les images d'immunofluorescence confirment l'expression du CD9 et du CD143 dans les cellules adhérentes et rondes, morphologiquement distinctes des fibroblastes qui sont négatifs pour ces marqueurs (Figure 1C). Les colonies hémogéniques humaines expriment également CD49f et CD3414. L'analyse ScRNA-seq des HDF, des cellules non triées du jour 2 et des cellules reprogrammées purifiées au jour 15 (CD49f-CD34-) et le jour 25 (CD49fetCD34) montrent une augmentation progressive de l'expression CD49f, CD9 et CD143 du jour 2 au jour 25. Les cellules positives CD49f et CD9 apparaissent en premier au cours du processus de reprogrammation, entre le jour 2 et le 15, ce qui indique que ces molécules peuvent représenter des marqueurs de l'hémogénèse humaine précoce. L'expression CD143 commence à être détectée au jour 15 et les cellules exprimant le CD34 ne sont détectées qu'à des moments ultérieurs (jour 25). Les cellules CD34et ombilical sang de cordon ombilical (UCB) ont été utilisées comme référence (Figure 1D).
La figure 2A décrit un protocole modifié pour générer un nombre suffisant de cellules pour l'analyse ChIP-seq aux premiers stades de la reprogrammation hémogénique (jour 2). Tout d'abord, les HDF sont plaqués à une densité deux fois plus élevée que dans le protocole standard (300 000 cellules contre 150 000 cellules par plaque). Après la transduction, chaque puits est re-plaqué dans un plat de 100 mm permettant aux cellules de se développer pendant 6 jours avant de compléter le milieu avec DOX. Les cellules sont analysées 2 jours après l'ajout de DOX et l'expression TF qui en résulte. La figure 2B montre les profils de navigateur sinique de GATA2 se liant aux régions de régulation génomique de l'ITGA6 et de l'ACE lorsque les cellules sont co-transducées avec les trois facteurs (3TF) ou GATA2 individuellement. GATA2 se lie également aux régions de chromatine ouverte des gènes CD9 et CD34 14.
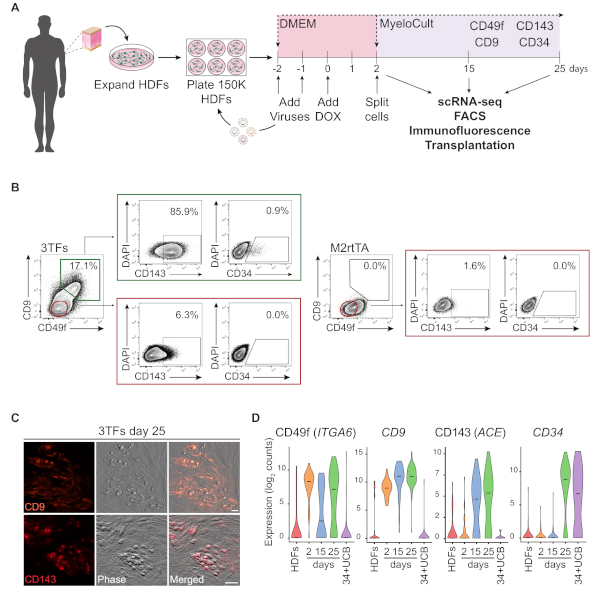
Figure 1 : Induction du destin hémogène dans les fibroblastes dermiques humains. (A) Stratégie expérimentale de reprogrammation hémogène des fibroblastes dermiques humains (HDF). Les fibroblastes provenant de biopsies de poinçon sourdeur sont prélevés auprès de donneurs, élargis et transducés avec des lentivirus GATA2, GFI1B, FOS et M2rtTA. La doxycycline (DOX) est ajoutée à la culture au jour 0 de la reprogrammation et les cellules sont analysées à plusieurs moments jusqu'au jour 25. scRNA-seq, séquençage de l'ARN à cellule unique. FACS, Tri cellulaire activé par fluorescence. (B) Stratégie de gating utilisée pour évaluer l'expression des marqueurs hémogènes/hématopoïétiques par cytométrie de flux au jour 25 après transduction avec les trois facteurs de transcription (3TFs). Les parcelles de cytométrie montrent le pourcentage de cellules doublement positives pour CD49f et CD9, fermées dans la population de cellules vivantes (DAPI-négatif). Au sein de la double population positive, l'expression de CD143 et CD34 est montrée. Les HDF transduisés uniquement par le virus M2rtTA dans les mêmes conditions de culture sont utilisés comme contrôle. (C) Images immunofluorescences des colonies reprogrammées du jour 25 confirmant l'expression du CD9 (panneau supérieur) et du CD143 (panneau inférieur). Les cellules ont été tachées avec des anticorps (Table of Materials) dilué 1:100 en PBS/2% FBS avec sérum de souris, incubé 20 min à 37 oC, 5% CO2, lavé trois fois et représenté en PBS/2% FBS. Phase, contraste phase-gradient. Barres d'échelle de 50 m. (D) Analyse ScRNA-seq de 253 cellules à différents moments. L'expression d'ITGA6, CD9, ACE et CD34 est activée lors de la reprogrammation. Les cellules sont collectées au jour 2 (non triés), au jour 15 (CD49fetCD34-) et au jour 25 (CD49fetCD34). Les hDF et les cellules CD34et le sang de cordon ombilical (34 UCB) sont utilisés comme références. Veuillez cliquer ici pour voir une version plus grande de ce chiffre.
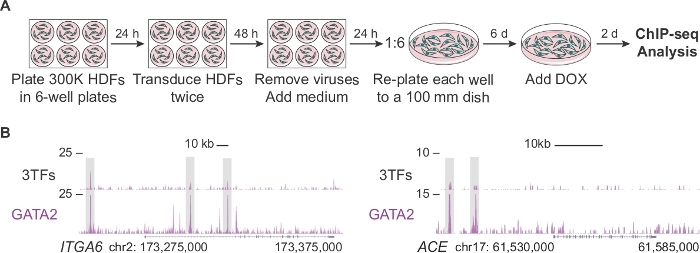
Figure 2 : Expansion des fibroblastes dermiques humains pour l'analyse ChIP-seq. (A) Stratégie expérimentale représentant un protocole modifié pour générer un grand nombre de fibroblastes cutanés humains transducteurs (HDF) pour ChIP-seq au deuxième jour de la reprogrammation. 300 000 cellules sont plaquées dans des plaques de 6 puits et transduisent deux fois avec des facteurs individuels (pFUW-tetO-FOS, pLV-tetO-HA-GFI1B ou pFUW-tetO-3xFLAG-GATA2) ou une combinaison des trois facteurs (plus M2rtTA). Après avoir enlevé les virus, les fibroblastes sont étendus pendant six jours dans des plats de 100 mm. La doxycycline (DOX) est ajoutée au jour 0 et les cellules sont collectées deux jours après l'ajout de DOX. (B) Profils de navigateur sinigies des sites de liaison GATA2 (boîtes grises) à ITGA6 et ACE loci deux jours après la transduction avec les trois facteurs de transcription (3TF) ou avec GATA2 seul. Le nombre total de lectures cartographiées est représenté sur l'axe y. Veuillez cliquer ici pour voir une version plus grande de ce chiffre.
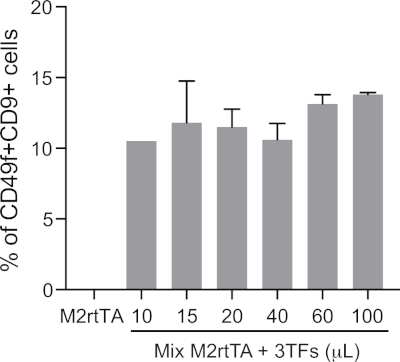
Figure supplémentaire 1 : Définir un volume lentiviral optimisé pour une reprogrammation hémogène efficace. Des volumes croissants de particules lentivirales concentrées (10 à 100 l) produites en piscine (3TF : GATA2, GFI1B et FOS) sont utilisés pour transduire les fibroblastes dermiques humains (HDF), ainsi que M2rtTA à un ratio de 1:1, suivant les étapes 4.5-4.12 du protocole. Les cellules reprogrammées sont analysées au jour 25 pour définir un volume optimal de transduction pour la reprogrammation hémogénique, donné par le pourcentage decellules CD49fetCD9 fermées dans des cellules vivantes (DAPI-négatif). La viabilité des cellules peut être évaluée en quantifiant le nombre absolu de cellules vivantes au jour 25. Les HDF transduisés par M2rtTA (100 l) sont utilisés comme contrôle négatif. Veuillez cliquer ici pour voir une version plus grande de ce chiffre.

Figure supplémentaire 2 : Changements de morphologie pendant la reprogrammation hémogène des fibroblastes dermiques humains. Les cultures de fibroblaste sécarcieux humain (HDF) sont représentées le jour de la première transduction (jour -2), lorsque le DOX est ajouté aux cultures (jour 0), deux jours (jour 2) et quinze jours (jour 15) après la supplémentation DOX, et au point final de l'expérience (jour 25). Les colonies hémogéniques aux jours 15 et 25 sont mises en évidence. Barres d'échelle de 100 m. Veuillez cliquer ici pour voir une version plus grande de ce chiffre.
Discussion
Dans cet article, une méthode est décrite pour produire des cellules hématopoïétiques progénitrices directement à partir des fibroblastes humains, qui passent par une cellule HP intermédiaire, de la même manière que les HSC définitifs14.
La production en piscine de particules lentivirales codant gatA2, GFI1B et FOS a été préférée à la production individuelle, car dans nos mains, il en résulte une plus grande efficacité de reprogrammation (données non publiées). Les lentivirus, en tant que membres de la famille des Rétroviridae, contiennent normalement deux copies d'ARN positif à brin unique19. L'efficacité accrue de la reprogrammation peut être due à l'emballage de deux transgènes différents dans la même particule lentivirale, ce qui entraîne une augmentation du nombre de cellules co-cédées avec les trois facteurs de transcription. Pour assurer le succès de ce protocole, il est nécessaire de transmettre des HDF avec une quantité suffisante de virus en fonction du passage cellulaire pour obtenir un équilibre optimal entre l'efficacité de la reprogrammation et la viabilité cellulaire, comme recommandé à l'étape 4.6. De plus, des virus frais non concentrés peuvent être utilisés. Il est recommandé de transduire les cellules avec 0,5-3 mL de piscine 3TFs et M2rtTA. En outre, la densité cellulaire doit être ajustée en fonction de l'application. 150 000 HDF par plaque de 6 puits (étape 4.4) ont fourni la densité optimale pour effectuer l'analyse de FACS, de transplantation et de cytométrie d'écoulement des cellules reprogrammées. Pour les expériences ChIP-seq, plus de cellules ont été nécessaires dès le début (étape 5.1). Il est important de vérifier régulièrement les cellules pour les changements morphologiques et remplacer le milieu hématopoïétique deux fois par semaine pour soutenir l'émergence de cellules hématopoïétiques induites. L'ajout de cytokines hématopoïétiques ou de co-culture dans les couches d'engraissement peut augmenter l'efficacité de la reprogrammation.
Avec cette méthode, nous pouvons identifier de nouveaux marqueurs hématopoïétiques qui sont exprimés dynamiquement lors de la reprogrammation hémogène. Le CD9, qui s'est avéré être régulé dans les cellules reprogrammées au niveau transcriptionnel14,s'exprime rapidement à la surface de la cellule dans les phases initiales de reprogrammation avec CD49f et CD143, servant de marqueur nouveau des précurseurs humains de HSC. Nous montrons également que ITGA6 et ACE sont des cibles directes de GATA2 au cours des premières étapes de la reprogrammation hémogène, en plus de CD9 et CD3414, fournissant un lien mécaniste direct entre l'homme hémogène phénotype précurseur et GATA2.
L'un des avantages de ce système réside dans l'utilisation de cultures de fibroblastes relativement homogènes. Tandis que les CFP sont facilement étendus et maintenus in vitro,les protocoles de différenciation génèrent des populations hétérogènes qui incluent les progéniteurs hématopoïétiques, qui s'engraftent mal5,6,7. En outre, il y a un risque de tumorigenesis en transplantant des HSPc PSC-dérivés de PSC, puisque les PSC indifférenciés peuvent toujours rester dans la culture même après employer des protocoles de différenciation. Alternativement aux fibroblastes, la reprogrammation directe aux HSC a été appliquée aux progéniteurs sanglants20 et aux cellules endothéliales21. Cependant, en commençant par les cellules progénitrices dans le sang restreint entrave l'application thérapeutique des HSC résultantsi le patient porte des mutations qui affectent la population hématopoïétique de tige/progénitrice22. Dans le cas des cellules endothéliales, celles-ci sont plus difficiles à obtenir par rapport aux fibroblastes, et constituent une population cellulaire très hétérogène en termes de phénotype, de fonction et de structure, qui sont tributaires des organes23. D'autres études ont réussi à reprogrammer les fibroblastes de souris en progéniteurs hématopoïétiques engraftables24,25 pourtant, jusqu'à présent, aucun autre protocole ne décrit la génération de cellules de HSPC-comme des fibroblastes humains.
Cette approche, associée à l'inhibition pharmacologique, aux permis d'élimination génétique ou d'élimination des gènes pour définir des facteurs individuels ou combinés qui sont nécessaires pour induire directement les HSC humains. L'utilisation de méthodes de dépistage à haut rendement fondées sur des CrispR-Cas9 technologies dans hDFs avant la reprogrammation, représente un effort passionnant pour définir de nouveaux régulateurs de l'hématopoiesis humain définitif. À l'avenir, la reprogrammation de types de cellules humaines non liées au sang, comme les fibroblastes, servira de plate-forme pour générer des cellules progénitrices hématopoïétiques saines adaptées aux patients pour des applications cliniques.
Déclarations de divulgation
Les auteurs n'ont rien à révéler.
Remerciements
La fondation Knut et Alice Wallenberg, la Faculté de médecine de l'Université de Lund et la région de Skône sont reconnues pour leur généreux soutien financier. Ce travail a été soutenu par une subvention d'Olle Engkvists Stiftelse (194-0694 à Filipe Pereira) et des bourses de doctorat de Fundaçao para a Ciência e Tecnologia (PTDC/BIM-MED/0075/2014 à Filipe Pereira, et SFRH/BD/135725/2018 et SFRH/BD/51968/2012 à Rita Alves et Andreia Gomes). Cette étude a également été soutenue par des fonds des NIH et du NYSTEM (1R01HL119404 et C32597GG à Kateri A. Moore).
matériels
| Name | Company | Catalog Number | Comments |
| 0.45 μm low-protein binding filter, 150 mL Bottle Top Vacuum Filter | Corning | #430625 | |
| 2-Mercaptoethanol | Sigma-Aldrich | #M6250 | |
| Alexa Fluor 488 anti-human CD34 clone 581 | BioLegend | #343518 | |
| BD Pharmingen APC Mouse Anti-Human Angiotensin Converting Enzyme (CD143) clone BB9 | BD Biosciences | #557929 | |
| BES buffered saline | Sigma-Aldrich | #14280 | |
| Calcium chloride (CaCl2) | Sigma-Aldrich | #449709 | |
| Centrifugal filter unit, Amicon Ultra-15 Centrifugal Filter Unit | Sigma-Aldrich | #UFC903096 | |
| Dissociation solution, TrypLE Express Enzyme (1x) no phenol red | Gibco | #12604-021 | |
| Doxycycline hyclate (DOX) | Sigma-Aldrich | #D9891 | |
| eBioscience CD49f (Integrin alpha 6) Monoclonal Antibody (eBioGoH3 (GoH3)), PE-Cyanine7 | Invitrogen | #25-0495-82 | |
| FUW-M2rtTA | Addgene | #20342 | |
| Gelatin from Porcine Skin Type A | Sigma-Aldrich | #G1890 | |
| Gibco L-Glutamine (200 mM) | ThermoFisher Scientific | #25030-024 | |
| Gibco MEM Non-Essential Amino Acids Solution (100x) | ThermoFisher Scientific | #11140-035 | |
| Hematopoietic medium, MyeloCult H5100 | STEMCELL Technologies | #05150 | |
| Hexadimethrine bromide (polybrene) | Sigma-Aldrich | #H9268 | |
| Human Dermal Fibroblasts (HDFs) | ScienCell | #2320 | |
| HyClone Dulbecco's Modified Eagles Medium (DMEM) | GE Healthcare | #SH30243.01 | |
| HyClone Fetal Bovine Serum (FBS) | GE Healthcare | #SV30160.03 | |
| HyClone Penicillin Streptomycin 100x Solution (Pen/Strep) | GE Healthcare | #SV30010 | |
| HyClone Phosphate Buffered Saline solution (PBS) | GE Healthcare | #SH30256.01 | |
| Hydrocortisone | STEMCELL Technologies | #7904 | |
| Mouse serum | Sigma-Aldrich | #M5905 | |
| PE anti-human CD9 Antibody clone HI9a | BioLegend | #312105 | |
| pFUW-tetO-3xFLAG-GATA2 | Addgene | #125600 | |
| pFUW-tetO-FOS | Addgene | #125598 | |
| pFUW-tetO-GATA2 | Addgene | #125028 | |
| pFUW-tetO-GFI1B | Addgene | #125597 | |
| pLV-tetO-HA-GFI1B | Addgene | #125599 | |
| pMD2.G | Addgene | #12259 | |
| psPAX2 | Addgene | #12260 |
Références
- Ivanovs, A., et al. Highly potent human hematopoietic stem cells first emerge in the intraembryonic aorta-gonad-mesonephros region. Journal of Experimental Medicine. 208, 2417-2427 (2011).
- Tavian, M., Biasch, K., Sinka, L., Vallet, J., Péault, B. Embryonic origin of human hematopoiesis. International Journal of Developmental Biology. 1065, 1061-1065 (2010).
- Medvinsky, A., Rybtsov, S., Taoudi, S. Embryonic origin of the adult hematopoietic system: advances and questions. Development. 138, 1017-1031 (2011).
- Ivanovs, A., et al. Human haematopoietic stem cell development: from the embryo to the dish. Development. 144, 2323-2337 (2017).
- Daniel, M. G., Pereira, C. F., Lemischka, I. R., Moore, K. A. Making a Hematopoietic Stem Cell. Trends in Cell Biology. 26, 202-214 (2016).
- Vo, L., Daley, G. De novo generation of HSCs from somatic and pluripotent stem cell sources. Blood. 125, 2641-2648 (2015).
- Rafii, S., et al. Human ESC-derived hemogenic endothelial cells undergo distinct waves of endothelial to hematopoietic transition. Blood. 121, 770-781 (2013).
- Ebina, W., Rossi, D. J. Transcription factor-mediated reprogramming toward hematopoietic stem cells. EMBO Journal. 34, 694-709 (2015).
- Sugimura, R., et al. Haematopoietic stem and progenitor cells from human pluripotent stem cells. Nature. 545, 432-438 (2017).
- Pereira, C. F., et al. Induction of a Hemogenic Program in Mouse Fibroblasts. Cell Stem Cell. 13, 205-218 (2013).
- Pereira, C. F., et al. Hematopoietic Reprogramming In vitro Informs In Vivo Identification of Hemogenic Precursors to Definitive Hematopoietic Stem Cells. Developmental Cell. 36, 525-539 (2016).
- Notta, F., et al. Isolation of Single Human Hematopoietic Stem Cells Capable of Long-Term Multilineage Engraftment. Science. 333, 218-221 (2011).
- Sinka, L., Biasch, K., Khazaal, I., Péault, B., Tavian, M. Angiotensin-converting enzyme (CD143) specifies emerging lympho-hematopoietic progenitors in the human embryo. Blood. 119, 3712-3724 (2012).
- Gomes, A. M., et al. Cooperative Transcription Factor Induction Mediates Hemogenic Reprogramming. Cell Reports. 25, 2821-2835 (2018).
- Karlsson, G., et al. Report The Tetraspanin CD9 Affords High-Purity Capture of All Murine Hematopoietic Stem Cells. Cell Reports. 4, 642-648 (2013).
- Leung, K. T., et al. The tetraspanin CD9 regulates migration, adhesion, and homing of human cord blood CD34+ hematopoietic stem and progenitor cells. Blood. 117, 1840-1851 (2011).
- Hockemeyer, D., et al. A drug-inducible system for direct reprogramming of human somatic cells to pluripotency. Cell Stem Cell. 3, 346-353 (2008).
- Kutner, R. H., Zhang, X., Reiser, J. Production concentration and titration of pseudotyped HIV-1-based lentiviral vectors. Nature Protocols. 4, 495-505 (2009).
- Suzuki, Y. S., Suzuki, Y., Ke, X. Gene Regulatable Lentiviral Vector System. Viral Gene Therapy. , (2011).
- Riddell, J., et al. Reprogramming committed murine blood cells to induced hematopoietic stem cells with defined factors. Cell. 157, 549-564 (2014).
- Lis, R., et al. Conversion of adult endothelium to immunocompetent haematopoietic stem cells. Nature. 545, 439-445 (2017).
- Pereira, C., Lemischka, I. R., Moore, K. From blood to blood’: de-differentiation of hematopoietic progenitors to stem cells. EMBO Journal. 33, 1511-1513 (2014).
- Nolan, D. J., et al. Molecular Signatures of Tissue-Specific Microvascular Endothelial Cell Heterogeneity in Organ Maintenance and Regeneration. Developmental Cell. 26, 204-219 (2013).
- Batta, K., Florkowska, M., Kouskoff, V., Lacaud, G. Direct Reprogramming of Murine Fibroblasts to Hematopoietic Progenitor Cells. Cell Reports. 9, 1871 (2014).
- Cheng, H., et al. Reprogramming mouse fibroblasts into engraftable myeloerythroid and lymphoid progenitors. Nature Communications. 7, 1-15 (2016).
Erratum
Formal Correction: Erratum: Hemogenic Reprogramming of Human Fibroblasts by Enforced Expression of Transcription Factors
Posted by JoVE Editors on 12/03/2019. Citeable Link.
An erratum was issued for: Hemogenic Reprogramming of Human Fibroblasts by Enforced Expression of Transcription Factors. The author affiliations were updated.
The second author affiliation was updated from:
2Wallenberg Center for Molecular, Lund University
to:
2Wallenberg Center for Molecular Medicine, Lund University
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon