Method Article
Riprogrammazione emogenica dei fibroblasti umani mediante l'espressione forzata dei fattori di trascrizione
In questo articolo
Erratum Notice
Riepilogo
Questo protocollo dimostra l'induzione di un programma emogenico nei fibroblasti dermici umani mediante l'espressione forzata dei fattori di trascrizione GATA2, GFI1B e FOS per generare cellule staminali e progenitrici ematopoietiche.
Abstract
I meccanismi cellulari e molecolari alla base delle specifiche delle cellule staminali ematopoietiche umane (HSC) rimangono sfuggenti. Le strategie per ricapitolare l'emergere umana di HSC in vitro sono necessarie per superare i limiti nello studio di questo complesso processo di sviluppo. Qui, descriviamo un protocollo per generare cellule staminali ematopoietiche e progenitrici da fibroblasti dermici umani utilizzando un approccio diretto di riprogrammazione cellulare. Queste cellule transitano attraverso un tipo di cellula intermedio emogenico, simile alla transizione endoteliale-ematopoietica (EHT) caratteristica della specifica HSC. I fibroblasti sono stati riprogrammati in cellule emogeniche tramite trasduzione con fattori di trascrizione GATA2, GFI1B e FOS. Questa combinazione di tre fattori ha indotto cambiamenti morfologici, espressione di marcatori emogeni ed ematopoietici e programmi trascrizionali EHT dinamici. Le cellule riprogrammate generano progenie ematopoietica e ripopolano topi immunodeficienti per tre mesi. Questo protocollo può essere adattato alla dissezione meccanicistica del processo EHT umano, come esemplificato in questo caso definendo gli obiettivi GATA2 durante le prime fasi della riprogrammazione. Pertanto, la riprogrammazione emogenica umana fornisce un approccio semplice e trattabile per identificare nuovi marcatori e regolatori dell'emergenza Umana dell'HSC. In futuro, l'induzione fedele del destino emogenico nei fibroblasti può portare alla generazione di HSC specifici per il paziente per il trapianto.
Introduzione
Le cellule staminali ematopoietiche definitive (HPG) emergono nella regione aorta-gonad-mesonephros (AGM) e in placenta da precursori endoteliali con capacità emogenetica, attraverso una transizione endoteliale-ematopoietica (EHT)1, 2. I precursori emogenici (HP) esprimono marcatori sia endoteliali che ematopoietici, ma la loro precisa identificazione rimane sfuggente, in particolare nel sistema umano. Nonostante sia un processo relativamente conservato nei mammiferi, lo sviluppo di cellule staminali ematopoietiche (HSC) differisce ancora in modo significativo tra gli esseri umani e i modelli murini3,4. Pertanto, sono necessari approcci in vitro per riassumere lo sviluppo umano dell'HSC.
La differenziazione delle cellule staminali pluripotenti (PSC) agli HSC, sebbene promettente, ha incontrato un successo limitato negli ultimi 20 anni, principalmente a causa dei protocolli di differenziazione disponibili, che si traducono in primitivi progenitori ematopoietici con scarso innesto abilità5,6,7. In alternativa, sono state applicate metodologie di riprogrammazione diretta delle cellule per generare celle simili a HSPC da più tipi di cellule, utilizzando fattori di trascrizione (TF)8,9. In particolare, la sovraespressione di tre TF, Gata2, Gfi1b e cFos, ha convertito i fibroblasti embrionali di topo in HSPC attraverso un intermedio HP con un fenotipo definito (Prom1-Sca-1-CD34-CD45-)10. Questo processo assomigliava all'EHT che si verifica nell'embrione e nella placenta, durante la specifica dell'ematopoiesi definitiva. Questo fenotipo ha permesso l'identificazione e l'isolamento di una popolazione di NP nella placenta del topo che dopo la coltura a breve termine e l'attivazione di Notch ha generato HSC trapiantabili serialmente11.
Finora non è stato stabilito alcun fenotipo che distingua gli HSC umani dai loro precursori, ma alcune molecole sono note per essere espresse nelle HSC emergenti. cellule nel compartimento HSC12e enzima di conversione dell'angiotensina (ACE o CD143) è presente nei precursori ematopoietici negativi CD34 nei tessuti embrionali ematopoietici13.
Recentemente, abbiamo dimostrato che le versioni umane dei tre TF, GATA2, FOS e GFI1B riprogrammano i fibroblasti dermici umani (HDF) in HP con capacità di innesto a breve termine14. Nelle fasi iniziali della riprogrammazione, GATA2 impegna la cromatina aperta e recluta GFI1B e FOS per reprimere i geni fibroblasti e attivare i geni endoteliali ed ematopoietici. Cellule indotte altamente espresso CD49f e ACE, e conteneva una piccola percentuale di cellule che esprimono il marcatore HSPC CD34. Il gene CD9, espresso negli HSC15 ed è importante per l'HSC homing16, si è dimostrato un bersaglio diretto di GATA2 e tra i geni più regolati nelle cellule riprogrammate14. Il CD9 può quindi costituire un ulteriore indicatore per gli ALTRI dell'ematopoiesi umana definitiva.
In questo protocollo, descriviamo la generazione di cellule simili all'HSPC dai fibroblasti umani attraverso l'espressione forzata di GATA2, GFI1B e FOS, nonché un metodo adattato per l'immunoprecipitazioni della cromatina (seq) (seq) Riprogrammazione. I TF sono stati codificati in un vettore lentivirale inducibile (pFUW-tetO) che contiene un elemento di risposta tetraciclina (TRE) e un promotore CMV minimo, e sono stati trasdotti insieme a un vettore costitutivo contenente la tetraciclina inversa proteina trasattivatore (pFUW-M2rtTA). Quando DOX (analogico di tetraciclina) viene aggiunto dopo la trasduzione, si lega alla proteina RTTA che interagisce con la trascrizione TRE permettendo TF (sistema Tet-On). Il completamento della procedura richiede 25 giorni. Per gli esperimenti ChIP-seq, gli MF sono stati transdotti con versioni taggate di GATA2 (pFUW-tetO-3xFLAG-GATA2) e GFI1B (pLV-tetO-HA-GFI1B), oltre a siti di binding pFUW-tetO-e TF analizzati due giorni dopo il completamento di DOX.
In definitiva, la riprogrammazione emogenica dei fibroblasti umani fornisce un sistema in vitro trattabile per studiare i meccanismi alla base dell'ematopoiesi dello sviluppo umano e una potenziale fonte di HPG specifici del paziente per l'applicazione clinica futura.
Protocollo
Questo protocollo è stato eseguito secondo le linee guida del comitato etico della ricerca umana dell'Università di Lund e dovrebbe essere fatto in conformità con le singole linee guida istituzionali.
1. Preparazione del reagente
- Per il siero bovino bovino fetale (FBS) modificato da Dulbecco, miscelare il DMEM ad alto glucosio contenente pirava di sodio con il 20% di FBS, l'1% di penicillina-streptomicina (pen/streptomici), l'1% di l-glutamine, il 1% di acidi non essenziali di amminom e 10-4 -2-M- Mercaptoetanolo.
- Per il DMEM completo, mescolare il DMEM ad alto glucosio contenente piravate di sodio con 10% FBS, 1% Penna/strepe e 1% L-glutamina.
- Per il mezzo ematopoietico, mescolare il mezzo ematopoietico (Tabella dei materiali) con 10-6 M idrocortisone e 1% penna/strep.
- Utilizzare la salina tampone fosfato (PBS) senza calcio o magnesio.
2. Isolamento del fibroblasto dermico umano
NOT: Gli HDF possono essere acquistati presso fornitori certificati (Table of Materials). In tal caso, espandere i fibroblasti e utilizzarli direttamente negli esperimenti di riprogrammazione (sezione 4). In alternativa, gli HDF possono essere isolati dai donatori. Se i fibroblasti sono isolati da donatori diversi, tenere i campioni separati l'uno dall'altro in tutte le fasi del protocollo. Etichettare piastre/pozzi e tubi di raccolta con il numero di identificazione di ciascun donatore.
- Ottenere HDF da biopsie punzone pelle rotonda da 3 mm eseguite da medici qualificati.
- Cappotto tre pozze di una piastra di 6 pozze di tessuto trattata con 500 L di 0,1% di gelatina e incubare per 20 min a 37 gradi centigradi.
- Aspirati la soluzione di gelatina rimanente e aggiungete 750 -L di DMEM/20% di FBS ad ogni pozzo. L'intera superficie del pozzo deve essere coperta con il mezzo.
- Aggiungere 1,5 mL di DMEM/20% FBS alla superficie interna di un coperchio sterile da 100 mm per parabola di Petri e stendere la goccia con l'aiuto di una pipetta sierologica da 5 mL.
- Posizionare la biopsia cutanea nel mezzo sul coperchio con pinze sterilizzate.
- Sezionare la biopsia cutanea in nove sezioni identiche, utilizzando un bisturi sterilizzato per tenere la biopsia in posizione e un secondo bisturi da tagliare.
- Posizionare tre pezzi di biopsia per pozzo utilizzando pinze appuntite. Assicurarsi che i pezzi si attacchino alla parte inferiore del pozzo.
- Posare un coperchio da 22 mm sopra i pezzi e applicare un po 'di pressione.
- Incubare la piastra a 37 gradi centigradi, 5% CO2, per una settimana. Controllare le cellule ogni giorno e aggiungere 200 L di DMEM/20% FBS ogni 2 giorni per sostituire il mezzo evaporato.
- Dopo una settimana, aggiungere fino a 2 mL di DMEM/20% FBS e sostituire medio ogni 2/3 giorni.
-
Celle di passaggio a rapporto 1:4 quando i pozzi sono confluenti (circa 4-8 settimane).
- Preparare lo 0,1% di tessuto rivestito di gelatina trattati con 6 pozze di acqua.
- Mezzo aspirato da pozzi all'80% di confluenza e lavare una volta con 1 mL di PBS.
- Rimuovere il coperchio con pinze sterili e posizionare il coperchio in un nuovo pozzo di una piastra di 6 pozzetti, con il lato del tessuto verso l'alto.
NOT: Saranno raccolte anche le cellule rimaste attaccate al coperchio. - Aggiungere 500 l di soluzione di dissociazione (Tabella dei materiali) per pozzo (compresi i pozzetti con i coprilabbra) e incubare a 37 c, 5% CO2 per 5-10 min. Controllare quando le cellule iniziano a salire dal fondo del pozzo e inattivare la soluzione di dissociazione aggiungendo 500 L di DMEM/20% FBS ad ogni pozzo.
- Raccogliere i fibroblasti da tutti i pozzi in un tubo conico da 15 mL. Aggiungere un mezzo extra ai pozzetto per raccogliere le cellule rimanenti. Centrifugare il tubo a 350 x g per 5 min.
- Nel frattempo, aggiungere 500 L di DMEM/20% FBS ad ogni pozzo di piatti precedentemente rivestiti di gelatina.
- Aspirati media e resuspend fibroblasts in 6 mL di DMEM/20% FBS.
- Aggiungere 500 l di sospensione del fibroblasto ad ogni pozzo (due piastre 6-pozze per campione/donatore in totale). Incubare le cellule durante la notte a 37 gradi centigradi, 5% DI CO2.
- Il giorno successivo, aggiungere 1 mL di DMEM/20% FBS a ogni pozzo. Sostituire il supporto con 2 mL di DMEM/20% FBS ogni 2/3 giorni fino a quando i pozzi sono confluenti dell'80%.
- Ripetere la sezione 2.11 per tre pozzi confluenti fino al raggiungito terzo passaggio.
-
Congelare i fibroblasti dai pozzi confluenti (passaggi 1 e 3).
- Aspirare mezzo dai pozzi e lavare una volta con 1 mL di PBS.
- Dissociare e raccogliere i fibroblasti come descritto nei passaggi 2.11.4 e 2.11.5.
- Contare le cellule con un emocitometro e centrifugare il tubo a 350 x g per 5 min.
- Dopo la centrifugazione, aspirare mezzo e resuspend fibroblasts in FBS con 10% DMSO ad una densità di 5 x 105 cellule / mL.
- Aggiungere 1 mL della sospensione cellulare per crioviale e congelare le celle durante la notte a -80 gradi centigradi utilizzando un contenitore di congelamento. Spostare le fiale a -150 gradi centigradi (azoto liquido) per lo stoccaggio a lungo termine.
3. Produzione Lentivirale
- Coltivare le cellule HEK293T in un piatto di 100 mm trattato con coltura di tessuto con 10 mL di DMEM completo, a 37 c, 5% DI CO2, fino a raggiungere la confluenza.
- Il giorno prima della trasfezione, aspirare mezzo e lavare il piatto con attenzione con 5 mL di PBS.
- Dopo aver rimosso PBS, aggiungere 1,5 mL di soluzione di dissociazione e incubare a 37 gradi centigradi, 5% di CO2 per 5/10 min per dissociare le cellule dal piatto.
NOT: Si raccomanda di riscaldare sia PBS che la soluzione di dissociazione prima dell'uso, in modo che le cellule non subiscano uno shock termico. - Inattivare la soluzione di dissociazione con 3 mL di DMEM completo e trasferire la sospensione cellulare in un tubo conico da 15 mL. Lavare il piatto con 5 mL di DMEM completo per rimuovere le celle rimanenti collegate e trasferire questo volume al tubo conico da 15 mL.
- Sospensione cellulare centrifuga a 350 x g per 5 min.
- Aspirato supernatante e dividere il pellet cellulare in modo uniforme tra sei piatti trattati con coltura di tessuti da 100 mm in un volume finale di 10 mL di DMEM completo per piatto. Le cellule devono essere circa il 60% confluenti al momento della trasfezione.
- Il giorno successivo, le cellule transfett con le miscele di plasmidi come segue:
NOTA: Questa parte del protocollo descrive la produzione di lentivirus in un piatto trattato con 100 mm come coltura di tessuto per mix di plasmide. Per ottenere volumi più elevati di supernatali lentivirali per la concentrazione, utilizzare almeno quattro piatti di coltura cellulare HEK293T da 100 mm per mix.- In un tubo conico da 15 mL, aggiungere insieme 10 g di tre plasmidi di trasferimento: 3,33 g di pFUW-tetO-GATA2 (Addgene plasmid #125028)14, 3,33 g di pFUW-tetO-GFI1B (Addgene #125597)14 e 3,33 g di pFUW-tetO-FOS (Add #125598gene) 10 g del vettore di imballaggio psPAX2 di 2di nd generazione che codifica i geni Gag, Pol, Tat e Rev (Addgene #12260) e 5 g di vettore di inviluppo pMD2.G che codifica il gene VSV-G (Addgene #12259). Aggiungere acqua fino a 500 l.
- In due nuovi tubi conici da 15 mL si aggiungono 10 g di plasmide FUW-M2rtTA (Addgene #20342)17, 10 g di vettore di imballaggio psPAX2 e 5 g di vettore di inviluppo pMD2.G ad ogni tubo. Aggiungere acqua fino a 500 l. Un tubo verrà usato come controllo.
- Ad ogni tubo aggiungere 62,5 gradi di 2 M CaCl2. Successivamente, rilasciare le bolle in ogni miscela utilizzando un pipet Pasteur inserito in un controller pipet. Mentre si stanno formando bolle, pipette 500 - L di N,N-bis(2-idrossitohyl)-2-aminoethanesulfonico acido (BES) bufferato salina (pH 7.1, 25 ) ), con una pipetta P1000, gocciolanti contro il pipet Past e sulla miscela.
- Incubare i tubi a temperatura ambiente per almeno 15 min. Le miscele appariranno leggermente nuvolose dopo qualche tempo.
- Nel frattempo, mezzo aspirato da piatti cellulari HEK293T (passato il giorno prima) e aggiungere 10 mL di DMEM completo senza antibiotici. Fare attenzione e aggiungere media lentamente come cellule HEK293T sono semi-aderenti.
- Distribuire ogni miscela individuale (circa 1 mL) in modo uniforme e drop-saggio in piatti separati e incubare durante la notte a 37 , 5% CO2.
- Sostituire il supporto con 4 mL di DMEM completo, 24 h dopo l'incubazione. Incubare pernottamento a 37 gradi centigradi, 5% CO2. Se disponibile, incubare invece a 32 gradi centigradi, 5% di CO2, poiché la temperatura ridotta aumenterà l'emivita delle particelle lentivirali.
- Raccogliere supernatante con particelle lentivirali tre volte in un tubo conico da 50 mL. Non mescolare diverse particelle lentivirali a questo punto. Ogni piatto si tradurrà in 12 mL di supernatante lentivirale. Quattro piatti della stessa preparazione virale si inseriscono in un tubo conico da 50 ml.
PRUDENTE: Eseguire la raccolta lentivirale in un laboratorio di biosicurezza livello 2 in una cappa di flusso laminare dedicata al lavoro lentivirale e posizionare i rifiuti contaminati virali (tubi, punte, piatti) in un contenitore appropriato per materiali biopericolosi.- Fare la prima collezione 16 h dopo l'ultima incubazione e aggiungere 4 mL di DMEM completo. Incubare a 37 gradi centigradi, 5% CO2.
- Fate la seconda collezione 8 h dopo la prima allo stesso tubo, aggiungete 4 mL di DMEM completa e incubate a 37 gradi centigradi, 5% CO2.
- Fate la terza collezione 16 h dopo la seconda allo stesso tubo e scartate i piatti.
NOT: Conservare i supernatanti lentivirali a 4 gradi centigradi dopo ogni collezione.
- Filtrare ogni supernatante lentivirale utilizzando un filtro legante a bassa proteina di 0,45 m con una membrana di acetato di cellulosa (Tabella dei materiali) in un tubo pulito.
- Aggiungere un massimo di 15 mL di supernatante filtrato a un'unità filtrante centrifuga con una membrana di cellulosa rigenerata (Tabella dei materiali) e girare a 4.000 x g per 25 min, a 4 gradi centigradi. Scartare il flow-through. Un liquido viscoso contenente lentivirus rimarrà nell'unità filtro.
- Ripetere il passaggio 3.13 aggiungendo 15 mL di supernatante sopra l'unità filtro, fino a quando non rimane più supernatante lentivirale.
NOT: Quando ci sono solo pochi millilitri di supernatante da concentrazione, diminuire il tempo di filatura a 10 min. Se sul filtro è ancora presente un liquido aggiuntivo (non viscoso), centrifugare per altri 10 min. - Crea delle aliquote di ogni tipo di lentivirus concentrate e conserva a -80 gradi centigradi per lo stoccaggio a lungo termine (1o2 anni) o a 4 gradi centigradi per lo stoccaggio a breve termine (1/2 settimane).
NOT: Anche i lentivirus concentrati o non concentrati possono essere utilizzati freschi. Non ricongelare e scongelare in quanto ciò comporta una riduzione del titro.
4. Riprogrammazione emogenica
NOT: Utilizzare HDF con un passaggio di tre (P3) o superiore (fino a P10) per eseguire esperimenti di riprogrammazione.
- Rivestire un piatto di 100 mm trattato con 5 mL di gelatina dello 0,1% e incubare a 37 gradi centigradi per 20 minuti. Aspirate la soluzione di gelatina rimanente.
- Scongelare una fiala fibroblasta e le cellule della piastra nel piatto rivestito di gelatina dello 0,1%. Incubare pernottamento a 37 gradi centigradi, 5% CO2. Se necessario, espandere i fibroblasti per un periodo di tempo più lungo fino a raggiungere il passaggio e la confluenza desiderati.
- Rivestire una piastra trattata con una coltura tissutale di 6 pozze con 500 gradi di gelatina dello 0,1% e incubare a 37 gradi centigradi per 20 min. Rimuovere la gelatina supplementare.
- Appiatti a densità di 150.000 cellule per piastra (25.000 cellule per pozzo) in 2 mL di DMEM completo per pozzo. Incubare peruna durante la notte a 37 gradi centigradi, 5% CO2, per consentire l'attaccamento cellulare.
- Sostituire il supporto con 2 mL di DMEM completo più 8 g/mL di polibrene. Preparare un mix di rapporto 1:1 di lentivirus TF prodotti in piscina e M2rtTA in un nuovo tubo di microcentrifuga.
NOT: In questo protocollo, viene eseguita la produzione in pool di lentivirus per le tre TF, che, nelle mani degli autori, si traduce in una maggiore efficienza di riprogrammazione. In alternativa, si suggerisce di eseguire una titolazione delle singole particelle lentivirali da qPCR18, su una linea cellulare standard. Questo sarà utilizzato per definire il volume dei singoli virus necessari per soddisfare una molteplicità di infezione (MOI) ottimale per la co-trasduzione e la riprogrammazione emogenica. - Distribuire da 10 a 100 l di miscela lentivirale per pozzo, per traducire gli HDF. Questo è il giorno -2 della riprogrammazione.
NOT: La definizione del volume ottimale della miscela lentivirale per una riprogrammazione efficiente, senza compromettere la vitalità cellulare, richiede l'ottimizzazione (vedere Figura supplementare 1 per ulteriori dettagli). Gli HDF con più di 7 passaggi possono richiedere volumi di virus più elevati rispetto alle cellule con passaggi inferiori. - Dopo 16 h di incubazione, rimuovere i virus e aggiungere DMEM completo. Consentire al ripristino delle celle per 6-8 h.
- Dopo il recupero, aspirare il mezzo e aggiungere 2 mL di DMEM completo con 8 g/mL di polibrene.
- Eseguire una seconda trasduzione come descritto al punto 4.6 e incubare a 37 , 5% CO2 per 16 h. Questo è il giorno -1 della riprogrammazione. La miscela lentivirale può essere preparata il giorno -2 per entrambe le trasduzioni e mantenuta a 4 gradi centigradi.
- Il giorno successivo, rimuovere i virus e aggiungere il DMEM completo integrato con 1 og/mL DOX. Questo è il giorno 0 della riprogrammazione. Incubare a 37 gradi centigradi, 5% CO2 per 48 h.
-
Al giorno 2 della riprogrammazione, dividere ogni pozzo a 1:2 rapporto.
- Mezzo aspirato e cellule di lavaggio con 1 mL di PBS.
- Cellule PBS e dissociate aspirata con 500 litri di soluzione di dissociazione. Incubare 5/10 min a 37 gradi centigradi, 5% CO2.
- Disattivare la soluzione di dissociazione con 1 mL di DMEM completo e raccogliere le cellule in un tubo conico. Centrifuga a 350 x g per 5 min.
- Risospendere il pellet in mezzo ematopoietico (vedi passo 1.3), integrato con 1 g/mL DOX, e le cellule piatte in nuove lastre di 6 pozzetti trattati con coltura dei tessuti rivestiti con 0,1% di gelatina ad un volume finale di 2 mL per pozzo.
- Cambiare il mezzo (mezzo ematopoietico più DOX) due volte a settimana per la durata delle culture di riprogrammazione (25 giorni).
- Analizzare le cellule riprogrammate risultanti in diversi punti temporali mediante microscopia a campo luminoso o a fluorescenza (vedere Figura supplementare 2), citometria di flusso, sequenziamento di massa e RNA unicellulare e analisi dei trapianti per l'acquisizione di morfologia ematopoietica, presenza di marcatori endoteliali ed ematopoietici, acquisizione del profilo di espressione genica endoteliale/ematopoietica e capacità di rigenerazione14.
5. Ottimizzazione dell'espansione fibroblasta per l'analisi ChIP-seq all'inizio della riprogrammazione emogenica
- Piastra 300.000 HDF (2.
- Il giorno successivo, sostituire il supporto con dMEM completo integrato con 8 g/mL di polibrene.
- Traduci cellule con fattori individuali: pFUW-tetO-FOS14, pLV-tetO-HA-GFI1B (Addgene #125599)14 e pFUW-tetO-3xFLAG-GATA2 (Addgene #125600)14 o con un pool dei tre fattori, più FUW-M2rtTA a 1:1 rapporto. Utilizzare il virus totale di 10-20 ll (tF individuale - M2rtTA o tre TF - M2rtTA). Incubare le cellule durante la notte a 37 gradi centigradi, 5% DI CO2.
NOT: Si consiglia di utilizzare dodici piastre 6-pono per condizione (per ogni singolo TF e le tre TF combinate). - Rimuovere i lentivirus e aggiungere il DMEM completo 16 h dopo la prima trasduzione. Lascia che le cellule si riprendano per 6/8 h.
- Trasduci le cellule una seconda volta con la stessa quantità di virus per condizione e incuba a 37 gradi centigradi, 5% di CO2.
- Il giorno successivo rimuovere i virus e aggiungere DMEM completo. Incubare a 37 gradi centigradi, 5% CO2 per 24 h.
- Ri-piastra ogni pozzo in un 0.1% gelatina rivestito tessuto tessuto rivestito 100 mm piatto con DMEM completo ad un volume finale di 10 mL per piatto. Questo rappresenta approssimativamente un passaggio 1:6.
- Consentire alle cellule di crescere per 6 giorni a 37 , 5% CO2.
- Il giorno 6 dopo la ri-placcatura, mezzo aspirato e aggiungere dMEM completo con 1 :g/mL DOX. Incubare le cellule a 37 gradi centigradi, il 5% di CO2 per 2 giorni.
- Raccogliere i fibroblasti e analizzare i siti di legame genomico dei tre TF trasmessi singolarmente o in combinazione, da ChIP-seq 2 giorni dopo il completamento di DOX14.
NOT: Gli ultimi settantadue piatti da 100 mm conterranno tra 20-50 x 106 celle, sufficienti per eseguire esperimenti ChIP-seq e repliche.
Risultati
Una rappresentazione schematica dell'approccio di riprogrammazione mediante HDF è illustrata nella Figura 1A. I fibroblasti vengono acquisiti da fonti commerciali o raccolti da donatori umani e espansi in vitro prima della riprogrammazione. Dopo la placcatura, le cellule vengono trasdotte due volte con lentivirus GATA2, GFI1B e FOS (e M2rtTA) e la doxycycline viene aggiunta al giorno 0 della riprogrammazione. Il giorno 2, le cellule sono divise e placcate in mezzo ematopoietico fino al giorno 25 della coltura. Le cellule riprogrammate possono essere generate in diversi momenti temporali per più applicazioni, tra cui il trapianto in topi immunocompromessi, il sequenziamento di RNA a singola cellula (scRNA-seq) di popolazioni di cellule purificate (giorno 2 non ordinato, giorno 15 CD49f giorno 25 CD49f-CD34- cellule), così come l'analisi di microscopia e citometria di flusso per i marcatori di superficie cellulare CD49f, CD34, CD9 e CD143. Le trame di citometria rappresentativa mostrano il 17% delle celle riprogrammate che esprimono sia CD49f che CD9 (Figura 1B, pannello sinistro),dopo 25 giorni di riprogrammazione. La maggior parte delle cellule a doppio positivo esprimono il CD143 (-86%), e una piccola popolazione esprime CD34 (0,9%), suggerendo una dinamica induzione del destino emogenico. Questi marcatori non sono attivati negli HDF traslati M2rtTA coltivati per 25 giorni (Figura 1B, pannello destro). Le immagini dell'immunofluorescenza confermano l'espressione di CD9 e CD143 nelle cellule aderenti e rotonde, morfologicamente distinte dai fibroblasti negativi per questi marcatori (Figura 1C). Le colonie emogeniche umane esprimono anche CD49f e CD3414. L'analisi ScRNA-seq degli HDF, delle cellule non ordinate del secondo giorno e delle cellule riprogrammate purificate al giorno 15(CD49f-CD34-) e del giorno 25 (CD49f)mostrano un aumento graduale nell'espressione CD49f, CD9 e CD143 dal giorno 2 al giorno 25. Le cellule positive CD49f e CD9 appaiono per prime durante il processo di riprogrammazione, tra il giorno 2 e il 15, indicando che queste molecole possono rappresentare marcatori dell'emogenesi umana precoce. L'espressione CD143 inizia a essere rilevata al giorno 15 e le celle esprimibili CD34 vengono rilevate solo in momenti successivi (giorno 25). Le cellule del sangue del cordone ombelicale (UCB) sono state utilizzate come riferimento(Figura 1D).
La figura 2descrive un protocollo modificato per generare un numero sufficiente di cellule per l'analisi ChIP-seq nelle fasi iniziali della riprogrammazione emogenica (giorno 2). In primo luogo, gli HDF sono placcati ad una densità due volte superiore rispetto al protocollo standard (300.000 cellule contro 150.000 celle per piastra). Dopo la trasduzione, ogni pozzo viene ri-placcato in un piatto da 100 mm permettendo alle cellule di espandersi per 6 giorni prima di integrare il mezzo con DOX. Le celle vengono analizzate 2 giorni dopo l'aggiunta di DOX e conseguente espressione TF. La figura 2B mostra i profili del browser del genoma del binding GATA2 alle aree regolatorie genomiche di ITGA6 e ACE quando le cellule sono co-transdotte con i tre fattori (3TF) o GATA2 singolarmente. GATA2 si lega anche alle regioni di cromatina aperte dei geni CD9 e CD34 14.
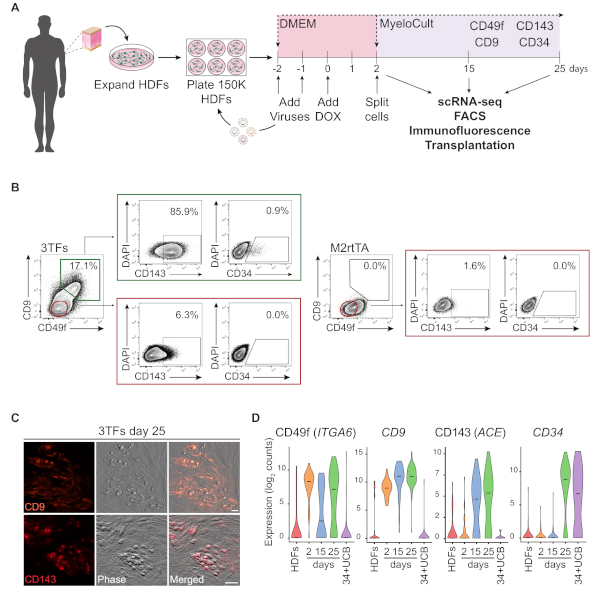
Figura 1: Induzione del destino emogenico nei fibroblasti dermici umani. (A) Strategia sperimentale per la riprogrammazione emogenica dei fibroblasti dermici umani (HDF). I fibroblasti da biopsie punzonabili cutanee vengono raccolti dai donatori, espansi e trasdotti con GATA2, GFI1B, FOS e lentivirus M2rtTA. Dox (DoX) viene aggiunto alla coltura al giorno 0 di riprogrammazione e le cellule vengono analizzate in diversi punti temporali fino al giorno 25. scRNA-seq, sequenziamento dell'RNA a cella singola. FACS, Ordinamento cellulare attivato dalla fluorescenza. (B) Strategia di Gating utilizzata per valutare l'espressione di marcatori emogeni/ematopoietici per citometria di flusso al giorno 25 dopo la trasduzione con i tre fattori di trascrizione (3TF). Le trame citometriche mostrano una percentuale di cellule doppie positivi per CD49f e CD9, gated nella popolazione di cellule vive (DAPI-negativo). All'interno della doppia popolazione positiva, viene mostrata l'espressione di CD143 e CD34. Gli HDF trasdotti solo con il virus M2rtTA nelle stesse condizioni di coltura vengono utilizzati come controllo. (C) Immagini immunofluorescenze del giorno 25 colonie riprogrammate confermando l'espressione di CD9 (pannello superiore) e CD143 (pannello inferiore). Le cellule sono state macchiate di anticorpi (Table of Materials) diluite 1:100 in PBS/2% FBS con siero di topo, incubate 20 min a 37 gradi centigradi, 5% CO2,lavate tre volte e immagini in PBS/2% FBS. Fase, contrasto fase-gradiente. Barre di scala - 50 scRNA-seq analisi di 253 cellule in punti temporali diversi. L'espressione di ITGA6, CD9, ACE e CD34 viene attivata durante la riprogrammazione. Le celle vengono raccolte il giorno 2 (non ordinato), il giorno 15 (CD49f-CD34-) e il giorno 25 (CD49f,CD34). Le cellule di FSE e CD34e sangue del cordone ombelicale (34-UCB) sono usate come riferimenti. Fare clic qui per visualizzare una versione più grande di questa figura.
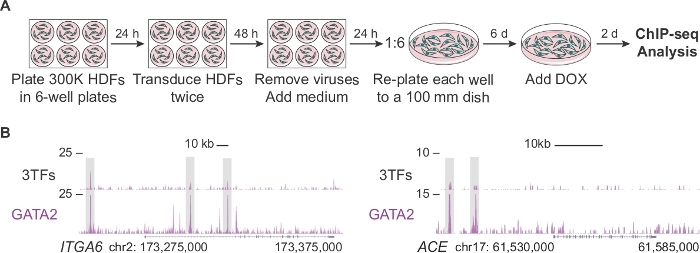
Figura 2: Espansione dei fibroblasti dermici umani per l'analisi ChIP-seq. (A) Strategia sperimentale raffigurante un protocollo modificato per generare un numero elevato di fibroblasti dermici umani tradotti (HDF) trasdotti per ChIP-seq al secondo giorno della riprogrammazione. 300.000 cellule sono placcate in piastre a 6 pozzetti e trasdotte due volte con fattori individuali (pFUW-tetO-FOS, pLV-tetO-HA-GFI1B o pFUW-tetO-3xFLAG-GATA2) o una combinazione dei tre fattori (più M2rtTA). Dopo la rimozione dei virus, i fibroblasti vengono espansi per sei giorni in piatti da 100 mm. Doxycycline (DOX) viene aggiunto al giorno 0 e le cellule vengono raccolte due giorni dopo l'aggiunta di DOX. (B) I profili del browser genoma che evidenziano i siti di legame GATA2 (caselle grigie) presso ITGA6 e ACE loci due giorni dopo la trasduzione con i tre fattori di trascrizione (3TF) o con GATA2 da solo. Il numero totale di letture mappate è rappresentato sull'asse y. Fare clic qui per visualizzare una versione più grande di questa figura.
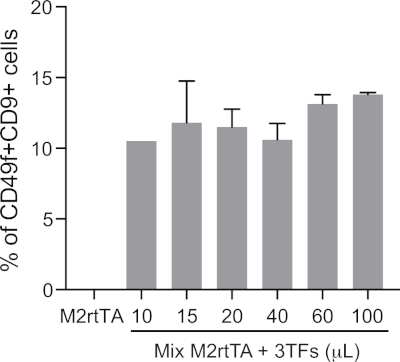
Figura supplementare 1: Definizione di un volume lentivirale ottimizzato per una riprogrammazione emogenica efficiente. L'aumento dei volumi di particelle lentivirali concentrate (3TF: GATA2, GFI1B e FOS) vengono utilizzate per trasdurre i fibroblasti dermici umani (HDF), insieme a M2rtTA con un rapporto di 1:1, seguendo i passaggi 4.5.4.12 del protocollo. Le cellule riprogrammate vengono analizzate al giorno 25 per definire un volume ottimale di trasduzione per la riprogrammazione emogenica, data dalla percentuale di CD49f-CD9- cellule recintate in cellule vive (DAPI-negative). La vitalità cellulare può essere valutata quantificando il numero assoluto di cellule vive al giorno 25. Gli HdF trasdotti con M2rtTA (100) vengono utilizzati come controllo negativo. Fare clic qui per visualizzare una versione più grande di questa figura.

Figura supplementare 2: Cambiamenti della morfologia durante la riprogrammazione emogenica dei fibroblasti dermici umani. Le colture umane del fibroblasto dermico (HDF) sono immagini al giorno della prima trasduzione (giorno -2), quando DOX viene aggiunto alle culture (giorno 0), due giorni (giorno 2) e quindici giorni (giorno 15) dopo il completamento doX e al punto finale dell'esperimento (giorno 25). Le colonie emogeniche nei giorni 15 e 25 sono evidenziate. Barre di scala : 100 m. Fare clic qui per visualizzare una versione più grande di questa figura.
Discussione
In questo articolo, viene descritto un metodo per generare cellule progenitrici ematopoietiche direttamente da fibroblasti umani, che passano attraverso un intermedio cellulare HP, in modo simile agli HSC definitivi14.
La produzione di particelle lentivirali che codificano GATA2, GFI1B e FOS è stata preferita rispetto alla produzione individuale, poiché nelle nostre mani si traduce in una maggiore efficienza di riprogrammazione (dati inediti). I lentivirus, in quanto membri della famiglia Retroviridae, normalmente contengono due copie di RNA positivo a filamento singolo19. La maggiore efficienza di riprogrammazione può essere dovuta all'imballaggio di due diversi transgeni nella stessa particella lentivirale, con conseguente aumento del numero di cellule co-transdotte con i tre fattori di trascrizione. Per garantire il successo di questo protocollo, è necessario trasdurre gli HDF con una quantità adeguata di virus a seconda del passaggio cellulare per ottenere un equilibrio ottimale tra efficienza di riprogrammazione e vitalità cellulare, come raccomandato nel passaggio 4.6. Inoltre, possono essere utilizzati nuovi virus non concentrati. Si raccomanda di trasdurare le cellule con 0,5-3 mL di piscina 3TFs e M2rtTA. Inoltre, la densità delle celle deve essere regolata in base all'applicazione. 150 000 HDF per piastra a 6 pozzetti (passaggio 4.4) hanno fornito la densità ottimale per eseguire l'analisi FACS, il trapianto e la citometria di flusso delle cellule riprogrammate. Per gli esperimenti ChIP-seq, sono state richieste più cellule fin dall'inizio (passaggio 5.1). È importante controllare regolarmente le cellule per i cambiamenti morfologici e sostituire il mezzo ematopoietico due volte alla settimana per sostenere l'emergere di cellule ematopoietiche indotte. L'aggiunta di citochine ematopoietiche o la cocoltura negli strati feeder può aumentare l'efficienza di riprogrammazione.
Con questo metodo, possiamo identificare nuovi marcatori ematopoietici che vengono espressi dinamicamente durante la riprogrammazione emogenica. Il CD9, che è stato dimostrato essere up-regolato in cellule riprogrammate al livello trascrizionale14,è rapidamente espresso sulla superficie cellulare nelle fasi iniziali della riprogrammazione insieme a CD49f e CD143, servendo come un nuovo marcatore dei precursori umani dell'HSC. Dimostriamo inoltre che ITGA6 e ACE sono obiettivi diretti di GATA2 durante le fasi iniziali della riprogrammazione emogenica, oltre a CD9 e CD3414, fornendo un collegamento meccanicistico diretto tra emogenici umani fenotipo precursore e GATA2.
Un vantaggio di questo sistema risiede nell'uso di colture fibroblaste relativamente omogenee. Mentre i PSC sono facilmente espandibili e mantenuti in vitro,i protocolli di differenziazione generano popolazioni eterogenee che includono progenitori ematopoietici, che ne innevano male5,6,7. Inoltre, c'è il rischio di tumorigenesi durante il trapianto di HSPC derivati da PSC, poiché i PSC indifferenziati possono ancora rimanere in coltura anche dopo l'impiego di protocolli di differenziazione. In alternativa ai fibroblasti, la riprogrammazione diretta agli HSC è stata applicata ai progenitori commessi nel sangue20 e alle cellule endoteliali21. Tuttavia, a partire dalle cellule progenitrici a sbarramento del sangue ostacola l'applicazione terapeutica degli HSC risultanti se il paziente porta con sé mutazioni che colpiscono la popolazione ematopoietica staminale/progenitrice22. Nel caso delle cellule endoteliali, queste sono più difficili da ottenere rispetto ai fibroblasti e costituiscono una popolazione cellulare molto eterogenea in termini di fenotipo, funzione e struttura, che dipendono dall'organo23. Altri studi sono riusciti a riprogrammare i fibroblasti di topi in progenitori ematopoietici ingenati24,25 ma, finora, nessun altro protocollo descrive la generazione di cellule simili a HSPC dai fibroblasti umani.
Questo approccio, unito all'inibizione farmacologica, al gene knock-out o al knock-down, consente di definire singoli o una combinazione di fattori necessari per indurre direttamente gli HSC umani. Le tecnologie CRISPR-Cas9 negli HDF prima della riprogrammazione, rappresentano un'impresa entusiasmante per la definizione di nuovi regolatori dell'ematopoiesi definitiva umana. In futuro, la riprogrammazione di tipi di cellule umane non correlate al sangue come i fibroblasti servirà come piattaforma per generare cellule progenitrici ematopoietiche sane per applicazioni cliniche.
Divulgazioni
Gli autori non hanno nulla da rivelare.
Riconoscimenti
La fondazione Knut e Alice Wallenberg, la Facoltà di Medicina presso l'Università di Lund e la Regione di Skàne sono riconosciuti per un generoso sostegno finanziario. Questo lavoro è stato sostenuto da una sovvenzione di Olle Engkvists Stiftelse (194-0694 a Filipe Pereira) e da borse di dottorato da Fundao para a Ciància e Tecnologia (PTDC/BIM-MED/0075/2014 a Filipe Pereira, e SFRH/BD/135725/2018 e SFRH/BD/51968/2012 a Rita Alves e Andreia Gomes). Questo studio è stato sostenuto anche da fondi di NIH e NYSTEM (1R01HL119404 e C32597GG a Kateri A. Moore).
Materiali
| Name | Company | Catalog Number | Comments |
| 0.45 μm low-protein binding filter, 150 mL Bottle Top Vacuum Filter | Corning | #430625 | |
| 2-Mercaptoethanol | Sigma-Aldrich | #M6250 | |
| Alexa Fluor 488 anti-human CD34 clone 581 | BioLegend | #343518 | |
| BD Pharmingen APC Mouse Anti-Human Angiotensin Converting Enzyme (CD143) clone BB9 | BD Biosciences | #557929 | |
| BES buffered saline | Sigma-Aldrich | #14280 | |
| Calcium chloride (CaCl2) | Sigma-Aldrich | #449709 | |
| Centrifugal filter unit, Amicon Ultra-15 Centrifugal Filter Unit | Sigma-Aldrich | #UFC903096 | |
| Dissociation solution, TrypLE Express Enzyme (1x) no phenol red | Gibco | #12604-021 | |
| Doxycycline hyclate (DOX) | Sigma-Aldrich | #D9891 | |
| eBioscience CD49f (Integrin alpha 6) Monoclonal Antibody (eBioGoH3 (GoH3)), PE-Cyanine7 | Invitrogen | #25-0495-82 | |
| FUW-M2rtTA | Addgene | #20342 | |
| Gelatin from Porcine Skin Type A | Sigma-Aldrich | #G1890 | |
| Gibco L-Glutamine (200 mM) | ThermoFisher Scientific | #25030-024 | |
| Gibco MEM Non-Essential Amino Acids Solution (100x) | ThermoFisher Scientific | #11140-035 | |
| Hematopoietic medium, MyeloCult H5100 | STEMCELL Technologies | #05150 | |
| Hexadimethrine bromide (polybrene) | Sigma-Aldrich | #H9268 | |
| Human Dermal Fibroblasts (HDFs) | ScienCell | #2320 | |
| HyClone Dulbecco's Modified Eagles Medium (DMEM) | GE Healthcare | #SH30243.01 | |
| HyClone Fetal Bovine Serum (FBS) | GE Healthcare | #SV30160.03 | |
| HyClone Penicillin Streptomycin 100x Solution (Pen/Strep) | GE Healthcare | #SV30010 | |
| HyClone Phosphate Buffered Saline solution (PBS) | GE Healthcare | #SH30256.01 | |
| Hydrocortisone | STEMCELL Technologies | #7904 | |
| Mouse serum | Sigma-Aldrich | #M5905 | |
| PE anti-human CD9 Antibody clone HI9a | BioLegend | #312105 | |
| pFUW-tetO-3xFLAG-GATA2 | Addgene | #125600 | |
| pFUW-tetO-FOS | Addgene | #125598 | |
| pFUW-tetO-GATA2 | Addgene | #125028 | |
| pFUW-tetO-GFI1B | Addgene | #125597 | |
| pLV-tetO-HA-GFI1B | Addgene | #125599 | |
| pMD2.G | Addgene | #12259 | |
| psPAX2 | Addgene | #12260 |
Riferimenti
- Ivanovs, A., et al. Highly potent human hematopoietic stem cells first emerge in the intraembryonic aorta-gonad-mesonephros region. Journal of Experimental Medicine. 208, 2417-2427 (2011).
- Tavian, M., Biasch, K., Sinka, L., Vallet, J., Péault, B. Embryonic origin of human hematopoiesis. International Journal of Developmental Biology. 1065, 1061-1065 (2010).
- Medvinsky, A., Rybtsov, S., Taoudi, S. Embryonic origin of the adult hematopoietic system: advances and questions. Development. 138, 1017-1031 (2011).
- Ivanovs, A., et al. Human haematopoietic stem cell development: from the embryo to the dish. Development. 144, 2323-2337 (2017).
- Daniel, M. G., Pereira, C. F., Lemischka, I. R., Moore, K. A. Making a Hematopoietic Stem Cell. Trends in Cell Biology. 26, 202-214 (2016).
- Vo, L., Daley, G. De novo generation of HSCs from somatic and pluripotent stem cell sources. Blood. 125, 2641-2648 (2015).
- Rafii, S., et al. Human ESC-derived hemogenic endothelial cells undergo distinct waves of endothelial to hematopoietic transition. Blood. 121, 770-781 (2013).
- Ebina, W., Rossi, D. J. Transcription factor-mediated reprogramming toward hematopoietic stem cells. EMBO Journal. 34, 694-709 (2015).
- Sugimura, R., et al. Haematopoietic stem and progenitor cells from human pluripotent stem cells. Nature. 545, 432-438 (2017).
- Pereira, C. F., et al. Induction of a Hemogenic Program in Mouse Fibroblasts. Cell Stem Cell. 13, 205-218 (2013).
- Pereira, C. F., et al. Hematopoietic Reprogramming In vitro Informs In Vivo Identification of Hemogenic Precursors to Definitive Hematopoietic Stem Cells. Developmental Cell. 36, 525-539 (2016).
- Notta, F., et al. Isolation of Single Human Hematopoietic Stem Cells Capable of Long-Term Multilineage Engraftment. Science. 333, 218-221 (2011).
- Sinka, L., Biasch, K., Khazaal, I., Péault, B., Tavian, M. Angiotensin-converting enzyme (CD143) specifies emerging lympho-hematopoietic progenitors in the human embryo. Blood. 119, 3712-3724 (2012).
- Gomes, A. M., et al. Cooperative Transcription Factor Induction Mediates Hemogenic Reprogramming. Cell Reports. 25, 2821-2835 (2018).
- Karlsson, G., et al. Report The Tetraspanin CD9 Affords High-Purity Capture of All Murine Hematopoietic Stem Cells. Cell Reports. 4, 642-648 (2013).
- Leung, K. T., et al. The tetraspanin CD9 regulates migration, adhesion, and homing of human cord blood CD34+ hematopoietic stem and progenitor cells. Blood. 117, 1840-1851 (2011).
- Hockemeyer, D., et al. A drug-inducible system for direct reprogramming of human somatic cells to pluripotency. Cell Stem Cell. 3, 346-353 (2008).
- Kutner, R. H., Zhang, X., Reiser, J. Production concentration and titration of pseudotyped HIV-1-based lentiviral vectors. Nature Protocols. 4, 495-505 (2009).
- Suzuki, Y. S., Suzuki, Y., Ke, X. Gene Regulatable Lentiviral Vector System. Viral Gene Therapy. , (2011).
- Riddell, J., et al. Reprogramming committed murine blood cells to induced hematopoietic stem cells with defined factors. Cell. 157, 549-564 (2014).
- Lis, R., et al. Conversion of adult endothelium to immunocompetent haematopoietic stem cells. Nature. 545, 439-445 (2017).
- Pereira, C., Lemischka, I. R., Moore, K. From blood to blood’: de-differentiation of hematopoietic progenitors to stem cells. EMBO Journal. 33, 1511-1513 (2014).
- Nolan, D. J., et al. Molecular Signatures of Tissue-Specific Microvascular Endothelial Cell Heterogeneity in Organ Maintenance and Regeneration. Developmental Cell. 26, 204-219 (2013).
- Batta, K., Florkowska, M., Kouskoff, V., Lacaud, G. Direct Reprogramming of Murine Fibroblasts to Hematopoietic Progenitor Cells. Cell Reports. 9, 1871 (2014).
- Cheng, H., et al. Reprogramming mouse fibroblasts into engraftable myeloerythroid and lymphoid progenitors. Nature Communications. 7, 1-15 (2016).
Erratum
Formal Correction: Erratum: Hemogenic Reprogramming of Human Fibroblasts by Enforced Expression of Transcription Factors
Posted by JoVE Editors on 12/03/2019. Citeable Link.
An erratum was issued for: Hemogenic Reprogramming of Human Fibroblasts by Enforced Expression of Transcription Factors. The author affiliations were updated.
The second author affiliation was updated from:
2Wallenberg Center for Molecular, Lund University
to:
2Wallenberg Center for Molecular Medicine, Lund University
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon