Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Größenbestimmung und phänotypische Analyse extrazellulärer Vesikel im Urin mittels Durchflusszytometrie
In diesem Artikel
Zusammenfassung
Dieses Protokoll beschreibt ein Verfahren zur Isolierung von extrazellulären Vesikeln (uEVs) im Urin von gesunden humanen Spendern und deren phänotypische Charakterisierung durch die Größe und Oberflächenmarkerexpression mittels Durchflusszytometrie.
Zusammenfassung
Extrazelluläre Vesikel, EVs, sind ein heterogener Komplex von Lipidmembranen, die von jedem Zelltyp in jeder Flüssigkeit wie dem Urin sezerniert werden. EVs können verschiedene Größen haben, die von einem Durchmesser von 40 bis 100 nm (z. B. in Exosomen) bis zu 100 bis 1000 nm (z. B. in Mikrovesikel) reichen. Sie können auch verschiedene Moleküle enthalten, die als Biomarker für die Prognose und Diagnose vieler Krankheiten verwendet werden können. Es wurden viele Techniken entwickelt, um diese Vesikel zu charakterisieren. Eine davon ist die Durchflusszytometrie. Es gibt jedoch keine Berichte, die zeigen, wie die Konzentration von Elektrofahrzeugen quantifiziert und nach Größe sowie nach Erkennung von Biomarkern unterschieden werden kann. Ziel dieser Arbeit ist es, ein Verfahren zur Isolierung, Quantifizierung und Phänotypisierung von extrazellulären Vesikeln (uEVs) im Urin zu beschreiben, wobei ein konventionelles Zytometer für die Analyse verwendet wird, ohne dass dessen Konfiguration verändert wird. Zu den Einschränkungen der Methode gehört die Färbung von maximal vier verschiedenen Biomarkern pro Probe. Die Methode ist auch durch die Menge der in der Probe verfügbaren EVs begrenzt. Trotz dieser Einschränkungen können wir mit diesem Protokoll und seiner anschließenden Analyse mehr Informationen über die Anreicherung von EVs-Markern und die Häufigkeit dieser Vesikel in Urinproben, bei Erkrankungen mit Nieren- und Hirnschäden erhalten.
Einleitung
Bei Säugetieren wird das Blut filtriert, indem es 250 - 300 Mal durch die Nieren fließt. Während dieser Zeit wird Urin gebildet. Die Herstellung dieses Biofluids ist das Ergebnis einer Reihe von Prozessen, darunter glomeruläre Filtration, tubuläre Reabsorption und Sekretion. Stoffwechselendprodukte und Elektrolyte sind die Hauptbestandteile des Urins. Auch andere Nebenprodukte wie Peptide, funktionelle Proteine und extrazelluläre Vesikel (EVs) werden ausgeschieden 1,2,3,4,5,6. Zunächst wurden extrazelluläre Vesikel (uEVs) im Urin in Urinproben von Patienten mit Wasserhaushaltsstörungen identifiziert. Diese Patienten wiesen das Vorhandensein von Molekülen wie Aquaporin-2 (AQP2) auf, das dann als Biomarker für diese Krankheit verwendet wurde7. Mehrere nachfolgende Studien konzentrierten sich auf die Identifizierung des zellulären Ursprungs von uEVs und beschrieben, dass diese Strukturen von Nierenzellen (Glomerulus, Podozyten usw.) und anderen Zelltypen endothelialer oder leukozytärer Linien sezerniert werden können. Darüber hinaus kann die Anzahl und Molekülanreicherung in uEVs mit dem Status vieler Krankheiten und Störungen korrelieren 8,9,10,11,12,13,14.
Insgesamt bilden EVs eine sehr heterogene Familie von Partikeln, die von Lipiddoppelschichten umschlossen sind und von Zellen durch passive oder aktive Mechanismen in verschiedene Flüssigkeiten freigesetzt werden. Abhängig von ihrer Herkunft können EVs als Endosomen-Exosomen oder als von Plasmamembranen abgeleitete Mikrovesikel/Mikropartikel klassifiziert werden. Dieses Klassifizierungskriterium kann jedoch nur angewendet werden, wenn die Biogenese der Partikel direkt beobachtet wird. Daher wurden andere nicht-triviale Kriterien, einschließlich physikalischer, biochemischer und zellulärer Herkunft, von mehreren Forschern auf diesem Gebiet befürwortet 15,16,17. Abhängig von der Art des analysierten Isolats wurden unterschiedliche Analysetechniken für die Charakterisierung von EVs vorgeschlagen. Basierend auf der Anreicherung von großen (≥100 nm) oder kleinen (≤100 nm) EVs wird beispielsweise eine Quantifizierung mittels Durchflusszytometrie bzw. Nanopartikel-Tracking vorgeschlagen18.
Heutzutage ist die Verwendung von EVs als Biomarker für viele Krankheiten relevant geworden, so dass die Suche nach verschiedenen Quellen untersucht wurde. Eine der vielversprechendsten Quellen ist der Urin, da er auf einfache und nicht-invasive Weise gewonnen werden kann. Daher beschreibt dieses Protokoll ein Verfahren zur Isolierung von uEVs durch Differenzialzentrifugation, die Verarbeitung mit Fluorochrom-konjugierten Antikörpern und die nachgeschaltete Analyse unter Verwendung eines herkömmlichen 2-Laser/4-Farben-Zytometers.
Protokoll
Die menschlichen Urinproben wurden von gesunden Freiwilligen entnommen, die eine Einverständniserklärung des Spenders unterschrieben hatten. Diese Verfahren wurden auch von der Forschungsethikkommission des Instituto Nacional de Ciencias Médicas y Nutrición Salvador Zubirán genehmigt.
1. Isolierung extrazellulärer Vesikel im Urin
HINWEIS: Das Isolationsprotokoll von uEVs wurde gegenüber Ref.19 geändert. Abbildung 1 zeigt die Darstellung des Protokolls zur Isolierung von uEVs.
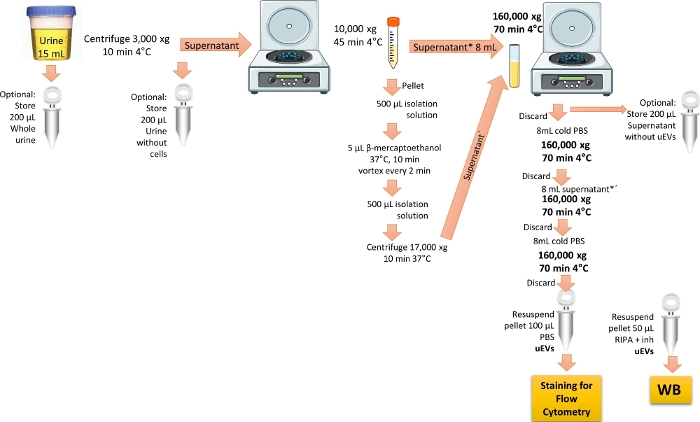
Abbildung 1: Überblick über die Isolierung von uEVs für die durchflusszytometrische Analyse. In diesem Protokoll zentrifugieren Sie zunächst den ersten Urin des Tages, um die Zellen und Ablagerungen zu entfernen. Zentrifugieren Sie dann, um die großen Vesikel zu entfernen, mit einer Behandlung, um das THP-Protein zu entfernen, und führen Sie schließlich eine Ultrazentrifugation durch, um die uEVs mit einer einzigen Wäsche anzureichern und zu erhalten. Die Schritte zum Aufbewahren der Urinfraktionen für die WB-Validierung sind markiert. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
- Verwenden Sie den Urin des ersten Morgens (15 ml) von gesunden Freiwilligen. Zentrifugieren Sie den Urin bei 3.000 x g für 10 min bei 4 °C, um alle Zellen und Ablagerungen zu entfernen.
HINWEIS: Verwenden Sie vorzugsweise frischen Urin; Falls nicht verfügbar, verwenden Sie Urin, der für einen Zeitraum von maximal 3 Monaten bei -20 °C oder für einen Zeitraum von maximal 6 Monaten bei -70 °C gelagert wird. Tauen Sie die Urinprobe auf Eis auf und wirbeln Sie sie kräftig ein. Führen Sie alle Eingriffe auf Eis oder bei 4 °C durch. Ein optionaler Schritt für die Western-Blot-Analyse besteht darin, ein doppeltes Röhrchen herzustellen und 200 μl des Urins des ersten Morgens in einem separaten Röhrchen zu aliquotieren. Lagern Sie die Probe bis zur Verwendung bei -20 °C und kennzeichnen Sie sie als "Vollurin", um sie auf extrazelluläre Vesikelmarker zu überprüfen. - Der in Schritt 1.1 erhaltene Überstand wird in ein neues konisches 15-ml-Zentrifugenröhrchen überführt und anschließend bei 10.000 x g für 45 min bei 4 °C zentrifugiert. Den Überstand in ein 8-ml-Polycarbonat-Ultrazentrifugationsröhrchen umfüllen und auf Eis konservieren.
ANMERKUNG: Optional aliquotieren Sie 200 μl des in Schritt 1.1 erhaltenen Überstands in einem separaten Röhrchen mit der Bezeichnung "Urin ohne Zellen" für die Western-Blot-Analyse.- Entfernung von Tamm-Horsfall-Protein, THP
HINWEIS: THP ist im Urin vorhanden und wird angereichert, wenn eine Person an einer Nierenerkrankung leidet. Es wurde berichtet, dass THP die Ausbeute von uEVs verringert, da es an uEVs binden kann. Um dieses Protein zu entfernen, ist die Verwendung eines Reduktionsmittels erforderlich 3,6.- Bereiten Sie die Isolierlösung vor, indem Sie 250 mM Saccharose mit 10 mM Triethanolamin mischen. Stellen Sie den pH-Wert auf 7,6 ein.
- Das in Schritt 1.2 erhaltene Pellet wird mit 500 μl Isolierlösung gemischt und anschließend 5 μl β-Mercaptoethanol zugegeben.
- Die Pelletmischung bei 37 °C 10 min inkubieren und alle 2 min vortexen. 500 μl Isolierlösung zugeben und anschließend 10 min bei 37 °C bei 17.000 x g zentrifugieren. Sammeln Sie den erhaltenen Überstand (der reduzierte, nicht aggregierte THP plus uEV enthält).
- Entfernung von Tamm-Horsfall-Protein, THP
- Mischen Sie beide reservierten Überstände (aus den Schritten 1.2 und 1.2.1.3) in demselben 8-ml-Polycarbonat-Ultrazentrifugationsröhrchen und zentrifugieren Sie dann 70 Minuten lang bei 4 °C bei 160.000 x g mit einem Ultrazentrifugen-Festwinkelrotor.
HINWEIS: Optional können Sie für die Western-Blot-Analyse 200 μl des oben erhaltenen Überstands aufbewahren und ihn als "Überstand ohne EVs" bezeichnen. Diese Probe dient als Negativkontrolle bei der Suche nach extrazellulären Vesikelmarkern. - Entsorgen Sie den Überstand. Geben Sie eiskaltes 1x PBS in das 8 mL Polycarbonat-Röhrchen und zentrifugieren Sie bei 160.000 x g für 70 min bei 4 °C.
HINWEIS: Achten Sie darauf, das Pellet nicht zu entsorgen, da es die uEVs enthält. Das 1x PBS muss sterilisiert und mit mindestens 0,22 μm Porenspritzenfilter filtriert werden. - Entsorgen Sie den Überstand. Den Rest des Überstands in das 8-ml-Polycarbonat-Röhrchen geben und bei 160.000 x g für 70 min bei 4 °C zentrifugieren.
- Entsorgen Sie den Überstand und fügen Sie dem Pellet 8 ml eiskaltes PBS hinzu, um die uEVs zu waschen.
- Entsorgen Sie den Überstand. Lassen Sie das Pellet trocknen und suspendieren Sie es dann mit 1 mL eiskaltem PBS. Bis zur Verwendung bei -70 °C lagern.
HINWEIS: Wenn Sie eine Western-Blot-Analyse durchführen, resuspendieren Sie das Pellet des Duplikatsrohrs mit 50 μl RIPA-Puffer plus Proteaseinhibitoren. Bis zur Verwendung bei -20 °C lagern.
2. Färbung von uEVs
HINWEIS: Vor dem Färben und Analysieren von uEVs ist es wichtig, mindestens eine der inMISEV2018 18 empfohlenen Methoden durchzuführen, um die ordnungsgemäße Isolierung von uEVs zu überprüfen. hier wird die Western-Blot-Analyse dargestellt. Abbildung 2 zeigt ein repräsentatives Protokoll zur Färbung von uEVs.
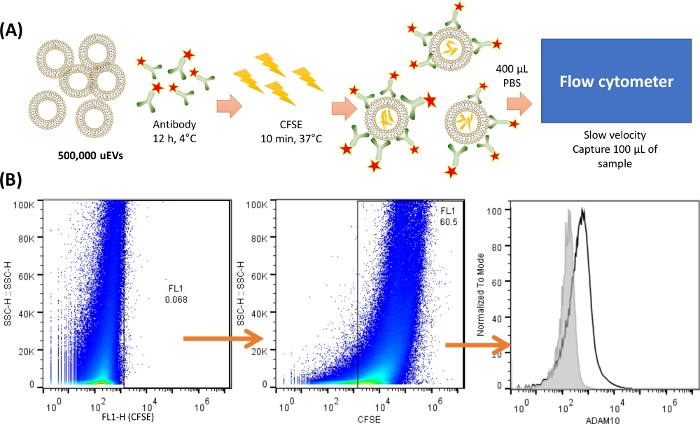
Abbildung 2: Überblick über die Färbung und Erfassung der uEVs im Zytometer. (A) Darstellung der uEV-Färbung. Für 500.000 μEV wurde der Antikörper gemischt und 12 Stunden lang bei 4 °C inkubiert. Dann wurde CFSE zugegeben und bei 37 °C für 10 min inkubiert. Die uEVs enthielten das CFSE im Inneren, und der Antikörper bindet an die Oberfläche des Antigens. 400 μl kaltes PBS wurden verwendet, um 100 μl der Probe mit langsamer Geschwindigkeit im Durchflusszytometer zu resuspendieren und einzufangen. (B) Analysestrategie. Das erste Punktdiagramm (SSC-H VS FL-X) zeigt die Negativkontrolle für uEVs, gefolgt von dem Punktdiagramm, das die Färbung von uEVs mit CFSE zeigt, und schließlich ein Histogramm mit der Antikörperfärbung von uEVs (schwarze Linie), die Negativkontrolle ist in der grauen Linie dargestellt. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
- Berechnen Sie die relative Anzahl der uEVs, indem Sie den Proteingehalt mit einem herkömmlichen kolorimetrischen Proteinassay quantifizieren, wie in der Materialtabelle erwähnt. Führen Sie eine 1:5-Verdünnung der uEVs durch und befolgen Sie die Anweisungen im Datenblatt im Assay-Proteinkit.
- Basierend auf einer zuvor berichteten Formel20 ist 1 μg/ml uEVs-Protein gleich 800.000 uEVs/μl. Stellen Sie sicher, dass 500.000 uEVs in 20 μl eiskaltem PBS vorhanden sind.
- Beschriften Sie die Röhrchen, wie in Tabelle 1 angegeben.
HINWEIS: Die Anzahl der verwendeten Röhrchen hängt von der Anzahl der verwendeten Antikörper ab, die Begrenzung für 2-Laser/4-Farben-Zytometer beträgt maximal 4 Antikörper pro Röhrchen, die verwendet werden können; daher konnten nur FL1, FL2, FL3 und FL4 im Zytometer abgelesen werden, obwohl eine Kompensation erforderlich ist. Verwenden Sie für diese Verfahren 1,5-ml-Mikrozentrifugenröhrchen oder 5-ml-Röhrchen mit rundem Boden für die Durchflusszytometrie. Die benötigten Rohre sind in Tabelle 1 dargestellt. Die Röhrchen 4 und 5 bestehen aus einem Cocktail aus allen Antikörpern, die in diesem Protokoll verwendet werden sollen. Als Beispiel werden zwei Problemröhren (8 und 9) genannt; Daher muss dieser Satz von Steuerelementen die Kombination haben, die in jedem Rohr verwendet werden soll.
| Röhre 1. | Megamix FSC Perlen | |
| Röhre 2. | PBS | |
| Röhre 3. | PBS mit CFSE | |
| Röhre 4. | PBS mit allen Antikörpern von Problem 1 | |
| Röhre 5. | PBS mit allen Antikörpern von Problem 2 | |
| Röhre 6. | Kontrolle der Autofluoreszenz | uEVs ohne Reagenz, nur in PBS. |
| Röhre 7. | #uEVs | uEVs mit CFSE |
| Röhre 8. | Aufgabe 1 | uEVs mit CD37 FITC, CD53 PE, ADAM10 APC |
| Röhre 9. | Aufgabe 2 | uEVs mit CD9 FITC, TSPAN33 APC |
Tabelle 1: Etikettierung der Tuben. Beispiel, das zeigt, wie die Tuben beschriftet werden. Die ersten Röhren sind alles, was für die Steuerung benötigt wird. Die Röhrchen mit den Antikörper-Fluorochromen hängen von der Färbung ab.
- Geben Sie 20 μl PBS mit 500.000 μV in die markierten Röhrchen.
- Fügen Sie die Antikörper hinzu, die wie in Tabelle 1 angegeben sind und zuvor titriert wurden. Über Nacht bei 4 °C inkubieren.
HINWEIS: Vor der Färbung wird empfohlen, die Antikörper mindestens 5 Minuten lang bei 4 °C bei voller Geschwindigkeit zu zentrifugieren, um die Aggregate zu verhindern. Die hier verwendeten Antikörper sind ein Beispiel für Proteine, die in den uEVs vorhanden sind, und gehören zu einem unabhängigen Manuskript, das derzeit in Vorbereitung ist. - 0,4 μl Carboxyfluorescein-Succinimidylester (CFSE) [5 nM] in das Röhrchen Nr. 3 geben. 10 min bei 37 °C inkubieren.
HINWEIS: CFSE ist ein Farbstoff, der verwendet wird, um alle in einer Probe vorhandenen EVs zu färben und zwischen dem Hintergrundrauschen zu unterscheiden, wenn eine Durchflusszytometeranalyse durchgeführt wird. Weitere Informationen finden Sie im Abschnitt Diskussion . - Geben Sie 400 μl eiskaltes 1x PBS in alle Röhrchen.
- Halten Sie alle Röhrchen bei 4 °C.
3. Erfassung von uEVs mit einem herkömmlichen Zytometer
HINWEIS: Anweisungen für die Verwendung des Durchflusszytometers (siehe Materialtabelle) sind hier beschrieben.
- Führen Sie die Qualitätskalibrierung des Zytometers mit den 6- und 8-Peak-Kügelchen durch.
HINWEIS: Der %CV des letzten Peaks muss einen Wert unter 6 haben; Der Zytometertechniker kümmert sich darum. - Öffnen Sie die Durchflusszytometer-Software.
HINWEIS: Sobald die Software geöffnet ist, wird ein neues Experiment geöffnet, das ein Screening mit einer Vorlage für 96 Proben, den Parametern für die Ausführung und einem "leeren" Abschnitt zum Erstellen von Punktdiagrammen oder Histogrammen zeigt. - Passen Sie die Parameter auf dem Bildschirm der laufenden Software an: 100 μl der zu erfassenden Probe, langsamer Betrieb, stellen Sie den Schwellenwert auf 10.000 für FSC-H und 2.000 für SSC-H ein.
HINWEIS: Der hier empfohlene Schwellenwert ist ein Beispiel. Es ist erforderlich, den Schwellenwert auf der Grundlage der analysierten Proben festzulegen. - Laden Sie das Rohr Nummer 1 (Megamix-Rohr), wie in Tabelle 1 angegeben. Fange die Perlen ein. Erstellen Sie zwei Punktdiagramme im "leeren" Teil des Softwarebildschirms, indem Sie auf die Option Punktdiagramm klicken. Erstellen Sie als Bereich A und B der Abbildung 3.
HINWEIS: Die fluoreszierenden Megamix-Kügelchen (Kügelchen, die zur Abgrenzung der Größen 0,1, 0,3, 0,5 und 0,9 μm und zur Erstellung der Vorlage für die Größen verwendet werden) können mit dem FL1 (FITC)-Detektor analysiert werden. Alle Punktdiagramme und Histogramme müssen in Höhenwerten angezeigt werden. Zeichnen Sie so viele Ereignisse wie möglich auf; Durch Änderungen im Arbeitsblatt werden die Daten nicht geändert.
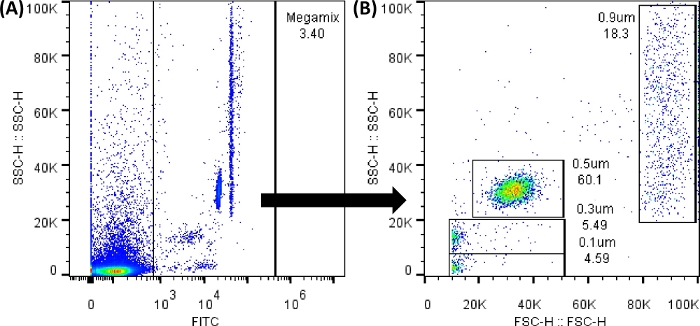
Abbildung 3: Megamix FSC-Perlen-Punktdiagramme. Die gezeigten Punktdiagramme wurden mit der Durchflusszytometer-Software erstellt. Im Durchflusszytometer ist das Bild sehr ähnlich. (A) Der erste Punktplot, der generiert wird, um die Perlen auszuwählen, um Hintergrundgeräusche zu vermeiden. (SSC-H GEGEN FL1-H). (B) Das Punktdiagramm, das durch die Auswahl des vorherigen Gates erzeugt wird und die verschiedenen Größen der Perlen zeigt. (SSC-H GEGEN FSC-H). Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
- Erstellen Sie im "leeren" Bereich des Software-Bildschirms ein Punktdiagramm und ein Histogramm für jedes verwendete Fluorochrom. Verwenden Sie den Algorithmus in der ergänzenden Abbildung 1 als Beispiel. Klicken Sie dazu für jedes Fluorochrom auf die Option Punktdiagramm oder Histogramm .
HINWEIS: Hintergrundgeräusche sind immer vorhanden, wie in Abbildung 3A gezeigt. Alle Punkte zwischen 0 und 1.000 von FL1 (x-Achse) befinden sich außerhalb des erstellten Gates. Daher ist es wichtig, alle Kontrollröhrchen zu verwenden, um angemessene Ergebnisse zu erzielen. Spülen Sie die Mantelflüssigkeit zwischen jeder Schlauchbelastung zurück und führen Sie einen Verstopfungsauflösungszyklus durch. Schütteln Sie jedes Rohr vor dem Laden vorsichtig. Nachdem Sie fünf aufeinanderfolgende Röhrchen geladen haben, lassen Sie 100 μl PBS 1x laufen. - Laden Sie die Rohre Nr. 2 und 5, um die Abschaltwerte (negativ) einzustellen. Wählen Sie dazu das Symbol Rechteck-Gate (unter dem erstellten Punktdiagramm) oder das Symbol Line Gate (unter dem erstellten Histogramm) aus. und platzieren Sie es an einem Ort, an dem kein Signal vorhanden ist, um Punktdiagramme und Histogramme zu erhalten, wie sie in der ergänzenden Abbildung 1 gezeigt sind.
HINWEIS: Bei allen diesen Röhren muss der positive Bereich nicht weiter als 0,70 % platziert werden, da es keine "Fluoreszenz" von mehr als 103 (1.500) gibt. Wenn eine Fluoreszenz weiter als dieser Bereich vorhanden ist, verdünnen Sie die verwendeten Reagenzien. Daher ist es wichtig, die Reagenzien vor den endgültigen Messungen zu titern. - Laden Sie die Röhre Nummer 6 (Autofluoreszenzröhre), um die negativen Bereiche für die Probe einzustellen. Wählen Sie dazu das Symbol Rechteck-Gate (unter dem erstellten Punktdiagramm) oder das Symbol Line Gate (unter dem erstellten Histogramm) aus. Platzieren Sie es an einem Ort, an dem kein Signal vorhanden ist.
- Laden Sie die nächsten Rohre (Rohre 7 bis 9, siehe Tabelle 1).
- Speichern Sie das Experiment.
HINWEIS: Für die hier verwendete Zytometer-Software bezieht sich der Befehl Save Experiment auf das Speichern der generierten Daten. Mit anderen Worten, alle Röhrchen, die vom Durchflusszytometer erfasst werden, und die für jedes Röhrchen generierten Daten müssen gespeichert werden. Dazu ist es notwendig, auf die Schaltfläche Experiment speichern zu klicken. - Exportieren Sie Daten als FSC-Dateien.
4. Analyse der Daten mit einer Durchflusszytometer-Software.
HINWEIS: Anweisungen zur Verwendung der Durchflusszytometer-Software, die in der Materialtabelle dargestellt ist, werden in diesem Abschnitt beschrieben. Abbildung 4 zeigt den Arbeitsbereich mit den Schritten zum Erstellen der Größengates.
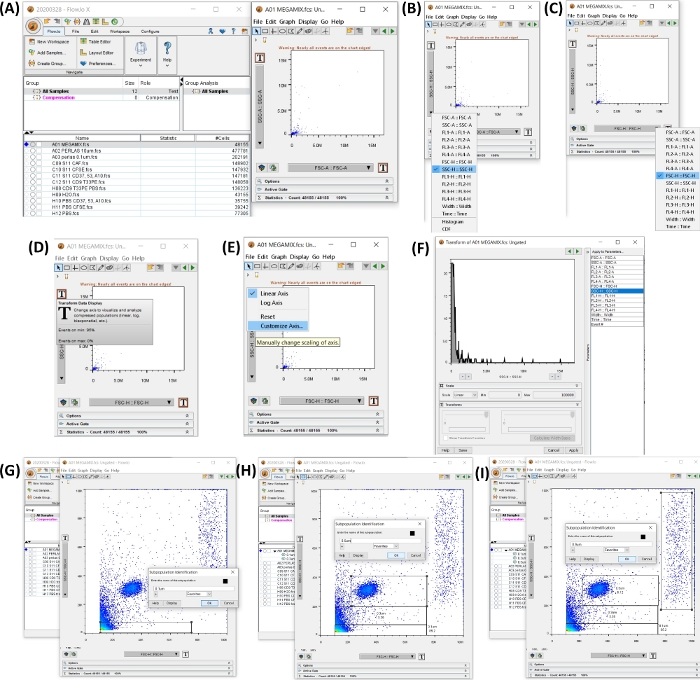
Abbildung 4: Arbeitsbereich mit allen Schritten, um mit der Analyse der Daten zu beginnen. Alle Bilder wurden durch Siebdruck des Arbeitsbereichs erzeugt. (A) Arbeitsbereich, der mit den hinzugefügten Beispieldaten erstellt wurde (links), Punktdiagramm, das durch die Auswahl des Rohrs 1 erzeugt wurde, FSC-Kügelchen, (rechts). (B,C) zeigen die Modifikation der Achse, um SSC-H und FSC-H zu haben. (D-F) zeigen eine schrittweise Anpassung beider Achsen. (G-I) zeigen die Auswahl und Generierung der verschiedenen Perlengrößen. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
- Öffnen Sie die Durchflusszytometer-Software. Fügen Sie die Samples hinzu, indem Sie auf die Schaltfläche Add Samples klicken , und wählen Sie die exportierten FCS-Dateien aus.
- Klicken Sie auf die Megamix-Röhre , um ein Punktdiagramm zu öffnen: SSC-H (y-Achse) VS FSC-H (X-Achse). Stellen Sie beide Achsen so ein, dass sie 0 bis 100.000 anzeigen, indem Sie auf die Schaltfläche T (Datenanzeige transformieren) klicken, klicken Sie auf Achse anpassen, klicken Sie auf SSC-H oder FSSC-H und ändern Sie den Skalenwert auf min 0 und max 100.000 (siehe Abbildung 4A-F).
- Legen Sie die Gates für 0,1, 0,3, 0,5 und 0,9 μm in dem in Schritt 4.2 generierten Punktdiagramm fest. Wählen Sie die Schaltfläche Rechteckiges Gate aus, um ein Gate zu erstellen, wie in Abbildung 4G-I dargestellt.
HINWEIS: Abbildung 5 zeigt den Arbeitsbereich mit den Schritten zum Erstellen der positiven Bereiche und zum Ermitteln der mittleren Intensität für das Fluorochrom.

Abbildung 5: Arbeitsbereich zur Analyse der erhaltenen Daten. Alle Bilder wurden durch den Siebdruck des Arbeitsbereichs erzeugt. (A) Arbeitsbereich, der mit dem Größen-Gate erstellt wurde, das auf alle Samples angewendet wurde. (B) Autofluoreszenzröhre ausgewählt, Punktdiagramm mit dem Größengate und das Histogramm für eine ausgewählte Größe (0,1 μm), verwenden Sie dieses Histogramm, um das positive Gate für jedes Fluorochrom und jede Größe zu erhalten. (C) Arbeitsbereich, der mit den positiven Gates für jedes Fluorochrom und jede Größe erzeugt wird. (D) Arbeitsbereich (links) und Layout-Editor (rechts), die für die Beispiele generiert wurden. Im Layout-Editor wird das Histogramm für die Autofluoreszenzröhre und die positive Röhre für FL1-H gezeigt und wie Sie das Eigenschaftenfenster abrufen, um sie zu ändern. (E) Das Bild zeigt, wie der mittlere Fluoreszenzwert der Intensität ermittelt wird. (F) Histogramme, die für drei verschiedene Fluorochrome erstellt wurden und alle Änderungen zeigen, die die Software mit den statistischen Informationen vornehmen kann. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
5. Analyse zur Ermittlung der Anzahl der uEVs pro Probe.
HINWEIS: Abbildung 6 zeigt den Arbeitsbereich mit den Schritten zum Abrufen der Anzahl von uEVs pro Probe.
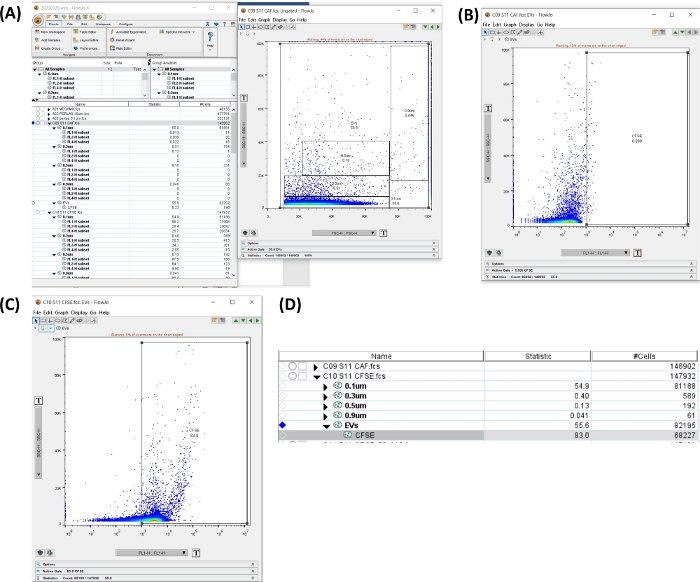
Abbildung 6: Arbeitsbereich zur Analyse der CFSE-Röhre. Alle Bilder wurden durch Siebdruck des Arbeitsbereichs erzeugt. (A) Arbeitsbereich, der durch die Auswahl aller Größenbereiche generiert wird, uEVs insgesamt (links), Punktdiagramm mit dem ausgewählten Gate (rechts). (B) Punktdiagramm SSC-H VS FL1-H für den CFSE-negativen Bereich in der Autofluoreszenzröhre. (C) Punktdiagramm SSC-H VS FL1-H für CFSE im Färberöhrchen. (D) Bild der Tabelle, die mit den Statistiken der CFSE-Färbung erstellt wurde und die Anzahl der uEV in der Probe zeigt. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Ergebnisse
Es gibt mehrere Kontrollpunkte durch das Protokoll und vor der Färbung von uEVs. Daher ist es wichtig, zunächst die Menge an Protein zu überprüfen, die im Extrakt von uEVs enthalten ist. Alle Forschungsgruppen, die mit extrazellulären Vesikeln arbeiten, quantifizieren das Protein, wie in Schritt 2.1 gezeigt. Ergänzende Abbildung 2 zeigt eine repräsentative 96-Well-Platte, die den uEV-Anteil in den Wells 4E, 5E und 6E enthält. Die Vertiefungen 1A, 2A und 3A besteh...
Diskussion
Heutzutage hat die Verwendung von extrazellulären Vesikeln als Biomarker für verschiedene Krankheiten zugenommen, insbesondere für solche, die aus nicht-invasiven Quellen wie Urin isoliert werden können 5,21,22,23,24. Es hat sich gezeigt, dass die Isolierung von uEVs eine wichtige Ressource ist, um den Status eines gesunde...
Offenlegungen
Die Autoren erklären, dass die Forschung in Abwesenheit einer finanziellen oder kommerziellen Beziehung durchgeführt wurde, die als potenzieller Interessenkonflikt ausgelegt werden könnte.
Danksagungen
Diese Arbeit wurde durch Zuschüsse von CONACyT (A3-S-36875) und dem UNAM-DGAPA-PAPIIT-Programm (IN213020 und IA202318) unterstützt. NH-I wurde durch das Stipendium 587790 von CONACyT unterstützt.
Die Autoren danken Leopoldo Flores-Romo†, Vianney Ortiz-Navarrete, Antony Boucard Jr. und Diana Gómez-Martin für ihre wertvollen Ratschläge zur Umsetzung dieses Protokolls und allen gesunden Personen für ihre Urinproben.
Materialien
| Name | Company | Catalog Number | Comments |
| APC anti human CD156c (ADAM10) antibody | BioLegend | 352706 | Add 5 µL to the 20 µL of uEVs in PBS |
| APC anti human TSPAN33 (BAAM) antibody | BioLegend | 395406 | Add 5 µL to the 20 µL of uEVs in PBS |
| Avanti centrifuge with JA-25.5O fixed angle rotor | Beckamn Coulter | J-26S XPI | |
| BD Accuri C6 Flow Cytometer | BD Biosciences | ||
| β-mercaptoethanol | SIGMA-Aldrich | M3148 | |
| Benchtop centrifuge with A-4-44 rotor | Eppendorf | 5804 | |
| BLUEstain 2 protein ladder | GOLDBIO | P008 | |
| CD9 (C-4) mouse monoclonal antibody | Santa Cruz Biotechnology | sc-13118 | |
| CD63 (MX-49.129.5) mouse monoclonal antibody | Santa Cruz Biotechnology | sc-5275 | |
| Cell Trace CFSE cell proliferation kit for flow cytometry | Thermo Scientific | C34554 | |
| Chemidoc XRS+ system | BIORAD | 5837 | |
| FITC anti human CD9 antibody | BioLegend | 312104 | Add 5 µL to the 20 µL of uEVs in PBS |
| FITC anti human CD37 antibody | BioLegend | 356304 | Add 5 µL to the 20 µL of uEVs in PBS |
| Fluorescent yellow particles | Spherotech | FP-0252-2 | |
| Fluorescent yellow particles | Spherotech | FP-0552-2 | |
| Fluorescent yellow particles | Spherotech | FP-1552-2 | |
| FlowJo Software | Becton, Dickinson and Company | ||
| Goat anti-mouse immunoglobulins/HRP | Dako | P0447 | |
| Halt protease inhibitor cocktail | Thermo Scientific | 78429 | |
| Immun-Blot PVDF membrane 0.22µm | BIORAD | 1620177 | |
| Megamix-Plus FSC beads | COSMO BIO CO.LTD | 7802 | |
| NuPAGE LDS sample buffer 4X | Thermo Scientific | NP0007 | |
| Optima ultracentrifuge with rotor 90Ti fixed angle 355530 | Beckamn Coulter | XPN100 | |
| Page Blue protein staining solution | Thermo Scientific | 24620 | |
| PE anti human CD53 antibody | BioLegend | 325406 | Add 5 µL to the 20 µL of uEVs in PBS |
| Pierce BCA Protein assay kit | Thermo Scientific | 23227 | |
| Pierce RIPA buffer | Thermo Scientific | 89900 | |
| Polycarbonate thick wall centrifuge tubes | Beckamn Coulter | 355630 | |
| Spherotech 8-Peak validation beads (FL1-FL3) | BD Accuri | 653144 | |
| Spherotech 6-Peak validation beads (FL4) | BD Accuri | 653145 | |
| Sucrose | SIGMA-Aldrich | 59378 | |
| Triethanolamine | SIGMA-Aldrich | 90279 |
Referenzen
- Decramer, S., et al. Urine in clinical proteomics. Molecular & Cellular Proteomics. 7 (10), 1850-1862 (2008).
- Nawaz, M., et al. The emerging role of extracellular vesicles as biomarkers for urogenital cancers. Nature Reviews Urology. 11 (12), 688-701 (2014).
- Pisitkun, T., Johnstone, R., Knepper, M. A. Discovery of urinary biomarkers. Molecular & Cellular Proteomics. 5 (10), 1760-1771 (2006).
- Urinology Think Tank Writing Group. Urine: Waste product or biologically active tissue. Neurourology and Urodynamics. 37 (3), 1162-1168 (2018).
- Wang, S., Kojima, K., Mobley, J. A., West, A. B. Proteomic analysis of urinary extracellular vesicles reveal biomarkers for neurologic disease. EBioMedicine. 45, 351-361 (2019).
- Merchant, M. L., Rood, I. M., Deegens, J. K. J., Klein, J. B. Isolation and characterization of urinary extracellular vesicles: implications for biomarker discovery. International Urology and Nephrology. 13 (12), 731-749 (2017).
- Alvarez, M. L., Khosroheidari, M., Kanchi Ravi, R., DiStefano, J. K. Comparison of protein, microRNA, and mRNA yields using different methods of urinary exosome isolation for the discovery of kidney disease biomarkers. Kidney International. 82 (9), 1024-1032 (2012).
- Jing, H., et al. The role of extracellular vesicles in renal fibrosis. Cell Death and Disease. 10 (5), 367 (2019).
- Liu, X., et al. Tubule-derived exosomes play a central role in fibroblast activation and kidney fibrosis. Kidney International. 97 (6), 1181-1195 (2020).
- Quaglia, M., et al. Extracellular Vesicles as Mediators of Cellular Crosstalk Between Immune System and Kidney Graft. Frontiers in Immunology. 11, 74 (2020).
- Simpson, R. J., Lim, J. W., Moritz, R. L., Mathivanan, S. Exosomes: proteomic insights and diagnostic potential. Expert Review of Proteomics. 6 (3), 267-283 (2009).
- Street, J. M., Koritzinsky, E. H., Glispie, D. M., Star, R. A., Yuen, P. S. Urine Exosomes: An Emerging Trove of Biomarkers. Advances in Clinical Chemistry. 78, 103-122 (2017).
- Thongboonkerd, V. Roles for Exosome in Various Kidney Diseases and Disorders. Frontiers in Pharmacology. 10, 1655 (2019).
- Watanabe, Y., et al. Molecular Network Analysis of the Urinary Proteome of Alzheimer's Disease Patients. Dementia and Geriatric Cognitive Disorders Extra. 9 (1), 53-65 (2019).
- Jadli, A. S., Ballasy, N., Edalat, P., Patel, V. B. Inside(sight) of tiny communicator: exosome biogenesis, secretion, and uptake. Molecular and Cellular Biochemistry. 467 (1-2), 77-94 (2020).
- Svenningsen, P., Sabaratnam, R., Jensen, B. L. Urinary extracellular vesicles: Origin, role as intercellular messengers and biomarkers; efficient sorting and potential treatment options. Acta Physiologica. 228 (1), 13346 (2020).
- Witwer, K. W., Thery, C. Extracellular vesicles or exosomes? On primacy, precision, and popularity influencing a choice of nomenclature. Journal of Extracellular Vesicles. 8 (1), 1648167 (2019).
- Thery, C., et al. Minimal information for studies of extracellular vesicles 2018 (MISEV2018): a position statement of the International Society for Extracellular Vesicles and update of the MISEV2014 guidelines. Journal of Extracellular Vesicles. 7 (1), 1535750 (2018).
- Perez-Hernandez, J., et al. Increased Urinary Exosomal MicroRNAs in Patients with Systemic Lupus Erythematosus. PLoS One. 10 (9), 0138618 (2015).
- Freyssinet, J. M., Toti, F. Membrane microparticle determination: at least seeing what's being sized. Journal of Thrombosis and Haemostasis. 8 (2), 311-314 (2010).
- D'Anca, M., et al. Exosome Determinants of Physiological Aging and Age-Related Neurodegenerative Diseases. Frontiers in Aging Neuroscience. 11, 232 (2019).
- Lawson, C., Vicencio, J. M., Yellon, D. M., Davidson, S. M. Microvesicles and exosomes: new players in metabolic and cardiovascular disease. Journal of Endocrinology. 228 (2), 57-71 (2016).
- Lee, S., Mankhong, S., Kang, J. H. Extracellular Vesicle as a Source of Alzheimer's Biomarkers: Opportunities and Challenges. International Journal of Molecular Sciences. 20 (7), (2019).
- Mori, M. A., Ludwig, R. G., Garcia-Martin, R., Brandao, B. B., Kahn, C. R. Extracellular miRNAs: From Biomarkers to Mediators of Physiology and Disease. Cell Metabolism. 30 (4), 656-673 (2019).
- Hoorn, E. J., et al. Prospects for urinary proteomics: exosomes as a source of urinary biomarkers. Nephrology (Carlton). 10 (3), 283-290 (2005).
- Ranghino, A., Dimuccio, V., Papadimitriou, E., Bussolati, B. Extracellular vesicles in the urine: markers and mediators of tissue damage and regeneration. Clinical Kidney Journal. 8 (1), 23-30 (2015).
- Zhang, W., et al. Extracellular vesicles in diagnosis and therapy of kidney diseases. American Journal of Physiology-Renal Physiology. 311 (5), 844-851 (2016).
- Fernandez-Llama, P., et al. Tamm-Horsfall protein and urinary exosome isolation. Kidney International. 77 (8), 736-742 (2010).
- Royo, F., et al. Different EV enrichment methods suitable for clinical settings yield different subpopulations of urinary extracellular vesicles from human samples. Journal of Extracellular Vesicles. 5, 29497 (2016).
- Rider, M. A., Hurwitz, S. N., Meckes, D. G. ExtraPEG: A Polyethylene Glycol-Based Method for Enrichment of Extracellular Vesicles. Scientific Reports. 6, 23978 (2016).
- Lv, C. Y., et al. A PEG-based method for the isolation of urinary exosomes and its application in renal fibrosis diagnostics using cargo miR-29c and miR-21 analysis. International Urology and Nephrology. 50 (5), 973-982 (2018).
- Alvarez, M. L., Khosroheidari, M., Kanchi Ravi, R., DiStefano, J. K. Comparison of protein, microRNA, and mRNA yields using different methods of urinary exosome isolation for the discovery of kidney disease biomarkers. Kidney International. 82 (9), 1024-1032 (2012).
- Wang, D., Sun, W. Urinary extracellular microvesicles: isolation methods and prospects for urinary proteome. Proteomics. 14 (16), 1922-1932 (2014).
- Pospichalova, V., et al. Simplified protocol for flow cytometry analysis of fluorescently labeled exosomes and microvesicles using dedicated flow cytometer. Journal of Extracellular Vesicles. 4, 25530 (2015).
- van der Pol, E., et al. Standardization of extracellular vesicle measurements by flow cytometry through vesicle diameter approximation. Journal of Thrombosis and Haemostasis. 16 (6), 1236-1245 (2018).
- Huyan, T., Du, Y., Huang, Q., Huang, Q., Li, Q. Uptake Characterization of Tumor Cell-derived Exosomes by Natural Killer Cells. Iranian Journal of Public Health. 47 (6), 803-813 (2018).
- Puzar Dominkus, P., et al. PKH26 labeling of extracellular vesicles: Characterization and cellular internalization of contaminating PKH26 nanoparticles. Biochimica et Biophysica Acta (BBA) - Biomembranes. 1860 (6), 1350-1361 (2018).
- Morales-Kastresana, A., et al. Labeling Extracellular Vesicles for Nanoscale Flow Cytometry. Scientific Reports. 7 (1), 1878 (2017).
- de Rond, L., et al. Comparison of Generic Fluorescent Markers for Detection of Extracellular Vesicles by Flow Cytometry. Clinical Chemistry. 64 (4), 680-689 (2018).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten