Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Détermination de la taille et analyse phénotypique des vésicules extracellulaires urinaires par cytométrie en flux
Dans cet article
Résumé
Ce protocole décrit une méthode d’isolement des vésicules extracellulaires urinaires, les uEVs, à partir de donneurs humains sains et leur caractérisation phénotypique par l’expression de la taille et du marqueur de surface à l’aide de la cytométrie en flux.
Résumé
Les vésicules extracellulaires sont un complexe hétérogène de membranes lipidiques, sécrétées par tout type de cellule, dans n’importe quel liquide tel que l’urine. Les VE peuvent être de différentes tailles, allant de 40 à 100 nm de diamètre, comme dans les exosomes, à 100-1000 nm dans les microvésicules. Ils peuvent également contenir différentes molécules qui peuvent être utilisées comme biomarqueurs pour le pronostic et le diagnostic de nombreuses maladies. De nombreuses techniques ont été développées pour caractériser ces vésicules. L’une d’entre elles est la cytométrie en flux. Cependant, il n’existe aucun rapport montrant comment quantifier la concentration des VE et les différencier en fonction de leur taille, ainsi que de la détection de biomarqueurs. Ce travail vise à décrire une procédure d’isolement, de quantification et de phénotypification des vésicules extracellulaires urinaires, les uEVs, à l’aide d’un cytomètre conventionnel pour l’analyse sans aucune modification de sa configuration. Les limites de la méthode comprennent la coloration d’un maximum de quatre biomarqueurs différents par échantillon. La méthode est également limitée par la quantité de VE disponibles dans l’échantillon. Malgré ces limites, avec ce protocole et son analyse ultérieure, nous pouvons obtenir plus d’informations sur l’enrichissement des marqueurs EVs et l’abondance de ces vésicules présentes dans les échantillons d’urine, dans les maladies impliquant des lésions rénales et cérébrales.
Introduction
Chez les mammifères, le sang est filtré en passant par les reins 250 à 300 fois ; Pendant ce temps, de l’urine se forme. La production de ce biofluide est le résultat d’une série de processus, notamment la filtration glomérulaire, la réabsorption tubulaire et la sécrétion. Les déchets métaboliques et les électrolytes sont les principaux composants de l’urine. De plus, d’autres sous-produits tels que les peptides, les protéines fonctionnelles et les vésicules extracellulaires (VE) sont excrétés 1,2,3,4,5,6. Initialement, des vésicules extracellulaires urinaires (vec) ont été identifiées dans des échantillons d’urine de patients souffrant de troubles de l’équilibre hydrique. Ces patients ont montré la présence de molécules telles que l’aquaporine-2 (AQP2), qui a ensuite été utilisée comme biomarqueur de cette maladie7. Plusieurs études ultérieures ont porté sur l’identification de l’origine cellulaire des uEV, décrivant que ces structures peuvent être sécrétées par les cellules rénales (glomérule, podocytes, etc.) et d’autres types de cellules de lignées endothéliales ou leucocytaires. De plus, le nombre et l’enrichissement en molécules dans les uEV peuvent être corrélés avec l’état de nombreuses maladies et troubles 8,9,10,11,12,13,14.
Dans l’ensemble, les VE constituent une famille très hétérogène de particules enfermées dans des bicouches lipidiques et libérées par les cellules par des mécanismes passifs ou actifs dans différents fluides. Selon leur origine, les VE peuvent être classés comme des exosomes provenant d’endosomes ou des microvésicules/microparticules dérivées de la membrane plasmique. Cependant, ce critère de classification ne peut être appliqué que lorsque la biogenèse des particules est directement observée. Par conséquent, d’autres critères non triviaux, notamment l’origine physique, biochimique et cellulaire, ont été approuvés par plusieurs chercheurs dans le domaine 15,16,17. Selon la nature de l’isolat analysé, différentes techniques analytiques ont été suggérées pour la caractérisation des VE. Par exemple, sur la base de l’enrichissement de VE de grande taille (≥100 nm) ou de petite taille (≤100 nm), une quantification par cytométrie en flux ou suivi de nanoparticules est suggérée, respectivement18.
De nos jours, l’utilisation des VE comme biomarqueurs pour de nombreuses maladies est devenue pertinente, de sorte que la recherche de différentes sources est étudiée. L’une des sources les plus prometteuses est l’urine car elle peut être obtenue de manière facile et non invasive. Par conséquent, ce protocole décrit une procédure pour l’isolement des uEVs par centrifugation différentielle, traitement avec des anticorps conjugués au fluorochrome et analyse en aval à l’aide d’un cytomètre conventionnel à 2 lasers/4 couleurs.
Access restricted. Please log in or start a trial to view this content.
Protocole
Les échantillons d’urine humaine ont été prélevés sur des volontaires en bonne santé qui avaient signé le consentement éclairé du donneur. Ces procédures ont également été approuvées par le Comité d’éthique de la recherche de l’Instituto Nacional de Ciencias Médicas y Nutrición Salvador Zubirán.
1. Isolement des vésicules extracellulaires urinaires
REMARQUE : Le protocole d’isolement des uEV est modifié à partir de la réf.19. La figure 1 illustre la représentation du protocole d’isolement des uEV.
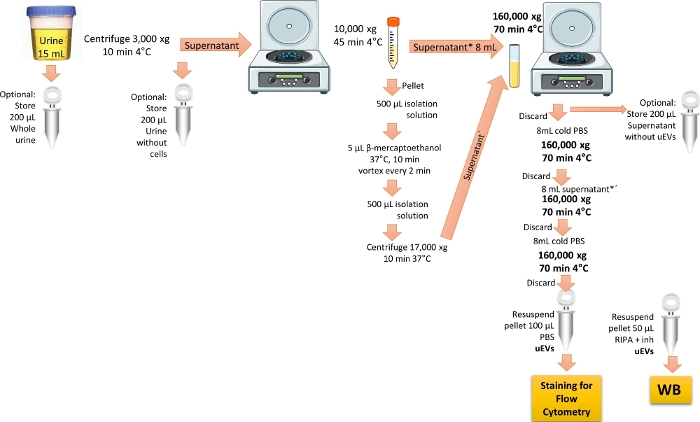
Figure 1 : Vue d’ensemble de l’isolement des uEVs pour l’analyse par cytométrie en flux. Dans ce protocole, centrifugez d’abord la première urine de la journée pour éliminer les cellules et les débris. Puis centrifuger pour retirer les grandes vésicules avec un traitement pour éliminer la protéine THP et enfin effectuer une ultracentrifugation pour enrichir et obtenir les uEVs en un seul lavage. Les étapes pour conserver les fractions urinaires pour la validation de la WB sont indiquées. Veuillez cliquer ici pour voir une version agrandie de cette figure.
- Utilisez l’urine du premier matin (15 ml) provenant de volontaires en bonne santé. Centrifugez l’urine à 3 000 x g pendant 10 min à 4 °C, pour éliminer toutes les cellules et les débris.
REMARQUE : Utilisez de préférence de l’urine fraîche ; si elle n’est pas disponible, utiliser l’urine conservée pendant une période maximale de 3 mois à -20 °C, ou conservée pendant une période maximale de 6 mois à -70 °C. Décongelez l’échantillon d’urine sur de la glace et agitez-le vigoureusement. Effectuez toutes les procédures sur glace ou à 4 °C. Une étape facultative de l’analyse par transfert Western consiste à faire une copie du tube et à aliquote 200 μL de l’urine du premier matin dans un tube séparé. Conservez l’échantillon à -20 °C jusqu’à utilisation, en l’étiquetant comme « urine entière », pour vérifier la présence de marqueurs de vésicules extracellulaires. - Transvaser le surnageant obtenu à l’étape 1.1 dans un nouveau tube à centrifuger conique de 15 mL, puis centrifuger à 10 000 x g pendant 45 min à 4 °C. Transférez le surnageant dans un tube d’ultracentrifugation en polycarbonate de 8 ml et conservez-le sur de la glace.
REMARQUE : Facultatif, aliquote de 200 μL du surnageant obtenu à l’étape 1.1 dans un tube séparé étiqueté « urine sans cellules » pour l’analyse par transfert Western.- Élimination de la protéine de Tamm-Horsfall, THP
REMARQUE : Le THP est présent dans l’urine et est enrichi lorsqu’une personne souffre d’une maladie rénale. Il a été rapporté que le THP diminue le rendement des uEV parce qu’il peut se lier aux uEV. Pour éliminer cette protéine, l’utilisation d’un agent réducteur est nécessaire 3,6.- Préparez la solution d’isolement en mélangeant 250 mM de saccharose avec 10 mM de triéthanolamine. Ajustez le pH à 7,6.
- Mélangez la pastille obtenue à l’étape 1.2 avec 500 μL de solution d’isolement, puis ajoutez 5 μL de β-mercaptoéthanol.
- Incuber le mélange de granulés à 37 °C pendant 10 min, en tourbillonnant toutes les 2 min. Ajouter 500 μL de solution d’isolement, puis centrifuger à 17 000 x g pendant 10 min à 37 °C. Récupérez le surnageant obtenu (contenant des THP non agrégés réduits plus uEV).
- Élimination de la protéine de Tamm-Horsfall, THP
- Mélanger les deux surnageants réservés (selon les étapes 1.2 et 1.2.1.3) dans le même tube d’ultracentrifugation en polycarbonate de 8 mL, puis centrifuger à 160 000 x g pendant 70 min à 4 °C, à l’aide d’un rotor à angle fixe de l’ultracentrifugeuse.
REMARQUE : Optionnellement, pour l’analyse par transfert de Western, stocker 200 μL du surnageant obtenu ci-dessus en l’étiquetant comme « surnageant sans VE ». Cet échantillon servira de contrôle négatif lors de la recherche de marqueurs de vésicules extracellulaires. - Jetez le surnageant. Ajoutez du PBS 1x glacé dans le tube en polycarbonate de 8 ml et centrifugez à 160 000 x g pendant 70 min à 4 °C.
REMARQUE : Veillez à ne pas jeter le granulé, car il contient les uEV. Le 1x PBS doit être stérilisé et filtré avec un filtre à seringue à pores d’au moins 0,22 μm. - Jetez le surnageant. Ajouter le reste du surnageant dans le tube en polycarbonate de 8 mL et centrifuger à 160 000 x g pendant 70 min à 4 °C.
- Jetez le surnageant et ajoutez 8 ml de PBS glacé à la pastille pour laver les uEV.
- Jetez le surnageant. Laissez sécher la pastille puis remettez-la en suspension avec 1 mL de PBS glacé. Conserver à -70 °C jusqu’à utilisation.
REMARQUE : Si vous effectuez une analyse par transfert Western, remettre en suspension la pastille du tube dupliqué avec 50 μL de tampon RIPA et des inhibiteurs de protéase. Conserver à -20 °C jusqu’à utilisation.
2. Coloration des uEV
REMARQUE : Avant la coloration et l’analyse des uEV, il est essentiel d’effectuer au moins une méthodologie recommandée par l’MISEV201818 pour vérifier l’isolation correcte des uEV ; ici, l’analyse par transfert de Western est représentée. La figure 2 montre un protocole représentatif de la coloration des uEV.
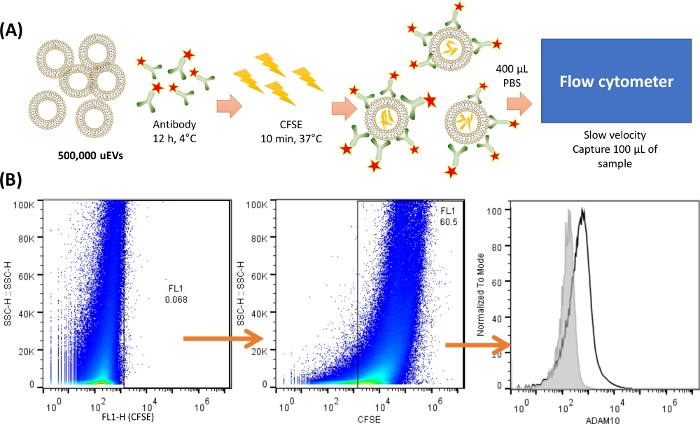
Figure 2 : Vue d’ensemble de la coloration et de la capture des uEVs dans le cytomètre. (A) Représentation de la coloration uEV. Pour 500 000 uEVs, l’anticorps a été mélangé et incubé à 4 °C pendant 12 h. Ensuite, le CFSE a été ajouté et incubé à 37 °C pendant 10 min. Les uEV avaient le CFSE à l’intérieur, et l’anticorps se liera à la surface de l’antigène. 400 μL de PBS froid ont été utilisés pour remettre en suspension et capturer 100 μL de l’échantillon dans le cytomètre en flux à une vitesse lente. (B) Stratégie d’analyse. Le premier graphique à points (SSC-H VS FL-X) représente le contrôle négatif pour les uEVs, suivi du graphique à points montrant la coloration des uEVs avec CFSE, et enfin, un histogramme avec la coloration des anticorps des uEVs (ligne noire), le contrôle négatif est montré dans la ligne grise. Veuillez cliquer ici pour voir une version agrandie de cette figure.
- Calculez le nombre relatif d’uEV en quantifiant la teneur en protéines à l’aide de n’importe quel test colorimétrique conventionnel des protéines, comme mentionné dans la Table des matériaux. Effectuez une dilution 1:5 des uEVs et suivez les instructions de la fiche technique fournie dans le kit de dosage des protéines.
- D’après la formule20 déjà rapportée, considérer que 1 μg/mL de protéine uEVs équivaut à 800 000 uEVs/μL. Assurez-vous que 500 000 uEVs sont présents dans 20 μL de PBS glacé.
- Étiquetez les tubes, comme indiqué dans le tableau 1.
REMARQUE : Le nombre de tubes utilisés dépendra du nombre d’anticorps utilisés, la limitation pour le cytomètre 2-lasers/4 couleurs est un maximum de 4 anticorps par tube qui pourraient être utilisés ; par conséquent, seuls les FL1, FL2, FL3 et FL4 dans le cytomètre ont pu être lus, bien qu’une compensation soit nécessaire. Pour ces procédures, utilisez des tubes de microcentrifugation de 1,5 ml ou des tubes à fond rond de 5 ml pour la cytométrie en flux. Les tubes nécessaires sont illustrés dans le tableau 1. Les tubes 4 et 5 sont constitués d’un cocktail de tous les anticorps à utiliser dans ce protocole. Deux tubes problématiques (8 et 9) sont donnés à titre d’exemple ; Par conséquent, cet ensemble de commandes doit avoir la combinaison nécessaire pour être utilisé dans chaque tube.
| Tube 1. | Perles Megamix FSC | |
| Tube 2. | PBS | |
| Tube 3. | PBS avec CFSE | |
| Tube 4. | PBS avec tous les anticorps du problème 1 | |
| Tube 5. | PBS avec tous les anticorps du problème 2 | |
| Tube 6. | Contrôle de l’autofluorescence | uEVs sans aucun réactif, uniquement en PBS. |
| Tube 7. | #uEVs | uEVs avec CFSE |
| Métro 8. | Problème 1 | uEV avec CD37 FITC, CD53 PE, ADAM10 APC |
| Tube 9. | Problème 2 | uEV avec CD9 FITC, TSPAN33 APC |
Tableau 1 : Étiquetage des tubes. Exemple montrant comment étiqueter les tubes. Les premiers tubes sont tous les contrôles nécessaires. Les tubes avec les anticorps-fluorochromes dépendront de la coloration.
- Ajoutez 20 μL de PBS contenant 500 000 uEV dans les tubes étiquetés.
- Ajouter les anticorps indiqués dans le tableau 1, préalablement titrés. Incuber toute la nuit à 4 °C.
REMARQUE : Avant la coloration, il est recommandé de centrifuger les anticorps à 4 °C à pleine vitesse pendant au moins 5 min, afin d’éviter les agrégats. Les anticorps utilisés ici sont un exemple de protéines présentes dans les uEVs et appartiennent à un manuscrit indépendant actuellement en préparation. - Ajouter 0,4 μL d’ester succinimidylique de carboxyfluorescéine (CFSE) [5 nM] dans le tube numéro 3. Incuber pendant 10 min à 37 °C.
REMARQUE : Le CFSE est un colorant utilisé pour colorer tous les VE présents dans un échantillon et faire la distinction entre le bruit de fond lorsqu’une analyse par cytomètre en flux est effectuée. Pour plus d’informations, consultez la section Discussion . - Ajoutez 400 μL de 1x PBS glacé à tous les tubes.
- Maintenez tous les tubes à 4 °C.
3. Acquisition d’uEV à l’aide d’un cytomètre classique
REMARQUE : Les instructions d’utilisation du cytomètre en flux (voir tableau des matériaux) sont décrites ici.
- Effectuez l’étalonnage de la qualité du cytomètre, à l’aide des billes à 6 et 8 pics.
REMARQUE : Le % CV du dernier pic doit être une valeur inférieure à 6 ; Le technicien en cytomètre s’en chargera. - Ouvrez le logiciel du cytomètre en flux.
REMARQUE : Une fois le logiciel ouvert, une nouvelle expérience s’ouvrira, montrant un criblage avec un modèle pour 96 échantillons, les paramètres de fonctionnement et une section « vide » pour créer des diagrammes à points ou des histogrammes. - Ajustez les paramètres sur l’écran du logiciel en cours d’exécution : 100 μL d’échantillon à capturer, fonctionnement lent, réglage du seuil à 10 000 sur FSC-H, et 2 000 sur SSC-H.
REMARQUE : Le seuil recommandé ici est un exemple ; Il est nécessaire de fixer le seuil en fonction des échantillons analysés. - Tube de charge numéro 1 (tube Megamix), comme indiqué dans le tableau 1. Capturez les perles. Créez deux diagrammes de points dans la partie « vide » de l’écran du logiciel en cliquant sur l’option Dot Plot . Créez les panneaux A et B de la figure 3.
REMARQUE : Les billes fluorescentes Megamix (billes utilisées pour délimiter les tailles de 0,1, 0,3, 0,5 et 0,9 μm et créer le modèle pour les tailles), peuvent être analysées à l’aide du détecteur FL1 (FITC). Tous les diagrammes à points et histogrammes doivent être affichés en valeurs de hauteur. Enregistrer le plus d’événements possible ; Toute modification dans la feuille de calcul n’altère pas les données.
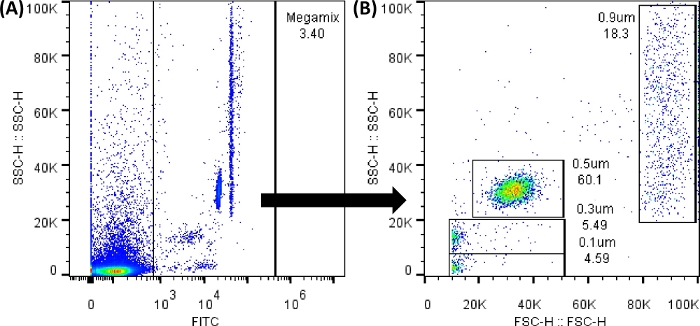
Figure 3 : Graphiques à points des billes Megamix FSC.Les diagrammes à points ont été générés à l’aide du logiciel de cytomètre en flux ; Dans le cytomètre en flux, l’image sera très similaire. (A) Le premier pointage généré pour sélectionner les perles en évitant le bruit de fond. (SSC-H CONTRE FL1-H). (B) Le graphique à points généré par la sélection de la porte précédente, montrant les différentes tailles des perles. (SSC-H CONTRE FSC-H). Veuillez cliquer ici pour voir une version agrandie de cette figure.
- Dans la section « vide » de l’écran du logiciel, créez un diagramme à points et un histogramme pour chaque fluorochrome utilisé ; utilisez l’algorithme de la figure supplémentaire 1 à titre d’exemple. Pour ce faire, cliquez sur l’option Dot Plot ou Histogram pour chaque fluorochrome.
REMARQUE : Le bruit de fond sera toujours présent, comme illustré à la Figure 3A. Tous les points compris entre 0 et 1 000 de FL1 (axe des x) se trouvent à l’extérieur de la porte créée. Par conséquent, il est essentiel d’utiliser tous les tubes de contrôle pour obtenir des résultats adéquats. Entre chaque chargement de tube, rincez à contre-courant le liquide de gaine et effectuez un cycle de débouchage. Secouez doucement chaque tube avant de le charger. Après avoir chargé cinq tubes consécutifs, faites fonctionner 100 μL de PBS 1x. - Chargez les tubes numéros 2 et 5, pour régler les valeurs de coupure (négatives). Pour ce faire, sélectionnez l’icône Porte rectangulaire (située sous le graphique à points créé) ou l’icône Porte linéaire (située sous l’histogramme créé) ; et le placer là où il n’y a pas de signal, afin d’obtenir des diagrammes à points et des histogrammes comme ceux illustrés dans la figure supplémentaire 1.
REMARQUE : Tous ces tubes nécessitent le placement de la région positive pas plus de 0,70 % car il n’y aura pas de « fluorescence » plus de 103 (1 500). S’il y a une fluorescence au-delà de cette région, diluez les réactifs utilisés. Par conséquent, il est important de titrer les réactifs avant les mesures finales. - Tube de charge numéro 6 (tube d’autofluorescence), pour régler les régions négatives de l’échantillon. Pour ce faire, sélectionnez l’icône Porte rectangulaire (située sous le graphique à points créé) ou l’icône Porte linéaire (située sous l’histogramme créé) ; Placez-le là où il n’y a pas de signal.
- Chargez les tubes suivants (tubes 7 à 9, illustrés dans le tableau 1).
- Enregistrez l’expérience.
REMARQUE : Pour le logiciel de cytomètre utilisé ici, la commande Enregistrer l’expérience fait référence à la sauvegarde des données générées. En d’autres termes, tous les tubes acquis par le cytomètre en flux, ainsi que les données générées pour chaque tube, doivent être enregistrés. Pour ce faire, il est nécessaire de cliquer sur le bouton Enregistrer l’expérience . - Exportez les données sous forme de fichiers FSC.
4. Analyse des données avec un logiciel de cytomètre en flux.
REMARQUE : Les instructions d’utilisation du logiciel de cytomètre en flux illustré dans le tableau des matériaux sont décrites dans cette section. La figure 4 montre l’espace de travail avec les étapes de création des portes de taille.
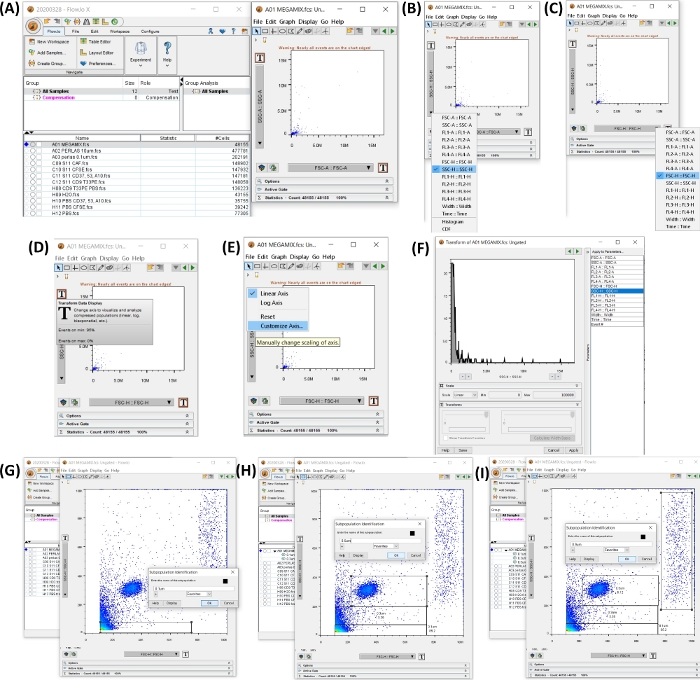
Figure 4 : Espace de travail avec toutes les étapes pour commencer l’analyse des données. Toutes les images ont été générées par sérigraphie de l’espace de travail. (A) Espace de travail généré avec les données de l’échantillon ajoutées (à gauche), diagramme à points généré par la sélection du tube 1, billes FSC, (à droite). (B,C) montrer la modification de l’axe, pour avoir SSC-H et FSC-H. (D-F) montrent la personnalisation étape par étape des deux axes. (G-I) montrent la sélection et la génération des différentes tailles de billes. Veuillez cliquer ici pour voir une version agrandie de cette figure.
- Ouvrez le logiciel du cytomètre en flux. Ajoutez les échantillons, en cliquant sur le bouton Ajouter des échantillons , et sélectionnez les fichiers FCS exportés.
- Cliquez sur le tube Megamix pour ouvrir un graphique SSC-H (axe y) VS FSC-H (axe x). Ajustez les deux axes pour afficher de 0 à 100 000 en cliquant sur le bouton T (Transformer l’affichage des données), cliquez sur Personnaliser l’axe, cliquez sur SSC-H ou FSSC-H et modifiez la valeur de l’échelle sur min 0 et max 100 000 (voir Figure 4A-F).
- Réglez les portes pour 0,1, 0,3, 0,5 et 0,9 μm dans le diagramme à points généré à l’étape 4.2. Sélectionnez le bouton Rectangle Gate pour créer un gate comme illustré à la Figure 4G-I.
REMARQUE : La figure 5 montre l’espace de travail avec les étapes permettant de créer les régions positives et d’obtenir l’intensité moyenne du fluorochrome.

Figure 5 : Espace de travail pour analyser les données obtenues. Toutes les images ont été générées par la sérigraphie de l’espace de travail. (A) Espace de travail généré avec la porte de taille appliquée à tous les échantillons. (B) Tube d’autofluorescence sélectionné, graphique à points montrant la porte de taille et l’histogramme pour une taille sélectionnée (0,1 μm), utilisez cet histogramme pour obtenir la porte positive pour chaque fluorochrome et taille. (C) Espace de travail généré avec les portes positives pour chaque fluorochrome et taille. (D) Espace de travail (à gauche) et Éditeur de mise en page (à droite) générés pour les échantillons. Dans l’éditeur de mise en page, l’histogramme pour le tube d’autofluorescence et le tube positif pour FL1-H est affiché, et comment obtenir le panneau des propriétés pour les modifier. (E) L’image montre comment obtenir la valeur de fluorescence d’intensité moyenne. (F) Histogrammes générés pour trois fluorochromes différents, montrant toutes les modifications que le logiciel permet d’effectuer avec les informations statistiques. Veuillez cliquer ici pour voir une version agrandie de cette figure.
- Appliquez les portes générées (0,1, 0,3, 0,5 et 0,9 μm) à tous les échantillons. Sélectionnez les portes, faites glisser et déposez l’option Tous les échantillons , comme illustré à la figure 5A.
- Cliquez sur le tube d’autofluorescence. Définissez les régions positives pour chaque fluorochrome, pour chaque taille. Ouvrez le graphique à points SSC-H VS FSC-H avec les portes de taille, cliquez sur Taille de porte unique. Dans la nouvelle fenêtre, cliquez sur l’axe des y pour sélectionner l’option d’histogramme. Sur l’axe x, sélectionnez FL1-H, puis sélectionnez l’icône Plage pour créer la région positive. Répétez l’opération pour FL2-H et FL4-H (voir Figure 5B).
- Appliquez les portes à tous les échantillons. Sélectionnez Gates, faites glisser et déposez l’option Tous les échantillons , comme illustré à la figure 5C.
- Ouvrez l’éditeur de mise en page.
- Faites glisser et déposez chaque échantillon dans l’éditeur. Sélectionnez la taille du tube « autofluorescence », glissez-déposez, puis sélectionnez la même taille que le tube coloré, glissez-déposez.
- Cliquez sur le bas à droite de l’histogramme. Ouvrez le menu Propriétés . Cliquez sur Légende. Ajoutez l’intensité moyenne du fluorochrome.
REMARQUE : Dans les propriétés, il existe d’autres outils disponibles pour modifier l’apparence de l’histogramme. - Répétez la même procédure pour les fluorochromes et les tailles restants.
5. Analyse pour obtenir le nombre d’uEV par échantillon.
REMARQUE : La figure 6 montre l’espace de travail avec les étapes permettant d’obtenir le nombre d’uEV par échantillon.
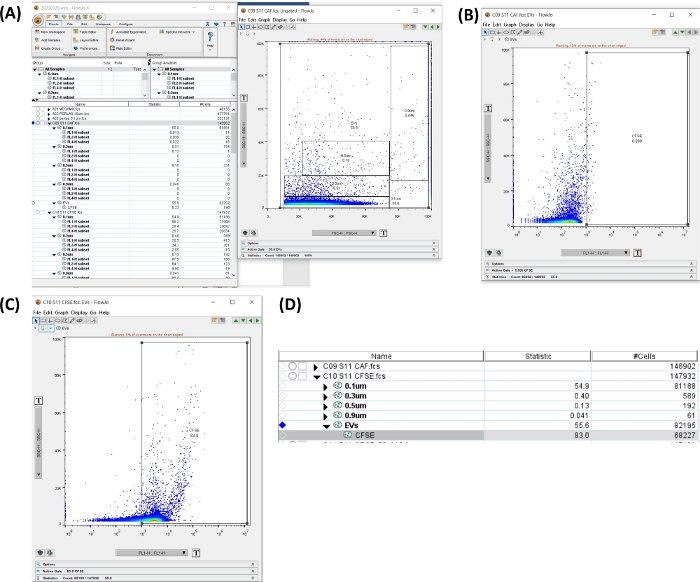
Figure 6 : Espace de travail pour analyser le tube CFSE. Toutes les images ont été générées par sérigraphie de l’espace de travail. (A) Espace de travail généré par la sélection de toutes les tailles de la région, du total des uEV (à gauche), du graphique à points montrant la porte sélectionnée (à droite). (B) Diagramme à points SSC-H VS FL1-H pour la région négative CFSE dans le tube d’autofluorescence. (C) Diagramme à points SSC-H VS FL1-H pour CFSE dans le tube de coloration. (D) Image du tableau obtenu avec les statistiques de la coloration CFSE, montrant le nombre d’uEV dans l’échantillon. Veuillez cliquer ici pour voir une version agrandie de cette figure.
- Cliquez sur le tube d’autofluorescence. Dans le graphique à points SSC-H VS FSC-H avec les portes de taille, créez une région comprenant toutes les tailles. Cliquez sur Nouvelle région. Réglez la région positive (voir Figure 6A,B).
- Appliquez les portes à tous les échantillons. Sélectionnez les portes, faites-les glisser et déposez-les dans l’option Tous les échantillons .
- Cliquez sur le tube CFSE + uEVs. Ouvrez la nouvelle région et vérifiez la région positive pour CFSE (FL1-H), comme illustré à la figure 6C.
- Dans l’espace de travail de la figure 6D, copiez les données des # cellules pour le tube. Ce nombre est le #uEVs obtenu par cytométrie en flux (#uEVs FC).
- Appliquez la formule suivante pour calculer le nombre d’uEV par microlitre :
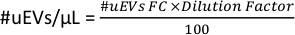
- Pour avoir le #uEVs par microlitre pour chaque fluorochrome (#uEVs/μL FLX), au niveau de l’espace de travail, copiez les statistiques du sous-ensemble FLX-H, ce nombre est le pourcentage (sous-ensemble %FLX).
- Appliquez la formule suivante pour calculer le nombre d’uEV par microlitre pour chaque fluorochrome.
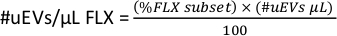
REMARQUE : Selon que la porte sélectionnée sera pour une taille unique ou pour tous les uEV, vérifiez les données sélectionnées.
Access restricted. Please log in or start a trial to view this content.
Résultats
Il y a plusieurs points de contrôle à travers le protocole, et avant la coloration des uEV. Par conséquent, il est essentiel de vérifier d’abord la quantité de protéines présentes dans l’extrait d’uEVs. Tous les groupes de recherche qui travaillent avec des vésicules extracellulaires quantifient la protéine, comme indiqué à l’étape 2.1. La figure supplémentaire 2 montre une plaque représentative de 96 puits contenant la fraction d’uEV dans les pui...
Access restricted. Please log in or start a trial to view this content.
Discussion
De nos jours, l’utilisation de vésicules extracellulaires comme biomarqueurs pour plusieurs maladies a augmenté, en particulier pour celles qui peuvent être isolées à partir de sources non invasives telles que l’urine 5,21,22,23,24. Il a été prouvé que l’isolement des uEVs est une ressource vitale pour connaîtr...
Access restricted. Please log in or start a trial to view this content.
Déclarations de divulgation
Les auteurs déclarent que la recherche a été menée en l’absence de toute relation financière ou commerciale qui pourrait être interprétée comme un conflit d’intérêts potentiel.
Remerciements
Ce travail a été soutenu par des subventions de la CONACyT (A3-S-36875) et du programme UNAM-DGAPA-PAPIIT (IN213020 et IA202318). NH-I a été soutenu par la bourse 587790 de CONACyT.
Les auteurs tiennent à remercier Leopoldo Flores-Romo†, Vianney Ortiz-Navarrete, Antony Boucard Jr et Diana Gómez-Martin pour leurs précieux conseils pour la réalisation de ce protocole, et à tous les individus en bonne santé pour leurs échantillons d’urine.
Access restricted. Please log in or start a trial to view this content.
matériels
| Name | Company | Catalog Number | Comments |
| APC anti human CD156c (ADAM10) antibody | BioLegend | 352706 | Add 5 µL to the 20 µL of uEVs in PBS |
| APC anti human TSPAN33 (BAAM) antibody | BioLegend | 395406 | Add 5 µL to the 20 µL of uEVs in PBS |
| Avanti centrifuge with JA-25.5O fixed angle rotor | Beckamn Coulter | J-26S XPI | |
| BD Accuri C6 Flow Cytometer | BD Biosciences | ||
| β-mercaptoethanol | SIGMA-Aldrich | M3148 | |
| Benchtop centrifuge with A-4-44 rotor | Eppendorf | 5804 | |
| BLUEstain 2 protein ladder | GOLDBIO | P008 | |
| CD9 (C-4) mouse monoclonal antibody | Santa Cruz Biotechnology | sc-13118 | |
| CD63 (MX-49.129.5) mouse monoclonal antibody | Santa Cruz Biotechnology | sc-5275 | |
| Cell Trace CFSE cell proliferation kit for flow cytometry | Thermo Scientific | C34554 | |
| Chemidoc XRS+ system | BIORAD | 5837 | |
| FITC anti human CD9 antibody | BioLegend | 312104 | Add 5 µL to the 20 µL of uEVs in PBS |
| FITC anti human CD37 antibody | BioLegend | 356304 | Add 5 µL to the 20 µL of uEVs in PBS |
| Fluorescent yellow particles | Spherotech | FP-0252-2 | |
| Fluorescent yellow particles | Spherotech | FP-0552-2 | |
| Fluorescent yellow particles | Spherotech | FP-1552-2 | |
| FlowJo Software | Becton, Dickinson and Company | ||
| Goat anti-mouse immunoglobulins/HRP | Dako | P0447 | |
| Halt protease inhibitor cocktail | Thermo Scientific | 78429 | |
| Immun-Blot PVDF membrane 0.22µm | BIORAD | 1620177 | |
| Megamix-Plus FSC beads | COSMO BIO CO.LTD | 7802 | |
| NuPAGE LDS sample buffer 4X | Thermo Scientific | NP0007 | |
| Optima ultracentrifuge with rotor 90Ti fixed angle 355530 | Beckamn Coulter | XPN100 | |
| Page Blue protein staining solution | Thermo Scientific | 24620 | |
| PE anti human CD53 antibody | BioLegend | 325406 | Add 5 µL to the 20 µL of uEVs in PBS |
| Pierce BCA Protein assay kit | Thermo Scientific | 23227 | |
| Pierce RIPA buffer | Thermo Scientific | 89900 | |
| Polycarbonate thick wall centrifuge tubes | Beckamn Coulter | 355630 | |
| Spherotech 8-Peak validation beads (FL1-FL3) | BD Accuri | 653144 | |
| Spherotech 6-Peak validation beads (FL4) | BD Accuri | 653145 | |
| Sucrose | SIGMA-Aldrich | 59378 | |
| Triethanolamine | SIGMA-Aldrich | 90279 |
Références
- Decramer, S., et al. Urine in clinical proteomics. Molecular & Cellular Proteomics. 7 (10), 1850-1862 (2008).
- Nawaz, M., et al. The emerging role of extracellular vesicles as biomarkers for urogenital cancers. Nature Reviews Urology. 11 (12), 688-701 (2014).
- Pisitkun, T., Johnstone, R., Knepper, M. A. Discovery of urinary biomarkers. Molecular & Cellular Proteomics. 5 (10), 1760-1771 (2006).
- Urinology Think Tank Writing Group. Urine: Waste product or biologically active tissue. Neurourology and Urodynamics. 37 (3), 1162-1168 (2018).
- Wang, S., Kojima, K., Mobley, J. A., West, A. B. Proteomic analysis of urinary extracellular vesicles reveal biomarkers for neurologic disease. EBioMedicine. 45, 351-361 (2019).
- Merchant, M. L., Rood, I. M., Deegens, J. K. J., Klein, J. B. Isolation and characterization of urinary extracellular vesicles: implications for biomarker discovery. International Urology and Nephrology. 13 (12), 731-749 (2017).
- Alvarez, M. L., Khosroheidari, M., Kanchi Ravi, R., DiStefano, J. K. Comparison of protein, microRNA, and mRNA yields using different methods of urinary exosome isolation for the discovery of kidney disease biomarkers. Kidney International. 82 (9), 1024-1032 (2012).
- Jing, H., et al. The role of extracellular vesicles in renal fibrosis. Cell Death and Disease. 10 (5), 367(2019).
- Liu, X., et al. Tubule-derived exosomes play a central role in fibroblast activation and kidney fibrosis. Kidney International. 97 (6), 1181-1195 (2020).
- Quaglia, M., et al. Extracellular Vesicles as Mediators of Cellular Crosstalk Between Immune System and Kidney Graft. Frontiers in Immunology. 11, 74(2020).
- Simpson, R. J., Lim, J. W., Moritz, R. L., Mathivanan, S. Exosomes: proteomic insights and diagnostic potential. Expert Review of Proteomics. 6 (3), 267-283 (2009).
- Street, J. M., Koritzinsky, E. H., Glispie, D. M., Star, R. A., Yuen, P. S. Urine Exosomes: An Emerging Trove of Biomarkers. Advances in Clinical Chemistry. 78, 103-122 (2017).
- Thongboonkerd, V. Roles for Exosome in Various Kidney Diseases and Disorders. Frontiers in Pharmacology. 10, 1655(2019).
- Watanabe, Y., et al. Molecular Network Analysis of the Urinary Proteome of Alzheimer's Disease Patients. Dementia and Geriatric Cognitive Disorders Extra. 9 (1), 53-65 (2019).
- Jadli, A. S., Ballasy, N., Edalat, P., Patel, V. B. Inside(sight) of tiny communicator: exosome biogenesis, secretion, and uptake. Molecular and Cellular Biochemistry. 467 (1-2), 77-94 (2020).
- Svenningsen, P., Sabaratnam, R., Jensen, B. L. Urinary extracellular vesicles: Origin, role as intercellular messengers and biomarkers; efficient sorting and potential treatment options. Acta Physiologica. 228 (1), 13346(2020).
- Witwer, K. W., Thery, C. Extracellular vesicles or exosomes? On primacy, precision, and popularity influencing a choice of nomenclature. Journal of Extracellular Vesicles. 8 (1), 1648167(2019).
- Thery, C., et al. Minimal information for studies of extracellular vesicles 2018 (MISEV2018): a position statement of the International Society for Extracellular Vesicles and update of the MISEV2014 guidelines. Journal of Extracellular Vesicles. 7 (1), 1535750(2018).
- Perez-Hernandez, J., et al. Increased Urinary Exosomal MicroRNAs in Patients with Systemic Lupus Erythematosus. PLoS One. 10 (9), 0138618(2015).
- Freyssinet, J. M., Toti, F. Membrane microparticle determination: at least seeing what's being sized. Journal of Thrombosis and Haemostasis. 8 (2), 311-314 (2010).
- D'Anca, M., et al. Exosome Determinants of Physiological Aging and Age-Related Neurodegenerative Diseases. Frontiers in Aging Neuroscience. 11, 232(2019).
- Lawson, C., Vicencio, J. M., Yellon, D. M., Davidson, S. M. Microvesicles and exosomes: new players in metabolic and cardiovascular disease. Journal of Endocrinology. 228 (2), 57-71 (2016).
- Lee, S., Mankhong, S., Kang, J. H. Extracellular Vesicle as a Source of Alzheimer's Biomarkers: Opportunities and Challenges. International Journal of Molecular Sciences. 20 (7), (2019).
- Mori, M. A., Ludwig, R. G., Garcia-Martin, R., Brandao, B. B., Kahn, C. R. Extracellular miRNAs: From Biomarkers to Mediators of Physiology and Disease. Cell Metabolism. 30 (4), 656-673 (2019).
- Hoorn, E. J., et al. Prospects for urinary proteomics: exosomes as a source of urinary biomarkers. Nephrology (Carlton). 10 (3), 283-290 (2005).
- Ranghino, A., Dimuccio, V., Papadimitriou, E., Bussolati, B. Extracellular vesicles in the urine: markers and mediators of tissue damage and regeneration. Clinical Kidney Journal. 8 (1), 23-30 (2015).
- Zhang, W., et al. Extracellular vesicles in diagnosis and therapy of kidney diseases. American Journal of Physiology-Renal Physiology. 311 (5), 844-851 (2016).
- Fernandez-Llama, P., et al. Tamm-Horsfall protein and urinary exosome isolation. Kidney International. 77 (8), 736-742 (2010).
- Royo, F., et al. Different EV enrichment methods suitable for clinical settings yield different subpopulations of urinary extracellular vesicles from human samples. Journal of Extracellular Vesicles. 5, 29497(2016).
- Rider, M. A., Hurwitz, S. N., Meckes, D. G. ExtraPEG: A Polyethylene Glycol-Based Method for Enrichment of Extracellular Vesicles. Scientific Reports. 6, 23978(2016).
- Lv, C. Y., et al. A PEG-based method for the isolation of urinary exosomes and its application in renal fibrosis diagnostics using cargo miR-29c and miR-21 analysis. International Urology and Nephrology. 50 (5), 973-982 (2018).
- Alvarez, M. L., Khosroheidari, M., Kanchi Ravi, R., DiStefano, J. K. Comparison of protein, microRNA, and mRNA yields using different methods of urinary exosome isolation for the discovery of kidney disease biomarkers. Kidney International. 82 (9), 1024-1032 (2012).
- Wang, D., Sun, W. Urinary extracellular microvesicles: isolation methods and prospects for urinary proteome. Proteomics. 14 (16), 1922-1932 (2014).
- Pospichalova, V., et al. Simplified protocol for flow cytometry analysis of fluorescently labeled exosomes and microvesicles using dedicated flow cytometer. Journal of Extracellular Vesicles. 4, 25530(2015).
- van der Pol, E., et al. Standardization of extracellular vesicle measurements by flow cytometry through vesicle diameter approximation. Journal of Thrombosis and Haemostasis. 16 (6), 1236-1245 (2018).
- Huyan, T., Du, Y., Huang, Q., Huang, Q., Li, Q. Uptake Characterization of Tumor Cell-derived Exosomes by Natural Killer Cells. Iranian Journal of Public Health. 47 (6), 803-813 (2018).
- Puzar Dominkus, P., et al. PKH26 labeling of extracellular vesicles: Characterization and cellular internalization of contaminating PKH26 nanoparticles. Biochimica et Biophysica Acta (BBA) - Biomembranes. 1860 (6), 1350-1361 (2018).
- Morales-Kastresana, A., et al. Labeling Extracellular Vesicles for Nanoscale Flow Cytometry. Scientific Reports. 7 (1), 1878(2017).
- de Rond, L., et al. Comparison of Generic Fluorescent Markers for Detection of Extracellular Vesicles by Flow Cytometry. Clinical Chemistry. 64 (4), 680-689 (2018).
Access restricted. Please log in or start a trial to view this content.
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon