È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Determinazione delle dimensioni e analisi fenotipica delle vescicole extracellulari urinarie mediante citometria a flusso
In questo articolo
Riepilogo
Questo protocollo descrive un metodo per l'isolamento di vescicole extracellulari urinarie, uEV, da donatori umani sani e la loro caratterizzazione fenotipica in base alle dimensioni e all'espressione del marcatore di superficie mediante citometria a flusso.
Abstract
Le vescicole extracellulari, EV, sono un complesso eterogeneo di membrane lipidiche, secrete da qualsiasi tipo di cellula, in qualsiasi fluido come l'urina. Le vescicole extracellulari possono essere di diverse dimensioni, che vanno da 40-100 nm di diametro, come negli esosomi, a 100-1000 nm nelle microvescicole. Possono anche contenere diverse molecole che possono essere utilizzate come biomarcatori per la prognosi e la diagnosi di molte malattie. Molte tecniche sono state sviluppate per caratterizzare queste vescicole. Uno di questi è la citometria a flusso. Tuttavia, non esistono rapporti che mostrino come quantificare la concentrazione di vescicole extracellulari e differenziarle in base alle dimensioni, insieme al rilevamento dei biomarcatori. Questo lavoro si propone di descrivere una procedura per l'isolamento, la quantificazione e la fenotipizzazione di vescicole extracellulari urinarie, uEVs, utilizzando un citometro convenzionale per l'analisi senza alcuna modifica alla sua configurazione. I limiti del metodo includono la colorazione di un massimo di quattro diversi biomarcatori per campione. Il metodo è limitato anche dalla quantità di vescicole extracellulari disponibili nel campione. Nonostante queste limitazioni, con questo protocollo e la sua successiva analisi, possiamo ottenere maggiori informazioni sull'arricchimento dei marcatori delle vescicole extracellulari e sull'abbondanza di queste vescicole presenti nei campioni di urina, nelle malattie che comportano danni renali e cerebrali.
Introduzione
Nei mammiferi, il sangue viene filtrato passando attraverso i reni 250 - 300 volte; Durante questo periodo, si forma l'urina. La produzione di questo biofluido è il risultato di una serie di processi, tra cui la filtrazione glomerulare, il riassorbimento tubulare e la secrezione. I prodotti di scarto metabolici e gli elettroliti sono i componenti principali dell'urina. Inoltre, altri sottoprodotti come peptidi, proteine funzionali e vescicole extracellulari (EV) vengono escreti 1,2,3,4,5,6. Inizialmente, le vescicole extracellulari urinarie (uEV) sono state identificate in campioni di urina di pazienti affetti da disturbi dell'equilibrio idrico. Questi pazienti hanno mostrato la presenza di molecole come l'acquaporina-2 (AQP2), che è stata poi utilizzata come biomarcatore per questa malattia7. Diversi studi successivi si sono concentrati sull'identificazione dell'origine cellulare delle uEV, descrivendo che queste strutture possono essere secrete dalle cellule renali (glomeruli, podociti, ecc.) e da altri tipi di cellule di linee endoteliali o leucocitarie. Inoltre, il numero e l'arricchimento di molecole nelle uEV possono essere correlati con lo stato di molte malattie e disturbi 8,9,10,11,12,13,14.
Complessivamente, le vescicole extracellulari costituiscono una famiglia altamente eterogenea di particelle racchiuse da doppi strati lipidici e rilasciate dalle cellule attraverso meccanismi passivi o attivi in diversi fluidi. A seconda della loro origine, le vescicole extracellulari possono essere classificate come esosomi originati da endosomi o microvescicole/microparticelle derivate dalla membrana plasmatica. Tuttavia, questo criterio di classificazione può essere applicato solo quando la biogenesi delle particelle è osservata direttamente. Pertanto, altri criteri non banali, tra cui l'origine fisica, biochimica e cellulare, sono stati approvati da diversi ricercatori nel campo 15,16,17. A seconda della natura dell'isolato analizzato, sono state suggerite diverse tecniche analitiche per la caratterizzazione delle vescicole extracellulari. Ad esempio, sulla base dell'arricchimento di vescicole extracellulari grandi (≥100 nm) o piccole (≤100 nm), si suggerisce la quantificazione tramite citometria a flusso o tracciamento di nanoparticelle, rispettivamente18.
Al giorno d'oggi, l'uso delle vescicole extracellulari come biomarcatori per molte malattie è diventato rilevante, quindi è stata studiata la ricerca di diverse fonti. Una delle fonti più promettenti è l'urina in quanto può essere ottenuta in modo facile e non invasivo. Pertanto, questo protocollo descrive una procedura per l'isolamento delle uEV mediante centrifugazione differenziale, il trattamento con anticorpi coniugati al fluorocromo e l'analisi a valle utilizzando un citometro convenzionale a 2 laser/4 colori.
Access restricted. Please log in or start a trial to view this content.
Protocollo
I campioni di urina umana sono stati ottenuti da volontari sani che avevano firmato il consenso informato del donatore. Queste procedure sono state approvate anche dal Comitato Etico per la Ricerca dell'Instituto Nacional de Ciencias Médicas y Nutrición Salvador Zubirán.
1. Isolamento delle vescicole extracellulari urinarie
NOTA: Il protocollo di isolamento degli uEV è stato modificato rispetto al rif.19. La Figura 1 illustra la rappresentazione del protocollo per isolare gli uEV.
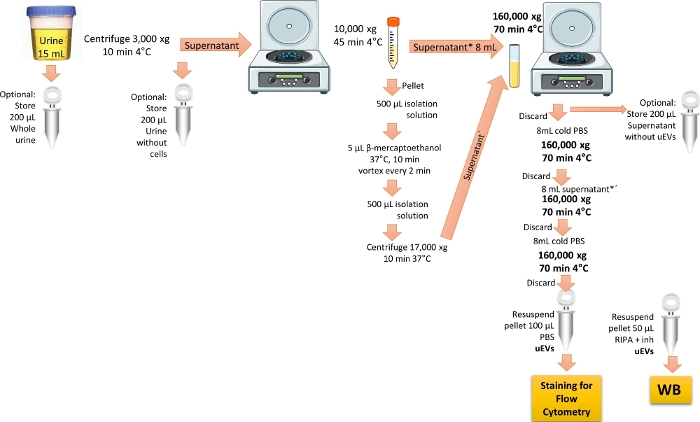
Figura 1: Panoramica dell'isolamento delle uEV per l'analisi della citometria a flusso. In questo protocollo, centrifugare prima la prima urina della giornata per rimuovere le cellule e i detriti. Quindi centrifugare per rimuovere le grandi vescicole con trattamento per rimuovere la proteina THP e infine eseguire l'ultracentrifugazione per arricchire e ottenere gli uEV con un solo lavaggio. I passaggi per mantenere le frazioni urinarie per la convalida WB sono contrassegnati. Clicca qui per visualizzare una versione più grande di questa figura.
- Utilizzare l'urina del primo mattino (15 ml) di volontari sani. Centrifugare l'urina a 3.000 x g per 10 minuti a 4 °C, per rimuovere tutte le cellule e i detriti.
NOTA: Utilizzare preferibilmente urina fresca; se non disponibile, utilizzare urina conservata per un periodo massimo di 3 mesi a -20 °C, o conservata per un periodo massimo di 6 mesi a -70 °C. Scongelare il campione di urina sul ghiaccio e farlo roteare energicamente. Eseguire tutte le procedure su ghiaccio o a 4 °C. Un passaggio facoltativo per l'analisi Western blot consiste nel creare una provetta duplicata e aliquotare 200 μl delle urine del primo mattino in una provetta separata. Conservare il campione a -20 °C fino al momento dell'uso, etichettandolo come "urina intera", per verificare la presenza di marcatori di vescicole extracellulari. - Trasferire il surnatante ottenuto al punto 1.1 in una nuova provetta da centrifuga conica da 15 mL, quindi centrifugare a 10.000 x g per 45 min a 4 °C. Trasferire il surnatante in una provetta per ultracentrifugazione in policarbonato da 8 ml e conservarla con ghiaccio.
NOTA: Facoltativamente, aliquotare 200 μl del surnatante ottenuto nella fase 1.1 in una provetta separata etichettata come "urina senza cellule" per l'analisi Western blot.- Rimozione della proteina Tamm-Horsfall, THP
NOTA: Il THP è presente nelle urine ed è arricchito quando un individuo ha una malattia renale. È stato riportato che la THP diminuisce la resa delle uEV perché può legarsi alle uEV. Per rimuovere questa proteina, è necessario l'uso di un agente riducente 3,6.- Preparare la soluzione di isolamento mescolando 250 mM di saccarosio con 10 mM di trietanolammina. Regolare il pH a 7,6.
- Miscelare il pellet ottenuto nella fase 1.2 con 500 μL di soluzione isolante, quindi aggiungere 5 μL di β-mercaptoetanolo.
- Incubare la miscela di pellet a 37 °C per 10 minuti, agitando ogni 2 minuti. Aggiungere 500 μl di soluzione isolante e centrifugare a 17.000 x g per 10 minuti a 37 °C. Raccogliere il surnatante ottenuto (contenente THP ridotto-non aggregato più uEV).
- Rimozione della proteina Tamm-Horsfall, THP
- Miscelare entrambi i surnatanti riservati (dai passaggi 1.2 e 1.2.1.3) nella stessa provetta per ultracentrifugazione in policarbonato da 8 mL, quindi centrifugare a 160.000 x g per 70 minuti a 4 °C, utilizzando un rotore ad angolo fisso per ultracentrifuga.
NOTA: Facoltativamente, per l'analisi Western blot, conservare 200 μl del surnatante ottenuto sopra etichettandolo come "surnatante senza EV". Questo campione servirà come controllo negativo durante la ricerca di marcatori di vescicole extracellulari. - Scartare il surnatante. Aggiungere 1x PBS ghiacciato alla provetta in policarbonato da 8 mL e centrifugare a 160.000 x g per 70 minuti a 4 °C.
NOTA: Fare attenzione a non scartare il pellet, poiché contiene gli uEV. Il PBS 1x deve essere sterilizzato e filtrato con un filtro per siringa a pori da almeno 0,22 μm. - Scartare il surnatante. Aggiungere il resto del surnatante alla provetta in policarbonato da 8 mL e centrifugare a 160.000 x g per 70 min a 4 °C.
- Scartare il surnatante e aggiungere 8 ml di PBS ghiacciato al pellet per lavare gli uEV.
- Scartare il surnatante. Lasciare asciugare il pellet e poi risospenderlo con 1 mL di PBS ghiacciato. Conservare a -70 °C fino al momento dell'uso.
NOTA: Se si esegue l'analisi Western blot, risospendere il pellet della provetta duplicata con 50 μl di tampone RIPA più inibitori della proteasi. Conservare a -20 °C fino al momento dell'uso.
2. Colorazione degli uEV
NOTA: Prima della colorazione e dell'analisi delle uEV, è essenziale eseguire almeno una metodologia raccomandata da MISEV201818 per verificare il corretto isolamento delle uEV; qui è raffigurata l'analisi del Western blot. La Figura 2 mostra un protocollo rappresentativo per la colorazione degli uEV.
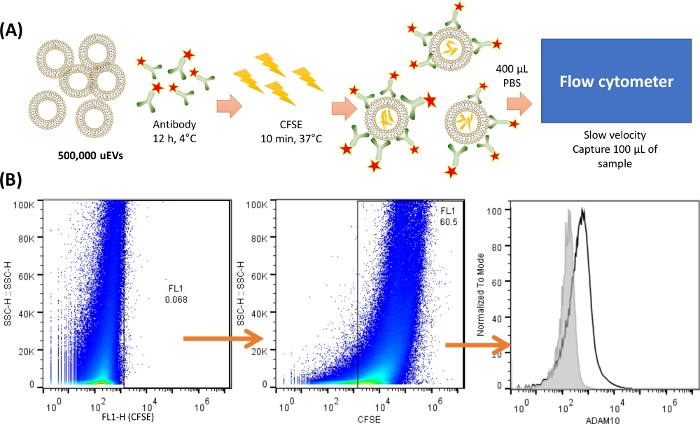
Figura 2: Panoramica della colorazione e della cattura delle uEV nel citometro. (A) Rappresentazione della colorazione uEV. Per 500.000 uEV, l'anticorpo è stato miscelato e incubato a 4 °C per 12 ore. Quindi è stato aggiunto CFSE e incubato a 37 °C per 10 minuti. Gli uEV avevano il CFSE all'interno e l'anticorpo si legherà alla superficie dell'antigene. Sono stati utilizzati 400 μL di PBS freddo per risospendere e catturare 100 μL del campione nel citometro a flusso a bassa velocità. (B) Strategia di analisi. Il primo grafico a punti (SSC-H VS FL-X) rappresenta il controllo negativo per le uEV, seguito dal grafico a punti che mostra la colorazione delle uEV con CFSE e, infine, un istogramma con la colorazione anticorpale delle uEV (linea nera), il controllo negativo è mostrato nella linea grigia. Clicca qui per visualizzare una versione più grande di questa figura.
- Calcolare il numero relativo di uEV quantificando il contenuto proteico utilizzando qualsiasi test proteico colorimetrico convenzionale, come indicato nella Tabella dei materiali. Eseguire una diluizione 1:5 degli uEV e seguire le istruzioni della scheda tecnica fornita nel kit di analisi delle proteine.
- Sulla base di una formula20 precedentemente riportata, considerare 1 μg/mL di proteina uEVs pari a 800.000 uEVs/μL. Assicurarsi che 500.000 uEVs siano presenti in 20 μL di PBS ghiacciato.
- Etichettare i tubi, come indicato nella Tabella 1.
NOTA: Il numero di provette utilizzate dipenderà dal numero di anticorpi impiegati, il limite per il citometro a 2 laser/4 colori è un massimo di 4 anticorpi per provetta che potrebbero essere utilizzati; pertanto, è stato possibile leggere solo FL1, FL2, FL3 e FL4 nel citometro, sebbene sia necessaria una compensazione. Per queste procedure, utilizzare provette per microcentrifuga da 1,5 mL o provette a fondo tondo da 5 mL per la citometria a flusso. I tubi necessari sono illustrati nella Tabella 1. Le provette 4 e 5 sono costituite da un cocktail di tutti gli anticorpi da utilizzare in questo protocollo. Due tubi problematici (8 e 9) sono forniti a titolo di esempio; Pertanto, questo set di controlli deve avere la combinazione da utilizzare in ciascun tubo.
| Tubo 1. | Perline Megamix FSC | |
| Tubo 2. | PBS | |
| Tubo 3. | PBS con CFSE | |
| Tubo 4. | PBS con tutti gli anticorpi del problema 1 | |
| Tubo 5. | PBS con tutti gli anticorpi del problema 2 | |
| Tubo 6. | Controllo dell'autofluorescenza | uEV senza alcun reagente, solo in PBS. |
| Tubo 7. | #uEVs | uEV con CFSE |
| Tubo 8. | Problema 1 | uEV con CD37 FITC, CD53 PE, ADAM10 APC |
| Tubo 9. | Problema 2 | uEV con CD9 FITC, TSPAN33 APC |
Tabella 1: Etichettatura delle provette. Esempio che mostra come etichettare i tubi. I primi tubi sono tutti i controlli necessari. Le provette con gli anticorpi-fluorocromi dipenderanno dalla colorazione.
- Aggiungere 20 μl di PBS contenente 500.000 uEV alle provette etichettate.
- Aggiungere gli anticorpi come indicato nella Tabella 1, precedentemente titolata. Incubare per una notte a 4 °C.
NOTA: Prima della colorazione, si consiglia di centrifugare gli anticorpi a 4 °C alla massima velocità per almeno 5 minuti, per evitare la formazione di aggregati. Gli anticorpi qui utilizzati sono un esempio di proteine presenti nelle uEV e appartengono a un manoscritto indipendente attualmente in preparazione. - Aggiungere 0,4 μl di carbossifluoresceina succinimidil estere (CFSE) [5 nM] alla provetta numero 3. Incubare per 10 minuti a 37 °C.
NOTA: Il CFSE è un colorante utilizzato per colorare tutte le vescicole extracellulari presenti in un campione e discriminare tra il rumore di fondo quando viene eseguita un'analisi con citometro a flusso. Per ulteriori informazioni, vedere la sezione Discussione . - Aggiungere 400 μl di 1x PBS ghiacciato a tutte le provette.
- Mantenere tutti i tubi a 4 °C.
3. Acquisizione di uEV utilizzando un citometro convenzionale
NOTA: Le istruzioni per l'uso del citometro a flusso (vedere la Tabella dei materiali) sono descritte qui.
- Eseguire la calibrazione di qualità del citometro, utilizzando le perle a 6 e 8 picchi.
NOTA: La %CV dell'ultimo picco deve essere un valore inferiore a 6; Il tecnico del citometro si occuperà di questo. - Aprire il software del citometro a flusso.
NOTA: Una volta aperto il software, si aprirà un nuovo esperimento, che mostra uno screening con un modello per 96 campioni, i parametri per l'esecuzione e una sezione "vuota" per creare grafici a punti o istogrammi. - Regolare i parametri sullo schermo del software in esecuzione: 100 μL di campione da acquisire, funzionamento lento, impostare la soglia a 10.000 su FSC-H e 2.000 su SSC-H.
NOTA: La soglia consigliata qui è un esempio; E' necessario impostare la soglia in base ai campioni analizzati. - Caricare il tubo numero 1 (tubo Megamix), come indicato nella Tabella 1. Cattura le perline. Crea due grafici a punti nella parte "vuota" della schermata del software facendo clic sull'opzione Grafico a punti . Creare come pannello A e B della Figura 3.
NOTA: Le microsfere fluorescenti Megamix (perle utilizzate per delimitare le dimensioni di 0,1, 0,3, 0,5 e 0,9 μm e creare il modello per le dimensioni), possono essere analizzate utilizzando il rivelatore FL1 (FITC). Tutti i grafici a punti e gli istogrammi devono essere visualizzati in valori di altezza. Registra il maggior numero possibile di eventi; Qualsiasi modifica nel foglio di lavoro non altera i dati.
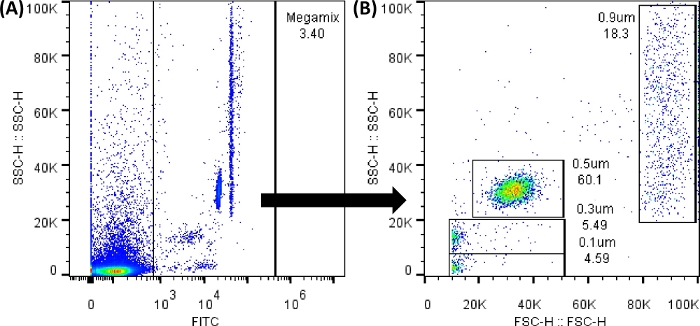
Figura 3: Grafici a punti di perline Megamix FSC. I dot plot mostrati sono stati generati utilizzando il software del citometro a flusso; Nel citometro a flusso, l'immagine sarà molto simile. (A) Il primo grafico a punti generato per selezionare le perline evitando il rumore di fondo. (SSC-H CONTRO FL1-H). (B) Il grafico a punti generato dalla selezione del cancello precedente, che mostra le diverse dimensioni delle perline. (SSC-H CONTRO FSC-H). Clicca qui per visualizzare una versione più grande di questa figura.
- Nella sezione "vuota" della schermata del software creare un dot plot e un istogramma per ogni fluorocromo utilizzato; utilizzare l'algoritmo nella Figura 1 supplementare come esempio. Per fare ciò, fare clic sull'opzione Dot Plot o Istogramma per ogni fluorocromo.
NOTA: Il rumore di fondo sarà sempre presente, come mostrato nella Figura 3A. Tutti i punti compresi tra 0 e 1.000 di FL1 (asse x) si trovano all'esterno del gate creato. Pertanto, è essenziale utilizzare tutti i tubi di controllo per ottenere risultati adeguati. Tra un caricamento e l'altro del tubo, risciacquare il fluido della guaina ed eseguire un ciclo di disostruzione. Agitare delicatamente ogni tubo prima di caricarlo. Dopo aver caricato cinque provette consecutive, eseguire 100 μl di PBS 1x. - Caricare i tubi numero 2 e 5, per impostare i valori di cut-off (negativi). Per fare ciò, selezionare l'icona Cancello rettangolo (situata sotto il grafico a punti creato) o l'icona Cancello linea (situata sotto l'istogramma creato); e posizionarlo dove non c'è segnale, in modo da ottenere grafici a punti e istogrammi come quelli mostrati nella Figura 1 supplementare.
NOTA: Tutti questi tubi richiedono il posizionamento della regione positiva non oltre lo 0,70% perché non ci sarà "fluorescenza" superiore a 103 (1.500). Se c'è una fluorescenza oltre questa regione, diluire i reagenti utilizzati. Pertanto, è importante titolare i reagenti prima delle misurazioni finali. - Caricare la provetta numero 6 (provetta in autofluorescenza), per impostare le regioni negative per il campione. Per fare ciò, selezionare l'icona Cancello rettangolo (situata sotto il grafico a punti creato) o l'icona Cancello linea (situata sotto l'istogramma creato); Posizionalo dove non c'è segnale.
- Caricare i tubi successivi (tubi da 7 a 9, illustrati nella Tabella 1).
- Salva l'esperimento.
NOTA: Per il software del citometro qui utilizzato, il comando Salva esperimento, si riferisce al salvataggio dei dati generati. In altre parole, tutte le provette che vengono acquisite dal citometro a flusso, e i dati generati per ogni provetta, devono essere salvati. Per fare ciò, è necessario fare clic sul pulsante Salva esperimento . - Esporta i dati come file FSC.
4. Analisi dei dati con un software citofluorimetrico.
NOTA: Le istruzioni per l'uso del software del citometro a flusso illustrate nella tabella dei materiali sono descritte in questa sezione. La Figura 4 mostra l'area di lavoro con i passaggi per creare i gate di dimensione.
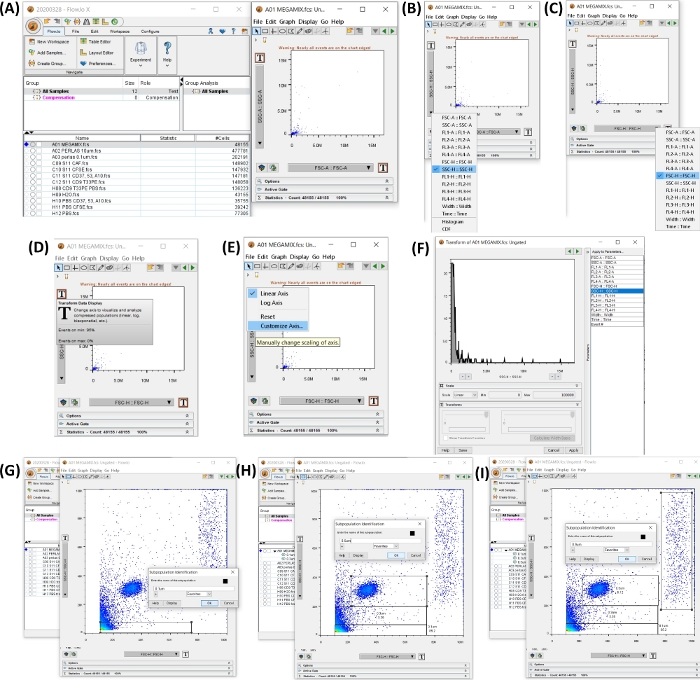
Figura 4: Area di lavoro con tutti i passaggi per iniziare l'analisi dei dati. Tutte le immagini sono state generate tramite serigrafia dell'area di lavoro. (A) Area di lavoro generata con l'aggiunta dei dati del campione (a sinistra), dot plot generato dalla selezione del tubo 1, perline FSC, (a destra). (B,C) mostrano la modifica dell'asse, per avere SSC-H e FSC-H. (D-F) mostrano la personalizzazione passo-passo di entrambi gli assi. (G-I) mostrano la selezione e la generazione delle diverse dimensioni delle perline. Clicca qui per visualizzare una versione più grande di questa figura.
- Aprire il software del citometro a flusso. Aggiungere i campioni, facendo clic sul pulsante Aggiungi campioni , e selezionare i file FCS esportati.
- Fare clic sul tubo Megamix per aprire un grafico a punti SSC-H (asse y) VS FSC-H (asse x). Regolare entrambi gli assi per visualizzare da 0 a 100.000 facendo clic sul pulsante T (Trasforma visualizzazione dati), fare clic su Personalizza asse, fare clic su SSC-H o FSSC-H e modificare il valore della scala in min 0 e max 100.000 (vedere la Figura 4A-F).
- Impostare i gate per 0,1, 0,3, 0,5 e 0,9 μm nel grafico a punti generato nel passaggio 4.2. Selezionare il pulsante Cancello rettangolare per creare un cancello come illustrato nella Figura 4G-I.
NOTA: La Figura 5 mostra l'area di lavoro con i passaggi per creare le regioni positive e per ottenere l'intensità media per il fluorocromo.

Figura 5: Area di lavoro per analizzare i dati ottenuti. Tutte le immagini sono state generate dalla serigrafia dell'area di lavoro. (A) Area di lavoro generata con il gate delle dimensioni applicato a tutti i campioni. (B) Tubo di autofluorescenza selezionato, dot plot che mostra il gate dimensionale e l'istogramma per una dimensione selezionata (0,1 μm), utilizzare questo istogramma per ottenere il gate positivo per ciascun fluorocromo e dimensione. (C) Area di lavoro generata con i gate positivi per ogni fluorocromo e dimensione. (D) Area di lavoro (a sinistra) e Editor layout (a destra) generati per gli esempi. Nell'Editor di Layout viene mostrato l'istogramma per il tubo di autofluorescenza e il tubo positivo per FL1-H, e come ottenere il pannello delle proprietà per modificarli. (E) L'immagine mostra come ottenere il valore medio di fluorescenza dell'intensità. (F) Istogrammi generati per tre diversi fluorocromi, che mostrano tutte le modifiche che il software consente di apportare alle informazioni statistiche. Clicca qui per visualizzare una versione più grande di questa figura.
- Applicare le porte generate (0,1, 0,3, 0,5 e 0,9 μm) a tutti i campioni. Selezionare i cancelli, trascinare e rilasciare l'opzione Tutti i campioni , come illustrato nella Figura 5A.
- Fare clic sul tubo di autofluorescenza. Imposta le regioni positive per ogni fluorocromo, per ogni dimensione. Apri il dot plot SSC-H VS FSC-H con le dimensioni dei cancelli, fai clic su Single Gate Size. Nella nuova finestra, fare clic sull'asse y per selezionare l'opzione dell'istogramma. Nell'asse x, selezionare FL1-H, quindi selezionare l'icona Intervallo per creare la regione positiva. Ripetere l'operazione per FL2-H e FL4-H (vedi Figura 5B).
- Applicare i gate a tutti i campioni. Selezionare Cancelli, trascinare e rilasciare l'opzione Tutti i campioni , come illustrato nella Figura 5C.
- Aprire l'editor di layout.
- Trascina e rilascia ogni campione nell'editor. Seleziona la dimensione del tubo "autofluorescenza", trascina e rilascia, quindi seleziona la stessa dimensione del tubo colorato, trascina e rilascia.
- Fare clic in basso a destra sull'istogramma. Aprire il menu Proprietà . Fare clic su Legenda. Aggiungi l'intensità media per il fluorocromo.
NOTA: In Proprietà, sono disponibili altri strumenti per modificare l'aspetto dell'istogramma. - Ripetere la stessa procedura per i fluorocromi e le dimensioni rimanenti.
5. Analisi per ottenere il numero di uEV per campione.
NOTA: La Figura 6 mostra l'area di lavoro con i passaggi per ottenere il numero di uEV per campione.
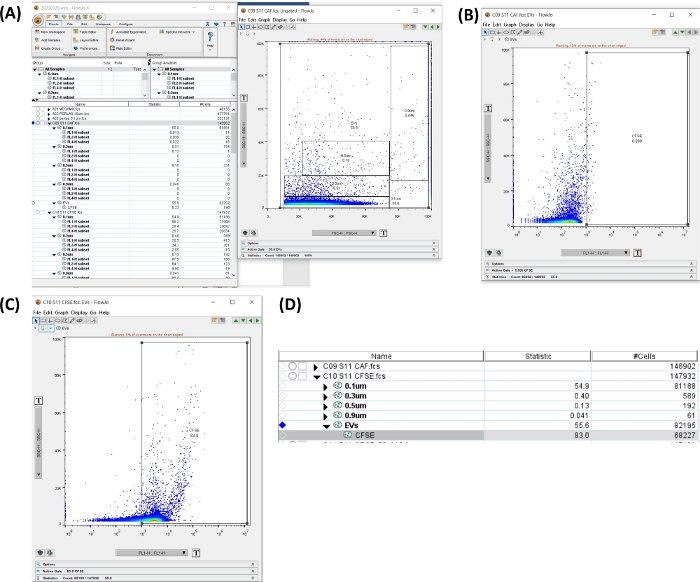
Figura 6: Area di lavoro per analizzare il tubo CFSE. Tutte le immagini sono state generate tramite serigrafia dell'area di lavoro. (A) Area di lavoro generata dalla selezione di tutte le dimensioni, uEVs totali (a sinistra), dot plot che mostra il gate selezionato (a destra). (B) Dot plot SSC-H VS FL1-H per la regione negativa CFSE nel tubo di autofluorescenza. (C) Dot plot SSC-H VS FL1-H per CFSE nel tubo di colorazione. (D) Immagine della tabella ottenuta con le statistiche della colorazione CFSE, che mostra il numero di uEV nel campione. Clicca qui per visualizzare una versione più grande di questa figura.
- Fare clic su Tubo di autofluorescenza. Nel dot plot SSC-H VS FSC-H con i gate delle dimensioni, crea una regione che includa tutte le dimensioni. Fare clic su Nuova regione. Impostare la regione positiva (vedere Figura 6A,B).
- Applicare i gate a tutti i campioni. Seleziona i cancelli, trascina e rilascia nell'opzione Tutti i campioni .
- Fare clic su CFSE + uEVs tube. Aprire la nuova area e verificare la regione positiva per CFSE (FL1-H), come illustrato nella Figura 6C.
- Nell'area di lavoro della Figura 6D, copiare i dati delle # celle per il tubo. Questo numero è il #uEVs ottenuto mediante citometria a flusso (#uEVs FC).
- Applicare la seguente formula per calcolare il numero di uEV per microlitro:
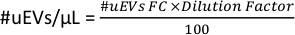
- Per avere il #uEVs per microlitro per ogni fluorocromo (#uEVs/μL FLX), nell'area di lavoro, copiare le statistiche per il sottoinsieme FLX-H, questo numero è la percentuale (sottoinsieme %FLX).
- Applicare la seguente formula per calcolare il numero di uEV per microlitro per ciascun fluorocromo.
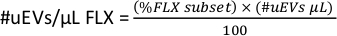
NOTA: A seconda che il gate selezionato sia per una taglia o per tutti gli uEV, verificare i dati selezionati.
Access restricted. Please log in or start a trial to view this content.
Risultati
Ci sono diversi punti di controllo attraverso il protocollo e prima della colorazione degli uEV. Pertanto, è essenziale verificare prima la quantità di proteine presenti nell'estratto di uEV. Tutti i gruppi di ricerca che lavorano con le vescicole extracellulari quantificano la proteina, come indicato nel passaggio 2.1. La Figura 2 supplementare mostra una piastra rappresentativa a 96 pozzetti contenente la frazione di uEV nei pozzetti 4E, 5E e 6E. I pozzetti 1A, 2A e ...
Access restricted. Please log in or start a trial to view this content.
Discussione
Al giorno d'oggi, l'uso di vescicole extracellulari come biomarcatori per diverse malattie è aumentato, soprattutto per quelle che possono essere isolate da fonti non invasive come l'urina 5,21,22,23,24. È stato dimostrato che l'isolamento delle uEV è una risorsa vitale per conoscere lo stato di un individuo sano e la diag...
Access restricted. Please log in or start a trial to view this content.
Divulgazioni
Gli autori dichiarano che la ricerca è stata condotta in assenza di qualsiasi relazione finanziaria o commerciale che possa essere interpretata come un potenziale conflitto di interessi.
Riconoscimenti
Questo lavoro è stato sostenuto da sovvenzioni di CONACyT (A3-S-36875) e del programma UNAM-DGAPA-PAPIIT (IN213020 e IA202318). NH-I è stato supportato dalla borsa di studio 587790 di CONACyT.
Gli autori ringraziano Leopoldo Flores-Romo†, Vianney Ortiz-Navarrete, Antony Boucard Jr e Diana Gómez-Martin per i loro preziosi consigli per la realizzazione di questo protocollo, e a tutti gli individui sani per i loro campioni di urina.
Access restricted. Please log in or start a trial to view this content.
Materiali
| Name | Company | Catalog Number | Comments |
| APC anti human CD156c (ADAM10) antibody | BioLegend | 352706 | Add 5 µL to the 20 µL of uEVs in PBS |
| APC anti human TSPAN33 (BAAM) antibody | BioLegend | 395406 | Add 5 µL to the 20 µL of uEVs in PBS |
| Avanti centrifuge with JA-25.5O fixed angle rotor | Beckamn Coulter | J-26S XPI | |
| BD Accuri C6 Flow Cytometer | BD Biosciences | ||
| β-mercaptoethanol | SIGMA-Aldrich | M3148 | |
| Benchtop centrifuge with A-4-44 rotor | Eppendorf | 5804 | |
| BLUEstain 2 protein ladder | GOLDBIO | P008 | |
| CD9 (C-4) mouse monoclonal antibody | Santa Cruz Biotechnology | sc-13118 | |
| CD63 (MX-49.129.5) mouse monoclonal antibody | Santa Cruz Biotechnology | sc-5275 | |
| Cell Trace CFSE cell proliferation kit for flow cytometry | Thermo Scientific | C34554 | |
| Chemidoc XRS+ system | BIORAD | 5837 | |
| FITC anti human CD9 antibody | BioLegend | 312104 | Add 5 µL to the 20 µL of uEVs in PBS |
| FITC anti human CD37 antibody | BioLegend | 356304 | Add 5 µL to the 20 µL of uEVs in PBS |
| Fluorescent yellow particles | Spherotech | FP-0252-2 | |
| Fluorescent yellow particles | Spherotech | FP-0552-2 | |
| Fluorescent yellow particles | Spherotech | FP-1552-2 | |
| FlowJo Software | Becton, Dickinson and Company | ||
| Goat anti-mouse immunoglobulins/HRP | Dako | P0447 | |
| Halt protease inhibitor cocktail | Thermo Scientific | 78429 | |
| Immun-Blot PVDF membrane 0.22µm | BIORAD | 1620177 | |
| Megamix-Plus FSC beads | COSMO BIO CO.LTD | 7802 | |
| NuPAGE LDS sample buffer 4X | Thermo Scientific | NP0007 | |
| Optima ultracentrifuge with rotor 90Ti fixed angle 355530 | Beckamn Coulter | XPN100 | |
| Page Blue protein staining solution | Thermo Scientific | 24620 | |
| PE anti human CD53 antibody | BioLegend | 325406 | Add 5 µL to the 20 µL of uEVs in PBS |
| Pierce BCA Protein assay kit | Thermo Scientific | 23227 | |
| Pierce RIPA buffer | Thermo Scientific | 89900 | |
| Polycarbonate thick wall centrifuge tubes | Beckamn Coulter | 355630 | |
| Spherotech 8-Peak validation beads (FL1-FL3) | BD Accuri | 653144 | |
| Spherotech 6-Peak validation beads (FL4) | BD Accuri | 653145 | |
| Sucrose | SIGMA-Aldrich | 59378 | |
| Triethanolamine | SIGMA-Aldrich | 90279 |
Riferimenti
- Decramer, S., et al. Urine in clinical proteomics. Molecular & Cellular Proteomics. 7 (10), 1850-1862 (2008).
- Nawaz, M., et al. The emerging role of extracellular vesicles as biomarkers for urogenital cancers. Nature Reviews Urology. 11 (12), 688-701 (2014).
- Pisitkun, T., Johnstone, R., Knepper, M. A. Discovery of urinary biomarkers. Molecular & Cellular Proteomics. 5 (10), 1760-1771 (2006).
- Urinology Think Tank Writing Group. Urine: Waste product or biologically active tissue. Neurourology and Urodynamics. 37 (3), 1162-1168 (2018).
- Wang, S., Kojima, K., Mobley, J. A., West, A. B. Proteomic analysis of urinary extracellular vesicles reveal biomarkers for neurologic disease. EBioMedicine. 45, 351-361 (2019).
- Merchant, M. L., Rood, I. M., Deegens, J. K. J., Klein, J. B. Isolation and characterization of urinary extracellular vesicles: implications for biomarker discovery. International Urology and Nephrology. 13 (12), 731-749 (2017).
- Alvarez, M. L., Khosroheidari, M., Kanchi Ravi, R., DiStefano, J. K. Comparison of protein, microRNA, and mRNA yields using different methods of urinary exosome isolation for the discovery of kidney disease biomarkers. Kidney International. 82 (9), 1024-1032 (2012).
- Jing, H., et al. The role of extracellular vesicles in renal fibrosis. Cell Death and Disease. 10 (5), 367(2019).
- Liu, X., et al. Tubule-derived exosomes play a central role in fibroblast activation and kidney fibrosis. Kidney International. 97 (6), 1181-1195 (2020).
- Quaglia, M., et al. Extracellular Vesicles as Mediators of Cellular Crosstalk Between Immune System and Kidney Graft. Frontiers in Immunology. 11, 74(2020).
- Simpson, R. J., Lim, J. W., Moritz, R. L., Mathivanan, S. Exosomes: proteomic insights and diagnostic potential. Expert Review of Proteomics. 6 (3), 267-283 (2009).
- Street, J. M., Koritzinsky, E. H., Glispie, D. M., Star, R. A., Yuen, P. S. Urine Exosomes: An Emerging Trove of Biomarkers. Advances in Clinical Chemistry. 78, 103-122 (2017).
- Thongboonkerd, V. Roles for Exosome in Various Kidney Diseases and Disorders. Frontiers in Pharmacology. 10, 1655(2019).
- Watanabe, Y., et al. Molecular Network Analysis of the Urinary Proteome of Alzheimer's Disease Patients. Dementia and Geriatric Cognitive Disorders Extra. 9 (1), 53-65 (2019).
- Jadli, A. S., Ballasy, N., Edalat, P., Patel, V. B. Inside(sight) of tiny communicator: exosome biogenesis, secretion, and uptake. Molecular and Cellular Biochemistry. 467 (1-2), 77-94 (2020).
- Svenningsen, P., Sabaratnam, R., Jensen, B. L. Urinary extracellular vesicles: Origin, role as intercellular messengers and biomarkers; efficient sorting and potential treatment options. Acta Physiologica. 228 (1), 13346(2020).
- Witwer, K. W., Thery, C. Extracellular vesicles or exosomes? On primacy, precision, and popularity influencing a choice of nomenclature. Journal of Extracellular Vesicles. 8 (1), 1648167(2019).
- Thery, C., et al. Minimal information for studies of extracellular vesicles 2018 (MISEV2018): a position statement of the International Society for Extracellular Vesicles and update of the MISEV2014 guidelines. Journal of Extracellular Vesicles. 7 (1), 1535750(2018).
- Perez-Hernandez, J., et al. Increased Urinary Exosomal MicroRNAs in Patients with Systemic Lupus Erythematosus. PLoS One. 10 (9), 0138618(2015).
- Freyssinet, J. M., Toti, F. Membrane microparticle determination: at least seeing what's being sized. Journal of Thrombosis and Haemostasis. 8 (2), 311-314 (2010).
- D'Anca, M., et al. Exosome Determinants of Physiological Aging and Age-Related Neurodegenerative Diseases. Frontiers in Aging Neuroscience. 11, 232(2019).
- Lawson, C., Vicencio, J. M., Yellon, D. M., Davidson, S. M. Microvesicles and exosomes: new players in metabolic and cardiovascular disease. Journal of Endocrinology. 228 (2), 57-71 (2016).
- Lee, S., Mankhong, S., Kang, J. H. Extracellular Vesicle as a Source of Alzheimer's Biomarkers: Opportunities and Challenges. International Journal of Molecular Sciences. 20 (7), (2019).
- Mori, M. A., Ludwig, R. G., Garcia-Martin, R., Brandao, B. B., Kahn, C. R. Extracellular miRNAs: From Biomarkers to Mediators of Physiology and Disease. Cell Metabolism. 30 (4), 656-673 (2019).
- Hoorn, E. J., et al. Prospects for urinary proteomics: exosomes as a source of urinary biomarkers. Nephrology (Carlton). 10 (3), 283-290 (2005).
- Ranghino, A., Dimuccio, V., Papadimitriou, E., Bussolati, B. Extracellular vesicles in the urine: markers and mediators of tissue damage and regeneration. Clinical Kidney Journal. 8 (1), 23-30 (2015).
- Zhang, W., et al. Extracellular vesicles in diagnosis and therapy of kidney diseases. American Journal of Physiology-Renal Physiology. 311 (5), 844-851 (2016).
- Fernandez-Llama, P., et al. Tamm-Horsfall protein and urinary exosome isolation. Kidney International. 77 (8), 736-742 (2010).
- Royo, F., et al. Different EV enrichment methods suitable for clinical settings yield different subpopulations of urinary extracellular vesicles from human samples. Journal of Extracellular Vesicles. 5, 29497(2016).
- Rider, M. A., Hurwitz, S. N., Meckes, D. G. ExtraPEG: A Polyethylene Glycol-Based Method for Enrichment of Extracellular Vesicles. Scientific Reports. 6, 23978(2016).
- Lv, C. Y., et al. A PEG-based method for the isolation of urinary exosomes and its application in renal fibrosis diagnostics using cargo miR-29c and miR-21 analysis. International Urology and Nephrology. 50 (5), 973-982 (2018).
- Alvarez, M. L., Khosroheidari, M., Kanchi Ravi, R., DiStefano, J. K. Comparison of protein, microRNA, and mRNA yields using different methods of urinary exosome isolation for the discovery of kidney disease biomarkers. Kidney International. 82 (9), 1024-1032 (2012).
- Wang, D., Sun, W. Urinary extracellular microvesicles: isolation methods and prospects for urinary proteome. Proteomics. 14 (16), 1922-1932 (2014).
- Pospichalova, V., et al. Simplified protocol for flow cytometry analysis of fluorescently labeled exosomes and microvesicles using dedicated flow cytometer. Journal of Extracellular Vesicles. 4, 25530(2015).
- van der Pol, E., et al. Standardization of extracellular vesicle measurements by flow cytometry through vesicle diameter approximation. Journal of Thrombosis and Haemostasis. 16 (6), 1236-1245 (2018).
- Huyan, T., Du, Y., Huang, Q., Huang, Q., Li, Q. Uptake Characterization of Tumor Cell-derived Exosomes by Natural Killer Cells. Iranian Journal of Public Health. 47 (6), 803-813 (2018).
- Puzar Dominkus, P., et al. PKH26 labeling of extracellular vesicles: Characterization and cellular internalization of contaminating PKH26 nanoparticles. Biochimica et Biophysica Acta (BBA) - Biomembranes. 1860 (6), 1350-1361 (2018).
- Morales-Kastresana, A., et al. Labeling Extracellular Vesicles for Nanoscale Flow Cytometry. Scientific Reports. 7 (1), 1878(2017).
- de Rond, L., et al. Comparison of Generic Fluorescent Markers for Detection of Extracellular Vesicles by Flow Cytometry. Clinical Chemistry. 64 (4), 680-689 (2018).
Access restricted. Please log in or start a trial to view this content.
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon