É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Determinação do Tamanho e Análise Fenotípica de Vesículas Extracelulares Urinárias por Citometria de Fluxo
Neste Artigo
Resumo
Este protocolo descreve um método para o isolamento de vesículas extracelulares urinárias, uEVs, de doadores humanos saudáveis e sua caracterização fenotípica pela expressão de marcadores de tamanho e superfície usando citometria de fluxo.
Resumo
As vesículas extracelulares, EVs, são um complexo heterogêneo de membranas lipídicas, secretadas por qualquer tipo de célula, em qualquer fluido, como a urina. Os EVs podem ser de diferentes tamanhos, variando de 40-100 nm de diâmetro, como em exossomos, a 100-1000 nm em microvesículas. Eles também podem conter diferentes moléculas que podem ser usadas como biomarcadores para o prognóstico e diagnóstico de muitas doenças. Muitas técnicas foram desenvolvidas para caracterizar essas vesículas. Uma delas é a citometria de fluxo. No entanto, não há relatórios existentes para mostrar como quantificar a concentração de EVs e diferenciá-los por tamanho, juntamente com a detecção de biomarcadores. Este trabalho tem como objetivo descrever um procedimento para o isolamento, quantificação e fenotipificação de vesículas extracelulares urinárias, uEVs, utilizando um citômetro convencional para a análise sem qualquer modificação em sua configuração. As limitações do método incluem a coloração de no máximo quatro biomarcadores diferentes por amostra. O método também é limitado pela quantidade de EVs disponíveis na amostra. Apesar dessas limitações, com este protocolo e sua posterior análise, podemos obter mais informações sobre o enriquecimento dos marcadores EVs e a abundância dessas vesículas presentes em amostras de urina, em doenças envolvendo danos renais e cerebrais.
Introdução
Nos mamíferos, o sangue é filtrado passando pelos rins 250 a 300 vezes; Durante esse período, a urina é formada. A produção desse biofluido é o resultado de uma série de processos, incluindo filtração glomerular, reabsorção tubular e secreção. Resíduos metabólicos e eletrólitos são os principais componentes da urina. Além disso, outros subprodutos, como peptídeos, proteínas funcionais e vesículas extracelulares (EVs), são excretados 1,2,3,4,5,6. Inicialmente, vesículas extracelulares urinárias (uEVs) foram identificadas em amostras de urina de pacientes que sofriam de distúrbios do equilíbrio hídrico. Esses pacientes apresentaram a presença de moléculas como a aquaporina-2 (AQP2), que foi então utilizada como biomarcador para essa doença7. Vários estudos subsequentes se concentraram na identificação da origem celular dos uEVs, descrevendo que essas estruturas podem ser secretadas por células renais (glomérulo, podócitos, etc.) e outros tipos de células de linhagens endoteliais ou leucocíticas. Além disso, o número e o enriquecimento molecular em uEVs podem se correlacionar com o status de muitas doenças e distúrbios 8,9,10,11,12,13,14.
Ao todo, os EVs compõem uma família altamente heterogênea de partículas envolvidas por bicamadas lipídicas e liberadas pelas células por meio de mecanismos passivos ou ativos em diferentes fluidos. Dependendo de sua origem, os EVs podem ser classificados como exossomos originados em endossomas ou microvesículas/micropartículas derivadas da membrana plasmática. No entanto, esse critério de classificação só pode ser aplicado quando a biogênese das partículas é observada diretamente. Portanto, outros critérios não triviais, incluindo origem física, bioquímica e celular, foram endossados por vários pesquisadores da área 15,16,17. Dependendo da natureza do isolado analisado, diferentes técnicas analíticas foram sugeridas para a caracterização das VEs. Por exemplo, com base no enriquecimento de EVs grandes (≥100 nm) ou pequenos (≤100 nm), sugere-se quantificação via citometria de fluxo ou rastreamento de nanopartículas, respectivamente18.
Atualmente, o uso de EVs como biomarcadores para muitas doenças tornou-se relevante, por isso a busca por diferentes fontes está sendo investigada. Uma das fontes mais promissoras é a urina, pois pode ser obtida de maneira fácil e não invasiva. Portanto, este protocolo descreve um procedimento para o isolamento de uEVs por centrifugação diferencial, processamento com anticorpos conjugados com fluorocromo e análise a jusante usando um citômetro convencional de 2 lasers/4 cores.
Protocolo
As amostras de urina humana foram obtidas de voluntários saudáveis que assinaram o consentimento informado do doador. Esses procedimentos também foram aprovados pelo Comitê de Ética em Pesquisa do Instituto Nacional de Ciencias Médicas y Nutrición Salvador Zubirán.
1. Isolamento de vesículas extracelulares urinárias
NOTA: O protocolo de isolamento de uEVs é modificado a partir da ref.19. A Figura 1 mostra a representação do protocolo para isolar uEVs.
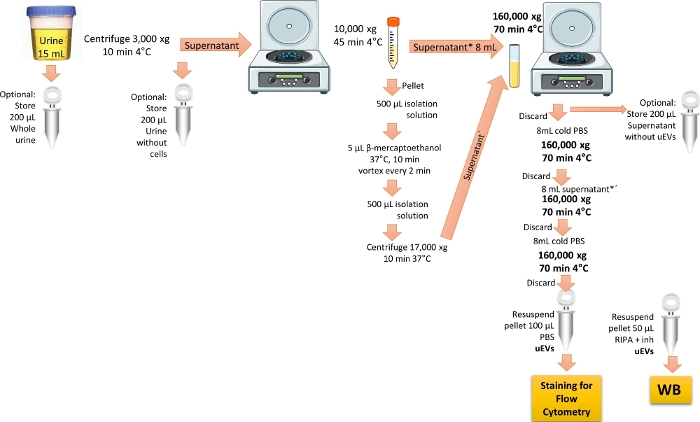
Figura 1: Visão geral do isolamento de uEVs para análise de citometria de fluxo. Neste protocolo, primeiro centrifugue a primeira urina do dia para remover as células e detritos. Em seguida, centrifugue para remover as grandes vesículas com tratamento para remover a proteína THP e, finalmente, realize a ultracentrifugação para enriquecer e obter os uEVs com uma única lavagem. As etapas para manter as frações de urina para a validação do WB são marcadas. Clique aqui para ver uma versão maior desta figura.
- Use a urina da primeira manhã (15 mL) de voluntários saudáveis. Centrifugue a urina a 3.000 x g por 10 min a 4 °C, para remover todas as células e detritos.
NOTA: Utilizar preferencialmente urina fresca; se não estiver disponível, use urina armazenada por um período máximo de 3 meses a -20 ° C ou armazenada por um período máximo de 6 meses a -70 ° C. Descongele a amostra de urina no gelo e vortex-a vigorosamente. Efectuar todos os procedimentos no gelo ou a 4 °C. Uma etapa opcional para a análise de Western blot é fazer um tubo duplicado e alíquota de 200 μL da primeira urina da manhã em um tubo separado. Armazene a amostra a -20 °C até o uso, rotulando-a como "urina inteira", para verificar se há marcadores de vesículas extracelulares. - Transferir o sobrenadante obtido no passo 1.1 para um novo tubo de centrifugação cónico de 15 ml e, em seguida, centrifugar a 10.000 x g durante 45 min a 4 °C. Transferir o sobrenadante para um tubo de ultracentrifugação de policarbonato de 8 ml e conservá-lo sobre gelo.
NOTA: Opcionalmente, alíquota de 200 μL do sobrenadante obtido na etapa 1.1 em um tubo separado rotulado como "urina sem células" para análise de Western blot.- Remoção da proteína de Tamm-Horsfall, THP
NOTA: O THP está presente na urina e é enriquecido quando um indivíduo tem doença renal. Foi relatado que o THP diminui o rendimento dos uEVs porque pode se ligar aos uEVs. Para remover essa proteína, é necessário o uso de um agente redutor 3,6.- Preparar a solução de isolamento misturando sacarose 250 mM com trietanolamina 10 mM. Ajuste o pH para 7,6.
- Misturar o sedimento obtido no passo 1.2 com 500 μl de solução de isolamento e, em seguida, adicionar 5 μl de β-mercaptoetanol.
- Incubar a mistura de pellets a 37 °C durante 10 min, agitando a cada 2 min. Adicione 500 μL de solução de isolamento e centrifugue a 17.000 x g por 10 min a 37 °C. Colete o sobrenadante obtido (contendo THP não agregado reduzido e uEVs).
- Remoção da proteína de Tamm-Horsfall, THP
- Misturar ambos os sobrenadantes reservados (dos passos 1.2 e 1.2.1.3) no mesmo tubo de ultracentrifugação de policarbonato de 8 ml e, em seguida, centrifugar a 160.000 x g durante 70 min a 4 °C, utilizando um rotor de ângulo fixo por ultracentrifugação.
NOTA: Opcionalmente, para análise de Western blot, armazene 200 μL do sobrenadante obtido acima, rotulando-o como "sobrenadante sem EVs". Esta amostra servirá como um controle negativo na busca de marcadores de vesículas extracelulares. - Descarte o sobrenadante. Adicione 1x PBS gelado ao tubo de policarbonato de 8 mL e centrifugue a 160.000 x g por 70 min a 4 °C.
NOTA: Tenha cuidado para não descartar o pellet, pois ele contém os uEVs. O 1x PBS precisa ser esterilizado e filtrado com pelo menos 0,22 μm de filtro de poro. - Descarte o sobrenadante. Adicione o resto do sobrenadante ao tubo de policarbonato de 8 ml e centrifugue a 160.000 x g durante 70 min a 4 °C.
- Descarte o sobrenadante e adicione 8 mL de PBS gelado ao pellet para lavar os uEVs.
- Descarte o sobrenadante. Deixe o pellet secar e ressuspenda-o com 1 mL de PBS gelado. Conservar a -70 °C até à utilização.
NOTA: Se estiver realizando a análise de Western blot, ressuspenda o pellet do tubo duplicado com 50 μL de tampão RIPA mais inibidores de protease. Conservar a -20 °C até à utilização.
2. Coloração de uEVs
NOTA: Antes da coloração e análise dos uEVs, é essencial realizar pelo menos uma metodologia recomendada pela MISEV201818 para verificar o isolamento adequado dos uEVs; aqui, a análise de Western blot é representada. A Figura 2 mostra um protocolo representativo para a coloração de uEVs.
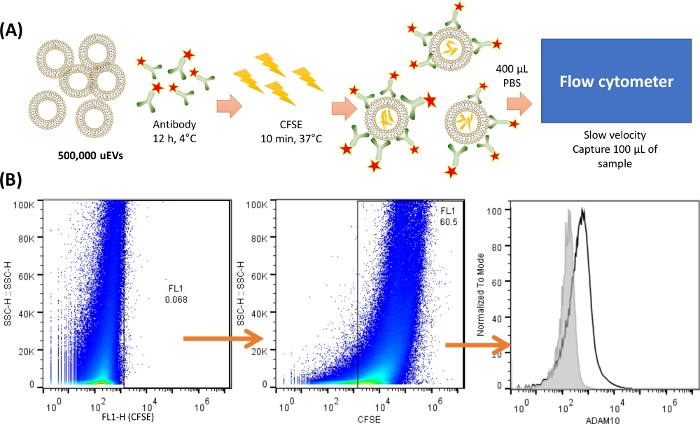
Figura 2: Visão geral da coloração e captura de uEVs no citômetro. (A) Representação da coloração uEV. Para 500.000 uEVs, o anticorpo foi misturado e incubado a 4 ° C por 12 h. Em seguida, o CFSE foi adicionado e incubado a 37 ° C por 10 min. Os uEVs tinham o CFSE dentro e o anticorpo se ligará à superfície do antígeno. 400 μL de PBS frio foram usados para ressuspender e capturar 100 μL da amostra no citômetro de fluxo em baixa velocidade. (B) Estratégia de análise. O primeiro gráfico de pontos (SSC-H VS FL-X) mostra o controle negativo para uEVs, seguido pelo gráfico de pontos mostrando uEVs corando com CFSE e, finalmente, um histograma com a coloração de anticorpos de uEVs (linha preta), o controle negativo é mostrado na linha cinza. Clique aqui para ver uma versão maior desta figura.
- Calcule o número relativo de uEVs quantificando o teor de proteína usando qualquer ensaio de proteína colorimétrico convencional, conforme mencionado na Tabela de Materiais. Realize uma diluição de 1:5 dos uEVs e siga as instruções da folha de dados fornecida no kit de proteínas do ensaio.
- Com base em uma fórmula20 relatada anteriormente, considere 1 μg/mL de proteína uEVs igual a 800.000 uEVs/μL. Certifique-se de que 500.000 uEVs estejam presentes em 20 μL de PBS gelado.
- Rotule os tubos, conforme indicado na Tabela 1.
NOTA: O número de tubos usados dependerá do número de anticorpos empregados, a limitação para citômetro de 2 lasers/4 cores é de no máximo 4 anticorpos por tubo que podem ser usados; portanto, apenas FL1, FL2, FL3 e FL4 no citômetro podem ser lidos, embora a compensação seja necessária. Para esses procedimentos, use tubos de microcentrífuga de 1,5 mL ou tubos de fundo redondo de 5 mL para citometria de fluxo. Os tubos necessários estão representados na Tabela 1. Os tubos 4 e 5 consistem em um coquetel de todos os anticorpos a serem usados neste protocolo. Dois tubos problemáticos (8 e 9) são dados como exemplo; Portanto, este conjunto de controles deve ter a combinação a ser usada em cada tubo.
| Tubo 1. | Miçangas Megamix FSC | |
| Tubo 2. | PBS | |
| Tubo 3. | PBS com CFSE | |
| Tubo 4. | PBS com todos os anticorpos do problema 1 | |
| Tubo 5. | PBS com todos os anticorpos do problema 2 | |
| Tubo 6. | Controle de autofluorescência | uEVs sem qualquer reagente, apenas em PBS. |
| Tubo 7. | #uEVs | uEVs com CFSE |
| Tubo 8. | Problema 1 | uEVs com CD37 FITC, CD53 PE, ADAM10 APC |
| Tubo 9. | Problema 2 | uEVs com FITC CD9, TSPAN33 APC |
Tabela 1: Rotulagem de tubos. Exemplo mostrando como rotular os tubos. Os primeiros tubos são todos os controles necessários. Os tubos com os anticorpos-fluorocromos dependerão da coloração.
- Adicione 20 μL de PBS contendo 500.000 uEVs aos tubos rotulados.
- Adicionar os anticorpos indicados no quadro 1, previamente titulados. Incubar durante a noite a 4 °C.
NOTA: Antes da coloração, recomenda-se centrifugar os anticorpos a 4 °C em velocidade máxima por pelo menos 5 min, para evitar os agregados. Os anticorpos usados aqui são um exemplo de proteínas presentes nos uEVs e pertencem a um manuscrito independente atualmente em preparação. - Adicione 0,4 μL de éster succinimidílico carboxifluoresceína (CFSE) [5 nM] ao tubo número 3. Incubar durante 10 min a 37 °C.
NOTA: CFSE É UM CORANTE USADO PARA CORAR TODOS OS EVs presentes em uma amostra e discriminar entre o ruído de fundo quando uma análise de citômetro de fluxo é realizada. Para obter mais informações, consulte a seção Discussão . - Adicione 400 μL de PBS 1x gelado a todos os tubos.
- Manter todos os tubos a 4 °C.
3. Aquisição de uEVs usando um citômetro convencional
NOTA: As instruções para o uso do citômetro de fluxo (consulte a Tabela de Materiais) são descritas aqui.
- Efectuar a calibração da qualidade do citómetro, utilizando os grânulos de 6 e 8 picos.
NOTA: O %CV do último pico deve ser um valor inferior a 6; O técnico do citômetro cuidará disso. - Abra o software do citômetro de fluxo.
NOTA: Assim que o software estiver aberto, um novo experimento será aberto, mostrando uma triagem com um modelo para 96 amostras, os parâmetros para execução e uma seção "vazia" para criar gráficos de pontos ou histogramas. - Ajuste os parâmetros na tela do software que está sendo executado: 100 μL de amostra para capturar, execução lenta, defina o limite em 10.000 no FSC-H e 2.000 no SSC-H.
NOTA: O limite recomendado aqui é um exemplo; É necessário definir o limite com base nas amostras analisadas. - Tubo de carga número 1 (tubo Megamix), conforme indicado na Tabela 1. Capture as contas. Crie dois gráficos de pontos na parte "vazia" da tela do software clicando na opção Dot Plot . Crie como painel A e B da Figura 3.
NOTA: As esferas fluorescentes Megamix (esferas usadas para delimitar os tamanhos de 0,1, 0,3, 0,5 e 0,9 μm e criar o modelo para tamanhos) podem ser analisadas usando o detector FL1 (FITC). Todos os gráficos de pontos e histogramas precisam ser exibidos em valores de altura. Registre o maior número possível de eventos; Qualquer modificação na planilha não altera os dados.
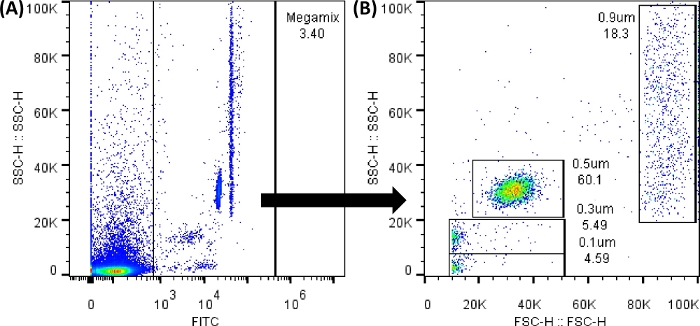
Figura 3: Gráficos de pontos de contas Megamix FSC. Os gráficos de pontos mostrados foram gerados usando o software do citômetro de fluxo; No citômetro de fluxo, a imagem será muito semelhante. (A) O primeiro gráfico de pontos gerado para selecionar as contas evitando o ruído de fundo. (SSC-H VS FL1-H). (B) O gráfico de pontos gerado pela seleção do portão anterior, mostrando os diferentes tamanhos das contas. (SSC-H VS FSC-H). Clique aqui para ver uma versão maior desta figura.
- Na seção "vazia" da tela do software, crie um gráfico de pontos e histograma para cada fluorocromo usado; use o algoritmo na Figura Suplementar 1 como exemplo. Para fazer isso, clique na opção Dot Plot ou Histograma para cada fluorocromo.
NOTA: O ruído de fundo estará sempre presente, conforme mostrado na Figura 3A. Todos os pontos entre 0 a 1.000 de FL1 (eixo x) estão fora da porta criada. Portanto, é essencial usar todos os tubos de controle para obter resultados adequados. Entre cada carregamento de tubo, lave novamente o fluido da bainha e execute um ciclo de desentupimento. Agite suavemente todos os tubos antes de carregar. Depois de carregar cinco tubos consecutivos, execute 100 μL de PBS 1x. - Tubos de carga número 2 e 5, para definir os valores de corte (negativos). Para fazer isso, selecione o ícone Rectangle Gate (localizado sob o gráfico de pontos criado) ou o ícone Line Gate (localizado sob o histograma criado); e coloque-o onde não haja sinal, a fim de obter gráficos de pontos e histogramas como os mostrados na Figura Suplementar 1.
NOTA: Todos esses tubos requerem a colocação da região positiva não superior a 0,70%, pois não haverá "fluorescência" superior a 103 (1.500). Se houver fluorescência além dessa região, dilua os reagentes usados. Portanto, é importante titular os reagentes antes das medições finais. - Carregue o tubo número 6 (tubo de autofluorescência), para definir as regiões negativas para a amostra. Para fazer isso, selecione o ícone Rectangle Gate (localizado sob o gráfico de pontos criado) ou o ícone Line Gate (localizado sob o histograma criado); coloque-o onde não haja sinal.
- Carregue os próximos tubos (tubos 7 a 9, representados na Tabela 1).
- Salve o experimento.
NOTA: Para o software do citômetro usado aqui, o comando Save Experiment, refere-se a salvar os dados gerados. Em outras palavras, todos os tubos que são adquiridos pelo citômetro de fluxo e os dados gerados para cada tubo precisam ser salvos. Para fazer isso, é necessário clicar no botão Salvar experimento . - Exporte dados como arquivos FSC.
4. Análise dos dados com um software de citômetro de fluxo.
NOTA: As instruções para usar o software do citômetro de fluxo representado na tabela de materiais são descritas nesta seção. A Figura 4 mostra o espaço de trabalho com as etapas para criar os portões de tamanho.
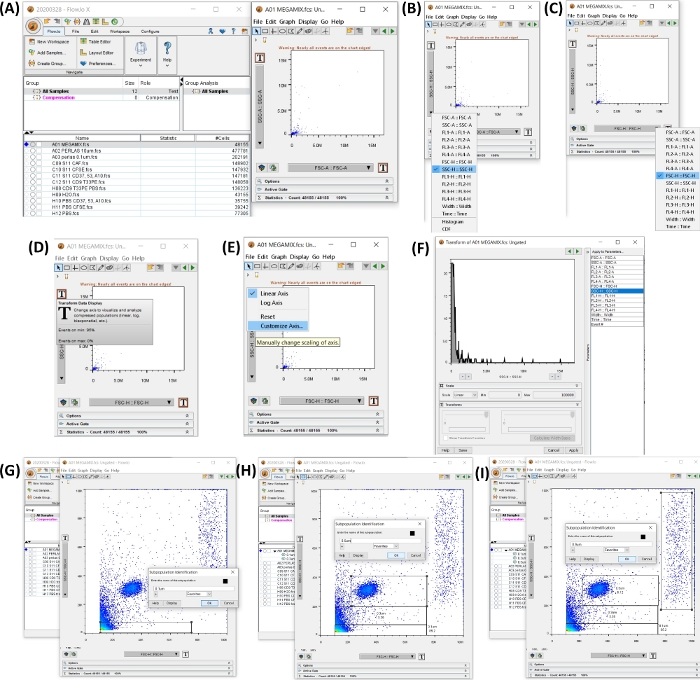
Figura 4: Área de trabalho com todas as etapas para iniciar a análise dos dados. Todas as imagens foram geradas por serigrafia do espaço de trabalho. (A) Área de trabalho gerada com os dados de amostra adicionados (esquerda), gráfico de pontos gerado pela seleção do tubo 1, contas FSC, (direita). (B,C) mostram a modificação do eixo, para ter SSC-H e FSC-H. (D-F) mostra a personalização passo a passo de ambos os eixos. (G-I) mostram a seleção e geração dos diferentes tamanhos de contas. Clique aqui para ver uma versão maior desta figura.
- Abra o software do citômetro de fluxo. Adicione as amostras, clicando no botão Adicionar amostras , e selecione os arquivos FCS exportados.
- Clique no tubo Megamix para abrir um gráfico de pontos SSC-H (eixo y) VS FSC-H (eixo x). Ajuste ambos os eixos para view 0 a 100.000 clicando no botão T (Transform Data Display), clique em Customize Axis, clique em SSC-H ou FSSC-H e altere o valor da escala para min 0 e max 100.000 (veja a Figura 4A-F).
- Defina as portas para 0,1, 0,3, 0,5 e 0,9 μm, no gráfico de pontos gerado na etapa 4.2. Selecione o botão Rectangle Gate para criar um portão, conforme ilustrado na Figura 4G-I.
NOTA: A Figura 5 mostra a área de trabalho com as etapas para criar as regiões positivas e obter a intensidade média para o fluorocromo.

Figura 5: Espaço de trabalho para analisar os dados obtidos. Todas as imagens foram geradas pela serigrafia do espaço de trabalho. (A) Espaço de trabalho gerado com o portão de tamanho aplicado a todas as amostras. (B) Tubo de autofluorescência selecionado, gráfico de pontos mostrando a porta de tamanho e o histograma para um tamanho selecionado (0,1 μm), use este histograma para obter a porta positiva para cada fluorocromo e tamanho. (C) Espaço de trabalho gerado com as portas positivas para cada fluorocromo e tamanho. (D) Espaço de trabalho (esquerda) e Editor de layout (direita) gerados para as amostras. No Editor de Layout é mostrado o histograma para tubo de autofluorescência e tubo positivo para FL1-H, e como obter o painel de propriedades para modificá-los. (E) A imagem mostra como obter o valor médio de fluorescência de intensidade. (F) Histogramas gerados para três fluorocromos diferentes, mostrando todas as alterações que o software permite fazer com as informações estatísticas. Clique aqui para ver uma versão maior desta figura.
- Aplique as portas geradas (0,1, 0,3, 0,5 e 0,9 μm) a todas as amostras. Selecione Gates, arraste e solte na opção All Samples , conforme ilustrado na Figura 5A.
- Clique no tubo de autofluorescência. Defina as regiões positivas para cada fluorocromo, para cada tamanho. Abra o gráfico de pontos SSC-H VS FSC-H com as portas de tamanho, clique em Tamanho de porta única. Na nova janela, clique no eixo y para selecionar a opção de histograma. No eixo x, selecione FL1-H e, em seguida, selecione o ícone Intervalo para criar a região positiva. Repita a operação para FL2-H e FL4-H (consulte a Figura 5B).
- Aplique as portas a todas as amostras. Selecione Gates, arraste e solte na opção All Samples , conforme ilustrado na Figura 5C.
- Abra o Editor de layout.
- Arraste e solte cada amostra no editor. Selecione o tamanho do tubo de "autofluorescência", arraste e solte, depois selecione o mesmo tamanho do tubo manchado, arraste e solte.
- Clique na parte inferior direita do histograma. Abra o menu Propriedades . Clique em Legenda. Adicione a intensidade média para o fluorocromo.
NOTA: Em Propriedades, existem outras ferramentas disponíveis para modificar a aparência do histograma. - Repita o mesmo procedimento para os fluorocromos e tamanhos restantes.
5. Análise para obter o número de uEVs por amostra.
NOTA: A Figura 6 mostra o espaço de trabalho com as etapas para obter o número de uEVs por amostra.
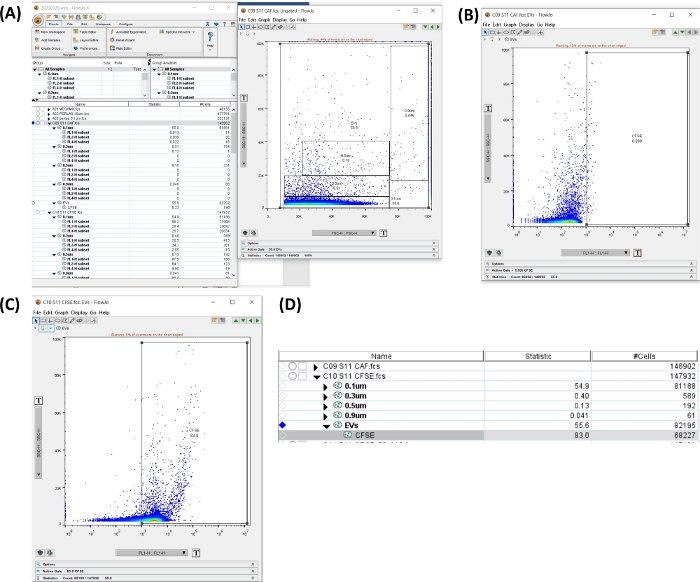
Figura 6: Espaço de trabalho para analisar o tubo CFSE. Todas as imagens foram geradas por serigrafia do espaço de trabalho. (A) Área de trabalho gerada pela seleção de todos os tamanhos da região, total de uEVs (esquerda), gráfico de pontos mostrando a porta selecionada (direita). (B) Gráfico de pontos SSC-H VS FL1-H para a região negativa CFSE no tubo de autofluorescência. (C) Gráfico de pontos SSC-H VS FL1-H para CFSE no tubo de coloração. (D) Imagem da tabela obtida com as estatísticas da coloração CFSE, mostrando o número de uEVs na amostra. Clique aqui para ver uma versão maior desta figura.
- Clique em Tubo de autofluorescência. No gráfico de pontos SSC-H VS FSC-H com as portas de tamanho, crie uma região incluindo todos os tamanhos. Clique em Nova Região. Defina a região positiva (veja a Figura 6A,B).
- Aplique as portas a todas as amostras. Selecione as portas, arraste e solte na opção Todas as amostras .
- Clique no tubo CFSE + uEVs. Abra a nova região e verifique a região positiva para CFSE (FL1-H), conforme ilustrado na Figura 6C.
- Na área de trabalho na Figura 6D, copie os dados # cells para o tubo. Este número é o #uEVs obtido por citometria de fluxo (#uEVs FC).
- Aplique a seguinte fórmula para calcular o número de uEVs por microlitro:
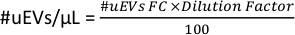
- Para ter o #uEVs por microlitro para cada fluorocromo (#uEVs/μL FLX), no espaço de trabalho, copie as estatísticas do subconjunto FLX-H, esse número é a porcentagem (subconjunto %FLX).
- Aplique a seguinte fórmula para calcular o número de uEVs por microlitro para cada fluorocromo.
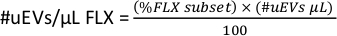
NOTA: Dependendo se a porta selecionada será para um tamanho ou para todos os uEVs, verifique os dados selecionados.
Resultados
Existem vários pontos de verificação através do protocolo e antes da coloração de uEVs. Portanto, é essencial verificar primeiro a quantidade de proteína presente no extrato de uEVs. Todos os grupos de pesquisa que trabalham com vesículas extracelulares quantificam a proteína, conforme indicado na etapa 2.1. A Figura 2 suplementar mostra uma placa representativa de 96 poços contendo a fração de uEVs nos poços 4E, 5E e 6E. Os poços 1A, 2A e 3A consistem em ...
Discussão
Atualmente, o uso de vesículas extracelulares como biomarcadores para diversas doenças tem aumentado, principalmente para aquelas que podem ser isoladas de fontes não invasivas, como a urina5,21,22,23,24. Está comprovado que o isolamento de EVs é um recurso vital para conhecer o estado de um indivíduo saudável e o diagn...
Divulgações
Os autores declaram que a pesquisa foi conduzida na ausência de qualquer relação financeira ou comercial que pudesse ser interpretada como um potencial conflito de interesses.
Agradecimentos
Este trabalho foi apoiado por doações do CONACyT (A3-S-36875) e do Programa UNAM-DGAPA-PAPIIT (IN213020 e IA202318). NH-I foi apoiado pela bolsa 587790 do CONACyT.
Os autores querem agradecer a Leopoldo Flores-Romo†, Vianney Ortiz-Navarrete, Antony Boucard Jr e Diana Gómez-Martin por seus valiosos conselhos para a realização deste protocolo, e a todos os indivíduos saudáveis por suas amostras de urina.
Materiais
| Name | Company | Catalog Number | Comments |
| APC anti human CD156c (ADAM10) antibody | BioLegend | 352706 | Add 5 µL to the 20 µL of uEVs in PBS |
| APC anti human TSPAN33 (BAAM) antibody | BioLegend | 395406 | Add 5 µL to the 20 µL of uEVs in PBS |
| Avanti centrifuge with JA-25.5O fixed angle rotor | Beckamn Coulter | J-26S XPI | |
| BD Accuri C6 Flow Cytometer | BD Biosciences | ||
| β-mercaptoethanol | SIGMA-Aldrich | M3148 | |
| Benchtop centrifuge with A-4-44 rotor | Eppendorf | 5804 | |
| BLUEstain 2 protein ladder | GOLDBIO | P008 | |
| CD9 (C-4) mouse monoclonal antibody | Santa Cruz Biotechnology | sc-13118 | |
| CD63 (MX-49.129.5) mouse monoclonal antibody | Santa Cruz Biotechnology | sc-5275 | |
| Cell Trace CFSE cell proliferation kit for flow cytometry | Thermo Scientific | C34554 | |
| Chemidoc XRS+ system | BIORAD | 5837 | |
| FITC anti human CD9 antibody | BioLegend | 312104 | Add 5 µL to the 20 µL of uEVs in PBS |
| FITC anti human CD37 antibody | BioLegend | 356304 | Add 5 µL to the 20 µL of uEVs in PBS |
| Fluorescent yellow particles | Spherotech | FP-0252-2 | |
| Fluorescent yellow particles | Spherotech | FP-0552-2 | |
| Fluorescent yellow particles | Spherotech | FP-1552-2 | |
| FlowJo Software | Becton, Dickinson and Company | ||
| Goat anti-mouse immunoglobulins/HRP | Dako | P0447 | |
| Halt protease inhibitor cocktail | Thermo Scientific | 78429 | |
| Immun-Blot PVDF membrane 0.22µm | BIORAD | 1620177 | |
| Megamix-Plus FSC beads | COSMO BIO CO.LTD | 7802 | |
| NuPAGE LDS sample buffer 4X | Thermo Scientific | NP0007 | |
| Optima ultracentrifuge with rotor 90Ti fixed angle 355530 | Beckamn Coulter | XPN100 | |
| Page Blue protein staining solution | Thermo Scientific | 24620 | |
| PE anti human CD53 antibody | BioLegend | 325406 | Add 5 µL to the 20 µL of uEVs in PBS |
| Pierce BCA Protein assay kit | Thermo Scientific | 23227 | |
| Pierce RIPA buffer | Thermo Scientific | 89900 | |
| Polycarbonate thick wall centrifuge tubes | Beckamn Coulter | 355630 | |
| Spherotech 8-Peak validation beads (FL1-FL3) | BD Accuri | 653144 | |
| Spherotech 6-Peak validation beads (FL4) | BD Accuri | 653145 | |
| Sucrose | SIGMA-Aldrich | 59378 | |
| Triethanolamine | SIGMA-Aldrich | 90279 |
Referências
- Decramer, S., et al. Urine in clinical proteomics. Molecular & Cellular Proteomics. 7 (10), 1850-1862 (2008).
- Nawaz, M., et al. The emerging role of extracellular vesicles as biomarkers for urogenital cancers. Nature Reviews Urology. 11 (12), 688-701 (2014).
- Pisitkun, T., Johnstone, R., Knepper, M. A. Discovery of urinary biomarkers. Molecular & Cellular Proteomics. 5 (10), 1760-1771 (2006).
- Urinology Think Tank Writing Group. Urine: Waste product or biologically active tissue. Neurourology and Urodynamics. 37 (3), 1162-1168 (2018).
- Wang, S., Kojima, K., Mobley, J. A., West, A. B. Proteomic analysis of urinary extracellular vesicles reveal biomarkers for neurologic disease. EBioMedicine. 45, 351-361 (2019).
- Merchant, M. L., Rood, I. M., Deegens, J. K. J., Klein, J. B. Isolation and characterization of urinary extracellular vesicles: implications for biomarker discovery. International Urology and Nephrology. 13 (12), 731-749 (2017).
- Alvarez, M. L., Khosroheidari, M., Kanchi Ravi, R., DiStefano, J. K. Comparison of protein, microRNA, and mRNA yields using different methods of urinary exosome isolation for the discovery of kidney disease biomarkers. Kidney International. 82 (9), 1024-1032 (2012).
- Jing, H., et al. The role of extracellular vesicles in renal fibrosis. Cell Death and Disease. 10 (5), 367 (2019).
- Liu, X., et al. Tubule-derived exosomes play a central role in fibroblast activation and kidney fibrosis. Kidney International. 97 (6), 1181-1195 (2020).
- Quaglia, M., et al. Extracellular Vesicles as Mediators of Cellular Crosstalk Between Immune System and Kidney Graft. Frontiers in Immunology. 11, 74 (2020).
- Simpson, R. J., Lim, J. W., Moritz, R. L., Mathivanan, S. Exosomes: proteomic insights and diagnostic potential. Expert Review of Proteomics. 6 (3), 267-283 (2009).
- Street, J. M., Koritzinsky, E. H., Glispie, D. M., Star, R. A., Yuen, P. S. Urine Exosomes: An Emerging Trove of Biomarkers. Advances in Clinical Chemistry. 78, 103-122 (2017).
- Thongboonkerd, V. Roles for Exosome in Various Kidney Diseases and Disorders. Frontiers in Pharmacology. 10, 1655 (2019).
- Watanabe, Y., et al. Molecular Network Analysis of the Urinary Proteome of Alzheimer's Disease Patients. Dementia and Geriatric Cognitive Disorders Extra. 9 (1), 53-65 (2019).
- Jadli, A. S., Ballasy, N., Edalat, P., Patel, V. B. Inside(sight) of tiny communicator: exosome biogenesis, secretion, and uptake. Molecular and Cellular Biochemistry. 467 (1-2), 77-94 (2020).
- Svenningsen, P., Sabaratnam, R., Jensen, B. L. Urinary extracellular vesicles: Origin, role as intercellular messengers and biomarkers; efficient sorting and potential treatment options. Acta Physiologica. 228 (1), 13346 (2020).
- Witwer, K. W., Thery, C. Extracellular vesicles or exosomes? On primacy, precision, and popularity influencing a choice of nomenclature. Journal of Extracellular Vesicles. 8 (1), 1648167 (2019).
- Thery, C., et al. Minimal information for studies of extracellular vesicles 2018 (MISEV2018): a position statement of the International Society for Extracellular Vesicles and update of the MISEV2014 guidelines. Journal of Extracellular Vesicles. 7 (1), 1535750 (2018).
- Perez-Hernandez, J., et al. Increased Urinary Exosomal MicroRNAs in Patients with Systemic Lupus Erythematosus. PLoS One. 10 (9), 0138618 (2015).
- Freyssinet, J. M., Toti, F. Membrane microparticle determination: at least seeing what's being sized. Journal of Thrombosis and Haemostasis. 8 (2), 311-314 (2010).
- D'Anca, M., et al. Exosome Determinants of Physiological Aging and Age-Related Neurodegenerative Diseases. Frontiers in Aging Neuroscience. 11, 232 (2019).
- Lawson, C., Vicencio, J. M., Yellon, D. M., Davidson, S. M. Microvesicles and exosomes: new players in metabolic and cardiovascular disease. Journal of Endocrinology. 228 (2), 57-71 (2016).
- Lee, S., Mankhong, S., Kang, J. H. Extracellular Vesicle as a Source of Alzheimer's Biomarkers: Opportunities and Challenges. International Journal of Molecular Sciences. 20 (7), (2019).
- Mori, M. A., Ludwig, R. G., Garcia-Martin, R., Brandao, B. B., Kahn, C. R. Extracellular miRNAs: From Biomarkers to Mediators of Physiology and Disease. Cell Metabolism. 30 (4), 656-673 (2019).
- Hoorn, E. J., et al. Prospects for urinary proteomics: exosomes as a source of urinary biomarkers. Nephrology (Carlton). 10 (3), 283-290 (2005).
- Ranghino, A., Dimuccio, V., Papadimitriou, E., Bussolati, B. Extracellular vesicles in the urine: markers and mediators of tissue damage and regeneration. Clinical Kidney Journal. 8 (1), 23-30 (2015).
- Zhang, W., et al. Extracellular vesicles in diagnosis and therapy of kidney diseases. American Journal of Physiology-Renal Physiology. 311 (5), 844-851 (2016).
- Fernandez-Llama, P., et al. Tamm-Horsfall protein and urinary exosome isolation. Kidney International. 77 (8), 736-742 (2010).
- Royo, F., et al. Different EV enrichment methods suitable for clinical settings yield different subpopulations of urinary extracellular vesicles from human samples. Journal of Extracellular Vesicles. 5, 29497 (2016).
- Rider, M. A., Hurwitz, S. N., Meckes, D. G. ExtraPEG: A Polyethylene Glycol-Based Method for Enrichment of Extracellular Vesicles. Scientific Reports. 6, 23978 (2016).
- Lv, C. Y., et al. A PEG-based method for the isolation of urinary exosomes and its application in renal fibrosis diagnostics using cargo miR-29c and miR-21 analysis. International Urology and Nephrology. 50 (5), 973-982 (2018).
- Alvarez, M. L., Khosroheidari, M., Kanchi Ravi, R., DiStefano, J. K. Comparison of protein, microRNA, and mRNA yields using different methods of urinary exosome isolation for the discovery of kidney disease biomarkers. Kidney International. 82 (9), 1024-1032 (2012).
- Wang, D., Sun, W. Urinary extracellular microvesicles: isolation methods and prospects for urinary proteome. Proteomics. 14 (16), 1922-1932 (2014).
- Pospichalova, V., et al. Simplified protocol for flow cytometry analysis of fluorescently labeled exosomes and microvesicles using dedicated flow cytometer. Journal of Extracellular Vesicles. 4, 25530 (2015).
- van der Pol, E., et al. Standardization of extracellular vesicle measurements by flow cytometry through vesicle diameter approximation. Journal of Thrombosis and Haemostasis. 16 (6), 1236-1245 (2018).
- Huyan, T., Du, Y., Huang, Q., Huang, Q., Li, Q. Uptake Characterization of Tumor Cell-derived Exosomes by Natural Killer Cells. Iranian Journal of Public Health. 47 (6), 803-813 (2018).
- Puzar Dominkus, P., et al. PKH26 labeling of extracellular vesicles: Characterization and cellular internalization of contaminating PKH26 nanoparticles. Biochimica et Biophysica Acta (BBA) - Biomembranes. 1860 (6), 1350-1361 (2018).
- Morales-Kastresana, A., et al. Labeling Extracellular Vesicles for Nanoscale Flow Cytometry. Scientific Reports. 7 (1), 1878 (2017).
- de Rond, L., et al. Comparison of Generic Fluorescent Markers for Detection of Extracellular Vesicles by Flow Cytometry. Clinical Chemistry. 64 (4), 680-689 (2018).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados