Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Ein umfassendes hocheffizientes Protokoll für die Isolierung, Kultur, Polarisation und glykolytische Charakterisierung von Makrophagen aus dem Knochenmark
In diesem Artikel
Zusammenfassung
Dieses Protokoll bietet detaillierte und umfassende Methoden für die Isolierung, Kultur, Polarisation und Messung des glykolytischen Stoffwechselzustands von lebenden Makrophagen (BMDMs) aus dem Knochenmark. Dieses Dokument bietet Schritt-für-Schritt-Anleitungen mit realistischen visuellen Illustrationen für den Arbeitsablauf und die glykolytische Bewertung von BMDMs in Echtzeit.
Zusammenfassung
Makrophagen gehören zu den wichtigsten Antigen-präsentierenden Zellen. Viele Untergruppen von Makrophagen wurden mit einzigartigen metabolischen Signaturen identifiziert. Makrophagen werden allgemein in M1-ähnliche (entzündliche) und M2-ähnliche (entzündungshemmende) Subtypen eingeteilt. M1-ähnliche Makrophagen sind proinflammatorische Makrophagen, die durch LPS und/oder entzündungsfördernde Zytokine wie INF-γ, IL-12 und IL-2 aktiviert werden. M1-ähnliche polarisierte Makrophagen sind an verschiedenen Krankheiten beteiligt, indem sie die Abwehr des Wirts an eine Vielzahl von Bakterien und Viren vermitteln. Dies ist sehr wichtig, um LPS-induzierte M1-ähnliche Makrophagen und ihre Stoffwechselzustände bei entzündlichen Erkrankungen zu untersuchen. M2-ähnliche Makrophagen gelten als entzündungshemmende Makrophagen, die durch entzündungshemmende Zytokine und Stimulatoren aktiviert werden. Im proinflammatorischen Zustand zeigen Makrophagen eine erhöhte Glykolyse in der glykolytischen Funktion. Die glykolytische Funktion wurde aktiv im Zusammenhang mit der Glykolyse, der glykolytischen Kapazität, der glykolytischen Reserve, der kompensatorischen Glykolyse oder der nicht-glykolytischen Ansäuerung mit extrazellulären Flussanalysatoren (XF) untersucht.
In diesem Artikel wird gezeigt, wie die glykolytischen Zustände in Echtzeit mit leicht verständlichen Schritten bewertet werden können, wenn die aus dem Knochenmark stammenden Makrophagen (BMDMs) atmen, verbrauchen und Energie produzieren. Anhand spezifischer Inhibitoren und Aktivatoren der Glykolyse zeigen wir in diesem Protokoll, wie man einen systemischen und vollständigen Überblick über die glykolytischen Stoffwechselprozesse in den Zellen erhält und genauere und realistischere Ergebnisse liefert. Um mehrere glykolytische Phänotypen messen zu können, bieten wir eine einfache, sensitive, DNA-basierte Normalisierungsmethode zur Polarisationsbestimmung von BMDMs an. Die Kultivierung, Aktivierung/Polarisation und Identifizierung des Phänotyps und des Stoffwechselzustands der BMDMs sind entscheidende Techniken, die bei der Untersuchung vieler verschiedener Arten von Krankheiten helfen können.
In dieser Arbeit polarisierten wir die naiven M0-Makrophagen zu M1-ähnlichen und M2-ähnlichen Makrophagen mit LPS bzw. IL4 und maßen einen umfassenden Satz glykolytischer Parameter in BMDMs in Echtzeit und longitudinal über die Zeit, wobei wir extrazelluläre Flussanalysen und glykolytische Aktivatoren und Inhibitoren verwendeten.
Einleitung
Makrophagen sind eine der kritischsten Zellen des M1-ähnlichen angeborenen Immunsystems. Sie sind an der Beseitigung von Infektionskrankheiten, Phagozytose, Antigenpräsentation und Entzündungsregulation beteiligt2. Darüber hinaus sind Makrophagen erforderlich, um andere Immunzellen über verschiedene Zytokine, die sie freisetzen, zu regulieren3. Es gibt ein großes Spektrum an Makrophagen-Phänotypen4. Abhängig von den Signalen, denen Makrophagen ausgesetzt sind, polarisieren sie in Richtung unterschiedlicher Entzündungs- und Stoffwechselzustände5. Makrophagen manifestieren metabolische Veränderungen bei verschiedenen Krankheiten, je nachdem, in welchem Gewebe sich die Makrophagen befinden6. Polarisierte Makrophagen haben die Fähigkeit, ihren glykolytischen Stoffwechsel, ihren Lipidstoffwechsel, ihren Aminosäurestoffwechsel und ihre mitochondriale oxidative Phosphorylierung (OXPHOS) umzuprogrammieren oder umzuschalten7,8. Klassisch aktivierte M1-ähnliche Makrophagen und alternativ aktivierte M2-ähnliche Makrophagen sind die beiden am besten untersuchten Phänotypen von Makrophagen3. Nicht aktivierte ruhende Makrophagen werden als M0-Makrophagen bezeichnet. Die Polarisation von M0-Makrophagen in Richtung eines M1-ähnlichen Phänotyps kann durch Stimulation naiver BMDMs mit bakteriellen Lipopolysacchariden (LPS) induziert werden9. Der PI3K-AKT-mTOR-HIF1a-Signalweg kann in Makrophagen in Gegenwart von inflammatorischen Zytokinen, Interferon-gamma (IFN γ,) oder Tumornekrosefaktor (TNF)10 aktiviert werden. M1-ähnliche Makrophagen haben einen erhöhten Glykolysestoffwechsel, eine verringerte oxidative Phosphorylierung (OXPHOS) und produzieren entzündliche Zytokine, die an Infektions- und Entzündungskrankheiten beteiligt sind8. Andererseits kann die Polarisation in Richtung des M2-ähnlichen Phänotyps durch Interleukin (IL)-4, über die JAK-STAT-, PPAR- und AMPK-Signalwege oder durch (IL)-13 und TGFβ-Signalwege induziert werden11,12.
Im Gegensatz zu M1-ähnlichen Makrophagen haben M2-ähnliche Makrophagen eine verminderte Glykolyse und ein erhöhtes OXPHOS und sind an antiparasitären und gewebereparierenden Aktivitäten beteiligt 8,13. BMDMs sind ein weit verbreitetes System zur Untersuchung von Makrophagen, die aus Knochenmarkstammzellen gewonnen werden. Glykolyse und OXPHOS sind die beiden führenden Energieproduktionswege in den Zellen14. Basierend auf ihrer Mikroumgebung können BMDMs sich für einen dieser Wege entscheiden. Wechseln Sie in einigen Fällen von einem zum anderen oder verwenden Sie beide Wege14. In dieser Studie konzentrierten wir uns auf den Glykolysestoffwechsel in aktivierten proinflammatorischen Makrophagen. Wenn die Glukose im Zytoplasma in Pyruvat umgewandelt wird und dann Laktat wird, produzieren die Zellen Protonen im Medium, die eine Erhöhung der Versauerungsrate im umgebenden Medium von M1-ähnlichen Zellen verursachen5. Ein extrazellulärer Flussanalysator wurde verwendet, um die Versauerungsrate der Zellmedien zu messen. Die Ergebnisse werden als extrazelluläre Ansäuerungsrate (ECAR) oder als Protonenausflussrate angegeben.
Eine optimierte, schnelle und einfache Methode zum Zugriff auf die Glykolyseniveaus in polarisierten Makrophagen ist unerlässlich, um den glykolytischen Phänotyp, Metabolitenveränderungen und die Auswirkungen von Inhibitoren/Aktivatoren und Medikamenten auf die polarisierten Makrophagen zu bestimmen. Die in diesem Manuskript beschriebene Methode wurde optimiert, um Informationen über spezifische Glykolysefaktoren (Glykolyse, glykolytische Kapazität, glykolytische Reserve und nicht-glykolytische Ansäuerung) sowie über die metabolische Reprogrammierung des glykolytischen Stoffwechsels zu erhalten. Der in dieser Studie verwendete Inhibitor (2DG) zielt explizit auf den Glykolyseweg ab.
Dieses optimierte Protokoll wurde auf der Grundlage der Kombination eines veröffentlichten Protokolls16, der extrazellulären Flussanalyse von glykolytischen Assays in den Benutzerhandbüchern des Herstellers und der direkten Kommunikation mit den Forschungs- und Entwicklungswissenschaftlern des Herstellers modifiziert und verbessert.
Protokoll
Die Mäuse wurden gemäß den Richtlinien der Assessment and Accreditation of Laboratory Animal Care (AAALAC) und der American Association for Laboratory Animal Science (AALAS) und unter Verwendung von Protokollen, die vom Texas A&M University Institutional Animal Care and Use Committee (IACUC) genehmigt wurden, auf humane Weise getötet.
1. Knochenmarkentnahme von Mäusen und Kultivierung von BMDMs
- Opfern Sie die Maus (6-10 Wochen alte C57Bl/6 Mäuse waren in diesem Protokoll) und legen Sie sie auf die Bauchseite, schneiden Sie die Haut und die Bauchfellschicht ab und schälen Sie vorsichtig die Beine ab.
HINWEIS: Verwenden Sie die CO2 -Gaseinwirkung, um die Maus einzuschläfern. - Trennen Sie beide Hinterbeine von der Hüfte abwärts und achten Sie darauf, den Knochen nicht zu schneiden.
- Legen Sie das gesamte Bein in ein leeres konisches 50-ml-Röhrchen (mit den Füßen nach oben, um es später leicht herausziehen zu können) auf Eis und fahren Sie mit der Entnahme beider Beine aus der Maus fort.
2. Freilegung des Oberschenkelknochens
HINWEIS: Führen Sie die folgenden Schritte in einer Biosicherheitswerkbank durch.
- Entnehmen Sie den Oberschenkelknochen, indem Sie die Tibia von jedem Bein abschneiden und so viel Gewebe wie möglich um den Oberschenkelknochen herum mit einer Schere und Laborpapier entfernen.
- Legen Sie entnommene, "gereinigte" Oberschenkelknochen in eine 10 cm große Platte, die ein Stück Laborpapier enthält, das mit Gewebekulturmedium (TC) oder PBS getränkt ist. Lege sie auf Eis.
- Fahren Sie mit der Entnahme von Femuren fort und entfernen Sie Gewebe aus allen Femuren, bevor Sie mit der Spülphase fortfahren (Abbildung 1A).
3. Mark-Spülung
- Um das Knochenmark aus den Oberschenkelknochen zu spülen, verwenden Sie eine 3-ml-Spritze, die mit TC-Medium oder PBS gefüllt ist, mit einer 23G-Nadel. Füllen Sie die Spritze, bevor Sie das Knochenmark freilegen.
- Lege das Knochenmark mit einer Schere frei, indem du das Ende des Oberschenkelknochens an beiden Epiphysen durchschneidest.
- Führen Sie die Nadelspitze in den Oberschenkelknochen ein und spülen Sie das Mark vorsichtig in eine 10 cm dicke Schale.
- Führen Sie die Nadel durch die gesamte Länge des Oberschenkelknochens und spülen Sie, bis die Knochenfarbe weiß wird. Normalerweise kann das meiste Knochenmark mit 2-3 ml Medium ausgespült werden.
- Spülen Sie alle Oberschenkelknochen und das Knochenmark des Pools in der Schale. Verwende eine Nadel, um sichtbare Klumpen aufzubrechen. Das Mark in ein konisches 50-ml-Röhrchen abseihen (Abbildung 1A).
4. Erythrozyten-Lyse
- Mark bei 190 x g für 10 min schleudern. Aspirieren Sie den Überstand.
- Resuspendieren Sie das Pellet mit einer Pipette in 4 ml ACK-Lysepuffer. Lassen Sie den RBC-Lysepuffer 5 Minuten lang bei Raumtemperatur einwirken.
- Geben Sie 4 mL TC-Medium RPMI-C 10% (RPMI 1640-GlutaMAX), ergänzt mit 2-Mercaptoethanol, Gentamicin, Streptomycin und 10% FCS, in die Knochenmarksuspension und schleudern Sie bei 1300 x g für 10 min.
- Erneut abseihen, um Erythrozytenablagerungen zu entfernen, und zum Zählen in einem kleinen Volumen von RPMI-C 10 % wieder suspendieren.
- Zählen Sie Zellen mit einem Zellzähler (Abbildung 1B). Ein Vi-Cell Counter wurde verwendet, um die Anzahl und Lebensfähigkeit der Zellen in der Suspension zu bestimmen.
5. Anrichten und Kultur
- Geben Sie 10 ml RPMI-C 10% + 10 ng/ml m-CSF (Macrophage Colony Stimulating Factor, ein essentieller Regulator der Proliferation, Differenzierung und des Überlebens von Monozyten/Makrophagen) zu so vielen 10-cm-Platten wie gewünscht.
- Fügen Sie ein entsprechendes Volumen an gezählten Zellen hinzu, so dass jede 10-cm-Platte 1 x 106 Zellen enthält. Legen Sie die Platten in einen 37 °C heißen Inkubator (definiert als Tag 0).
- Geben Sie an Tag 3 vorsichtig 5 ml frisches RPMI-C 10% + 10 ng/mL M-CSF auf jede Platte.
HINWEIS: An Tag 7 sollten BMDMs für den Test bereit sein (Abbildung 1C).
6. Ernte von Tellern
- Verwenden Sie ein Lichtmikroskop, um zu bestätigen, dass die meisten Zellen an den Platten haften.
- Sanftes Ansaugen von Medien. Fügen Sie dann 3 mL PBS hinzu und schwenken Sie die Platte vorsichtig. Aspirieren Sie diese Vertiefung, um alle verbleibenden nicht adhärenten Zellen zu entfernen.
- Geben Sie 7-10 ml kaltes PBS in die Platte, waschen Sie mit einer P1000-Pipette den Boden der Platten und ernten Sie alle verbleibenden Zellen in ein Sammelröhrchen.
HINWEIS: Bewahren Sie die Röhrchen auf Eis auf, da Makrophagen sehr fest haften und an der Innenseite der Röhrchen haften. Würden die Zellen kalt bleiben, würden sie weniger fest haften. - Zentrifuge-, Zähl- und Plattenzellen für Experimente (Abbildung 1D). Mit Hilfe der Durchflusszytometrie sollten die resultierenden Zellen zu >95 % positiv für CD11b und F4/80 sein. (Die Makrophagenpolarisation wurde durch Färbung mit M1-ähnlichen Markern von CD38, TNF-a und MCP-1 und M2-ähnlichen Markern von CD206 bestimmt.
HINWEIS: Führen Sie die Schritte 6.1-6.3 in der Biosicherheitswerkbank und Schritt 6.4 auf dem Labortisch durch. Behalten Sie während des gesamten Verfahrens aseptische Techniken bei.
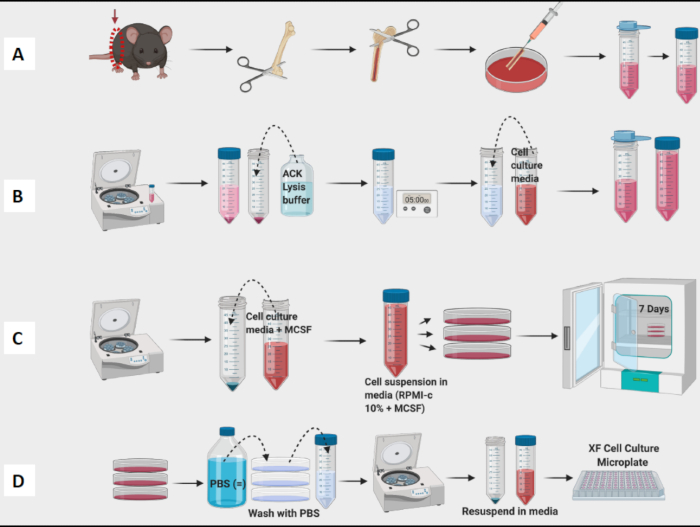
Abbildung 1: Grafischer Arbeitsablauf der Knochenmarkkultur von Mäusen mit BM-abgeleiteten Makrophagen. (A) Beinentnahme, Femur-Exposition und Knochenmarkspülung; (b) Erythrozyten-Lyse; (C) Beschichtung und Kultur; (D) Zellernte aus den Platten. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
7. Am Tag vor dem Metabolic Flux Analyzer Assay: Aussaat und Polarisation der Zellen für den glykolytischen Test
- Erwärmen Sie den Metabolic Flux Analyzer auf 37 °C, indem Sie das Gerät einschalten.
- Hydratisieren Sie eine Kartusche durch Zugabe von 200 μl einer kalibrierten Lösung und inkubieren Sie die Kartusche über Nacht in einem Nicht-CO2 -Inkubator (Abbildung 2A). Die Feuchtigkeit des Nicht-CO2 -Inkubators ist für die Hydratation der Kartusche nicht wichtig.
- Tauchen Sie die Platte eine Stunde vor dem Experiment einige Male auf und ab, um Luftblasen zu entfernen.
- Entwerfen Sie die Plattenkarte in der Software im Standard-Glykolyse-Stresstest – akute Injektion, indem Sie den Anweisungen des Tests folgen.
- Klicken Sie auf das Software-Symbol und dann auf Glykolyse-Stress-akuter Injektionstest. Generieren Sie auf dem Symbol für die Gruppendefinition Gruppennamen.
- Es gibt fünf Messzyklen mit einer Dauer von 18 Minuten und vier Injektionen. Ändern Sie die Injektion von Port A auf Glukose, Port B auf Oligomycin, Port C auf Rotenon und Antimycin A (Rot/AA) und Port D auf 2DG.
- Resuspendieren Sie die Zellen in RPMI-C 10% Medium und säen Sie 50k Zellen pro Well, mit Ausnahme der vier Ränder der Platte (A1, A12, H1 und H12; Geben Sie nur Medien hinzu, keine Zellen) in einer Mikroplatte mit Metabolic Flux Analyzer auf ein Endvolumen von 100 μl. Normalerweise sind mindestens 40.000 Zellen erforderlich, um diesen Assay durchzuführen.
- Lassen Sie die Zellen 45 Minuten bei Raumtemperatur ruhen, um den Kanteneffekt der Zellen zu vermeiden. Der Kanteneffekt tritt auf, wenn das Medium um den Umfang der Platte herum teilweise verdampft, was zu Volumen- und Konzentrationsänderungen führt und die Lebensfähigkeit der Zellen verringert.
- Fügen Sie 10 ng/ml LPS hinzu, um die naiven Makrophagen in Richtung M1-ähnlicher Zellen zu polarisieren, und fügen Sie 20 ng/ml IL-4 hinzu, um sie in Richtung M2-ähnlicher Zellen zu polarisieren. Verwenden Sie mindestens 3 bis 6 Wells pro Bedingung (Abbildung 2B).
- Überprüfen Sie die Zellen unter dem Mikroskop und legen Sie die Platte 24 Stunden lang bei 37 °C und 5 % CO2 in einen Inkubator.
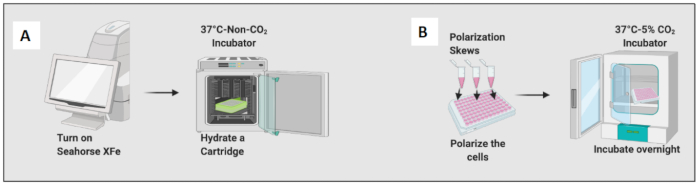
Abbildung 2: Grafische Demonstration der Aussaat und Polarisation der Zellen. (A) Einrichtung des extrazellulären Flussanalysators und Kartuschenhydratation; (B) Polarisation der Zellen und Inkubation über Nacht. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
8. Tag des Assays: Herstellung von XF Medium und Verbindung
- Ergänzen Sie 100 ml XF RPMI (pH 7,4) Testmedium mit 2 mM Glutamin.
- Filtersterilisieren Sie die Medien mit einem 0,2 μm Vakuumfiltersystem.
- Legen Sie das Testmedium für 20 Minuten in ein 37 °C heißes Wasserbad.
- Nehmen Sie die plattierten Zellen aus dem 37 °C und 5 % CO2 -Inkubator. Waschen Sie die Zellen zweimal mit Assay-Medien und ersetzen Sie das vorherige Medium durch Assay-Medien bis zum endgültigen Volumen von 180 μl.
- Stellen Sie mit einem Mikroskop sicher, dass alle Vertiefungen konfluente Zellen aufweisen, und markieren Sie alle Vertiefungen, die vom Pipettieren Kratzer aufweisen. Wenn Kratzer vorhanden sind, entfernen Sie diese Platte, bevor Sie sie analysieren.
- Stellen Sie die zellhaltige Platte für 45 Minuten in einen Nicht-CO2 -Inkubator (Abbildung 3A).
- Verwendung der Verbindungen und des Assay-Mediums zur Herstellung von Stammlösungen von Glucose (100 mM), Oligomycin (100 μM), Rot/AA (50 μM) und 2DG (500 mM) (Tabelle 1).
- Stellen Sie eine 10-fache Injektionsmischung jeder Verbindung unter Verwendung von Testmedien her (Tabelle 2).
| Injektionsschäfte (in den Kits enthalten) | Vollständiges Assay-Medium (ml) hinzufügen | Endkonzentration des Stammes (μM) |
| Traubenzucker | 3 | 100 Tsd. |
| Oligomycin | 0.72 | 100 |
| 2-GD | 3 | 100 Tsd. |
Tabelle 1. Injektions-Bestände
| Anschlüsse an der Patrone | Lagerlösungen | Hinzufügen des Lagervolumens | Hinzufügen von Assay-Medien | Endkonzentration der Injektionen (10x) | Fügen Sie dieses Volumen zum angegebenen Port hinzu (μL) | Endkonzentration nach der Injektion in jeder Vertiefung |
| Ein | Glukose (100 mM) | 3000 μL + 0 μL | ca. 100 mM | 20 | ca. 10 mM | |
| B | Oligomycin (100 μM) | 300 μL + 2700 μL | 10 μM | 22 | 1,0 μM | |
| C | Rotenon/ Antimycin A (50 μM) | 300 μL + 2700 μL | 5 μM | 25 | 0,5 μM | |
| D | 2-DG (500 mM) | 300 μL + 0 μL | 500 mM | 28 | 50 mM | |
Tabelle 2. Konzentrationen der abschließenden Injektion
9. Tag des Assays: Durchführung des akuten glykolytischen Tests an polarisierten Makrophagen
- Öffnen Sie die gespeicherte Vorlage für den Glykolyse-Stress-Assay (akute Injektion) aus der Software. Der standardmäßige akute Glyko-Stresstest hat eine 3-minütige Mischung und Messung vor jeder Injektion.
- Überprüfen Sie die Vorlage und die Assay-Details, und wenn Sie fertig sind, klicken Sie auf Ausführen und folgen Sie den Anweisungen des Standard-Assays. Alle Parameter können jedoch angepasst werden.
- Nehmen Sie die Sensorkassette aus dem Nicht-CO2 -Inkubator, entfernen Sie den Deckel und setzen Sie das Gerät so ein, dass die Vertiefung A1 der Kartuschenplatte in die obere linke Ecke der Einschubplatte des Geräts fällt. In der Regel dauert die Kalibrierung zwischen 20 und 45 Minuten.
- Nach Abschluss der Kalibrierung wirft das Gerät die Platte mit der Kalibrierungslösung aus und hält die Sensorpatrone. Entfernen Sie das Kalibrant, das die Platte enthält.
- Nehmen Sie die Zellplatte aus dem Nicht-CO2 -Inkubator, entfernen Sie den Plattendeckel und setzen Sie sie in die Maschine ein. Klicken Sie auf Ausführen (Abbildung 3B).
- Wenn der Assay abgeschlossen ist, wirft das Gerät die Zellplatte und die Sensorpatrone aus.
- Nehmen Sie das Medium von der Platte und frieren Sie es zur weiteren Normalisierung bei -20 °C ein.
- Verwenden Sie das kommerzielle Zellproliferations-Assay-Kit (z. B. CyQUANT) zur Normalisierung der Zellen.
- Fügen Sie 1 ml Verbindung B oder Lysepuffer zu 19 ml nukleasefreiem destilliertem Wasser hinzu.
- Geben Sie 100 μl der Arbeitslösung der Verbindung A oder GR zu der oben genannten Lösung.
- Stellen Sie sicher, dass die Zellen in der Platte aufgetaut sind, und geben Sie dann 200 μl der Lösung in jede Vertiefung.
- 5 min bei Raumtemperatur (RT) inkubieren.
- Messen Sie die Fluoreszenz in 480 nm Anregungs- und 520 nm Emissionswellenlängen mit einem Plattenleser.
- Normalisieren Sie die Zellen auf dem Normalisierungsfeld der Software.
- Normalisieren Sie Zellen basierend auf der Zellzahl naiver Makrophagen (Abbildung 3C). Betrachten Sie den Durchschnitt der naiven Makrophagen als 1 (indem Sie die Zellzahl jeder Vertiefung durch die durchschnittliche Zellzahl der naiven Makrophagen dividieren) und wenden Sie sie auf alle Makrophagen an.
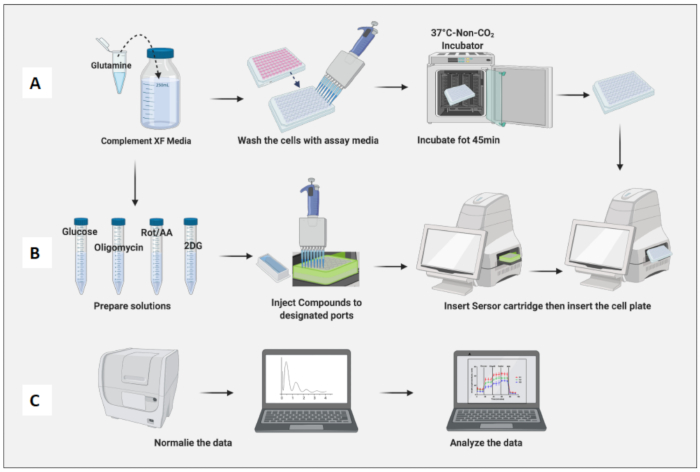
Abbildung 3: Tag des Assays: Vorbereitung des Mediums und der Verbindungen und Durchführung des Assays. (A) Vorbereitung der Zellen für den Assay; (B) Vorbereitung, Kalibrierung und Durchführung des Assays; (C) Normalisierung und Datenanalyse. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Ergebnisse
Glykolyse und mitochondriale oxidative Phosphorylierung sind die beiden wichtigsten ATP-Produktionswege in den Zellen (Abbildung 4A). Einige Zellen haben die Fähigkeit, zwischen diesen beiden Wegen zu wechseln, um ihren Energiebedarf zu decken. Die Umwandlung von Glukose in Pyruvat im Zytoplasma wird als Glykolyse bezeichnet. Pyruvat hat zwei Schicksale; Es wird entweder in Laktat umgewandelt oder durch den TCA-Zyklus und sc...
Diskussion
Wie bereits erwähnt, kann das extrazelluläre Flussanalysator Echtzeitinformationen über zwei wichtige Energieerzeugungswege der Zellen liefern, indem es die OCR (Sauerstoffverbrauchsrate), einen Indikator für die mitochondriale OXPHOS-Aktivität, und ECAR (extrazelluläre Ansäuerungsrate), die ein Indikator für die Glykolyse ist, misst. Makrophagen können beide Wege nutzen, abhängig von ihrer Mikroumgebung. Sie können auch ihre Energieerzeugungspfade umschalten...
Offenlegungen
Die Autoren haben nichts offenzulegen.
Danksagungen
Wir danken Frau Joanna Rocha für die redaktionelle Unterstützung. Die Arbeit wurde teilweise von den National Institutes of Health (NIH) R01DK118334 (an Dr. Sun und Alaniz) und (NIH) R01A11064Z (an Dr. Jayaraman und Alaniz) unterstützt.
Materialien
| Name | Company | Catalog Number | Comments |
| 23G needles | VWR | BD305145 | |
| 2-mercaptoethanol | Life Technologies | 21985023 | |
| 50ml Conical Tube | VWR | 21008-951 | |
| ACK lysis buffer | Thermo Fisher Scientific | A1049201 | It can be lab-made |
| Agilent Seahorse XF glycolysis stress test kit | Agilent Technologies | 103020-100 | |
| Agilent Seahorse XF Glycolysis Stress Test Kit User Guide | Agilent Technologies | 103020-400 | |
| Agilent Seahorse XF Glycolytic Rate Assay Kit | Agilent Technologies | 103344-100 | |
| Agilent Seahorse XF Glycolytic Rate Assay Kit User Guide | Agilent Technologies | 103344-100 | |
| Alexa Fluor 488 anti-mouse CD206 (MMR) Antibody | BioLegend | 141710 | |
| anti-mouse CD11b eFluor450 100ug | eBioscience | 48-0112-82 | |
| BD 3ML - SYRINGE | VWR | BD309657 | Other syringes are acceptable too |
| Cell counter-Vi-CELL- XR Complete System | BECKMAN COULTER Life Sciences | 731050 | Cells can be manually counted too |
| Cell Strainer-70µm | VWR | 10199-656 | |
| CyQUANT Cell Proliferation Assay Kit | Thermo Fisher Scientific | C7026 | |
| F4/80 monoclonal antibody (BM8) pe-Cyanine7 | eBioscience | 25-4801-82 | |
| Fetal Bovine Serum | Life Technologies | 16000-044 | |
| Flow cytometer: BD LSFRFortessa X-20 | BD | 656385 | |
| Kim Wipes | VWR | 82003-822 | |
| LPS-SM ultrapure (tlrl-smpls) 5 mg | Invivogen | tlrl-smlps | |
| MCSF | Peprotech | 315-02 | |
| Murine IL-4 | Peprotech | 214-14 | |
| PE Rat Anti-Mouse CD38 | BD Biosciences | 553764 | |
| Penicillin-Streptomycin (10,000 U/mL) | Life Technologies | 15140122 | |
| Petri Dish 100mm x 15 mm | Fisher Scientific | F80875712 | |
| RPMI, Glutamax, HEPES | Invitrogen | 72400-120 | |
| Seahorse Calibrant Solution | Agilent Technologies | 103059-000 | |
| Seahorse XF 200mM Glutamine Solution | Agilent Technologies | 103579-100 | |
| Seahorse XF Glycolytic Rate Assay Kit | Agilent Technologies | 103344-100 | |
| Seahorse XFe96 FluxPaks | Agilent Technologies | 102416-100 | |
| XF Glycolysis Stress Test Kit | Agilent Technologies | 103020-100 | |
| XF RPMI Medium, pH 7.4 without phenol Red | Agilent Technologies | 103336-100 |
Referenzen
- Rosowski, E. E. Determining macrophage versus neutrophil contributions to innate immunity using larval zebrafish. Disease Models & Mechanisms. 13 (1), (2020).
- Martinez-Pomares, L., Gordon, S. . The Autoimmune Diseases. , 191-212 (2020).
- Orecchioni, M., Ghosheh, Y., Pramod, A. B., Ley, K. Macrophage polarization: different gene signatures in M1 (LPS+) vs. classically and M2- (LPS–) vs. alternatively activated macrophages. Frontiers in immunology. 10, 1084 (2019).
- Barrett, T. J. Macrophages in atherosclerosis regression. Arteriosclerosis, Thrombosis, and Vascular Biology. 40 (1), 20-33 (2020).
- Ni, Y., et al. Adipose tissue macrophage phenotypes and characteristics: the key to insulin resistance in obesity and metabolic disorders. Obesity. 28 (2), 225-234 (2020).
- Chu, C., et al. Modulation of foreign body reaction and macrophage phenotypes concerning microenvironment. Journal of Biomedical Materials Research Part A. 108 (1), 127-135 (2020).
- Batista-Gonzalez, A., Vidal, R., Criollo, A., Carreño, L. J. New insights on the role of lipid metabolism in the metabolic reprogramming of macrophages. Frontiers in Immunology. 10, 2993 (2020).
- Kang, S., Kumanogoh, A. The spectrum of macrophage activation by immunometabolism. International Immunology. , (2020).
- Shi, Y., et al. M1 But Not M0 Extracellular Vesicles Induce Polarization of RAW264. 7 Macrophages Via the TLR4-NFκB Pathway In Vitro. Inflammation. , 1-9 (2020).
- Zhang, L., Li, S. Lactic acid promotes macrophage polarization through MCT-HIF1α signaling in gastric cancer. Experimental Cell Research. 388 (2), 111846 (2020).
- Geng, T., et al. CD137 signaling induces macrophage M2 polarization in atherosclerosis through STAT6/PPARδ pathway. Cellular Signalling. , 109628 (2020).
- Liu, Q., et al. Combined blockade of TGf-β1 and GM-CSF improves chemotherapeutic effects for pancreatic cancer by modulating tumor microenvironment. Cancer Immunology & Immunotherapy. , 1-16 (2020).
- Rigoni, T. S., et al. RANK Ligand Helps Immunity to Leishmania major by Skewing M2 Into M1 Macrophages. Frontiers in Immunology. 11, 886 (2020).
- Viola, A., Munari, F., Sánchez-Rodríguez, R., Scolaro, T., Castegna, A. The metabolic signature of macrophage responses. Frontiers in Immunology. 10, (2019).
- Ivashkiv, L. B. The hypoxia–lactate axis tempers inflammation. Nature Reviews Immunology. 20 (2), 85-86 (2020).
- Van den Bossche, J., Baardman, J., de Winther, M. P. Metabolic characterization of polarized M1 and M2 bone marrow-derived macrophages using real-time extracellular flux analysis. Journal of Visualized Experiments. (105), e53424 (2015).
- Van den Bossche, J., et al. Mitochondrial dysfunction prevents repolarization of inflammatory macrophages. Cell reports. 17 (3), 684-696 (2016).
- Liu, P. S., Ho, P. C. . Metabolic Signaling. , 173-186 (2019).
- Wang, F., et al. Glycolytic stimulation is not a requirement for M2 macrophage differentiation. Cell metabolism. 28 (3), 463-475 (2018).
- Romero, N., Rogers, G., Neilson, A., Dranka, B. . Quantifying Cellular ATP Production Rate Using Agilent Seahorse XF Technology. , (2018).
- Mookerjee, S. A., Brand, M. D. Measurement and analysis of extracellular acid production to determine glycolytic rate. Journal of Visualized Experiments. (106), e53464 (2015).
- Mookerjee, S. A., Goncalves, R. L., Gerencser, A. A., Nicholls, D. G., Brand, M. D. The contributions of respiration and glycolysis to extracellular acid production. Biochimica Et Biophysica Acta-Bioenergetics. 1847 (2), 171-181 (2015).
- Yang, S., et al. Macrophage polarization in atherosclerosis. Clinica Chimica Acta. 501, 142-146 (2020).
- Hörhold, F., et al. Reprogramming of macrophages employing gene regulatory and metabolic network models. PLoS Computational Biology. 16 (2), e1007657 (2020).
- Han, X., Ma, W., Zhu, Y., Sun, X., Liu, N. Advanced glycation end products enhance macrophage polarization to the M1 phenotype via the HIF-1α/PDK4 pathway. Molecular and Cellular Endocrinology. , 110878 (2020).
- Müller, E., et al. Toll-like receptor ligands and interferon-γ synergize for induction of antitumor M1 macrophages. Frontiers in Immunology. 8, 1383 (2017).
- Soto-Heredero, G., Gómez de las Heras, M. M., Gabandé-Rodríguez, E., Oller, J., Mittelbrunn, M. Glycolysis–a key player in the inflammatory response. The FEBS Journal. , (2020).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten