É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Um protocolo abrangente de alta eficiência para isolamento, cultura, polarização e caracterização glicolítica de macrófagos derivados da medula óssea
Neste Artigo
Resumo
Este protocolo fornece métodos detalhados e abrangentes para o isolamento, cultura, polarização e medição do estado metabólico glicolítico de macrófagos vivos derivados da medula óssea (BMDMs). Este artigo fornece instruções passo a passo com ilustrações visuais realistas para fluxo de trabalho e avaliação glicolítica de BMDMs em tempo real.
Resumo
Os macrófagos estão entre as células apresentadoras de antígenos mais importantes. Muitos subconjuntos de macrófagos foram identificados com assinaturas metabólicas únicas. Os macrófagos são comumente classificados como subtipos semelhantes a M1 (inflamatórios) e semelhantes a M2 (antiinflamatórios). Macrófagos semelhantes a M1 são macrófagos pró-inflamatórios que são ativados por LPS e / ou citocinas pró-inflamatórias, como INF-γ, IL-12 e IL-2. Macrófagos polarizados do tipo M1 estão envolvidos em várias doenças, mediando a defesa do hospedeiro a uma variedade de bactérias e vírus. Isso é muito importante para estudar macrófagos semelhantes a M1 induzidos por LPS e seus estados metabólicos em doenças inflamatórias. Os macrófagos do tipo M2 são considerados macrófagos anti-inflamatórios, ativados por citocinas e estimuladores anti-inflamatórios. Sob o estado pró-inflamatório, os macrófagos apresentam glicólise aumentada na função glicolítica. A função glicolítica tem sido ativamente investigada no contexto de glicólise, capacidade glicolítica, reserva glicolítica, glicólise compensatória ou acidificação não glicolítica usando analisadores de fluxo extracelular (XF).
Este artigo demonstra como avaliar os estados glicolíticos em tempo real com etapas fáceis de seguir quando os macrófagos derivados da medula óssea (BMDMs) estão respirando, consumindo e produzindo energia. Usando inibidores e ativadores específicos da glicólise neste protocolo, mostramos como obter uma visão sistêmica e completa dos processos metabólicos glicolíticos nas células e fornecer resultados mais precisos e realistas. Para poder medir vários fenótipos glicolíticos, fornecemos um método de normalização fácil, sensível e baseado em DNA para avaliação da polarização de BMDMs. Cultura, ativação/polarização e identificação do fenótipo e estado metabólico dos BMDMs são técnicas cruciais que podem ajudar a investigar muitos tipos diferentes de doenças.
Neste artigo, polarizamos os macrófagos M0 virgens para macrófagos semelhantes a M1 e M2 com LPS e IL4, respectivamente, e medimos um conjunto abrangente de parâmetros glicolíticos em BMDMs em tempo real e longitudinalmente ao longo do tempo, usando análise de fluxo extracelular e ativadores e inibidores glicolíticos.
Introdução
Os macrófagos são uma das células mais críticas do sistema imunológico inato semelhante ao M1. Eles estão envolvidos na eliminação de doenças infecciosas, fagocitose, apresentação de antígenos e regulação da inflamação2. Além disso, os macrófagos são necessários para regular outras células imunes por meio de várias citocinas que liberam3. Existe um grande espectro nos fenótipos de macrófagos4. Dependendo dos sinais aos quais os macrófagos são expostos, eles se polarizam em direção a diferentes estados inflamatórios e metabólicos5. Os macrófagos manifestam alterações metabólicas em várias doenças, dependendo do tecido em que os macrófagos residem6. Os macrófagos polarizados têm a capacidade de reprogramar ou mudar seu metabolismo glicolítico, metabolismo lipídico, metabolismo de aminoácidos e fosforilação oxidativa mitocondrial (OXPHOS)7,8. Macrófagos do tipo M1 classicamente ativados e macrófagos do tipo M2 ativados alternativamente são os dois fenótipos de macrófagos mais estudados3. Macrófagos quiescentes não ativados são chamados de macrófagos M0. A polarização de macrófagos M0 em direção a um fenótipo semelhante ao M1 pode ser induzida pela estimulação de BMDMs virgens com lipopolissacarídeo bacteriano (LPS)9. A via de sinalização PI3K-AKT-mTOR-HIF1a pode ser ativada em macrófagos na presença de citocinas inflamatórias, interferon-gama (IFN γ) ou fator de necrose tumoral (TNF)10. Macrófagos semelhantes a M1 apresentam níveis aumentados de metabolismo da glicólise, diminuição dos níveis de fosforilação oxidativa (OXPHOS), produzindo citocinas inflamatórias envolvidas em doenças infecciosas e inflamatórias8. Por outro lado, a polarização em direção ao fenótipo M2-like pode ser induzida pela interleucina (IL)-4, através das vias JAK-STAT, PPAR e AMPK, ou pelas vias (IL)-13 e TGFβ11,12.
Em contraste com os macrófagos semelhantes a M1, os macrófagos semelhantes a M2 diminuíram a glicólise e aumentaram o OXPHOS e estão envolvidos em atividades antiparasitárias e de reparo tecidual 8,13. Os BMDMs são um sistema amplamente utilizado para o estudo de macrófagos derivados de células-tronco da medula óssea. A glicólise e o OXPHOS são as duas principais vias de produção de energia nas células14. Com base em seu microambiente, os BMDMs podem optar por usar qualquer uma dessas vias; em alguns casos, mude de um para outro ou use os dois caminhos14. Neste estudo, nos concentramos no metabolismo da glicólise em macrófagos pró-inflamatórios ativados. Quando a glicose no citoplasma é convertida em piruvato e depois em lactato, as células produzem prótons no meio que causam uma elevação na taxa de acidificação no meio cercado de células semelhantes a M15. Um analisador de fluxo extracelular foi usado para medir a taxa de acidificação do meio celular. Os resultados são relatados como Taxa de Acidificação Extracelular (ECAR) ou como Taxa de Efluxo de Prótons.
Um método otimizado, rápido e fácil para acessar os níveis de glicólise em macrófagos polarizados é essencial para determinar o fenótipo glicolítico, alterações de metabólitos e os efeitos de inibidores / ativadores e drogas nos macrófagos polarizados. O método descrito neste manuscrito foi otimizado para fornecer informações sobre fatores específicos de glicólise (glicólise, capacidade glicolítica, reserva glicolítica e acidificação não glicolítica), bem como a reprogramação metabólica do metabolismo glicolítico. O inibidor (2DG) que foi usado neste estudo visa explicitamente a via da glicólise.
Este protocolo otimizado foi modificado e aprimorado com base na combinação de um protocolo publicado16, análise de fluxo extracelular de ensaios glicolíticos de guias do usuário do fabricante e comunicação direta com cientistas de P&D do fabricante.
Protocolo
Os camundongos foram sacrificados humanamente de acordo com as diretrizes de Avaliação e Acreditação de Cuidados com Animais de Laboratório (AAALAC) e Associação Americana de Ciência de Animais de Laboratório (AALAS) e usando protocolos aprovados pelo comitê institucional de cuidados e uso de animais da Texas A&M University (IACUC).
1. Colheita de medula óssea de camundongos e cultura de BMDMs
- Sacrifique o camundongo (camundongos C57Bl / 6 de 6 semanas de idade estavam neste protocolo) e coloque-o em seu lado ventral, corte a pele e a camada peritoneal e retire suavemente as pernas.
NOTA: Use a exposição ao gás CO2 para sacrificar o mouse. - Separe as duas patas traseiras do quadril para baixo, tomando cuidado para não cortar o osso.
- Coloque a perna inteira em um tubo cônico vazio de 50 mL (com os pés voltados para cima para ter uma pegada fácil de puxar mais tarde) no gelo e prossiga com a colheita de ambas as pernas do mouse.
2. Exposição do fêmur
NOTA: Execute as etapas a seguir em um gabinete de biossegurança.
- Colha o fêmur cortando a tíbia de cada perna e remova o máximo de tecido possível ao redor do fêmur com uma tesoura e papel de laboratório.
- Coloque os fêmures colhidos e "limpos" em uma placa de 10 cm contendo um pedaço de papel de laboratório saturado com meio de cultura de tecidos (TC) ou PBS. Coloque-os no gelo.
- Continue colhendo fêmures e removendo o tecido de todos os fêmures antes de prosseguir para o estágio de lavagem (Figura 1A).
3. Rubor de medula
- Para lavar a medula dos fêmures, use uma seringa de 3 mL cheia de meio TC ou PBS com uma agulha 23G. Encha a seringa antes de expor a medula.
- Use uma tesoura para expor a medula cortando a extremidade do fêmur em ambas as epífises.
- Insira a ponta da agulha no fêmur e lave suavemente a medula em um prato de 10 cm.
- Passe a agulha por todo o comprimento do fêmur e lave até que a cor do osso fique branca. Normalmente, a maior parte da medula pode ser lavada com 2-3 mL de meio.
- Lave todos os fêmures e medula óssea no prato. Use uma agulha para quebrar quaisquer aglomerados visíveis. Coe a medula em um tubo cônico de 50 mL (Figura 1A).
4. Lise de hemácias
- Gire a medula a 190 x g por 10 min. Aspire o sobrenadante.
- Ressuspenda o pellet em 4 ml de tampão de lise ACK com uma pipeta. Deixe o tampão de lise de hemácias funcionar por 5 min em temperatura ambiente.
- Adicione 4 mL de meio TC RPMI-C 10% (RPMI 1640 -GlutaMAX) suplementado com 2-mercaptoetanol, gentamicina, estreptomicina e 10% FCS à suspensão medular e gire a 1300 x g por 10 min.
- Coe novamente para remover os detritos de hemácias e ressuspenda em um pequeno volume de RPMI-C 10% para contagem.
- Conte as células com um contador de células (Figura 1B). Um contador de células Vi foi usado para determinar a contagem e a viabilidade das células na suspensão.
5. Chapeamento e cultura
- Adicione 10 mL de RPMI-C 10% + 10 ng/mL M-CSF (Fator Estimulador de Colônias de Macrófagos, um regulador essencial da proliferação, diferenciação e sobrevivência de monócitos/macrófagos) a quantas placas de 10 cm desejar.
- Adicione um volume apropriado de células contadas para que cada placa de 10 cm contenha 1 x 106 células. Coloque as placas em uma incubadora a 37 °C (definida como dia 0).
- No dia 3, adicione suavemente 5 mL de RPMI-C fresco a 10% + 10 ng/mL M-CSF a cada placa.
NOTA: No dia 7, os BMDMs devem estar prontos para teste (Figura 1C).
6. Colheita de pratos
- Use um microscópio óptico para confirmar que a maioria das células aderiu às placas.
- Aspire suavemente o meio. Em seguida, adicione 3 mL de PBS e gire suavemente a placa. Aspire bem para remover quaisquer células não aderentes restantes.
- Adicione 7-10 mL de PBS frio à placa, use uma pipeta P1000 para lavar o fundo das placas e colha todas as células restantes em um tubo de coleta.
NOTA: Mantenha os tubos no gelo, pois os macrófagos são muito aderentes e aderem ao interior do tubo. Se as células se mantivessem frias, elas seriam menos aderentes. - Centrifugar, contar e células de placa para experimentos (Figura 1D). Usando citometria de fluxo, as células resultantes devem ser >95% positivas para CD11b e F4/80. (A polarização dos macrófagos foi determinada pela coloração com marcadores semelhantes a M1 de CD38, TNF-a e marcadores semelhantes a MCP-1 e M2 de CD206.
NOTA: Execute as etapas 6.1-6.3 no gabinete de biossegurança e execute a etapa 6.4 na bancada. Mantenha as técnicas assépticas durante todo o procedimento.
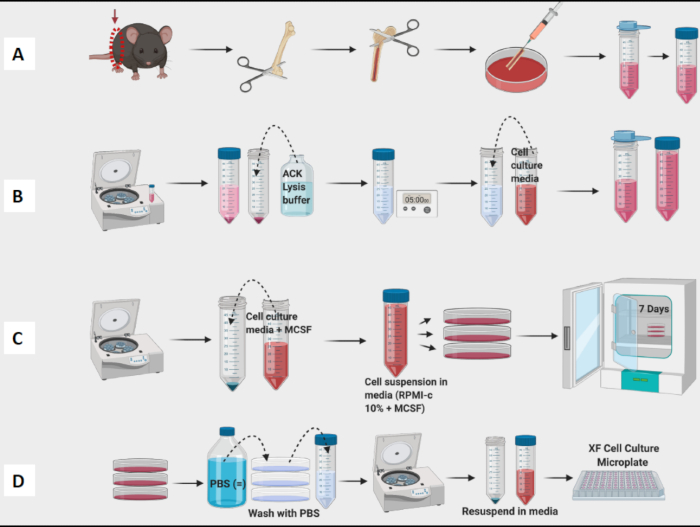
Figura 1: Fluxo de trabalho gráfico da cultura de medula óssea de camundongos de macrófagos derivados de BM. (A) Colheita da perna, exposição do fêmur e rubor da medula; (b) lise de hemácias; (C) Revestimento e cultura; (D) Colheita de células das placas. Clique aqui para ver uma versão maior desta figura.
7. Um dia antes do ensaio do analisador de fluxo metabólico: semeadura e polarização das células para o teste glicolítico
- Aqueça o Metabolic Flux Analyzer a 37 °C ligando o instrumento.
- Hidrate um cartucho adicionando 200 μL de uma solução calibrante e incube o cartucho em uma incubadora não-CO2 durante a noite (Figura 2A). A umidade da incubadora não-CO2 não é importante para a hidratação do cartucho.
- Uma hora antes do experimento, mergulhe a placa algumas vezes para cima e para baixo, o que ajudará a remover as bolhas de ar.
- Projete o mapa da placa no software no teste de estresse de glicólise padrão - injeção aguda, seguindo as instruções do teste.
- Clique no ícone do software e, em seguida, clique em Teste de injeção aguda de estresse glicólise. No ícone de definição de grupo, gere nomes de grupo.
- Existem cinco ciclos de medição com duração de 18 minutos e quatro injeções. Altere a injeção da porta A para glicose, porta B para oligomicina, porta C para rotenona e antimicina A (Rot / AA) e porta D para 2DG.
- Ressuspenda as células em meio RPMI-C 10% e semeie 50k células por poço, exceto as quatro bordas da placa (A1, A12, H1 e H12; Adicione apenas meios, sem células) em uma microplaca do Metabolic Flux Analyzer a um volume final de 100 μL. Normalmente, é necessário um mínimo de 40k células para realizar este ensaio.
- Deixe as células descansarem em temperatura ambiente por 45 min para evitar o efeito de borda das células. O efeito de borda é quando o meio ao redor do perímetro da placa evapora parcialmente, o que causa mudanças de volume e concentração e reduz a viabilidade celular.
- Adicione 10 ng/mL de LPS para polarizar os macrófagos virgens em direção a células semelhantes a M1 e adicione 20 ng/ml de IL-4 para polarizá-los em direção a células semelhantes a M2. Use pelo menos 3 a 6 poços por condição (Figura 2B).
- Verificar as células ao microscópio e colocar a placa numa incubadora a 37 °C e 5% de CO2 durante 24 horas.
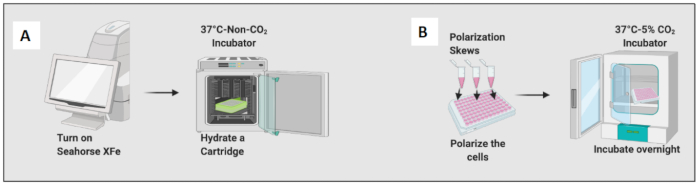
Figura 2: Demonstração gráfica da semeadura e polarização das células. (A) Configuração do analisador de fluxo extracelular e hidratação do cartucho; (B) Polarização das células e incubação durante a noite. Clique aqui para ver uma versão maior desta figura.
8. Dia do ensaio: XF Preparação de meio e composto
- Complemente 100 mL de meio de ensaio XF RPMI (pH 7,4) com 2 mM de glutamina.
- Filtre e esterilize a mídia usando um sistema de filtro a vácuo de 0,2 μm.
- Coloque o meio de ensaio em banho-maria a 37°C por 20 min.
- Remover as células plaqueadas da incubadora de CO2 a 37 °C, 5%. Lave as células com meio de ensaio duas vezes e substitua o meio anterior por meio de ensaio até o volume final de 180 μL.
- Use um microscópio para certificar-se de que todos os poços tenham células confluentes e marque todos os poços que tenham arranhões de pipetagem. Se houver algum arranhão, remova essa placa antes de analisar.
- Posicione a placa contendo células em uma incubadora sem CO2 por 45 min (Figura 3A).
- Usando os compostos e os meios de ensaio para fazer soluções de estoque de glicose (100 mM), oligomicina (100 μM), rot / AA (50 μM) e 2DG (500 mM) (Tabela 1).
- Faça uma mistura de injeção de 10x de cada composto usando meio de ensaio (Tabela 2).
| Estoques de injeção (fornecidos nos kits) | Adicionar meio de ensaio completo (mL) | Concentração de estoque final (μM) |
| Glicose | 3 | 100 mil |
| Oligomicina | 0.72 | 100 |
| 2-DG | 3 | 100 mil |
Tabela 1. Estoques de injeção
| Portas no cartucho | Soluções de estoque | Adicionar volume de estoque | Adicionar meio de ensaio | Concentração final de injeções (10x) | Adicione este volume à porta designada (μL) | Concentração final após a injeção em cada poço |
| Um | Glicose (100 mM) | 3000 μL + 0 μL | 100 mM | 20 | 10 mM | |
| B | Oligomicina (100 μM) | 300 μL + 2700 μL | 10 μM | 22 | 1,0 μM | |
| C | Rotenona/Antimicina A (50 μM) | 300 μL + 2700 μL | 5 μM | 25 | 0,5 μM | |
| D | 2-DG (500 mM) | 300 μL + 0 μL | 500 mM | 28 | 50 mM | |
Tabela 2. Concentrações finais de injeção
9. Dia do ensaio: Executando o teste glicolítico agudo em macrófagos polarizados
- Abra o modelo salvo de ensaio de estresse de glicólise (injeção aguda) no software. O teste de glicoestresse agudo padrão tem 3 minutos de mistura e medição antes de cada injeção.
- Verifique o modelo e os detalhes do ensaio e, quando estiver pronto, clique em Executar e siga as instruções do ensaio padrão. No entanto, todos os parâmetros podem ser personalizados.
- Remova o cartucho do sensor da incubadora não-CO2 , remova a tampa e insira no instrumento de forma que o poço A1 da placa do cartucho caia no canto superior esquerdo do painel de inserção da máquina. Normalmente, a calibração leva entre 20 a 45 min.
- Após terminar a calibração, o dispositivo ejetará a placa contendo a solução calibrante e segurará o cartucho do sensor. Remova o calibrante que contém a placa.
- Remova a placa celular da incubadora não-CO2 , remova a tampa da placa e insira-a na máquina. Clique em Executar (Figura 3B).
- Quando o ensaio for concluído, a máquina ejetará a placa da célula e o cartucho do sensor.
- Remova a mídia da placa e congele-a a -20 °C para posterior normalização.
- Use o kit de ensaio de proliferação celular comercial (por exemplo, CyQUANT) para normalizar as células.
- Adicione 1 mL de Composto B ou tampão de lise a 19 mL de água destilada livre de nuclease.
- Adicione 100 μL de solução de trabalho Composto A ou GR à solução acima mencionada.
- Certifique-se de que as células da placa estejam descongeladas e, em seguida, adicione 200 μL da solução a cada poço.
- Incube por 5 min em temperatura ambiente (RT).
- Meça a fluorescência em comprimentos de onda de excitação de 480 nm e emissão de 520 nm usando um leitor de placas.
- Normalize as células no painel de normalização do software.
- Normalize as células com base na contagem de células de macrófagos ingênuos ( Figura 3C ). Considere a média dos macrófagos ingênuos como 1 (dividindo o número de células de cada poço pelo número médio de células de macrófagos ingênuos) e aplique-os a todos os macrófagos.
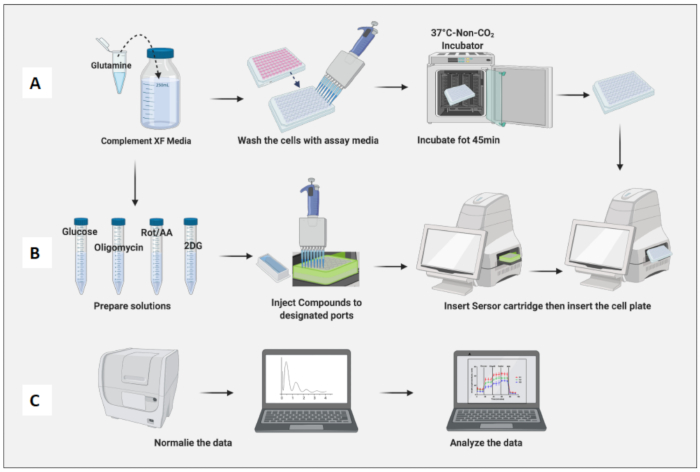
Figura 3: Dia do ensaio: preparação do meio e do composto e execução do ensaio. (A) Preparação das células para o ensaio; (B) Preparação, calibração e execução do ensaio de compostos; (C) Normalização e análise de dados. Clique aqui para ver uma versão maior desta figura.
Resultados
A glicólise e a fosforilação oxidativa mitocondrial são as duas principais vias de produção de ATP nas células (Figura 4A). Algumas células têm a capacidade de alternar entre esses dois caminhos para atender às suas demandas de energia. A conversão de glicose em piruvato no citoplasma é chamada de glicólise. O piruvato tem dois destinos; ele será convertido em lactato ou metabolizado através do ciclo de TCA e, ...
Discussão
Como mencionado anteriormente, a máquina analisadora de fluxo extracelular pode fornecer informações em tempo real sobre duas principais vias de produção de energia das células, medindo o OCR (taxa de consumo de oxigênio), um indicador da atividade mitocondrial do OXPHOS, e o ECAR (taxa de acidificação extracelular), que é um indicador de glicólise. Os macrófagos podem usar ambas as vias, dependendo de seu microambiente. Eles também podem mudar suas vias de produção de ene...
Divulgações
Os autores não têm nada a divulgar.
Agradecimentos
Agradecemos à Sra. Joanna Rocha pela assistência editorial. O trabalho foi parcialmente apoiado pelos Institutos Nacionais de Saúde (NIH) R01DK118334 (para os Drs. Sun e Alaniz) e (NIH) R01A11064Z (para os Drs. Jayaraman e Alaniz).
Materiais
| Name | Company | Catalog Number | Comments |
| 23G needles | VWR | BD305145 | |
| 2-mercaptoethanol | Life Technologies | 21985023 | |
| 50ml Conical Tube | VWR | 21008-951 | |
| ACK lysis buffer | Thermo Fisher Scientific | A1049201 | It can be lab-made |
| Agilent Seahorse XF glycolysis stress test kit | Agilent Technologies | 103020-100 | |
| Agilent Seahorse XF Glycolysis Stress Test Kit User Guide | Agilent Technologies | 103020-400 | |
| Agilent Seahorse XF Glycolytic Rate Assay Kit | Agilent Technologies | 103344-100 | |
| Agilent Seahorse XF Glycolytic Rate Assay Kit User Guide | Agilent Technologies | 103344-100 | |
| Alexa Fluor 488 anti-mouse CD206 (MMR) Antibody | BioLegend | 141710 | |
| anti-mouse CD11b eFluor450 100ug | eBioscience | 48-0112-82 | |
| BD 3ML - SYRINGE | VWR | BD309657 | Other syringes are acceptable too |
| Cell counter-Vi-CELL- XR Complete System | BECKMAN COULTER Life Sciences | 731050 | Cells can be manually counted too |
| Cell Strainer-70µm | VWR | 10199-656 | |
| CyQUANT Cell Proliferation Assay Kit | Thermo Fisher Scientific | C7026 | |
| F4/80 monoclonal antibody (BM8) pe-Cyanine7 | eBioscience | 25-4801-82 | |
| Fetal Bovine Serum | Life Technologies | 16000-044 | |
| Flow cytometer: BD LSFRFortessa X-20 | BD | 656385 | |
| Kim Wipes | VWR | 82003-822 | |
| LPS-SM ultrapure (tlrl-smpls) 5 mg | Invivogen | tlrl-smlps | |
| MCSF | Peprotech | 315-02 | |
| Murine IL-4 | Peprotech | 214-14 | |
| PE Rat Anti-Mouse CD38 | BD Biosciences | 553764 | |
| Penicillin-Streptomycin (10,000 U/mL) | Life Technologies | 15140122 | |
| Petri Dish 100mm x 15 mm | Fisher Scientific | F80875712 | |
| RPMI, Glutamax, HEPES | Invitrogen | 72400-120 | |
| Seahorse Calibrant Solution | Agilent Technologies | 103059-000 | |
| Seahorse XF 200mM Glutamine Solution | Agilent Technologies | 103579-100 | |
| Seahorse XF Glycolytic Rate Assay Kit | Agilent Technologies | 103344-100 | |
| Seahorse XFe96 FluxPaks | Agilent Technologies | 102416-100 | |
| XF Glycolysis Stress Test Kit | Agilent Technologies | 103020-100 | |
| XF RPMI Medium, pH 7.4 without phenol Red | Agilent Technologies | 103336-100 |
Referências
- Rosowski, E. E. Determining macrophage versus neutrophil contributions to innate immunity using larval zebrafish. Disease Models & Mechanisms. 13 (1), (2020).
- Martinez-Pomares, L., Gordon, S. . The Autoimmune Diseases. , 191-212 (2020).
- Orecchioni, M., Ghosheh, Y., Pramod, A. B., Ley, K. Macrophage polarization: different gene signatures in M1 (LPS+) vs. classically and M2- (LPS–) vs. alternatively activated macrophages. Frontiers in immunology. 10, 1084 (2019).
- Barrett, T. J. Macrophages in atherosclerosis regression. Arteriosclerosis, Thrombosis, and Vascular Biology. 40 (1), 20-33 (2020).
- Ni, Y., et al. Adipose tissue macrophage phenotypes and characteristics: the key to insulin resistance in obesity and metabolic disorders. Obesity. 28 (2), 225-234 (2020).
- Chu, C., et al. Modulation of foreign body reaction and macrophage phenotypes concerning microenvironment. Journal of Biomedical Materials Research Part A. 108 (1), 127-135 (2020).
- Batista-Gonzalez, A., Vidal, R., Criollo, A., Carreño, L. J. New insights on the role of lipid metabolism in the metabolic reprogramming of macrophages. Frontiers in Immunology. 10, 2993 (2020).
- Kang, S., Kumanogoh, A. The spectrum of macrophage activation by immunometabolism. International Immunology. , (2020).
- Shi, Y., et al. M1 But Not M0 Extracellular Vesicles Induce Polarization of RAW264. 7 Macrophages Via the TLR4-NFκB Pathway In Vitro. Inflammation. , 1-9 (2020).
- Zhang, L., Li, S. Lactic acid promotes macrophage polarization through MCT-HIF1α signaling in gastric cancer. Experimental Cell Research. 388 (2), 111846 (2020).
- Geng, T., et al. CD137 signaling induces macrophage M2 polarization in atherosclerosis through STAT6/PPARδ pathway. Cellular Signalling. , 109628 (2020).
- Liu, Q., et al. Combined blockade of TGf-β1 and GM-CSF improves chemotherapeutic effects for pancreatic cancer by modulating tumor microenvironment. Cancer Immunology & Immunotherapy. , 1-16 (2020).
- Rigoni, T. S., et al. RANK Ligand Helps Immunity to Leishmania major by Skewing M2 Into M1 Macrophages. Frontiers in Immunology. 11, 886 (2020).
- Viola, A., Munari, F., Sánchez-Rodríguez, R., Scolaro, T., Castegna, A. The metabolic signature of macrophage responses. Frontiers in Immunology. 10, (2019).
- Ivashkiv, L. B. The hypoxia–lactate axis tempers inflammation. Nature Reviews Immunology. 20 (2), 85-86 (2020).
- Van den Bossche, J., Baardman, J., de Winther, M. P. Metabolic characterization of polarized M1 and M2 bone marrow-derived macrophages using real-time extracellular flux analysis. Journal of Visualized Experiments. (105), e53424 (2015).
- Van den Bossche, J., et al. Mitochondrial dysfunction prevents repolarization of inflammatory macrophages. Cell reports. 17 (3), 684-696 (2016).
- Liu, P. S., Ho, P. C. . Metabolic Signaling. , 173-186 (2019).
- Wang, F., et al. Glycolytic stimulation is not a requirement for M2 macrophage differentiation. Cell metabolism. 28 (3), 463-475 (2018).
- Romero, N., Rogers, G., Neilson, A., Dranka, B. . Quantifying Cellular ATP Production Rate Using Agilent Seahorse XF Technology. , (2018).
- Mookerjee, S. A., Brand, M. D. Measurement and analysis of extracellular acid production to determine glycolytic rate. Journal of Visualized Experiments. (106), e53464 (2015).
- Mookerjee, S. A., Goncalves, R. L., Gerencser, A. A., Nicholls, D. G., Brand, M. D. The contributions of respiration and glycolysis to extracellular acid production. Biochimica Et Biophysica Acta-Bioenergetics. 1847 (2), 171-181 (2015).
- Yang, S., et al. Macrophage polarization in atherosclerosis. Clinica Chimica Acta. 501, 142-146 (2020).
- Hörhold, F., et al. Reprogramming of macrophages employing gene regulatory and metabolic network models. PLoS Computational Biology. 16 (2), e1007657 (2020).
- Han, X., Ma, W., Zhu, Y., Sun, X., Liu, N. Advanced glycation end products enhance macrophage polarization to the M1 phenotype via the HIF-1α/PDK4 pathway. Molecular and Cellular Endocrinology. , 110878 (2020).
- Müller, E., et al. Toll-like receptor ligands and interferon-γ synergize for induction of antitumor M1 macrophages. Frontiers in Immunology. 8, 1383 (2017).
- Soto-Heredero, G., Gómez de las Heras, M. M., Gabandé-Rodríguez, E., Oller, J., Mittelbrunn, M. Glycolysis–a key player in the inflammatory response. The FEBS Journal. , (2020).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoExplore Mais Artigos
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados