Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Un protocole complet à haute efficacité pour l’isolement, la culture, la polarisation et la caractérisation glycolytique des macrophages dérivés de la moelle osseuse
Dans cet article
Résumé
Ce protocole fournit des méthodes détaillées et complètes pour l’isolement, la culture, la polarisation et la mesure de l’état métabolique glycolytique des macrophages vivants dérivés de la moelle osseuse (BMDM). Ce document fournit des instructions étape par étape avec des illustrations visuelles réalistes pour le flux de travail et l’évaluation glycolytique des BMDM en temps réel.
Résumé
Les macrophages sont parmi les cellules présentatrices d’antigènes les plus importantes. De nombreux sous-ensembles de macrophages ont été identifiés avec des signatures métaboliques uniques. Les macrophages sont généralement classés en sous-types de type M1 (inflammatoire) et de type M2 (anti-inflammatoire). Les macrophages de type M1 sont des macrophages pro-inflammatoires qui sont activés par le LPS et/ou les cytokines pro-inflammatoires telles que INF-γ, IL-12 et IL-2. Les macrophages polarisés de type M1 sont impliqués dans diverses maladies en médiant la défense de l’hôte contre une variété de bactéries et de virus. C’est très important pour étudier les macrophages de type M1 induits par le LPS et leurs états métaboliques dans les maladies inflammatoires. Les macrophages de type M2 sont considérés comme des macrophages anti-inflammatoires, activés par des cytokines et des stimulateurs anti-inflammatoires. Sous l’état pro-inflammatoire, les macrophages montrent une glycolyse accrue dans la fonction glycolytique. La fonction glycolytique a été activement étudiée dans le contexte de la glycolyse, de la capacité glycolytique, de la réserve glycolytique, de la glycolyse compensatoire ou de l’acidification non glycolytique à l’aide d’analyseurs de flux extracellulaire (XF).
Cet article démontre comment évaluer les états glycolytiques en temps réel avec des étapes faciles à suivre lorsque les macrophages dérivés de la moelle osseuse (BMDM) respirent, consomment et produisent de l’énergie. En utilisant des inhibiteurs et des activateurs spécifiques de la glycolyse dans ce protocole, nous montrons comment obtenir une vue systémique et complète des processus métaboliques glycolytiques dans les cellules et fournir des résultats plus précis et réalistes. Pour être en mesure de mesurer plusieurs phénotypes glycolytiques, nous fournissons une méthode de normalisation simple, sensible et basée sur l’ADN pour l’évaluation de la polarisation des BMDM. La culture, l’activation/polarisation et l’identification du phénotype et de l’état métabolique des BMDM sont des techniques cruciales qui peuvent aider à étudier de nombreux types de maladies différentes.
Dans cet article, nous avons polarisé les macrophages M0 naïfs en macrophages de type M1 et M2 avec LPS et IL4, respectivement, et mesuré un ensemble complet de paramètres glycolytiques dans les BMDM en temps réel et longitudinalement au fil du temps, en utilisant l’analyse des flux extracellulaires et les activateurs et inhibiteurs glycolytiques.
Introduction
Les macrophages sont l’une des cellules les plus critiques du système immunitaire inné, de type M1. Ils sont impliqués dans l’élimination des maladies infectieuses, la phagocytose, la présentation d’antigènes et la régulation de l’inflammation2. De plus, les macrophages sont nécessaires pour réguler d’autres cellules immunitaires via diverses cytokines qu’elles libèrent3. Il existe un large spectre dans les phénotypes de macrophages4. En fonction des signaux auxquels les macrophages sont exposés, ils se polarisent vers différents états inflammatoires et métaboliques5. Les macrophages manifestent des altérations métaboliques dans diverses maladies, selon le tissu où ils résident6. Les macrophages polarisés ont la capacité de reprogrammer ou de changer leur métabolisme glycolytique, leur métabolisme lipidique, leur métabolisme des acides aminés et leur phosphorylation oxydative mitochondriale (OXPHOS)7,8. Les macrophages de type M1 activés de manière classique et les macrophages de type M2 alternativement activés sont les deux phénotypes de macrophages3 les plus étudiés. Les macrophages quiescents non activés sont appelés macrophages M0. La polarisation des macrophages M0 vers un phénotype de type M1 peut être induite par la stimulation de DMBO naïfs avec un lipopolysaccharide bactérien (LPS)9. La voie de signalisation PI3K-AKT-mTOR-HIF1a peut être activée dans les macrophages en présence de cytokines inflammatoires, d’interféron-gamma (IFN γ,) ou de facteur de nécrose tumorale (TNF)10. Les macrophages de type M1 ont des niveaux accrus de métabolisme de glycolyse, des niveaux diminués de phosphorylation oxydative (OXPHOS), produisant des cytokines inflammatoires impliquées dans les maladies infectieuses et inflammatoires8. D’autre part, la polarisation vers le phénotype de type M2 peut être induite par l’interleukine (IL)-4, via les voies JAK-STAT, PPAR et AMPK, ou par les voies (IL)-13 et TGFβ11,12.
Contrairement aux macrophages de type M1, les macrophages de type M2 ont une glycolyse réduite et une augmentation de l’OXPHOS et sont impliqués dans des activités antiparasitaires et de réparation tissulaire 8,13. Les BMDM sont un système largement utilisé pour l’étude des macrophages dérivés des cellules souches de la moelle osseuse. La glycolyse et l’OXPHOS sont les deux principales voies de production d’énergie dans les cellules14. En fonction de leur microenvironnement, les BMDM peuvent choisir d’utiliser l’une ou l’autre de ces voies ; Dans certains cas, passez de l’un à l’autre ou utilisez les deux voies14. Dans cette étude, nous nous sommes concentrés sur le métabolisme de la glycolyse dans les macrophages pro-inflammatoires activés. Lorsque le glucose dans le cytoplasme est converti en pyruvate puis en lactate, les cellules produisent des protons dans le milieu qui provoquent une élévation du taux d’acidification dans le milieu entouré de cellules de type M15. Un analyseur de flux extracellulaire a été utilisé pour mesurer le taux d’acidification des milieux cellulaires. Les résultats sont exprimés sous forme de taux d’acidification extracellulaire (ECAR) ou de taux d’efflux de protons.
Une méthode optimisée, rapide et facile pour accéder aux niveaux de glycolyse dans les macrophages polarisés est essentielle pour déterminer le phénotype glycolytique, les changements de métabolites et les effets des inhibiteurs/activateurs et des médicaments sur les macrophages polarisés. La méthode décrite dans ce manuscrit a été optimisée pour fournir des informations sur des facteurs de glycolyse spécifiques (glycolyse, capacité glycolytique, réserve glycolytique et acidification non glycolytique), ainsi que sur la reprogrammation métabolique du métabolisme glycolytique. L’inhibiteur (2DG) qui a été utilisé dans cette étude cible explicitement la voie de la glycolyse.
Ce protocole optimisé a été modifié et amélioré sur la base de la combinaison d’un protocole16 publié, de l’analyse du flux extracellulaire des dosages glycolytiques des guides d’utilisation du fabricant et d’une communication directe avec les scientifiques de la R&D du fabricant.
Protocole
Les souris ont été sacrifiées sans cruauté conformément aux directives de l’Assessment and Accreditation of Laboratory Animal Care (AAALAC) et de l’American Association for Laboratory Animal Science (AALAS) et en utilisant des protocoles approuvés par le comité institutionnel de protection et d’utilisation des animaux de l’Université Texas A&M (IACUC).
1. Prélèvement de moelle osseuse chez la souris et culture de DMO.
- Sacrifiez la souris (âgées de 6 à 10 semaines C57Bl/6 souris étaient dans ce protocole) et posez-la sur sa face ventrale, coupez la peau et la couche péritonéale et retirez doucement les pattes.
REMARQUE : Utilisez l’exposition au gaz CO2 pour euthanasier la souris. - Séparez les deux pattes arrière de la hanche vers le bas, en faisant attention de ne pas couper l’os.
- Placez la patte entière dans un tube conique vide de 50 ml (avec les pieds vers le haut pour une prise facile à retirer plus tard) sur de la glace et procédez à la récolte des deux pattes de la souris.
2. Exposition du fémur
REMARQUE : Effectuez les étapes suivantes dans une enceinte de biosécurité.
- Récoltez le fémur en coupant le tibia de chaque patte et retirez autant de tissu entourant le fémur que possible avec des ciseaux et du papier de laboratoire.
- Placez les fémurs récoltés et « nettoyés » dans une plaque de 10 cm contenant un morceau de papier de laboratoire saturé de milieu de culture tissulaire (TC) ou PBS. Placez-les sur de la glace.
- Continuer à prélever les fémurs et à retirer les tissus de tous les fémurs avant de passer à l’étape du rinçage (figure 1A).
3. Rinçage de la moelle osseuse
- Pour rincer la moelle osseuse des fémurs, utilisez une seringue de 3 mL remplie de milieu TC ou de PBS avec une aiguille 23G. Remplissez la seringue avant d’exposer la moelle.
- Utilisez des ciseaux pour exposer la moelle en coupant l’extrémité du fémur aux deux épiphyses.
- Insérez la pointe de l’aiguille dans le fémur et rincez doucement la moelle dans un plat de 10 cm.
- Passez l’aiguille sur toute la longueur du fémur et rincez jusqu’à ce que la couleur de l’os devienne blanche. Habituellement, la plupart de la moelle osseuse peut être rincée avec 2 à 3 ml de milieu.
- Rincez tous les fémurs et la moelle osseuse de la flaque dans le plat. Utilisez une aiguille pour briser les amas visibles. Filtrer la moelle dans un tube conique de 50 mL (figure 1A).
4. Lyse des globules rouges
- Essorer la moelle à 190 x g pendant 10 min. Aspirez le surnageant.
- Remettre la pastille en suspension dans 4 mL de tampon de lyse ACK à l’aide d’une pipette. Laisser agir le tampon de lyse des globules rouges pendant 5 minutes à température ambiante.
- Ajouter 4 mL de TC medium RPMI-C 10 % (RPMI 1640 -GlutaMAX) complété par du 2-mercaptoéthanol, de la gentamicine, de la streptomycine et 10 % de FCS dans la suspension de moelle et tourner à 1300 x g pendant 10 min.
- Filtrer à nouveau pour enlever les débris de globules rouges et les remettre en suspension dans un petit volume de RPMI-C 10 % pour le comptage.
- Comptez les cellules à l’aide d’un compteur de cellules (Figure 1B). Un compteur Vi-Cell a été utilisé pour déterminer le nombre et la viabilité des cellules dans la suspension.
5. Placage et culture
- Ajouter 10 mL de RPMI-C 10 % + 10 ng/mL DE M-CSF (Macrophage Colony Stimulating Factor, un régulateur essentiel de la prolifération, de la différenciation et de la survie des monocytes/macrophages) à autant de plaques de 10 cm que vous le souhaitez.
- Ajouter un volume approprié de cellules comptées de manière à ce que chaque plaque de 10 cm contienne 1 x 106 cellules. Mettez les plaques dans un incubateur à 37 °C (défini comme le jour 0).
- Le 3e jour, ajouter délicatement 5 ml de RPMI-C 10 % + 10 ng/mL DE M-CSF frais dans chaque assiette.
REMARQUE : Le jour 7, les BMDM doivent être prêtes pour les essais (figure 1C).
6. Récolter dans les assiettes
- Utilisez un microscope optique pour confirmer que la plupart des cellules ont adhéré aux plaques.
- Aspirez doucement le support. Ajouter ensuite 3 ml de PBS et agiter doucement l’assiette. Aspirez-le bien pour éliminer toutes les cellules non adhérentes restantes.
- Ajoutez 7 à 10 ml de PBS froid dans la plaque, utilisez une pipette P1000 pour laver le fond des plaques et récoltez toutes les cellules restantes dans un tube de collecte.
REMARQUE : Gardez les tubes sur de la glace car les macrophages sont très adhérents et adhéreront à l’intérieur du tube. Si les cellules restaient froides, elles auraient une adhérence moins stricte. - Centrifugeuses, cellules de comptage et cellules sur plaque pour les expériences (figure 1D). À l’aide de la cytométrie en flux, les cellules résultantes doivent être positives à >95 % pour CD11b et F4/80. (La polarisation des macrophages a été déterminée par coloration avec des marqueurs de type M1 de CD38, TNF-a et MCP-1 et marqueur de type M2 de CD206.
REMARQUE : Effectuez les étapes 6.1 à 6.3 dans l’enceinte de sécurité biologique et effectuez l’étape 6.4 sur la paillasse. Maintenez les techniques d’asepsie tout au long de la procédure.
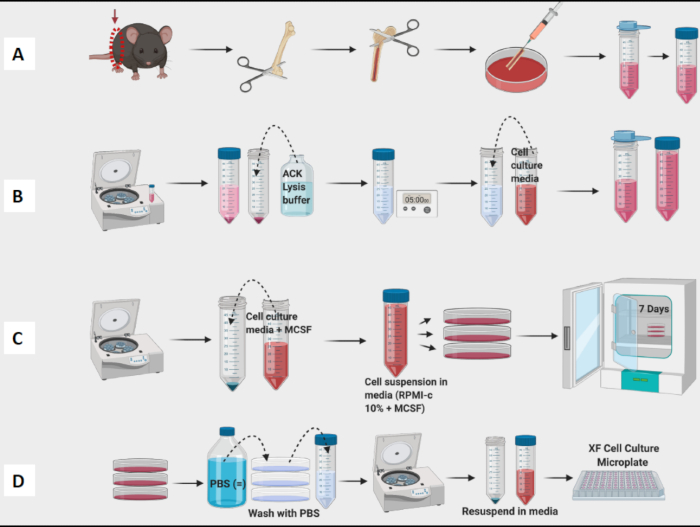
Figure 1 : Flux de travail graphique de la culture de moelle osseuse de souris de macrophages dérivés de BM. (A) Prélèvement des pattes, exposition du fémur et rinçage de la moelle osseuse ; (b) lyse des globules rouges ; (C) Placage et culture ; (D) Récolte de cellules à partir des plaques. Veuillez cliquer ici pour voir une version agrandie de cette figure.
7. La veille du dosage de l’analyseur de flux métabolique : ensemencement et polarisation des cellules pour le test glycolytique
- Réchauffez l’analyseur de flux métabolique à 37 °C en allumant l’instrument.
- Hydratez une cartouche en ajoutant 200 μL d’une solution d’étalonnage et incubez la cartouche dans un incubateur sans CO2 pendant la nuit (figure 2A). L’humidité de l’incubateur sans CO2 n’est pas importante pour l’hydratation de la cartouche.
- Une heure avant l’expérience, trempez la plaque plusieurs fois de haut en bas, ce qui aidera à éliminer les bulles d’air.
- Concevez la carte des plaques sur le logiciel dans le test de stress de glycolyse par défaut-injection aiguë, en suivant les instructions du test.
- Cliquez sur l’icône du logiciel, puis cliquez sur Test d’injection aiguë de stress de glycolyse. Sur l’icône de définition de groupe, générez des noms de groupe.
- Il y a cinq cycles de mesure d’une durée de 18 minutes et quatre injections. Changez l’injection de la porte A en glucose, de la voie B en oligomycine, de la porte C en roténone et de l’antimycine A (Rot/AA) et de la porte D en 2DG.
- Remettre les cellules en suspension dans le milieu RPMI-C à 10 % et amorcer 50 000 cellules par puits, à l’exception des quatre bords de la plaque (A1, A12, H1 et H12 ; Ajouter uniquement du milieu (pas de cellules) dans une microplaque d’analyseur de flux métabolique jusqu’à un volume final de 100 μL. Normalement, un minimum de 40 000 cellules est nécessaire pour effectuer ce test.
- Laissez les cellules reposer à température ambiante pendant 45 minutes pour éviter l’effet de bord des cellules. L’effet de bord se produit lorsque le milieu autour du périmètre de la plaque s’évapore partiellement, ce qui provoque des changements de volume et de concentration et réduit la viabilité cellulaire.
- Ajoutez 10 ng/mL de LPS pour polariser les macrophages naïfs vers les cellules de type M1 et ajoutez 20 ng/ml d’IL-4 pour les polariser vers les cellules de type M2. Utilisez au moins 3 à 6 puits par condition (figure 2B).
- Vérifiez les cellules au microscope et placez la plaque dans un incubateur à 37 °C et 5 % de CO2 pendant 24 heures.
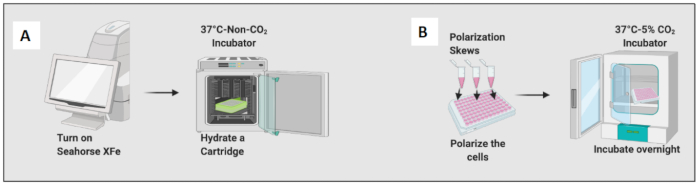
Figure 2 : Démonstration graphique de l’ensemencement et de la polarisation des cellules. (A) Configuration de l’analyseur de flux extracellulaire et hydratation de la cartouche ; (B) Polarisation des cellules et incubation pendant la nuit. Veuillez cliquer ici pour voir une version agrandie de cette figure.
8. Jour du test : Préparation du milieu XF et du composé
- Compléter 100 mL de milieu de dosage XF RPMI (pH 7,4) avec 2 mM de glutamine.
- Stérilisez le média à l’aide d’un système de filtre sous vide de 0,2 μm.
- Placez le milieu de dosage dans un bain-marie à 37°C pendant 20 min.
- Retirez les cellules plaquées de l’incubateur à 37 °C à 5 % de CO2 . Lavez les cellules avec un milieu de test deux fois et remplacez le milieu précédent par un milieu de test jusqu’à ce qu’il atteigne le volume final de 180 μL.
- Utilisez un microscope pour vous assurer que tous les puits ont des cellules confluentes et marquez tous les puits qui ont des rayures dues au pipetage. S’il y a des rayures, retirez cette plaque avant d’analyser.
- Placez la plaque contenant la cellule dans un incubateur sans CO2 pendant 45 min (Figure 3A).
- Utilisation des composés et du milieu de dosage pour fabriquer des solutions mères de glucose (100 mM), d’oligomycine (100 μM), de pourriture/AA (50 μM) et de 2DG (500 mM) (tableau 1).
- Faire un mélange d’injection 10x de chaque composé à l’aide d’un milieu de dosage (tableau 2).
| Stocks d’injection (fournis dans les kits) | Ajouter un milieu de test complet (mL) | Concentration finale du stock (μM) |
| Glucose | 3 | 100K |
| Oligomycine | 0.72 | 100 |
| 2-DG | 3 | 100 000 |
Tableau 1. Crosses d’injection
| Ports sur la cartouche | Solutions mères | Ajouter du volume de stock | Ajouter un milieu de test | Concentration finale des injections (10x) | Ajoutez ce volume au port désigné (μL) | Concentration finale après injection dans chaque puits |
| Un | Glucose (100 mM) | 3000 μL + 0 μL | 100 millions d’euros | 20 | 10 millions d’euros | |
| B | Oligomycine (100 μM) | 300 μL + 2700 μL | 10 μM | 22 | 1,0 μM | |
| C | Roténone/Antimycine A (50 μM) | 300 μL + 2700 μL | 5 μM | 25 | 0,5 μM | |
| D | 2-DG (500 mM) | 300 μL + 0 μL | 500 mM | 28 | 50 millions d’euros | |
Tableau 2. Concentrations finales par injection
9. Le jour du test : Exécution du test glycolytique aigu sur des macrophages polarisés
- Ouvrez le modèle de test de stress de glycolyse (injection aiguë) enregistré à partir du logiciel. Le test de glyco-stress aigu par défaut comporte 3 minutes de mélange et de mesure avant chaque injection.
- Vérifiez le modèle et les détails du test, et lorsque vous êtes prêt, cliquez sur Exécuter et suivez les instructions du test par défaut. Cependant, tous les paramètres peuvent être personnalisés.
- Retirez la cartouche de capteur de l’incubateur sans CO2 , retirez le couvercle et insérez-la dans l’instrument de manière à ce que le puits A1 de la plaque de cartouche tombe dans le coin supérieur gauche du panneau d’insertion de la machine. Habituellement, l’étalonnage prend entre 20 et 45 min.
- Une fois l’étalonnage terminé, l’appareil éjectera la plaque contenant la solution d’étalonnage et maintiendra la cartouche du capteur. Retirez le calibrateur contenant la plaque.
- Retirez la plaque cellulaire de l’incubateur sans CO2 , retirez le couvercle de la plaque et insérez-la dans la machine. Cliquez sur Exécuter (Figure 3B).
- Une fois le test terminé, la machine éjecte la plaque cellulaire et la cartouche du capteur.
- Retirez le support de la plaque et congelez-le à -20 °C pour une normalisation supplémentaire.
- Utilisez le kit de test de prolifération cellulaire commercial (par exemple, CyQUANT) pour normaliser les cellules.
- Ajouter 1 mL de composé B ou de tampon de lyse à 19 mL d’eau distillée sans nucléases.
- Ajouter 100 μL de solution de travail du composé A ou GR à la solution susmentionnée.
- Assurez-vous que les cellules de la plaque sont décongelées, puis ajoutez 200 μL de la solution dans chaque puits.
- Incuber 5 min à température ambiante (RT).
- Mesurez la fluorescence dans des longueurs d’onde d’excitation de 480 nm et d’émission de 520 nm à l’aide d’un lecteur de plaques.
- Normalisez les cellules sur le panneau de normalisation du logiciel.
- Normaliser les cellules en fonction du nombre de macrophages naïfs (Figure 3C). Considérons la moyenne des macrophages naïfs comme 1 (en divisant le nombre de cellules de chaque puits par le nombre moyen de cellules de macrophages naïfs) et appliquez-les à tous les macrophages.
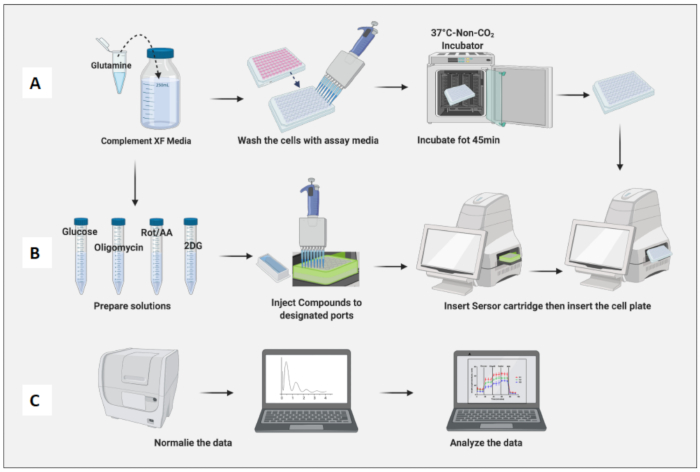
Figure 3 : Jour de l’essai : préparation du milieu et du composé et exécution de l’essai. (A) Préparation des cellules pour l’essai ; (B) la préparation des composés, l’étalonnage et l’exécution de l’essai ; (C) Normalisation et analyse des données. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Résultats
La glycolyse et la phosphorylation oxydative mitochondriale sont les deux principales voies de production d’ATP dans les cellules (Figure 4A). Certaines cellules ont la capacité de passer d’une voie à l’autre pour répondre à leurs besoins énergétiques. La conversion du glucose en pyruvate dans le cytoplasme s’appelle la glycolyse. Le pyruvate a deux destins ; il sera soit converti en lactate, soit métabolisé d...
Discussion
Comme mentionné précédemment, l’analyseur de flux extracellulaire peut fournir des informations en temps réel sur deux principales voies de production d’énergie des cellules en mesurant l’OCR (taux de consommation d’oxygène), un indicateur de l’activité mitochondriale OXPHOS, et l’ECAR (taux d’acidification extracellulaire) qui est un indicateur de la glycolyse. Les macrophages peuvent utiliser les deux voies, en fonction de leur microenvironnement. Ils peuvent égal...
Déclarations de divulgation
Les auteurs n’ont rien à divulguer.
Remerciements
Nous remercions Mme Joanna Rocha pour son aide rédactionnelle. Le travail a été partiellement soutenu par les National Institutes of Health (NIH) R01DK118334 (aux Drs Sun et Alaniz) et (NIH) R01A11064Z (aux Drs Jayaraman et Alaniz).
matériels
| Name | Company | Catalog Number | Comments |
| 23G needles | VWR | BD305145 | |
| 2-mercaptoethanol | Life Technologies | 21985023 | |
| 50ml Conical Tube | VWR | 21008-951 | |
| ACK lysis buffer | Thermo Fisher Scientific | A1049201 | It can be lab-made |
| Agilent Seahorse XF glycolysis stress test kit | Agilent Technologies | 103020-100 | |
| Agilent Seahorse XF Glycolysis Stress Test Kit User Guide | Agilent Technologies | 103020-400 | |
| Agilent Seahorse XF Glycolytic Rate Assay Kit | Agilent Technologies | 103344-100 | |
| Agilent Seahorse XF Glycolytic Rate Assay Kit User Guide | Agilent Technologies | 103344-100 | |
| Alexa Fluor 488 anti-mouse CD206 (MMR) Antibody | BioLegend | 141710 | |
| anti-mouse CD11b eFluor450 100ug | eBioscience | 48-0112-82 | |
| BD 3ML - SYRINGE | VWR | BD309657 | Other syringes are acceptable too |
| Cell counter-Vi-CELL- XR Complete System | BECKMAN COULTER Life Sciences | 731050 | Cells can be manually counted too |
| Cell Strainer-70µm | VWR | 10199-656 | |
| CyQUANT Cell Proliferation Assay Kit | Thermo Fisher Scientific | C7026 | |
| F4/80 monoclonal antibody (BM8) pe-Cyanine7 | eBioscience | 25-4801-82 | |
| Fetal Bovine Serum | Life Technologies | 16000-044 | |
| Flow cytometer: BD LSFRFortessa X-20 | BD | 656385 | |
| Kim Wipes | VWR | 82003-822 | |
| LPS-SM ultrapure (tlrl-smpls) 5 mg | Invivogen | tlrl-smlps | |
| MCSF | Peprotech | 315-02 | |
| Murine IL-4 | Peprotech | 214-14 | |
| PE Rat Anti-Mouse CD38 | BD Biosciences | 553764 | |
| Penicillin-Streptomycin (10,000 U/mL) | Life Technologies | 15140122 | |
| Petri Dish 100mm x 15 mm | Fisher Scientific | F80875712 | |
| RPMI, Glutamax, HEPES | Invitrogen | 72400-120 | |
| Seahorse Calibrant Solution | Agilent Technologies | 103059-000 | |
| Seahorse XF 200mM Glutamine Solution | Agilent Technologies | 103579-100 | |
| Seahorse XF Glycolytic Rate Assay Kit | Agilent Technologies | 103344-100 | |
| Seahorse XFe96 FluxPaks | Agilent Technologies | 102416-100 | |
| XF Glycolysis Stress Test Kit | Agilent Technologies | 103020-100 | |
| XF RPMI Medium, pH 7.4 without phenol Red | Agilent Technologies | 103336-100 |
Références
- Rosowski, E. E. Determining macrophage versus neutrophil contributions to innate immunity using larval zebrafish. Disease Models & Mechanisms. 13 (1), (2020).
- Martinez-Pomares, L., Gordon, S. . The Autoimmune Diseases. , 191-212 (2020).
- Orecchioni, M., Ghosheh, Y., Pramod, A. B., Ley, K. Macrophage polarization: different gene signatures in M1 (LPS+) vs. classically and M2- (LPS–) vs. alternatively activated macrophages. Frontiers in immunology. 10, 1084 (2019).
- Barrett, T. J. Macrophages in atherosclerosis regression. Arteriosclerosis, Thrombosis, and Vascular Biology. 40 (1), 20-33 (2020).
- Ni, Y., et al. Adipose tissue macrophage phenotypes and characteristics: the key to insulin resistance in obesity and metabolic disorders. Obesity. 28 (2), 225-234 (2020).
- Chu, C., et al. Modulation of foreign body reaction and macrophage phenotypes concerning microenvironment. Journal of Biomedical Materials Research Part A. 108 (1), 127-135 (2020).
- Batista-Gonzalez, A., Vidal, R., Criollo, A., Carreño, L. J. New insights on the role of lipid metabolism in the metabolic reprogramming of macrophages. Frontiers in Immunology. 10, 2993 (2020).
- Kang, S., Kumanogoh, A. The spectrum of macrophage activation by immunometabolism. International Immunology. , (2020).
- Shi, Y., et al. M1 But Not M0 Extracellular Vesicles Induce Polarization of RAW264. 7 Macrophages Via the TLR4-NFκB Pathway In Vitro. Inflammation. , 1-9 (2020).
- Zhang, L., Li, S. Lactic acid promotes macrophage polarization through MCT-HIF1α signaling in gastric cancer. Experimental Cell Research. 388 (2), 111846 (2020).
- Geng, T., et al. CD137 signaling induces macrophage M2 polarization in atherosclerosis through STAT6/PPARδ pathway. Cellular Signalling. , 109628 (2020).
- Liu, Q., et al. Combined blockade of TGf-β1 and GM-CSF improves chemotherapeutic effects for pancreatic cancer by modulating tumor microenvironment. Cancer Immunology & Immunotherapy. , 1-16 (2020).
- Rigoni, T. S., et al. RANK Ligand Helps Immunity to Leishmania major by Skewing M2 Into M1 Macrophages. Frontiers in Immunology. 11, 886 (2020).
- Viola, A., Munari, F., Sánchez-Rodríguez, R., Scolaro, T., Castegna, A. The metabolic signature of macrophage responses. Frontiers in Immunology. 10, (2019).
- Ivashkiv, L. B. The hypoxia–lactate axis tempers inflammation. Nature Reviews Immunology. 20 (2), 85-86 (2020).
- Van den Bossche, J., Baardman, J., de Winther, M. P. Metabolic characterization of polarized M1 and M2 bone marrow-derived macrophages using real-time extracellular flux analysis. Journal of Visualized Experiments. (105), e53424 (2015).
- Van den Bossche, J., et al. Mitochondrial dysfunction prevents repolarization of inflammatory macrophages. Cell reports. 17 (3), 684-696 (2016).
- Liu, P. S., Ho, P. C. . Metabolic Signaling. , 173-186 (2019).
- Wang, F., et al. Glycolytic stimulation is not a requirement for M2 macrophage differentiation. Cell metabolism. 28 (3), 463-475 (2018).
- Romero, N., Rogers, G., Neilson, A., Dranka, B. . Quantifying Cellular ATP Production Rate Using Agilent Seahorse XF Technology. , (2018).
- Mookerjee, S. A., Brand, M. D. Measurement and analysis of extracellular acid production to determine glycolytic rate. Journal of Visualized Experiments. (106), e53464 (2015).
- Mookerjee, S. A., Goncalves, R. L., Gerencser, A. A., Nicholls, D. G., Brand, M. D. The contributions of respiration and glycolysis to extracellular acid production. Biochimica Et Biophysica Acta-Bioenergetics. 1847 (2), 171-181 (2015).
- Yang, S., et al. Macrophage polarization in atherosclerosis. Clinica Chimica Acta. 501, 142-146 (2020).
- Hörhold, F., et al. Reprogramming of macrophages employing gene regulatory and metabolic network models. PLoS Computational Biology. 16 (2), e1007657 (2020).
- Han, X., Ma, W., Zhu, Y., Sun, X., Liu, N. Advanced glycation end products enhance macrophage polarization to the M1 phenotype via the HIF-1α/PDK4 pathway. Molecular and Cellular Endocrinology. , 110878 (2020).
- Müller, E., et al. Toll-like receptor ligands and interferon-γ synergize for induction of antitumor M1 macrophages. Frontiers in Immunology. 8, 1383 (2017).
- Soto-Heredero, G., Gómez de las Heras, M. M., Gabandé-Rodríguez, E., Oller, J., Mittelbrunn, M. Glycolysis–a key player in the inflammatory response. The FEBS Journal. , (2020).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon