Method Article
Kryo-EM- und Einzelpartikelanalyse mit Scipion
In diesem Artikel
Zusammenfassung
Die Einzelpartikelanalyse in der Kryo-Elektronenmikroskopie ist eine der wichtigsten Techniken, um die Struktur biologischer Ensembles mit hoher Auflösung zu bestimmen. Scipion bietet die Werkzeuge, um die gesamte Pipeline zu erstellen, um die vom Mikroskop erfassten Informationen zu verarbeiten und eine 3D-Rekonstruktion der biologischen Probe zu erreichen.
Zusammenfassung
Die Kryo-Elektronenmikroskopie ist zu einem der wichtigsten Werkzeuge in der biologischen Forschung geworden, um die Strukturinformationen von Makromolekülen mit nahezu atomarer Auflösung aufzudecken. Bei der Einzelpartikelanalyse wird die verglaste Probe durch einen Elektronenstrahl abgebildet und die Detektoren am Ende der Mikroskopsäule produzieren Filme dieser Probe. Diese Filme enthalten Tausende von Bildern identischer Partikel in zufälligen Orientierungen. Die Daten müssen einen Bildverarbeitungs-Workflow mit mehreren Schritten durchlaufen, um das endgültige rekonstruierte 3D-Volumen zu erhalten. Ziel des Bildverarbeitungs-Workflows ist es, die Erfassungsparameter zu identifizieren, um die untersuchte Probe rekonstruieren zu können. Scipion stellt alle Werkzeuge zur Verfügung, um diesen Workflow mit mehreren Bildverarbeitungspaketen in einem integrativen Rahmen zu erstellen, was auch die Rückverfolgbarkeit der Ergebnisse ermöglicht. In diesem Artikel wird der gesamte Bildverarbeitungsablauf in Scipion vorgestellt und mit Daten aus einem echten Testfall diskutiert, der alle details enthält, die notwendig sind, um von den vom Mikroskop erhaltenen Filmen zu einer hochauflösenden endgültigen 3D-Rekonstruktion zu gelangen. Außerdem wird die Leistungsfähigkeit der Verwendung von Konsenswerkzeugen diskutiert, die es ermöglichen, Methoden zu kombinieren und Ergebnisse in jedem Schritt des Workflows zu bestätigen, wodurch die Genauigkeit der erhaltenen Ergebnisse verbessert wird.
Einleitung
In der Kryo-Elektronenmikroskopie (Kryo-EM) ist die Einzelpartikelanalyse (SPA) von vitrifizierten gefrorenen hydratisierten Proben eine der am weitesten verbreiteten und erfolgreichsten Varianten der Bildgebung für biologische Makromoleküle, da sie es ermöglicht, molekulare Wechselwirkungen und die Funktion biologischer Ensembles zu verstehen1. Dies ist den jüngsten Fortschritten in dieser Bildgebungstechnik zu verdanken, die zur "Auflösungsrevolution"2 geführt haben und die erfolgreiche Bestimmung biologischer 3D-Strukturen mit nahezu atomarer Auflösung ermöglicht haben. Derzeit betrug die höchste Auflösung, die in SPA-Kryo-EM erreicht wurde, 1,15 Å für Apoferritin3 (EMDB-Eintrag: 11668). Diese technologischen Fortschritte umfassen Verbesserungen in der Probenvorbereitung4, der Bildaufnahme5 und den Bildverarbeitungsmethoden6. Dieser Artikel konzentriert sich auf diesen letzten Punkt.
Kurz gesagt, das Ziel der Bildverarbeitungsmethoden ist es, alle Erfassungsparameter zu identifizieren, um den Bildgebungsprozess des Mikroskops umzukehren und die 3D-Struktur der untersuchten biologischen Probe wiederherzustellen. Diese Parameter sind der Gewinn der Kamera, die strahlinduzierte Bewegung, die Aberrationen des Mikroskops (hauptsächlich die Defokussierung), die 3D-Winkelorientierung und -verschiebung jedes Partikels und der Konformationszustand im Falle einer Probe mit Konformationsänderungen. Die Anzahl der Parameter ist jedoch sehr hoch und Kryo-EM erfordert die Verwendung von niedrig dosierten Bildern, um Strahlenschäden zu vermeiden, was das Signal-Rausch-Verhältnis (SNR) der aufgenommenen Bilder erheblich reduziert. Somit kann das Problem nicht eindeutig gelöst werden und alle zu berechnenden Parameter können nur Schätzungen sein. Entlang des Bildverarbeitungs-Workflows sollten die richtigen Parameter identifiziert und die verbleibenden verworfen werden, um schließlich eine hochauflösende 3D-Rekonstruktion zu erhalten.
Die vom Mikroskop erzeugten Daten werden in Frames gesammelt. Vereinfacht ausgedrückt enthält ein Rahmen die Anzahl der Elektronen, die an einer bestimmten Position (Pixel) im Bild angekommen sind, wenn Elektronenzähldetektoren verwendet werden. In einem bestimmten Sichtfeld werden mehrere Frames gesammelt und dies wird als Film bezeichnet. Da niedrige Elektronendosen verwendet werden, um Strahlenschäden zu vermeiden, die die Probe zerstören könnten, ist der SNR sehr niedrig und die Rahmen, die demselben Film entsprechen, müssen gemittelt werden, um ein Bild zu erhalten, das strukturelle Informationen über die Probe enthüllt. Es wird jedoch nicht nur ein einfacher Mittelwert angewendet, die Probe kann während der Bildgebungszeit aufgrund der strahlinduzierten Bewegung, die kompensiert werden muss, Verschiebungen und andere Arten von Bewegungen erleiden. Die shift-kompensierten und gemittelten Frames stammen aus einer Mikroaufnahme.
Sobald die Mikroaufnahmen erhalten sind, müssen wir die vom Mikroskop für jeden von ihnen eingeführten Aberrationen abschätzen, die als Kontrasttransferfunktion (CTF) bezeichnet werden und die die Änderungen des Kontrasts der Mikroaufnahme als Funktion der Frequenz darstellen. Dann können die Partikel ausgewählt und extrahiert werden, was als Partikelpicking bezeichnet wird. Jedes Partikel sollte ein kleines Bild sein, das nur eine Kopie der untersuchten Probe enthält. Es gibt drei Familien von Algorithmen für die Partikelauswahl: 1) diejenigen, die nur eine grundlegende Parametrisierung des Aussehens des Partikels verwenden, um sie in der gesamten Menge von Mikroaufnahmen (z. B. Partikelgröße) zu finden, 2) diejenigen, die lernen, wie die Partikel vom Benutzer oder einem vortrainierten Satz aussehen, und 3) diejenigen, die Bildvorlagen verwenden. Jede Familie hat unterschiedliche Eigenschaften, die später angezeigt werden.
Der extrahierte Satz von Partikeln, die in den Mikroaufnahmen gefunden wurden, wird in einem 2D-Klassifizierungsprozess verwendet, der zwei Ziele verfolgt: 1) Reinigung des Partikelsatzes durch Verwerfen der Teilmenge, die reine Rauschbilder, überlappende Partikel oder andere Artefakte enthält, und 2) die gemittelten Partikel, die jede Klasse darstellen, könnten als Ersteinformation zur Berechnung eines 3D-Anfangsvolumens verwendet werden.
Die 3D-Anfangsvolumenberechnung ist der nächste entscheidende Schritt. Das Problem, die 3D-Struktur zu erhalten, kann als Optimierungsproblem in einer mehrdimensionalen Lösungslandschaft angesehen werden, in der das globale Minimum das beste 3D-Volumen ist, das die ursprüngliche Struktur darstellt, aber mehrere lokale Minima, die suboptimale Lösungen darstellen, gefunden werden können und wo es sehr leicht ist, gefangen zu werden. Das Anfangsvolumen stellt den Ausgangspunkt für den Suchprozess dar, so dass eine schlechte anfängliche Volumenschätzung uns daran hindern könnte, das globale Minimum zu finden. Vom Anfangsvolumen aus hilft ein 3D-Klassifikationsschritt, verschiedene Konformationszustände zu entdecken und den Partikelsatz erneut zu reinigen. Ziel ist es, eine strukturell homogene Population von Partikeln zu erhalten. Danach ist ein 3D-Verfeinerungsschritt dafür verantwortlich, die Winkel- und Translationsparameter für jedes Partikel zu verfeinern, um das bestmögliche 3D-Volumen zu erhalten.
Schließlich kann in den letzten Schritten die erhaltene 3D-Rekonstruktion geschärft und poliert werden. Das Schärfen ist ein Prozess der Steigerung der hohen Frequenzen des rekonstruierten Volumens, und das Polieren ist ein Schritt, um einige Parameter wie CTF oder strahlinduzierte Bewegungskompensation auf der Ebene von Partikeln weiter zu verfeinern. Außerdem könnten einige Validierungsverfahren verwendet werden, um die erreichte Auflösung am Ende des Workflows besser zu verstehen.
Nach all diesen Schritten werden die Tracing- und Docking-Prozesse7 dazu beitragen, der erhaltenen 3D-Rekonstruktion eine biologische Bedeutung zu verleihen, indem atomare Modelle de novo gebaut oder bestehende Modelle angepasst werden. Wenn eine hohe Auflösung erreicht wird, werden uns diese Prozesse die Positionen der biologischen Strukturen, sogar der verschiedenen Atome, in unserer Struktur mitteilen.
Scipion8 ermöglicht es, den gesamten Workflow zu erstellen und die relevantesten Bildverarbeitungspakete auf integrative Weise zu kombinieren. Xmipp9, Relion10, CryoSPARC11, Eman12, Spider13, Cryolo14, Ctffind15, CCP416, Phenix17 und viele weitere Pakete können in Scipion enthalten sein. Darüber hinaus enthält es alle notwendigen Werkzeuge, um die Integration, Interoperabilität, Rückverfolgbarkeit und Reproduzierbarkeit zu unterstützen, um eine vollständige Verfolgung des gesamten Bildverarbeitungs-Workflows zu ermöglichen8.
Eines der mächtigsten Werkzeuge, die Scipion uns ermöglicht, ist der Konsens, was bedeutet, die Ergebnisse mit mehreren Methoden in einem Schritt der Verarbeitung zu vergleichen und eine Kombination der durch verschiedene Methoden vermittelten Informationen zu erstellen, um eine genauere Ausgabe zu erzeugen. Dies könnte dazu beitragen, die Leistung zu steigern und die erreichte Qualität in den geschätzten Parametern zu verbessern. Beachten Sie, dass ein einfacherer Workflow ohne die Verwendung von Konsensmethoden erstellt werden kann. Wir haben jedoch die Leistungsfähigkeit dieses Tools gesehen22,25 und der in diesem Manuskript vorgestellte Workflow wird es in mehreren Schritten verwenden.
Alle Schritte, die in den vorherigen Absätzen zusammengefasst wurden, werden im folgenden Abschnitt ausführlich erläutert und mit Scipion in einem kompletten Workflow zusammengefasst. Außerdem wird gezeigt, wie die Konsensus-Tools verwendet werden können, um eine höhere Übereinstimmung in den generierten Ergebnissen zu erzielen. Dazu wurde der Beispieldatensatz des Plasmodium falciparum 80S Ribosoms ausgewählt (EMPIAR-Eintrag: 10028, EMDB-Eintrag: 2660). Der Datensatz besteht aus 600 Filmen mit 16 Bildern der Größe 4096x4096 Pixel bei einer Pixelgröße von 1,34 Å, die an einer FEI POLARA 300 mit einer FEI FALCON II-Kamera aufgenommen wurden, wobei die gemeldete Auflösung bei EMDB 3,2 Å18 beträgt.
Protokoll
1. Erstellen eines Projekts in Scipion und Importieren der Daten
- Öffnen Sie Scipion und klicken Sie auf Projekt erstellen, geben Sie den Namen für das Projekt und den Speicherort an (ergänzende Abbildung 1). Scipion öffnet das Projektfenster mit einer Leinwand mit auf der linken Seite einem Bereich mit einer Liste der verfügbaren Methoden, von denen jede ein Bildverarbeitungswerkzeug darstellt, das zur Verwaltung von Daten verwendet werden kann.
Hinweis: Strg+F kann verwendet werden, um eine Methode zu finden, wenn sie nicht in der Liste angezeigt wird. - Um die vom Mikroskop aufgenommenen Filme zu importieren, wählen Sie das pwem - Importieren Sie Filme im linken Bereich (oder geben Sie es ein, wenn Sie Strg + F drücken).
- Es wird ein neues Fenster geöffnet (ergänzende Abbildung 2). Geben Sie dort den Pfad zu den Daten und die Erfassungsparameter an. Verwenden Sie in diesem Beispiel den folgenden Aufbau: Mikroskopspannung 300 kV, Sphärische Aberration 2,0 mm, Amplitudenkontrast 0,1, Vergrößerungsrate 50000, Abtastrate in Vom Bild und Pixelgröße 1,34 Å. Wenn alle Parameter im Formular ausgefüllt sind, klicken Sie auf die Schaltfläche Ausführen .
HINWEIS: Wenn eine Methode gestartet wird, wird auf der Leinwand ein Feld in gelber Farbe mit der Bezeichnung "Ausgeführt" angezeigt. Wenn eine Methode abgeschlossen ist, ändert sich das Feld in Grün und die Beschriftung in "Fertig". Im Falle eines Fehlers während der Ausführung einer Methode wird das Feld rot und als fehlgeschlagen gekennzeichnet angezeigt. Überprüfen Sie in diesem Fall den unteren Teil der Canvas, auf der Registerkarte Ausgabeprotokoll wird eine Erklärung des Fehlers angezeigt. - Wenn die Methode abgeschlossen ist, überprüfen Sie die Ergebnisse im unteren Teil der Arbeitsfläche auf der Registerkarte Zusammenfassung . Hier werden die von der Methode erzeugten Ausgaben dargestellt, in diesem Fall die Menge der Filme. Klicken Sie auf die Schaltfläche Ergebnisse analysieren und ein neues Fenster mit der Liste der Filme erscheint.
2. Filmausrichtung: von Filmen zu Mikroaufnahmen
- Verwenden Sie die Methode xmipp3 - optical alignment , die Optical flow19 implementiert. Verwenden Sie die folgenden Parameter, um das Formular auszufüllen (ergänzende Abbildung 3): Die Eingabefilme sind diejenigen, die in Schritt 1 erhalten wurden, der Bereich in Frames to ALIGN liegt zwischen 2 und 13, die anderen Optionen bleiben bei den Standardwerten. Führen Sie das Programm aus.
HINWEIS: Die fett gedruckten Parameter in einem Formular müssen immer ausgefüllt werden. Die anderen haben einen Standardwert oder sind nicht zwingend erforderlich. Im oberen Teil des Formularfensters befinden sich die Felder, in denen die Rechenressourcen verteilt sind, als Threads, MPIs oder GPUs. - Klicken Sie auf Ergebnisse analysieren , um die erhaltenen Mikroaufnahmen und die Trajektorie der geschätzten Verschiebungen zu überprüfen (Abbildung 1). Für jede gesehene Mikroaufnahme: Schauen Sie sich die spektrale Leistungsdichte (PSD), die Trajektorien an, die erhalten wurden, um den Film (einen Punkt pro Bild) in kartesischen und polaren Koordinaten auszurichten, und den Dateinamen der erhaltenen Mikroaufnahme (wenn Sie darauf klicken, kann die Mikroaufnahme inspiziert werden). Beachten Sie, dass die Partikel der Probe in der Mikroaufnahme viel besser sichtbar sind als in einem einzelnen Bild des Films.
3. CTF-Schätzung: Berechnung der Aberrationen des Mikroskops
- Verwenden Sie zunächst die Methode grigoriefflab - ctffind15. Der Aufbau ist: Die Eingangsmikroskope sind die Ausgabe von Schritt 2, der manuelle CTF-Downsampling-Faktor ist auf 1,5 eingestellt und der Auflösungsbereich reicht von 0,06 bis 0,42. Legen Sie außerdem in den erweiterten Optionen (die Sie finden, indem Sie diese Auswahl in der Expertenebene des Formulars auswählen) die Fenstergröße auf 256 fest. Die übrigen Parameter bleiben bei den Standardwerten (Ergänzende Abbildung 4).
HINWEIS: In den meisten Methoden in Scipion zeigt die Option Erweitert mehr Konfigurationsparameter an. Verwenden Sie diese Optionen mit Bedacht, wenn das zu startende Programm vollständig bekannt ist und die Bedeutung der Parameter verstanden ist. Einige Parameter können schwierig zu füllen sein, ohne einen Blick auf die Daten zu werfen. In diesem Fall zeigt Scipion auf der rechten Seite einen Zauberstab, der ein Assistentenfenster anzeigt (ergänzende Abbildung 5). Zum Beispiel ist im Feld Auflösung dieses Formulars besonders nützlich, da diese Werte ausgewählt werden sollten, um den Bereich von der ersten Null bis zum letzten wahrnehmbaren Ring der PSD ungefähr abzudecken. - Klicken Sie auf Ausführen und auf Ergebnisse analysieren (Abbildung 2), wenn die Methode abgeschlossen ist. Überprüfen Sie, ob die geschätzte CTF mit der experimentellen übereinstimmt. Schauen Sie sich zu diesem Zweck die PSD an und vergleichen Sie die geschätzten Ringe in der Ecke mit denen, die aus den Daten stammen. Überprüfen Sie auch die erhaltenen Defokussierungswerte, um unerwartete Werte zu finden, und die entsprechenden Mikroaufnahmen können verworfen oder neu berechnet werden. In diesem Beispiel kann der gesamte Satz von Mikroaufnahmen verwendet werden.
HINWEIS: Verwenden Sie die Schaltflächen im unteren Teil des Fensters, um eine Teilmenge der Mikroaufnahmen zu erstellen (mit der roten Schaltfläche Mikroaufnahmen ) und bei Bedarf eine CTF neu zu berechnen (mit der roten Schaltfläche CTFs neu berechnen ). - Um die vorherige Schätzung zu verfeinern, verwenden Sie xmipp3 - ctf estimation20. Wählen Sie als Eingabemikroskope die Ausgabe von Schritt 2 aus, wählen Sie die Option Defoci aus einer früheren CTF-Schätzung verwenden, als Vorherige CTF-Schätzung wählen Sie die Ausgabe von grigoriefflab - ctffind und ändern Sie in der Stufe Erweitert die Fenstergröße auf 256 (Ergänzende Abbildung 6). Führen Sie es aus.
- Klicken Sie auf Ergebnisse analysieren , um die erhaltenen CTFs zu überprüfen. Mit dieser Methode werden mehr Daten geschätzt und in einigen zusätzlichen Spalten dargestellt. Da keiner von ihnen falsche Schätzwerte zeigt, werden alle Mikroaufnahmen in den folgenden Schritten verwendet.
4. Partikelpicking: Finden von Partikeln in den Mikroaufnahmen
- Bevor Sie mit der Kommissionierung beginnen, führen Sie einen Vorprozess der Mikroaufnahmen durch. Öffnen Sie xmipp3 - Preprocess micrographs, setze als Input micrographs die in Schritt 2 erhaltenen und wähle die Optionen Remove bad pixels? with Multiple of Stddev to 5, und Downsample micrographs? with a Downsampling factor of 2 (Ergänzende Abbildung 7). Klicken Sie auf Ausführen und überprüfen Sie, ob die Größe der resultierenden Mikroaufnahmen reduziert wurde.
- Für die Kommissionierung verwenden Sie xmipp3 - manuelle Kommissionierung (Schritt 1) und xmipp3 - automatische Kommissionierung (Schritt 2)21. Die manuelle Kommissionierung ermöglicht die manuelle Vorbereitung eines Partikelsatzes, mit dem der Auto-Picking-Schritt lernt und den vollständigen Partikelsatz generiert. Führen Sie zunächst xmipp3 - manuelles Picking (Schritt 1) mit Eingabemikroskopen wie die im vorherigen Vorprozess erhaltenen Mikroaufnahmen aus. Klicken Sie auf Ausführen und ein neues interaktives Fenster erscheint (Abbildung 3).
- In diesem Fenster wird eine Liste der Mikroaufnahmen (Abbildung 3a) und anderer Optionen angezeigt. Ändern Sie die Größe (px) in 150, dies ist die Größe der Box, die jedes Partikel enthält. Die ausgewählte Mikroaufnahme wird in einem größeren Fenster angezeigt. Wählen Sie einen Bereich aus, und wählen Sie alle sichtbaren Partikel darin aus (Abbildung 3b). Klicken Sie dann auf Training aktivieren , um das Lernen zu starten. Die verbleibenden Bereiche der Mikroaufnahme werden automatisch ausgewählt (Abbildung 3c). Überprüfen Sie die ausgewählten Partikel und fügen Sie weitere hinzu, indem Sie darauf klicken, oder entfernen Sie die falschen bei Bedarf mit Umschalt + Klicken.
- Wählen Sie im ersten Fenster die nächste Mikroaufnahme aus. Die Mikroaufnahme wird automatisch ausgewählt. Überprüfen Sie erneut, ob Sie bei Bedarf einige Partikel einschließen oder entfernen. Wiederholen Sie diesen Schritt mit ungefähr 5 Mikroaufnahmen, um ein repräsentatives Trainingsset zu erstellen.
- Sobald dies erledigt ist, klicken Sie im Hauptfenster auf Koordinaten , um die Koordinaten aller ausgewählten Partikel zu speichern. Der Trainingssatz der Partikel ist bereit, zum automatischen Picking zu gehen, um den Prozess für alle Mikroaufnahmen abzuschließen.
- Öffnen Sie xmipp3 - Auto-Picking (Schritt 2), das in Xmipp partikel picking die vorherige manuelle Kommissionierung ausführt und Mikrographen als Wie überwacht auswählt. Klicken Sie auf Ausführen. Diese Methode generiert als Ausgabe einen Satz von etwa 100000 Koordinaten.
- Wenden Sie einen Konsensansatz an, also führen Sie eine zweite Picking-Methode durch, um die Partikel auszuwählen, in denen beide Methoden übereinstimmen. Öffnen Sie sphire - cryolo picking14 und wählen Sie die vorverarbeiteten Mikroaufnahmen als Eingabemikroskope, Allgemeines Modell verwenden? bis Ja, mit einem Konfidenzschwellenwert von 0,3 und einer Boxgröße von 150 (ergänzende Abbildung 8). Führen Sie es aus. Diese Methode sollte auch etwa 100000 Koordinaten erzeugen.
- Führen Sie xmipp3 aus - deep consensus picking22. Da die Eingabekoordinaten die Ausgabe von Sphire - Kryolo-Picking (Schritt 4.7) und xmipp3 - Auto-Picking (Schritt 4.6) enthalten, wählen Sie Modelltyp auswählen auf Vortrainiert und Überspringen Sie das Training und bewerten Sie direkt mit dem vortrainierten Modell? Zu Ja (ergänzende Abbildung 9). Führen Sie es aus.
- Klicken Sie auf Ergebnisse analysieren und im neuen Fenster auf das Augensymbol neben Partikel/Koordinaten mit hohen 'zScoreDeepLearning1'-Werten auswählen. Es öffnet sich ein neues Fenster mit einer Liste aller Partikel (Abbildung 4). Die zScore-Werte in der Spalte geben einen Einblick in die Qualität eines Partikels, niedrige Werte bedeuten schlechte Qualität.
- Klicken Sie auf das Etikett_xmipp_zScoreDeepLearning , um die Partikel vom höchsten zum niedrigsten zScore zu ordnen. Wählen Sie die Partikel mit einem höheren zScore als 0,75 aus und klicken Sie auf Koordinaten , um die neue Teilmenge zu erstellen. Dadurch sollte eine Teilmenge mit ungefähr 50000 Koordinaten erstellt werden.
- Öffnen Sie xmipp3 - Deep Micrograph Cleaner. Wählen Sie als Eingabekoordinaten die im vorherigen Schritt erhaltene Teilmenge, Die Quelle der Mikroaufnahmen entspricht den Koordinaten aus, und halten Sie den Schwellenwert bei 0,75. Führen Sie es aus. Überprüfen Sie auf der Registerkarte Zusammenfassung , ob die Anzahl der Koordinaten reduziert wurde, obwohl in diesem Fall nur wenige Koordinaten entfernt werden.
HINWEIS: Dieser Schritt ist in der Lage, den Koordinatensatz zusätzlich zu bereinigen und könnte sehr nützlich sein, um andere Datensätze mit mehr Filmartefakten wie Kohlenstoffzonen oder großen Verunreinigungen zu reinigen. - Führen Sie xmipp3 - Partikel extrahieren aus (ergänzende Abbildung 10). Geben Sie als Eingabekoordinaten die nach dem vorherigen Schritt erhaltenen Koordinaten an, die Quelle der Mikroaufnahmen als andere, die Eingabemikroskopie als Ausgabe von Schritt 2, die CTF-Schätzung als Ausgabe der xmipp3- ctf-Schätzung, den Downsampling-Faktor auf 3 und die Partikelfeldgröße auf 100. Wählen Sie auf der Registerkarte Vorverarbeitung des Formulars Ja für alle aus. Führen Sie es aus.
- Überprüfen Sie, ob die Ausgabe die Partikel in einer reduzierten Größe von 100x100 Pixel und einer Pixelgröße von 4,02 Å/px enthalten soll.
- Führen Sie xmipp3 erneut aus - extrahieren Sie Partikel und ändern Sie die folgenden Parameter: Downsampling-Faktor auf 1 und Partikelboxgröße auf 300. Überprüfen Sie, ob die Ausgabe derselbe Satz von Partikeln ist, aber jetzt mit der vollen Auflösung.
5. 2D-Klassifikation: Gruppierung ähnlicher Partikel
- Öffnen Sie die Methode kryosparc2 - 2d classification11 mit Eingabepartikeln wie in Schritt 4.11 erhalten und behalten Sie auf der Registerkarte 2D-Klassifikation die Anzahl der Klassen bis 128 alle anderen Parameter mit den Standardwerten bei. Führen Sie es aus.
- Klicken Sie auf Ergebnisse analysieren und dann auf das Augensymbol neben Partikelklassen mit Scipion anzeigen (Abbildung 5). Diese Klassifizierung hilft, den Partikelsatz zu reinigen, da mehrere Klassen laut oder mit Artefakten erscheinen. Wählen Sie die Klassen mit guten Ansichten aus. Klicken Sie auf Partikel (rote Schaltfläche im unteren Teil des Fensters), um die sauberere Teilmenge zu erstellen.
- Öffnen Sie nun xmipp3 - cl2d23 und legen Sie als Eingabebilder die im vorherigen Schritt erhaltenen Bilder und die Anzahl der Klassen auf 128 fest. Klicken Sie auf Ausführen.
HINWEIS: Diese zweite Klassifizierung wird als zusätzlicher Reinigungsschritt des Partikelsatzes verwendet. Normalerweise ist es nützlich, so viele laute Partikel wie möglich zu entfernen. Wenn jedoch ein einfacherer Workflow gewünscht wird, kann nur eine 2D-Klassifizierungsmethode verwendet werden. - Wenn die Methode abgeschlossen ist, überprüfen Sie die 128 generierten Klassen, indem Sie auf Ergebnisse analysieren und auf Was angezeigt werden soll: Klassen klicken. Die meisten der generierten Klassen zeigen eine Projektion des Makromoleküls mit einem gewissen Detaillierungsgrad. Einige von ihnen erscheinen jedoch laut (in diesem Beispiel ungefähr 10 Klassen). Wählen Sie alle guten Klassen aus und klicken Sie auf die Schaltfläche Klassen , um eine neue Teilmenge mit nur den guten zu generieren. Diese Teilmenge wird als Eingabe für eine der Methoden zum Generieren eines Anfangsvolumes verwendet. Klicken Sie bei den gleichen ausgewählten Klassen auf Partikel , um eine sauberere Teilmenge zu erstellen, nachdem Sie diejenigen entfernt haben, die zu den fehlerhaften Klassen gehören.
- Öffnen Sie pwem - Teilmenge mit vollständiger Menge von Elementen als Ausgabe von 4.13 (alle Partikel in voller Größe), Zufällige Teilmenge auf Nein, Andere Menge als Teilmenge der im vorherigen Schritt erstellten Partikel und Vorgang als Schnittpunkt festlegen. Dadurch wird die vorherige Teilmenge aus den Partikeln mit voller Auflösung extrahiert.
6. Anfängliche Volumenschätzung: Erstellen der ersten Schätzung des 3D-Volumens
- Schätzen Sie in diesem Schritt zwei Anfangsvolumina mit unterschiedlichen Methoden und verwenden Sie dann ein Konsensuswerkzeug, um das endgültige geschätzte 3D-Volumen zu generieren. Öffnen Sie xmipp3 - rekonstruieren Sie die significant24-Methode mit Input-Klassen wie nach Schritt 5, Symmetriegruppe als c1, und behalten Sie die verbleibenden Parameter mit ihren Standardwerten bei (Ergänzende Abbildung 11). Führen Sie es aus.
- Klicken Sie auf Ergebnisse analysieren. Überprüfen Sie, ob ein Volumen mit niedriger Auflösung der Größe 100x100x100 Pixel und einer Pixelgröße von 4,02 Å/px erreicht wird.
- Öffnen Sie xmipp3 - Volumes zuschneiden/größe ändern (ergänzende Abbildung 12) und verwenden Sie als Eingabe-Volumes das im vorherigen Schritt erhaltene Volume ändern? auf Ja, Option Größe ändern auf Abtastrate und Größe ändern auf 1,34 Å/px. Führen Sie es aus. Überprüfen Sie auf der Registerkarte Zusammenfassung , ob das Ausgabevolumen die richtige Größe hat.
- Erstellen Sie nun das zweite Anfangsvolume. Open relion - 3D-Ausgangsmodell10, da Eingabepartikel die guten Partikel bei voller Auflösung (Ausgabe von 5,5) verwenden und Partikelmaskendurchmesser auf 402Å einstellen, behalten Sie die verbleibenden Parameter mit den Standardwerten bei. Führen Sie es aus.
- Klicken Sie auf Ergebnisse analysieren und dann auf Volumen anzeigen mit: Slices. Überprüfen Sie, ob ein Volumen mit niedriger Auflösung, aber mit der Hauptform der Struktur erhalten wird (ergänzende Abbildung 13).
- Öffnen Sie nun pwem - join sets , um die beiden generierten Anfangsvolumina zu kombinieren, um die Eingabe für die Konsensmethode zu erstellen. Geben Sie einfach Volumes als Eingabetyp an und wählen Sie die beiden Anfangsvolumes im Eingabesatz aus. Führen Sie es aus. Die Ausgabe sollte ein Satz sein, der zwei Elemente mit beiden Volumes enthält.
- Das Konsensus-Tool ist das in xmipp3 enthaltene - schwarm consensus25. Öffnen Sie es. Verwenden Sie als Bilder in voller Größe die guten Partikel bei voller Auflösung (Ausgabe von 5,5), als Anfangsvolumen den Satz mit zwei im vorherigen Schritt generierten Elementen, und stellen Sie sicher, dass die Symmetriegruppe c1 ist. Klicken Sie auf Ausführen.
- Klicken Sie auf Ergebnisse analysieren. Überprüfen Sie, ob ein detaillierteres Ausgabevolumen ermittelt wird (Abbildung 6). Obwohl es mehr Lärm um die Struktur herum gibt, helfen mehr Details in der Strukturkarte den folgenden Verfeinerungsschritten, um lokale Minima zu vermeiden.
HINWEIS: Wenn UCSF Chimera26 verfügbar ist, verwenden Sie das letzte Symbol im oberen Teil des Fensters, um eine 3D-Visualisierung des erhaltenen Volumens zu erstellen. - Relion öffnen und ausführen - 3D auto-refine10, um eine erste 3D-Winkelzuordnung der Partikel vorzunehmen. Wählen Sie als Eingabepartikel die Ausgabe von 5,5 aus, und legen Sie den Partikelmaskendurchmesser auf 402 Å fest. Wählen Sie auf der Registerkarte Referenz-3D-Karte als Eingabevolumen das im vorherigen Schritt erhaltene Volume, Symmetrie als c1 und Anfänglicher Tiefpassfilter auf 30Å aus (Ergänzende Abbildung 14).
- Klicken Sie auf Ergebnisse analysieren. Wählen Sie im neuen Fenster final als Volume aus, um es zu visualisieren, und klicken Sie auf Display volume with: slices, um das erhaltene Volume anzuzeigen. Überprüfen Sie auch die Fourier-Schalenkorrelation (FSC), indem Sie im Ergebnisfenster auf Darstellungsauflösungsdiagramme und im Ergebnisfenster auf Winkelverteilung anzeigen: 2D-Diagramm (Abbildung 7) klicken. Das rekonstruierte Volumen enthält viel mehr Details (wahrscheinlich mit einigen verschwommenen Bereichen im äußeren Teil der Struktur), und der FSC überschreitet die Schwelle von 0,143 um 4,5Å. Die Winkelabdeckung deckt die gesamte 3D-Kugel ab.
7.3D Klassifikation: Exterieurzustände entdecken
- Mit einem Konsensansatz können unterschiedliche Konformationszustände in den Daten ermittelt werden. Open relion - 3D-Klassifikation10 (ergänzende Abbildung 15). Als Eingangspartikel verwenden Sie die gerade in 6.10 erhaltenen Partikelmasken und setzen Sie den Partikelmaskendurchmesser auf 402Å. Verwenden Sie auf der Registerkarte Referenz-3D-Karte als Eingabevolumen das nach Schritt 6.10 erhaltene Volumen, legen Sie Symmetrie auf c1 und anfänglichen Tiefpassfilter auf 15 Å fest. Legen Sie schließlich auf der Registerkarte Optimierung die Anzahl der Klassen auf 3 fest. Führen Sie es aus.
- Überprüfen Sie die Ergebnisse, indem Sie auf Ergebnisse analysieren klicken und Klassifizierung in Scipion anzeigen auswählen. Die drei generierten Klassen und einige interessante Messgrößen werden gezeigt. Die ersten beiden Klassen sollten eine ähnliche Anzahl von zugewiesenen Bildern (Größenspalte ) haben und sehr ähnlich aussehen, während die dritte Klasse weniger Bilder und ein verschwommeneres Erscheinungsbild aufweist. Auch die rlnAccuracyRotations und rlnAccuracyTranslations sollten für die ersten beiden Klassen deutlich besser sein. Wählen Sie die beiden besten Klassen aus und klicken Sie auf die Schaltfläche Klassen , um eine Teilmenge zu generieren, die sie enthält.
- Wiederholen Sie die Schritte 7.1 und 7.2, um eine zweite Gruppe guter Klassen zu generieren. Beides wird der Input des Konsens-Tools sein.
- Öffnen und führen Sie xmipp3 - Konsensusklassen 3D aus und wählen Sie als Eingabeklassen die beiden Teilmengen aus, die in den vorherigen Schritten generiert wurden.
- Klicken Sie auf Ergebnisse analysieren. Die Anzahl der koinzidierenden Teilchen zwischen den Klassen wird dargestellt: Der erste Wert ist die Anzahl der koinzidierenden Teilchen in der ersten Klasse der Teilmenge 1 und der ersten Klasse der Teilmenge 2, der zweite Wert ist die Anzahl der zufälligen Teilchen in der ersten Klasse der Teilmenge 1 und der zweiten Klasse der Teilmenge 2 usw. Überprüfen Sie, ob die Partikel zufällig den Klassen eins oder zwei zugeordnet sind, was bedeutet, dass die 3D-Klassifizierungsmethode nicht in der Lage ist, Konformationsänderungen zu finden. Angesichts dieses Ergebnisses wird der gesamte Satz von Partikeln verwendet, um die Verarbeitung fortzusetzen.
8.3D Verfeinerung: Verfeinerung von Winkelzuordnungen einer homogenen Grundgesamtheit
- Wenden Sie auch in diesem Schritt einen Konsensansatz an. Öffnen und führen Sie zunächst pwem - Subset mit Full set of items als Ausgabe von 6.9, Make random subset to Yes und Number of elements to 5000 aus. Damit wird eine Teilmenge von Bildern mit einer vorherigen Ausrichtung erstellt, um die im folgenden Schritt verwendete Methode zu trainieren.
- Öffnen Sie xmipp3 - deep align, Set Input images as the output of good particles received in 5.5, Volume as one received after 6.10, Input training set as the one created in the previous step, Target resolution to 10Å, and keep the remaining parameters with the default values (Supplemental Figure 16). Klicken Sie auf Ausführen.
- Klicken Sie auf Ergebnisse analysieren , um die erhaltene Winkelverteilung zu überprüfen, bei der keine Richtungen fehlen und sich die Winkelabdeckung im Vergleich zu 6,10 leicht verbessert (Abbildung 8).
- Öffnen und führen Sie xmipp3 aus - vergleichen Sie die Winkel und wählen Sie als Eingabeteilchen 1 den Ausgang von 6,9 und Eingangsteilchen 2 den Ausgang von 8,2 aus, stellen Sie sicher, dass die Symmetriegruppe c1 ist. Diese Methode berechnet die Übereinstimmung zwischen xmipp3 - deep align und relion - 3D auto refine.
- Klicken Sie auf Ergebnisse analysieren, die Liste der Partikel mit den erhaltenen Unterschieden in Verschiebungen und Winkeln wird angezeigt. Klicken Sie auf das Balkensymbol im oberen Teil des Fensters, ein weiteres Fenster wird geöffnet, in dem Sie Plots der berechneten Variablen erstellen können. Wählen Sie _xmipp_angleDiff und klicken Sie auf Plot , um eine Darstellung der Winkelunterschiede pro Partikel anzuzeigen. Machen Sie dasselbe mit _xmipp_shiftDiff. In diesen Zahlen stimmen etwa in der Hälfte der Teilchen beide Methoden überein (Abbildung 9). Wählen Sie die Partikel mit Winkeldifferenzen unter 10º aus und erstellen Sie eine neue Teilmenge.
- Öffnen Sie nun xmipp3 - highres27, um eine lokale Verfeinerung der zugewiesenen Winkel vorzunehmen. Wählen Sie zunächst die im vorherigen Schritt erhaltenen Bilder als Bilder in voller Größe aus, und als Anfangsvolumen die Ausgabe von 6,9, legen Sie den Partikelradius auf 150 Pixel und die Symmetriegruppe auf c1 fest. Legen Sie auf der Registerkarte Winkelzuweisung die Bildausrichtung auf Lokal, Anzahl der Iterationen auf 1 und Max fest. Zielauflösung als 5Å/px (ergänzende Abbildung 17). Führen Sie es aus.
- Überprüfen Sie auf der Registerkarte Zusammenfassung , ob das Ausgabevolumen kleiner als 300x300x300 Pixel und mit einer etwas höheren Pixelgröße ist.
- Klicken Sie auf Ergebnisse analysieren , um die erhaltenen Ergebnisse anzuzeigen. Klicken Sie auf Bildschirmauflösungsdiagramme , um den FSC anzuzeigen, und auf Anzeigevolumen: Rekonstruiert , um das erhaltene Volumen anzuzeigen (ergänzende Abbildung 18). Es wird ein gutes Auflösungsvolumen in der Nähe von 4-3,5 Å erreicht.
- Klicken Sie auf Ausgabepartikel anzeigen und klicken Sie im Fenster mit der Partikelliste auf das Balkensymbol. Wählen Sie im neuen Fenster Als Histogramm eingeben mit 100 Behältern aus, wählen Sie _xmipp_cost Beschriftung aus, und drücken Sie schließlich Plot (Ergänzende Abbildung 19). Auf diese Weise wird das Histogramm des Kostenlabels dargestellt, das die Korrelation des Partikels mit der dafür gewählten Projektionsrichtung enthält. In diesem Fall erhält man eine unimodale Dichtefunktion, die ein Zeichen dafür ist, dass es keine unterschiedlichen Populationen in der Menge der Teilchen gibt. So werden alle von ihnen verwendet, um die Verfeinerung fortzusetzen
HINWEIS: Wenn eine multimodale Dichtefunktion angezeigt wird, sollte der Satz von Partikeln, die zum höheren Maximum gehören, ausgewählt werden, um den Workflow nur mit ihnen fortzusetzen. - Öffnen und erneut ausführen xmipp3 - highres mit Weiter von einem vorherigen Lauf? Auf Ja, legen Sie die nach 8.5 erhaltenen Images als Full-Size-Images fest und wählen Sie vorherige Ausführung mit der vorherigen Ausführung von Xmipp Highres aus. Legen Sie auf der Registerkarte Winkelzuweisung die Bildausrichtung auf Lokal fest, mit 1 Iteration und 2,6 Å/px als Zielauflösung (volle Auflösung).
- Nun sollte die Ausgabe ein Volume bei voller Auflösung (Größe 300x300x300 Pixel) enthalten. Klicken Sie auf Ergebnisse analysieren , um das erhaltene Volumen und den FSC erneut zu überprüfen, der nun ein hochauflösendes Volumen bei etwa 3Å sein sollte (Abbildung 10).
9. Auswertung und Nachbearbeitung
- Öffnen Sie xmipp3 - lokales MonoRes28. Diese Methode berechnet die Auflösung lokal. Legen Sie als Eingangslautstärke das nach 8.10 erhaltene Volume fest, setzen Sie Möchten Sie halbe Volumes verwenden? auf Ja und Auflösungsbereich von 1 bis 10Å. Führen Sie es aus.
- Klicken Sie auf Ergebnisse analysieren , und wählen Sie Auflösungshistogramm anzeigen und Farbige Segmente anzeigen aus (Abbildung 11). Die Auflösung in den verschiedenen Teilen des Volumens wird angezeigt. Die meisten Voxelle des zentralen Teils der Struktur sollten Auflösungen um 3Å aufweisen, während die schlechtesten Auflösungen in den äußeren Teilen erreicht werden. Außerdem wird ein Histogramm der Auflösungen pro Voxel mit einem Peak um (sogar unter) 3Å angezeigt.
- Öffnen und führen Sie xmipp3 - localdeblur sharpening29 aus, um eine Schärfung anzuwenden. Wählen Sie als Input Map diejenige aus, die in 8.10 erhalten wurde, und als Resolution Map diejenige, die im vorherigen Schritt mit MonoRes erhalten wurde.
- Klicken Sie auf Ergebnisse analysieren , um die erhaltenen Volumina zu überprüfen. Öffnen Sie die letzte, die der letzten Iteration des Algorithmus entspricht. Es wird empfohlen, das Volume mit anderen Tools wie UCSF Chimera26 zu öffnen, um die Eigenschaften des Volumes in 3D besser zu sehen (Abbildung 12).
- Öffnen Sie abschließend das in xmipp3 enthaltene Validierungstool - validate overfitting30, das zeigt, wie sich die Auflösung mit der Anzahl der Partikel ändert. Öffnen Sie es und fügen Sie die in Schritt 8.5 erhaltenen Partikel als Eingabepartikel hinzu, setzen Sie Berechnung des für die Auflösung gebundenen Rauschens? auf Ja, mit anfänglichem 3D-Referenzvolumen als Ausgabe von 8,10. Legen Sie unter Erweiterte Optionen die Anzahl der Partikel auf "500 1000 1500 2000 3000 5000 10000 15000 20000" fest (Ergänzende Abbildung 20). Führen Sie es aus.
- Klicken Sie auf Ergebnisse analysieren. Es erscheinen zwei Diagramme (Abbildung 13) mit der Entwicklung der Auflösung in der grünen Linie, wenn die Anzahl der bei der Rekonstruktion verwendeten Partikel wächst. Die rote Linie stellt die Auflösung dar, die mit einer Rekonstruktion des ausgerichteten Gaußschen Rauschens erreicht wurde. Die Auflösung verbessert sich mit der Anzahl der Partikel und es wird ein großer Unterschied der Rekonstruktion von Partikeln im Vergleich zu der von Rauschen beobachtet, was ein Indikator für Partikel mit guten Strukturinformationen ist.
- Aus den bisherigen Ergebnissen konnte eine Anpassung eines Modells in das nachbearbeitete Volumen durchgeführt werden, was die Entdeckung der biologischen Strukturen des Makromoleküls ermöglichen würde.
Ergebnisse
Wir haben den Datensatz des Plasmodium falciparum 80S Ribosoms (EMPIAR-Eintrag: 10028, EMDB-Eintrag: 2660) verwendet, um den Test durchzuführen, und mit dem im vorherigen Abschnitt vorgestellten Scipion-Protokoll wurde ein hochauflösendes 3D-rekonstruiertes Volumen des Makromoleküls in diesem speziellen Beispiel erreicht, beginnend mit den vom Mikroskop gesammelten Informationen, die aus sehr verrauschten Bildern bestehen, die 2D-Projektionen in jeder Ausrichtung der Probe enthalten.
Die wichtigsten Ergebnisse, die nach dem Ausführen des gesamten Protokolls erzielt wurden, sind in Abbildung 10, Abbildung 11 und Abbildung 12 dargestellt. Abbildung 10 stellt das erhaltene 3D-Volumen vor der Nachbearbeitung dar. In Abbildung 10a ist ein FSC von 3 Å zu sehen, dass er sehr nahe an der Nyquist-Grenze liegt (bei Daten mit einer Pixelgröße von 1,34 Å beträgt die Nyquist-Grenze 2,6 Å). Abbildung 10b zeigt einige Scheiben des rekonstruierten 3D-Volumens mit hohem Detaillierungsgrad und klar definierten Strukturen. In Abbildung 11 sind die Ergebnisse nach lokaler Analyse der Auflösung des erhaltenen 3D-Volumens dargestellt. Es ist zu sehen, dass die meisten Voxel in der Struktur eine Auflösung unter 3 Å erreichen, hauptsächlich diejenigen, die sich im zentralen Teil der Struktur befinden. Der äußere Teil zeigt jedoch schlechtere Auflösungen, was mit der Unschärfe übereinstimmt, die in diesen Bereichen in den Scheiben von Abbildung 10b auftritt. Abbildung 12 zeigt nach der Nachbearbeitung die gleiche 3D-Karte, die in der Lage ist, die höheren Frequenzen des Volumens hervorzuheben, mehr Details zu enthüllen und die Darstellung zu verbessern, was insbesondere in der 3D-Präsentation in Abbildung 12c zu sehen ist.
In Abbildung 14 wurde Chimera26 verwendet, um eine 3D-Darstellung des erhaltenen Volumens (Abbildung 14a), der nachbearbeiteten (Abbildung 14b) und der Auflösungskarte (Abbildung 14c) zu sehen, die mit dem Farbcode der lokalen Auflösungen gefärbt sind. Dies kann noch mehr Informationen über die erhaltene Struktur geben. Dieses Tool ist sehr nützlich, um einen Einblick in die Qualität des erhaltenen Volumens zu erhalten, da sehr kleine Details im gesamten 3D-Kontext der Struktur zu sehen sind. Wenn die erreichte Auflösung ausreicht, können sogar einige biochemische Teile der Struktur gefunden werden (z. B. Alpha-Helices in Abbildung 14d. In dieser Abbildung muss die hohe Auflösung hervorgehoben werden, die in allen zentralen Teilen der 3D-Struktur erreicht wurde, die als dunkelblaue Bereiche in Abbildung 14c zu sehen sind.
Alle bisherigen Ergebnisse wurden dank einer guten Leistung des gesamten Protokolls erzielt, aber dies ist möglicherweise nicht der Fall. Es gibt mehrere Möglichkeiten, ein schlechtes Verhalten zu identifizieren. Im allgemeinsten Fall geschieht dies, wenn die erhaltene Struktur eine niedrige Auflösung hat und sich nicht zu einer besseren entwickeln kann. Ein Beispiel dafür ist in Abbildung 15 dargestellt. Ein unscharfes Volumen (Abbildung 15c) führt zu einem niedrigen FSC, was in der FSC-Kurve (Abbildung 15a) und dem Histogramm der lokalen Schätzung (Abbildung 15b) zu sehen ist. Dieses Beispiel wurde mit einer 3D-Verfeinerungsmethode mit falschen Eingabedaten generiert, da einige spezifische Eigenschaften im Eingabesatz von Partikeln erwartet wurden, die sie nicht erfüllen. Wie man sieht, ist es immer sehr wichtig zu wissen, wie die verschiedenen Methoden die Daten erhalten und richtig aufbereiten. Wenn eine Ausgabe wie die in Abbildung 15 abgerufen wird, liegt im Allgemeinen ein Problem im Verarbeitungsworkflow oder den zugrunde liegenden Daten vor.
Es gibt mehrere Prüfpunkte entlang des Workflows, die analysiert werden können, um zu wissen, ob sich das Protokoll ordnungsgemäß entwickelt oder nicht. Zum Beispiel können einige der zuvor besprochenen Methoden direkt nach der Auswahl die Partikel einstufen und für jeden von ihnen eine Punktzahl geben. Im Falle von schlechten Partikeln ermöglichen diese Methoden, sie zu identifizieren und zu entfernen. Außerdem kann die 2D-Klassifizierung ein guter Indikator für einen schlechten Satz von Partikeln sein. Abbildung 16 zeigt ein Beispiel für eine solche schlechte Menge. In Abbildung 16a sind gute Klassen mit einigen Details der Struktur dargestellt, während Abbildung 16b schlechte Klassen zeigt, die verrauscht oder unzentriert sind, in diesem letzten Fall kann man sehen, dass die Auswahl falsch war und zwei Partikel zusammen zu erscheinen scheinen. Ein weiterer Prüfpunkt ist die anfängliche Volumenschätzung, Abbildung 17 zeigt ein Beispiel für gute (Abbildung 17a) und schlechte (Abbildung 17b) Anfangsschätzungen. Die fehlerhafte Schätzung wurde mit einem falschen Setup für die Methode erstellt. Es muss berücksichtigt werden, dass alle Setups sorgfältig durchgeführt werden sollten, wobei jeder Parameter entsprechend den analysierten Daten entsprechend ausgewählt werden sollte. Wenn Sie keine Karte mit minimalen strukturellen Informationen haben, kann die folgende Verfeinerung keine gute Rekonstruktion erhalten.
Wenn das Problem eine schlechte Akquisition ist, bei der die Filme keine strukturellen Informationen bewahren, wird es unmöglich sein, gute Partikel aus ihnen zu extrahieren und eine erfolgreiche Verarbeitung zu erhalten. In diesem Fall sollten mehr Filme gesammelt werden, um eine hochauflösende 3D-Rekonstruktion zu erhalten. Wenn dies jedoch nicht der Fall ist, gibt es mehrere Möglichkeiten, Probleme entlang des Verarbeitungsworkflows zu verwalten. Wenn die Kommissionierung nicht gut genug ist, gibt es mehrere Möglichkeiten, um zu versuchen, sie zu beheben, z. B. die Kommissionierung zu wiederholen, verschiedene Methoden zu verwenden oder zu versuchen, mehr Partikel manuell auszuwählen, um den Methoden zu helfen, daraus zu lernen. Wenn während der 2D-Klassifizierung nur wenige Klassen gut sind, sollten Sie auch den Kommissioniervorgang wiederholen. Versuchen Sie bei der anfänglichen Volumenschätzung, mehrere Methoden zu verwenden, wenn einige von ihnen ungenaue Ergebnisse lieferten. Gleiches gilt für die 3D-Veredelung. Nach dieser Argumentation wurden in diesem Manuskript mehrere Konsenswerkzeuge vorgestellt, die sehr nützlich sein könnten, um Probleme zu vermeiden und die Verarbeitung mit genauen Daten fortzusetzen. Dank der Verwendung eines Konsenses zwischen mehreren Methoden können wir Daten verwerfen, die schwer auszuwählen, zu klassifizieren, auszurichten usw. sind, was wahrscheinlich ein Indikator für schlechte Daten ist. Wenn sich jedoch mehrere Methoden in der generierten Ausgabe einigen können, enthalten diese Daten wahrscheinlich wertvolle Informationen, mit denen die Verarbeitung fortgesetzt werden kann.
Wir empfehlen dem Leser, mehr Datensätze herunterzuladen und zu versuchen, sie gemäß den empfehlungen in diesem Manuskript zu verarbeiten und einen ähnlichen Workflow zu erstellen, der Verarbeitungspakete mit Scipion kombiniert. Der Versuch, einen Datensatz zu verarbeiten, ist der beste Weg, um die Leistungsfähigkeit der im State-of-the-Art in Cryo-EM verfügbaren Verarbeitungswerkzeuge zu erlernen, die besten Regeln zu kennen, um die möglichen Nachteile während der Verarbeitung zu überwinden und die Leistung der verfügbaren Methoden in jedem spezifischen Testfall zu steigern.
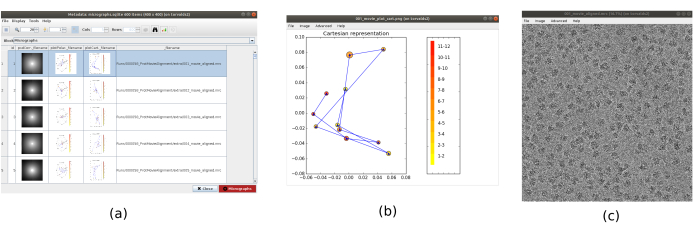
Abbildung 1. Ergebnis der Filmausrichtung. (a) Das Hauptfenster der Ergebnisse mit einer Liste aller erzeugten Mikroaufnahmen und zusätzlichen Informationen: die spektrale Leistungsdichte, die Flugbahn der geschätzten Ausrichtung in Polarkoordinaten, die gleiche in kartesischen Koordinaten, den Dateinamen der erzeugten Mikroaufnahme. (b) Die Ausrichtungskurve, dargestellt in kartesischen Koordinaten. (c) Die erzeugte Mikroaufnahme. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
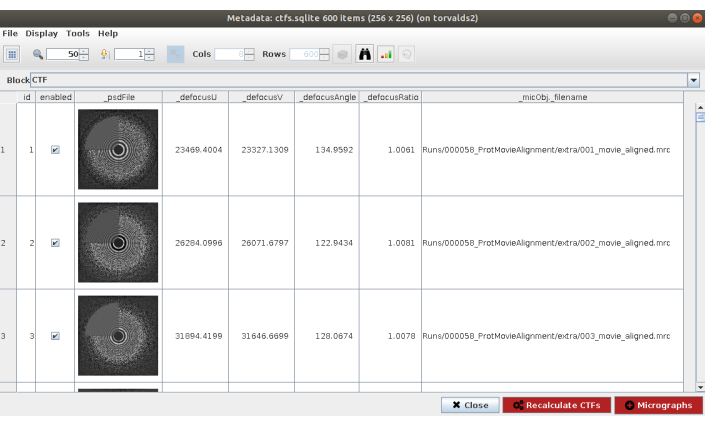
Abbildung 2. CTF-Schätzung mit Ctffind-Ergebnis. Das Hauptfenster mit den Ergebnissen enthält eine Zahl mit der geschätzten PSD (in einer Ecke) zusammen mit der PSD, die aus den Daten stammt, und mehreren Defokussierungsparametern. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
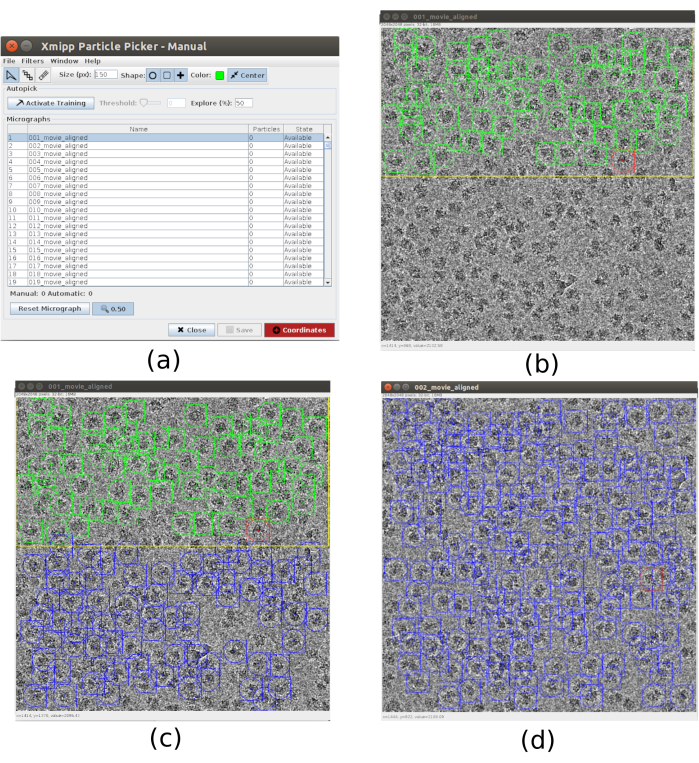
Abbildung 3. Manuelle Kommissionierfenster mit Xmipp. (a) Das Hauptfenster mit der Liste der zu verarbeitenden Mikroaufnahmen und einigen anderen Parametern. (b) Manuelles Pflücken von Partikeln in einem Bereich einer Mikroaufnahme. (c) und (d) Automatisch zu überwachende Partikel, um eine Reihe von Trainingspartikeln für die automatische Xmipp-Kommissioniermethode zu erstellen. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
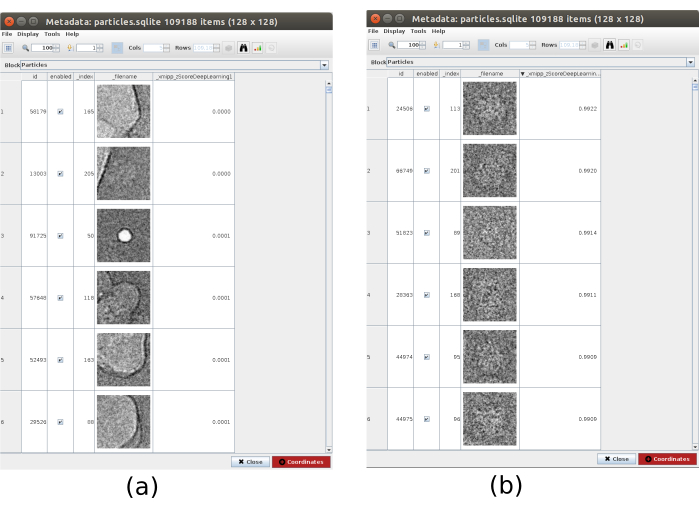
Abbildung 4. Deep Consensus Picking mit Xmipp-Ergebnis. Der Parameter zScoreDeepLearning gibt der Güte eines Teilchens Gewicht und ist der Schlüssel zur Entdeckung schlechter Teilchen. (a) Die niedrigsten zScores-Werte sind Artefakten zugeordnet. (b) Die höchsten zScores sind mit Partikeln assoziiert, die das Makromolekül enthalten. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
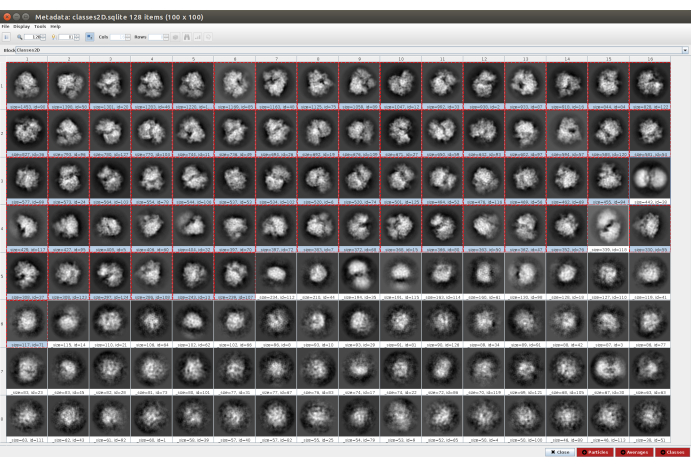
Abbildung 5. 2D-Klassifizierung mit Cryosparc-Ergebnis. Die erzeugten Klassen (Mittelwerte von Teilmengen von Teilchen, die aus der gleichen Ausrichtung kommen) werden angezeigt. Mehrere gute Klassen, die rot ausgewählt wurden (mit einem gewissen Detaillierungsgrad) und einige schlechte Klassen nicht ausgewählt (verrauschte und unzentrierte Klassen). Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
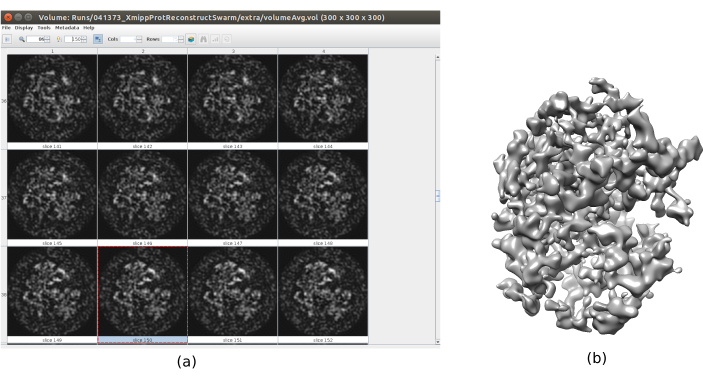
Abbildung 6. 3D-Anfangsvolumen mit Schwarmkonsensergebnis. Eine Ansicht des 3D-Anfangsvolumens, die nach dem Ausführen des Konsensus-Tools xmipp3 - Schwarmkonsens unter Verwendung der vorherigen 3D-Anfangsvolumenschätzungen von Xmipp und Relion erhalten wurde. (a) Das Volumen wird durch Scheiben dargestellt. (b) 3D-Visualisierung des Volumens. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
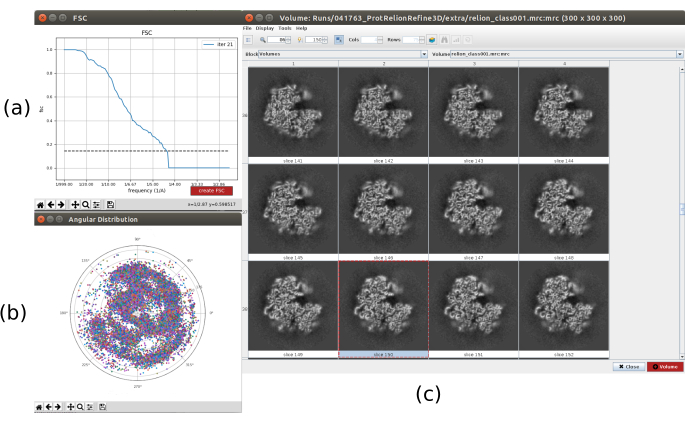
Abbildung 7. Verfeinerung eines 3D-Ausgangsvolumens mit Relion-Ergebnis. (a) FSC-Kurve erhalten, die den Schwellenwert bei etwa 4,5 Å überschreitet. (b) Winkelabdeckung, dargestellt als obere Ansicht der 3D-Kugel. In diesem Fall, da es keine Symmetrie gibt, sollten die zugewiesenen Partikel die gesamte Kugel abdecken. c) Raffiniertes Volumen, dargestellt durch Scheiben. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
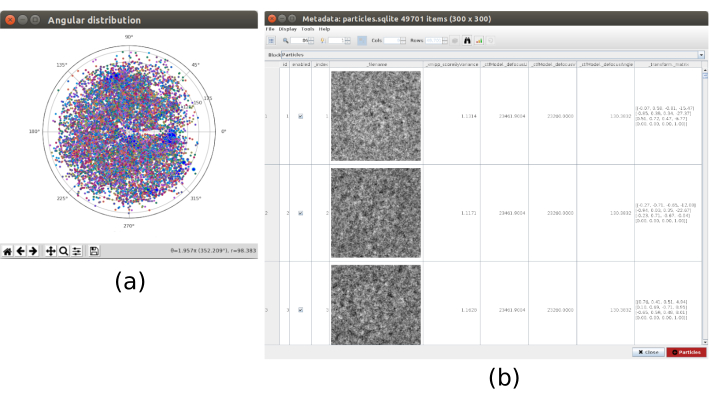
Abbildung 8. 3D-Ausrichtung basierend auf Deep Learning mit Xmipp-Ergebnis. Die ergebnisse generiert von xmipp3 - deep align methode for 3D alignment. (a) Die Winkelbelegung für jedes Teilchen in Form einer Transformationsmatrix. b) Die Winkelabdeckung. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
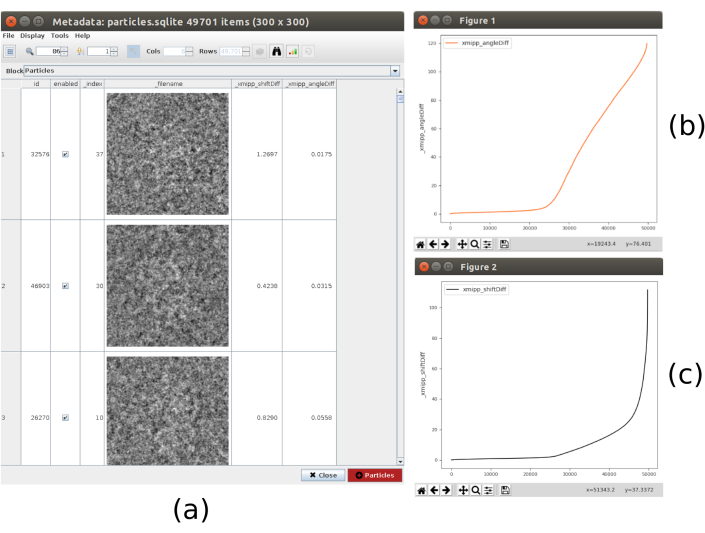
Abbildung 9. 3D-Alignment-Konsensergebnis. (a) Liste der Partikel mit den erhaltenen Unterschieden in den Verschiebungs- und Winkelparametern. (b) Darstellung der Winkeldifferenzen je Teilchen. (c) Darstellung der Verschiebungsdifferenz pro Partikel. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
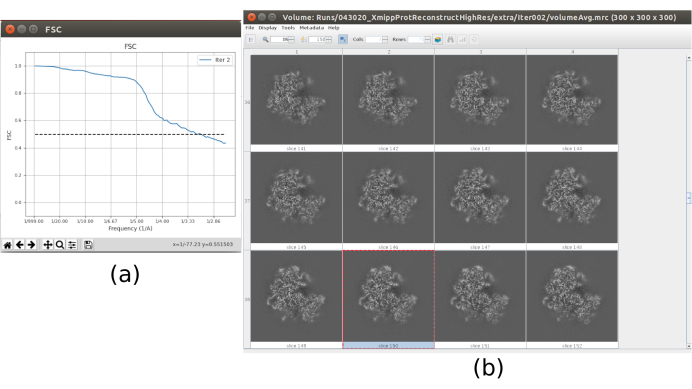
Abbildung 10. Abschließende Iteration des 3D-Verfeinerungsergebnisses. a) FSC-Kurve. (b) Erhaltenes Volumen bei voller Auflösung durch Scheiben. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
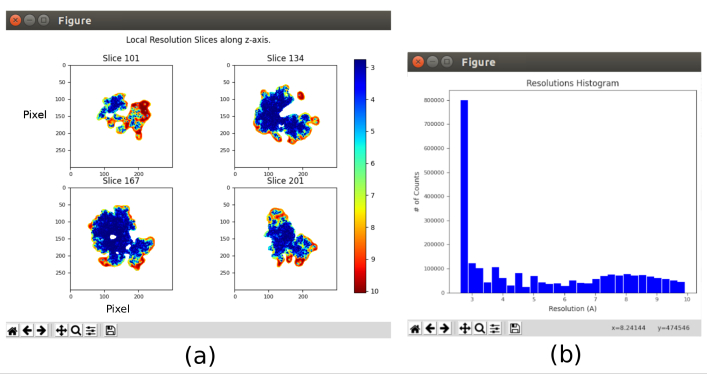
Abbildung 11. Lokale Auflösungsanalyse mit Xmipp-Ergebnis. Ergebnisse der Methode xmipp3 - lokale MonoRes. (a) Einige repräsentative Scheiben, die mit dem Auflösungswert pro Voxel gefärbt sind, wie im Farbcode angegeben. b) Histogramm der lokalen Auflösung. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
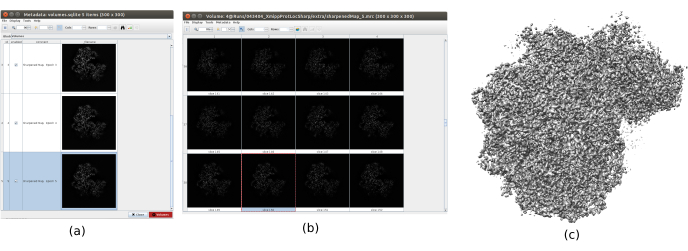
Abbildung 12. Schärfen mit Xmipp-Ergebnis. Ergebnisse von xmipp3 - localdeblur Schärfmethode . (a) Liste der erhaltenen Volumes pro Iteration. (b) 3D-Volumen, das nach der letzten Iteration erhalten wurde, dargestellt durch Slices. (c) Eine 3D-Darstellung des endgültigen Bandes. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
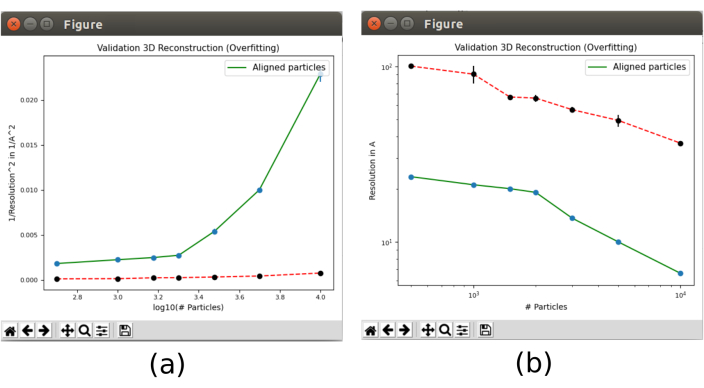
Abbildung 13. Überanpassungswerkzeug im Xmipp-Ergebnis validieren. Ergebnisse von xmipp3 - Validierungsüberanpassung. Die grüne Linie entspricht der Rekonstruktion aus Daten, die rote Linie aus Rauschen. (a) Umkehrung der quadrierten Auflösung mit dem Logarithmus der Anzahl der Teilchen. b) Auflösung mit der Anzahl der Partikel. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
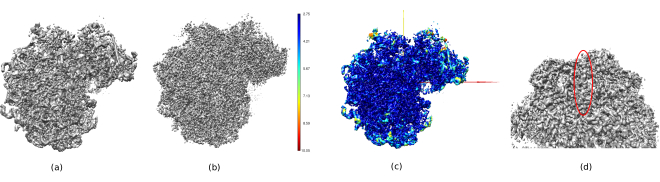
Abbildung 14. Mehrere 3D-Darstellungen des erhaltenen Volumens. (a) Vorverarbeitetes Volumen. b) Nachbearbeitetes Volumen. (c) Lokale Auflösung, dunkelblaue Voxel sind solche mit höherer Auflösung (2,75Å) und dunkelrote Voxel sind solche mit niedrigerer Auflösung (10,05Å). (d) Zoomen Sie in das nachbearbeitete Volumen, in dem eine Alpha-Helix (rotes Oval) zu sehen ist. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
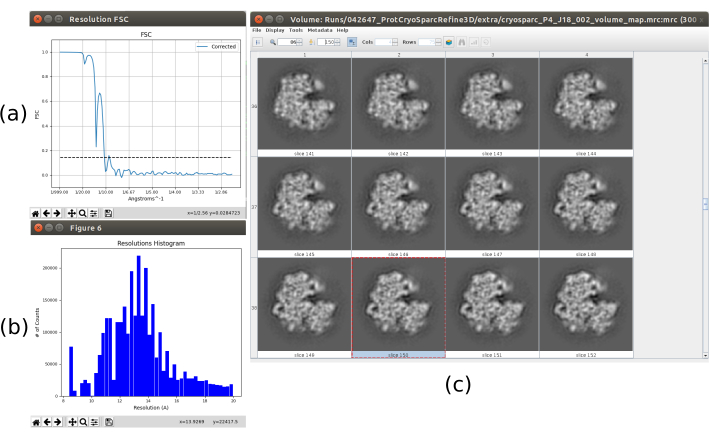
Abbildung 15. Beispiel für eine schlechte 3D-Rekonstruktion. (a) FSC-Kurve mit einem starken Abfall und Überschreiten der Schwelle bei niedriger Auflösung. b) Histogramm der lokalen Auflösung. (c) 3D-Volumen in Scheiben. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
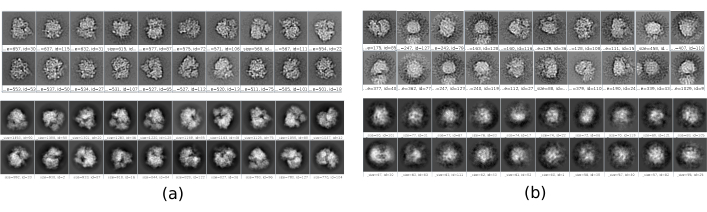
Abbildung 16. Beispiel für 2D-Klassen. (a) Gute Klassen mit einem gewissen Detaillierungsgrad. (b) Schlechte Klassen, die Rauschen und Artefakte enthalten (oberer Teil mit Xmipp, unterer teil mit CryoSparc). Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
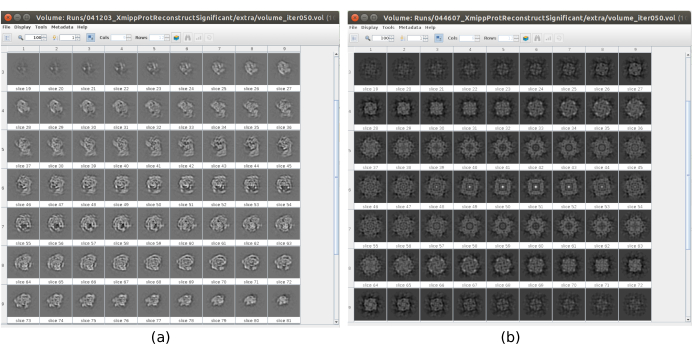
Abbildung 17. Beispiel für ein 3D-Anfangsvolumen mit unterschiedlichen Qualitäten. (a) Gutes Anfangsvolumen, bei dem die Form des Makromoleküls beobachtet werden kann. (b) Schlechtes Anfangsvolumen, wenn die erhaltene Form völlig anders ist als die erwartete. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Ergänzende Abbildung 1. Erstellen eines Scipion-Projekts. Von Scipion angezeigtes Fenster, in dem ein altes Projekt ausgewählt oder ein neues erstellt werden kann, das einen Namen und einen Speicherort für dieses Projekt enthält. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Ergänzende Abbildung 2. Methode zum Importieren von Filmen. Fenster, das von Scipion angezeigt wird, wenn pwem - Filme importieren geöffnet ist. Hier müssen die wichtigsten Erfassungsparameter enthalten sein, damit die Filme in Scipion verarbeitet werden können. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Ergänzende Abbildung 3. Filmausrichtungsmethode. Fenster, das von Scipion angezeigt wird, wenn xmipp3 - optische Ausrichtung verwendet wird. Die Eingabefilme, der Bereich der für die Ausrichtung berücksichtigten Frames und einige andere Parameter zum Verarbeiten der Filme sollten gefüllt werden. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Ergänzende Abbildung 4. CTF-Schätzmethode mit Ctffind. Das Formular in Scipion mit allen notwendigen Feldern, um das Programm Ctffind auszuführen. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Ergänzende Abbildung 5. Wizard in Scipion. Ein Assistent, der dem Benutzer beim Ausfüllen einiger Parameter im Formular hilft. In diesem Fall muss der Assistent das Auflösungsfeld in der Methode grigoriefflab - ctffind vervollständigen. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Ergänzende Abbildung 6. CTF-Verfeinerungsmethode mit Xmipp. Die Form der xmipp3 - ctf Schätzung mit allen Parametern, um eine Verfeinerung einer zuvor geschätzten CTF vorzunehmen.
Ergänzende Abbildung 7. Preprocess Micrographs Methode. Die Form von xmipp3 - Vorverarbeitung von Mikroaufnahmen , die es ermöglicht, einige Operationen über sie durchzuführen. In diesem Beispiel ist das Entfernen fehlerhafter Pixel und das Downsampeln von Mikroaufnahmen das nützliche Beispiel. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Ergänzende Abbildung 8. Kommissioniermethode mit Cryolo. Das Formular zum Ausführen der Cryolo-Kommissioniermethode mithilfe eines vortrainierten Netzwerks. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Ergänzende Abbildung 9. Konsensfindungsmethode mit Xmipp. Die Form von xmipp3 - Deep Consensus Picking basierend auf Deep Learning, um einen Konsens von Koordinaten zu berechnen, unter Verwendung eines vortrainierten Netzwerks über mehrere Koordinatensätze, die mit verschiedenen Picking-Methoden erhalten wurden. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Ergänzende Abbildung 10. Extraktionspartikel-Methode. Eingabe- und Vorverarbeitungsregister von xmipp3 - Partikel extrahieren. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Ergänzende Abbildung 11.3D Anfängliche Volumenmethode mit Xmipp. Die Form der Methode xmipp3 - rekonstruieren Sie signifikant , um eine erste 3D-Karte zu erhalten. Die Registerkarten Eingabe und Kriterien werden angezeigt. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Ergänzende Abbildung 12. Volume-Methode zum Ändern der Größe. Das Formular zum Zuschneiden oder Ändern der Größe eines Volumes. In diesem Beispiel wird diese Methode verwendet, um ein Volume in voller Größe nach xmipp3 zu generieren - rekonstruieren Sie signifikant. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Ergänzende Abbildung 13.3D Anfangsvolumen mit Relion-Ergebnis. Eine Ansicht des erhaltenen 3D-Anfangsvolumens mit Relion - 3D-Anfangsmodellmethode durch Slices. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Ergänzende Abbildung 14. Verfeinerung des Ausgangsvolumens mit Relion. Die Form der Methode relion - 3D auto-refine. In diesem Beispiel wurde es verwendet, um ein anfängliches Volumen zu verfeinern, das nach Konsens geschätzt wurde. Die Registerkarten Eingabe und Referenz der 3D-Karte werden angezeigt. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Ergänzende Abbildung 15.3D Klassifizierungsmethode. Form der Relion - 3D-Klassifizierung. Die Registerkarten Eingabe, Referenz-3D-Karte und Optimierung werden angezeigt. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Ergänzende Abbildung 16.3D Ausrichtung basierend auf einer Deep-Learning-Methode. Das Formular wurde für die Methode xmipp3 - deep align geöffnet. Hier ist es notwendig, ein Netzwerk mit einem Trainingssatz zu trainieren, dann wird dieses Netzwerk die Winkelbelegung pro Teilchen vorhersagen. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Ergänzende Abbildung 17.3D Verfeinerungsmethode. Form der xmipp3 - highres Methode. Die Registerkarten Eingabe und Winkelzuweisung werden angezeigt. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Ergänzende Abbildung 18. Erste Iteration des 3D-Verfeinerungsergebnisses. a) FSC-Kurve. (b) Erhaltenes Volumen (kleiner als die volle Auflösung), dargestellt als Scheiben. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Ergänzende Abbildung 19. Erste Iteration der 3D-Verfeinerungskorrelationsanalyse. Ein neues Fenster erscheint, indem Sie auf das Balkensymbol im oberen Teil des Fensters mit der Liste der Partikel klicken. Im Fenster Spalten plotten kann ein Histogramm des gewünschten geschätzten Parameters erstellt werden. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Ergänzende Abbildung 20. Validierungs-Overfitting-Tool. Form von xmipp3 - Validierung der Überanpassungsmethode . Bitte klicken Sie hier, um diese Datei herunterzuladen.
Diskussion
Derzeit ist Kryo-EM ein Schlüsselwerkzeug, um die 3D-Struktur biologischer Proben aufzudecken. Wenn gute Daten mit dem Mikroskop gesammelt werden, können wir mit den verfügbaren Verarbeitungswerkzeugen eine 3D-Rekonstruktion des untersuchten Makromoleküls erhalten. Die Kryo-EM-Datenverarbeitung ist in der Lage, eine nahezu atomare Auflösung zu erreichen, die für das Verständnis des funktionellen Verhaltens eines Makromoleküls von entscheidender Bedeutung ist und auch für die Wirkstoffforschung von entscheidender Bedeutung ist.
Scipion ist eine Software, die es ermöglicht, den gesamten Workflow zu erstellen und die relevantesten Bildverarbeitungspakete auf integrative Weise zu kombinieren, was die Rückverfolgbarkeit und Reproduzierbarkeit des gesamten Bildverarbeitungsworkflows unterstützt. Scipion bietet einen sehr vollständigen Satz von Werkzeugen, um die Verarbeitung durchzuführen; Die Erzielung hochauflösender Rekonstruktionen hängt jedoch vollständig von der Qualität der erfassten Daten und der Art und Weise ab, wie diese Daten verarbeitet werden.
Um eine hochauflösende 3D-Rekonstruktion zu erhalten, ist die erste Voraussetzung, gute Filme aus dem Mikroskop zu erhalten, die strukturelle Informationen auf hoher Auflösung erhalten. Ist dies nicht der Fall, kann der Workflow keine hochauflösenden Informationen aus den Daten extrahieren. Dann sollte ein erfolgreicher Verarbeitungsworkflow in der Lage sein, Partikel zu extrahieren, die wirklich der Struktur entsprechen, und die Orientierungen dieser Partikel im 3D-Raum zu finden. Wenn einer der Schritte im Workflow fehlschlägt, verschlechtert sich die Qualität des rekonstruierten Volumes. Scipion ermöglicht die Verwendung verschiedener Pakete in jedem der Verarbeitungsschritte, was dazu beiträgt, den am besten geeigneten Ansatz zur Verarbeitung der Daten zu finden. Darüber hinaus können dank der Verfügbarkeit vieler Pakete Konsenswerkzeuge verwendet werden, die die Genauigkeit erhöhen, indem sie eine Übereinstimmung in den geschätzten Ergebnissen verschiedener Methoden finden. Außerdem wurden im Abschnitt Repräsentative Ergebnisse mehrere Validierungstools ausführlich diskutiert und wie man genaue und ungenaue Ergebnisse in jedem Schritt des Workflows identifiziert, potenzielle Probleme erkennt und versucht, sie zu lösen. Es gibt mehrere Prüfpunkte entlang des Protokolls, die helfen können zu erkennen, ob das Protokoll ordnungsgemäß ausgeführt wird oder nicht. Einige der relevantesten sind: Kommissionierung, 2D-Klassifizierung, anfängliche Volumenschätzung und 3D-Ausrichtung. Das Überprüfen der Eingaben, das Wiederholen des Schritts mit einer anderen Methode oder das Verwenden eines Konsenses sind Optionen, die in Scipion verfügbar sind, mit denen der Benutzer Lösungen finden kann, wenn Probleme auftreten.
In Bezug auf die bisherigen Ansätze zur Paketintegration im Cryo-EM-Bereich ist Appion31 der einzige, der eine echte Integration verschiedener Softwarepakete ermöglicht. Appion ist jedoch eng mit Leginon32 verbunden, einem System zur automatisierten Erfassung von Elektronenmikroskopen. Der Hauptunterschied zu Scipion besteht darin, dass Datenmodell und Speicher weniger gekoppelt sind. Um ein neues Protokoll in Scipion zu erstellen, muss nur ein Python-Skript entwickelt werden. In Appion muss der Entwickler jedoch das Skript schreiben und die zugrunde liegende Datenbank ändern. Zusammenfassend lässt sich sagen, dass Scipion entwickelt wurde, um die Wartung und Erweiterbarkeit zu vereinfachen.
Wir haben in diesem Manuskript einen vollständigen Workflow für die Kryo-EM-Verarbeitung vorgestellt, der den realen Falldatensatz des Plasmodium falciparum 80S Ribosoms (EMPIAR-Eintrag: 10028, EMDB-Eintrag: 2660) verwendet. Die hier behandelten und diskutierten Schritte können als Filmausrichtung, CTF-Schätzung, Partikelauswahl, 2D-Klassifizierung, anfängliche Kartenschätzung, 3D-Klassifizierung, 3D-Verfeinerung, Bewertung und Nachbearbeitung zusammengefasst werden. Verschiedene Pakete wurden verwendet und Konsensus-Tools wurden in mehreren dieser Schritte angewendet. Das endgültige rekonstruierte 3D-Volumen erreichte eine Auflösung von 3 Å und im nachbearbeiteten Volumen können einige Sekundärstrukturen unterschieden werden, wie Alpha-Helices, was hilft zu beschreiben, wie Atome im Raum angeordnet sind.
Der in diesem Manuskript vorgestellte Workflow zeigt, wie Scipion verwendet werden kann, um verschiedene Cryo-EM-Pakete auf einfache und integrative Weise zu kombinieren, um die Verarbeitung zu vereinfachen und gleichzeitig ein zuverlässigeres Ergebnis zu erzielen.
In Zukunft wird die Entwicklung neuer Methoden und Pakete weiter wachsen und Software wie Scipion, um sie alle einfach zu integrieren, wird für die Forscher noch wichtiger sein. Konsensansätze werden auch dann relevanter sein, wenn viele Methoden mit unterschiedlicher Basis zur Verfügung stehen werden, die dazu beitragen, genauere Schätzungen aller Parameter zu erhalten, die am Rekonstruktionsprozess in Cryo-EM beteiligt sind. Tracking und Reproduzierbarkeit sind der Schlüssel im Forschungsprozess und dank eines gemeinsamen Frameworks für die Ausführung kompletter Workflows mit Scipion einfacher zu erreichen.
Offenlegungen
Die Autoren haben nichts preiszugeben.
Danksagungen
Die Autoren danken der wirtschaftlichen Unterstützung von: Dem spanischen Ministerium für Wissenschaft und Innovation durch Zuschüsse: PID2019-104757RB-I00/AEI/10.13039/501100011033, der "Comunidad Autónoma de Madrid" durch Zuschuss: S2017/BMD-3817, Instituto de Salud Carlos III, PT17/0009/0010 (ISCIII-SGEFI/EFRE), Europäische Union (EU) und Horizon 2020 durch Zuschuss: INSTRUCT - ULTRA (INFRADEV-03-2016-2017, Vorschlag: 731005), EOSC Life (INFRAEOSC-04-2018, Vorschlag: 824087), iNEXT - Discovery (Vorschlag: 871037) und HighResCells (ERC - 2018 - SyG, Vorschlag: 810057). Das Projekt, das zu diesen Ergebnissen führte, wurde von der Stiftung "la Caixa" (ID 100010434) mit einem Stipendium unterstützt. Der Stipendiencode lautet LCF/BQ/DI18/11660021. Dieses Projekt wurde aus dem Forschungs- und Innovationsprogramm Horizon 2020 der Europäischen Union im Rahmen der Marie-Skłodowska-Curie-Finanzhilfevereinbarung Nr. 713673 gefördert. Die Autoren würdigen die Unterstützung und den Einsatz von Ressourcen von Instruct, einem Landmark ESFRI-Projekt.
Materialien
| Name | Company | Catalog Number | Comments |
| no material is used in this article | - | - | - |
Referenzen
- Nogales, E. The development of cryo-EM into a mainstream structural biology technique. Nature Methods. 13 (1), 24-27 (2016).
- Kühlbrandt, W. The Resolution Revolution. Science. 343 (6178), 1443-1444 (2014).
- . 1.15 A structure of human apoferritin obtained from Titan Mono- BCOR microscope Available from: https://www.rcsb.org/structure/7A6A (2021)
- Arnold, S. A., et al. Miniaturizing EM Sample Preparation: Opportunities, Challenges, and "Visual Proteomics". PROTEOMICS. 18 (5-6), 1700176 (2018).
- Faruqi, A. R., McMullan, G. Direct imaging detectors for electron microscopy. Nuclear Instruments and Methods in Physics Research Section A: Accelerators, Spectrometers, Detectors and Associated Equipment. 878, 180-190 (2018).
- Vilas, J. L., et al. Advances in image processing for single-particle analysis by electron cryomicroscopy and challenges ahead. Current Opinion in Structural Biology. 52, 127-145 (2018).
- Martinez, M., et al. Integration of Cryo-EM Model Building Software in Scipion. Journal of Chemical Information and Modeling. 60, 2533-2540 (2020).
- de la Rosa-Trevín, J. M., et al. Scipion: A software framework toward integration, reproducibility and validation in 3D electron microscopy. Journal of Structural Biology. 195, 93-99 (2016).
- de la Rosa-Trevín, J. M., et al. Xmipp 3.0: an improved software suite for image processing in electron microscopy. Journal of Structural Biology. 184, 321-328 (2013).
- Scheres, S. H. W. . Methods in Enzymology. The Resolution Revolution: Recent Advances In cryoEM. , 125-157 (2016).
- Punjani, A., Rubinstein, J. L., Fleet, D. J., Brubaker, M. A. cryoSPARC: algorithms for rapid unsupervised cryo-EM structure determination. Nature Methods. 14, 290-296 (2017).
- Ludtke, S. J. 3-D structures of macromolecules using single-particle analysis in EMAN. Methods in Molecular Biology. 673, 157-173 (2010).
- Shaikh, T. R., et al. SPIDER image processing for single-particle reconstruction of biological macromolecules from electron micrographs. Nature Protocols. 3, 1941-1974 (2008).
- Wagner, T., et al. SPHIRE-crYOLO is a fast and accurate fully automated particle picker for cryo-EM. Communications Biology. 2, (2019).
- Mindell, J. A., Grigorieff, N. Accurate determination of local defocus and specimen tilt in electron microscopy. Journal of Structural Biology. 142, 334-347 (2003).
- Winn, M. D., et al. Overview of the CCP4 suite and current developments. Acta crystallographica. Section D, Biological crystallography. 67, 235-242 (2011).
- Liebschner, D., et al. Macromolecular structure determination using X-rays, neutrons and electrons: recent developments in Phenix. Acta Crystallographica Section D. 75, 861-887 (2019).
- Wong, W., et al. Cryo-EM structure of the Plasmodium falciparum 80S ribosome bound to the anti-protozoan drug emetine. eLife. 3, 03080 (2014).
- Abrishami, V., et al. Alignment of direct detection device micrographs using a robust Optical Flow approach. Journal of Structural Biology. 189, 163-176 (2015).
- Sorzano, C. O. S., Jonic, S., Nunez Ramirez, R., Boisset, N., Carazo, J. M. Fast, robust and accurate determination of transmission electron microscopy contrast transfer function. Journal of Structural Biology. 160, 249-262 (2007).
- Abrishami, V., et al. A pattern matching approach to the automatic selection of particles from low-contrast electron micrographs. Bioinformatics. 29, 2460-2468 (2013).
- Sanchez-Garcia, R., Segura, J., Maluenda, D., Carazo, J. M., Sorzano, C. O. S. Deep Consensus, a deep learning-based approach for particle pruning in cryo-electron microscopy. IUCrJ. 5, 854-865 (2018).
- Sorzano, C. O. S., et al. A clustering approach to multireference alignment of single-particle projections in electron microscopy. Journal of Structural Biology. 171, 197-206 (2010).
- Sorzano, C. O. S., et al. A statistical approach to the initial volume problem in Single Particle Analysis by Electron Microscopy. Journal of Structural Biology. 189, 213-219 (2015).
- Sorzano, C. O. S., et al. Swarm optimization as a consensus technique for Electron Microscopy Initial Volume. Applied Analysis and Optimization. 2, 299-313 (2018).
- Pettersen, E. F., et al. UCSF Chimera--a visualization system for exploratory research and analysis. Journal of computational chemistry. 25, 1605-1612 (2004).
- Sorzano, C. O. S., et al. A new algorithm for high-resolution reconstruction of single particles by electron microscopy. Journal of Structural Biology. 204, 329-337 (2018).
- Vilas, J. L., et al. MonoRes: Automatic and Accurate Estimation of Local Resolution for Electron Microscopy Maps. Structure. 26, 337-344 (2018).
- Ramirez-Aportela, E., et al. Automatic local resolution-based sharpening of cryo-EM maps. Bioinformatics. 36, 765-772 (2020).
- Heymann, J. B. Validation of 3D EM Reconstructions: The Phantom in the Noise. AIMS Biophys. 2, 21-35 (2015).
- Lander, G. C., et al. Appion: An integrated, database-drive pipeline to facilitate EM image processing. Journal of Structural Biology. 166, 95-102 (2009).
- Suloway, C., et al. Automated molecular microscopy: The new Leginon system. Journal of Structural Biology. 151, 41-60 (2005).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten