Method Article
Crio-EM e Análise de Partículas Únicas com Scipion
Neste Artigo
Resumo
A análise de partículas únicas na microscopia crio-elétron é uma das principais técnicas utilizadas para determinar a estrutura dos conjuntos biológicos em alta resolução. A Scipion fornece as ferramentas para criar todo o gasoduto para processar as informações adquiridas pelo microscópio e alcançar uma reconstrução 3D do espécime biológico.
Resumo
A microscopia crio-elétron tornou-se uma das ferramentas mais importantes na pesquisa biológica para revelar as informações estruturais das macromoléculas em resolução quase atômica. Na análise de partículas únicas, a amostra vitrificada é imagens por um feixe de elétrons e os detectores no final da coluna de microscópio produzem filmes dessa amostra. Estes filmes contêm milhares de imagens de partículas idênticas em orientações aleatórias. Os dados precisam passar por um fluxo de trabalho de processamento de imagem com várias etapas para obter o volume 3D reconstruído final. O objetivo do fluxo de trabalho de processamento de imagens é identificar os parâmetros de aquisição para poder reconstruir o espécime em estudo. O Scipion fornece todas as ferramentas para criar esse fluxo de trabalho usando vários pacotes de processamento de imagens em uma estrutura integrativa, permitindo também a rastreabilidade dos resultados. Neste artigo, todo o fluxo de trabalho de processamento de imagem no Scipion é apresentado e discutido com dados provenientes de um caso de teste real, dando todos os detalhes necessários para ir desde os filmes obtidos pelo microscópio até uma reconstrução 3D final de alta resolução. Além disso, discute-se o poder de usar ferramentas de consenso que permitam combinar métodos e confirmar resultados ao longo de cada etapa do fluxo de trabalho, melhorando a precisão dos resultados obtidos.
Introdução
Na microscopia crio-elétron (crio-EM), a análise de partículas únicas (SPA) de espécimes congelados vitrificados é uma das variantes mais utilizadas e bem sucedidas de imagem para macromoléculas biológicas, pois permite compreender as interações moleculares e a função dos conjuntos biológicos1. Isso se deve aos recentes avanços nesta técnica de imagem que deu origem à "revolução da resolução"2 e permitiram a determinação bem sucedida de estruturas biológicas 3D com resolução quase atômica. Atualmente, a maior resolução alcançada no SPA crio-EM foi de 1,15 Å para apoferritin3 (entrada EMDB: 11668). Esses avanços tecnológicos compreendem melhorias na preparação da amostra4, na aquisição de imagens5 e nos métodos de processamento de imagens6. Este artigo está focado neste último ponto.
Resumidamente, o objetivo dos métodos de processamento de imagens é identificar todos os parâmetros de aquisição para inverter o processo de imagem do microscópio e recuperar a estrutura 3D do espécime biológico em estudo. Esses parâmetros são o ganho da câmera, o movimento induzido pelo feixe, as aberrações do microscópio (principalmente o desfoco), a orientação angular 3D e a tradução de cada partícula, e o estado conformacional em caso de ter um espécime com alterações conformais. No entanto, o número de parâmetros é muito alto e o crio-EM requer o uso de imagens de baixa dose para evitar danos à radiação, o que reduz significativamente a Relação Sinal-Ruído (SNR) das imagens adquiridas. Assim, o problema não pode ser resolvido inequivocamente e todos os parâmetros a serem calculados só podem ser estimativas. Ao longo do fluxo de trabalho de processamento de imagem, devem ser identificados os parâmetros corretos, descartando os restantes para finalmente obter uma reconstrução 3D de alta resolução.
Os dados gerados pelo microscópio são coletados em quadros. Simplificando, um quadro contém o número de elétrons que chegaram a uma posição específica (pixel) na imagem, sempre que detectores de contagem de elétrons são usados. Em um determinado campo de visão, vários quadros são coletados e isso é chamado de filme. Como doses baixas de elétrons são usadas para evitar danos à radiação que poderiam destruir a amostra, o SNR é muito baixo e os quadros correspondentes ao mesmo filme precisam ser mediados para obter uma imagem revelando informações estruturais sobre a amostra. No entanto, não apenas uma média simples é aplicada, a amostra pode sofrer mudanças e outros tipos de movimentos durante o tempo de imagem devido ao movimento induzido pelo feixe que precisa ser compensado. Os quadros compensados e médios originários de um micrografo.
Uma vez obtidos os micrógrafos, precisamos estimar as aberrações introduzidas pelo microscópio para cada um deles, chamada Função de Transferência de Contraste (CTF), que representa as mudanças no contraste do micrógrafo em função da frequência. Em seguida, as partículas podem ser selecionadas e extraídas, o que é chamado de colheita de partículas. Cada partícula deve ser uma pequena imagem contendo apenas uma cópia do espécime em estudo. Existem três famílias de algoritmos para colheita de partículas: 1) as que só usam alguma parametrização básica da aparência da partícula para encontrá-las em todo o conjunto de micrografos (por exemplo, tamanho de partículas), 2) as que aprendem como as partículas se parecem com o usuário ou um conjunto pré-treinado, e 3) as que usam modelos de imagem. Cada família tem propriedades diferentes que serão mostradas mais tarde.
O conjunto extraído de partículas encontradas nos micrografos será usado em um processo de classificação 2D que tem dois objetivos: 1) limpar o conjunto de partículas descartando o subconjunto contendo imagens de ruído puro, partículas sobrepostas ou outros artefatos, e 2) as partículas médias que representam cada classe poderiam ser usadas como informações iniciais para calcular um volume inicial 3D.
O cálculo inicial do volume 3D é o próximo passo crucial. O problema da obtenção da estrutura 3D pode ser visto como um problema de otimização em um cenário de solução multidimensional, onde o mínimo global é o melhor volume 3D que representa a estrutura original, mas vários minimas locais representando soluções subótimas podem ser encontradas, e onde é muito fácil ficar preso. O volume inicial representa o ponto de partida para o processo de busca, de modo que a estimativa de volume inicial ruim poderia nos impedir de encontrar o mínimo global. A partir do volume inicial, uma etapa de classificação 3D ajudará a descobrir diferentes estados conformacionais e a limpar novamente o conjunto de partículas; o objetivo é obter uma população estruturalmente homogênea de partículas. Depois disso, uma etapa de refinamento 3D será responsável por refinar os parâmetros angulares e de tradução de cada partícula para obter o melhor volume 3D possível.
Finalmente, nas últimas etapas, a reconstrução 3D obtida pode ser afiada e polida. O afiação é um processo de aumentar as altas frequências do volume reconstruído, e o polimento é um passo para refinar ainda mais alguns parâmetros, como a compensação de movimento induzido pelo CTF ou pelo feixe, ao nível das partículas. Além disso, alguns procedimentos de validação poderiam ser utilizados para entender melhor a resolução alcançada no final do fluxo de trabalho.
Após todas essas etapas, os processos de rastreamento e acoplamento7 ajudarão a dar um significado biológico à reconstrução 3D obtida, construindo modelos atômicos de novo ou adaptando modelos existentes. Se a alta resolução for alcançada, esses processos nos dirão as posições das estruturas biológicas, mesmo dos diferentes átomos, em nossa estrutura.
O Scipion8 permite criar todo o fluxo de trabalho combinando os pacotes de processamento de imagem mais relevantes de forma integrativa. Xmipp9, Relion10, CryoSPARC11, Eman12, Spider13, Cryolo14, Ctffind15, CCP416, Phenix17, e muitos outros pacotes podem ser incluídos no Scipion. Além disso, incorpora todas as ferramentas necessárias para beneficiar a integração, interoperabilidade, rastreabilidade e reprodutibilidade para fazer um acompanhamento completo de todo o fluxo de trabalho de processamento de imagem8.
Uma das ferramentas mais poderosas que o Scipion nos permite usar é o consenso, que significa comparar os resultados obtidos com vários métodos em uma etapa do processamento, fazendo uma combinação das informações transmitidas por diferentes métodos para gerar uma saída mais precisa. Isso poderia ajudar a aumentar o desempenho e melhorar a qualidade alcançada nos parâmetros estimados. Observe que um fluxo de trabalho mais simples pode ser construído sem o uso de métodos de consenso; no entanto, vimos o poder desta ferramenta22,25 e o fluxo de trabalho apresentado neste manuscrito irá usá-lo em várias etapas.
Todas as etapas que foram resumidas nos parágrafos anteriores serão explicadas detalhadamente na seção a seguir e combinadas em um fluxo de trabalho completo usando scipion. Além disso, será mostrado como usar as ferramentas de consenso para alcançar um acordo maior nas saídas geradas. Para isso, foi escolhido o conjunto de dados do Plasmodium falciparum 80S Ribosome (entrada EMPIAR: 10028, entrada EMDB: 2660). O conjunto de dados é formado por 600 filmes de 16 quadros de tamanho 4096x4096 pixels em um tamanho de pixel de 1.34Å tirado em um FEI POLARA 300 com uma câmera FEI FALCON II, com uma resolução relatada no EMDB é de 3.2Å18 .
Protocolo
1. Criar um projeto em Scipion e importar os dados
- Abra o Scipion e clique em Criar Projeto, especifique o nome do projeto e o local onde ele será salvo (Figura Suplementar 1). O Scipion abrirá a janela do projeto mostrando uma tela com, no lado esquerdo, um painel com uma lista de métodos disponíveis, cada um deles representa uma ferramenta de processamento de imagem que pode ser usada para gerenciar dados.
NOTA: Ctrl+F pode ser usado para encontrar um método se ele não aparecer na lista. - Para importar os filmes levados pelo microscópio selecione o pwem - importe filmes no painel esquerdo (ou digite-o ao pressionar Ctrl+F).
- Uma nova janela será aberta (Figura Suplementar 2). Lá, inclua o caminho para os dados e os parâmetros de aquisição. Neste exemplo, use a seguinte configuração: Tensão de microscópio 300 kV, aberração esférica 2.0 mm, Amplitude Contraste 0.1, Taxa de Ampliação 50000, Modo de taxa de amostragem para Imagem e Tamanho pixel 1.34 Å. Quando todos os parâmetros do formulário estiverem preenchidos, clique no botão Executar .
NOTA: Quando um método é iniciado, uma caixa aparece na tela na cor amarela rotulada como running. Quando um método termina, a caixa muda para verde, e o rótulo muda para final. Em caso de erro durante a execução de um método, a caixa aparecerá em vermelho, rotulada como falha. Nesse caso, verifique a parte inferior da tela, na guia Registro de saída uma explicação do erro aparecerá. - Quando o método terminar, verifique os resultados na parte inferior da tela na guia Resumo . Aqui, as saídas geradas pelo método são apresentadas, neste caso, o conjunto de filmes. Clique no botão Analisar resultados e uma nova janela aparecerá com a lista de filmes.
2. Alinhamento de filmes: de filmes a micrografias
- Use o método xmipp3 - alinhamento óptico que implementa fluxo óptico19. Use os seguintes parâmetros para preencher o formulário (Figura Suplementar 3): os Filmes de Entrada são aqueles obtidos na etapa 1, a gama em Quadros para ALINHAR é de 2 a 13, as outras opções permanecem com os valores padrão. Execute o programa.
NOTA: Os parâmetros em negrito em forma devem ser sempre preenchidos. Os outros terão um valor padrão ou não serão obrigatoriamente exigidos. Na parte superior da janela de formulário, os campos onde os recursos computacionais são distribuídos podem ser encontrados, como roscas, MPIs ou GPUs. - Clique em Analisar resultados para verificar os micrografos obtidos e a trajetória dos turnos estimados (Figura 1). Para cada micrografo visto: olhar para a densidade espectral de poder (PSD), as trajetórias obtidas para alinhar o filme (um ponto por quadro) em coordenadas cartesianas e polares, e o nome do arquivo do micrografo obtido (clicando nele, o micrografo pode ser inspecionado). Observe que as partículas do espécime são muito mais visíveis no micrografo, em comparação com um único quadro do filme.
3. Estimativa ctf: cálculo das aberrações do microscópio
- Primeiro, use o método grigoriefflab - ctffind15. A configuração é: os Micrografos de Entrada são a saída da etapa 2, o fator de baixa do CTF manual está definido como 1,5 e a faixa de resolução vai de 0,06 a 0,42. Além disso, nas opções Avançadas (que podem ser encontradas selecionando esta escolha no Nível de Especialista do formulário), defina o tamanho da janela para 256. Os demais parâmetros permanecem com os valores padrão (Figura Suplementar 4).
NOTA: Na maioria dos métodos no Scipion, a opção Advanced mostra mais parâmetros de configuração. Use essas opções com cuidado, quando o programa a ser lançado for completamente conhecido e o significado dos parâmetros for compreendido. Alguns parâmetros podem ser difíceis de preencher sem dar uma olhada nos dados; nesse caso, Scipion mostra uma varinha mágica no lado direito que mostrará uma janela de mago (Figura Suplementar 5). Por exemplo, no campo de Resolução desta forma é especialmente útil, pois esses valores devem ser selecionados para cobrir aproximadamente a região do primeiro zero ao último anel perceptível do PSD. - Clique em Executar e em Analisar resultados (Figura 2) quando o método terminar. Verifique se o CTF estimado corresponde com o experimental. Para isso, olhe para o PSD e compare os anéis estimados no canto com os provenientes dos dados. Verifique também os valores de desfocto obtidos para encontrar valores inesperados e os respectivos micrografos podem ser descartados ou recalculados. Neste exemplo, todo o conjunto de micrografos pode ser usado.
NOTA: Use os botões na parte inferior da janela para fazer um subconjunto de micrografos (com botão vermelho Micrographs ) e para recalcular um CTF (com botão vermelho Recalculado CTFs ), em caso de necessidade. - Para refinar a estimativa anterior, utilize xmipp3 - estimativa ctf20. Selecione como Micrografo de Entrada a saída da etapa 2, selecione a opção Use defoci a partir de uma estimativa anterior do CTF, pois a estimativa anterior do CTF escolhe a saída de grigoriefflab - ctffind e, no nível Avançado, altera o tamanho da janela para 256 (Figura Suplementar 6). Executá-lo.
- Clique em Analisar resultados para verificar os CTFs obtidos. Com esse método, mais dados são estimados e representados em algumas colunas extras. Como nenhum deles mostra valores estimados incorretos, todos os micrografos serão usados nas etapas seguintes.
4. Colheita de partículas: encontrar partículas nos micrografos
- Antes de iniciar a coleta, realize um pré-processo dos micrografos. Abra xmipp3 - micrografos pré-processo, definidos como micrografos de entrada aqueles obtidos na etapa 2 e selecione as opções Remova pixels ruins? com Múltiplos de Stddev a 5, e micrografos Downsample? Clique em Executar e verifique se o tamanho dos micrografos resultantes foi reduzido.
- Para a escolha use xmipp3 - escolha manual (etapa 1) e xmipp3 - auto-picking (etapa 2)21. A escolha manual permite preparar manualmente um conjunto de partículas com as quais a etapa de colheita automática aprenderá e gerará o conjunto completo de partículas. Primeiro, execute xmipp3 - escolha manual (etapa 1) com micrografos de entrada como os micrografos obtidos no pré-processo anterior. Clique em Execute e uma nova janela interativa aparecerá (Figura 3).
- Nesta janela é apresentada uma lista dos micrografos (Figura 3a) e outras opções. Tamanho da mudança (px) para 150, este será o tamanho da caixa contendo cada partícula. O micrografo selecionado aparece em uma janela maior. Escolha uma região e escolha todas as partículas visíveis nela (Figura 3b). Em seguida, clique em Ativar treinamento para iniciar o aprendizado. As demais regiões do micrografo são automaticamente escolhidas (Figura 3c). Verifique as partículas escolhidas e inclua mais clicando nela, ou remova as incorretas com shift+clicking, se necessário.
- Selecione o próximo micrografo na primeira janela. O micrografo será automaticamente escolhido. Verifique novamente para incluir ou remover algumas partículas, se necessário. Repita esta etapa com, aproximadamente, 5 micrografos para criar um conjunto de treinamento representativo.
- Feito isso, clique em Coordenadas na janela principal para salvar as coordenadas de todas as partículas colhidas. O conjunto de treinamento de partículas está pronto para ir para a auto-colheita para completar o processo para todos os micrografos.
- Abra xmipp3 - auto-picking (etapa 2) indicando na coleta de partículas Xmipp executar a escolha manual anterior, e Micrografos para escolher como mesmo supervisionado. Clique em Executar. Este método irá gerar como saída um conjunto de cerca de 100000 coordenadas.
- Aplique uma abordagem de consenso, por isso realize um segundo método de escolha para selecionar as partículas em que ambos os métodos concordam. Torre aberta - criolo picking14 e selecione os micrografos pré-processados como Micrografos de Entrada, Use modelo geral? Executá-lo. Este método deve gerar também cerca de 100000 coordenadas.
- Run xmipp3 - consenso profundo picking22. Como as coordenadas de entrada incluem a saída de sphire - picking cryolo (passo 4.7) e xmipp3 - auto-picking (passo 4.6), definir tipo de modelo Select para Pré-treinado, e Treinar e pontuar diretamente com o modelo pré-treinado? Para Sim (Figura Suplementar 9). Executá-lo.
- Clique em Analisar resultados e, na nova janela, no ícone ocular ao lado de Selecionar partículas/coordenadas com altos valores 'zScoreDeepLearning1'. Uma nova janela será aberta com uma lista de todas as partículas (Figura 4). Os valores zScore na coluna dão uma visão da qualidade de uma partícula, valores baixos significam má qualidade.
- Clique no rótulo_xmipp_zScoreDeepLearning para encomendar as partículas do mais alto ao nível mais baixo do zScore. Selecione as partículas com zScore acima de 0,75 e clique em Coordenadas para criar o novo subconjunto. Isso deve criar um subconjunto com aproximadamente 50000 coordenadas.
- Open xmipp3 - limpador de micrografo profundo. Selecione como As coordenadas de entrada obtidas no subconjunto obtido na etapa anterior, micrografos fonte como as coordenadas e mantenha Threshold em 0,75. Executá-lo. Verifique na guia Resumo que o número de coordenadas foi reduzido, embora neste caso, apenas algumas coordenadas sejam removidas.
NOTA: Esta etapa é capaz de limpar adicionalmente o conjunto de coordenadas e pode ser muito útil na limpeza de outros conjuntos de dados com mais artefatos de filme como zonas de carbono ou grandes impurezas. - Executar xmipp3 - partículas extratos (Figura Suplementar 10). Indique como a entrada coordena as coordenadas obtidas após a etapa anterior, micrografias fonte como outras, micrografias de entrada como a saída da etapa 2, estimativa ctf como a saída do xmipp3 - estimativa ctf, fator downsampling para 3, e tamanho da caixa de partículas para 100. Na guia Pré-processo do formulário selecione Sim para todos. Executá-lo.
- Verifique se a saída deve conter as partículas em tamanho reduzido de 100x100 pixels e um tamanho de pixel de 4.02Å/px.
- Execute novamente xmipp3 - extraia partículas alterando os seguintes parâmetros: Fator de baixa para 1, e tamanho da caixa de partículas para 300. Verifique se a saída é o mesmo conjunto de partículas, mas agora na resolução completa.
5. Classificação 2D: agrupando partículas semelhantes
- Abra o método criosparc2 - classificação 2d11 com partículas de entrada como as obtidas na etapa 4.11 e, na aba classificação 2D, o número de classes para 128, mantenha todos os outros parâmetros com os valores padrão. Executá-lo.
- Clique em Analisar resultados e, em seguida, no ícone dos olhos ao lado das classes de partículas exibir com Scipion (Figura 5). Esta classificação ajudará a limpar o conjunto de partículas, pois várias classes parecerão ruidas ou com artefatos. Selecione as classes que contenham boas visualizações. Clique em Partículas (botão vermelho na parte inferior da janela) para criar o subconjunto mais limpo.
- Agora, abra xmipp3 - cl2d23 e defina como imagens de entrada as imagens obtidas na etapa anterior e número de classes como 128. Clique em Executar.
NOTA: Esta segunda classificação é usada como etapa de limpeza adicional do conjunto de partículas. Normalmente é útil para remover o máximo de partículas ruidas possível. No entanto, se um fluxo de trabalho mais simples for desejado, apenas um método de classificação 2D pode ser usado. - Quando o método terminar, verifique as 128 classes geradas clicando em Analisar resultados e no que mostrar: classes. A maioria das classes geradas mostra uma projeção da macromolécula com algum nível de detalhes. No entanto, alguns deles parecem barulhentos (neste exemplo aproximadamente 10 classes). Selecione todas as boas classes e clique no botão Classes para gerar um novo subconjunto apenas com as boas. Este subconjunto será usado como entrada para um dos métodos para gerar um volume inicial. Com as mesmas classes selecionadas clique em Partículas para criar um subconjunto mais limpo depois de remover aqueles pertencentes às classes ruins.
- Pwem aberto - subconjunto com conjunto completo de itens como a saída de 4.13 (todas as partículas em tamanho real), Faça subconjunto aleatório para No, Outro conjunto como o subconjunto de partículas criado na etapa anterior e ajuste a operação como intersecção. Isso extrairá o subconjunto anterior das partículas em resolução total.
6. Estimativa inicial de volume: construindo o primeiro palpite do volume 3D
- Nesta etapa, estime dois volumes iniciais com métodos diferentes e, em seguida, use uma ferramenta de consenso para gerar o volume 3D estimado final. Abra xmipp3 - reconstrua método significativo24 com classes de Entrada como as obtidas após a etapa 5, grupo de simetria como c1, e mantenha os parâmetros restantes com seus valores padrão (Figura Suplementar 11). Executá-lo.
- Clique em Analisar resultados. Verifique se um volume de baixa resolução de tamanho 100x100x100 pixels e um tamanho de pixel de 4.02Å/px é obtido.
- Abrir xmipp3 - volumes de cultura/redimensionar (Figura Suplementar 12) utilizando como Volumes de Entrada o obtido na etapa anterior, Resize volumes? Executá-lo. Verifique na guia Resumo que o volume de saída tem o tamanho correto.
- Agora, crie o segundo volume inicial. Relion aberto - modelo inicial 3D10, como partículas de entrada usam as partículas boas em resolução total (saída de 5,5) e definem o diâmetro da máscara de partículas para 402Å, mantenha os parâmetros restantes com os valores padrão. Executá-lo.
- Clique em Analisar resultados e, em seguida, em Volume de exibição com: fatias. Verifique se um volume de baixa resolução, mas com a forma principal da estrutura, é obtido (Figura Suplementar 13).
- Agora, abra pwem - junte conjuntos para combinar os dois volumes iniciais gerados para criar a entrada para o método de consenso. Basta indicar volumes como tipo de entrada e selecionar os dois volumes iniciais no conjunto De entrada. Executá-lo. A saída deve ser um conjunto contendo dois itens com ambos os volumes.
- A ferramenta de consenso é a incluída no xmipp3 - consenso do enxame25. Abra. Use como Imagens em tamanho real as partículas boas em resolução total (saída de 5,5), como volumes iniciais do conjunto com dois itens gerados na etapa anterior, e certifique-se de que o grupo Simetria é c1. Clique em Executar.
- Clique em Analisar resultados. Verifique se um volume de saída mais detalhado é obtido (Figura 6). Embora haja mais ruído em torno da estrutura, ter mais detalhes no mapa da estrutura ajudará as seguintes etapas de refinamento para evitar minima local.
NOTA: Se o UCSF Chimera26 estiver disponível, use o último ícone na parte superior da janela para fazer uma visualização 3D do volume obtido. - Abra e execute relion - 3D auto-refine10 para fazer uma primeira atribuição angular 3D das partículas. Selecione como partículas de entrada a saída de 5.5 e defina o diâmetro da máscara de partículas para 402Å. Na guia de mapa 3D de referência, selecione como volume de entrada o obtido na etapa anterior, Simetria como c1 e filtro de passe baixo inicial para 30Å (Figura Suplementar 14).
- Clique em Analisar resultados. Na nova janela selecione final como Volume para visualizar e clicar em Exibir volume com: fatias para ver o volume obtido. Verifique também a correlação da concha Fourier (FSC) clicando em gráficos de resolução display na janela de resultados e na cobertura angular na distribuição angular do Display: gráfico 2D (Figura 7). O volume reconstruído contém muito mais detalhes (provavelmente com algumas áreas borradas na parte externa da estrutura), e o FSC cruza o limiar de 0,143 em torno de 4,5Å. A cobertura angular cobre toda a esfera 3D.
Classificação .3D 7: descobrir estados conformais
- Usando uma abordagem de consenso, se diferentes estados conformais estiverem nos dados podem ser descobertos. Reluão aberto - classificação 3D10 (Figura Suplementar 15). Como partículas de entrada usam essas obtidas apenas em 6.10, e definem o diâmetro da máscara de partículas para 402Å. Na guia de mapa Referência 3D, use como volume de entrada o obtido após a etapa 6.10, defina simetria para c1 e filtro de passe baixo inicial para 15Å. Por fim, na guia Otimização, defina o número de classes para 3. Executá-lo.
- Confira os resultados clicando em Analisar resultados, selecione Mostrar classificação no Scipion. As três classes geradas e algumas medidas interessantes são mostradas. As duas primeiras classes devem ter um número semelhante de imagens atribuídas (coluna de tamanho ) e muito semelhantes, enquanto a terceira tem menos imagens e uma aparência mais borrada. Além disso, as rlnAccuracyRotations e rlnAccuracyTranslations devem ser claramente melhores para as duas primeiras classes. Selecione as duas melhores classes e clique no botão Classes para gerar um subconjunto contendo-as.
- Repita as etapas 7.1 e 7.2 para gerar um segundo grupo de boas classes. Ambos serão a entrada da ferramenta de consenso.
- Abra e execute xmipp3 - classes de consenso 3D e selecione como Classes de Entrada os dois subconjuntos gerados nas etapas anteriores.
- Clique em Analisar resultados. O número de partículas coincidentes entre as classes é apresentado: o primeiro valor é o número de partículas coincidentes na primeira classe do subconjunto 1 e na primeira classe do subconjunto 2, o segundo valor é o número de partículas coincidentes na primeira classe do subconjunto 1 e da segunda classe do subconjunto 2, etc. Verifique se as partículas são atribuídas aleatoriamente às classes um ou dois, o que significa que o método de classificação 3D não é capaz de encontrar alterações conformais. Dado este resultado, todo o conjunto de partículas será usado para continuar o processamento.
8.3D refinamento: refinar atribuições angulares de uma população homogênea
- Mais uma vez, aplique uma abordagem de consenso nesta etapa. Primeiro, abra e execute pwem - subconjunto com conjunto completo de itens como a saída de 6.9, Faça subconjunto aleatório para Sim e Número de elementos para 5000. Com isso, cria-se um subconjunto de imagens com um alinhamento prévio para treinar o método utilizado na etapa seguinte.
- Abra xmipp3 - alinhado profundo, defina imagens de entrada como a saída de boas partículas obtidas em 5.5, Volume como o obtido após 6.10, Conjunto de treinamento de entrada como o criado na etapa anterior, resolução de destino para 10Å, e mantenha os parâmetros restantes com os valores padrão (Figura Suplementar 16). Clique em Executar.
- Clique em Analisar resultados para verificar a distribuição angular obtida, onde não há direções ausentes e a cobertura angular melhora ligeiramente em comparação com a de 6,10 (Figura 8).
- Abra e execute xmipp3 - compare ângulos e selecione como partículas de entrada 1 a saída de partículas de 6,9 e partículas de entrada 2 a saída de 8.2, certifique-se de que o grupo Simetria é c1. Este método calcula o acordo entre xmipp3 - linha profunda e relion - refino automático 3D.
- Clique em Analisar resultados, mostra-se a lista de partículas, com as diferenças obtidas em turnos e ângulos. Clique no ícone da barra na parte superior da janela, outra janela será aberta que permite fazer parcelas das variáveis calculadas. Selecione _xmipp_angleDiff e clique em Plot para ver uma representação das diferenças angulares por partícula. Faça o mesmo com _xmipp_shiftDiff. Nessas figuras, aproximadamente em metade das partículas ambos os métodos concordam (Figura 9). Selecione as partículas com diferenças angulares inferiores a 10º e crie um novo subconjunto.
- Agora, abra xmipp3 - highres27 para fazer um refinamento local dos ângulos atribuídos. Primeiro, selecione como Imagens em tamanho real as imagens obtidas na etapa anterior, e como inicial volumes a saída de 6.9, definir raio de partícula para 150 pixels e grupo Simetria como c1. Na guia de atribuição Angular, defina o alinhamento de imagem para Local, Número de iterações para 1 e Max. Resolução-alvo como 5Å/px (Figura Suplementar 17). Executá-lo.
- Na guia Resumo verifique se o volume de saída é menor que 300x300x300 pixels e com tamanho de pixel ligeiramente maior.
- Clique em Analisar resultados para ver os resultados obtidos. Clique em Exibir gráficos de resolução para ver o FSC e no volume de exibição: Reconstruído para ver o volume obtido (Figura Suplementar 18). Um bom volume de resolução próximo a 4-3.5Å é obtido.
- Clique em Exibir partículas de saída e, na janela com a lista de partículas, clique no ícone da barra. Na nova janela, selecione Tipo como Histograma, com 100 Bins, selecione _xmipp_cost rótulo e, finalmente, pressione Plot (Figura Suplementar 19). Dessa forma, é apresentado o histograma do rótulo de custo , que contém a correlação da partícula com a direção de projeção selecionada para ele. Neste caso, uma função de densidade unimodal é obtida, o que é um sinal de não ter populações diferentes no conjunto de partículas. Assim, todos eles serão usados para continuar o refinamento
NOTA: No caso de ver uma função de densidade multimodal, o conjunto de partículas pertencentes ao máximo superior deve ser selecionado para continuar o fluxo de trabalho apenas com elas. - Abrir e executar novamente xmipp3 - highres com Continue de uma corrida anterior? Sim, definir como Imagens em tamanho real aquelas obtidas após 8,5 e Selecionar execução anterior com a execução anterior de Xmipp Highres. Na guia de atribuição Angular , defina o alinhamento de imagem para Local, com 1 iteração e 2,6Å/px como resolução de destino (resolução completa).
- Agora, a saída deve conter um volume em resolução total (tamanho 300x300x300 pixels). Clique em Analisar resultados para verificar novamente o volume obtido e o FSC, que agora deve ser um volume de alta resolução em torno de 3Å (Figura 10).
9. Avaliação e pós-processamento
- Open xmipp3 - MonoRes28 local. Este método calculará a resolução localmente. Definir como Volume de Entrada o obtido após 8.10, definir Gostaria de usar metade dos volumes? Executá-lo.
- Clique em Analisar resultados e selecione Mostrar resolução histograma e mostrar fatias coloridas (Figura 11). A resolução nas diferentes partes do volume é mostrada. A maioria dos voxels da parte central da estrutura deve apresentar resoluções em torno de 3Å, enquanto as piores resoluções são alcançadas nas partes externas. Além disso, um histograma das resoluções por voxel é mostrado com um pico em torno (mesmo abaixo) 3Å.
- Abra e execute xmipp3 - localdeblur afiando29 para aplicar um afiação. Selecione como Mapa de Entrada o obtido em 8.10, e como Mapa de Resolução o obtido na etapa anterior com MonoRes.
- Clique em Analisar resultados para verificar os volumes obtidos. Abra o último, correspondendo à última iteração do algoritmo. Recomenda-se abrir o volume com outras ferramentas, como a UCSF Quimera26, para ver melhor as características do volume em 3D (Figura 12).
- Por fim, abra a ferramenta de validação incluída no xmipp3 - valide o overfitting30 que mostrará como a resolução muda com o número de partículas. Abra-o e inclua como partículas de entrada as partículas obtidas na etapa 8.5, definir Calcular o ruído com destino à resolução? Em opções avançadas, defina o número de partículas para "500 1000 1500 2000 3000 5000 10000 15000 20000" (Figura Suplementar 20). Executá-lo.
- Clique em Analisar resultados. Dois lotes aparecerão (Figura 13) com a evolução da resolução, na linha verde, à medida que cresce o número de partículas utilizadas na reconstrução. A linha vermelha representa a resolução alcançada com uma reconstrução do ruído gaussiano alinhado. A resolução melhora com o número de partículas e uma grande diferença da reconstrução das partículas em relação à do ruído, que é um indicador de ter partículas com boas informações estruturais.
- A partir dos resultados anteriores, poderia ser realizado um ajuste de um modelo no volume pós-processado, o que permitiria a descoberta das estruturas biológicas da macromolécula.
Resultados
Utilizamos o conjunto de dados do Plasmodium falciparum 80S Ribosome (entrada EMPIAR: 10028, Entrada emDB: 2660) para realizar o teste e, com o protocolo Scipion apresentado na seção anterior, foi alcançado um volume reconstruído de alta resolução 3D do macromolécula neste exemplo específico, começando com as informações coletadas pelo microscópio que consistem em imagens muito ruidosas contendo projeções 2D em qualquer orientação da amostra.
Os principais resultados obtidos após a execução de todo o protocolo são apresentados na Figura 10, Figura 11 e Figura 12. A Figura 10 representa o volume 3D obtido antes do pós-processamento. Na Figura 10a, um FSC de 3 Å pode ser visto, que está muito próximo do limite nyquist (com dados com um tamanho de pixel de 1,34 Å, o limite de Nyquist é de 2,6 Å). A Figura 10b mostra algumas fatias do volume 3D reconstruído com altos níveis de detalhes e estruturas bem definidas. Na Figura 11 são apresentados os resultados após análise local da resolução do volume 3D obtido. Pode-se ver que a maioria dos voxels na estrutura alcança uma resolução abaixo de 3 Å, principalmente aqueles localizados na parte central da estrutura. No entanto, a parte externa mostra resoluções piores, o que é consistente com o borrão que aparece nessas áreas nas fatias da Figura 10b. A Figura 12 mostra o mesmo mapa 3D após o pós-processamento que é capaz de destacar as frequências mais altas do volume, revelando mais detalhes e melhorando a representação, o que pode ser visto especialmente na apresentação 3D na Figura 12c.
Na Figura 14, Quimera26 foi utilizada para ver uma representação 3D do volume obtido (Figura 14a), do pós-processado (Figura 14b) e do mapa de resolução (Figura 14c), colorido com o código de cores das resoluções locais. Isso pode dar ainda mais informações sobre a estrutura obtida. Esta ferramenta é muito útil para obter uma visão da qualidade do volume obtido, pois pequenos detalhes em todo o contexto 3D da estrutura podem ser vistos. Quando a resolução alcançada é suficiente, até mesmo algumas partes bioquímicas da estrutura podem ser encontradas (por exemplo, alfa-helices na Figura 14d. Nesta figura, deve-se destacar a alta resolução alcançada em todas as partes centrais da estrutura 3D, que podem ser vistas como as áreas azuis escuras na Figura 14c.
Todos os resultados anteriores foram alcançados graças a um bom desempenho de todo o protocolo, mas isso pode não ser o caso. Há várias maneiras de identificar um mau comportamento. No caso mais geral, isso acontece quando a estrutura obtida tem baixa resolução e não é capaz de evoluir para uma melhor. Um exemplo disso é apresentado na Figura 15. Um volume embaçado (Figura 15c) resulta em um FSC baixo, que pode ser visto na curva FSC (Figura 15a) e no histograma da estimativa local (Figura 15b). Este exemplo foi gerado usando um método de refinamento 3D com dados de entrada incorretos, pois esperava algumas propriedades específicas no conjunto de entrada de partículas que elas não cumprem. Como se pode ver, é sempre muito importante saber como os diferentes métodos esperam receber os dados e prepará-los corretamente. Em geral, quando uma saída como a da Figura 15 é obtida, pode haver um problema no fluxo de trabalho de processamento ou nos dados subjacentes.
Existem vários pontos de verificação ao longo do fluxo de trabalho que podem ser analisados para saber se o protocolo evolui corretamente ou não. Por exemplo, logo após a colheita, vários dos métodos discutidos anteriormente podem classificar as partículas e dar uma pontuação para cada uma delas. No caso de ter partículas ruins, esses métodos permitem identificá-las e removê-las. Além disso, a classificação 2D pode ser um bom indicador de ter um conjunto ruim de partículas. A Figura 16 mostra um exemplo de um conjunto tão ruim. Na Figura 16a, boas classes contendo alguns detalhes da estrutura são mostradas, enquanto a Figura 16b mostra classes ruins, que são ruidosas ou não centraladas, neste último caso pode-se ver que a colheita foi incorreta e duas partículas parecem aparecer juntas. Outro ponto de verificação é a estimativa inicial de volume, a Figura 17 mostra um exemplo de estimativas iniciais boas (Figura 17a) e ruins (Figura 17b). A estimativa ruim foi criada usando uma configuração incorreta para o método. Deve-se levar em conta que todas as configurações devem ser feitas com cuidado, escolhendo adequadamente todos os parâmetros de acordo com os dados analisados. Em caso de não ter um mapa com algumas informações estruturais mínimas, o refinamento a seguir não será capaz de obter uma boa reconstrução.
Quando o problema é uma aquisição ruim, na qual os filmes não preservam informações estruturais, será impossível extrair partículas boas delas e obter um processamento bem sucedido. Nesse caso, mais filmes devem ser coletados para obter uma reconstrução 3D de alta resolução. Mas, se esse não for o caso, existem várias maneiras de gerenciar problemas ao longo do fluxo de trabalho de processamento. Se a escolha não for boa o suficiente, existem várias maneiras de tentar corrigi-la, por exemplo, repetindo a escolha, usando diferentes métodos ou tentando escolher manualmente mais partículas para ajudar os métodos a aprender com eles. Durante a classificação 2D, se apenas algumas classes forem boas, considere também repetir o processo de escolha. Na estimativa inicial de volume, tente usar vários métodos se alguns deles derem resultados imprecisos. O mesmo se aplica ao refinamento 3D. Após esse raciocínio, neste manuscrito, foram apresentadas várias ferramentas de consenso, que poderiam ser muito úteis para evitar problemas e continuar o processamento com dados precisos. Graças ao uso de um consenso entre vários métodos, podemos descartar dados difíceis de escolher, classificar, alinhar, etc., que provavelmente é um indicador de dados ruins. No entanto, se vários métodos forem capazes de concordar na saída gerada, provavelmente esses dados contêm informações valiosas para continuar o processamento.
Encorajamos o leitor a baixar mais conjuntos de dados e tentar processá-los seguindo as recomendações apresentadas neste manuscrito e criar um fluxo de trabalho semelhante combinando pacotes de processamento usando o Scipion. Tentar processar um conjunto de dados é a melhor maneira de aprender o poder das ferramentas de processamento disponíveis no estado da arte no Cryo-EM, conhecer as melhores regras para superar as possíveis desvantagens que aparecem durante o processamento e aumentar o desempenho dos métodos disponíveis em cada caso de teste específico.
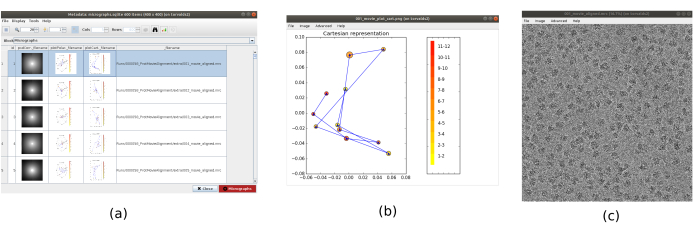
Figura 1. Resultado do alinhamento do filme. a A janela principal dos resultados, com uma lista de todos os micrografos gerados e informações adicionais: a densidade espectral de energia, a trajetória do alinhamento estimado em coordenadas polares, o mesmo nas coordenadas cartesianas, o nome do arquivo do micrografo gerado. b A trajetória de alinhamento representada nas coordenadas cartesianas. c O micrografo gerado. Clique aqui para ver uma versão maior desta figura.
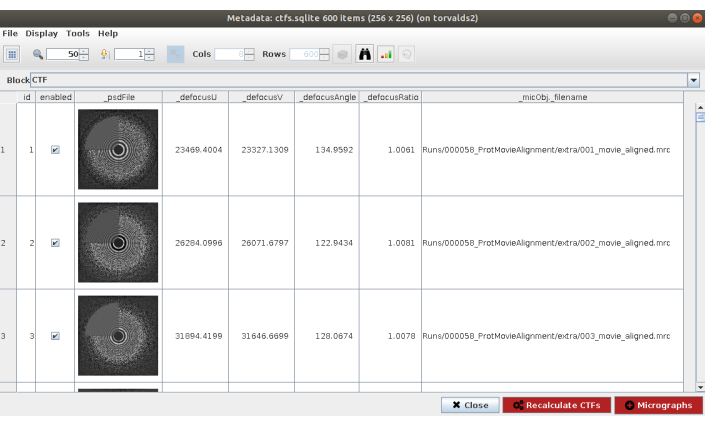
Figura 2. Estimativa ctf com resultado ctffind. A principal janela com os resultados inclui um valor com o PSD estimado (em um canto) junto com o PSD proveniente dos dados, e vários desfoque params. Clique aqui para ver uma versão maior desta figura.
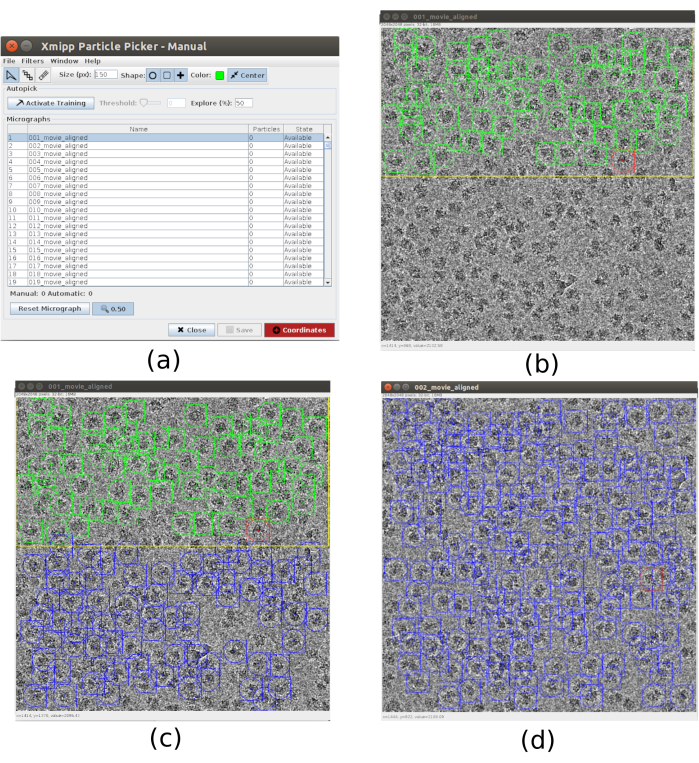
Figura 3. Manual de escolher janelas com Xmipp. a A janela principal com a lista de micrografos para processar e alguns outros parâmetros. b Colher manualmente partículas dentro de uma região de um micrografo. c e (d) Partículas escolhidas automaticamente para serem supervisionadas para criar um conjunto de partículas de treinamento para o método de colheita automática Xmipp. Clique aqui para ver uma versão maior desta figura.
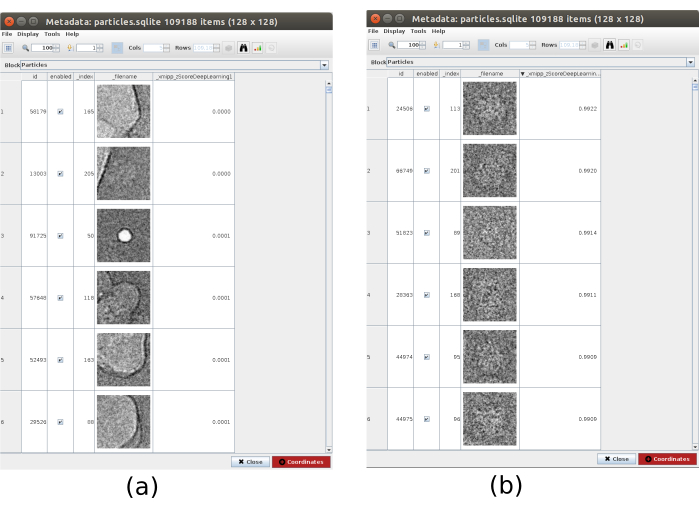
Figura 4. Profunda escolha de consenso com o resultado de Xmipp. O parâmetro zScoreDeepLearning dá peso à bondade de uma partícula e é fundamental para descobrir partículas ruins. a Os valores zScores mais baixos estão associados a artefatos. b Os zScores mais altos estão associados a partículas que contêm a macromolécula. Clique aqui para ver uma versão maior desta figura.
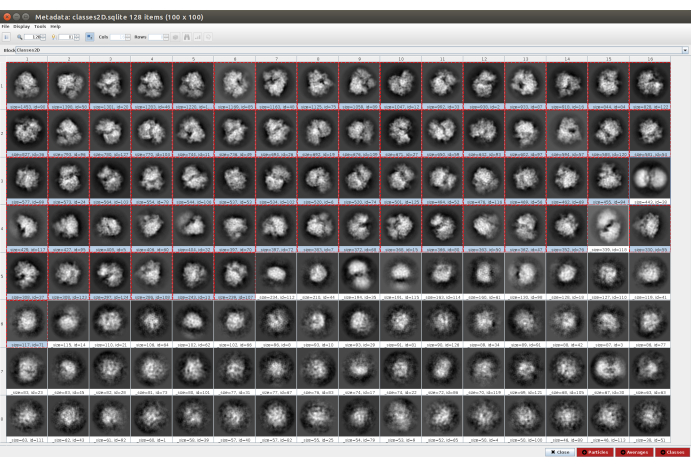
Figura 5. Classificação 2D com resultado cryosparc. As classes geradas (médias de subconjuntos de partículas provenientes da mesma orientação) são mostradas. Várias boas aulas selecionadas em vermelho (com algum nível de detalhe) e algumas classes ruins não selecionadas (aulas ruidosas e não centraladas). Clique aqui para ver uma versão maior desta figura.
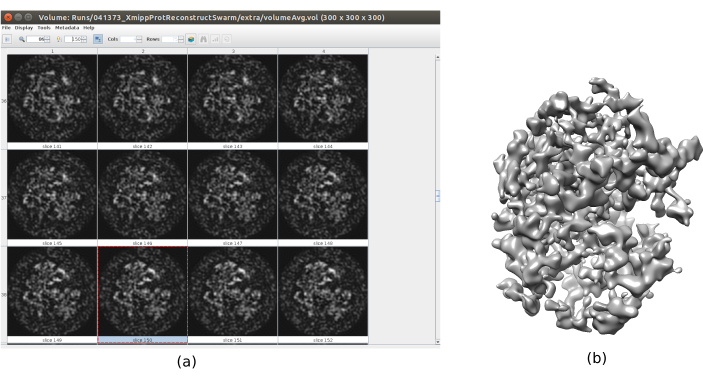
Figura 6. Volume inicial 3D com resultado de consenso de enxame. Uma visão do volume inicial 3D obtido após a execução da ferramenta de consenso xmipp3 - consenso de enxame, usando as estimativas anteriores de volume inicial 3D de Xmipp e Relion. a O volume é representado por fatias. b Visualização 3D do volume. Clique aqui para ver uma versão maior desta figura.
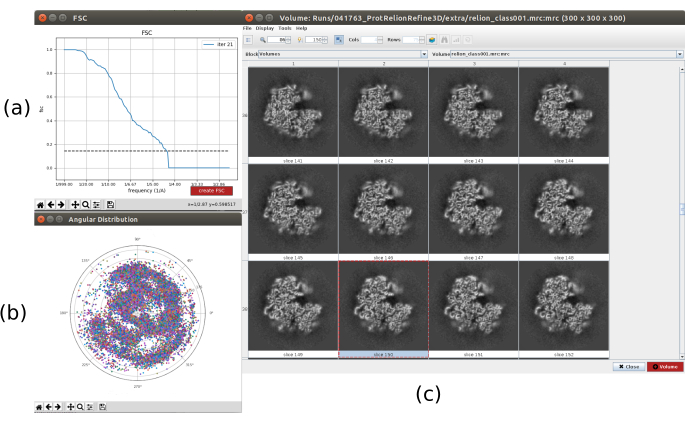
Figura 7. Refinamento de um volume inicial 3D com resultado Relion. a Curva FSC obtida, cruzando o limiar em um 4,5Å, aproximadamente. b Cobertura angular mostrada como visão superior da esfera 3D. Neste caso, como não há simetria, as partículas atribuídas devem cobrir toda a esfera. c Volume refinado representado por fatias. Clique aqui para ver uma versão maior desta figura.
Figura 8. Alinhamento 3D baseado no aprendizado profundo com o resultado Xmipp. Os resultados gerados por xmipp3 - método de alinhamento profundo para alinhamento 3D. a A atribuição angular para cada partícula na forma de matriz de transformação. b A cobertura angular. Clique aqui para ver uma versão maior desta figura.
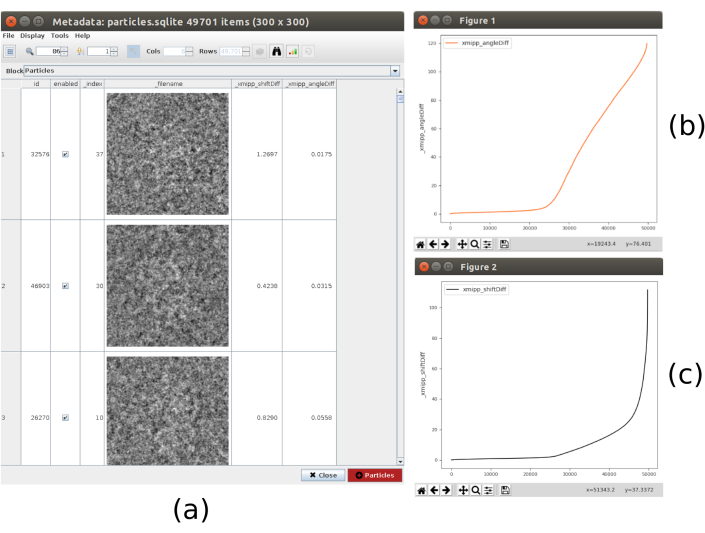
Figura 9. Resultado de consenso de alinhamento 3D. a Lista de partículas com as diferenças obtidas nos parâmetros de mudança e ângulos. b Traçar as diferenças angulares por partícula. c Parcelar a diferença de turno por partícula. Clique aqui para ver uma versão maior desta figura.
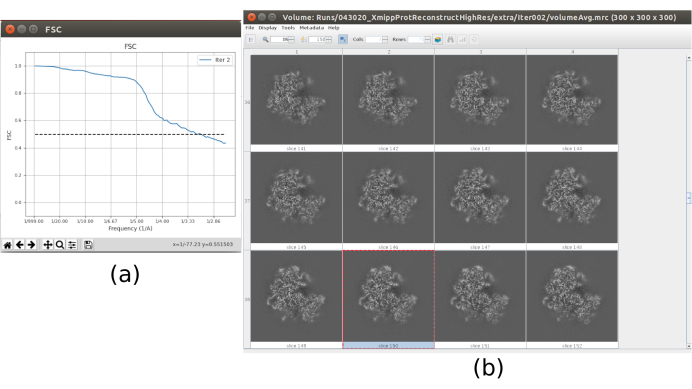
Figura 10. Iteração final do resultado de refinamento 3D. a Curva FSC. b Volume obtido em resolução total por fatias. Clique aqui para ver uma versão maior desta figura.
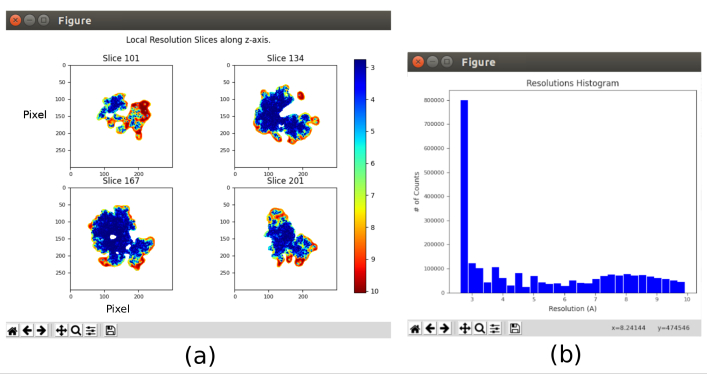
Figura 11. Análise de resolução local com resultado Xmipp. Resultados do método xmipp3 - MonoRes locais. a Algumas fatias representativas coloridas com o valor de resolução por voxel, conforme indicado no código de cores. b Histograma de resolução local. Clique aqui para ver uma versão maior desta figura.
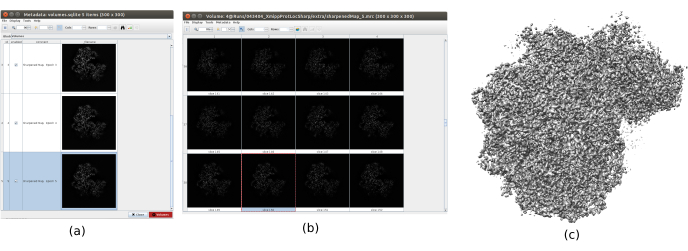
Figura 12. Afiação com o resultado de Xmipp. Resultados do método de afiação xmipp3 - localdeblur . a Lista de volumes obtidos por iteração. b Volume 3D obtido após a última iteração representada por fatias. c Uma representação 3D do volume final. Clique aqui para ver uma versão maior desta figura.
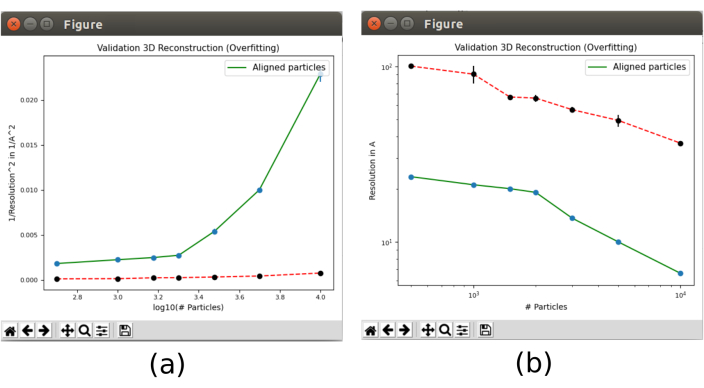
Figura 13. Validar a ferramenta de overfit no resultado Xmipp. Resultados de xmipp3 - superfmontagem de validação. A linha verde corresponde à reconstrução de dados, a linha vermelha do ruído. a Inverso da resolução quadrada com o logaritmo do número de partículas. b Resolução com o número de partículas. Clique aqui para ver uma versão maior desta figura.
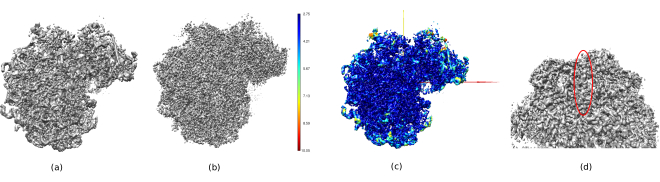
Figura 14. Várias representações 3D do volume obtido. a Volume pré-processado. b Volume pós-processado. (c) Resolução local, voxels azuis escuros são aqueles com maior resolução (2.75Å) e voxels vermelho escuro são aqueles com resolução mais baixa (10.05Å). d Zoom no volume pós-processado onde um alfa-hélice (oval vermelho) pode ser visto. Clique aqui para ver uma versão maior desta figura.
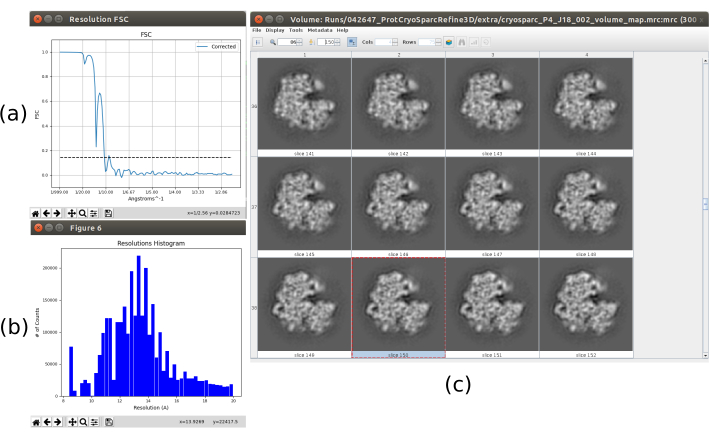
Figura 15. Exemplo de uma má reconstrução 3D. a Curva FSC com queda acentuada e cruzando o limiar em baixa resolução. b Histograma de resolução local. c Volume 3D por fatias. Clique aqui para ver uma versão maior desta figura.
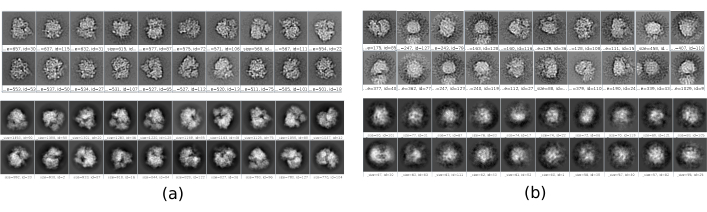
Figura 16. Exemplo de classes 2D. a Boas aulas mostrando algum nível de detalhes. b Aulas ruins contendo ruído e artefatos (parte superior obtida com Xmipp, inferior com CryoSparc). Clique aqui para ver uma versão maior desta figura.
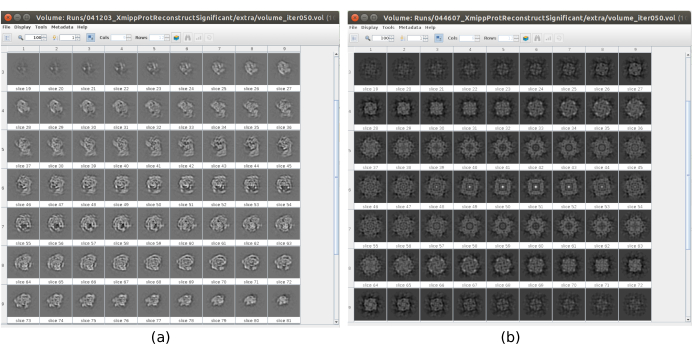
Figura 17. Exemplo de volume inicial 3D com diferentes qualidades. a Bom volume inicial onde a forma da macromolécula pode ser observada. b Volume inicial ruim onde a forma obtida é completamente diferente do esperado. Clique aqui para ver uma versão maior desta figura.
Figura suplementar 1. Criando um projeto Scipion. Janela exibida pelo Scipion onde um projeto antigo pode ser selecionado ou um novo pode ser criado dando um nome e um local para esse projeto. Clique aqui para baixar este Arquivo.
Figura suplementar 2. Importar filmes. Janela exibida pelo Scipion quando pwem - filmes de importação está aberto. Aqui, os principais parâmetros de aquisição devem ser incluídos para deixar os filmes disponíveis para serem processados no Scipion. Clique aqui para baixar este Arquivo.
Figura Suplementar 3. Método de alinhamento de filmes. Janela exibida pelo Scipion quando xmipp3 - alinhamento óptico é usado. Os filmes de entrada, a gama de quadros considerados para alinhamento, e alguns outros parâmetros para processar os filmes devem ser preenchidos. Clique aqui para baixar este Arquivo.
Figura suplementar 4. Método de estimativa ctf com Ctffind. A forma em Scipion com todos os campos necessários para executar o programa Ctffind. Clique aqui para baixar este Arquivo.
Figura suplementar 5. Mago em Scipion. Um assistente para ajudar o usuário a preencher alguns parâmetros no formulário. Neste caso, o assistente deve completar o campo de resolução no método grigoriefflab - ctffind . Clique aqui para baixar este Arquivo.
Figura Suplementar 6. Método de refinamento CTF com Xmipp. A forma de xmipp3 - estimativa ctf com todos os parâmetros para fazer um refinamento de um CTF previamente estimado.
Figura Suplementar 7. Método de micrografia pré-processo. A forma de xmipp3 - micrografos pré-processos que permite a realização de algumas operações sobre eles. Neste exemplo, Remover pixels ruins e micrografos downsample é o útil. Clique aqui para baixar este Arquivo.
Figura Suplementar 8. Método de escolha com Cryolo. O formulário para executar o método de escolha Cryolo usando uma rede pré-treinada. Clique aqui para baixar este Arquivo.
Figura Suplementar 9. Método de escolha de consenso com Xmipp. A forma de xmipp3 - profunda escolha de consenso com base no aprendizado profundo para calcular um consenso de coordenadas, usando uma rede pré-treinada em vários conjuntos de coordenadas obtidas com diferentes métodos de escolha. Clique aqui para baixar este Arquivo.
Figura suplementar 10. Extrair o método de partículas. Guias de entrada e pré-processo de xmipp3 - partículas de extrato. Clique aqui para baixar este Arquivo.
Figura suplementar 11.3D método de volume inicial com Xmipp. A forma do método xmipp3 - reconstruir significativa para obter um mapa 3D inicial. As guias Entrada e Critérios são mostradas. Clique aqui para baixar este Arquivo.
Figura Suplementar 12. Redimensionar o método de volume. A forma de fazer uma colheita ou redimensionar um volume. Neste exemplo, este método é usado para gerar um volume de tamanho total após xmipp3 - reconstruir significativo. Clique aqui para baixar este Arquivo.
Figura suplementar 13.3D volume inicial com resultado de Relion. Uma visão do volume inicial 3D obtido com relion - método de modelo inicial 3D por fatias. Clique aqui para baixar este Arquivo.
Figura suplementar 14. Refinamento do volume inicial com Relion. A forma do método relion - 3D refinar automaticamente. Neste exemplo, foi usado para refinar um volume inicial estimado após consenso. As guias de mapas 3D de entrada e referência são mostradas. Clique aqui para baixar este Arquivo.
Figura suplementar 15.3D método de classificação. Forma de relion - classificação 3D. As guias Entrada, mapa 3D de referência e otimização são mostradas. Clique aqui para baixar este Arquivo.
Figura Suplementar 16.3D alinhamento baseado em um método de aprendizagem profunda. O formulário foi aberto para o método xmipp3 - alinhamento profundo. Aqui é necessário treinar uma rede com um conjunto de treinamento, então essa rede irá prever a atribuição angular por partícula. Clique aqui para baixar este Arquivo.
Figura suplementar 17.3D método de refinamento. Forma do método xmipp3 - highres . Guias Entrada e atribuição angular são mostrados. Clique aqui para baixar este Arquivo.
Figura Suplementar 18. Primeira iteração do resultado de refinamento 3D. a Curva FSC. b O volume obtido (de tamanho menor que a resolução completa) representado como fatias. Clique aqui para baixar este Arquivo.
Figura Suplementar 19. Primeira iteração da análise de correlação de refinamento 3D. Uma nova janela aparece clicando no ícone da barra na parte superior da janela com a lista de partículas. Nas colunas Plot , um histograma do parâmetro estimado desejado pode ser criado. Clique aqui para baixar este Arquivo.
Figura Suplementar 20. Ferramenta de ajuste de validade. Forma de xmipp3 - validar método de overfitting . Clique aqui para baixar este Arquivo.
Discussão
Atualmente, o crio-EM é uma ferramenta-chave para revelar a estrutura 3D de amostras biológicas. Quando bons dados são coletados com o microscópio, as ferramentas de processamento disponíveis nos permitirão obter uma reconstrução 3D da macromolécula em estudo. O processamento de dados Crio-EM é capaz de alcançar uma resolução quase atômica, que é fundamental para entender o comportamento funcional de uma macromolécula e também é crucial na descoberta de drogas.
O Scipion é um software que permite criar todo o fluxo de trabalho combinando os pacotes de processamento de imagem mais relevantes de forma integrativa, o que ajuda a rastreabilidade e reprodutibilidade de todo o fluxo de trabalho de processamento de imagem. O Scipion fornece um conjunto muito completo de ferramentas para realizar o processamento; no entanto, a obtenção de reconstruções de altas resoluções depende completamente da qualidade dos dados adquiridos e de como esses dados são processados.
Para obter uma reconstrução 3D de alta resolução, o primeiro requisito é obter bons filmes do microscópio, que preservam informações estruturais para alta resolução. Se este não for o caso, o fluxo de trabalho não será capaz de extrair informações de alta definição dos dados. Em seguida, um fluxo de trabalho de processamento bem-sucedido deve ser capaz de extrair partículas que realmente correspondem à estrutura e encontrar as orientações dessas partículas no espaço 3D. Se alguma das etapas do fluxo de trabalho falhar, a qualidade do volume reconstruído será degradada. O Scipion permite o uso de diferentes pacotes em qualquer uma das etapas de processamento, o que ajuda a encontrar a abordagem mais adequada para processar os dados. Além disso, graças à disponibilização de muitos pacotes, podem ser utilizadas ferramentas de consenso que aumentam a precisão ao encontrar um acordo nas saídas estimadas de diferentes métodos. Além disso, tem sido discutido detalhadamente na seção Resultados Representativos várias ferramentas de validação e como identificar resultados precisos e imprecisos em cada etapa do fluxo de trabalho, para detectar possíveis problemas e como tentar resolvê-los. Existem vários pontos de verificação ao longo do protocolo que podem ajudar a perceber se o protocolo está funcionando corretamente ou não. Alguns dos mais relevantes são: escolha, classificação 2D, estimativa inicial de volume e alinhamento 3D. Verificar as entradas, repetir a etapa com um método diferente ou usar consenso, são opções disponíveis no Scipion que o usuário pode usar para encontrar soluções quando os problemas aparecem.
Em relação às abordagens anteriores para a integração de pacotes no campo Cryo-EM, o Appion31 é o único que permite a integração real de diferentes pacotes de software. No entanto, a Appion está fortemente conectada com leginon32, um sistema para coleta automatizada de imagens a partir de microscópios eletrônicos. A principal diferença com o Scipion é que o modelo de dados e o armazenamento são menos acoplados. Dessa forma, para criar um novo protocolo no Scipion, apenas um script Python precisa ser desenvolvido. No entanto, no Appion, o desenvolvedor deve escrever o script e alterar o banco de dados subjacente. Em resumo, o Scipion foi desenvolvido para simplificar a manutenção e a extensibilidade.
Apresentamos neste manuscrito um fluxo de trabalho completo para o processamento cryo-EM, utilizando o conjunto de dados de caso real do Plasmodium falciparum 80S Ribosome (entrada EMPIAR: 10028, entrada EMDB: 2660). As etapas aqui abordadas podem ser resumidas como alinhamento de filme, estimativa ctf, captação de partículas, classificação 2D, estimativa inicial do mapa, classificação 3D, refinamento 3D, avaliação e pós-processamento. Diferentes pacotes foram utilizados e ferramentas de consenso foram aplicadas em várias dessas etapas. O volume reconstruído 3D final alcançou uma resolução de 3 Å e, no volume pós-processado, algumas estruturas secundárias podem ser distinguidas, como as helices alfa, o que ajuda a descrever como os átomos são dispostos no espaço.
O fluxo de trabalho apresentado neste manuscrito mostra como o Scipion pode ser usado para combinar diferentes pacotes Cryo-EM de forma simples e integrativa para simplificar o processamento e obter resultados mais confiáveis ao mesmo tempo.
No futuro, o desenvolvimento de novos métodos e pacotes continuará crescendo e softwares como o Scipion para integrar facilmente todos eles serão ainda mais importantes para os pesquisadores. Abordagens de consenso serão mais relevantes mesmo assim, quando muitos métodos com bases diferentes estarão disponíveis, ajudando a obter estimativas mais precisas de todos os parâmetros envolvidos no processo de reconstrução no Cryo-EM. O rastreamento e a reprodutibilidade são fundamentais no processo de pesquisa e mais fáceis de alcançar com o Scipion, graças a ter uma estrutura comum para a execução de fluxos de trabalho completos.
Divulgações
Os autores não têm nada a revelar.
Agradecimentos
Os autores gostariam de reconhecer o apoio econômico de: Ministério espanhol da Ciência e Inovação através de Bolsas: PID2019-104757RB-I00/AEI/10.13039/501100011033, o "Comunidad Autónoma de Madrid" através de Grant: S2017/BMD-3817, Instituto de Salud Carlos III, PT17/0009/0010 (ISCIII-SGEFI/ERDF), União Europeia (UE) e Horizon 2020 através de subvenção: INSTRUIR - ULTRA (INFRADEV-03-2016-2017, Proposta: 731005), EOSC Life (INFRAEOSC-04-2018, Proposta: 824087), iNEXT - Discovery (Proposta: 871037) e HighResCells (ERC - 2018 - SyG, Proposta: 810057). O projeto que deu origem a esses resultados recebeu o apoio de uma bolsa da Fundação "la Caixa" (ID 100010434). O código de companheirismo é LCF/BQ/DI18/11660021. Este projeto recebeu financiamento do programa de pesquisa e inovação Horizon 2020 da União Europeia no âmbito do acordo de subvenção Marie Skłodowska-Curie nº 713673. Os autores reconhecem o apoio e o uso dos recursos do Instruct, um projeto da Landmark ESFRI.
Materiais
| Name | Company | Catalog Number | Comments |
| no material is used in this article | - | - | - |
Referências
- Nogales, E. The development of cryo-EM into a mainstream structural biology technique. Nature Methods. 13 (1), 24-27 (2016).
- Kühlbrandt, W. The Resolution Revolution. Science. 343 (6178), 1443-1444 (2014).
- . 1.15 A structure of human apoferritin obtained from Titan Mono- BCOR microscope Available from: https://www.rcsb.org/structure/7A6A (2021)
- Arnold, S. A., et al. Miniaturizing EM Sample Preparation: Opportunities, Challenges, and "Visual Proteomics". PROTEOMICS. 18 (5-6), 1700176 (2018).
- Faruqi, A. R., McMullan, G. Direct imaging detectors for electron microscopy. Nuclear Instruments and Methods in Physics Research Section A: Accelerators, Spectrometers, Detectors and Associated Equipment. 878, 180-190 (2018).
- Vilas, J. L., et al. Advances in image processing for single-particle analysis by electron cryomicroscopy and challenges ahead. Current Opinion in Structural Biology. 52, 127-145 (2018).
- Martinez, M., et al. Integration of Cryo-EM Model Building Software in Scipion. Journal of Chemical Information and Modeling. 60, 2533-2540 (2020).
- de la Rosa-Trevín, J. M., et al. Scipion: A software framework toward integration, reproducibility and validation in 3D electron microscopy. Journal of Structural Biology. 195, 93-99 (2016).
- de la Rosa-Trevín, J. M., et al. Xmipp 3.0: an improved software suite for image processing in electron microscopy. Journal of Structural Biology. 184, 321-328 (2013).
- Scheres, S. H. W. . Methods in Enzymology. The Resolution Revolution: Recent Advances In cryoEM. , 125-157 (2016).
- Punjani, A., Rubinstein, J. L., Fleet, D. J., Brubaker, M. A. cryoSPARC: algorithms for rapid unsupervised cryo-EM structure determination. Nature Methods. 14, 290-296 (2017).
- Ludtke, S. J. 3-D structures of macromolecules using single-particle analysis in EMAN. Methods in Molecular Biology. 673, 157-173 (2010).
- Shaikh, T. R., et al. SPIDER image processing for single-particle reconstruction of biological macromolecules from electron micrographs. Nature Protocols. 3, 1941-1974 (2008).
- Wagner, T., et al. SPHIRE-crYOLO is a fast and accurate fully automated particle picker for cryo-EM. Communications Biology. 2, (2019).
- Mindell, J. A., Grigorieff, N. Accurate determination of local defocus and specimen tilt in electron microscopy. Journal of Structural Biology. 142, 334-347 (2003).
- Winn, M. D., et al. Overview of the CCP4 suite and current developments. Acta crystallographica. Section D, Biological crystallography. 67, 235-242 (2011).
- Liebschner, D., et al. Macromolecular structure determination using X-rays, neutrons and electrons: recent developments in Phenix. Acta Crystallographica Section D. 75, 861-887 (2019).
- Wong, W., et al. Cryo-EM structure of the Plasmodium falciparum 80S ribosome bound to the anti-protozoan drug emetine. eLife. 3, 03080 (2014).
- Abrishami, V., et al. Alignment of direct detection device micrographs using a robust Optical Flow approach. Journal of Structural Biology. 189, 163-176 (2015).
- Sorzano, C. O. S., Jonic, S., Nunez Ramirez, R., Boisset, N., Carazo, J. M. Fast, robust and accurate determination of transmission electron microscopy contrast transfer function. Journal of Structural Biology. 160, 249-262 (2007).
- Abrishami, V., et al. A pattern matching approach to the automatic selection of particles from low-contrast electron micrographs. Bioinformatics. 29, 2460-2468 (2013).
- Sanchez-Garcia, R., Segura, J., Maluenda, D., Carazo, J. M., Sorzano, C. O. S. Deep Consensus, a deep learning-based approach for particle pruning in cryo-electron microscopy. IUCrJ. 5, 854-865 (2018).
- Sorzano, C. O. S., et al. A clustering approach to multireference alignment of single-particle projections in electron microscopy. Journal of Structural Biology. 171, 197-206 (2010).
- Sorzano, C. O. S., et al. A statistical approach to the initial volume problem in Single Particle Analysis by Electron Microscopy. Journal of Structural Biology. 189, 213-219 (2015).
- Sorzano, C. O. S., et al. Swarm optimization as a consensus technique for Electron Microscopy Initial Volume. Applied Analysis and Optimization. 2, 299-313 (2018).
- Pettersen, E. F., et al. UCSF Chimera--a visualization system for exploratory research and analysis. Journal of computational chemistry. 25, 1605-1612 (2004).
- Sorzano, C. O. S., et al. A new algorithm for high-resolution reconstruction of single particles by electron microscopy. Journal of Structural Biology. 204, 329-337 (2018).
- Vilas, J. L., et al. MonoRes: Automatic and Accurate Estimation of Local Resolution for Electron Microscopy Maps. Structure. 26, 337-344 (2018).
- Ramirez-Aportela, E., et al. Automatic local resolution-based sharpening of cryo-EM maps. Bioinformatics. 36, 765-772 (2020).
- Heymann, J. B. Validation of 3D EM Reconstructions: The Phantom in the Noise. AIMS Biophys. 2, 21-35 (2015).
- Lander, G. C., et al. Appion: An integrated, database-drive pipeline to facilitate EM image processing. Journal of Structural Biology. 166, 95-102 (2009).
- Suloway, C., et al. Automated molecular microscopy: The new Leginon system. Journal of Structural Biology. 151, 41-60 (2005).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados