Method Article
Direkte Agroinokulation von Maissämlingen durch Injektion mit rekombinanten Foxtail Mosaic Virus und Sugarcane Mosaic Virus Infectious Clones
* Diese Autoren haben gleichermaßen beigetragen
In diesem Artikel
Zusammenfassung
Ein Agrobacterium-basiertes Injektionsprotokoll (Agroinjektion) wird für die Impfung von Fuchsschwanzmosaikvirus und Zuckerrohrmosaikvirusclone in Maissämlinge vorgestellt. Die Impfung auf diese Weise führt zu einer Virusinfektion, einem virusinduzierten Gen-Silencing von Markergenen und einer viralen Überexpression von GFP.
Zusammenfassung
Agrobakterium-basierte Impfansätze werden häufig verwendet, um virale Vektoren in Pflanzengewebe einzuführen. Diese Studie beschreibt ein Protokoll für die Injektion von Maissämlingen in der Nähe von meristematischem Gewebe mit Agrobacterium , das einen viralen Vektor trägt. Rekombinante Foxtail Mosaic Virus (FoMV) Klone, die für Gen-Silencing und Genexpression entwickelt wurden, wurden verwendet, um diese Methode zu optimieren, und ihre Verwendung wurde um ein rekombinantes Zuckerrohrmosaikvirus (SCMV) erweitert, das für die Genexpression entwickelt wurde. Genfragmente oder kodierende Sequenzen von Interesse werden in ein modifiziertes, infektiöses virales Genom eingefügt, das in den binären T-DNA-Plasmidvektor pCAMBIA1380 kloniert wurde. Die resultierenden Plasmidkonstrukte werden in Agrobacterium tumefaciens Stamm GV3101 umgewandelt. Maissämlinge, die erst 4 Tage alt sind, können in der Nähe des Coleoptilar-Knotens mit Bakterien injiziert werden, die in MgSO4-Lösung resuspendiert sind. Während der Infektion mit Agrobacterium wird die T-DNA, die das virale Genom trägt, auf Maiszellen übertragen, was die Transkription des viralen RNA-Genoms ermöglicht. Da sich das rekombinante Virus repliziert und systemisch in der Pflanze ausbreitet, können virale Symptome und phänotypische Veränderungen, die sich aus der Stummschaltung der Zielgene ergeben , läsionsimmiert 22 (les22) oder Phytoen-Desaturase (pds) auf den Blättern beobachtet werden, oder die Expression von grün fluoreszierendem Protein (GFP) kann bei Beleuchtung mit UV-Licht oder Fluoreszenzmikroskopie nachgewiesen werden. Um das Virus nachzuweisen und gleichzeitig die Integrität des Inserts zu beurteilen, wird RNA aus den Blättern der injizierten Pflanze extrahiert und die RT-PCR unter Verwendung von Primern durchgeführt, die die multiple Klonierungsstelle (MCS) flankieren, die die eingefügte Sequenz trägt. Dieses Protokoll wurde in mehreren Maisgenotypen effektiv eingesetzt und kann leicht auf andere virale Vektoren erweitert werden, wodurch ein zugängliches Werkzeug für die Einführung viraler Vektoren in Mais geboten wird.
Einleitung
Infektiöse Klone vieler Pflanzenviren wurden für virusinduziertes Gen-Silencing (VIGS), Gen-Overexpression (VOX) und zuletzt virusfähiges Gen-Editing (VEdGE) entwickelt 1,2,3,4,5,6,7,8,9,10,11 . Mit der Entwicklung neuer viraler Konstrukte müssen auch Methoden in Betracht gezogen werden, um Pflanzengewebe erfolgreich mit diesen modifizierten Viren zu infizieren. Aktuelle Methoden zur Einleitung von Virusinfektionen in Pflanzen umfassen Partikelbeschuss, Reibungsimpfung von In-vitro-RNA-Transkripten oder DNA-Klonen, vaskuläre Punktionsimpfung oder Agrobacterium tumefaciens-Impfung (Agroinokulation)5,12,13,14,15,16,17 . Jede dieser Impfmethoden hat inhärente Vor- und Nachteile, zu denen Kosten, der Bedarf an Spezialausrüstung und die Machbarkeit innerhalb eines bestimmten Pflanzenvirussystems gehören. Methoden, die die Infiltration oder Injektion von Agrobacterium-Stämmen verwenden, die binäre T-DNA-Konstrukte enthalten, die rekombinante Viren liefern sollen, werden bevorzugt, da sie einfach und kostengünstig sind. Detaillierte Agroinokulationsmethoden für monokotyle Arten wie Zea mays (Mais) fehlen jedoch.
Einer der ersten Berichte über die Agroinokulation zur Virusabgabe wurde 1986 veröffentlicht, als das Genom des Blumenkohlmosaikvirus (CaMV) in ein T-DNA-Konstrukt eingefügt wurde und das resultierende Agrobakterium, das dieses Konstrukt trug, auf Rübenpflanzen gerieben wurde18. Seitdem wurden weitere Methoden zur Agroinokulation entwickelt. Im Falle des Fuchsschwanzmosaikvirus (FoMV) kann Nicotiana benthamiana beispielsweise als Zwischenwirt verwendet werden, um Viruspartikel in Blättern zu erzeugen, die eine Inokulumquelle bilden6. Die Reibung von Mais mit infizierten N. benthamiana-Blättern ist effizient, schnell und einfach, aber die Verwendung eines Zwischenwirts funktioniert nicht bei allen maisinfizierenden Viren. Das Zuckerrohrmosaikvirus (SCMV) zum Beispiel kann N. benthamiana nicht infizieren, was die Verwendung alternativer Inokulumquellen für Vektoren erfordert, die von diesem Virus abgeleitet sind. Im Jahr 1988 wurde Agrobacterium, das das Maisstreifenvirus (MSV), ein DNA-Virus, enthält, durch Injektion (Agroinjektion) in Maissämlinge eingeführt, was zeigt, dass Agrobacterium-basierte Impfmethoden auch für Monokotyledonen nützlich sind19. Trotz dieses frühen Erfolgs mit der Agroinjektion wurden nur wenige Studien veröffentlicht, die diese Technik bei Mais anwenden, was Fragen zur Anwendbarkeit dieser Methode auf RNA-Viren und VIGS-, VOX- und VEdGE-Vektoren offen lässt20,21,22. Die breite Anwendung der Agroinjektion bei Monokotyledonenarten ist jedoch vielversprechend, da dieser allgemeine Ansatz in Orchidee, Reis und Weizen verwendet wurde23,24,25,26,27,28.
Dieses Protokoll wurde für FoMV und Agrobacterium Stamm GV3101 optimiert und wurde auch auf einen SCMV-Vektor angewendet. FoMV ist ein Potexvirus mit einem breiten Wirtsbereich, der 56 Monokotyledonen- und Dikotyledonenarten umfasst29. FoMV hat ein 6,2 Kilobase (kb) positives, einzelsträngiges RNA-Genom, das fünf verschiedene Proteine aus fünf offenen Leserahmen (ORFs) kodiert)30,31,32,33,34,35. FoMV wurde zuvor sowohl als VIGS- als auch als VOX-Vektor für Mais entwickelt, indem ein infektiöser Klon auf ein T-DNA-Plasmid-Rückgrat eingebaut wurde6,36,37. Das virale Genom wurde für VIGS-Anwendungen modifiziert, indem eine Klonierungsstelle (MCS1*) unmittelbar nach dem Mantelprotein (CP) hinzugefügt wurde (Abbildung 1A)36. Für VOX- und VEdGE-Anwendungen wurde der CP-Promotor dupliziert und eine zweite Klonierungsstelle (MCS2) hinzugefügt, um das Einfügen von Sequenzen von Interesse zwischen ORF 4 und cp zu ermöglichen (Abbildung 1B)6. Der FoMV-Vektor, der sowohl MCS1 als auch MCS2 ohne Inserts enthält, ist der FoMV-Leervektor (FoMV-EV) (Abbildung 1).
SCMV ist ein nicht verwandtes Virus, das für VOX in Mais38 entwickelt wurde. Es ist ein Mitglied der Potyviridae-Familie, von denen mehrere Mitglieder so konstruiert wurden, dass sie Fremdproteine in Pflanzen exprimieren39,40,41,42,43,44. Das Wirtsspektrum von SCMV umfasst Mais, Sorghum und Zuckerrohr45,46, was es für Genfunktionsstudien in diesen wichtigen Kulturpflanzen wertvoll macht36,38. SCMV hat ein positives, einzelsträngiges RNA-Genom von etwa 10 kb Länge47,48. Zur Erstellung des SCMV-VOX-Vektors wurde der etablierte P1/HCPro-Übergang als Insertionsstelle für heterologe Sequenzen verwendet38. Auf diese Klonierungsstelle folgt eine Sequenz, die für eine NIa-Protease-Spaltungsstelle kodiert, was zur Produktion von Proteinen unabhängig vom SCMV-Polyprotein führt (Abbildung 1C).
T-DNA-Plasmide, die infektiöse cDNA dieser rekombinanten Viren tragen, wurden in den Agrobacterium-Stamm GV3101 umgewandelt. GV3101 ist ein Noalin-Stamm, von dem bekannt ist, dass er T-DNA auf monokotyle Spezies, einschließlich Mais, übertragen kann26,28,49. Darüber hinaus wurden in früheren Agroinjektionsstudien die Stämme C58 oder sein Derivat GV3101 sowie 19,20,22,27 verwendet.
Bei der Entwicklung dieses Protokolls wurden drei Markergene verwendet: zwei für das Gen-Silencing und eines für die Genexpression. Ein 329 Basenpaar (bp) Fragment aus der Maisgenläsion Mimic 22 (les22, GRMZM2G044074) wurde verwendet, um den Stummschaltungsvektor FoMV-LES22 zu konstruieren. Wenn les22 in Mais zum Schweigen gebracht wird, erscheinen kleine, runde Flecken nekrotischer Zellen entlang des Gefäßsystems von Blättern, die sich ausdehnen und zu großen Bereichen nekrotischen Blattgewebes verschmelzen50. FoMV-PDS, das ein 313 bp-Fragment aus dem Sorghum-Gen Phytoen-Desaturase (pds, LOC110436156, 96% Sequenzidentität zu Mais pds, GRMZM2G410515) enthält, induziert das Stummschalten von PDS in Mais, was zu kleinen Streifen von photogebleichten Zellen entlang des Gefäßsystems der Blätter führt, die sich im Laufe der Zeit verlängern51. Die intakte kodierende Sequenz für grün fluoreszierendes Protein (GFP) wurde verwendet, um die Proteinexpression sowohl für FoMV (FoMV-GFP) als auch für SCMV (SCMV-GFP) nachzuweisen. Die GFP-Expression in den Blättern ist in der Regel 14 Tage nach der Impfung (DPI) am nachweisbarsten 6. Obwohl es frühere Studien gab, in denen die Agroinjektion viraler Vektoren in Mais verwendet wurde, haben diese Experimente nur gezeigt, dass die Agroinjektion die Virusinfektion durch einen infektiösen Klon in Maissämlingen erleichtern und sich nicht auf rekombinante Viren ausdehnen kann, die für VIGS- oder VOX-Anwendungen entwickelt wurden19,20,21,22. Das hier vorgestellte Protokoll baut auf früheren Agroinjektionsmethoden auf, insbesondere grismley et al.19. Insgesamt ist diese Agroinjektionsmethode mit VIGS- und VOX-Vektoren kompatibel, erfordert keine spezielle Ausrüstung oder alternative Wirte als Inokulumquellen und verringert die Gesamtzeit und -kosten für die Einrichtung und Durchführung von Impfungen im Vergleich zu anderen gängigen Methoden, die Biolistik oder In-vitro-Transkription erfordern. Dieses Protokoll wird funktionelle Genomikstudien in Mais mit Anwendungen wie VIGS, VOX und VEdGE erleichtern.
Protokoll
1. Plasmidkonstruktion
HINWEIS: Dieses Protokoll kann auf andere virale Vektoren oder Agrobacterium-Stämme angewendet werden, dies kann jedoch den Gesamterfolg der Impfung durch Agroinjektion beeinflussen. Führen Sie immer bakterielle Impf- und Beschichtungsschritte in einer Laminar-Flow-Haube durch.
- FoMV-Stummschaltungskonstrukt
HINWEIS: Luria-Bertani (LB) Medien (Miller) werden für alle Medien verwendet, sofern nicht anders angegeben. Flüssiges LB wird hergestellt, indem 25 g Granulat in 1.000 ml destilliertes Wasser suspendiert und 15 minuten bei 121 ° C autoklaviert werden. Feste LB-Medien werden in ähnlicher Weise unter Zugabe von 1,5% Agar vor dem Autoklavieren hergestellt. Antibiotika werden zugegeben, nachdem LB auf ~ 60 ° C abgekühlt ist, und die Lösung wird in 95 x 15 mm Petriplatten gegossen. Die zu verwendenden Antibiotikakonzentrationen sind wie folgt: Rifampicin (rif) bei 25 μg / ml, Gentamycin (gent) bei 50 μg / ml und Kanamycin (kan) bei 50 μg / ml.- PCR amplifizieren Fragmente aus dem zum Schweigen zu bringenden Maisgen (z. B. les22 oder pds) mit einem Vorwärtsprimer mit einer PacI-Restriktionsstelle und einem Reverse-Primer mit einer XbaI-Restriktionsstelle. Dies ermöglicht die Ligation der Genfragmente in den MCS1* des FoMV-pCAMBIA1380-Binärvektors in der Antisense-Orientierung.
HINWEIS: Richten Sie die PCR mit einer High-Fidelity-DNA-Polymerase, Vorwärts- und Rückwärtsprimern bei jeweils 10 μM, Plasmid-DNA-Vorlage und Wasser gemäß den DNA-Polymerase-Spezifikationen ein. Amplifizieren Sie für 35 Zyklen unter Verwendung einer Glühtemperatur entsprechend der DNA-Polymerase- und Primerschmelztemperatur (Tm) und einer zu amplifizierenden Erweiterung von 30 s pro Kilobase. - Führen Sie die PCR-Reinigung mit einem PCR-Reinigungskit gemäß den Kit-Spezifikationen durch.
- Verdauen Sie das gereinigte PCR-Produkt und das FoMV-EV mit den Restriktionsenzymen XbaI und PacI. Verwenden Sie 1 μg Plasmid oder das gesamte gereinigte PCR-Produkt, 2 μL 10x Puffer, 1 μL Restriktionsenzym und fügen Sie Wasser hinzu, um ein endgültiges Reaktionsvolumen von 20 μL zu erhalten. Inkubieren Sie nach Enzymspezifikation.
- Ligatieren Sie das verdaute PCR-Produkt und FoMV-EV zusammen mit der T4-DNA-Ligase gemäß dem Protokoll des Herstellers.
- Wandeln Sie das ligierte Plasmid mit der Hitzeschockmethode in chemisch kompetente DH5α-E. coli-Zellen um.
- Zellen auf Eis auftauen und 3 μL Plasmid in die Röhre geben. 30 min auf Eis inkubieren, dann 30 s bei 42 °C hitzeschockieren.
- Für 5 min auf Eis legen, 200 μL superoptimale Brühe mit kataboler Repression (SOC) hinzufügen und E. coli-Zellen 1 h bei 37 °C mit Schütteln bei 225 U/min in SOC-Medien erholen lassen.
- Auf Kanamycin selektiven LB-Medien auftragen und über Nacht bei 37 °C inkubieren.
- Überprüfen Sie die Kolonien auf genaue Klone durch Sanger-Sequenzierung mit den Primern FM-5840F und FM-6138R (ergänzende Tabelle 1). Reichen Sie 250 ng Plasmid-DNA an eine Einrichtung ein, die die Sanger-Sequenzierung durchführt. Für dieses Experiment wurden Proben an die IOWA State University DNA Core Facility geschickt.
- Impfen Sie 2 ml flüssige LB mit der gewählten Kolonie und inkubieren Sie bei 37 ° C über Nacht unter Schütteln bei 225 U / min. Extrahieren Sie Plasmid-DNA aus der Nachtkultur durch ein alkalisches Lyseplasmid-DNA-Präparat52.
- Wandeln Sie Plasmid-DNA mit der Freeze-Thaw-Methode in Zellen des Agrobacterium-Stammes GV3101 um. Lassen Sie 100 μL chemisch kompetente Zellen auf Eis auftauen, fügen Sie 1-5 μL Plasmid hinzu und inkubieren Sie 30 minuten lang auf Eis. 1 min in flüssigen Stickstoff geben und dann 3 min bei 37 °C inkubieren. Fügen Sie 1 ml SOC hinzu, lassen Sie sich für 2-3 h bei 28 ° C mit Schütteln, Platten auf Rif, Gent und Kan selektiven LB-Medien erholen und inkubieren Sie bei 28 ° C für 2 Tage.
- Screen-Kolonien auf das Vorhandensein von Insert mit Kolonie-PCR. Wählen Sie eine einzelne Bakterienkolonie und mischen Sie sie in 30 μL Wasser. Richten Sie eine PCR-Reaktion ein, indem Sie 12,5 μL Polymerase-Master-Mix, 1,25 μL von je 10 μM Primer, FM-5840F und FM-6138R, 3 μL der Bakterienkoloniesuspension und Wasser zu einem Endvolumen von 25 μL hinzufügen. Zyklus 35 mal mit einer Glühtemperatur von 64 °C und einer Verlängerungszeit von 1 min (1 min für jeden kbit amplifizierten KB).
- Impfen Sie 2-5 ml flüssiges LB (rif, gent, kan) mit der richtigen Agrobacterium-Kolonie . Über Nacht bei 28 °C wachsen lassen und bei 225 U/min schütteln.
- Mischen Sie die Übernachtungskultur mit einer 50% igen Glycerinlösung 1:1. Für eine Langzeitlagerung bei -80 °C lagern.
- PCR amplifizieren Fragmente aus dem zum Schweigen zu bringenden Maisgen (z. B. les22 oder pds) mit einem Vorwärtsprimer mit einer PacI-Restriktionsstelle und einem Reverse-Primer mit einer XbaI-Restriktionsstelle. Dies ermöglicht die Ligation der Genfragmente in den MCS1* des FoMV-pCAMBIA1380-Binärvektors in der Antisense-Orientierung.
- FoMV-Ausdruckskonstrukt
- Die PCR verstärkt die Kodierungssequenz von Interesse, einschließlich Start- und Stoppcodons (z. B. GFP), wie in 1.1.1 beschrieben, und fügt eine Bsu36I-Restriktionsstelle auf dem Vorwärtsprimer und eine PspOMI-Restriktionsstelle auf der Umgekehrten Grundierung hinzu, um ein gerichtetes Klonen in der Sinnesorientierung in MCS2 zu ermöglichen.
- Führen Sie die PCR-Reinigung mit einem PCR-Reinigungskit gemäß den Spezifikationen des Kits durch.
- Verdauung des PCR-Produkts und des FoMV-EV mit den Restriktionsenzymen Bsu36I und PspOMI, wie in 1.1.3 beschrieben.
- Ligatieren Sie das verdaute PCR-Produkt und FoMV-EV zusammen mit der T4-DNA-Ligase gemäß dem Protokoll des Herstellers.
- Umwandlung in chemisch kompetente E. coli-Zellen dh5α unter Verwendung der Hitzeschockmethode gemäß 1.1.5. Auf Kanamycin selektiven LB-Medien auftragen und über Nacht bei 37 °C inkubieren.
- Überprüfen Sie die Kolonien auf genaue Klone durch Sanger-Sequenzierung wie in 1.1.6 beschrieben mit den Primern 5AmuS2 und 5AmuA2 (Ergänzende Tabelle 1).
- Impfen Sie 2 ml flüssiges LB mit der gewählten Kolonie und inkubieren Sie bei 37 ° C über Nacht unter Schütteln bei 225 U / min. Extrahieren Sie Plasmid-DNA aus der Nachtkultur durch ein alkalisches Lyseplasmid-DNA-Präparat52.
- Umwandlung von Plasmid-DNA in chemisch kompetente Zellen des Agrobacterium-Stammes GV3101 unter Verwendung der Freeze-Thaw-Methode gemäß 1.1.8. Platte auf rif, gent und kan selektiven LB-Medien und inkubieren bei 28 ° C für 2 Tage.
- Screen-Kolonien auf das Vorhandensein von Insert mit Kolonie-PCR unter Verwendung der Primer 5AmuS2 und 5AmuA2.
- Impfen Sie 2-5 ml flüssige LB (rif, gent, kan) mit der richtigen Agrobacterium-Kolonie . Über Nacht bei 225 U/min bei 28 °C schütteln.
- Mischen Sie die Übernachtungskultur mit einer 50% igen Glycerinlösung 1:1. Für eine Langzeitlagerung bei -80 °C lagern.
- SCMV-Ausdruckskonstrukt
- PCR amplifizieren das interessierende Gen (z. B. GFP) mit Ausnahme des Stopcodons wie in 1.1.1 beschrieben, einschließlich einer PspOMI-Aufschlussstelle auf dem Vorwärtsprimer und einer SbfI-Aufschlussstelle auf der Umgekehrten Grundierung, um ein gerichtetes Klonen in den binären SCMV-pCAMBIA1380-Vektor zu ermöglichen.
HINWEIS: Insert muss im Rahmen mit dem viralen Polyprotein kloniert werden. - Führen Sie die PCR-Reinigung mit einem PCR-Reinigungskit gemäß den Spezifikationen des Kits durch.
- Verdauung des PCR-Produkts und des SCMV-EV mit den Restriktionsenzymen PspOMI und SbfI, wie in 1.1.3 beschrieben.
- Ligatieren Sie das verdaute PCR-Produkt und SCMV-EV zusammen mit der T4-DNA-Ligase gemäß dem Protokoll des Herstellers.
- Das Produkt wird unter Verwendung der hitzeschockartigen Methode gemäß 1.1.5 in chemisch kompetente DH5α-E. coli-Zellen umgewandelt. Auf kan selektiven LB-Medien plattieren und über Nacht bei 37 °C inkubieren.
- Screening von Kolonien auf genaue Klone durch Sanger-Sequenzierung wie in 1.1.6 beschrieben unter Verwendung der Primer SC-745F und HCProR1 (Ergänzende Tabelle 1).
- Impfen Sie 2 ml flüssige LB mit der gewählten Kolonie und inkubieren Sie bei 37 ° C über Nacht unter Schütteln bei 225 U / min. Extrahieren Sie die Plasmid-DNA aus der Nachtkultur durch eine alkalische Lyseplasmid-DNA-Präparation52.
- Umwandlung der Plasmid-DNA in chemisch kompetente Zellen des Agrobacterium-Stammes GV3101 unter Verwendung der Freeze-Thaw-Methode wie in 1.1.8 beschrieben. Platte auf rif, gent und kan selektiven LB-Medien und inkubieren bei 28 ° C für 2 Tage.
- Screening-Kolonien auf Vorhandensein von Insert mit Kolonie-PCR mit den Primern SC-745F und HCProR1, wie in 1.1.9 beschrieben.
- Impfen Sie 2-5 ml flüssiges LB (rif, gent, kan) mit der richtigen Agrobacterium-Kolonie . Über Nacht bei 225 U/min bei 28 °C schütteln.
- Mischen Sie die Übernachtungskultur mit einer 50% igen Glycerinlösung 1:1. Für eine Langzeitlagerung bei -80 °C lagern.
- PCR amplifizieren das interessierende Gen (z. B. GFP) mit Ausnahme des Stopcodons wie in 1.1.1 beschrieben, einschließlich einer PspOMI-Aufschlussstelle auf dem Vorwärtsprimer und einer SbfI-Aufschlussstelle auf der Umgekehrten Grundierung, um ein gerichtetes Klonen in den binären SCMV-pCAMBIA1380-Vektor zu ermöglichen.
2. Zubereitung des Sämlings
- Pflanzen Sie 1-2 Maissamen ("Golden Bantam" Zuckermais, FR1064, B73 usw.) in torfbasiertem Wachstumsmedium in kleinen Einsätzen, die 4-7 Tage vor der Injektion in Schalen platziert werden. Platz in einer Wachstumskammer unter 16 h Tagen bei 25 °C und 8 h Nächten bei 22 °C (~185 photosynthetisch aktive Strahlung (PAR)) oder in einem Gewächshaus unter 16 h Tagen bei 22-25 °C und 8 h Nächten bei 22-25 °C (350-400 PAR).
HINWEIS: Die Anfälligkeit für Agrobacterium variiert zwischen den Maisgenotypen, was sich auf die Erfolgsraten auswirkt. Darüber hinaus können einige virale Vektoren mit bestimmten Maisgenotypen unvereinbar sein. - Regelmäßig gießen und einmal pro Woche mit 15-5-15 Flüssigdünger bei 330 Teilen pro Million (PPM) düngen.
3. Herstellung von Agrobacterium
- Bereiten Sie einen Tag vor der Injektion LB flüssige Medien mit dem entsprechenden Antibiotikum (rif, gent, kan) vor und impfen Sie mit dem Agrobacterium-Stamm , der das gewünschte virale Konstrukt trägt. Es wird empfohlen, 20 μL Glycerinvorrat in 50 ml LB zu geben, was genügend Bakterienkultur ergeben sollte, um >100 Pflanzen zu impfen und nach Bedarf nach oben oder unten skaliert werden kann.
HINWEIS: Bereiten Sie genügend Inokulum vor, um eine endgültige Menge an Bakteriensuspension von mindestens 1 ml bei einer optischen Dichte von 600 nm (OD600) von 1,0 pro 4-5 Pflanzen zu erhalten. - 24 h bei 225 U/min bei 28 °C schütteln.
- Pelletbakterien für 10 min bei 4.000 x g bei Raumtemperatur. Verwerfen Sie den Überstand.
- Waschen Sie das Pellet gründlich mit 1 ml entionisiertem (DI) Wasser durch Pipettieren oder sanftes Wirbeln.
- Wiederholen Sie Schritt 3.3, um Bakterien zu pelletieren.
- Resuspendieren Sie das Pellet in 1 mL 10 mM MgSO4-Lösung durch Pipettieren oder sanftes Wirbeln.
- Optional werden der Lösung 200 μM Acetosyringon zugesetzt. Obwohl häufig verwendet, verbessert Acetosyringon nur die Transformationsfähigkeit einiger Agrobacterium-Stämme . Die Autoren haben nicht festgestellt, dass die Zugabe von Acetosyringon die Effizienz in diesem Protokoll beeinflusst (Ergänzende Tabelle 2).
HINWEIS: 10 mM MgSO4-Lösung kann aus einer 1 M Stammlösung mit einem pH-Wert von 6,3 hergestellt werden, die bei Raumtemperatur gelagert wird. Die Lösung erfordert wahrscheinlich keine pH-Einstellung.
- Optional werden der Lösung 200 μM Acetosyringon zugesetzt. Obwohl häufig verwendet, verbessert Acetosyringon nur die Transformationsfähigkeit einiger Agrobacterium-Stämme . Die Autoren haben nicht festgestellt, dass die Zugabe von Acetosyringon die Effizienz in diesem Protokoll beeinflusst (Ergänzende Tabelle 2).
- Od600 der Probe mit einem Spektralphotometer messen und mit 10 mM MgSO4-Lösung auf 1,0 OD600 verdünnen.
HINWEIS: Dies ist ein sicherer Haltepunkt. Die Bakteriensuspension kann vor der Injektion bis zu 5 h bei Raumtemperatur aufbewahrt werden.
4. Injektion
HINWEIS: Maissämlinge im Alter von 4-7 Tagen können zur Injektion verwendet werden. Die Wachstumsrate der Sämlinge wird stark von den Wachstumsbedingungen, der Par-Menge (dh einem höheren PAR im Gewächshaus als in der Wachstumskammer) und dem Genotyp beeinflusst, unter anderem, der unter Gewächshausbedingungen schwer zu kontrollieren sein kann. Pflanzen können so jung wie 4 Tage alt injiziert werden, wenn sie 2-3 cm groß sind, ohne Blätter erweitert und so alt wie 7 Tage, wenn das unterste runde Blatt erweitert wird. Die Erfolgsrate dieser Impfmethoden sinkt rapide, wenn die Pflanzen über 7 Tage nach der Aussaat altern. Die Injektionsstelle ist unabhängig vom Alter der Sämlinge gleich.
- Tragen Sie eine Schutzbrille und injizieren Sie die Bakteriensuspension 2-3 mm über dem Coleoptilar-Knoten in die Sämlinge mit einer 25G x 5/8 "Nadel, die an einer 1 ml Einwegspritze befestigt ist.
HINWEIS: Der Coleoptilar-Knoten ist der Ort, an dem sich schließlich Kronenwurzeln bilden. Dies ist der niedrigste Knoten auf der Anlage. In der Regel kommt es am und unterhalb des Knotens zu einem Farbwechsel von Grün zu Weiß. Die Injektionsstelle befindet sich knapp über dem Meristem. Das Sezieren einiger Sämlinge in diesem Stadium kann helfen, die Position des Meristems und folglich die richtige Injektionsstelle zu visualisieren. - Üben Sie sanften Druck auf die Spritze aus, bis die Suspension das Coleoptil füllt oder je nach Wachstumsstadium der Pflanzen im Wirbel sichtbar ist. Dies sind ungefähr 100-200 μL Suspension.
HINWEIS: Wenn es schwierig ist, die Suspension in den Sämling zu injizieren, kann die Injektionsstelle zu niedrig sein. Moderater Druck ist alles, was benötigt werden sollte, um die Suspension zu injizieren. - Injizieren Sie alle Sämlinge und wechseln Sie Spritzen und Nadeln für jedes Konstrukt.
5. Fortgesetzte Pflanzenpflege
- Verpflanzen Sie die injizierten Sämlinge in 13 x 13 x 15 cm oder größere Töpfe, wenn sie 7-8 Tage alt sind.
- Aufrechterhaltung der Wachstumsbedingungen (16 h Photoperiode und Düngung einmal pro Woche).
6. Bestätigung der Infektion (phänotypisch und RT-PCR)
- Phänotypisch bewerten Pflanzen zwischen 14-21 DPI. Läsionen durch Stummschaltung der Kontrollgene Läsion Mimik 22 oder Phytoen-Desaturase können leicht auf den Blättern gesehen werden und unterscheiden sich von FoMV-Symptomen. Die GFP-Expression kann über fluoreszierende Mikroskopaufnahmen oder andere UV-Lichtaufnahmen nachgewiesen werden.
HINWEIS: Einige Konstrukte / virale Vektoren können länger dauern, um Symptome zu zeigen, oder zeigen überhaupt keine Symptome. Hohe Lichtverhältnisse erhöhen die Phänotypen, die durch die Stummschaltung der Läsion Mimik 22 und die Phytoen-Desaturase verursacht werden, stark. Läsionen können weniger sichtbar oder nicht sichtbar sein, wenn Pflanzen unter niedrigeren Lichtverhältnissen wie einer Wachstumskammer gehalten werden, jedoch sollte die tatsächliche Infektionsrate, wie sie durch RT-PCR bestimmt wird, nicht beeinflusst werden (Tabelle 1). - Um die Infektion molekular zu bestätigen, entnehmen Sie Blatt 6 zwischen 14-21 DPI und extrahieren Sie die Gesamt-RNA mit einer Phenol-Chloroform-Extraktion gemäß herstellerseitiger Anweisung.
- Verwendung der extrahierten RNA als Vorlage zur Erzeugung der cDNA des ersten Strangs.
- Richten Sie die cDNA-Reaktion mit bis zu 5 μg Gesamt-RNA, 1 μL zufälligen Hexamer-Primern, 1 μL Oligo (dT)18-Primern , 1 μL dNTPs, 1 μL Reverse Transkriptase und Wasser für ein Endvolumen von 14,5 μL ein.
- Unter Verwendung von Primern, die für das virale Konstrukt und die cDNA als Vorlage entwickelt wurden, führen Sie an jeder Probe eine PCR durch, um die Virusinfektion zu bestätigen und die Integrität des Gens oder Genfragments von Interesse zu bestimmen, wie in 1.1.1 beschrieben, mit Ausnahme der Reduzierung der Zyklen auf 25 für FoMV und 30 für SCMV, um falsch positive Ergebnisse zu vermeiden.
- Verwenden Sie für FoMV-Stummschaltungskonstrukte die Primer FM-5840F und FM-6138R, um über das MCS1*, das das Mais-Genfragment enthält, zu amplifizieren. Verwenden Sie für FoMV-Expressionskonstrukte die Primer 5AmuS2 und 5AmuA2, um über das MCS2 zu amplifizieren, das das eingefügte Gen enthält.
- Verwenden Sie für SCMV-Expressionskonstrukte die Primer SC745-F und HCProR1, um über das MCS zu amplifizieren, das das eingefügte Gen enthält (ergänzende Abbildung 3).
- Verwenden Sie für ein endogenes Kontrollgen die Primer ZmActS und ZmActA, die ein mRNA-Fragment von Maisaktin amplifizieren (GRMZM2G126010) oder die Primer ZmUbiF und ZmUbiR, die ein mRNA-Fragment von Maispolyubiquitin (GRMZM2G409726_T01) amplifizieren.
- Visualisieren Sie das PCR-Produkt auf einem 1% igen Agarosegel, das eine Nukleinsäurefärbung enthält, um das Vorhandensein oder Fehlen von Virus und Gen oder Genfragment zu bestimmen.
Ergebnisse
Ziel dieser Studie war es, ein einfaches Protokoll für die direkte Einführung rekombinanter Viren zu entwickeln, die für das Stummschalten von Genen oder die Genexpression in Maissämlinge entwickelt wurden (Abbildung 2). Die Virusvektoren, die Inserts tragen, werden unter Verwendung molekularbiologischer Standardtechniken entworfen und kloniert. Genfragmente zur Stummschaltung werden in MCS1* in FoMV-EV eingefügt und kodierende Sequenzen für die Expression werden in FoMV-EV bei MCS2 oder SCMV-EV bei MCS eingefügt. Die resultierenden Plasmide werden auf den Agrobacterium-Stamm GV3101 übertragen. Anschließend werden Maissämlinge innerhalb einer Woche oder weniger nach dem Pflanzen injiziert. Zwei Wochen nach der Injektion können Pflanzen sowohl phänotypisch als auch molekular auf Virusinfektion, Gen-Silencing und Genexpression untersucht werden.
Maispflanzen werden in einem torfbasierten Medium für 4-7 Tage angebaut. In diesem Stadium befindet sich das apikale Meristem des Triebes knapp über dem Coleoptilar-Knoten (Abbildung 3A). Nachdem sich das Coleoptil um 2-3 Zentimeter oder bis zu 7 Tage nach der Aussaat ausgedehnt hat, werden die Pflanzen 2-3 mm über dem Coleoptilarknoten injiziert (Abbildung 3B-F). Etwa 12 Tage nach der Injektion beginnen die Pflanzen, Phänotypen auf ihren Blättern zum Schweigen zu bringen, die üblicherweise in der Nähe von Gefäßgewebe beobachtet werden, und diese Läsionen unterscheiden sich visuell von foMV-Virusmosaiksymptomen (Abbildung 4). Sowohl das Vorhandensein von FoMV als auch das Stummschalten von Zielgenen ist in injizierten Pflanzen nachweisbar (Abbildung 5). Die GFP-Expression kann 2 Wochen nach der Injektion unter einem Fluoreszenzmikroskop nachgewiesen werden und ist auf den Blättern 5-7 am stärksten (Abbildung 6). Bei der Beobachtung unter einem Fluoreszenzbildgebungssystem kann die GFP-Expression von FoMV als viele kleine, punktförmige Bereiche der Fluoreszenz sichtbar gemacht werden, die über Blätter in der Nähe von Gefäßgewebe verteilt sind, während die GFP-Expression von SCMV aus größeren Flecken besteht (Abbildung 6, Ergänzende Abbildung 1). Obwohl virale Mosaiksymptome oft bei Pflanzen sichtbar sind, die mit FoMV-Stummschaltungskonstrukten infiziert sind, haben Pflanzen, denen GFP-Expressionskonstrukte injiziert werden, die GFP erfolgreich exprimieren, diese Symptome oft nicht. Infolgedessen kann eine Pflanze ohne sichtbare Symptome immer noch positiv für die Virus- und GFP-Expression sein. Darüber hinaus sollte eine Punktion des Meristems während des Agroinjektionsverfahrens vermieden werden, da dies morphologische Defekte verursachen kann, aber die resultierenden Pflanzen überleben und sind oft symptomatisch (Abbildung 7).
Obwohl dieses Protokoll ursprünglich mit Zuckermais entwickelt wurde, können mehrere Mais-Inzuchtlinien erfolgreich mit FoMV-Gen-Silencing-Konstrukten mittels Agroinjektion geimpft werden. Zum Beispiel haben FR1064 und B73 typischerweise hohe Raten von Virusinfektionen (Tabelle 2). Bemerkenswert ist, dass Mo17, eine Linie mit bekannter genetischer Resistenz gegen FoMV, wie erwartet eine Infektionseffizienz von 0% aufwies36,53. Zusätzlich beeinflusst das verwendete Konstrukt die Infektionseffizienz (Tabelle 3). Im Falle von FoMV haben FoMV-EV und FoMV-LES22 typischerweise die höchsten Infektionswirkungsgrade von 53% bzw. 54%. FoMV-PDS hat mit 38% einen etwas niedrigeren Wirkungsgrad und FoMV-GFP ist mit 17% der niedrigste. SCMV-GFP hat eine Infektionseffizienz von 8%. Diese Prozentsätze sind Durchschnittswerte über mehrere Experimente; Einzelne Experimente können eine höhere oder niedrigere Infektionseffizienz aufweisen.
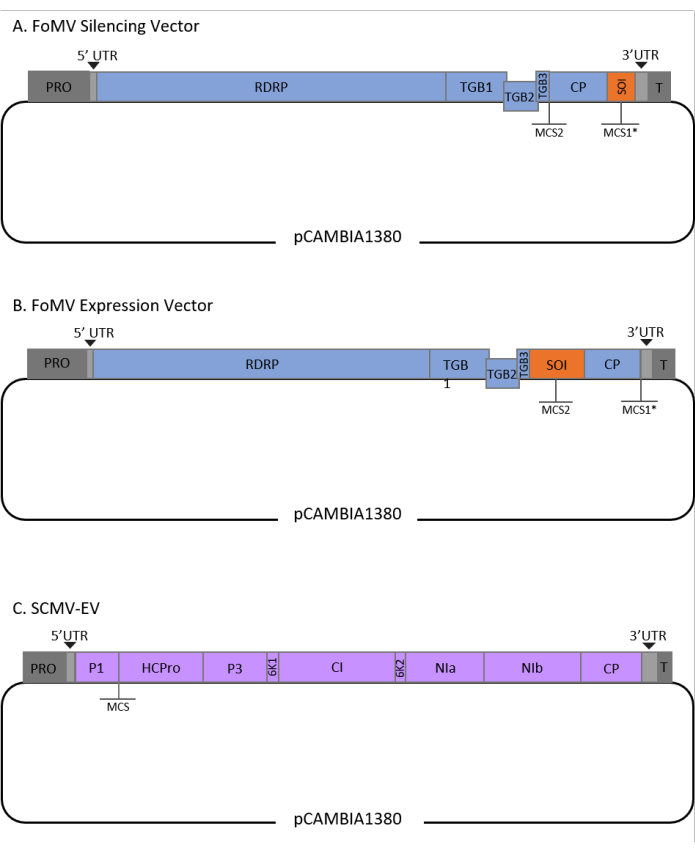
Abbildung 1: Schematische Darstellungen der FoMV- und SCMV-T-DNA-Klone, die für die Agroinjektion in Mais verwendet werden. Der FoMV-Vektor enthält zwei mehrfache Klonierungsstellen (MCS1* und MCS2). Der leere Vektor FoMV-EV ist 7.269 bp und enthält keine Inserts in beiden MCS. (A) Gen-Silencing unter Verwendung des FoMV-Vektors kann durch Einfügen von Genfragmenten in MCS1* erreicht werden, die als Sequence of Interest (SOI) bezeichnet werden, typischerweise in der Anti-Sense-Orientierung. (B) Die Genexpression unter Verwendung des FoMV-Vektors kann durch Einfügen von Gen-ORFs in das MCS2 in der Als SOI bezeichneten Sinnesorientierung erreicht werden. (C) Der SCMV-Vektor wurde so konstruiert, dass er einen MCS zwischen P1 und HCPro hat. Der leere Vektor SCMV-EV ist 11.015 bp und enthält keine Inserts im MCS. In das MCS eingefügte Gen-ORFs, die sich im Rahmen des SCMV-Polyproteins befinden, werden als Proteine exprimiert. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
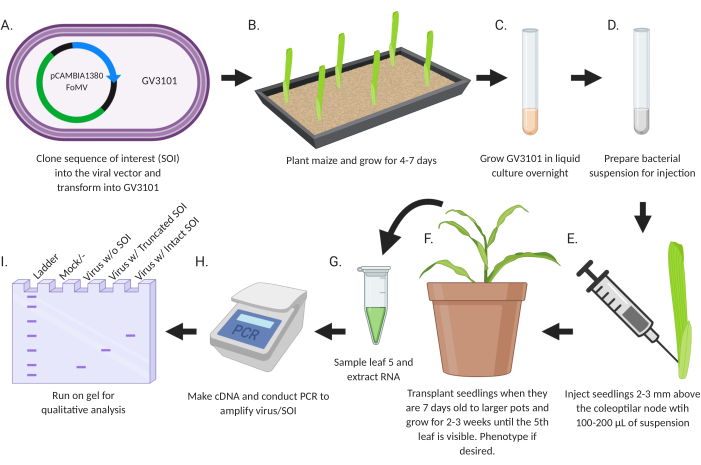
Abbildung 2: Schematische Zusammenfassung des Agroinjektionsprotokolls. (A) Klonen Sie SOI, entweder ein CDS oder ein Genfragment, in den viralen Vektor und wandeln Sie sich in den Agrobacterium-Stamm GV3101 um. (B) Mais anpflanzen und 4-7 Tage wachsen lassen. (C) GV3101 in flüssiger Kultur über Nacht bei 28 °C züchten. (D) Bakteriensuspension zur Injektion vorbereiten. (E) Sämlinge 2-3 mm über dem Coleoptilarknoten mit 100-200 μL Suspension injizieren. (F) Sämlinge, wenn sie 7 Tage alt sind, in größere Töpfe umpflanzen und 2-3 Wochen wachsen, bis das 5. Blatt sichtbar ist. Phänotyp, falls gewünscht. (G) Probe blatt 5 und Extrakt-RNA. (H) Machen Sie cDNA und führen Sie PCR durch, um Virus / SOI zu verstärken. (I) Auf Gel zur qualitativen Analyse ausführen, um das Vorhandensein/ Fehlen eines Virus und einer abgeschnittenen oder intakten SOI zu bestimmen. Diese Figur wurde mit BioRender.com erstellt. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 3: Agroinjektionsmethode zur Impfung von Sämlingen knapp über dem Coleoptil-Knoten. (A) 4-5 Tage alte Pflanzen. Das Coleoptil ist vollständig erweitert, und das erste echte Blatt kann teilweise sichtbar sein, wird aber nicht entfaltet. (B) 6-7 Tage alte Pflanzen. Das erste Blatt kann erweitert werden, aber es sind keine Kragen sichtbar. Das zweite Blatt wird ebenfalls sichtbar sein und kann sich in diesem Stadium zu entfalten beginnen. (C) Dissektion von 6-7 Tage alten Pflanzen, die die Lage des apikalen Meristems des Triebes in Bezug auf den Coleoptil-Knoten zeigen. (D) Injektion von 4-5 Tage alten Pflanzen. (E) Injektion von 6-7 Tage alten Pflanzen. (F) Injektion von 6-7 Tage alten Pflanzen mit einer Farbstofflösung, die eingefärbtes Inokulum zeigt, das aus dem Wirbel des Sämlings kommt. (G) Nahaufnahme der Injektionsstelle von 6-7 Tage alten Pflanzen in Bezug auf den Coleoptil-Knoten. (H) Nahaufnahme einer 6-7 Tage alten Pflanze nach der Injektion, die eingefärbtes Inokulum im Wirbel der Pflanze zeigt. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 4: Symptome der in den Agroinjektionsexperimenten verwendeten Stummschaltungskontrollgene. (A) Ein Blatt, das bei 17 DPI fotografiert wurde, nachdem der Pflanze FoMV-LES22 injiziert wurde. FoMV-LES22 trägt einen 329 bp Insert des 3' CDS des Läsionsimitat 22 Maisgens in der Antisense-Orientierung. Silencing führt zur Ansammlung eines toxischen Metaboliten, der wiederum die nekrotischen Läsionen verursacht, die zuerst als Streifen entlang des Gefäßsystems erscheinen und zu größeren Flecken wachsen, wie hier gezeigt. (B) Ein Blatt, das mit 17 DPI fotografiert wurde, nachdem der Pflanze FoMV-PDS injiziert wurde. FoMV-PDS trägt einen 313 Basenpaar-Insert des 3' CDS des Sorghum-Phytoen-Desaturase-Gens in der Antisense-Orientierung. Das Stummschalten von PDS in Mais verursacht einen systemischen photobleichenden Phänotyp, der als kleine, dünne Streifen entlang des Gefäßsystems beginnt, die zu längeren Streifen entlang der Blattlänge wachsen, wie hier gezeigt. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
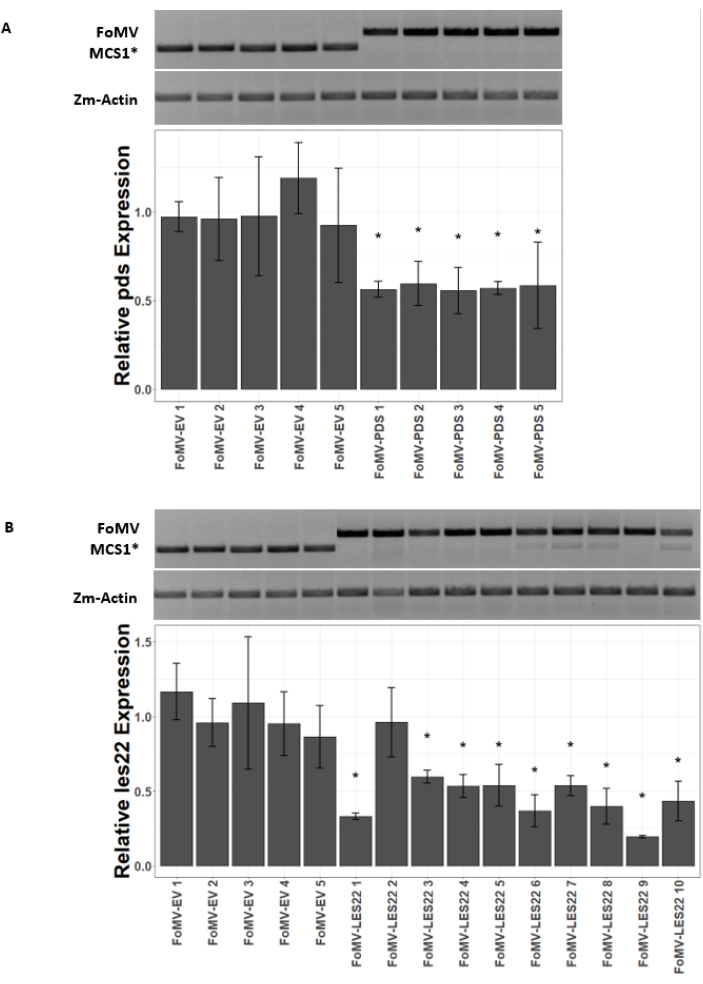
Abbildung 5: qRT-PCR von Pflanzen, denen FoMV-Gen-Silencing-Konstrukte injiziert wurden. Bestätigung der systemischen FoMV-Infektion und des Gen-Silencings, induziert durch die FoMV-LES22- und FoMV-PDS-Konstrukte, die durch Agroinjektion in Zuckermaispflanzen (Golden x Bantam) verabreicht werden. (A) Die Gelbilder zeigen RT-PCR-Analysen, die das Vorhandensein von FoMV-MCS1* leerem Vektor (315 bp Amplikon) und FoMV-PDS (625 bp Amplikon) in Blatt 6 von fünf Einzelpflanzen bestätigen. Die verwendeten PCR-Primer erzeugen ein Amplikon, das MCS1* überspannt. Als Referenzgen dient das Maisgenaktin (Zm-Actin) Amplicon. Das Balkendiagramm stellt die relativen qRT-PCR-Expressionswerte für die pds-Expression in Blatt 6 37 Tage nach der Impfung (DPI) durch Agroinjektion mit FoMV-MCS1* oder FoMV-PDS dar. Die Unterdrückung von pds ist in jedem der fünf biologischen Replikate nachweisbar (p = 0,003; Post-hoc-Dunnett-Test; Fehlerbalken zeigen die Standardabweichung (SD) von drei technischen Replikaten an). (B) Die Gelbilder zeigen RT-PCR-Analysen, die das Vorhandensein von FoMV-MCS1* (315 bp Amplikon) in Blatt 6 von fünf Einzelpflanzen bestätigen. FoMV-LES22 (625 bp Amplikon) wurde in Blatt 6 Gewebe (Proben FoMV-LES22 1-5, 38 DPI) und Blatt 4 (Proben FoMV-LES22 6-10, 20 DPI) für zehn einzelne Pflanzen nachgewiesen. Als Referenzgen diente das Zm-Actin-Amplikon. Das Balkendiagramm stellt die relativen qRT-PCR-Expressionswerte für die les22-Expression in Maisgewebe durch Agroinjektion von FoMV-MCS1*- oder FoMV-LES22-Viruskonstrukten dar. Les22-Unterdrückung tritt in 9 von 10 biologischen Replikaten auf (p =<0,0001; Post-hoc-Dunnett-Test; Fehlerbalken zeigen SD für drei technische Replikate an). Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
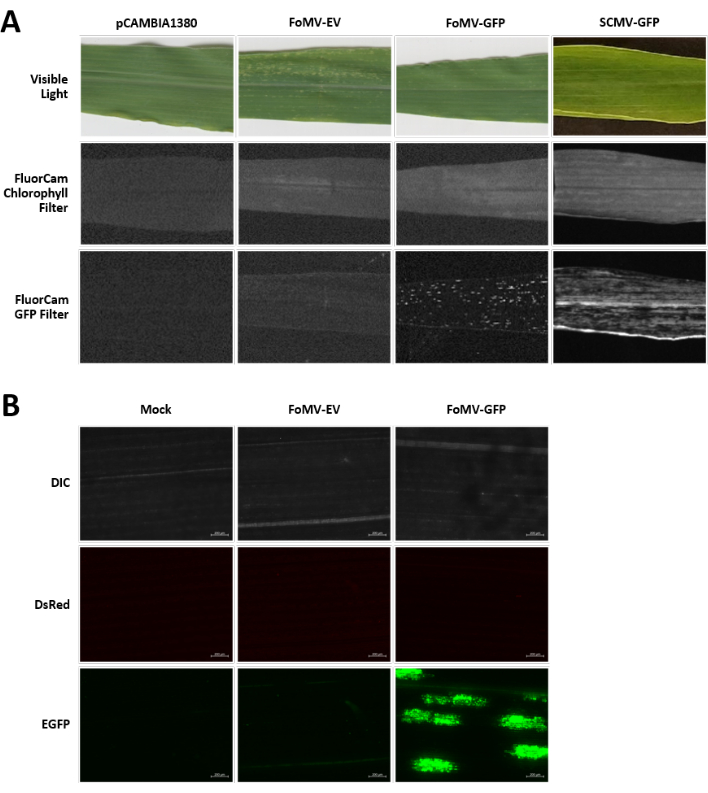
Abbildung 6. Phänotypen verschiedener Konstrukte, die in den Agroinjektionsexperimenten verwendet werden. Alle abgebildeten Pflanzen wurden injiziert, als sie 6-7 Tage alt waren, mit dem Agrobacterium-Stamm GV3101, der die angegebenen Konstrukte trug. Die Bilder wurden mit 16 DPI aufgenommen. (A) Blattsymptome von pCAMBIA1380 (leeres Plasmid-Rückgrat), FoMV-EV, FoMV-GFP und SCMV-GFP im sichtbaren Licht, unter dem FluorCam Chlorophyllfilter bei 250 μs Exposition und unter dem FluorCam GFP-Filter bei 10 ms Exposition. (B) Fluoreszierende mikroskopische Aufnahmen der Blätter von pseudobehandelten (nur mit MgSO4-Lösung injizierten), FoMV-EV und FoMV-GFP injizierten Pflanzen. Die DIC-, DsRed- und EGFP-Kanäle werden gezeigt und wurden jeweils bei 1500 ms Exposition aufgenommen. Der Maßstabsbalken beträgt 200 μm. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 7. Morphologische Wirkungen der Injektion. Ein Beispiel für die schwereren morphologischen Effekte, die durch direkte Injektion in meristematisches Gewebe auftreten können. Diese Verletzung kann zu einem "Schreddern" der Blätter und einer Spaltung des Stängels führen. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
| Virus | Wachstumsbedingungen | Genotyp | # Infizierte Pflanzen | Gesamtzahl der Pflanzen | % Infektion | Durchschnittlicher Prozentsatz der Infektion |
| FoMV-EV | Wachstumskammer | Zuckermais | 22 | 23 | 96% | 97% |
| B73 | 18 | 18 | 100% | |||
| B104 | 20 | 21 | 95% | |||
| Gewächshaus | Zuckermais | 20 | 23 | 87% | 89% | |
| B73 | 17 | 18 | 94% | |||
| B104 | 16 | 19 | 84% | |||
| SCMV-EV | Wachstumskammer | Zuckermais | 14 | 21 | 67% | 47% |
| B73 | 5 | 18 | 28% | |||
| B104 | 10 | 21 | 48% | |||
| Gewächshaus | Zuckermais | 14 | 23 | 61% | 49% | |
| B73 | 0 | 19 | 0% | |||
| B104 | 19 | 22 | 86% |
Tabelle 1: Einfluss der Treibhaus- und Wachstumskammerbedingungen auf die Impfeffizienz der Agroinjektion. Samen wurden unter identischen Wachstumsbedingungen gekeimt. Gekeimte Sämlinge wurden agroinjiziert und die Hälfte von ihnen wurde in eine Wachstumskammer gebracht (25 ° C 16 h Tageslicht / 22C 8 h Nacht; 185 PAR) und die andere Hälfte wurde in ein Gewächshaus gebracht (22-25 ° C 16 h Tageslicht / 22-25 ° C 8 h Nacht; 350-400 PAR). Diese Tabelle zeigt die Infektionsrate in Prozent, berechnet aus der Anzahl der Pflanzen, von denen durch RT-PCR bestätigt wurde, dass sie mit dem jeweiligen Virus infiziert sind, geteilt durch die Gesamtzahl der agroinjizierten Pflanzen. Es gibt keinen statistischen Unterschied in der Infektionseffizienz zwischen Wachstumskammer- und Gewächshausbedingungen (FoMV-Zweischwanz-t-Test p=0,08; SCMV zweiseitiger t-Test p=0,96).
| Mais-Genotyp | FoMV-EV | FoMV-LES22 | Gesamtsumme | ||||
| Infiziert | Gesamt | % Infizierte | Infiziert | Gesamt | % Infizierte | % Infizierte | |
| Zuckermais | 18 | 23 | 78% | 15 | 23 | 65% | 72% |
| MO47 | 7 | 22 | 32% | 1 | 21 | 5% | 19% |
| K55 | 1 | 15 | 7% | 3 | 17 | 18% | 13% |
| W64A | 10 | 22 | 45% | 8 | 20 | 40% | 43% |
| MO17 | 0 | 16 | 0% | 0 | 13 | 0% | 0% |
| B73 | 10 | 18 | 56% | 7 | 17 | 41% | 49% |
| B101 | 12 | 21 | 57% | 8 | 24 | 33% | 44% |
| FR1064 | 4 | 4 | 100% | 4 | 4 | 100% | 100% |
| B104 | 10 | 22 | 45% | 5 | 21 | 24% | 35% |
| ÖRK22 | 2 | 7 | 29% | 4 | 6 | 67% | 46% |
| A188 | 0 | 3 | 0% | 4 | 6 | 67% | 44% |
Tabelle 2: Infektionseffizienz von FoMV-Konstrukten über Maisgenotypen hinweg. FoMV-EV und FoMV-LES22 wurden in 11 Maisgenossen agroinjiziert. Nach der Injektion wurden die Sämlinge in das Gewächshaus gebracht. Diese Tabelle zeigt die Infektionsrate in Prozent, berechnet aus der Anzahl der mit FoMV infizierten Pflanzen, wie durch RT-PCR bestätigt, geteilt durch die Gesamtzahl der agroinjizierten Pflanzen. Die kombinierte Gesamtinfektionsrate zeigt die durchschnittlichen Infektionsraten jedes Genotyps für beide getesteten FoMV-Konstrukte.
| Anlagenstadium | 4-5 Tage alte Pflanzen | 6-7 Tage alte Pflanzen | Gesamtsumme | ||||
| Symptomatisch | Gesamtzahl der Anlagen | % Infizierte | Symptomatisch | Gesamtzahl der Anlagen | % Infizierte | % Infizierte | |
| FoMV-EV | 42 | 72 | 58% | 80 | 170 | 47% | 53% (A) |
| FoMV-PDS | 65 | 157 | 41% | 66 | 184 | 36% | 39% (B C) |
| FoMV-LES22 | 115 | 195 | 59% | 144 | 292 | 49% | 54% (A B) |
| FoMV-GFP | 16 | 103 | 16% | 37 | 217 | 17% | 16% (C) |
| SCMV-GFP | 10 | 95 | 11% | 5 | 82 | 6% | 8 % (C) |
Tabelle 3: Zusammenfassung der Injektionsexperimente. Diese Tabelle stellt eine Zusammenfassung der Injektionsexperimente dar, die von August 2017 bis August 2018 an Golden Bantam Zuckermaissämlingen durchgeführt wurden. Die Pflanzen wurden auf virale Symptome (FoMV-EV), Stummschaltungssymptome (pds und les22) oder GFP-Fluoreszenz (GFP) durch visuelles (FoMV-EV, FoMV-PDS und FoMV-LES22) oder FluorCam-Screening (FoMV-GFP und SCMV-GFP) untersucht. Die Ergebnisse werden einzeln für 4-5 Tage alte Pflanzen und 6-7 Tage alte Pflanzen sowie eine Zusammenfassung über alle Pflanzenalter hinweg angezeigt. Es gibt keinen signifikanten Unterschied zwischen 4-5 Tage alten Pflanzen und 6-7 Tage alten Pflanzen (Einweg-ANOVA, F = 0,6513). Es gibt einen Unterschied zwischen viralen Konstrukten (Onaway ANOVA, F = <0.0001), wobei die Buchstaben den Tukey-Kramer HSD-Verbindungsbuchstabenbericht darstellen.
Ergänzende Tabelle 1: Tabelle mit allen in diesem Protokoll verwendeten Primernamen und -sequenzen. Bitte klicken Sie hier, um diese Tabelle herunterzuladen.
Ergänzende Tabelle 2: Acetosyringon-Test. (A) Anfänglicher Acetosyringon-Test zum Vergleich der Symptomraten von Mock-, FoMV-EV- und FoMV-LES22-injizierten Pflanzen zwischen Impfsuspensionen mit 200 μM Acetosyringon (+) oder ohne Acetosyringon (-). (B) Vergleich der Infektionsraten von FoMV-LES22, bestimmt durch RT-PCR zwischen Impfsuspensionen ohne Acetosyron (-), mit 200 μM Acetosyringon (+), und Zugabe von 20 μM Acetosyringon zur Bakterienkultur 4 Stunden vor der Resuspension in Puffer zusammen mit der Zugabe von 200 μM Acetosyringon zur Endgültigen Suspension (++). Insgesamt wurde kein signifikanter Unterschied zwischen Aceotysyringon-Behandlungen gefunden (Oneway ANOVA, f=0,5452). Bitte klicken Sie hier, um diese Tabelle herunterzuladen.
Ergänzende Abbildung 1: Fluoreszenzbildgebung und molekulare Validierung von agroinjiziertem SCMV und Expression heterologer Proteine in Mais. Mais wurde mit einem modifizierten SCMV-Konstrukt agroinjiziert, das sowohl CDS von GFP als auch Nano-Luciferase (NLuc) enthielt. (A) Die Fluorcam-Bildgebung wurde zum Screening und nachweisen von GFP verwendet. Die linke ist eine simulierte injizierte Pflanze und die rechte ist eine SCMV-NLucGFP injizierte Pflanze. (B) Blattproteinextrakte wurden mittels SDS-PAGE getrennt und auf das Vorhandensein von NLuc-, GFP- und SCMV-Mantelprotein (CP) mittels In-Gel-Luciferase-Assay oder Immunoblot wie angegeben bewertet. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Diskussion
Agrobacterium ist ein wesentliches Werkzeug, das zahlreiche molekularbiologische Techniken in der pflanzenbezogenen Forschung ermöglicht. Diese Studie liefert ein Agroinjektionsprotokoll für die Impfung von FoMV- und SCMV-Virusvektoren direkt in Maisgewebe für VIGS- und VOX-Anwendungen. Das Hauptziel ist es, die Leichtigkeit und den Nutzen virusbasierter Technologien für die Forschung an Monokotyledonen-Kulturpflanzen zu erhöhen. Obwohl für einige Viren eine direkte Agroinokulation von Mais berichtet wurde, ist den Autoren kein detailliertes Protokoll bekannt, und es gibt keine Beispiele für VIGS- und VOX-Anwendungen in diesen Studien19,22.
Es wurde berichtet und während der Entwicklung dieses Protokolls bestätigt, dass der Injektionsort ein Schlüsselfaktor für die erfolgreiche Einleitung einer systemischen Virusinfektion mittels Agroinjektion ist19. Die konsequente Injektion der empfohlenen Position auf die Pflanze wird als die größte Variable angesehen, da die genaue Position des Meristems in Maissämlingen mit dem Auge praktisch nicht nachweisbar ist. Um zwischenmenschliche Variationen zu minimieren, wird empfohlen, einige Maissämlinge bis zum Meristem zu sezieren, um ihre Position besser zu visualisieren (Abbildung 3C). Die Position des Meristems in Bezug auf den Coleoptilar-Knoten sollte für Pflanzen im Alter von 4-7 Tagen ungefähr gleich sein. Darüber hinaus bietet das Üben der Injektion mit einer gefärbten Flüssigkeit eine leicht sichtbare Demonstration, wie das "Inokulum" den Blattwirbel füllt, und da die Injektionsstelle mit Farbstoff markiert ist, kann die Genauigkeit der Injektionsstelle bestätigt werden (Abbildung 3G, H). Meristematische Gewebe sind am anfälligsten für Agroinjektion, aber die Injektion von Agrobacterium-Suspensionen direkt in dieses Gewebe führt zu unerwünschten morphologischen Wirkungen (Abbildung 6)19. Pflanzen mit beschädigten Meristemen überleben, aber die daraus resultierenden Defekte sind unerwünscht, und daher sollte eine direkte Injektion dieses Gewebes vermieden werden.
Es gibt mehrere Variablen, die den erfolgreichen Start einer systemischen Virusinfektion durch Agroinjektion beeinflussen können, da drei komplexe biologische Systeme (Pflanze, Virus und Agrobacterium-Stamm ) in Koordination interagieren müssen. Dieses komplexe Zusammenspiel kann durch die sich schnell teilenden Zellen der meristematischen Region unterstützt werden, was sie zu einem idealen Standort für die Agroinokulation macht19. Der Agrobacterium-Stamm muss in der Lage sein, Zellen des Pflanzengewebes zu infizieren, um die T-DNA zu liefern, die das virale Genom trägt, und die Pflanze muss anfällig für das Virus sein, um die Virusreplikation und systemische Infektion einzuleiten. Mais-Genotypen unterscheiden sich in ihrer Anfälligkeit für Viren (z. B. Mo17 ist resistent gegen FoMV) oder Agrobacterium-Stämme , aber die Mehrheit, die getestet wurde, scheint sowohl für FoMV als auch für SCMV empfänglich zu sein (Tabelle 1 und Tabelle 2)53. Zum Beispiel können die Inzuchtlinie FR1064 und die Zuckermaissorte Golden Bantam besonders anfällig für GV3101 Agrobacterium und FoMV-basierte Vektoren sein.
Die Beprobung der Beprobung und der Zeitpunkt der Probenahme für die RT-PCR sind entscheidend für die genaue Beurteilung der Virusinfektion. In den hier gezeigten Beispielen wurde die Blattzahl bestimmt, indem beim ersten gerundeten Blatt (allgemein bekannt als "Daumenblatt") begonnen und nach oben gezählt wurde. Die Blätter wurden beprobt, sobald sie expandiert waren und das nächste Blatt zu entstehen begann. Welche Blätter für die Probenahme optimal sind, kann jedoch je nach verwendeter Virusart, Wachstumsbedingungen und Maisgenotyp variieren. Daher wird ein anfängliches Zeitkursexperiment empfohlen, wenn dieses Protokoll auf ein neues Virensystem angewendet wird, um die Stichprobenstrategie in Bezug auf Blätter und Timing zu optimieren.
Das verwendete spezifische Konstrukt beeinflusst die Effizienz dieses Protokolls erheblich. Zum Beispiel produzieren die leeren Vektoren FoMV-EV und SCMV-EV sowie FoMV-PDS und FoMV-LES22, die beide kleine Einsätze (313 bp bzw. 329 bp) enthalten, typischerweise die höchsten Prozentsätze von Pflanzen mit viralen Symptomen in diesen Experimenten (Tabellen 1 und Tabelle 2). Rekombinante Viren, die größere Einsätze des GFP ORF (720 bp) in FoMV-GFP und SCMV-GFP trugen, hatten jedoch im Vergleich zu Pflanzen, denen die leeren Vektor- oder Gen-Silencing-Konstrukte injiziert wurden, viel niedrigere Infektionsraten. Dieser Trend könnte auf die negativen Auswirkungen auf die virale Fitness zurückzuführen sein, die durch zunehmende Mengen an exogenem genetischem Material im viralen Genom verursacht werden. Mehrere Studien haben gezeigt, dass die Insert-Stabilität pflanzlicher viraler Vektoren weitgehend von der Insertgröße und -sequenz abhängt36,54,55,56,57. Darüber hinaus gab es einen bemerkenswerten Unterschied im Prozentsatz der Pflanzen, die sich nach der Impfung mit dem leeren FoMV- oder SCMV-Vektor infizieren, was darauf hindeutet, dass zusätzliche Arbeit erforderlich ist, um dieses Protokoll für SCMV zu optimieren (Tabelle 1). Diese Ergebnisse deuten darauf hin, dass bei der Entwicklung eines Konstrukts möglicherweise eine Fehlerbehebung erforderlich ist, da sowohl die Reihenfolge als auch die Länge des Fragments die Effizienz beeinträchtigen können.
Insgesamt hat diese Studie gezeigt, dass die Agroinjektion von Maissämlingen eine wirksame Impfmethode für zwei verschiedene RNA-Pflanzenviren, mehrere Vektorkonfigurationen und 11 Genotypen von Mais ist. Diese Arbeit mit FoMV und SCMV, gepaart mit früheren Arbeiten unter Verwendung der Injektion mit dem Chlorocemottle-Virus (MCMV) oder MSV, zeigt, dass die Agroinjektion für die Impfung von Maissämlingen mit infektiösen Klonen von RNA- und DNA-Viren geeignet ist19,20,21,22. Darüber hinaus zeigt diese Arbeit, dass die Agroinjektion eine praktikable Methode für VIGS- und VOX-Vektoren ist und auf Pflanzen ab vier Tagen angewendet werden kann (Tabelle 3). Es wird erwartet, dass das hier vorgestellte Protokoll von Maisbiologen leicht angepasst werden kann, um die Forschung in funktionellen Genomik-Studien mit transientem Gen-Silencing (VIGS) und Überexpression (VOX) zu erleichtern. Agroinjektion hat auch die Fähigkeit, virusbasierte Gen-Editing-Ansätze (VEdGE) zu erleichtern, die sonst durch die Abhängigkeit von der Pflanzentransformation eingeschränkt wären, was möglicherweise die Bearbeitungseffizienz sowie die Zugänglichkeit verbessert58,59,60. Angesichts des geeigneten Agrobacterium-Stammes, der Mais-Genotypen und der viralen Vektoren, die sorgfältig kombiniert werden, wird erwartet, dass die Impfung durch Agroinjektion zu einem wertvollen Werkzeug für transiente Genfunktionsanalysen in Mais wird.
Offenlegungen
Die Forscher haben keine Interessenkonflikte offenzulegen.
Danksagungen
Die Iowa State University ist Teil eines Teams, das das Insect Allies-Programm HR0011-17-2-0053 der DARPA unterstützt. Diese Arbeit wurde auch vom Iowa State University Plant Sciences Institute, dem Iowa State University Crop Bioengineering Center, dem USDA NIFA Hatch Projektnummer 3808 und den State of Iowa Funds unterstützt. K.L.H. wurde auch teilweise durch das von der National Science Foundation (DGE #1545453) finanzierte Graduiertenausbildungsprogramm Der Iowa State University Predictive Plant Phenomics und durch den Zuschuss Nr. 2019-07318 der Agricultural and Food Research Initiative des USDA National Institute of Food and Agriculture unterstützt. Die Geldgeber spielten keine Rolle bei der Gestaltung der Studie und Sammlung, Analyse und Interpretation der Daten und beim Schreiben des Manuskripts. Alle Meinungen, Ergebnisse und Schlussfolgerungen oder Empfehlungen, die in diesem Material zum Ausdruck gebracht werden, sind die der Autoren und spiegeln nicht unbedingt die Ansichten der Geldgeber wider.
Wir danken Nick Lauter (USDA-ARS, Ames, IA) für das Saatgut von Mais-Inzuchtlinien, Christian F. Montes-Serey (Iowa State University) für die Herstellung des FoMV-GFP-Klons und Tyler Austin (Iowa State University) für die technische Unterstützung.
Materialien
| Name | Company | Catalog Number | Comments |
| 1 mL syringes | Fisher Scientific | 14955450 | alternatively, BD 309659 |
| 15 mL Falcon Tubes | Corning Science | 352059 | |
| 1kb+ Ladder | ThermoFisher Scientific | 10787018 | For assessing sizes of PCR products |
| 25G x 5/8" PrecisionGlide Needles | Becton, Dickinson and Company (BD) | 305122 | |
| 28°C Incubator | For Agrobacterium | ||
| 37°C Incubator | For E. coli | ||
| Acetosyringone | MilliporeSigma | D134406 | Optional |
| Agar | MilliporeSigma | A4800 | |
| Agarose | GeneMate | E-3120 | For making gels to check for virus/insert stability |
| Agrobacterium tumefaciens Strain GV3101 | Carries vir plasmid encoding T-DNA transfer machinery, RifR, GmR, from lab stock | ||
| Bsu36I | New England Biolabs | R0524 | |
| cDNA Kit | ThermoFisher Scientific | K1672 | Maxima First Strand cDNA Synthesis Kit with Dnase |
| Chloroform | Fisher Scientific | C298 | For RNA extraction |
| Cuvettes | Fisher Scientific | 14955127 | 1.5 mL |
| D-(+)-Glucose | MilliporeSigma | G7528 | Alkaline Lysis |
| DH5alpha Competent E. coli Cells | New England Biolabs | C2987 | |
| DNA Ligase | ThermoFisher Scientific | K1422 | Rapid DNA Ligation Kit |
| EDTA (Ethylenediamine Tetraacetic Acid, Disodium Salt Dihydrate) | Fisher Scientific | S311 | Alkaline Lysis |
| Ethanol | For RNA extraction | ||
| Fertilizer | Peters Fertilizer 15-15-15 Concentrate | ||
| Flat Inserts | T.O. Plastics | 715357C | For germinating seeds in trays |
| Flats | T.O. Plastics | 710245C | For germinating seeds in trays |
| FluorCam | Photon Systems Instruments | To assess maize plants for GFP expression before microscope | |
| Fluorescence Microscope | |||
| Gel Electrophoresis Box | |||
| Gentamycin Sulfate | Fisher Scientific | BP918 | |
| Glacial Acetate | Fisher Scientific | A38 | Alkaline Lysis |
| Glycerol | Fisher Scientific | G33-500 | For saving frozen stocks of bacteria |
| Go-Taq, 2X | Promega | M7123 | |
| Hydrochloric Acid | Fisher Scientific | A144 | for pHing solutions |
| Isopropanol | Sigma-Aldrich | 109827 | For RNA extraction |
| Kanamycin, Monosulfate | Fisher Scientific | BP906 | |
| Large Pots | Kordlok | SQL0550 | 5x5x4" or bigger. For transplanting seedlings. |
| Luria Bertani (LB) Broth, Miller | Himedia | M1245 | |
| Magnesium Sulfate Heptahydrate | Amresco | 662 | |
| Maize Golden Bantam Sweet Corn Seed | American Meadows, West Coast Seeds | ||
| Maize Inbred Seed | Our seed comes from our institution, but we are not able to provide this for other researchers. | ||
| Maxima H Minus Reverse Transcriptase | ThermoFisher Scientific | EP0753 | |
| MilliQ | Elga | Purelab Ultra | |
| Monarch PCR & DNA Cleanup Kit | New England Biolabs | T1030 | |
| PacI | New England Biolabs | R0547 | |
| Peters Excel 15-5-15 Fertilizer | ICL Specialty Fertilizers | G99140 | |
| Petri Dish, 95 mm x 15 mm | Fisher Scientific | FB0875714G | |
| pH Meter | |||
| Potassium Acetate | Fisher Scientific | P171 | Alkaline Lysis |
| Primers | Our primers were synthesized through our institutional DNA facility or through IDT | ||
| PspOMI | New England Biolabs | R0653 | |
| Q5 High-Fidelity DNA Polymerase | New England Biolabs | M0491 | |
| Rifampicin | EMD Millipore Corp | 557303 | |
| Rnase A | ThermoFisher Scientific | 12091021 | Alkaline Lysis |
| SbfI | New England Biolabs | R0642 | |
| Scale | For weighing chemicals for media or buffers | ||
| SDS (Sodium Dodecyl Sulfate) | Fisher Scientific | BP166 | Alkaline Lysis |
| Sodium Hydroxide | Fisher Scientific | S318 | Alkaline Lysis |
| Soil Substrate | SunGro Horticulture | SS#1-F1P | Sunshine Mix #1/Fafard-1P, any soil mix that maize grows well in is sufficient |
| Spectrophotometer | For measuring OD600 | ||
| Sybr Safe, 10,000X | Invitrogen | S33102 | For making gels to check for virus/insert stability |
| Thermocycler | For PCR | ||
| Tris Base | Fisher Scientific | BP154 | Alkaline Lysis |
| Trizol | Ambion | 15596018 | For RNA extraction |
| Weigh Paper | For weighing chemicals for media or buffers | ||
| XbaI | New England Biolabs | R0145 |
Referenzen
- Zaidi, S. E. A., Mansoor, S. Viral vectors for plant genome engineering. Frontiers in Plant Science. 8, 539(2017).
- Kant, R., Dasgupta, I. Gene silencing approaches through virus-based vectors: speeding up functional genomics in monocots. Plant Molecular Biology. 100, 3-18 (2019).
- Hu, J., et al. A barley stripe mosaic virus-based guide RNA delivery system for targeted mutagenesis in wheat and maize. Molecular Plant Pathology. 20 (10), 1463-1474 (2019).
- Pasin, F., Menzel, W., Daròs, J. A. Harnessed viruses in the age of metagenomics and synthetic biology: an update on infectious clone assembly and biotechnologies of plant viruses. Plant Biotechnology Journal. 17 (6), 1010-1026 (2019).
- Cody, W. B., Scholthof, H. B. Plant virus vectors 3.0: Transitioning into synthetic genomics. Annual Review of Phytopathology. 57 (1), 211-230 (2019).
- Mei, Y., et al. Protein expression and gene editing in monocots using foxtail mosaic virus vectors. Plant Direct. 3 (11), 00181(2019).
- Ruiz, M. T., Voinnet, O., Baulcombe, D. C. Initiation and maintenance of virus-induced gene silencing. Plant Cell. 10 (6), 937-946 (1998).
- Bekele, D., Tesfaye, K., Fikre, A. Applications of virus induced gene silencing (VIGS) in plant functional genomics studies. Journal of Plant Biochemistry & Physiology. 07 (01), 1000229(2019).
- Scholthof, H. B., Scholthof, K. B. G., Jackson, A. O. Plant virus gene vectors for transient expression of foreign proteins in plants. Annual Review of Phytopathology. 34 (1), 299-323 (1996).
- Holzberg, S., Brosio, P., Gross, C., Pogue, G. P. Barley stripe mosaic virus-induced gene silencing in a monocot plant. Plant Journal. 30 (3), 315-327 (2002).
- Wang, R., et al. An efficient virus-induced gene silencing vector for maize functional genomics research. Plant Journal. 86 (1), 102-115 (2016).
- Redinbaugh, M. G., et al. Transmission of viral RNA and DNA to maize kernels by vascular puncture inoculation. Journal of Virological Methods. 98 (2), 135-143 (2001).
- Scholthof, H. B. The capsid protein gene of tomato bushy stunt virus is dispensable for systemic movement and can be replaced for localized expression of foreign genes. Molecular Plant-Microbe Interactions. 6 (3), 309(1993).
- Scholthof, H. B., Scholthof, K. B. G., Kikkert, M., Jackson, A. O. Tomato bushy stunt virus spread is regulated by two nested genes that function in cell-to-cell movement and host-dependent systemic invasion. Virology. 213 (2), 425-438 (1995).
- Scholthof, H. B. Rapid delivery of foreign genes into plants by direct rub-inoculation with intact plasmid dna of a tomato bushy stunt virus gene vector. Journal of Virology. 73 (9), 7823-7829 (1999).
- Zhang, J., et al. Vacuum and co-cultivation agroinfiltration of (germinated) seeds results in tobacco rattle virus (TRV) mediated whole-plant virus-induced gene silencing (VIGS) in wheat and maize. Frontiers in Plant Science. 8, 393(2017).
- Vaghchhipawala, Z., Rojas, C. M., Senthil-Kumar, M., Mysore, K. S. Agroinoculation and agroinfiltration: simple tools for complex gene function analyses. Methods in Molecular Biology. 678, Clifton, N.J. 65-76 (2011).
- Grimsley, N., Hohn, B., Hohn, T., Walden, R. "Agroinfection," an alternative route for viral infection of plants by using the Ti plasmid. Proceedings of the National Academy of Sciences. 83 (10), 3282-3286 (1986).
- Grimsley, N. H., Ramos, C., Hein, T., Hohn, B. Merisfematic tissues of maize plants are most suscepnsle to agroinfection with maize streak virus. Bio/Technology. 6 (2), 185-189 (1988).
- Martin, D. P., Rybicki, E. P. Improved efficiency of Zea mays agroinoculation with Maize streak virus. Plant Disease. 84 (10), 1096(2000).
- Martin, D. P., Willment, J. A., Rybicki, E. P. Evaluation of maize streak virus pathogenicity in differentially resistant Zea mays genotypes. Phytopathology. 89 (8), 695-700 (1999).
- Wang, Q., et al. Further characterization of Maize chlorotic mottle virus and its synergistic interaction with Sugarcane mosaic virus in maize. Scientific Reports. 7, 39960(2017).
- Hsieh, M. H., et al. Optimizing virus-induced gene silencing efficiency with Cymbidium mosaic virus in Phalaenopsis flower. Plant Science. 201-202 (1), 25-41 (2013).
- Hsieh, M. H., et al. Virus-induced gene silencing unravels multiple transcription factors involved in floral growth and development in Phalaenopsis orchids. Journal of Experimental Botany. 64 (12), 3869-3884 (2013).
- Zenna, N. S., et al. Genetic analysis of tolerance to rice tungro bacilliform virus in rice (Oryza sativa L.) through agroinoculation. Journal of Phytopathology. 154 (4), 197-203 (2006).
- Marks, M. S., Kemp, J. M., Woolston, C. J., Dale, P. J. Agroinfection of wheat: A comparison of Agrobacterium strains. Plant Science. 63 (2), 247-256 (1989).
- Dasgupta, I., et al. Rice tungro bacilliform virus DNA independently infects rice after Agrobacterium-mediated transfer. Journal of General Virology. 72 (6), 1215-1221 (1991).
- Boulton, M. I., Buchholz, W. G., Marks, M. S., Markham, P. G., Davies, J. W. Specificity of Agrobacterium-mediated delivery of maize streak virus DNA to members of the Gramineae. Plant Molecular Biology. 12 (1), 31-40 (1989).
- Paulsen, A. Q. Purification and properties of foxtail mosaic virus. Phytopathology. 77 (11), 1346(1977).
- Bancroft, J. B., Rouleau, M., Johnston, R., Prins, L., Mackie, G. A. The entire nucleotide sequence of foxtail mosaic virus RNA. Journal of General Virology. 72 (9), 2173-2181 (1991).
- Bruun-Rasmussen, M., Madsen, C. T., Johansen, E., Albrechtsen, M. Revised sequence of foxtail mosaic virus reveals a triple gene block structure similar to potato virus X. Archives of Virology. 153 (1), 223-226 (2008).
- Rouleau, M., Bancroft, J. B., Mackie, G. A. Partial purification and characterization of foxtail mosaic potexvirus RNA-dependent RNA polymerase. Virology. 197 (2), 695-703 (1993).
- Rouleau, M., Smith, R. J., Bancroft, J. B., Mackie, G. A. Purification, properties, and subcellular localization of foxtail mosaic potexvirus 26-kDa protein. Virology. 204 (1), 254-265 (1994).
- Samuels, T. D., et al. Subcellular targeting and interactions among the potato virus X TGB proteins. Virology. 367 (2), 375-389 (2007).
- Cho, S. Y., Kim, K. H. Identification of the capsid protein-binding region of the SL1(+) RNA located at the 5' region of the potato virus X genome. Plant Pathology Journal. 28 (1), 75-80 (2012).
- Mei, Y., Zhang, C., Kernodle, B. M., Hill, J. H., Whitham, S. A. A foxtail mosaic virus vector for virus-induced gene silencing in maize. Plant Physiology. 171 (2), 760-772 (2016).
- Bouton, C., et al. Foxtail mosaic virus: A viral vector for protein expression in cereals. Plant Physiology. 177 (4), 1352-1367 (2018).
- Mei, Y., Liu, G., Zhang, C., Hill, J. H., Whitham, S. A. A sugarcane mosaic virus vector for gene expression in maize. Plant Direct. 3 (8), 00158(2019).
- Gal-On, A., Meiri, E., Huet, H., Hua, W. J., Raccah, B., Gaba, V. Particle bombardment drastically increases the infectivity of cloned DNA of zucchini yellow mosaic potyvirus. Journal of General Virology. 76 (12), (1995).
- Gao, R., et al. Construction of an infectious cDNA clone and gene expression vector of Tobacco vein banding mosaic virus (genus Potyvirus). Virus Research. 169 (1), 276-281 (2012).
- López-Moya, J. J., García, J. A. Construction of a stable and highly infectious intron-containing cDNA clone of plum pox potyvirus and its use to infect plants by particle bombardment. Virus Research. 68 (2), (2000).
- Choi, I. R., French, R., Hein, G. L., Stenger, D. C. Fully biologically active in vitro transcripts of the eriophyid mite-transmitted wheat streak mosaic tritimovirus. Phytopathology. 89 (12), (1999).
- Kim, K. S., et al. Infectivity of in vitro transcripts of Johnsongrass mosaic potyvirus full-length cDNA clones in maize and sorghum. Archives of Virology. 148 (3), 563-574 (2003).
- Stewart, L. R., Bouchard, R., Redinbaugh, M. G., Meulia, T. Complete sequence and development of a full-length infectious clone of an Ohio isolate of Maize dwarf mosaic virus (MDMV). Virus Research. 165 (2), 219-224 (2012).
- Wylie, S. J., et al. ICTV virus taxonomy profile: Potyviridae. Journal of General Virology. 98 (3), 352-354 (2017).
- Shukla, D. D. taxonomy of potyviruses infecting maize, sorghum, and sugarcane in Australia and the United States as determined by reactivities of polyclonal antibodies directed towards virus-specific N-termini of coat proteins. Phytopathology. 79 (2), 223(1989).
- Shukla, D. D., Ward, C. W. Amino Acid sequence homology of coat proteins as a basis for identification and classification of the potyvirus group. Journal of General Virology. 69 (11), 2703-2710 (1988).
- Chung, B. Y. W., Miller, W. A., Atkins, J. F., Firth, A. E. An overlapping essential gene in the Potyviridae. Proceedings of the National Academy of Sciences of the United States of America. 105 (15), 5897-5902 (2008).
- Jarchow, E., Grimsley, N. H., Hohn, B. virF, the host-range-determining virulence gene of Agrobacterium tumefaciens, affects T-DNA transfer to Zea mays. Proceedings of the National Academy of Sciences of the United States of America. 88 (23), 10426-10430 (1991).
- Hu, G., Yalpani, N., Briggs, S. P., Johal, G. S. A porphyrin pathway impairment is responsible for the phenotype of a dominant disease lesion mimic mutant of maize. The Plant Cell. 10 (7), 1095(2007).
- Qin, G., et al. Disruption of phytoene desaturase gene results in albino and dwarf phenotypes in Arabidopsis by impairing chlorophyll, carotenoid, and gibberellin biosynthesis. Cell Research. 17 (5), 471-482 (2007).
- Jones, P. Isolation of plasmid DNA from E. coli. Encyclopedia of Life Sciences. , (2003).
- Ji, Q., Yang, B., Lee, M., Chen, Y., Lübberstedt, T. Mapping of quantitative trait loci/locus conferring resistance to foxtail mosaic virus in maize using the intermated B73-×-Mo17 population. Plant Breeding. 129 (6), 721-723 (2010).
- Pacak, A., et al. The brome mosaic virus-based recombination vector triggers a limited gene silencing response depending on the orientation of the inserted sequence. Archives of Virology. 155 (2), 169-179 (2010).
- Miché, L., Battistoni, F., Gemmer, S., Belghazi, M., Reinhold-Hurek, B. Host-dependent expression of Rhizobium leguminosarum bv. viciae hydrogenase is controlled at transcriptional and post-transcriptional levels in legume nodules. Molecular Plant-Microbe Interactions. 19 (5), 1323-1331 (2018).
- Yamagishi, M., Masuta, C., Suzuki, M., Netsu, O. Peanut stunt virus-induced gene silencing in white lupin (lupinus albus). Plant Biotechnology. 32 (3), 181-191 (2015).
- Avesani, L., et al. Stability of Potato virus X expression vectors is related to insert size: Implications for replication models and risk assessment. Transgenic Research. 16 (5), 587-597 (2007).
- Ali, Z., et al. Efficient virus-mediated genome editing in plants using the CRISPR/Cas9 system. Molecular Plant. 8 (8), 1288-1291 (2015).
- Cody, W. B., Scholthof, H. B., Mirkov, T. E. Multiplexed gene editing and protein overexpression using a tobacco mosaic virus viral vector. Plant Physiology. 175 (1), 23-35 (2017).
- Ali, Z., Eid, A., Ali, S., Mahfouz, M. M. Pea early-browning virus-mediated genome editing via the CRISPR/Cas9 system in Nicotiana benthamiana and Arabidopsis. Virus Research. 244, 333-337 (2018).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten