Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Eine robuste Entdeckungsplattform zur Identifizierung neuartiger Mediatoren der Melanommetastasierung
In diesem Artikel
Zusammenfassung
Dieser Artikel beschreibt einen Workflow von Techniken, die zum Testen neuartiger Kandidatenmediatoren von Melanommetastasen und deren Wirkmechanismus eingesetzt werden.
Zusammenfassung
Metastasierung ist ein komplexer Prozess, bei dem Zellen Barrieren überwinden müssen, die nur unvollständig durch In-vitro-Assays modelliert werden. Ein systematischer Workflow wurde unter Verwendung robuster, reproduzierbarer In-vivo-Modelle und standardisierter Methoden etabliert, um neue Akteure in der Melanommetastasierung zu identifizieren. Dieser Ansatz ermöglicht Datenrückschlüsse in bestimmten experimentellen Stadien, um die Rolle eines Gens bei Metastasen genau zu charakterisieren. Modelle werden erstellt, indem genetisch veränderte Melanomzellen über intrakardiale, intradermale oder subkutane Injektionen in Mäuse eingeführt werden, gefolgt von einer Überwachung mit serieller In-vivo-Bildgebung . Sobald vordefinierte Endpunkte erreicht sind, werden Primärtumoren und/oder metastasentragende Organe entnommen und für verschiedene Analysen verarbeitet. Tumorzellen können sortiert und einer von mehreren "Omics"-Plattformen unterzogen werden, einschließlich der Einzelzell-RNA-Sequenzierung. Organe werden bildgebenden und immunhistopathologischen Analysen unterzogen, um die Gesamtbelastung durch Metastasen zu quantifizieren und ihren spezifischen anatomischen Standort abzubilden. Diese optimierte Pipeline, einschließlich standardisierter Protokolle für Transplantation, Überwachung, Gewebeernte, -verarbeitung und -analyse, kann für patientenabgeleitete, kurzfristige Kulturen und etablierte menschliche und murine Zelllinien verschiedener solider Krebsarten übernommen werden.
Einleitung
Die hohe Mortalität im Zusammenhang mit metastasierendem Melanom in Kombination mit einer zunehmenden Inzidenz von Melanomen weltweit 1 (geschätzter Anstieg von 7,86% bis2025) erfordert neue Behandlungsansätze. Fortschritte in der Zielentdeckung hängen von reproduzierbaren Metastasenmodellen ab, einem hochkomplexen Prozess. Während der Schritte der metastasierenden Kaskade müssen Melanomzellen unzählige Barrieren überwinden, um eine Umgehung des Immunsystems und die Besiedlung entfernter Gewebe zu erreichen2. Die Widerstandsfähigkeit und Anpassungsfähigkeit von Melanomzellen ergibt sich aus einer Vielzahl von Faktoren, darunter ihre hohe genetische Mutationslast 3 und ihr Neuralkammursprung, die eine entscheidende phänotypische Plastizitätverleihen 3,4,5. Bei jedem Schritt ermöglichen Transkriptionsprogramme es metastasierenden Melanomzellen, von einem Zustand in einen anderen zu wechseln, basierend auf Hinweisen aus dem Übersprechen mit der Mikroumgebung, bestehend aus dem Immunsystem6, dem extrazellulären Milieu7,8 und der zellulären Architektur der physischen Barrieren9, mit denen sie in Kontakt kommen. Zum Beispiel entziehen sich Melanomzellen der Immunüberwachung, indem sie die Expression wichtiger immunprimierender tumorsezernierter Faktoren herunterregulieren6.
Studien beschreiben eine "prämetastasierende Nische", in der Melanomzellen Chemokine und Zytokine absondern, um das entfernte "Zielorgan" für Metastasen10 vorzubereiten. Diese Ergebnisse werfen wichtige Fragen über den Organtropismus metastasierender Melanomzellen und den anatomischen Weg auf, den sie nehmen, um auf entferntes Gewebe zuzugreifen. Nach der Intravasation ist bekannt, dass Melanomzellen durch Lymphatika (lymphatische Ausbreitung) und Blutgefäße (hämatogene Ausbreitung) metastasieren2,11. Während die meisten Patienten eine lokalisierte Erkrankung aufweisen, weist eine kleine Untergruppe von Fällen eine ferne metastasierende Erkrankung und keine lymphatische Verbreitung (negative Lymphknotenbeteiligung) auf11, was auf die Existenz alternativer metastasierender Wege für Melanome hindeutet.
Wenn sie eine metastasierende Stelle besiedeln, durchlaufen Melanomzellen epigenetische und metabolische Anpassungen12,13. Um auf neue Kompartimente zuzugreifen und in sie einzudringen, verwenden Melanomzellen Proteasen 14 und zytoskelettale Modifikationen 11,15, die es ihnen ermöglichen, zu ihrem neuen Standort zu gelangen und dort zu wachsen. Die Schwierigkeit bei der Bekämpfung von Melanomzellen liegt in der Komplexität und Anzahl solcher Anpassungen; Daher sollte sich das Feld bemühen, so viele Schritte und Anpassungen wie möglich experimentell nachzubilden. Trotz zahlreicher Fortschritte in in vitro Assays wie Organoiden und 3D-Kulturen16,17 rekapitulieren diese Modelle die in vivo metastatische Kaskade nur unvollständig.
Murine-Modelle haben sich als wertvoll erwiesen, indem sie ein Gleichgewicht zwischen Reproduzierbarkeit, technischer Machbarkeit und Simulation menschlicher Krankheiten gefunden haben. Intravaskuläre, orthotopisch und heterotopisch implantierte Melanomzellen aus patientenabgeleiteten Xenotransplantaten oder Kurzzeitkulturen in immungeschwächte oder humanisierte Mäuse stellen das Rückgrat der Zielentdeckung bei metastasierendem Melanom dar. Diesen Systemen fehlt jedoch oft eine entscheidende biologische Einschränkung der Metastasierung: das Immunsystem. Syngene Melanommetastasenmodelle, die diese Einschränkung besitzen, sind auf dem Gebiet relativ selten. Diese Systeme, die in immunkompetenten Mäusen entwickelt wurden, einschließlich B16-F1018, der YUMM-Familie der Zelllinien19, SM1 20, D4M3 21, RIM3 22 oder neuer, der RMS 23 und M1 (Mel114433), M3 (HCmel1274), M4 (B2905) 24 Melanom-Zelllinien, erleichtern die Untersuchung der komplexen Rolle der Immunantwort des Wirts bei der Melanomprogression.
Hier wird eine Pipeline zur Identifizierung von Melanommetastasenzielen vorgestellt. Mit zunehmenden und größeren "Omics" -Datensätzen, die aus Melanom-Patientenkohorten generiert werden, postulieren wir, dass Studien, die das klinischste Versprechen haben, diejenigen sind, die aus der Big-Data-Integration stammen, was zu einer akribischen funktionellen und mechanistischen Befragung führt25,26,27,28. Durch die Verwendung von Mausmodellen zur Untersuchung potenzieller Ziele im metastatischen Prozess können in vivo-spezifische Ereignisse und Gewebeinteraktionen berücksichtigt werden, wodurch die Wahrscheinlichkeit einer klinischen Translation erhöht wird. Mehrere Methoden zur Quantifizierung der metastatischen Belastung werden skizziert und liefern ergänzende Daten zu den Ergebnissen eines bestimmten Experiments. Ein Protokoll für die Einzelzellisolierung von Tumoren in verschiedenen Organen wird beschrieben, um die unvoreingenommene Charakterisierung der Genexpression in metastasierenden Zellen zu unterstützen, die der Einzelzell- oder Massen-RNA-Sequenzierung vorausgehen kann.
Protokoll
HINWEIS: Die Tierverfahren, die mit dem folgenden Protokoll verbunden sind, wurden vom New York University Institutional Animal Care and Use Committee (IACUC) genehmigt. Alle Verfahren werden in Einrichtungen durchgeführt, die von der Association for Assessment and Accreditation of Laboratory Animal Care International (AAALAC) zugelassen sind. Abbildung 1 zeigt den allgemeinen experimentellen Ansatz.
1. Von Patienten abgeleitete Melanom-Kurzzeitkulturen (STCs)
- Geben Sie das Gewebe in eine 60 mm große Petrischale mit 1 ml vollständigem RPMI (RPMI 1640 ergänzt mit 10% fetalem Rinderserum (FBS), 2 mM L-Glutamin, 1 mM Natriumpyruvat, 1x MEM nicht-essentieller Aminosäurelösung und Penicillin (100 IE / ml ) / Streptomycin (100 μg / ml)).
HINWEIS: Um das Verhältnis der Tumorzellen zu erhöhen, analysieren und entfernen Sie bei Bedarf das den Tumor umgebende Gewebe in der Petrischale unter einem Mikroskop mit sterilen chirurgischen Instrumenten. - Schneiden Sie das frische Gewebe mit sterilisierten Rasierklingen fein in 1-2 mm große Würfel. Fügen Sie 4 ml vollständige RPMI hinzu und pipettieren Sie den Inhalt der Platte 5-10 Mal mit einer serologischen Pipette von 10 ml auf und ab.
- Übertragen Sie die Zellsuspension auf ein 15 ml Polypropylen-Konionsröhrchen und drehen Sie die Zellen nach unten (180 × g für 5 min bei 4 ° C). Saugen Sie den Überstand ab, suspendieren Sie das Zellpellet in 1 ml frischem Medium und übertragen Sie die Suspension in einen 25 cm2 großen Gewebekulturkolben.
- Um den Gewebefragmenten zu helfen, sich am Boden festzusetzen, stellen Sie den Kolben in einem Gewebekultur-Inkubator in einem Gewebekultur-Inkubator in einem Winkel von 20 ° -30 ° C auf 5% CO2 für 20 min gekippt.
- Legen Sie den Kolben flach ab, damit das Medium das Gewebe bedecken kann, und überprüfen Sie täglich den Status der Kultur. Teilen Sie die Zellen auf, wenn sie eine 90-100% ige Konfluenz erreichen. Halten Sie kurzfristige Kulturen bei einer "niedrigen" Durchgangszahl aufrecht.
HINWEIS: STCs werden etwa 2 Monate nach der Zellisolierung und -kultur eingerichtet, obwohl der tatsächliche Zeitplan zwischen Proben und Tumorarten variiert. Nach 10 bis 14 Passagen erreichen die Zelllinien eine Reinheit von 100% und enthalten nur Melanomzellen29. Die Passage Number Threshold wird empirisch bestimmt, indem Veränderungen in der Zellmorphologie, der Verdopplungszeit und des Verhaltens in vivo beobachtet werden. Um die Heterogenität und andere Merkmale des Elterntumors zu erhalten, teilen Sie die Zellen nicht mehr als 1:5 auf. - Nach der Etablierung eines STC und mit einem Zelllinienmodell, das in die Tiere injiziert werden soll, wie in den folgenden Schritten beschrieben, transduzieren Sie Zellen mit einem Reporter.
HINWEIS: Ein fluoreszierendes Tag (z. B. rotfluoreszierendes Protein (RFP), grün fluoreszierendes Protein (GFP)) ermöglicht beispielsweise die Ex-vivo-Immunfluoreszenzbildgebung und die Sortierung von Tumorzellen durch fluoreszenzaktivierte Zellsortierung (FACS). Luciferase ermöglicht in vivo Biolumineszenz-Bildgebung, ein nützliches Werkzeug zur Überwachung der experimentellen Progression (Abschnitt 4).
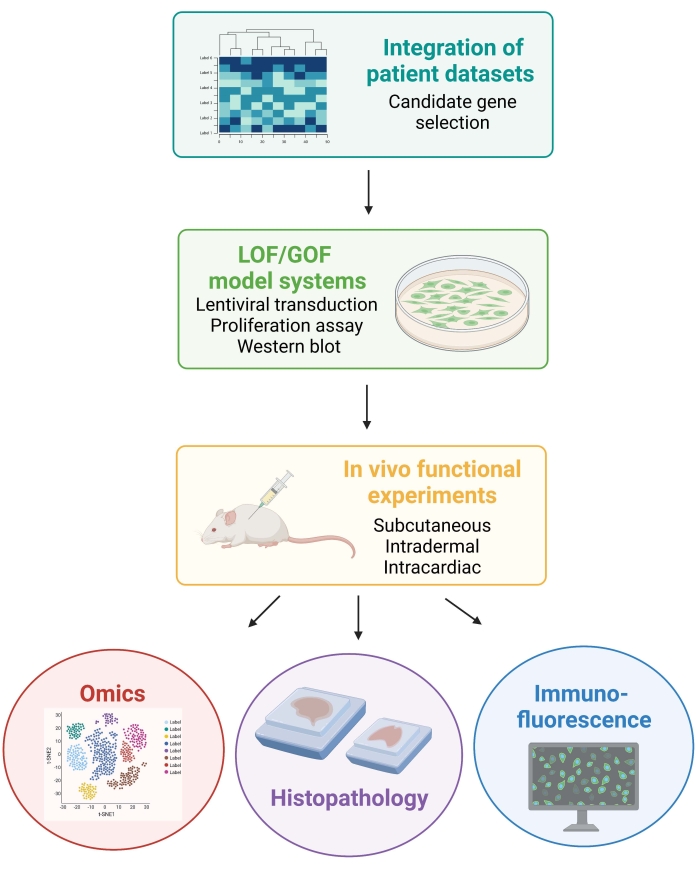
Abbildung 1: Schematische Darstellung des beschriebenen Workflows, von der Patientendatenintegration bis zur Generierung und Analyse von In-vivo-Daten von Mäusen. Abkürzungen: LOF = Funktionsverlust; GOF = Funktionsgewinn. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
2. Xenograft-Implantation
HINWEIS: Die hier beschriebenen experimentellen Verfahren werden an Mäusen durchgeführt, die ein geschwächtes adaptives und angeborenes Immunsystem haben, NOD.Cg-PrkdcscidIl2rg tm1Wjl/SzJ (NSG) Mäuse; oder bei Mäusen, denen nur eine adaptive Immunität fehlt, wie die T-Zell-defizienten athymischen/nackten (NU/J) Mäuse. Tiere sind männlichen Geschlechts, 8 bis 10 Wochen alt. Frauen zeigen oft eine hohe Inzidenz von Gonadenmetastasen bei intrakardialer Injektion von Tumorzellen, was ihr Überleben reduziert.
- Für subkutane und intradermale Injektionen ist eine 1:1-Zellsuspension herzustellen, indem ein Teil der in 1x Dulbeccos Phosphate-Buffered Saline (DPBS) suspendierten Zellen mit einem Teil aufgetautem extrazellulärem Matrixsubstrat (EMS) gemischt und bei 4 °C auf Eis gehalten wird. Für intravaskuläre (intrakardiale, intrakarotide, retro-orbitale, Schwanzvenen- oder splenische) Injektionen suspendieren Sie die Zellen nur in DPBS.
HINWEIS: Das geeignete Volumen für intradermale Injektionen sollte so gering wie möglich gehalten werden (30 μL). Bei subkutanen Injektionen kann das injizierte Volumen bis zu 150 μL und bei intravaskulären Injektionen bis zu 250 μL (basierend auf dem Gewicht des Tieres) betragen. Fügen Sie der endgültigen Zellsuspension 10-30% zusätzliches Injektionsvolumen hinzu, basierend auf der injizierten Menge und der verwendeten Spritze, um das tote Volumen in der Schlupfspitze und das der Nadel zu berücksichtigen (z. B. eine 1 ml Tuberkulinspritze mit einer 30 G, 25mm Nadel hat ein totes Volumen von 100 μL). - Führen Sie ein Pilotprojekt durch, um das Verhalten der verwendeten Zelllinien und den Zeitplan der Tumorprogression in vivo zu charakterisieren. Für intradermale Injektionen beginnen Sie mit der Injektion von 1.000 bis zu 50.000 Zellen / 30 μL. Beginnen Sie bei subkutanen Injektionen mit der Injektion von 10.000 bis zu 2 × 106 Zellen/150 μL. Für intravaskuläre (intrakardiale, intrakarotide, retro-orbitale und splenische) Injektionen beginnen Sie mit der Injektion von 50.000 Zellen / 150 μL.
HINWEIS: Intravaskuläre Injektionen prädisponieren die Tiere für embolische Ereignisse, entweder durch Einbringen von Luft in das Kreislaufsystem oder durch Verwendung einer übermäßigen Anzahl von Zellen, die die kleinen Gefäße verschließen. Mischen Sie die Zellsuspension gut, um ein Verklumpen zu vermeiden. Grundieren Sie die Spritze, bevor Sie die Zellsuspension laden. Entfernen Sie alle Luftblasen in der Spritze. Bewahren Sie die Zellsuspension/Spritzen bis zum Laden und zum Zeitpunkt der Injektion auf Eis auf. - Verabreichen Sie die Anästhesie durch Inhalation. Stellen Sie den Sauerstoffmessregler zwischen 1-2 l / min ein. Legen Sie das Tier in die Induktionskammer, wobei der Isofluran-Verdampfer auf 2,5-5% für die Induktion und 1,5-3% für die Wartung eingestellt ist.
HINWEIS: Überwachen Sie die Atmung und Herzfrequenz des Tieres während der Anästhesie-Induktionsphase. Lassen Sie das Tier nicht unbeaufsichtigt. Überwachen Sie nicht mehr als ein Tier gleichzeitig. Titern Sie die Menge der Anästhesie auf das Gewicht des Tieres. - Bewegen Sie das Tier aus der Induktionskammer in den Nasenzapfen. Tragen Sie sterile petrolatum ophthalmologische Salbe auf die Augen des Tieres auf, um Hornhauttrockenheit während des Eingriffs zu verhindern.
- Rasieren Sie die Verfahrensstelle mit einer geraden Rasierklinge, die in einem Winkel von 30 ° geneigt ist. Reinigen Sie die Haut des Verfahrensbereichs mit 70% Isopropylalkohol-Tupfern. Beurteilen Sie vor weiteren Schritten eine ausreichende Anästhesie durch Pedalreflex.
- Bei intradermalen Injektionen führen Sie das gesamte Verfahren in einer Biosicherheitskabine durch, um aseptische Bedingungen aufrechtzuerhalten.
- Betäuben und rasieren Sie das Tier wie in den Schritten 2.3-2.5 beschrieben.
- Greifen und ziehen Sie die Haut rückwärts gegen die Flugbahn des Nadelstichs zurück. Mit einer 31 G Insulinspritzennadel, 6 mm lang, in einem spitzen Winkel gehalten, punktieren Sie die Haut sanft mit der Fase nach oben.
- Spüren Sie die Druckentlastung an der Spitze der Nadel. Gehen Sie vorsichtig vor, um im intradermalen Kompartiment zu bleiben und nicht durch die gesamte Hauttiefe in die Unterhaut zu gelangen. Entscheidend: Wenn man in den subkutanen Raum schlüpft, entfernen Sie die Nadel, wechseln Sie den Injektionsbereich und setzen Sie die Nadel wieder ein. Injizieren Sie das gesamte Volumen (30 μL) der Zellsuspension langsam, bis eine kuppelförmige Quaddel beobachtet wird.
HINWEIS: Geringe Injektionsvolumina induzieren weniger Dissektion der Hautschichten und weniger architektonische Verzerrungen. - Halten Sie die Nadel drin und zählen Sie bis 5.
HINWEIS: EMS wird bei Körpertemperatur viskos und hilft, Rückfluss durch die Nadelstichwunde zu vermeiden. - Entfernen Sie die Nadel und legen Sie das Tier in einem Käfig auf einem warmen Pad auf, um sich zu erholen. Bringen Sie das Tier in den Vivariumkäfig zurück, nachdem Sie das Bewusstsein wiedererlangt haben, wenn es sternal und gehfähig ist.
HINWEIS: Überwachen Sie das Tier kontinuierlich während aller in diesem Protokoll beschriebenen Verfahren. Lassen Sie das Tier nicht unbeaufsichtigt und überwachen Sie nicht mehr als ein Tier gleichzeitig. - Überwachen Sie das Fortschreiten des Tumorwachstums, des Gewichtsverlusts und des allgemeinen Gesundheitszustands in Zusammenarbeit mit dem tierärztlichen Personal täglich in der Anfangswachstumsphase und bei Bedarf intensiver, nachdem die Tiere mit dem Abnehmen begonnen haben. Während dieser Überwachungssitzungen: Wiegen Sie die Tiere und zeichnen Sie ein Diagramm, um den Gewichtsverlust zu überwachen, und überprüfen Sie auf Anzeichen von Tumorgeschwüren, neurologischen, lokomotorischen und / oder Verhaltensmerkmalen (Lethargie, mangelnde Pflege, geringe Nahrungs- oder Wasseraufnahme).
HINWEIS: Euthanasie die Tiere unmittelbar nach der Beobachtung von Anzeichen einer fortgeschrittenen Krankheit (mehr als 20% Gewichtsverlust, ein Körperzustandswert von <2, extrem reduzierte Aktivitätsniveaus, Lähmungen oder Krampfanfälle). Verwenden Sie die von der IACUC der Institution genehmigte Euthanasiemethode (z. B. wird eine automatisierte Tisch-CO 2-Kammer verwendet, um die Tiere 15 Minuten lang CO2 auszusetzen, gefolgt von einer sekundären Euthanasiemethode, entweder zervikale Dislokation, Enthauptung oder Pneumothorax, der bilateral durch Einschneiden desBrustkorbs induziert wird). - Nehmen Sie Messungen mit Messschiebern vor und verwenden Sie die Länge (L) und die Breite (W) des Tumors, um das Volumen (V) mit der Formel zu berechnen:
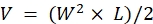
- Für subkutane Injektionen:
- Führen Sie das gesamte Verfahren in einer Biosicherheitskabine durch, um aseptische Bedingungen aufrechtzuerhalten26,27.
- Betäuben und rasieren Sie das Tier wie in den Schritten 2.3-2.5 beschrieben.
- Mit einer 6 mm langen Insulinspritzennadel von 28 g bis 31 g, die in einem spitzen Winkel gehalten wird, stichen Sie sanft die Haut mit der Fase nach oben ein. Spüren Sie die Druckentlastung an der Spitze der Nadel zweimal , während Sie durch die Epidermis, Dermis und Hypodermis gehen.
HINWEIS: Das zweite Mal, wenn eine Druckentlastung an der Spitze der Nadel gefühlt wird, zeigt an, dass das subkutane Kompartiment erreicht wurde. - Injizieren Sie das gesamte Volumen (30-150 μL) der Zellsuspension langsam, bis eine längliche ellipsenförmige Quaddel beobachtet wird. Halten Sie die Nadel drin und zählen Sie bis 5. Zählen Sie bis 10 für größere Volumina (mehr als 50 μL).
HINWEIS: EMS wird bei Körpertemperatur viskos und hilft, Rückfluss durch die Nadelstichwunde zu vermeiden. - Entfernen Sie die Nadel und legen Sie das Tier in einem Käfig auf einem warmen Pad auf, um sich zu erholen. Bringen Sie das Tier in seinen Vivariumkäfig zurück, nachdem Sie das Bewusstsein wiedererlangt haben, wenn es sternal und gehfähig ist.
HINWEIS: Achten Sie während der postprozeduralen Überwachung auf Anzeichen von Komplikationen (niedrige Atemfrequenz, Blutungen, langsame Genesung) und behandeln Sie sie angemessen. Wenn keine Besserung beobachtet wird, fahren Sie mit humanen Euthanasieverfahren fort, die in der ANMERKUNG zu Schritt 2.6.6 beschrieben sind. - Überwachen Sie das Tier auf Tumorwachstum, Gewichtsverlust und allgemeinen Gesundheitszustand, wie in den Schritten 2.6.6-2.6.7 beschrieben.
- Für intrakardiale Injektionen:
- Führen Sie das gesamte Verfahren in einer Biosicherheitskabine durch, um aseptische Bedingungen aufrechtzuerhalten26,30.
- Betäuben Sie das Tier wie in den Schritten 2.3-2.4 beschrieben.
- Bringen Sie das Tier auf die beheizte Plattform des Ultraschallgeräts und befestigen Sie es mit hypoallergenem Klebeband am Nasenkonus.
- Rasieren Sie den Thorax mit einer geraden Rasierklinge, die in einem Winkel von 30 ° geneigt ist. Reinigen Sie die Haut des Verfahrensbereichs mit 3 Anwendungen von 10% Povidon-Jod im Wechsel mit 3 Anwendungen von Isopropylalkohol.
- Beurteilen Sie vor weiteren Schritten eine ausreichende Anästhesie durch Pedalreflex. Tragen Sie das Ultraschallgel auf die Verfahrensstelle auf.
- Erfassen Sie das Herzfenster mit der Ultraschallsonde. Positionieren Sie die Ultraschallsonde in der Mitte des Thorax auf der linken Seite des Tieres, um ein horizontales Fenster zu erfassen, das auf eine Querschnittsansicht (kurze Achse) des linken Ventrikels ausgerichtet ist. Stellen Sie sicher, dass die Längsachse der Sonde nach oben zeigt, und fixieren Sie die Sonde in einem Winkel von 50 ° und die beheizte Plattform in einem Winkel von 20 °. Verriegeln Sie die Sonde und den Stützrahmen in Position.
- Ziehen Sie die Zellsuspension auf, während Sie in der Biosicherheitskabine in einer Tuberkulin-1-ml-Spritze mit einer 30 G, 25-mm-Nadel arbeiten. Entfernen Sie alle Luftblasen in der Spritze.
HINWEIS: Es ist wichtig, eine Einzelzellsuspension zu erstellen und beizubehalten, während die Zellen verarbeitet und injiziert werden. Das Entfernen von Luftblasen ist ein wichtiger Schritt, um Luftembolien zu vermeiden. Ein gut grundiertes Spritzennadelsystem verhindert vermeidbare Todesfälle in der experimentellen Gruppe. Ziehen Sie immer mehr Volumen in die Spritze, als injiziert wird. Das zusätzliche Volumen hilft, die Luft zu entfernen, indem ein Teil der Zellsuspension wieder in eine 1,5-ml-Röhre injiziert wird. - Verriegeln Sie die Spritze im stereotaktischen Injektor. Führen Sie die Nadel unter Ultraschallführung durch die Brustwand in den linken Ventrikel des Herzens. Injizieren Sie das gesamte Volumen (100-250 μL) der Zellsuspension langsam.
- Entfernen Sie die Nadel und legen Sie das Tier in einem Käfig auf einem warmen Pad auf, um sich zu erholen. Bringen Sie das Tier in seinen Vivariumkäfig zurück, nachdem Sie das Bewusstsein wiedererlangt haben, wenn es sternal und gehfähig ist. Überwachen Sie das Tier auf Tumorwachstum, Gewichtsverlust und allgemeinen Gesundheitszustand, wie in Schritt 2.6.6 beschrieben.
- Für intrakarotis-Injektionen:
- Führen Sie das gesamte Verfahren auf einer ordnungsgemäß desinfizierten Oberfläche durch, um die aseptischen Bedingungen aufrechtzuerhalten30.
- Betäuben Sie das Tier mit einem Cocktail aus Ketamin (100 mg/kg) und Xylazin (10 mg/kg) durch intraperitoneale Injektion mit einer Insulinspritze, 28 G Nadel. Tragen Sie sterile petrolatum ophthalmologische Salbe auf die Augen des Tieres auf, um Hornhauttrockenheit während des Eingriffs zu verhindern.
- Rasieren Sie den Verfahrensbereich mit einer geraden Rasierklinge, die in einem Winkel von 30 ° geneigt ist. Beurteilen Sie vor weiteren Schritten eine ausreichende Anästhesie durch Pedalreflex.
- Legen Sie das Tier unter ein Stereomikroskop auf ein wärmendes Pad. Reinigen Sie die Haut des Verfahrensbereichs mit 3 Anwendungen von 10% Povidon-Jod im Wechsel mit 3 Anwendungen von Isopropylalkohol.
- Tragen Sie sterile persönliche Schutzausrüstung (PSA) und sterile Handschuhe. Bereiten Sie das sterile Feld vor, indem Sie einen sterilen Vorhang über den Körper des Tieres legen.
HINWEIS: Wenn der sterile Vorhang kein Loch hat, das für die Größe und Lage des Einschnitts geeignet ist, falten Sie den Vorhang in zwei Hälften und schneiden Sie mit einer Metzenbaum-Schere das Loch in der Mitte des sterilen Vorhangs in die entsprechende Größe. - Verwenden Sie ein Skalpell oder eine Irisschere, um die Haut vom halben Hals bis zum Brustbein zu schneiden. Mit zwei mikrochirurgischen Pinzetten die 2 submandibulären Speicheldrüsen in der Mittellinienebene stumpf auseinander schneiden. Verwenden Sie bei Bedarf eine elektrische Kauterisation für die Hämostase.
- Sezieren Sie die Faszie, die die gemeinsame Halsschlagader (CCA) vom Manubrium in Richtung der Bifurkation umgibt, und fahren Sie medial fort, um die hintere Wand der äußeren Halsschlagader zu befreien. Befestigen Sie die äußere Halsschlagader (ECA) vor der Injektion vorübergehend.
HINWEIS: Bei der Sezierung um den Umfang des CCA muss darauf geachtet werden, dass der Vagusnerv (liegt seitlich zur Arterie) nicht beschädigt wird. - Laden Sie die Zellsuspension in eine 1 ml Spritze mit einer 33 G, 15 mm Nadel.
- Führen Sie zwei 7-0-Ligaturen unter der CCA durch und führen Sie für jede der beiden Ligaturen einen losen Instrumentenknoten aus. Verwenden Sie einen 5 mm, 10 G Druckbehälterclip und klemmen Sie den ECA vorübergehend an. Binden Sie die proximale Ligatur; dann binden Sie die distale Ligatur locker (neben der Verzweigung der CCA). Verwenden Sie die distale Schleife später, um die Blutung nach der Injektion zu kontrollieren.
- Verwenden Sie die Spritze mit einer 33 G, 15 mm Nadel, punktieren Sie vorsichtig die CCA mit der Fase der Nadel nach oben und in einem spitzen Winkel. Injizieren Sie das gesamte Volumen (50-150 μL) der Zellsuspension langsam.
- Greifen Sie die distale Schlaufe mit der Pinzette und heben Sie sie an, während Sie die Nadel entfernen, um das Lumen der CCA zu verschließen und die Blutung zu stoppen. Tauschen Sie die Spritze gegen eine #7 Juweliere-Pinzette aus und binden Sie die distale Schlaufe fest.
- Werfen Sie einen weiteren Instrumentenknoten auf die distale Ligatur und entfernen Sie den Gefäßclip von der ECA. Kontrollieren Sie das Operationsfeld für Blutungen und kauterisieren Sie alle blutenden Gefäße vor dem Schließen. Verwenden Sie eine 9-mm-Heftvorrichtung, um die Haut des Tieres zu schließen, und legen Sie das Tier auf ein warmes Pad, um sich zu erholen.
HINWEIS: Entfernen Sie die Klammern 7-10 Tage nach der Operation. - Verabreichen Sie Analgetika subkutan - Buprenorphin (0,3 mg / ml) alle 12 h für 72 h nach der Operation in einer Konzentration von 0,1 mg / kg.
HINWEIS: Alternativ können Sie ein Analgetikum mit verlängerter Freisetzung verwenden, das alle 72 Stunden 1 Dosis erfordert. - Bringen Sie das Tier in seinen Vivariumkäfig zurück, nachdem Sie das Bewusstsein wiedererlangt haben, wenn es sternal und gehfähig ist. Überwachen Sie die Tiere täglich postoperativ auf Anzeichen einer Infektion oder Schmerzen an der Operationsstelle, einen allgemeinen Gesundheitszustand und Komplikationen.
HINWEIS: Tiere, die sich von einer Überlebensoperation nicht gut erholen, können zusätzliche Dosen von Schmerzmitteln erhalten und werden human eingeschläfert, wenn sie sich nicht bis 72 Stunden nach der Operation vollständig erholen. - Überwachen Sie das Tier auf Tumorwachstum, Gewichtsverlust und allgemeinen Gesundheitszustand, wie in Schritt 2.6.6 beschrieben.
- Für retroorbitale Injektionen:
HINWEIS: Verwenden Sie diese Technik als Alternative zu Heckveneninjektionen, wenn der Bediener in dieser Technik geschult und kompetent ist und wenn es eine starke wissenschaftliche Begründung gibt. Zellsuspensionen, die über diesen Weg abgegeben werden, können Tumorwachstum im retroorbitalen Raum induzieren; Daher sollten bei der Wahl dieser Technik die Risiken und Vorteile sorgfältig berücksichtigt werden. Um beispielsweise die direkte zirkulatorische Verbindung des retroorbitalen Veneninus mit den intrazerebralen Venenvenen über Anastomosen zu nutzen, wählen Sie diese Methode, wenn die Hirntumorbildung auf anderen Injektionswegen fehlgeschlagen ist.- Führen Sie das gesamte Verfahren in einer Biosicherheitskabine durch, um aseptische Bedingungen aufrechtzuerhalten. Ziehen Sie sterile PSA und Handschuhe an.
- Betäuben Sie das Tier wie in den Schritten 2.3-2.4 beschrieben.
HINWEIS: Tragen Sie bei diesem Verfahren keine sterile petrolatum-Augensalbe auf die Augen des Tieres auf, da dies die Injektion behindert. Tragen Sie nur Lokalanästhesietropfen auf. - Laden Sie die Zellsuspension in eine Insulinspritze mit einer 28-31 G, 6 mm Nadel.
- Wenn sich das Tier in Bauchlage befindet, ziehen Sie die Augenlider zurück, bis das Auge hervorsteht. Tragen Sie 1 Tropfen Lokalanästhetikum auf die Seite auf, die dem Eingriff unterzogen wird.
- Führen Sie die Nadel in einem Winkel von 30-45° zwischen dem Auge und dem medialen Epicanthus mit der Fase nach unten ein. Injizieren Sie die Zellsuspension (10-150 μL) langsam.
HINWEIS: Langsamere Bewegungen verhindern eine Schädigung des Auges und den Rückfluss der Injektion. - Führen Sie die in 2.7.5-2.7.6 beschriebenen Schritte aus.
- Für Milzinjektionen:
- Führen Sie das gesamte Verfahren in einer Biosicherheitskabine durch, um aseptische Bedingungenaufrechtzuerhalten 31. Ziehen Sie sterile PSA und sterile Handschuhe an.
- Betäuben und rasieren Sie das Tier wie in den Schritten 2.3-2.5 beschrieben.
- Bringen Sie das Tier in eine rechte seitliche Liegeposition. Reinigen Sie die Haut des Verfahrensbereichs mit 3 Anwendungen von 10% Povidon-Jod im Wechsel mit 3 Anwendungen von Isopropylalkohol und bereiten Sie das Operationsfeld wie in Schritt 2.9.5 beschrieben vor.
- Machen Sie mit einer Metzenbaum-Schere oder einem Skalpell einen 1 cm großen Schnitt in der linken Flanke der Bauchdecke, gefolgt von einem Schnitt in das Peritoneum.
HINWEIS: Die Milz wird nach dem Hautschnitt durch die durchscheinende Bauchdecke gesehen. Führen Sie den Peritonealschnitt genau an dieser Stelle durch. - Legen Sie die Milz und das Milzhilum durch den Schnitt frei. Mit einer 28-31 G Insulinspritzennadel, 6 mm lang, punktieren Sie vorsichtig die Milz mit der Fase der Nadel nach oben und in einem spitzen Winkel.
HINWEIS: Wenn die Stichwunde blutet, kauterisieren Sie die Stelle, um Blutungen und Rückfluss zu begrenzen. - Injizieren Sie das gesamte Volumen (50-100 μL) der Zellsuspension langsam. Entfernen Sie die Nadel. Legen Sie eine kleine Gaze auf die Milz und üben Sie Druck mit einer Pinzette aus. Klemmen Sie die Milz mit einer feinen Moskitozange leicht zwischen die Gaze und warten Sie 15 min.
- Führen Sie eine Splenektomie durch, indem Sie das Milzhilum mit einer 3-0- oder 4-0-Seidennaht binden und die Gefäße bei Bedarf kauterisieren. Schließen Sie das Peritoneum mit einer 5-0 Polydioxanon (PDS) oder Polyglykolsäure resorbierbaren Naht.
- Führen Sie die in den Schritten 2.7.5-2.7.6 beschriebenen Schritte aus.
HINWEIS: Tiere, die Blutungskomplikationen aufweisen oder sich 72 Stunden nach der Operation nicht vollständig erholt haben, sollten human eingeschläfert werden. Denken Sie daran, dass das Wohlbefinden der Mäuse zu jeder Zeit Priorität hat.
3. Inszenierte Überlebenschirurgie (SSS)
- Basierend auf den experimentellen Schlussfolgerungen aus Schritt 2.2 bestimmen Sie den richtigen Zeitpunkt bis zur Überlebensoperation. Wählen Sie je nach Zelllinie und experimenteller Hypothese einen früheren Zeitpunkt für die Tumorresektion (bei einem Tumorvolumen = 150 mm 3) oder einen späteren Zeitpunkt (bei einem Tumorvolumen = 500 mm3)26.
HINWEIS: Die Tumorvolumengrenze beträgt 1.500 mm 3, wenn die Tumorlast hoch genug ist, um das Wohlbefinden des Tieres zu beeinträchtigen und für Komplikationen prädisponiert. - Betäuben und rasieren Sie das Tier wie in den Schritten 2.3-2.5 beschrieben.
HINWEIS: Das gesamte Verfahren wird in einer Biosicherheitskabine durchgeführt. - Reinigen Sie die Haut des Verfahrensbereichs mit 3 Anwendungen von 10% Povidon-Jod im Wechsel mit 3 Anwendungen von Isopropylalkohol und bereiten Sie das Operationsfeld wie in Schritt 2.9.5 beschrieben vor.
- Verwenden Sie eine Iris-Schere oder ein Skalpell, schneiden Sie die Haut ein und halten Sie einen 5-7 mm Resektionsrand vom Rand des Tumors aufrecht.
HINWEIS: Der Spielraum für die Resektion hängt von der Fähigkeit des Tumors ab, sich lokal auszubreiten. Erhöhen Sie bei aggressiven Tumoren den Resektionsrand, während Sie sicherstellen, dass genügend Haut übrig bleibt, um den Wundverschluss durchzuführen. - Bei intradermalen Tumoren resektieren Sie den Tumor zusammen mit der umlaufenden Haut.
- Bei subkutanen Tumoren sezieren und entfernen Sie den Tumor unter der Haut.
HINWEIS: Wenn der Tumor in das Peritoneum und / oder die Haut eindringt, resezieren Sie ihn en bloc mit dem Tumor und schließen Sie das Peritoneum mit 5-0/4-0 PDS oder Polyglykolsäure resorbierbaren Nähten. - Schließen Sie die Wunde mit der 9 mm Heftvorrichtung.
HINWEIS: Entfernen Sie die Klammern 7-10 Tage nach der Operation. Verabreichen Sie analgetische Medikamente und legen Sie das Tier auf ein warmes Pad, um sich zu erholen. Setzen Sie die Verabreichung von analgetischen Medikamenten für 72 Stunden nach der Operation fort, einmal alle 12 Stunden gemäß Schritt 2.9.13. Tiere mit Blutungskomplikationen oder die nach der Operation nicht wieder das volle Bewusstsein erlangt haben, sollten human eingeschläfert werden. - Legen Sie das Tier in einem Käfig auf, auf einem warmen Pad, um sich zu erholen. Bringen Sie das Tier in den Vivariumkäfig zurück, nachdem Sie das Bewusstsein wiedererlangt haben, wenn es sternal und gehfähig ist.
- Überwachen Sie das Tier nach der Operation weiterhin auf lokale Rezidive, Gewichtsverlust, neurologische, lokomotorische und / oder Verhaltensmerkmale (Lethargie, mangelnde Pflege, geringe Nahrungs- oder Wasseraufnahme) und den allgemeinen Gesundheitszustand.
4. In-vivo-Bildgebung (Abbildung 2A)
- Verabreichen Sie den Tieren D-Luciferin-Substrat (150 mg/kg) durch intraperitoneale Injektion mit einer 1 ml Insulinspritze, 28 G Nadel.
HINWEIS: Tumorzellen müssen stabil mit der Luciferase-cDNA transduziert werden. - Induzieren Sie die Anästhesie wie in den Schritten 2.3-2.4, 6 Minuten nach der D-Luciferin-Substratinjektion beschrieben.
- Führen Sie die Bildgebung mit einem Biolumineszenz-Bildgebungsscanner (BLI) (In-vivo-Bildgebungssystem )26 durch.
- Bewegen Sie das Tier in die Bildgebungskammer und in den Nasenkegel. Fotografieren Sie bis zu 5 Tiere gleichzeitig, abhängig von der Kapazität des Bildgebungssystems.
- Starten Sie das Instrument, indem Sie auf Initialisieren klicken. Stellen Sie die Einstellung der Belichtungszeit auf auto (1-120 s) ein.
- Nehmen Sie ein leeres Bild auf, um bei Bedarf einen beliebigen Hintergrund zu subtrahieren. Klicken Sie auf Erfassen und speichern Sie das Bild, nachdem die Aufnahmesequenz abgeschlossen ist.
- Legen Sie das Tier zurück in einen Käfig, der mit 50% der Grundfläche über einem Wärmekissen sitzt, um sich von der Anästhesie zu erholen. Bringen Sie das Tier in den Vivariumkäfig zurück, nachdem Sie das Bewusstsein wiedererlangt haben, wenn es sternal und gehfähig ist.
- Für die Datenanalyse in derselben In-vivo-Bildgebungssoftware , mit der die Bilder aufgenommen wurden, navigieren Sie zu dem Ordner, in dem die Bilder gespeichert sind, und öffnen Sie die Bilder aller Mäuse, die zum Experiment gehören, auf einmal.
HINWEIS: Die Analyse eines Bildes nach dem anderen ermöglicht keine gruppenübergreifende Normalisierung. - Stellen Sie die Einheiten auf Ausstrahlung ein (zählt nicht). Stellen Sie sicher, dass das Kontrollkästchen für die Einzelperson deaktiviert ist, da dies eine gruppenübergreifende Normalisierung des Signals verhindert.
- Zeichnen Sie mit dem Zeichenwerkzeug Region of Interest (ROI) kreisförmige ROIs für die Hirnregion und rechteckige ROIs für den Körper. Achten Sie darauf, die Ohren und die Nase vom ROI des Gehirns auszuschließen, da sie dazu neigen, unspezifische Lumineszenz auszusenden. Um die Verzerrung in diesem Prozess zu minimieren, zeichnen Sie ROIs nur auf den Fotos der Mäuse, ohne dass das lumineszierende Signal überlagert ist.
- Wählen Sie ROIs messen aus, um das Signal zu quantifizieren und die Daten in eine Tabelle zu exportieren. Analysieren Sie Unterschiede zwischen Gruppen, indem Sie den Gesamtlumineszenzfluss (p / sec / cm2 / sr) in Körperregionen von Interesse aufzeichnen.
HINWEIS: Um Unterschiede zwischen Gruppen im Gehirntropismus speziell zu beurteilen, berechnen Sie das Verhältnis zwischen dem Gehirnsignal und dem Körpersignal für jede Maus. Dies kontrolliert die Intermausvariation der Gesamttumorlast und die Unterschiede in der Luciferase-Expression zwischen experimentellen Gruppen.
5. Ex-vivo-Magnetresonanztomographie
- Führen Sie eine Ex-vivo-MRT unmittelbar nach der Euthanasie durch. Alternativ können Sie die Organe von Interesse entnehmen, sie bis zu 72 h in Formalin fixieren und die Bildgebung zu einem späteren Zeitpunkt durchführen.
- Erfassen Sie die Bilder mit einem 7-Tesla (7-T) (300-MHz) Mikro-MRT-System, das mit einer NMR-Konsole und einem Zero-Boil-off-Magneten mit horizontaler Bohrung oder ähnlichen Geräten ausgestattet ist.
HINWEIS: Die Notwendigkeit eines aktiv abgeschirmten Gradientenspuleneinsatzes mit dem richtigen Leistungskompromiss ist von entscheidender Bedeutung. Sie muss eine Gradientenlinearität von mindestens 50 mm dynamischem sphärischem Volumen (DSV) aufweisen, um die untersuchten Proben gleichzeitig ohne geometrische Verzerrung abzudecken. Die Kombination aus Gradientenfestigkeit (von 440 bis 750 mT/m) und Tastverhältnis, die maximale gleichzeitige Gleichströme von 3 x 30 A bis 3 x 87 A ermöglicht, ermöglicht eine angemessene Bildgebungsleistung. Der verwendete Gradientenspuleneinsatz (siehe Materialtabelle) ermöglicht folgende Leistung: 660 mT/m, 130 μs Anstiegszeit, 3 x 87 A und ein DSV = 80 mm. - Führen Sie die Scans mit einer kommerziellen, kreisförmig polarisierten Radiofrequenzspule für den gesamten Mauskörper (OD = 59 mm, ID = 38 mm, L = 40 mm) durch, die auf 300,16 MHz, die 1H Protonen-Larmor-Frequenz, abgestimmt ist.
HINWEIS: Diese HF-Sonde ermöglicht die Erfassung von 3D-Datensätzen mit submillimetermetrischer isotroper Auflösung (<150 μm) während nächtlicher Scans von 8-12 h. - Erkennen Sie die Tumorlast anhand mehrerer Sequenzen30.
HINWEIS: Hyperintensives Signal, das von einerT2-gewichteten, schnellen Bildgebung mit refokussierten Echos (RARE) Sequenz erkannt wird, erkennt Ödeme, die Tumore umgeben. - Führen Sie die 3D-RARE-Sequenz mit den folgenden Erfassungsparametern durch: [120 μm]3 isotrope Auflösung; Erfassungszeit 5 h, 27 min; Wiederholungszeit (TR) = 500 ms; Echoabstand (ES) = 12,7 min; Turbofaktor TFx = 12; effektive Echozeit (TEeff) = 76,2 ms; Bandbreite (BW) = 75 kHz; Matrixgröße = 2843; Sichtfeld (FOV) = [4,0 mm]3; Anzahl der Durchschnittswerte (Nav) = 6.
- Erkennen Sie Metastasen mit den folgenden Parametern.
- Für pigmentierte Metastasen mit Signalaufhellung verwenden Sie eineT1-gewichtete 3D-Gradienten-Echosequenz mit folgenden Parametern: [120 μm]3 isotrope Auflösung; Erfassungszeit 2 h, 41 min; TR = 20 ms; Echozeit (TE) = 4,0 ms; Flip-Winkel (FA) = 18°; BW = 75 KHz; Matrixgröße = 2843; Sichtfeld = [34,0 mm]3; Nav = 6.
- Verwenden Sie für unpigmentierte und/oder hämorrhagische Metastasen ein hypointensives Signal, wenn Sie unter einerT2*-gewichteten, multigradienten Echosequenz (MGE) (3D-MGE, [120 μm]3 isotrope Auflösung erfassen; Erfassungszeit 3 h, 35 min; TR = 40 ms; TE = 3,6 ms; ES = 3,2 ms; 4 Echos; FA = 20°; BW = 100 kHz; Matrixgröße = 2843; Sichtfeld = (34,0 mm)3; Nav = 4.
- Verwenden Sie alle 3 Sequenzen, um die Tumorlast zu quantifizieren.
- Querverweisen Sie die während der Analyse identifizierten Tumorbereiche mit histologischen Schnitten, um die Genauigkeit zu gewährleisten. Siehe Abschnitte 7 und 8
6. Gewebeverarbeitung für die Einzelzell- oder Bulk-RNA-Sequenzierung
- Euthanasie das Tier mit einer Methode, die von der IAUC der Institution genehmigt wurde. Sehen Sie sich eines der Verfahren an, die im HINWEIS zu Schritt 2.6.6 beschrieben sind.
- Sezieren Sie die interessierenden Organe und legen Sie sie in separate Vertiefungen einer Platte, die Hank's Balanced Salt Solution (HBSS) auf Eis enthält. Arbeiten Sie schnell und halten Sie das Gewebe jederzeit auf Eis, um die Lebensfähigkeit der Zellen zu maximieren.
HINWEIS: Die folgenden Schritte sind spezifisch für die Gehirnverarbeitung. Passen Sie den Kollagenase-Typ an das spezifische Gewebe an, basierend auf Ihren Bedürfnissen. - Bereiten Sie eine 6-Well-Platte mit 3 ml HBSS in jedem Well vor.
- Um die Dissektion zu visualisieren und weiter zu leiten, verwenden Sie ein Fluoreszenzmikroskop und identifizieren Sie die markierten Bereiche.
- Sezieren Sie die fluoreszierenden Bereiche und legen Sie die Gewebefragmente in die 6-Well-Platte (1 Fragment pro Well, wenn einzelne metastatische Herde analysiert werden sollen oder mehrere Fragmente aus einem Organ, wenn mehrere Metastasen im selben Organ analysiert werden sollen). Verwenden Sie sterile Rasierklingen, um das Gewebe so klein wie möglich in Fragmente zu zerkleinern (ohne mehr als 1-2 Minuten für diesen Schritt für jede Probe auszugeben).
HINWEIS: Die Begrenzung der Verarbeitungszeit der Tumoren hilft, die Lebensfähigkeit der Zellen zu erhalten. - Saugen Sie den Inhalt jedes Wells ab und übertragen Sie ihn in ein 15 ml konisches Rohr.
HINWEIS: Schneiden Sie die Spitze einer 1.000 μL Pipettenspitze ab, um den Transfer größerer Fragmente zu erleichtern. - Fügen Sie 1 ml HBSS in das Bohrloch hinzu und stellen Sie sicher, dass die verbleibenden Gewebefragmente / Zellen in die 15-ml-Röhre übertragen werden, die ein Endvolumen von 4 ml enthält. 50 μL Kollagenase Typ I (40 mg/ml) und 12,5 μL DNase I (2.000 Einheiten/ml) zu jeder Tube.
- Legen Sie die konischen Rohre für 45 min in ein auf 37 °C beheiztes Wasserbad. Alle 5 Minuten kurz die konischen Röhren umdrehen.
- Ein 70 μm Sieb mit HBSS vorbenetzen. Verwenden Sie das verschlossene Ende eines sterilisierten Mikrozentrifugenröhrchens oder den Kunststoffteil eines Spritzenkolbens und mahlen Sie das Gewebehomogen durch ein 70 μm langes Sieb zu einem neuen 50 ml konischen Röhrchen.
HINWEIS: Die Vorbenetzung der Siebe mit HBSS- oder FACS-Puffer erleichtert die Belastung. - Waschen Sie das Sieb mit 1 ml HBSS. Ein 40 μm Sieb mit HBSS vorbenetzen. Filtern Sie jede Probe erneut durch ein 40μm-Sieb in ein neues konisches 50-ml-Rohr. Geben Sie 1 ml FBS in das 40-μm-Sieb, um es zu waschen. Halten Sie die konischen Röhren die ganze Zeit auf Eis.
- Füllen Sie das konische Rohr auf 50 ml mit eiskalten DPBs. Drehen Sie die Zellen herunter (180 × g für 10 min bei 4 °C). Verwerfen Sie den Überstand und achten Sie darauf, das Zellpellet nicht zu verlieren.
HINWEIS: Für Gehirnproben resuspendieren Sie die Zellen in 2,5 ml 38% iger Dichte-Trennlösung (in HBSS verdünnen und bei Raumtemperatur (RT) lagern). Transfer auf 5 ml FACS-Röhren. Drehen Sie 20 Minuten bei 800 × g. Schneiden Sie die Spitze einer 1.000 μL Pipettenspitze ab und entfernen Sie die oberste Fettschicht. Lassen Sie kein Fett an den Wänden der Röhren. Wenn noch Fett übrig ist, drehen Sie sich erneut nach unten und wiederholen Sie den Vorgang. Dies ist ein kritischer Schritt. Entfernen Sie den Rest der flüssigen Phase (das Pellet ist durchscheinend und schwer zu visualisieren). - Resuspendieren Sie die Zellen in 1 ml Lysepuffer der roten Blutkörperchen (RBC) und inkubieren Sie für 60 s bei RT. Quen Sie die Lyselösung durch Zugabe von 20 ml DPBS.
- Drehen Sie die Zellen bei 180 × g für 10 min bei 4 °C herunter. Entfernen Sie den Überstand und suspendieren Sie die Zellen in 2 ml FACS-Puffer (5% FBS in DPBS).
- Fahren Sie mit der Sortierung markierter Zellen und / oder der Bibliotheksvorbereitung für die Massen- oder Einzelzell-RNA-Sequenzierung fort.
HINWEIS: Zellen können vor der RNA-Isolierung für Bulk-RNA-Seq bei -80 ° C gedreht, eingefroren und gelagert werden.
7. Durchblutung tierischen Gewebes und Vorbereitung für immunhistologische Analysen
- Anästhesieren Sie das Tier mit einer Überdosis Ketamin (300 mg/kg) und Xylazin (30 mg/kg) Cocktail durch intraperitoneale Injektion mit einer Insulinspritze und einer 28 G Nadel.
- Entblößen Sie das Herz durch grobe Dissektion und machen Sie einen Schnitt im rechten Vorhof. Halten Sie das Herz sanft an Ort und Stelle mit langen, gebogenen Pinzetten, die nach vorne gerichtet sind.
HINWEIS: Formalin und Paraformaldehyd (PFA) sind Karzinogene. Lesen Sie das Sicherheitsdatenblatt (SDB), vermeiden Sie die Exposition gegenüber Dämpfen und tragen Sie die entsprechende PSA. - Injizieren Sie mit einer 10-ml-Spritze mit einer 22-G-, 22-mm-Nadel 10 ml DPBS, gefolgt von 10 ml 4% PFA in den linken Ventrikel.
- Fahren Sie fort, die Organe zu entnehmen und sie in vorbeschriftete histologische Kassetten zu laden. Legen Sie die Kassetten in einen entsprechend großen Behälter, der genügend Fixiermittel (Formalin) aufnehmen kann, um das Gewebe zu bedecken.
HINWEIS: Idealerweise sollte das fixierende Volumen das 5-10-fache des Volumens der Gewebe betragen. - Fixieren Sie die Organe in den histologischen Kassetten in 10% Formalin für 48-72 h. Entsorgen Sie das 10% Formalin und waschen Sie die Kassetten zweimal mit 1x DPBS.
- Beginnen Sie den Dehydratisierungsprozess, indem Sie die Kassetten für 2 Stunden in 70% Ethanol tauchen. Tauchen Sie die Kassetten sukzessive in steigende Ethanolkonzentrationen ein: 80%, 95%, 100% für jeweils 1 h. Wechseln Sie die 100%ige Lösung zweimal nach 1,5 h. Tauchen Sie die Kassetten für 1,5 h in Xylol ein und führen Sie drei Änderungen der Lösung durch.
- Betten Sie die Kassetten bei 58-60 °C in Paraffinwachs ein. Schneiden Sie die Paraffinblöcke auf. Fahren Sie mit der Hämatoxylin- und Eosin- (H & E) oder Immunhistochemie-Färbung fort.
- Um Melanomzellen zu identifizieren, verwenden Sie einen dieser Marker oder ein Panel: S100, Melan-A, HMB-45, Tyrosinase, MITF. Wenn möglich, verwenden Sie die NuMA-Färbung (Nuclear Mitotic Apparatus Protein), da es sich um einen hochspezifischen menschlichen Zellmarker handelt.
HINWEIS: Die NuMA-Färbung bietet eine scharfe Abgrenzung zwischen Wirtszellen (Maus) und transplantierten Zellen (Mensch), die bei der Bildverarbeitung und den nachfolgenden Tumorquantifizierungsstadien hilft.

Abbildung 2: Beispiele für BLI-, Hellfeld-, Ex-vivo-Fluoreszenz- und H&E-Färbebilder, die den mehrgleisigen Ansatz zur Analyse der Wirkungen von Kandidatengenen bei Melanommetastasen veranschaulichen. (A) BLI-, (B) BF-, (C) Ex-vivo-Fluoreszenz- und (D) H&E-Färbebilder. Die zur Veranschaulichung verwendeten Bilder entsprechen einem Experiment, bei dem 131/6-4L-Melanomzellen, die mit einer Non-Targeting-Kontroll-shRNA (shNTC) oder einer shRNA-Targeting-FUT8-Mäuse transduziert wurden, in immunodefiziente (NSG) Mäuse injiziert wurden. FUT8-Stummschaltung beeinträchtigte die metastatische Verbreitung von Melanomzellen. Maßstabsleisten und Farbbalken = p/sec/cm2/sr × 106 (A), 100 mm (B, C), 100 μm (D). Abkürzungen: BLI = Biolumineszenzbildgebung; H&E = Hämatoxylin und Eosin; shRNA = Kurzhaarnadel-RNA; shNTC = non-targeting control shRNA; NSG = nicht-adipöser Diabetiker schwerer kombinierter Immunschwäche gamma; FUT8 = Fucosyltransferase 8; BF = Hellfeld. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
8. Färbung des nuklearen mitotischen Apparateproteins (NuMA) (Abbildung 3)
- Verwenden Sie einen Anti-NuMA-Antikörper als hochgradig humanspezifischen mitotischen Spindelmarker zur Identifizierung und Quantifizierung der metastatischen Belastung in Gewebeschnitten. 8.1. Es wird eine hochspezifische und empfindliche Identifizierung von Melanomzellen erreicht.
- Wenn die chromogene Immunhistochemie für NuMA an einem automatischen Immunfärbungsinstrument durchgeführt wird, führen Sie die folgenden Schritte wiebeschrieben aus 32:
- Deparaffinisieren Sie die Abschnitte in Xylol und rehydrieren Sie sie in sequentiell abnehmenden Ethanolkonzentrationen. Halten Sie die Objektträger für 15 min in Xylol unter Wasser und übertragen Sie sie für weitere 15 Minuten auf 100% Ethanol.
HINWEIS: Der Rest der Ethanol-Rehydratisierungsschritte (95%, 80%, 75%) dauert jeweils 3 bis 5 Minuten. - Spülen Sie die Objektträger in entionisiertem Wasser ab.
- Führen Sie die Epitopentnahme durch, indem Sie die Objektträger in einen Behälter (z. B. Coplin-Färbeglas) in einen 10-m-Natriumcitratpuffer mit einem pH-Wert von 6,0 m) in einem 1200-Watt-Mikrowellenherd mit 100% Leistung für 10 min tauchen.
- Verwenden Sie einen unkonjugierten, polyklonalen Kaninchen-Antihuman-NuMA-Antikörper zur Markierung, verdünnt 1:7.000 in Tris-Rinderserumalbumin (BSA) (25 mM Tris, 15 mM NaCl, 1% BSA, pH 7,2). Führen Sie die entsprechenden Positiv- und Negativkontrollen parallel zu den Studienabschnitten durch.
- Inkubieren Sie die Objektträger mit dem primären Antikörper für 12 h. Nachweis des primären Antikörpers mit einem Ziegen-Anti-Kaninchen-HRP-konjugierten Multimer und Visualisierung des Komplexes mit 3,3-Diaminobenzidin und einem Kupfersulfat-Enhancer.
- Waschen Sie die Objektträger in destilliertem Wasser, bekämpfen Sie sie mit Hämatoxylin, dehydrieren Sie sie und montieren Sie sie mit permanentem Medium.
HINWEIS: Die Dehydratisierungsschritte sind die Umkehrung der in Schritt 8.3 beschriebenen Rehydratationsschritte. Scannen Sie die Dias mit dem verfügbaren Scanner mit 20-facher oder 40-facher Vergrößerung und laden Sie sie in eine Datenbank hoch. - Ziehen Sie mithilfe von Software ROIs, um alle NuMA-gefärbten Zellen innerhalb des Organgewebes einzubeziehen, mit Ausnahme anderer Organparenchyme und leerer Räume.
- Passen Sie die Einstellungen an, um die NuMA-positiven und NuMA-negativen Zellen zu kategorisieren, während Sie für jedes Organ geeignete Positiv- und Negativkontrollen verwenden. Verwenden Sie einen etablierten Softwarealgorithmus, um die Gesamtzahl / den Prozentsatz der NuMA-positiven Zellen für jede Probe zu quantifizieren.
9. Immunfluoreszenz von Gewebescheiben
Um das metastatische Stadium zu identifizieren, in dem ein bestimmter Genkandidat erforderlich ist (z. B. Extravasation vs. Überleben nach der Aussaat), kann man die Immunfluoreszenz von Gewebeschnitten zu verschiedenen Zeitpunkten bestimmen, um die Progression der Tumorzellen von der Injektion bis zur entfernten Organinvasion, dem Seeding und dem Wachstum zu verfolgen. Dieser Ansatz ermöglicht die Addition von Markern für benachbarte Zellen, um das Extravasationsereignis und die Veränderungen der umgebenden Tumormikroumgebungzu erfassen 33.
- Betäuben Sie das Tier wie in Schritt 7.1 beschrieben.
- Injizieren Sie 100 μg fluorophorkonjugiertes Lycopersicon Esculentum (Tomaten) -Lektin 3 Minuten vor der Perfusion in den linken Ventrikel jedes Tieres, um das vaskuläre Endothel abzugrenzen.
HINWEIS: Lassen Sie dem Tomatenlekt Zeit, um im gesamten System zu zirkulieren. - Das Tier wie in den Schritten 7.2-7.3 beschrieben durchbluten. Entnehmen Sie die Organe von Interesse und geben Sie sie in vorbeschriftete Behälter, die mit 4% PFA gefüllt sind. Fixieren Sie das Gewebe für 24 bis 48 h. Schneiden Sie das Gewebe mit einem Vibratom in 30-50 μm dicke Scheiben.
HINWEIS: Die Dicke sollte optimiert werden. Scheiben zwischen 30 μm und 50 μm Dicke werden empfohlen, insbesondere bei der Durchführung von Z-Stack-Imaging. - Inkubieren Sie die Scheiben in Blockpuffer (10% normales Ziegenserum, 2% BSA, 0,25% Triton X-100 in DPBS) für 2 h bei RT.
- Führen Sie ein Färbeoptimierungsexperiment durch.
HINWEIS: Da die Antigenabrufzeit, die für die Antigengewinnung verwendeten Puffer, die Temperatur, verschiedene Antikörper / verschiedene Chargen und die Art des Gewebes die Färbung beeinflussen, sind Optimierungsexperimente erforderlich. - Fügen Sie primäre Antikörper bei einer optimierten Verdünnung hinzu und inkubieren Sie für eine optimierte Zeit bei einer optimierten Temperatur (siehe Beispiele in Tabelle 1).
HINWEIS: Verwenden Sie geeignete Kontrollen für primäre/sekundäre Antikörper und ungefärbte Gewebeproben. - Waschen Sie die Gewebescheiben 3 Mal für 5 min mit 0,25% Triton X-100 in DPBS.
- Inkubieren Sie die Gewebeschnitte in sekundären Antikörpern, verdünnt in Blockierlösung für die gewünschte Zeit (Tabelle 1).
- Waschen Sie die Gewebescheiben 3 Mal für 5 min mit 0,25% Triton X-100 in DPBS.
- Färben Sie die Kerne mit 4′,6-Diamidino-2-phenylindol (DAPI), verdünnt 1:1.000 in DPBS oder blockieren Sie Puffer für 5 min.
- Fügen Sie 2 Tropfen Antifade-Fluoreszenz-Montagemedium zu einem Deckglas hinzu und montieren Sie das Gewebe auf Glasobjektträger, um sicherzustellen, dass die Scheiben vollständig mit Montagemedium bedeckt sind.
HINWEIS: Stellen Sie sicher, dass sich keine Luftblasen direkt auf den Scheiben befinden, da dies die Mikroskopie verzerrt. - Nehmen Sie konfokale Bilder mit dem verfügbaren Mikroskop mit einem 60-fachen Öl-Tauchobjektiv auf.
HINWEIS: Wenn Sie konfokale Bilder aufnehmen, wenden Sie die gleichen Einstellungsparameter (Spannung, Lufteinheiten und Verstärkung) auf alle Bilder innerhalb des Experiments an. - Nehmen Sie nicht-konfokale Bilder mit dem Mikroskop bei 10x, 20x oder 40x auf.
- Laden Sie die Bilder in eine Bildanalysesoftware hoch und analysieren Sie sie, indem Sie die Parameter der Wahl vergleichen (z. B. Fläche, Anzahl, Intensität der Marker oder Kontakt mit benachbarten Zellen).
Ergebnisse
Die folgenden Abbildungen veranschaulichen, wie der beschriebene Workflow zur Identifizierung neuartiger Treiber der Melanommetastasierung angewendet wurde. Abbildung 2 fasst die Ergebnisse einer veröffentlichten Studie zusammen, in der die Auswirkungen der Stilllegung der Fucosyltransferase FUT8 in vivo Melanommetastasen untersucht wurden26. Kurz gesagt, die Analyse der glykomischen Daten des menschlichen Patienten (erhalten durch Lektin-Arrays) und die tra...
Diskussion
Ziel dieses technischen Berichts ist es, einen standardisierten, von oben nach unten gerichteten Workflow für die Untersuchung potenzieller Akteure bei Melanommetastasen anzubieten. Da In-vivo-Experimente kostspielig und zeitaufwendig sein können, sind Strategien zur Maximierung der Effizienz und zur Steigerung des Wertes der erhaltenen Informationen von größter Bedeutung.
Es ist unerlässlich, komplementäre Ansätze zu verwenden, um die Ergebnisse innerhalb desselben Experiments...
Offenlegungen
Die Autoren haben keine Interessenkonflikte zu erklären.
Danksagungen
Wir danken der Division of Advanced Research Technologies (DART) an der NYU Langone Health und insbesondere dem Experimental Pathology Research Laboratory, dem Genome Technology Center, dem Cytometry and Cell Sorting Laboratory, dem Pre-Clinical Imaging Core, die teilweise durch den Perlmutter Cancer Center Support Grant NIH / NCI 5P30CA016087 unterstützt werden. Wir danken der NYU Interdisciplinary Melanoma Cooperative Group (PI: Dr. Iman Osman) für den Zugang zu patientenabgeleiteten Melanom-Kurzzeitkulturen + (10-230BM und 12-273BM), die durch IRB-genehmigte Protokolle (Universal Consent Study #s16-00122 und Interdisciplinary Melanoma Cooperative Group Study # 10362) erhalten wurden. Wir danken Dr. Robert Kerbel (University of Toronto) für die Bereitstellung von 113/6-4L und 131/4-5B1 Melanom-Zelllinien* und Dr. Meenhard Herlyn (Wistar Institute) für die Bereitstellung von WM 4265-2, WM 4257s-1, WM 4257-2 Melanom-Kurzzeitkulturen**. E.H. wird unterstützt von NIH/NCI R01CA243446, P01CA206980, einem American Cancer Society-Melanoma Research Alliance Team Science Award und einem NIH Melanoma SPORE (NCI P50 CA225450; PI: U.A.). Abbildung 1 wurde mit Biorender.com erstellt.
Materialien
| Name | Company | Catalog Number | Comments |
| #15 Scapel Blade | WPI | 500242 | For surgical procedures |
| #3 Scapel Handle | WPI | 500236 | For surgical procedures |
| 1 mL Tuberculin syringe, slip tip | BD | 309626 | Injections |
| 10 mL syringe, slip tip | BD | 301029 | Perfusion |
| 10% Formalin Sodium Buffered | EK Industries | 4499-20L | For perfusion/tissue fixative |
| 15 mL Conical | Corning | 430052 | Cell culture |
| 15 mL Conical Polypropylene Centrifuge Tubes | Falcon | 352196 | Cell culture |
| 200 Proof Ethanol | Deacon Labs | 04-355-223 | Histology |
| 22G – 22mm needle | BD | 305156 | Perfusion |
| 4-0 Vicryl Suture | Ethicon | J464G | Suture |
| 4% Carson's phosphate buffered paraformaldehyde | EMS | 15733-10 | For perfusion/tissue fixative |
| 40µm | Corning | 431750 | Tissue processing |
| 5-0 Absorbable Suture | Ethicon | 6542000 | Closure |
| 50 mL Conical | Corning | 430828 | Cell culture |
| 50mL Conical Polypropylene Centrifuge Tubes | Falcon | 352070 | Cell culture |
| 7-0 Silk suture | FST | 18020-70 | Ligature |
| 70µm | Corning | 431751 | Tissue processing |
| Anti-fade mounting media | Vector Labs | H-1000-10 | Immunofluorescence |
| Approximator applying Forceps, 10cm | WPI | 14189 | For microsurgical procedures |
| Avance | Bruker | 3 HD | NMR Console |
| Biospec 7030 | Bruker | 7030 | Micro MRI |
| BSA | Bioreg | A941 | NuMA Staining |
| Castroviejo suturing forceps, straight tips 5.5mm tying platform, 11cm | WPI | WP5025501 | For microsurgical procedures |
| Coplin Staining Jar | Bel-Art | F44208-1000 | Histology |
| DAPI | Sigma-Aldrich | D9542-1MG | Immunofluorescence |
| dCas9-KRAB | Addgene | 110820 | Genetic manipulation |
| DNase I | NEB | M0303L | Tissue processing |
| DPBS | Corning | 21-030-CM | Tissue processing |
| Extra Sharp Uncoated Single Edge Blade | GEM | 62-0167 | Tissue processing |
| Extracellular Matrix Substrate | Corning | 354234 | Consider the Growth Factor Reduced ( as alternative |
| FBS | Cytiva | SH30910.03 | Cell culture |
| Fiji Image J | Fiji Image J | Software | Immunofluorescence |
| Goat anti-rabbit HRP conjugated multimer | Thermo Fisher | A16104 | NuMA Staining |
| Goat Serum | Gibco | PCN5000 | Immunofluorescence |
| HBSS | Corning | 21-020-CV | Tissue processing |
| Hematoxylin | Richard-Allan Scientific | 7231 | Histology |
| Illumina III | PerkinElmer | CLS136334 | BLI Instrument |
| Insulin syringe 28G - 8mm needle | BD | 329424 | Injections |
| Insulin syringe 31G - 6mm needle | BD | 326730 | Injections |
| Iris Forceps, 10.2cm, Full Curve, serrated | WPI | 504478 | For perfusion and surgical procedures |
| Isoflurane USP | Covetrus | 11695067772 | Anesthesia |
| Jewelers #7 Forceps Titanium 11 cm 0.07 x 0.01 mm Tip | WPI | WP6570 | For microsurgical procedures |
| Ketamine HCl 100mg/mL | Mylan Ind. | 1049007 | Anesthesia |
| lentiCRISPRv2 | Addgene | 98290 | Genetic manipulation |
| Lycopersicon Esculentum (Tomato) Lectin, DyLight 649 | Invitrogen | L32472 | Vascular endothelial cells marker |
| MEM non-essential amino acids X 100 | Corning | 25-025-CI | Cell culture |
| Metzenbaum Scissors | WPI | 503269 | For surgical procedures |
| Microinjection Unit | KOPF | 5000 | Intracardiac injections |
| NaCl | Fisher | S25877 | NuMA Staining |
| Needle 30G x 25mm | BD | 305128 | Intracardiac Injection |
| Needle 33G x 15mm | Hamilton | 7747-01 | Intracarotid Injection |
| Needle holder, Castroviejo, 14cm, with lock, 1.2mm Serrated Jaws | WPI | 14137-G | For microsurgical procedures |
| NOD.Cg-Prkdcscid Il2rgtm1Wjl/SzJ mice | The Jackson Laboratory | 005557 | Murine model |
| NU/J mice | The Jackson Laboratory | 002019 | Murine model |
| Nuclear Mitotic Apparatus Protein polyclonal rabbit anti-human | Abcam | 97585 | NuMA Staining |
| Penicillin-Streptomycin 10000U/mL | Gibco | 15140122 | Cell culture |
| Percoll | GE | 0891-01 | density separation solution |
| PI Classic Surgical Gloves | Cardinal Health | 2D72PT75X | Surgery |
| pLKO Tet-On | Addgene | 21915 | Genetic manipulation |
| Povidone-Iodine 10% Solution | Medline | MDS093943 | Surgery |
| Proparacaine Drops 0.5% | Akorn Pharma | AX0501 | Opthalmic local anesthetic |
| Puralube Petrolatum Opthalmic Ointment | Dechra | 83592 | Anesthesia |
| Razor Blade Double Edge Blades | EMS | 72000 | Shaving and Vibrotome Brain Slicing |
| Reflex 9mm EZ Clip | Braintree | EZC- KIT | Wound closure |
| RPMI 1640 | Corning | 10-040-CM | Cell culture |
| Scissors, Spring 10.5cm Str, 8mm Blades | WPI | 501235 | For microsurgical procedures |
| Semi-Automatic Vibrating Blade Microtome | Leica | VT1200 | Brain Slice Immunofluorescence |
| Single Channel Anesthesia Vaporizer System | Kent Scientific | VetFlo-1210S | Anesthesia |
| Smartbox Tabletop Chamber System and Exhaust Blower | EZ Systems | TT4000 | CO2 Euthanasia |
| Sterile Fenestrated Disposable Drape | Medline | NON21002 | Surgery |
| Sterile Non-Reinforced Aurora Surgical Gowns with Set-In Sleeves | Medline | DYNJP2715 | Surgery |
| T25 Flask | Corning | 430639 | Cell culture |
| Tris | Corning | 46-031-CM | NuMA Staining |
| Triton X-100 | Sigma-Aldrich | X100-500ML | Immunofluorescence |
| Troutman tying forceps, 10cm, Curved G pattern, 0.52mm tip with tying platform | WPI | WP505210 | For microsurgical procedures |
| Vessel clips 10G Pressure 5x 0.8mm Jaws, 5/pkg | WPI | 15911 | For microsurgical procedures |
| Visiopharm | Visiopharm | Visiopharm | NuMA Staining Quantification Software |
| Xylasine 100mg/mL | Akorn Pharma | 59399-111-50 | Anesthesia |
| Xylene | Fisher | X3P-1GAL | Histology |
Referenzen
- Sung, H., et al. Global Cancer Statistics 2020: GLOBOCAN Estimates of incidence and mortality worldwide for 36 cancers in 185 countries. CA: A Cancer Journal for Clinicians. 71 (3), 209-249 (2021).
- Adler, N. R., Haydon, A., McLean, C. A., Kelly, J. W., Mar, V. J. Metastatic pathways in patients with cutaneous melanoma. Pigment Cell Melanoma Research. 30 (1), 13-27 (2017).
- Platz, A., Egyhazi, S., Ringborg, U., Hansson, J. Human cutaneous melanoma; a review of NRAS and BRAF mutation frequencies in relation to histogenetic subclass and body site. Molecular Oncology. 1 (4), 395-405 (2008).
- Alonso, S. R., et al. A high-throughput study in melanoma identifies epithelial-mesenchymal transition as a major determinant of metastasis. Cancer Research. 67 (7), 3450-3460 (2007).
- Rowe, C. J., Khosrotehrani, K. Clinical and biological determinants of melanoma progression: Should all be considered for clinical management. Australasian Journal of Dermatology. 57 (3), 175-181 (2016).
- Plebanek, M. P., et al. Pre-metastatic cancer exosomes induce immune surveillance by patrolling monocytes at the metastatic niche. Nature Communications. 8 (1), 1319 (2017).
- Orgaz, J. L., et al. Loss of pigment epithelium-derived factor enables migration, invasion and metastatic spread of human melanoma. Oncogene. 28 (47), 4147-4161 (2009).
- Ladhani, O., Sanchez-Martinez, C., Orgaz, J. L., Jimenez, B., Volpert, O. V. Pigment epithelium-derived factor blocks tumor extravasation by suppressing amoeboid morphology and mesenchymal proteolysis. Neoplasia. 13 (7), 633-642 (2011).
- Ju, R. J., Stehbens, S. J., Haass, N. K. The role of melanoma cell-stroma interaction in cell motility, invasion, and metastasis. Frontiers in Medicine - Dermatology. 5, 307 (2018).
- Wiley, H. E., Gonzalez, E. B., Maki, W., Wu, M. T., Hwang, S. T. Expression of CC chemokine receptor-7 and regional lymph node metastasis of B16 murine melanoma. Journal of the National Cancer Institute. 93 (21), 1638-1643 (2001).
- Meier, F., et al. Metastatic pathways and time courses in the orderly progression of cutaneous melanoma. British Journal of Dermatology. 147 (1), 62-70 (2002).
- Turner, N., Ware, O., Bosenberg, M. Genetics of metastasis: melanoma and other cancers. Clinical & Experimental Metastasis. 35 (5-6), 379-391 (2018).
- Ubellacker, J. M., et al. Lymph protects metastasizing melanoma cells from ferroptosis. Nature. 585 (7823), 113-118 (2020).
- Cukierman, E., Pankov, R., Stevens, D. R., Yamada, K. M. Taking cell-matrix adhesions to the third dimension. Science. 294 (5547), 1708-1712 (2001).
- Cunningham, C. C., et al. Actin-binding protein requirement for cortical stability and efficient locomotion. Science. 255 (5042), 325-327 (1992).
- Unger, C., et al. Modeling human carcinomas: physiologically relevant 3D models to improve anti-cancer drug development. Advanced Drug Delivery Reviews. 79-80, 50-67 (2014).
- Fong, E. L., Harrington, D. A., Farach-Carson, M. C., Yu, H. Heralding a new paradigm in 3D tumor modeling. Biomaterials. 108, 197-213 (2016).
- Nakamura, K., et al. Characterization of mouse melanoma cell lines by their mortal malignancy using an experimental metastatic model. Life Science. 70 (7), 791-798 (2002).
- Meeth, K., Wang, J. X., Micevic, G., Damsky, W., Bosenberg, M. W. The YUMM lines: a series of congenic mouse melanoma cell lines with defined genetic alterations. Pigment Cell Melanoma Research. 29 (5), 590-597 (2016).
- Koya, R. C., et al. BRAF inhibitor vemurafenib improves the antitumor activity of adoptive cell immunotherapy. Cancer Research. 72 (16), 3928-3937 (2012).
- Jenkins, M. H. Multiple murine BRaf(V600E) melanoma cell lines with sensitivity to PLX4032. Pigment Cell Melanoma Research. 27 (3), 495-501 (2014).
- Tuncer, E., et al. SMAD signaling promotes melanoma metastasis independently of phenotype switching. The Journal of Clinical Investigation. 129 (7), 2702-2716 (2019).
- Schwartz, H., et al. Incipient Melanoma Brain Metastases Instigate Astrogliosis and Neuroinflammation. Cancer Research. 76 (15), 4359-4371 (2016).
- Perez-Guijarro, E., et al. Multimodel preclinical platform predicts clinical response of melanoma to immunotherapy. Nature Medicine. 26 (5), 781-791 (2020).
- Krepler, C., et al. A Comprehensive Patient-Derived Xenograft Collection Representing the Heterogeneity of Melanoma. Cell Reports. 21 (7), 1953-1967 (2017).
- Agrawal, P., et al. A systems biology approach identifies FUT8 as a driver of melanoma metastasis. Cell. 31 (6), 804-819 (2017).
- Hanniford, D., et al. Epigenetic silencing of CDR1as drives IGF2BP3-mediated melanoma invasion and metastasis. Cancer Cell. 37 (1), 55-70 (2020).
- Kim, H., et al. PRMT5 control of cGAS/STING and NLRC5 pathways defines melanoma response to antitumor immunity. Science Translational Medicine. 12 (551), (2020).
- de Miera, E. V., Friedman, E. B., Greenwald, H. S., Perle, M. A., Osman, I. Development of five new melanoma low passage cell lines representing the clinical and genetic profile of their tumors of origin. Pigment Cell Melanoma Research. 25 (3), 395-397 (2012).
- Morsi, A., et al. Development and characterization of a clinically relevant mouse model of melanoma brain metastasis. Pigment Cell Melanoma Research. 26 (5), 743-745 (2013).
- Huynh, C., et al. Efficient in vivo microRNA targeting of liver metastasis. Oncogene. 30 (12), 1481-1488 (2011).
- Zou, C., et al. Experimental variables that affect human hepatocyte AAV transduction in liver chimeric mice. Molecular Therapy Methods and Clinical Development. 18, 189-198 (2020).
- Kleffman, K., et al. Melanoma-secreted Amyloid Beta Suppresses Neuroinflammation and Promotes Brain Metastasis. bioRxiv. , 854885 (2019).
- Curtis, A., Calabro, K., Galarneau, J. R., Bigio, I. J., Krucker, T. Temporal variations of skin pigmentation in C57BL/6 mice affect optical bioluminescence quantitation. Molecular Imaging and Biology. 13 (6), 1114-1123 (2011).
- Sil, P., Wong, S. W., Martinez, J. More than skin deep: autophagy is vital for skin barrier function. Frontiers in Immunology. 9, 1376 (2018).
- Chen, S., et al. Genome-wide CRISPR screen in a mouse model of tumor growth and metastasis. Cell. 160 (6), 1246-1260 (2015).
- Hart, T., et al. High-resolution CRISPR screens reveal fitness genes and genotype-specific cancer liabilities. Cell. 163 (6), 1515-1526 (2015).
- Wang, T., et al. Identification and characterization of essential genes in the human genome. Science. 350 (6264), 1096-1101 (2015).
- Edgar, R., Domrachev, M., Lash, A. E. Gene Expression Omnibus: NCBI gene expression and hybridization array data repository. Nucleic Acids Research. 30 (1), 207-210 (2002).
- Lappalainen, I., et al. The European Genome-phenome Archive of human data consented for biomedical research. Nature Genetics. 47 (7), 692-695 (2015).
- Cerami, E., et al. The cBio cancer genomics portal: an open platform for exploring multidimensional cancer genomics data. Cancer Discovery. 2 (5), 401-404 (2012).
- Grossman, R. L., et al. Toward a shared vision for cancer genomic data. New England Journal of Medicine. 375 (12), 1109-1112 (2016).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten