È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Una solida piattaforma di scoperta per l'identificazione di nuovi mediatori delle metastasi del melanoma
In questo articolo
Riepilogo
Questo articolo descrive un flusso di lavoro di tecniche impiegate per testare nuovi potenziali mediatori di metastasi del melanoma e i loro meccanismi d'azione.
Abstract
Le metastasi sono un processo complesso, che richiede alle cellule di superare barriere che sono solo modellate in modo incompleto da saggi in vitro . È stato stabilito un flusso di lavoro sistematico utilizzando modelli in vivo robusti e riproducibili e metodi standardizzati per identificare nuovi attori nelle metastasi del melanoma. Questo approccio consente l'inferenza dei dati in specifiche fasi sperimentali per caratterizzare con precisione il ruolo di un gene nelle metastasi. I modelli vengono stabiliti introducendo cellule di melanoma geneticamente modificate tramite iniezioni intracardiache, intradermiche o sottocutanee nei topi, seguite dal monitoraggio con imaging seriale in vivo . Una volta raggiunti gli endpoint prestabiliti, i tumori primari e/o gli organi portatori di metastasi vengono prelevati ed elaborati per varie analisi. Le cellule tumorali possono essere ordinate e sottoposte a una qualsiasi delle diverse piattaforme "omiche", incluso il sequenziamento dell'RNA a singola cellula. Gli organi vengono sottoposti ad imaging e analisi immunoistopatologiche per quantificare il carico complessivo delle metastasi e mappare la loro specifica posizione anatomica. Questa pipeline ottimizzata, compresi i protocolli standardizzati per l'attecchimento, il monitoraggio, la raccolta dei tessuti, l'elaborazione e l'analisi, può essere adottata per colture derivate dal paziente, a breve termine e linee cellulari umane e murine consolidate di vari tipi di cancro solido.
Introduzione
L'elevata mortalità associata al melanoma metastatico combinata con una crescente incidenza di melanoma in tutto il mondo1 (un aumento stimato del 7,86% entro il 2025) richiede nuovi approcci terapeutici. I progressi nella scoperta del target dipendono da modelli riproducibili di metastasi, un processo altamente complesso. Durante le fasi della cascata metastatica, le cellule di melanoma devono superare innumerevoli barriere per raggiungere l'evasione del sistema immunitario e la colonizzazione di tessuti distanti2. La resilienza e l'adattabilità delle cellule di melanoma derivano da una moltitudine di fattori, tra cui il loro elevato carico genetico mutazionale3 e la loro origine della cresta neurale, che conferiscono plasticità fenotipica cruciale 3,4,5. Ad ogni fase, i programmi trascrizionali consentono alle cellule di melanoma metastatizzante di passare da uno stato all'altro in base a segnali provenienti dalla diafonia con il microambiente, comprendente il sistema immunitario6, l'ambiente extracellulare 7,8 e l'architettura cellulare delle barriere fisiche9 con cui entrano in contatto. Ad esempio, le cellule di melanoma sfuggono alla sorveglianza immunitaria sottoregolando l'espressione di importanti fattori secreti dal tumore immuno-priming6.
Gli studi descrivono una "nicchia premetastatica", in cui le cellule di melanoma secernono chemochine e citochine per innescare l'organo "bersaglio" distante per le metastasi10. Questi risultati sollevano importanti domande sul tropismo d'organo delle cellule di melanoma metastatico e sul percorso anatomico che intraprendono per accedere a tessuti distanti. Dopo l'intravaso, è noto che le cellule di melanoma metastatizzano attraverso i linfatici (diffusione linfatica) e i vasi sanguigni (diffusione ematogena)2,11. Mentre la maggior parte dei pazienti presenta una malattia localizzata, un piccolo sottogruppo di casi presenta una malattia metastatica a distanza e nessuna disseminazione linfatica (coinvolgimento linfonodale negativo)11, suggerendo l'esistenza di percorsi metastatici alternativi per il melanoma.
Quando colonizzano un sito metastatico, le cellule di melanoma subiscono adattamenti epigenetici e metabolici12,13. Per accedere e invadere nuovi compartimenti, le cellule di melanoma impiegano proteasi14 e modificazioni citoscheletriche 11,15, che consentono loro di attraversare e crescere nella loro nuova posizione. La difficoltà nel colpire le cellule di melanoma risiede nella complessità e nel numero di tali adattamenti; pertanto, il campo dovrebbe fare sforzi per ricreare sperimentalmente il maggior numero possibile di passaggi e adattamenti. Nonostante i numerosi progressi nei saggi in vitro come gli organoidi e le colture 3D16,17, questi modelli ricapitolano solo in modo incompleto la cascata metastatica in vivo.
I modelli murini hanno dimostrato valore trovando un equilibrio tra riproducibilità, fattibilità tecnica e simulazione della malattia umana. Cellule di melanoma impiantate per via intravascolare, ortotopica ed eterotopica da xenotrapianti derivati dal paziente o colture a breve termine in topi immunocompromessi o umanizzati rappresentano la spina dorsale della scoperta del bersaglio nel melanoma metastatico. Tuttavia, questi sistemi spesso mancano di un vincolo biologico cruciale sulle metastasi: il sistema immunitario. I modelli di metastasi del melanoma singenico che possiedono questo vincolo sono relativamente scarsi sul campo. Questi sistemi, sviluppati in topi immunocompetenti, tra cui B16-F1018, la famiglia YUMM di linee cellulari19, SM120, D4M321, RIM322 o più recentemente, RMS 23 e M1 (Mel114433), M3 (HCmel1274), M4 (B2905)24 linee cellulari di melanoma, facilitano lo studio del complesso ruolo della risposta immunitaria dell'ospite nella progressione del melanoma.
Qui, viene presentata una pipeline per l'identificazione del bersaglio delle metastasi del melanoma. Con l'aumento e più grandi set di dati "omici" generati da coorti di pazienti affetti da melanoma, postuliamo che gli studi che detengono la maggiore promessa clinica siano quelli che derivano dall'integrazione dei big data, portando a un meticoloso interrogatorio funzionale e meccanicistico 25,26,27,28. Utilizzando modelli murini per studiare potenziali bersagli nel processo metastatico, si possono tenere conto di eventi specifici in vivo e interazioni tissutali, aumentando così la probabilità di traduzione clinica. Sono delineati diversi metodi per quantificare il carico metastatico, fornendo dati complementari sui risultati di un dato esperimento. Viene descritto un protocollo per l'isolamento unicellulare dai tumori in vari organi per aiutare la caratterizzazione imparziale dell'espressione genica nelle cellule metastatiche, che può precedere il sequenziamento dell'RNA monocellulare o di massa.
Protocollo
NOTA: Le procedure animali coinvolte nel seguente protocollo sono state approvate dal New York University Institutional Animal Care and Use Committee (IACUC). Tutte le procedure sono condotte in strutture approvate dall'Association for Assessment and Accreditation of Laboratory Animal Care International (AAALAC). La Figura 1 illustra l'approccio sperimentale generale.
1. Colture a breve termine di melanoma derivate dal paziente (STCs)
- Posizionare il tessuto in una capsula di Petri da 60 mm con 1 mL di RPMI completo (RPMI 1640 integrato con siero bovino fetale al 10% (FBS), 2 mM di L-glutammina, 1 mM di piruvato di sodio, 1 soluzione di aminoacidi non essenziali MEM e penicillina (100 UI/mL)/streptomicina (100 μg/mL)).
NOTA: Per aumentare il rapporto tra le cellule tumorali, se necessario, sezionare e rimuovere il tessuto che circonda il tumore nella capsula di Petri, al microscopio, utilizzando strumenti chirurgici sterili. - Tagliare finemente il tessuto fresco usando lame di rasoio sterilizzate in cubetti da 1-2 mm. Aggiungere 4 mL di RPMI completo e pipettare il contenuto della piastra su e giù 5-10 volte con una pipetta sierologica da 10 ml.
- Trasferire la sospensione cellulare in un tubo conico in polipropilene da 15 mL e ruotare le celle verso il basso (180 × g per 5 minuti a 4 °C). Aspirare il surnatante, risospesciare il pellet cellulare in 1 mL di terreno fresco e trasferire la sospensione in un matraccio di coltura tissutale da25 cm.
- Per aiutare i frammenti di tessuto ad attaccarsi al fondo, impostare il pallone inclinato con un angolo di 20°-30° in un incubatore di colture tissutali a 37 °C, 5% CO2 per 20 min.
- Stendere il pallone in piano per consentire al mezzo di coprire il tessuto e controllare quotidianamente lo stato della coltura. Dividere le cellule quando raggiungono il 90-100% di confluenza. Mantenere le culture a breve termine a un numero di passaggio "basso".
NOTA: le STC saranno stabilite circa 2 mesi dopo l'isolamento cellulare e la coltura, anche se la tempistica effettiva varia tra campioni e tipi di tumore. Dopo 10-14 passaggi, le linee cellulari raggiungono il 100% di purezza, contenenti solo cellule di melanoma29. La soglia del numero di passaggio è determinata empiricamente osservando i cambiamenti nella morfologia cellulare, nel tempo di raddoppio e nel comportamento in vivo. Per preservare l'eterogeneità e altre caratteristiche del tumore genitore, non dividere le cellule più di 1: 5. - Al momento della creazione di un STC e con qualsiasi modello di linea cellulare da iniettare negli animali come descritto nelle fasi successive, trasdurre le cellule con un reporter.
NOTA: un tag fluorescente (ad esempio, proteina fluorescente rossa (RFP), proteina fluorescente verde (GFP)), ad esempio, consente l'imaging a immunofluorescenza ex vivo e lo smistamento delle cellule tumorali mediante selezione cellulare attivata dalla fluorescenza (FACS). La luciferasi consente l'imaging a bioluminescenza in vivo, uno strumento utile per monitorare la progressione sperimentale (sezione 4).
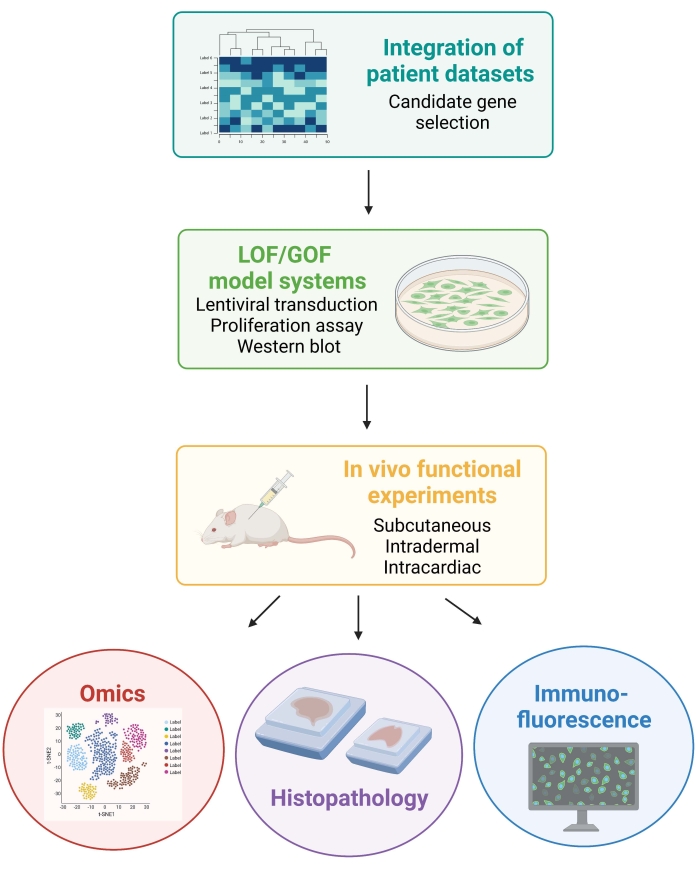
Figura 1: Schema che illustra il flusso di lavoro descritto, dall'integrazione dei dati dei pazienti alla generazione e analisi dei dati in vivo dai topi. Abbreviazioni: LOF = perdita di funzione; GOF = guadagno di funzione. Fare clic qui per visualizzare una versione più grande di questa figura.
2. Impianto di xenotrapianto
NOTA: Le procedure sperimentali qui descritte sono condotte in topi con sistema immunitario adattivo e innato compromesso, NOD.Cg-PrkdcscidIl2rgtm1Wjl/SzJ (NSG); o nei topi che mancano solo di immunità adattativa, come i topi atimici / nudi (NU / J) carenti di cellule T. Gli animali sono di sesso maschile, da 8 a 10 settimane di età. Le femmine spesso mostrano un'alta incidenza di metastasi gonadiche dopo l'iniezione intracardiaca di cellule tumorali, che riduce la loro sopravvivenza.
- Per le iniezioni sottocutanee e intradermiche, preparare una sospensione cellulare 1:1 mescolando una parte di cellule sospese in 1x Soluzione Salina tamponata con fosfato (DPBS) di Dulbecco con una parte di substrato della matrice extracellulare scongelata (EMS) e mantenere il ghiaccio a 4 °C. Per le iniezioni intravascolare (intracardiaca, intracarotide, retro-orbitale, vena della coda o splenica), sospendere le cellule solo in DPBS.
NOTA: Il volume appropriato per le iniezioni intradermiche deve essere mantenuto il più basso possibile (30 μL). Per le iniezioni sottocutanee, il volume iniettato può arrivare fino a 150 μL e per le iniezioni intravascolari fino a 250 μL (in base al peso dell'animale). Aggiungere alla sospensione cellulare finale il 10-30% di volume extra di iniettato, in base alla quantità iniettata e alla siringa utilizzata per tenere conto del volume morto all'interno della punta del tappo e di quello dell'ago (ad esempio, una siringa tubercolina da 1 mL con un ago da 30 G, 25 mm ha un volume morto di 100 μL). - Condurre un progetto pilota per caratterizzare il comportamento delle linee cellulari in uso e la timeline della progressione tumorale in vivo. Per le iniezioni intradermiche, iniziare iniettando da 1.000 a 50.000 cellule/30 μL. Per le iniezioni sottocutanee, iniziare iniettando 10.000 fino a 2 × 106 cellule/150 μL. Per le iniezioni intravascolare (intracardiaca, intracarotide, retro-orbitale e splenica), iniziare iniettando 50.000 cellule/ 150 μL.
NOTA: Le iniezioni intravascolari predispongono gli animali a eventi embolici, sia introducendo aria nel sistema circolatorio sia utilizzando un numero eccessivo di cellule che occludono i piccoli vasi. Mescolare bene la sospensione cellulare per evitare l'aggregazione. Accendere la siringa prima di caricare la sospensione cellulare. Rimuovere eventuali bolle d'aria all'interno della siringa. Tenere la sospensione cellulare/le siringhe su ghiaccio fino al momento del carico e dell'iniezione. - Somministrare l'anestesia per inalazione. Impostare il regolatore del livello di ossigeno tra 1-2 L/min. Posizionare l'animale nella camera di induzione con il vaporizzatore isoflurano impostato al 2,5-5% per l'induzione e all'1,5-3% per il mantenimento.
NOTA: Monitorare la respirazione e la frequenza cardiaca dell'animale durante la fase di induzione dell'anestesia. Non lasciare l'animale incustodito. Non monitorare più di un animale contemporaneamente. Titer la quantità di anestesia al peso dell'animale. - Spostare l'animale dalla camera di induzione nel cono del naso. Applicare un unguento oftalmico petrolato sterile sugli occhi dell'animale per prevenire la secchezza corneale durante la procedura.
- Rasare il sito della procedura con una lama di rasoio dritta inclinata di 30 °. Pulire la pelle dell'area della procedura con tamponi di alcol isopropilico al 70%. Prima di ulteriori passaggi, valutare un livello sufficiente di anestesia con il riflesso del pedale.
- Per le iniezioni intradermiche, eseguire l'intera procedura all'interno di un armadio di biosicurezza per mantenere le condizioni asettiche.
- Anestetizzare e radere l'animale come descritto nei passaggi 2.3-2.5.
- Afferrare e ritrarre la pelle all'indietro contro la traiettoria della pugnalata dell'ago. Utilizzando un ago per siringa da insulina da 31 G, lungo 6 mm, tenuto ad angolo acuto, perforare delicatamente la pelle con la smussatura rivolta verso l'alto.
- Senti il rilascio di pressione sulla punta dell'ago. Avanzare delicatamente per rimanere all'interno del compartimento intradermico e non passare attraverso l'intera profondità della pelle nel sottocute. Cruciale: se si scivola nello spazio sottocutaneo, rimuovere l'ago, cambiare l'area di iniezione e reinserire l'ago. Iniettare lentamente l'intero volume (30 μL) della sospensione cellulare fino a quando non si osserva un wheal a forma di cupola.
NOTA: bassi volumi di iniezione indurranno meno dissezione degli strati cutanei e meno distorsione architettonica. - Tenere l'ago dentro e contare fino a 5.
NOTA: EMS diventa viscoso a temperatura corporea, contribuendo ad evitare il riflusso attraverso la ferita da puntura dell'ago. - Rimuovere l'ago e mettere l'animale in una gabbia su un cuscinetto caldo per recuperare. Riportare l'animale nella gabbia del vivaio dopo aver ripreso conoscenza, quando sternale e ambulatoriale.
NOTA: Monitorare continuamente l'animale durante tutte le procedure descritte in questo protocollo. Non lasciare l'animale incustodito o monitorare più di un animale contemporaneamente. - Monitorare la progressione della crescita del tumore, la perdita di peso e lo stato di salute generale in collaborazione con il personale veterinario ogni giorno nella fase iniziale di crescita e più intensamente, se necessario, dopo che gli animali iniziano a perdere peso. Durante queste sessioni di monitoraggio: pesare gli animali e tracciare un grafico per monitorare la perdita di peso e verificare la presenza di segni di ulcerazione tumorale, segni neurologici, locomotori e / o comportamentali (letargia, mancanza di toelettatura, basso consumo di cibo o acqua).
NOTA: Eutanasia degli animali immediatamente dopo aver osservato i segni di malattia avanzata (perdita di peso superiore al 20%, un punteggio di condizione corporea di <2, livelli di attività estremamente ridotti, paralisi o convulsioni). Utilizzare il metodo di eutanasia approvato dalla IACUC dell'istituzione (ad esempio, una camera CO2 da tavolo automatizzata viene utilizzata per esporre gli animali a CO2 per 15 minuti seguita da un metodo secondario di eutanasia, lussazione cervicale, decapitazione o pneumotorace indotto bilateralmente dall'incisione della gabbia toracica). - Prendi le misure con le pinze e usa le dimensioni di lunghezza (L) e larghezza (W) del tumore per calcolare il volume (V) con la formula:
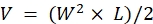
- Per iniezioni sottocutanee:
- Eseguire l'intera procedura all'interno di un armadio di biosicurezza per mantenere le condizioni asettiche26,27.
- Anestetizzare e radere l'animale come descritto nei passaggi 2.3-2.5.
- Utilizzando un ago per siringa per insulina da 28 G a 31 G, lungo 6 mm, tenuto ad angolo acuto, perforare delicatamente la pelle con la smussatura rivolta verso l'alto. Senti il rilascio di pressione sulla punta dell'ago due volte mentre passi attraverso l'epidermide, il derma e l'ipoderma.
NOTA: La seconda volta che si avverte un rilascio di pressione sulla punta dell'ago indica che il compartimento sottocutaneo è stato raggiunto. - Iniettare lentamente l'intero volume (30-150 μL) della sospensione cellulare fino a quando non si osserva un wheal allungato a forma di ellisse. Tenere l'ago dentro e contare fino a 5. Contare fino a 10 per volumi maggiori (più di 50 μL).
NOTA: EMS diventa viscoso a temperatura corporea, contribuendo ad evitare il riflusso attraverso la ferita da puntura dell'ago. - Rimuovere l'ago e mettere l'animale in una gabbia su un cuscinetto caldo per recuperare. Riportare l'animale nella sua gabbia vivaio dopo aver ripreso conoscenza, quando sternale e ambulatoriale.
NOTA: Durante il monitoraggio postprocedurale, osservare eventuali segni di complicanze (bassa frequenza respiratoria, sanguinamento, recupero lento) e affrontarli in modo appropriato. Se non si osserva alcun miglioramento, procedere alle procedure di eutanasia umane descritte nella NOTA del passo 2.6.6. - Monitorare l'animale per la crescita del tumore, la perdita di peso e lo stato di salute generale come descritto nei passaggi 2.6.6-2.6.7.
- Per iniezioni intracardiache:
- Eseguire l'intera procedura all'interno di un armadio di biosicurezza per mantenere le condizioni asettiche26,30.
- Anestetizzare l'animale come descritto nei passaggi 2.3-2.4.
- Trasferire l'animale sulla piattaforma riscaldata della macchina ad ultrasuoni e fissarlo con nastro ipoallergenico al cono del naso.
- Radere il torace con una lama di rasoio dritta inclinata di 30°. Pulire la pelle dell'area della procedura con 3 applicazioni di 10% di povidone-iodio alternate a 3 applicazioni di alcool isopropilico.
- Prima di ulteriori passaggi, valutare un livello sufficiente di anestesia con il riflesso del pedale. Applicare gel ad ultrasuoni sul sito della procedura.
- Cattura la finestra cardiaca con la sonda ad ultrasuoni. Posizionare la sonda ad ultrasuoni al centro del torace sul lato sinistro dell'animale per catturare una finestra orizzontale orientata per ottenere una vista in sezione trasversale (asse corto) del ventricolo sinistro. Assicurandosi che l'asse lungo della sonda sia rivolto verso l'alto, fissare la sonda con un angolo di 50° e la piattaforma riscaldata con un angolo di 20°. Bloccare la sonda e il telaio di supporto in posizione.
- Aspirare la sospensione cellulare mentre si lavora all'interno dell'armadio di biosicurezza in una siringa tubercolina da 1 mL con un ago da 30 G e 25 mm. Rimuovere eventuali bolle d'aria nella siringa.
NOTA: è importante creare e mantenere una sospensione a cella singola durante l'elaborazione e l'iniezione delle cellule. La rimozione delle bolle d'aria è un passo importante per evitare l'embolia aerea. Un sistema di siringa-ago ben innescato impedirà morti evitabili nel gruppo sperimentale. Aspirare sempre più volume nella siringa di quello che verrà iniettato. Il volume extra aiuterà a rimuovere l'aria iniettando parte della sospensione cellulare in un tubo da 1,5 ml. - Bloccare la siringa nell'iniettore stereotassico. Sotto la guida ecografica, far avanzare l'ago attraverso la parete toracica nel ventricolo sinistro del cuore. Iniettare lentamente l'intero volume (100-250 μL) della sospensione cellulare.
- Rimuovere l'ago e mettere l'animale in una gabbia su un cuscinetto caldo per recuperare. Riportare l'animale nella sua gabbia vivaio dopo aver ripreso conoscenza, quando sternale e ambulatoriale. Monitorare l'animale per la crescita del tumore, la perdita di peso e lo stato di salute generale come descritto nel passaggio 2.6.6.
- Per iniezioni intracarotidi:
- Eseguire l'intera procedura su una superficie adeguatamente disinfettata per aiutare a mantenere le condizioni asettiche30.
- Anestetizzare l'animale con un cocktail di ketamina (100 mg/kg) e xilazina (10 mg/kg) mediante iniezione intraperitoneale con una siringa da insulina, ago da 28 G. Applicare un unguento oftalmico petrolato sterile sugli occhi dell'animale per prevenire la secchezza corneale durante la procedura.
- Rasare l'area della procedura con una lama di rasoio dritta inclinata di 30 °. Prima di ulteriori passaggi, valutare un livello sufficiente di anestesia con il riflesso del pedale.
- Metti l'animale sotto uno stereomicroscopio su un pad riscaldante. Pulire la pelle dell'area della procedura con 3 applicazioni di 10% di povidone-iodio alternate a 3 applicazioni di alcool isopropilico.
- Indossare dispositivi di protezione individuale sterili (DPI) e guanti sterili. Preparare il campo sterile posando un drappo sterile sul corpo dell'animale.
NOTA: Se il drappo sterile non ha un foro appropriato per le dimensioni e la posizione dell'incisione, piegare il drappo a metà e utilizzare le forbici Metzenbaum per tagliare il foro di dimensioni appropriate nel mezzo del drappo sterile. - Utilizzare un bisturi o forbici dell'iride per incidere la pelle da metà collo fino allo sterno. Con due pinze microchirurgiche, sezionare senza mezzi termini le 2 ghiandole salivari sottomandibolari nel piano della linea mediana. Utilizzare una cauterizzazione elettrica per l'emostasi, se necessario.
- Sezionare la fascia che circonda l'arteria carotide comune (CCA) dal manubrio verso la biforcazione e continuare medialmente per liberare la parete posteriore della carotide esterna. Tagliare temporaneamente l'arteria carotide esterna (ECA) prima dell'iniezione.
NOTA: Quando si seziona intorno alla circonferenza del CCA, bisogna fare attenzione a non danneggiare il nervo vago (si trova lateralmente all'arteria). - Caricare la sospensione cellulare in una siringa da 1 mL con un ago da 33 G e 15 mm.
- Passa due legature 7-0 sotto il CCA ed esegui un nodo sciolto per ciascuna delle due legature. Utilizzare una clip per recipiente a pressione da 5 mm e 10 G e agganciare temporaneamente l'ECA. Legare la legatura prossimale; quindi, legare liberamente la legatura distale (accanto alla biforcazione del CCA). Utilizzare l'anello distale in un secondo momento per controllare il sanguinamento post-iniezione.
- Utilizzando la siringa con un ago da 33 G e 15 mm, perforare delicatamente il CCA con la smussatura dell'ago rivolta verso l'alto e ad angolo acuto. Iniettare lentamente l'intero volume (50-150 μL) della sospensione cellulare.
- Afferrare l'anello distale con la pinza e sollevarlo mentre si rimuove l'ago per occludere il lume del CCA e fermare l'emorragia. Scambia la siringa con una pinza #7 Jewelers e lega il cappio distale.
- Getta un altro nodo dello strumento sulla legatura distale e rimuovi la clip del vaso dall'ECA. Controllare il campo chirurgico per il sanguinamento e cauterizzare eventuali vasi sanguinanti prima della chiusura. Utilizzare un dispositivo di pinzatura da 9 mm per chiudere la pelle dell'animale e posizionare l'animale su un cuscinetto caldo per recuperare.
NOTA: Rimuovere le graffette 7-10 giorni dopo l'intervento chirurgico. - Somministrare farmaci analgesici per via sottocutanea-Buprenorfina (0,3 mg/ml) ogni 12 ore per 72 ore dopo l'intervento chirurgico ad una concentrazione di 0,1 mg/kg.
NOTA: In alternativa, prendere in considerazione l'utilizzo di un farmaco analgesico a rilascio prolungato, che richiede 1 dose ogni 72 ore. - Riportare l'animale nella sua gabbia vivaio dopo aver ripreso conoscenza, quando sternale e ambulatoriale. Monitorare gli animali postoperatoriamente ogni giorno per segni di infezione o dolore al sito chirurgico, stato di salute generale e complicazioni.
NOTA: Gli animali che non si riprendono bene dalla chirurgia di sopravvivenza possono ricevere dosi aggiuntive di farmaci antidolorifici e saranno sottoposti a eutanasia umana se non completamente recuperati entro 72 ore dopo l'intervento chirurgico. - Monitorare l'animale per la crescita del tumore, la perdita di peso e lo stato di salute generale come descritto nel passaggio 2.6.6.
- Per iniezioni retroorbitali:
NOTA: Utilizzare questa tecnica come alternativa alle iniezioni della vena della coda quando l'operatore è addestrato e competente in questa tecnica e quando vi è una forte giustificazione scientifica. Le sospensioni cellulari erogate attraverso questa via possono indurre la crescita tumorale nello spazio retro-orbitale; quindi, un'attenta considerazione dovrebbe essere data ai rischi e ai benefici quando si sceglie questa tecnica. Ad esempio, per sfruttare la connessione circolatoria diretta del seno venoso retro-orbitale con le vene intracerebrali tramite anastomosi, selezionare questo metodo quando la formazione di tumori cerebrali non è riuscita utilizzando altre vie di iniezione.- Eseguire l'intera procedura all'interno di un armadio di biosicurezza per mantenere le condizioni asettiche. Indossare DPI e guanti sterili.
- Anestetizzare l'animale come descritto nei passaggi 2.3-2.4.
NOTA: Per questa procedura, non applicare un unguento oftalmico petrolato sterile agli occhi dell'animale perché ciò impedirà l'iniezione; applicare solo gocce di anestetico locale. - Caricare la sospensione cellulare in una siringa da insulina con un ago da 28-31 G e 6 mm.
- Con l'animale in posizione prona, ritrarre le palpebre fino a quando l'occhio sporge. Applicare 1 goccia di anestetico locale nell'occhio sul lato sottoposto alla procedura.
- Inserire l'ago con un angolo di 30-45° tra l'occhio e l'epicanto mediale con la smussatura rivolta verso il basso. Iniettare lentamente la sospensione cellulare (10-150 μL).
NOTA: movimenti più lenti prevengono danni all'occhio e il riflusso dell'iniezione. - Eseguire i passaggi descritti in 2.7.5-2.7.6.
- Per le iniezioni di milza:
- Eseguire l'intera procedura all'interno di un armadio di biosicurezza per mantenere le condizioni asettiche31. Indossare DPI sterili e guanti sterili.
- Anestetizzare e radere l'animale come descritto nei passaggi 2.3-2.5.
- Posizionare l'animale in una posizione sdraiata laterale destra. Pulire la pelle dell'area della procedura con 3 applicazioni di povidone-iodio al 10% alternate a 3 applicazioni di alcool isopropilico e preparare il campo chirurgico come descritto nel passaggio 2.9.5.
- Usando le forbici Metzenbaum o un bisturi, fai un'incisione di 1 cm nel fianco sinistro della parete addominale seguita da un'incisione nel peritoneo.
NOTA: La milza sarà vista attraverso la parete addominale traslucida dopo aver praticato l'incisione cutanea. Eseguire l'incisione peritoneale esattamente in questo sito. - Esporre la milza e l'ilo splenico attraverso l'incisione. Utilizzando un ago per siringa da insulina da 28-31 G, lungo 6 mm, perforare delicatamente la milza con la smussatura dell'ago rivolta verso l'alto e ad angolo acuto.
NOTA: se la ferita da puntura sanguina, cauterizzare il sito per limitare il sanguinamento e il riflusso. - Iniettare lentamente l'intero volume (50-100 μL) della sospensione cellulare. Rimuovere l'ago. Posizionare una piccola garza sulla milza e applicare pressione con una pinza. Blocca leggermente la milza tra la garza usando una pinza per zanzare fini e attendi 15 minuti.
- Eseguire una splenectomia legando l'ilo splenico con una sutura di seta 3-0 o 4-0, cauterizzando i vasi se necessario. Chiudere il peritoneo con un polidioxanone 5-0 (PDS) o una sutura assorbibile con acido poliglicolico.
- Eseguire i passaggi descritti nei passaggi 2.7.5-2.7.6.
NOTA: Gli animali che presentano complicazioni emorragiche o che non hanno recuperato completamente 72 ore dopo l'intervento chirurgico devono essere sottoposti a eutanasia umana. Ricorda che il benessere dei topi è la priorità in ogni momento.
3. Chirurgia di sopravvivenza in stadio (SSS)
- Sulla base delle conclusioni sperimentali del passaggio 2.2, determinare il momento giusto per la chirurgia di sopravvivenza. A seconda della linea cellulare e dell'ipotesi sperimentale, selezionare un punto temporale precedente per la resezione del tumore (a un volume tumorale = 150 mm3) o un punto temporale successivo (a un volume tumorale = 500 mm3)26.
NOTA: Il limite di volume tumorale è di 1.500 mm3 quando il carico tumorale è abbastanza elevato da essere dannoso per il benessere dell'animale e predispone a complicanze. - Anestetizzare e radere l'animale come descritto nei passaggi 2.3-2.5.
NOTA: L'intera procedura viene eseguita all'interno di un armadio di biosicurezza. - Pulire la pelle dell'area della procedura con 3 applicazioni di povidone-iodio al 10% alternate a 3 applicazioni di alcool isopropilico e preparare il campo chirurgico come descritto nel passaggio 2.9.5.
- Usando le forbici dell'iride o un bisturi, incidere la pelle, mantenendo un margine di resezione di 5-7 mm dal bordo del tumore.
NOTA: Il margine per la resezione dipende dalla capacità del tumore di diffondersi localmente. Per i tumori aggressivi, aumentare il margine di resezione assicurandosi che rimanga abbastanza pelle per eseguire la chiusura della ferita. - Nel caso di tumori intradermici, resecare il tumore insieme alla pelle circonferenziale.
- Per i tumori sottocutanei, sezionare e rimuovere il tumore sotto la pelle.
NOTA: Se il tumore invade il peritoneo e/o la pelle, resecalo in blocco con il tumore e chiudere il peritoneo con 5-0/4-0 PDS o suture assorbibili con acido poliglicolico. - Chiudere la ferita con il dispositivo di pinzatura da 9 mm.
NOTA: Rimuovere le graffette 7-10 giorni dopo l'intervento chirurgico. Somministrare farmaci analgesici e posizionare l'animale su un cuscinetto caldo per recuperare. Continuare a somministrare farmaci analgesici per 72 ore dopo l'intervento chirurgico, una volta ogni 12 ore secondo il passo 2.9.13. Gli animali con complicanze emorragiche o che non hanno riacquistato piena consapevolezza dopo l'intervento chirurgico dovrebbero essere eutanasia umana. - Casa singola l'animale in una gabbia, su un cuscinetto caldo per recuperare. Riportare l'animale nella gabbia del vivaio dopo aver ripreso conoscenza, quando sternale e ambulatoriale.
- Continuare a monitorare l'animale post-operatorio per recidive locali, perdita di peso, segni neurologici, locomotori e / o comportamentali (letargia, mancanza di toelettatura, basso apporto di cibo o acqua) e stato di salute generale.
4. Imaging in vivo (Figura 2A)
- Somministrare il substrato di D-luciferina (150 mg/kg) agli animali mediante iniezione intraperitoneale con una siringa da 1 mL di insulina, ago da 28 G.
NOTA: Le cellule tumorali devono essere stabilmente trasdotte con il cDNA della luciferasi. - Indurre l'anestesia come descritto nei passaggi 2.3-2.4, 6 minuti dopo l'iniezione del substrato di D-luciferina.
- Eseguire l'imaging utilizzando uno scanner DI imaging a bioluminescenza (BLI) (sistema di imaging in vivo )26.
- Spostare l'animale all'interno della camera di imaging e nel cono del naso. Immagine fino a 5 animali contemporaneamente, a seconda della capacità del sistema di imaging.
- Avviare lo strumento premendo initialize. Impostare l'impostazione del tempo di esposizione su auto (1-120 s).
- Cattura un'immagine vuota per sottrarre qualsiasi sfondo, se necessario. Fare clic su Acquisisci e salvare l'immagine al termine della sequenza di acquisizione.
- Metti l'animale in una gabbia, che si trova con il 50% della superficie di base su un cuscinetto riscaldante per riprendersi dall'anestesia. Riportare l'animale nella gabbia del vivaio dopo aver ripreso conoscenza, quando sternale e ambulatoriale.
- Per l'analisi dei dati nello stesso software di imaging in vivo con cui sono state acquisite le immagini, passare alla cartella in cui vengono salvate le immagini e aprire contemporaneamente le immagini di tutti i mouse relativi all'esperimento.
NOTA: l'analisi di un'immagine alla volta non consente la normalizzazione tra i gruppi. - Impostare le unità su radiance (non conteggi). Assicurarsi che la casella di controllo che indica l'individuo sia deselezionata, in quanto ciò precluderà la normalizzazione del segnale tra i gruppi.
- Utilizzando lo strumento di disegno Region of Interest (ROI), disegna ROI circolari per la regione del cervello e ROI rettangolari per il corpo. Fai attenzione a escludere le orecchie e il naso dal ROI del cervello, poiché tendono a emettere luminescenza non specifica. Per ridurre al minimo i pregiudizi in questo processo, disegna i ROI solo sulle fotografie dei mouse, senza che il segnale luminescente sia sovrapposto.
- Selezionare Misura ROI per quantificare il segnale ed esportare i dati in un foglio di calcolo. Analizza le differenze tra i gruppi tracciando il flusso luminescente totale (p / sec / cm2 / sr) nelle regioni del corpo di interesse.
NOTA: Per valutare le differenze tra i gruppi nel tropismo cerebrale in particolare, calcolare il rapporto tra il segnale cerebrale e il segnale corporeo per ciascun topo. Questo controlla la variazione intermouse nel carico tumorale complessivo e le differenze nei livelli di espressione della luciferasi tra i gruppi sperimentali.
5. Risonanza magnetica ex vivo
- Eseguire la risonanza magnetica ex vivo immediatamente dopo l'eutanasia. In alternativa, prelevare gli organi di interesse, fissarli in formalina per un massimo di 72 ore ed eseguire l'imaging in un secondo momento.
- Acquisisci le immagini con un sistema micro-MRI da 7 Tesla (7 T) (300 MHz) dotato di una console NMR e di un magnete a foro orizzontale a zero emissioni o apparecchiature simili.
NOTA: la necessità di un inserto della bobina di gradiente schermato attivamente con il corretto compromesso di prestazioni è cruciale. Deve fornire una linearità del gradiente di almeno 50 mm di volume sferico dinamico (DSV) per coprire l'insieme di campioni esaminati contemporaneamente senza distorsioni geometriche. La combinazione di intensità del gradiente (che va da 440 a 750 mT/m) e ciclo di lavoro che consente correnti CC simultanee massime comprese tra 3 x 30 A e 3 x 87 A consentirà prestazioni di imaging adeguate. L'inserto a bobina sfumata utilizzato (vedere la Tabella dei materiali) consente le seguenti prestazioni: 660 mT/m, tempo di salita di 130 μs, 3 x 87 A e un DSV = 80 mm. - Eseguire le scansioni con una bobina di radiofrequenza commerciale di trasmissione-ricezione polarizzata circolarmente su tutto il corpo del mouse (OD = 59 mm, ID = 38 mm, L = 40 mm) sintonizzata su 300,16 MHz, la frequenza di Larmor del protone 1H.
NOTA: questa sonda rf consente l'acquisizione di set di dati 3D con risoluzione isotropa submillimetrica (< 150 μm) durante scansioni notturne che coprono 8-12 ore. - Rileva il carico tumorale utilizzando più sequenze30.
NOTA: Il segnale iperintenso rilevato da una sequenza RARE (Rapid Imaging with Refocused Echoes) ponderata T2 riconosce l'edema che circonda i tumori. - Eseguire la sequenza 3D RARE con i seguenti parametri di acquisizione: [120 μm]3 risoluzione isotropa; tempo di acquisizione 5 h, 27 min; tempo di ripetizione (TR) = 500 ms; echo spacing (ES) = 12,7 min; Fattore turbo TFx = 12; tempo di eco effettivo (TEeff) = 76,2 ms; larghezza di banda (BW) = 75 KHz; Dimensione matrice = 2843; campo visivo (FOV) = [4,0 mm]3; numero di medie (Nav) = 6.
- Rileva le metastasi utilizzando i seguenti parametri.
- Per le metastasi pigmentate con schiarimento del segnale, utilizzare una sequenza di eco gradiente 3D pesata T1 con i seguenti parametri: risoluzione isotropa [120 μm]3 ; tempo di acquisizione 2 h, 41 min; TR = 20 ms; tempo di eco (TE) = 4,0 ms; angolo di inversione (FA) = 18°; BW = 75 KHz; Dimensione matrice = 2843; FOV = [34,0 mm]3; Nav = 6.
- Per le metastasi non pigmentate e/o emorragiche, utilizzare un segnale ipointense quando si acquisisce sotto una sequenza di eco multigradiente (MGE) ponderata T2 * (MGE 3D, risoluzione isotropa [120 μm]3 ; tempo di acquisizione 3 h, 35 min; TR = 40 ms; TE = 3,6 ms; ES = 3,2 ms; 4 echi; FA = 20°; BW = 100 kHz; Dimensione matrice = 2843; FOV = (34,0 mm)3; Nav = 4.
- Utilizzare tutte e 3 le sequenze per quantificare il carico tumorale.
- Incrociare le aree tumorali identificate durante l'analisi con sezioni istologiche per garantire l'accuratezza. Vedere paragrafi 7 e 8.
6. Elaborazione tissutale per il sequenziamento di RNA monocellulare o di massa
- Eutanasia dell'animale utilizzando qualsiasi metodo approvato dalla IACUC dell'istituzione. Vedere una delle procedure descritte nella NOTA del passaggio 2.6.6.
- Seziona gli organi di interesse e mettili in pozzetti separati di una piastra contenente hank's Balanced Salt Solution (HBSS) su ghiaccio. Lavora rapidamente e mantieni il tessuto sul ghiaccio in ogni momento per massimizzare la vitalità cellulare.
NOTA: i seguenti passaggi sono specifici per l'elaborazione del cervello. Regola il tipo di collagenasi al tessuto specifico, in base alle tue esigenze. - Preparare una piastra a 6 pozzetti con 3 ml di HBSS in ogni pozzetto.
- Per visualizzare e aiutare ulteriormente a guidare la dissezione, utilizzare un microscopio a fluorescenza e identificare le aree etichettate.
- Sezionare le aree fluorescenti e posizionare i frammenti di tessuto nella piastra a 6 pozzetti (1 frammento per pozzetto se devono essere analizzati singoli focolai metastatici o più frammenti da un organo se devono essere analizzate più metastasi nello stesso organo). Utilizzare lame di rasoio sterili per tritare il tessuto in frammenti il più piccoli possibile (senza spendere più di 1-2 minuti in questo passaggio per ogni campione).
NOTA: Limitare il tempo di elaborazione dei tumori aiuta a preservare la vitalità cellulare. - Aspirare e trasferire il contenuto di ciascun pozzetto in un tubo conico da 15 ml.
NOTA: tagliare la punta di una punta della pipetta da 1.000 μL per facilitare il trasferimento di frammenti più grandi. - Aggiungere 1 mL di HBSS al pozzo e assicurarsi che i frammenti / cellule di tessuto rimanenti vengano trasferiti al tubo da 15 ml, che conterrà un volume finale di 4 ml. Aggiungere 50 μL di collagenasi di tipo I (40 mg/mL) e 12,5 μL di DNasi I (2.000 unità/ml) a ciascun tubo.
- Mettere i tubi conici in un bagno d'acqua riscaldato a 37 °C per 45 min. Ruotare brevemente i tubi conici ogni 5 min.
- Prewet un filtro da 70 μm con HBSS. Utilizzare l'estremità tappata di un tubo microcentrifuga sterilizzato o la parte in plastica di uno stantuffo a siringa e macinare il tessuto omogeneizzato attraverso un filtro da 70 μm in un nuovo tubo conico da 50 ml.
NOTA: La prebagnatura dei filtri con tampone HBSS o FACS facilita lo sforzo. - Lavare il filtro con 1 mL di HBSS. Prewet un filtro da 40 μm con HBSS. Filtrare nuovamente ogni campione attraverso un filtro da 40 μm in un nuovo tubo conico da 50 ml. Aggiungere 1 mL di FBS al filtro da 40 μm per lavarlo. Tenere i tubi conici sul ghiaccio tutto il tempo.
- Riempire il tubo conico a 50 ml con DPBS ghiacciato. Abbassare le cellule (180 × g per 10 minuti a 4 °C). Scartare il surnatante, facendo attenzione a non perdere il pellet cellulare.
NOTA: Per i campioni di cervello, risospesciare le cellule in 2,5 mL di soluzione di separazione della densità al 38% (diluire in HBSS e conservare a temperatura ambiente (RT)). Trasferire su tubi FACS da 5 mL. Girare per 20 minuti a 800 × g. Tagliare la punta di una punta della pipetta da 1.000 μL e rimuovere lo strato superiore di grasso. Non lasciare grasso sulle pareti dei tubi. Se rimane del grasso, gira di nuovo verso il basso e ripeti il processo. Questo è un passo critico . Rimuovere il resto della fase liquida (il pellet sarà traslucido e difficile da visualizzare). - Risospesere le cellule in 1 mL di tampone di lisi dei globuli rossi (RBC) e incubare per 60 s a RT. Spegnere la soluzione di lisi aggiungendo 20 mL di DPBS.
- Girare le cellule a 180 × g per 10 minuti a 4 °C. Rimuovere il surnatante e risospese le cellule in 2 mL di tampone FACS (5% FBS in DPBS).
- Procedere con l'ordinamento delle cellule etichettate e/o la preparazione della libreria per il sequenziamento dell'RNA di massa o a singola cellula.
NOTA: le cellule possono essere filate, congelate a scatto e conservate a -80 °C prima dell'isolamento dell'RNA per l'RNA-seq di massa.
7. Perfusione di tessuti animali e preparazione per analisi immunoistologiche
- Anestetizzare l'animale con un sovradosaggio di ketamina (300 mg/kg) e xylazine (30 mg/kg) cocktail mediante iniezione intraperitoneale con una siringa da insulina e un ago da 28 G.
- Esporre il cuore per dissezione grossolana e fare un'incisione nell'atrio destro. Tenere il cuore delicatamente in posizione con una pinza lunga e curva rivolta verso l'esterno.
NOTA: La formalina e la paraformaldeide (PFA) sono cancerogene. Leggere la scheda di dati di sicurezza (SDS), evitare l'esposizione ai fumi e indossare i DPI appropriati. - Utilizzando una siringa da 10 mL con un ago da 22 G e 22 mm, iniettare 10 mL di DPBS seguiti da 10 mL di PFA al 4% nel ventricolo sinistro.
- Procedere al prelievo degli organi e caricarli in cassette istologiche preetiche. Posizionare le cassette in un contenitore di dimensioni appropriate che contenga abbastanza fissativo (formalina) per coprire i tessuti.
NOTA: Idealmente, il volume fissativo dovrebbe essere 5-10 volte il volume dei tessuti. - Fissare gli organi all'interno delle cassette istologiche in formalina al 10% per 48-72 ore. Scartare la formalina al 10% e lavare le cassette due volte con 1x DPBS.
- Inizia il processo di disidratazione immergendo le cassette nel 70% di etanolo per 2 ore. Continuare a immergere le cassette successivamente in concentrazioni crescenti di etanolo: 80%, 95%, 100% per 1 ora ciascuna. Cambiare la soluzione al 100% due volte dopo 1,5 ore. Immergere le cassette in xilene per 1,5 ore ed eseguire tre cambi della soluzione.
- Incorporare le cassette in cera di paraffina a 58-60 °C. Sezionare i blocchi di paraffina. Procedere alla colorazione ematossilina ed eosina (H & E) orimmunoistochimica.
- Per identificare le cellule di melanoma, utilizzare uno di questi marcatori o un pannello: S100, Melan-A, HMB-45, tirosinasi, MITF. Quando possibile, utilizzare la colorazione Nuclear Mitotic Apparatus Protein (NuMA) in quanto è un marcatore cellulare umano altamente specifico.
NOTA: La colorazione NuMA offre una netta demarcazione tra l'ospite (topo) e le cellule innestate (umane) che aiuta nell'elaborazione delle immagini e nelle successive fasi di quantificazione del tumore.

Figura 2: Esempi di bli, campo luminoso, fluorescenza ex vivo e immagini di colorazione H&E che illustrano l'approccio su più fronti per l'analisi degli effetti dei geni candidati nelle metastasi del melanoma. (A) BLI, (B) BF, (C) fluorescenza ex vivo e (D) immagini di colorazione H&E. Le immagini utilizzate a scopo illustrativo corrispondono a un esperimento in cui 131/6-4L cellule di melanoma trasdotte con uno shRNA di controllo non mirato (shNTC) o uno shRNA mirato a FUT8 sono state iniettate in topi immunodeficienti (NSG). Il silenziamento FUT8 ha compromesso la diffusione metastatica delle cellule di melanoma. Barre di scala e barra di colore = p/sec/cm2/sr × 106 (A), 100 mm (B, C), 100 μm (D). Abbreviazioni: BLI = imaging a bioluminescenza; H&E = ematossilina ed eosina; shRNA = RNA a forcina corta; shNTC = shRNA di controllo non mirato; NSG = immunodeficienza gamma combinata grave diabetica non obesa; FUT8 = fucosiltransferasi 8; BF = campo luminoso. Fare clic qui per visualizzare una versione più grande di questa figura.
8. Colorazione della proteina dell'apparato mitotico nucleare (NuMA) (Figura 3)
- Utilizzare un anticorpo anti-NuMA come marcatore mitotico del fuso altamente specifico per l'uomo per l'identificazione e la quantificazione del carico metastatico nelle sezioni tissutali. 8.1. Si ottiene un'identificazione altamente specifica e sensibile delle cellule di melanoma.
- Se l'immunoistochimica cromogenica per NuMA viene eseguita su uno strumento di immunocolorazione automatica, attenersi alla seguente procedura come descritto32:
- Deparaffinizzare le sezioni in xilene e reidratarle in concentrazioni di etanolo in diminuzione sequenziale. Tenere i vetrini immersi per 15 minuti in xilene e trasferirli al 100% di etanolo per altri 15 minuti.
NOTA: Il resto delle fasi di reidratazione dell'etanolo (95%, 80%, 75%) dura da 3 a 5 minuti ciascuna. - Risciacquare i vetrini in acqua deionizzata.
- Eseguire il recupero dell'epitopo immergendo i vetrini in un contenitore (ad esempio, barattolo di colorazione Coplin) in tampone di citrato di sodio da 10 mM, pH 6,0, in un forno a microonde da 1200 Watt al 100% di potenza per 10 minuti.
- Utilizzare l'anticorpo NuMA anti-umano del coniglio policlonale non coniugato per l'etichettatura, diluito 1:7.000 in albumina sierica Tris-Bovine (BSA) (25 mM Tris, 15 mM NaCl, 1% BSA, pH 7,2). Eseguire i controlli positivi e negativi appropriati in parallelo con le sezioni di studio.
- Incubare i vetrini con l'anticorpo primario per 12 ore. Rileva l'anticorpo primario con multimero coniugato HRP anti-coniglio di capra e visualizza il complesso con 3,3-diaminobenzidina e un potenziatore di solfato di rame.
- Lavare i vetrini in acqua distillata, contromaccare con ematossilina, disidratare e montare con mezzo permanente.
NOTA: le fasi di disidratazione sono l'inverso delle fasi di reidratazione descritte nel passaggio 8.3. Scansiona le diapositive con lo scanner disponibile con un ingrandimento di 20x o 40x e caricale in un database. - Utilizzando il software, disegna ROI per includere tutte le cellule colorate di NuMA all'interno del tessuto dell'organo, escluso altri parenchima d'organo e spazi vuoti.
- Regolare le impostazioni per classificare le cellule NuMA-positive e NuMA-negative utilizzando controlli positivi e negativi appropriati per ciascun organo. Utilizzare un algoritmo software consolidato per quantificare il numero/percentuale totale di cellule NuMA-positive per ciascun campione.
9. Immunofluorescenza della fetta di tessuto
Per identificare lo stadio metastatico in cui è richiesto un particolare candidato gene (ad esempio, stravaso vs sopravvivenza dopo la semina), è possibile determinare l'immunofluorescenza della fetta di tessuto in diversi punti temporali per tracciare la progressione delle cellule tumorali dall'iniezione all'invasione di organi a distanza, semina e crescita. Questo approccio consente l'aggiunta di marcatori per le cellule vicine per catturare l'evento di stravaso e i cambiamenti del microambiente tumorale circostante33.
- Anestetizzare l'animale come descritto al punto 7.1.
- Iniettare 100 μg di fluoroforo coniugato Lycopersicon Esculentum (Pomodoro) Lectina nel ventricolo sinistro di ciascun animale, 3 minuti prima della perfusione, per delineare l'endotelio vascolare.
NOTA: Lasciare il tempo per la lectina di pomodoro di ricircolare nell'intero sistema. - Perfondere l'animale come descritto nei passaggi 7.2-7.3. Prelevare gli organi di interesse e trasferirli in contenitori preetichettati riempiti con il 4% di PFA. Fissare il tessuto per 24-48 ore. Sezionare il tessuto usando un vibratoma in fette spesse 30-50 μm.
NOTA: lo spessore deve essere ottimizzato. Si consigliano fette di spessore compreso tra 30 μm e 50 μm, soprattutto quando si esegue l'imaging z-stack. - Incubare le fette in tampone bloccante (10% Normal Goat Serum, 2% BSA, 0,25% Triton X-100 in DPBS) per 2 ore a RT.
- Eseguire un esperimento di ottimizzazione della colorazione.
NOTA: Poiché il tempo di recupero dell'antigene, i tamponi utilizzati per il recupero dell'antigene, la temperatura, diversi anticorpi / lotti diversi e il tipo di tessuto influenzano la colorazione, sono necessari esperimenti di ottimizzazione. - Aggiungere anticorpi primari a una diluizione ottimizzata e incubare per un tempo ottimizzato a una temperatura ottimizzata (vedere esempi nella Tabella 1).
NOTA: utilizzare controlli appropriati per gli anticorpi primari/secondari e i campioni di tessuto non macchiato. - Lavare le fette di tessuto 3 volte per 5 minuti con lo 0,25% di Triton X-100 in DPBS.
- Incubare le fette di tessuto in anticorpi secondari diluiti in soluzione bloccante per il tempo desiderato (Tabella 1).
- Lavare le fette di tessuto 3 volte per 5 minuti con lo 0,25% di Triton X-100 in DPBS.
- Colorare i nuclei con 4′,6-diamidino-2-fenilindolo (DAPI) diluito 1:1.000 in DPBS o tampone bloccante per 5 min.
- Aggiungere 2 gocce di mezzo di montaggio a fluorescenza antideflagrante a un coperchio e montare il tessuto su vetrini, assicurandosi che le fette siano completamente coperte dal mezzo di montaggio.
NOTA: assicurarsi che non ci siano bolle d'aria direttamente sulle fette in quanto ciò distorce la microscopia. - Cattura immagini confocali con il microscopio disponibile utilizzando un obiettivo ad immersione in olio 60x.
NOTA: quando si acquisiscono immagini confocali, applicare gli stessi parametri di impostazione (tensione, unità ariose e guadagno) su tutte le immagini all'interno dell'esperimento. - Cattura immagini non confocali usando il microscopio a 10x, 20x o 40x.
- Carica le immagini in un software di analisi delle immagini e analizzale confrontando i parametri di scelta (ad esempio, area, numero, intensità dei marcatori o contatto con celle adiacenti).
Risultati
Le figure seguenti illustrano come il flusso di lavoro descritto è stato applicato per l'identificazione di nuovi driver di metastasi del melanoma. La Figura 2 riassume i risultati di uno studio pubblicato in cui sono stati studiati gli effetti del silenziamento della fucosiltransferasi FUT8 nelle metastasi del melanoma in vivo 26. In breve, l'analisi dei dati glicemici dei pazienti umani (ottenuti da array di lectine) e il profilo trascrittomico hanno rivel...
Discussione
Lo scopo di questo rapporto tecnico è quello di offrire un flusso di lavoro standardizzato, dall'alto verso il basso per lo studio di potenziali attori nelle metastasi del melanoma. Poiché gli esperimenti in vivo possono essere costosi e dispendiosi in termini di tempo, le strategie per massimizzare l'efficienza e aumentare il valore delle informazioni ottenute sono fondamentali.
È imperativo utilizzare approcci complementari per incrociare i risultati all'interno dello stesso espe...
Divulgazioni
Gli autori non hanno conflitti di interesse da dichiarare.
Riconoscimenti
Ringraziamo la Division of Advanced Research Technologies (DART) della NYU Langone Health, e in particolare, il Experimental Pathology Research Laboratory, il Genome Technology Center, il Cytometry and Cell Sorting Laboratory, Pre-Clinical Imaging Core, che sono parzialmente supportati dal Perlmutter Cancer Center Support Grant NIH/NCI 5P30CA016087. Ringraziamo il NYU Interdisciplinary Melanoma Cooperative Group (PI: Dr. Iman Osman) per aver fornito l'accesso alle colture a breve termine di melanoma derivate dal paziente + (10-230BM e 12-273BM), che sono state ottenute attraverso protocolli approvati dall'IRB (Universal Consent study #s16-00122 e Interdisciplinary Melanoma Cooperative Group study #10362). Ringraziamo il Dr. Robert Kerbel (Università di Toronto) per aver fornito le linee cellulari di melanoma 113/6-4L e 131/4-5B1* e il Dr. Meenhard Herlyn (Wistar Institute) per aver fornito colture a breve termine di melanoma WM 4265-2, WM 4257s-1, WM 4257-2**. E.H. è supportato da NIH/NCI R01CA243446, P01CA206980, un American Cancer Society-Melanoma Research Alliance Team Science Award e un NIH Melanoma SPORE (NCI P50 CA225450; PI: I.O.). La Figura 1 è stata creata con Biorender.com.
Materiali
| Name | Company | Catalog Number | Comments |
| #15 Scapel Blade | WPI | 500242 | For surgical procedures |
| #3 Scapel Handle | WPI | 500236 | For surgical procedures |
| 1 mL Tuberculin syringe, slip tip | BD | 309626 | Injections |
| 10 mL syringe, slip tip | BD | 301029 | Perfusion |
| 10% Formalin Sodium Buffered | EK Industries | 4499-20L | For perfusion/tissue fixative |
| 15 mL Conical | Corning | 430052 | Cell culture |
| 15 mL Conical Polypropylene Centrifuge Tubes | Falcon | 352196 | Cell culture |
| 200 Proof Ethanol | Deacon Labs | 04-355-223 | Histology |
| 22G – 22mm needle | BD | 305156 | Perfusion |
| 4-0 Vicryl Suture | Ethicon | J464G | Suture |
| 4% Carson's phosphate buffered paraformaldehyde | EMS | 15733-10 | For perfusion/tissue fixative |
| 40µm | Corning | 431750 | Tissue processing |
| 5-0 Absorbable Suture | Ethicon | 6542000 | Closure |
| 50 mL Conical | Corning | 430828 | Cell culture |
| 50mL Conical Polypropylene Centrifuge Tubes | Falcon | 352070 | Cell culture |
| 7-0 Silk suture | FST | 18020-70 | Ligature |
| 70µm | Corning | 431751 | Tissue processing |
| Anti-fade mounting media | Vector Labs | H-1000-10 | Immunofluorescence |
| Approximator applying Forceps, 10cm | WPI | 14189 | For microsurgical procedures |
| Avance | Bruker | 3 HD | NMR Console |
| Biospec 7030 | Bruker | 7030 | Micro MRI |
| BSA | Bioreg | A941 | NuMA Staining |
| Castroviejo suturing forceps, straight tips 5.5mm tying platform, 11cm | WPI | WP5025501 | For microsurgical procedures |
| Coplin Staining Jar | Bel-Art | F44208-1000 | Histology |
| DAPI | Sigma-Aldrich | D9542-1MG | Immunofluorescence |
| dCas9-KRAB | Addgene | 110820 | Genetic manipulation |
| DNase I | NEB | M0303L | Tissue processing |
| DPBS | Corning | 21-030-CM | Tissue processing |
| Extra Sharp Uncoated Single Edge Blade | GEM | 62-0167 | Tissue processing |
| Extracellular Matrix Substrate | Corning | 354234 | Consider the Growth Factor Reduced ( as alternative |
| FBS | Cytiva | SH30910.03 | Cell culture |
| Fiji Image J | Fiji Image J | Software | Immunofluorescence |
| Goat anti-rabbit HRP conjugated multimer | Thermo Fisher | A16104 | NuMA Staining |
| Goat Serum | Gibco | PCN5000 | Immunofluorescence |
| HBSS | Corning | 21-020-CV | Tissue processing |
| Hematoxylin | Richard-Allan Scientific | 7231 | Histology |
| Illumina III | PerkinElmer | CLS136334 | BLI Instrument |
| Insulin syringe 28G - 8mm needle | BD | 329424 | Injections |
| Insulin syringe 31G - 6mm needle | BD | 326730 | Injections |
| Iris Forceps, 10.2cm, Full Curve, serrated | WPI | 504478 | For perfusion and surgical procedures |
| Isoflurane USP | Covetrus | 11695067772 | Anesthesia |
| Jewelers #7 Forceps Titanium 11 cm 0.07 x 0.01 mm Tip | WPI | WP6570 | For microsurgical procedures |
| Ketamine HCl 100mg/mL | Mylan Ind. | 1049007 | Anesthesia |
| lentiCRISPRv2 | Addgene | 98290 | Genetic manipulation |
| Lycopersicon Esculentum (Tomato) Lectin, DyLight 649 | Invitrogen | L32472 | Vascular endothelial cells marker |
| MEM non-essential amino acids X 100 | Corning | 25-025-CI | Cell culture |
| Metzenbaum Scissors | WPI | 503269 | For surgical procedures |
| Microinjection Unit | KOPF | 5000 | Intracardiac injections |
| NaCl | Fisher | S25877 | NuMA Staining |
| Needle 30G x 25mm | BD | 305128 | Intracardiac Injection |
| Needle 33G x 15mm | Hamilton | 7747-01 | Intracarotid Injection |
| Needle holder, Castroviejo, 14cm, with lock, 1.2mm Serrated Jaws | WPI | 14137-G | For microsurgical procedures |
| NOD.Cg-Prkdcscid Il2rgtm1Wjl/SzJ mice | The Jackson Laboratory | 005557 | Murine model |
| NU/J mice | The Jackson Laboratory | 002019 | Murine model |
| Nuclear Mitotic Apparatus Protein polyclonal rabbit anti-human | Abcam | 97585 | NuMA Staining |
| Penicillin-Streptomycin 10000U/mL | Gibco | 15140122 | Cell culture |
| Percoll | GE | 0891-01 | density separation solution |
| PI Classic Surgical Gloves | Cardinal Health | 2D72PT75X | Surgery |
| pLKO Tet-On | Addgene | 21915 | Genetic manipulation |
| Povidone-Iodine 10% Solution | Medline | MDS093943 | Surgery |
| Proparacaine Drops 0.5% | Akorn Pharma | AX0501 | Opthalmic local anesthetic |
| Puralube Petrolatum Opthalmic Ointment | Dechra | 83592 | Anesthesia |
| Razor Blade Double Edge Blades | EMS | 72000 | Shaving and Vibrotome Brain Slicing |
| Reflex 9mm EZ Clip | Braintree | EZC- KIT | Wound closure |
| RPMI 1640 | Corning | 10-040-CM | Cell culture |
| Scissors, Spring 10.5cm Str, 8mm Blades | WPI | 501235 | For microsurgical procedures |
| Semi-Automatic Vibrating Blade Microtome | Leica | VT1200 | Brain Slice Immunofluorescence |
| Single Channel Anesthesia Vaporizer System | Kent Scientific | VetFlo-1210S | Anesthesia |
| Smartbox Tabletop Chamber System and Exhaust Blower | EZ Systems | TT4000 | CO2 Euthanasia |
| Sterile Fenestrated Disposable Drape | Medline | NON21002 | Surgery |
| Sterile Non-Reinforced Aurora Surgical Gowns with Set-In Sleeves | Medline | DYNJP2715 | Surgery |
| T25 Flask | Corning | 430639 | Cell culture |
| Tris | Corning | 46-031-CM | NuMA Staining |
| Triton X-100 | Sigma-Aldrich | X100-500ML | Immunofluorescence |
| Troutman tying forceps, 10cm, Curved G pattern, 0.52mm tip with tying platform | WPI | WP505210 | For microsurgical procedures |
| Vessel clips 10G Pressure 5x 0.8mm Jaws, 5/pkg | WPI | 15911 | For microsurgical procedures |
| Visiopharm | Visiopharm | Visiopharm | NuMA Staining Quantification Software |
| Xylasine 100mg/mL | Akorn Pharma | 59399-111-50 | Anesthesia |
| Xylene | Fisher | X3P-1GAL | Histology |
Riferimenti
- Sung, H., et al. Global Cancer Statistics 2020: GLOBOCAN Estimates of incidence and mortality worldwide for 36 cancers in 185 countries. CA: A Cancer Journal for Clinicians. 71 (3), 209-249 (2021).
- Adler, N. R., Haydon, A., McLean, C. A., Kelly, J. W., Mar, V. J. Metastatic pathways in patients with cutaneous melanoma. Pigment Cell Melanoma Research. 30 (1), 13-27 (2017).
- Platz, A., Egyhazi, S., Ringborg, U., Hansson, J. Human cutaneous melanoma; a review of NRAS and BRAF mutation frequencies in relation to histogenetic subclass and body site. Molecular Oncology. 1 (4), 395-405 (2008).
- Alonso, S. R., et al. A high-throughput study in melanoma identifies epithelial-mesenchymal transition as a major determinant of metastasis. Cancer Research. 67 (7), 3450-3460 (2007).
- Rowe, C. J., Khosrotehrani, K. Clinical and biological determinants of melanoma progression: Should all be considered for clinical management. Australasian Journal of Dermatology. 57 (3), 175-181 (2016).
- Plebanek, M. P., et al. Pre-metastatic cancer exosomes induce immune surveillance by patrolling monocytes at the metastatic niche. Nature Communications. 8 (1), 1319 (2017).
- Orgaz, J. L., et al. Loss of pigment epithelium-derived factor enables migration, invasion and metastatic spread of human melanoma. Oncogene. 28 (47), 4147-4161 (2009).
- Ladhani, O., Sanchez-Martinez, C., Orgaz, J. L., Jimenez, B., Volpert, O. V. Pigment epithelium-derived factor blocks tumor extravasation by suppressing amoeboid morphology and mesenchymal proteolysis. Neoplasia. 13 (7), 633-642 (2011).
- Ju, R. J., Stehbens, S. J., Haass, N. K. The role of melanoma cell-stroma interaction in cell motility, invasion, and metastasis. Frontiers in Medicine - Dermatology. 5, 307 (2018).
- Wiley, H. E., Gonzalez, E. B., Maki, W., Wu, M. T., Hwang, S. T. Expression of CC chemokine receptor-7 and regional lymph node metastasis of B16 murine melanoma. Journal of the National Cancer Institute. 93 (21), 1638-1643 (2001).
- Meier, F., et al. Metastatic pathways and time courses in the orderly progression of cutaneous melanoma. British Journal of Dermatology. 147 (1), 62-70 (2002).
- Turner, N., Ware, O., Bosenberg, M. Genetics of metastasis: melanoma and other cancers. Clinical & Experimental Metastasis. 35 (5-6), 379-391 (2018).
- Ubellacker, J. M., et al. Lymph protects metastasizing melanoma cells from ferroptosis. Nature. 585 (7823), 113-118 (2020).
- Cukierman, E., Pankov, R., Stevens, D. R., Yamada, K. M. Taking cell-matrix adhesions to the third dimension. Science. 294 (5547), 1708-1712 (2001).
- Cunningham, C. C., et al. Actin-binding protein requirement for cortical stability and efficient locomotion. Science. 255 (5042), 325-327 (1992).
- Unger, C., et al. Modeling human carcinomas: physiologically relevant 3D models to improve anti-cancer drug development. Advanced Drug Delivery Reviews. 79-80, 50-67 (2014).
- Fong, E. L., Harrington, D. A., Farach-Carson, M. C., Yu, H. Heralding a new paradigm in 3D tumor modeling. Biomaterials. 108, 197-213 (2016).
- Nakamura, K., et al. Characterization of mouse melanoma cell lines by their mortal malignancy using an experimental metastatic model. Life Science. 70 (7), 791-798 (2002).
- Meeth, K., Wang, J. X., Micevic, G., Damsky, W., Bosenberg, M. W. The YUMM lines: a series of congenic mouse melanoma cell lines with defined genetic alterations. Pigment Cell Melanoma Research. 29 (5), 590-597 (2016).
- Koya, R. C., et al. BRAF inhibitor vemurafenib improves the antitumor activity of adoptive cell immunotherapy. Cancer Research. 72 (16), 3928-3937 (2012).
- Jenkins, M. H. Multiple murine BRaf(V600E) melanoma cell lines with sensitivity to PLX4032. Pigment Cell Melanoma Research. 27 (3), 495-501 (2014).
- Tuncer, E., et al. SMAD signaling promotes melanoma metastasis independently of phenotype switching. The Journal of Clinical Investigation. 129 (7), 2702-2716 (2019).
- Schwartz, H., et al. Incipient Melanoma Brain Metastases Instigate Astrogliosis and Neuroinflammation. Cancer Research. 76 (15), 4359-4371 (2016).
- Perez-Guijarro, E., et al. Multimodel preclinical platform predicts clinical response of melanoma to immunotherapy. Nature Medicine. 26 (5), 781-791 (2020).
- Krepler, C., et al. A Comprehensive Patient-Derived Xenograft Collection Representing the Heterogeneity of Melanoma. Cell Reports. 21 (7), 1953-1967 (2017).
- Agrawal, P., et al. A systems biology approach identifies FUT8 as a driver of melanoma metastasis. Cell. 31 (6), 804-819 (2017).
- Hanniford, D., et al. Epigenetic silencing of CDR1as drives IGF2BP3-mediated melanoma invasion and metastasis. Cancer Cell. 37 (1), 55-70 (2020).
- Kim, H., et al. PRMT5 control of cGAS/STING and NLRC5 pathways defines melanoma response to antitumor immunity. Science Translational Medicine. 12 (551), (2020).
- de Miera, E. V., Friedman, E. B., Greenwald, H. S., Perle, M. A., Osman, I. Development of five new melanoma low passage cell lines representing the clinical and genetic profile of their tumors of origin. Pigment Cell Melanoma Research. 25 (3), 395-397 (2012).
- Morsi, A., et al. Development and characterization of a clinically relevant mouse model of melanoma brain metastasis. Pigment Cell Melanoma Research. 26 (5), 743-745 (2013).
- Huynh, C., et al. Efficient in vivo microRNA targeting of liver metastasis. Oncogene. 30 (12), 1481-1488 (2011).
- Zou, C., et al. Experimental variables that affect human hepatocyte AAV transduction in liver chimeric mice. Molecular Therapy Methods and Clinical Development. 18, 189-198 (2020).
- Kleffman, K., et al. Melanoma-secreted Amyloid Beta Suppresses Neuroinflammation and Promotes Brain Metastasis. bioRxiv. , 854885 (2019).
- Curtis, A., Calabro, K., Galarneau, J. R., Bigio, I. J., Krucker, T. Temporal variations of skin pigmentation in C57BL/6 mice affect optical bioluminescence quantitation. Molecular Imaging and Biology. 13 (6), 1114-1123 (2011).
- Sil, P., Wong, S. W., Martinez, J. More than skin deep: autophagy is vital for skin barrier function. Frontiers in Immunology. 9, 1376 (2018).
- Chen, S., et al. Genome-wide CRISPR screen in a mouse model of tumor growth and metastasis. Cell. 160 (6), 1246-1260 (2015).
- Hart, T., et al. High-resolution CRISPR screens reveal fitness genes and genotype-specific cancer liabilities. Cell. 163 (6), 1515-1526 (2015).
- Wang, T., et al. Identification and characterization of essential genes in the human genome. Science. 350 (6264), 1096-1101 (2015).
- Edgar, R., Domrachev, M., Lash, A. E. Gene Expression Omnibus: NCBI gene expression and hybridization array data repository. Nucleic Acids Research. 30 (1), 207-210 (2002).
- Lappalainen, I., et al. The European Genome-phenome Archive of human data consented for biomedical research. Nature Genetics. 47 (7), 692-695 (2015).
- Cerami, E., et al. The cBio cancer genomics portal: an open platform for exploring multidimensional cancer genomics data. Cancer Discovery. 2 (5), 401-404 (2012).
- Grossman, R. L., et al. Toward a shared vision for cancer genomic data. New England Journal of Medicine. 375 (12), 1109-1112 (2016).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon