Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Beurteilung der Energiesubstratoxidation in vitro mit 14CO2 Trapping
In diesem Artikel
Zusammenfassung
Dieses Protokoll beschreibt eine einfach anzuwendende Methode zur Untersuchung der Substratoxidation durch Verfolgung der 14CO 2 -Produktion in vitro.
Zusammenfassung
Mitochondrien beherbergen die Maschinerie für den Tricarbonsäurezyklus (TCA) und die Elektronentransportkette (ETC), die Adenosintriphosphat (ATP) erzeugen, um die Energiehomöostase aufrechtzuerhalten. Glukose, Fettsäuren und Aminosäuren sind die wichtigsten Energiesubstrate, die die mitochondriale Atmung in den meisten Körperzellen antreiben. Es gibt Hinweise darauf, dass verschiedene Zelltypen eine deutliche Präferenz für bestimmte Substrate haben können. Die Substratverwertung durch verschiedene Zellen im Skelett wurde jedoch nicht im Detail untersucht. Da der Zellstoffwechsel auf physiologische und pathophysiologische Veränderungen abgestimmt ist, können direkte Bewertungen der Substratabhängigkeit in Skelettzellen wichtige Erkenntnisse über die Pathogenese von Knochenerkrankungen liefern.
Das folgende Protokoll basiert auf dem Prinzip der Kohlendioxidfreisetzung aus Substratmolekülen nach oxidativer Phosphorylierung. Durch die Verwendung von Substraten, die radioaktiv markierte Kohlenstoffatome (14C) enthalten, bietet die Methode einen empfindlichen und einfach zu verwendenden Assay für die Rate der Substratoxidation in Zellkultur. Eine Fallstudie mit primären kalvarialen Präosteoblasten im Vergleich zu aus dem Knochenmark abgeleiteten Makrophagen (BMMs) zeigt eine unterschiedliche Nutzung der Hauptsubstrate zwischen den beiden Zelltypen.
Einleitung
Oxidative Phosphorylierung (OXPHOS) in Eukaryoten ist der Prozess, bei dem Nährstoffe in Mitochondrien abgebaut werden, um chemische Energie in Form von ATP durch Sauerstoffverbrauch freizusetzen. Der Katabolismus verschiedener Substrate innerhalb der Mitochondrien durch den Tricarbonsäurezyklus (TCA) erzeugt nur wenige ATP-Moleküle direkt, sondern speichert Energie durch Reduktion der Elektronenträger Nicotinamidadenindinukleotid (NAD+) und Flavinadenindinukleotid (FAD+). Die reduzierten Träger werden dann durch ETC oxidiert, das sich auf der inneren Membran der Mitochondrien befindet, um einen Protonenkonzentrationsgradienten über die Membran zu erzeugen. Die Protonen fließen schließlich durch die ATP-Synthase in ihrem Gradienten zurück in die mitochondriale Matrix, um ATP zu erzeugen. OXPHOS ist das effizienteste Mittel zur ATP-Herstellung aus Energiesubstraten und wird im Allgemeinen in aeroben Umgebungen bevorzugt. Früher wurde angenommen, dass die aerobe Glykolyse - die Produktion von Laktat aus Glukose, während Sauerstoff vorhanden ist - pathophysiologisch ist, oft ein Kennzeichen von Krebszellen. Mehr und mehr wird entdeckt, dass einige normale Zelltypen die aerobe Glykolyse aus Gründen verwenden, die noch nicht vollständig entschlüsselt wurden.
Metabolische Flexibilität ist die Fähigkeit von Zellen oder Organismen, sich an veränderte Energiebedürfnisse und verfügbare Brennstoffquellen anzupassen. Zum Beispiel wird der energetische Bedarf der Skelettmuskulatur hauptsächlich durch OXPHOS im stationären Zustand, aber durch anaerobe Glykolyse während hochintensiver Übungen1 gedeckt. Wenn die Trainingsdauer zunimmt, tragen Glukose- und Fettsäureoxidation mehr zur Gesamtenergieproduktionbei 2. Der Substrateinsatz hängt jedoch nicht nur von der Verfügbarkeit ab, da Substrate während der Oxidation antagonistisch konkurrieren. Vor allem hat sich gezeigt, dass die Fettsäureoxidation die Glukoseverwertung durch die Skelettmuskulatur in einem Phänomen hemmt, das als Randle-Effekt3 bekannt ist. Eine Wechselwirkung wurde durch nachfolgende Studiengezeigt 4,5. Darüber hinaus sind viele Krankheiten mit einer Veränderung der Substratpräferenz und der Entwicklung einer metabolischen Inflexibilität in Zellen verbunden. Zum Beispiel ist die Fettsäureoxidation in der Skelettmuskulatur von Typ-II-Diabetikern im Vergleich zu normalen Kontrollpersonen reduziert6. Die metabolischen Veränderungen in den Krankheitseinstellungen sind Gegenstand intensiver Untersuchungen, da sie zur Pathogenese beitragen können.
Der Energiestoffwechsel in Skelettzelltypen ist relativ wenig erforscht, hat aber in den letzten Jahren an Aufmerksamkeit gewonnen7. Frühere Arbeiten haben gezeigt, dass die aerobe Glykolyse der dominante Energieweg in kalvarialen Osteoblasten ist, während die Glukoseoxidation durch den TCA-Zyklus eine Rolle bei der Osteoklastenbildung spielt 8,9. Andere haben Beweise für Fettsäuren als Energiequelle für Osteoblasten10 geliefert. Es wurde auch gezeigt, dass der Glutaminkatabolismus die Osteoblastendifferenzierung von den Vorläufernunterstützt 11,12. Ein umfassendes Verständnis der Substratnutzung durch verschiedene Skelettzelltypen fehlt jedoch noch. Darüber hinaus wird erwartet, dass Veränderungen im Zellstoffwechsel während der Zelldifferenzierung oder als Reaktion auf pathologische Signale die Nutzung des Brennstoffsubstrats verändern. Im Folgenden wird ein einfach zu bedienendes Protokoll zur Untersuchung der Substratoxidation in vitro beschrieben.
Protokoll
Die Verwendung radioaktiver Stoffe (RAM) bedarf der vorherigen Genehmigung durch einen benannten Sicherheitsausschuss an jeder Institution. Die in diesem Protokoll verwendeten RAMs wurden von Environmental Health & Radiation Safety (EHRS) an der University of Pennsylvania zugelassen. Die Verwendung von Tieren bedarf der vorherigen Genehmigung durch das Institutional Animal Care and Use Committee (IACUC) der Heimeinrichtung. Die folgende Studie wurde von IACUC am Children's Hospital of Philadelphia genehmigt.
1. Herstellung von Stammlösungen für 14C-markierte Substrate
- Stellen Sie 4 mM Rinderserumalbumin (BSA) Stammlösung her, indem Sie 11 g BSA und 33,1 ml Dulbeccos phosphatgepufferte Kochsalzlösung (DPBS) mischen und bei Raumtemperatur für ~ 3 h vorsichtig schütteln, bis BSA vollständig aufgelöst ist. Erwärmen Sie die BSA-Stammlösung vor Gebrauch in einem 70 °C warmen Wasserbad.
- 50 μCi 14C-oleat (50 μCi/μmol in Ethanol) in der Durchstechflasche bei abgenommenem Deckel für 8 h in einer dafür vorgesehenen RAM-Arbeitshaube an die Luft trocknen. 312,5 μLH2Ound dann 3,5 mg (11,5 μmol) Natriumoleat zugeben. Gründlich mischen.
- Erwärmen Sie die resultierende Lösung in einem Wasserbad auf 70 °C. 0,9375 ml der vorgewärmten BSA-Stammlösung hinzufügen, um 10 mM heiße Oleat-Stammlösung (mit einem Verhältnis von 1:11,5 von 14C-Oleaten zu unmarkiertem Oleat) zu erhalten. Aliquot die Lagerlösung und lagern Sie sie bei -20 °C für den langfristigen Gebrauch.
- Verwenden Sie 14 C-Glucose und 14C-Glutamin direkt nach dem Auftauen.
2. Herstellung von Medium, das 14C-markierte Substrate enthält
HINWEIS: Um zuverlässige Konzentrationen von Energiesubstraten in den Medien zu gewährleisten, müssen maßgeschneiderte Medien verwendet werden, wobei kurz vor der Verwendung frische Substrate hinzugefügt werden. Hier wird ein maßgeschneidertes Minimum Essential Medium (MEMα) ohne Glukose, Pyruvat, Glutamin, Phenolrot oder Natriumbicarbonat verwendet. Für jeden Zelltyp sollte jedoch ein optimales Medium bestimmt werden.
- Rekonstituieren Sie 8,67 g des Mediums in 1 l gereinigtem Wasser. Zugabe von 2,2 g Natriumbicarbonat, um einen pH-Wert von 7,4 zu erreichen, und filtere die Lösung mit einer Vakuumfiltration von 0,22 μm.
- Fügen Sie frische Zutaten hinzu, um das vollständige Medium (cMEMα) mit Endkonzentrationen von 5,5 mM Glukose, 2 mM Glutamin, 1 mM Pyruvat und 10% fetalem Rinderserum (FBS) zu erhalten. Weitere ergänzen cMEMα mit 100 mM L-Carnitin und 10 μM HEPES, um die Fettsäureoxidation zu erleichtern.
- Unmittelbar vor der Verwendung 14C-markierte Substrate zu frisch zubereitetem cMEMα hinzufügen, um heiße Medien herzustellen. Um das Oleat-Heißmedium herzustellen, fügen Sie die 10 mM heiße Oleat-Stammlösung zu cMEMα hinzu, um eine Endkonzentration von 100 μM Oleaten (1:100 Verdünnung, endgültige Radioaktivität 0,4 μCi/ml) zu erreichen.
HINWEIS: Der 10 mM heiße Oleatvorrat, der ein Verhältnis von 1:11,5 von heißem:kaltem Oleats enthält (siehe Schritt 1.3), wird verwendet, um ein physiologisches Niveau von 100 μM Gesamtoleat in den Medien zu erreichen und gleichzeitig die Menge an radioaktivem Material zu minimieren. - Machen Sie 10 mM unmarkierte Oleat-Brühe, indem Sie 24,36 mg Natriumoleat zu 2 ml H2O in einem Wasserbad bei 70 ° C für ~ 20 min hinzufügen, bis sie vollständig aufgelöst sind, bevor Sie schnell mit 6 ml der 4 mM BSA-Stammlösung mischen, die auf 70 ° C vorgewärmt sind. Für 1 h in ein 37 °C warmes Wasserbad überführen und durch einen 0,22 μm Filter filtern. Aliquot und bei -20 °C für den Langzeitgebrauch lagern.
- Um Glukose zu heißem Medium zu machen, fügen Sie 14C-Glukose zu einer Endkonzentration von 1,333 μM (1:4.125 Verdünnung, endgültige Radioaktivität 0,4 μCi/ml) hinzu. Um Glutamin zu heißem Medium zu machen, fügen Sie 14C-Glutamin zu der Endkonzentration von 2 μM (1:1.000 Verdünnung, endgültige Radioaktivität 0,4 μCi/ml) hinzu.
- Ergänzen Sie das heiße Glukose- und Glutaminmedium mit einer 10 mM Stammlösung aus unmarkiertem Oleat, aus Schritt 2.4, um die Endkonzentration von 100 μM Oleat, die der im heißen Oleatmedium entspricht, zu erreichen.
3. Vorbereitung der Zellen
HINWEIS: Kalvariale Präosteoblasten und Knochenmarkmakrophagen werden hier als Beispiele verwendet. Benutzer sollten ihren Zelltyp der Wahl nach geeigneten Protokollen vorbereiten. Optimieren Sie die Konzentration von Kollagenase II, die in Pilotversuchen für die Verdauung verwendet werden soll, da die enzymatische Aktivität zwischen verschiedenen Chargen variieren kann.
- Isolierung und Kultur von kalvarialen Präosteoblasten
- Opfern Sie postnatale Tag-3-5 (P3-5) Welpen durch Enthauptung und übertragen Sie sie auf eiskalte DPBS, die Penicillin-Streptomycin (P / S) enthalten.
- Legen Sie die Kalvarien frei, indem Sie die Haut und das Weichgewebe entfernen. Sammeln Sie den mittleren Bereich, indem Sie das umgebende Gewebe in DPBS von hinten nach vorne abschneiden (Abbildung 1A). Kratzen Sie die inneren und äußeren Oberflächen der Kalvaria vorsichtig mit einer Pinzette, um die Zellen bei der anschließenden Verdauung freizusetzen.
- Bereiten Sie eine 2 mg/ml und eine 4 mg/ml Lösung von Collagenase Typ II in DPBS vor und filtern Sie jede Lösung mit einem frischen 0,22 μm Filter. Fermentieren Sie die gereinigten Calvaria zuerst mit der 2 mg / ml Kollagenaselösung für 15 Minuten und entsorgen Sie die Verdauungslösung. Fermentieren Sie die gereinigten Proben in 4 mg / ml Kollagenaselösung 3 Mal für jeweils 15 Minuten, sammeln und speichern Sie die Aufschlusslösung.
- Filtern Sie die Aufschlusslösung durch 70 μm Zellsiebe und zentrifugieren Sie das Filtrat bei 300 × g für 5 min. Resuspendiert das Zellpellet in cMEMα (siehe Schritt 2.2), enthaltend 10% FBS und P/S.
- Zählen und säen Sie die Zellen bei 4 × 104 Zellen/cm2 in 10 cm Platten. Kultivieren Sie die Zellen in einem 37 °C Inkubator mit 5% CO2 für 3 Tage.
- Dissoziieren Sie die Zellen bei 37 °C mit 0,25% Trypsin-EDTA. Zählen und säen Sie die Zellen in 24-Well-Zellkulturplatten mit cMEMα mit 10% FBS und P / S bei 7,5 × 105 / cm2. Samen Sie mindestens 4 Vertiefungen pro Zelltyp pro Substrat und mindestens drei zusätzliche Vertiefungen für jeden Zelltyp für die Zellzählung vor dem Assay.
- Kultivieren Sie die Zellen bei 37 °C über Nacht, bevor die Substratoxidationstests durchgeführt werden.
- Isolation und Kultur von BMMs
HINWEIS: Die Kultur von BMMs erfordert einen Makrophagen-Kolonie-stimulierenden Faktor (M-CSF), der kommerziell als rekombinantes Protein erworben werden kann. Konditioniertes Medium aus der Zelllinie CMG14-12, das zur Expression von M-CSF entwickelt wurde, ist eine wirtschaftliche Alternative, die hier13 verwendet wird.- Sammeln Sie Femuren und Tibias von 8 Wochen alten Mäusen, nachdem Sie die Muskeln und das Bindegewebe entfernt haben. Schneiden und verwerfen Sie beide Enden der Knochen mit einer scharfen Schere.
- Spülen Sie das Knochenmark von einem Femur und einer Tibia in eine 10 cm große Petrischale mit einer Spritze, die mit einer 23-G-Nadel ausgestattet ist, die 15 ml cMEMα mit 10% FBS, P / S und 10% CMG14-12-konditioniertem Medium (BMM-Medium) enthält. Kultivieren Sie die Zellen in der Petrischale in einem 37 °C Inkubator mit 5% CO2.
- Nach 3 Tagen entsorgen Sie die Nährmedien und spülen Sie sie mit DPBS ab. Dissoziieren Sie die angeschlossenen Zellen bei 37 °C mit 0,25% Trypsin-EDTA für 5 min. Zentrifieren und resuspendieren Sie das Zellpellet im BMM-Medium.
- Die Zellen werden in 24-Well-Zellkulturplatten bei 7,5 × 105/cm2 auf die gleiche Weise gezählt und ausgesät, wie in 3.1.6 beschrieben. Kultur bei 37 °C über Nacht im BMM-Medium vor Beginn der Assays.
4. Substratoxidationstest mit CO2-Falle
HINWEIS: Für jeden Zelltyp sollte eine geeignete Seeding-Dichte bestimmt werden, um vor Beginn des Assays eine Konfluenz von 80-90% zu erreichen. Beachten Sie, dass die Zelldichte den Stoffwechselzustand der Zellen beeinflussen kann.
- Waschen Sie die Zellen in den zusätzlichen Vertiefungen zweimal mit DPBS. Dissoziieren Sie die Zellen mit 0,25% Trypsin-EDTA und gemischten 20 μL-Zellen, die in DPBS resuspendiert sind, mit 20 μL Acridin Orange / Propidiumiodid (AO / PI), gebrauchsfertiger kommerzieller Farbstofflösung. Bestimmen Sie die Anzahl der lebenden Zellen mit einem automatisierten Zellzähler und zeichnen Sie die Anzahl für endgültige Berechnungen auf.
- Waschen Sie die Zellen in den Assay-Vertiefungen zweimal mit DPBS. Geben Sie 500 μL heißes Medium zu jedem Assay in der RAM-designierten Gewebekulturhaube. Verschließen Sie die Platten mit Parafilm und inkubieren Sie die Zellen in einem RAM-designierten Inkubator bei 37 °C für 4 h.
- Schneiden Sie während der Inkubation Filterpapier in kreisförmige Stücke, die etwas größer sind als die Fläche in der Kappe von 1,5-ml-Mikrozentrifugenröhrchen, und legen Sie das Papier eng in die Kappe ein (Abbildung 1B). Geben Sie 200 μL 1 M Perchlorsäure zu jedem Röhrchen und 20 μL Natriumhydroxid zu dem Filterpapier, das in der Kappe angebracht ist.
- Nach der Inkubation der Zellen werden 400 μL Kulturmedium von jeder Vertiefung in die vorbereiteten Röhrchen übertragen und die Kappen sofort verschlossen. Lassen Sie die Rohre 1 Stunde bei Raumtemperatur in einem Rohrgestell.
- Stellen Sie während der Inkubation eine Szintillationsdurchstechflasche für jede Tube auf und füllen Sie sie mit 4 ml Szintillationsflüssigkeit. Geben Sie jedes Stück Filterpapier in ein Szintillationsfläschchen und inkubieren Sie es bei Raumtemperatur für 30 min.
- Führen Sie Wischtests an der Gewebekulturhaube, dem Wasserbad (für die 14-C-Oleat-Zubereitung), dem Kühlschrank, dem Inkubator, der Spüle, dem Boden und jedem anderen Arbeitsbereich für eine mögliche RAM-Kontaminationdurch. Legen Sie die Papiertücher in Szintillationsfläschchen, die Szintillationsflüssigkeit enthalten.
- Messen Sie 14° C Radioaktivität in den Szintillationsfläschchen mit einem Szintillationszähler. Notieren Sie die Leseergebnisse. Dekontaminieren Sie die Arbeitsumgebung bei Bedarf nach Strahlenschutzrichtlinien.
5. Datenanalyse
HINWEIS: Unter der Annahme, dass jedes Substrat vollständig oxidiert ist, um CO 2 freizusetzen, kann die Substratoxidationsrate aus der eingeschlossenen CO2-Radioaktivität berechnet werden.
- Berechnen Sie die Substratoxidationsrate unter Verwendung von Eq (1) und den in Tabelle 1 angegebenen X-, Y- und Z-Werten für verschiedene Substrate.
Substratoxidationsrate (μmol/Zelle/h) =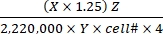 (1)
(1)- Berechnen Sie die Gesamtzerfälle pro min (DPM) für jede Reaktionsbohrung. Für X ist der DPM-Wert (Szintillationszählerwert) von 0,4 ml des Reaktionsmediums zu verwenden, das zumEinfangen von 0,5 ml des gesamten Reaktionsmediums verwendet wird. Daher beträgt der Gesamt-DPM aus jeder Reaktionsquelle X × 1,25.
- Teilen Sie den Gesamt-DPM durch den Faktor 2.220.000, um ihn in μCi umzuwandeln.
- Wandeln Sie die Radioaktivität in μCi in die Anzahl von 14C-markierten Molekülen um. Teilen Sie den μCi-Wert durch die spezifische Aktivität (Y) jedes markierten Substrats. Verwenden Sie die folgenden Y-Werte (Tabelle 1): 14C-Glukose: 300 mCi/mmol; 14 C-Glutamin: 200 mCi/mmol; 14 C-Oleat: 50 mCi/mmol
- Berechnen Sie die Gesamtzahl der oxidierten Moleküle aus den oxidierten 14C-markierten Molekülen, indem Sie letztere mit dem Hot-to-Total-Verdünnungsfaktor (Z) multiplizieren. Verwenden Sie die folgenden Z-Werte (Tabelle 1): Glukose: 4.125; Glutamin: 1.000; Oleat: 12,5
- Teilen Sie das resultierende Produkt durch Zellzahl (Zelle#) und 4 (für die Reaktionszeit von 4 h), um die Substratoxidationsrate pro Zelle zu bestimmen. Verwenden Sie die Zelle#, die in den parallelen Vertiefungen zu Beginn des Assays bestimmt wurde.
Ergebnisse
In diesem Beispiel wird die CO2 -Fangmethode verwendet, um die Substratoxidation durch primäre kalvariale Präosteoblasten mit BMMs zu vergleichen, die häufig für die In-vitro-Osteoblasten- bzw. Osteoblastendifferenzierung verwendet werden. Nachdem die Primärzellen über Nacht in cMEMα durchgangen und kultiviert wurden, erreichen sie typischerweise 80-90% des Zusammenflusses und zeigen ihre charakteristische Morphologie. Die kalvarialen Präosteoblasten sind deutlich größer als BMMs (
Diskussion
Das Protokoll bietet eine einfach zu bedienende Methode zur Bestimmung der Oxidationsrate wichtiger Energiesubstrate. Es ist eine einfachere Alternative zu anderen Protokollen, die Kolben verwenden, die einen zentralen Brunnen enthalten und mit Gummistopfen14,15,16 verschlossen sind. Obwohl die Beispielstudie hier mit Zellkultur durchgeführt wird, kann die Methode leicht für Gewebeexplantationen oder Gewebehomogenate angepasst...
Offenlegungen
Die Autoren erklären keine Interessenkonflikte.
Danksagungen
Die Arbeit wurde zum Teil durch den NIH-Zuschuss R01 AR060456 (FL) unterstützt. Wir danken Dr. Michael Robinson und Elizabeth Krizman (The Children's Hospital of Philadelphia) für ihre großzügige Hilfe bei der Szintillationstheke.
Materialien
| Name | Company | Catalog Number | Comments |
| 0.22 µm filters | Sigma-Aldrich | SLGVM33RS | Used to filter BSA solution |
| 0.25% Trypsin-EDTA | Gibco | 25200056 | Dissociate cells from cell culture plates |
| 1.5 mL Eppendorf tubes | PR1MA | PR MCT17 RB | Used for reaction incubation |
| 10 cm plates | TPP | 93100 | Used for cell culture |
| 10 mL syringe | BD | 302995 | Used to flush marrow from long bones |
| 10% FBS | Atlanta biologicals | S11550 | For Cell culture medium preparation |
| 14C-Glucose | PerkinElmer | NEC042X050UC | Used to make hot media |
| 14C-glutamine | PerkinElmer | NEC451050UC | Used to make hot media |
| 14C-oleate | PerkinElmer | NEC317050UC | Used to make hot media |
| 23 G needle | BD | 305120 | Used to flush marrow from long bones |
| 24-well plates | TPP | 92024 | Used for cell culture |
| 70 μm cell strainers | MIDSCI | 70CELL | Used to filter supernatant during cavarial digestion |
| Acridine Orange/Propidium Iodide (AO/PI) dye | Nexcelom Biosciences | CS2-0106 | Stains live cells to determine seed density |
| Bovine Serum Ablumin | Proliant Biologicals | 68700 | Used for fatty acid conjugation |
| Cellometer Auto 2000 | Nexcelom Biosciences | Determine the number of viable cells | |
| Centrifuge | Thermo Fisher | Legend Micro 21R | Used to pellet cells |
| Collagenase type II | Worthington | LS004176 | Dissociate cells from tissue |
| Custom MEM alpha | GIBCO | SKU: ME 18459P1 | Used to create custom hot media |
| Dulbecco's Phosphate-Buffered Saline | Gibco | 10010023 | Used to dissolve and dilute reagents, and wash culture dishes |
| Filter Paper | Millipore-Sigma | WHA1001090 | Traps CO2 with sodium hydroxide |
| Glucose | Sigma-Aldrich | g7528 | Used to make custom media |
| HEPES | Gibco | 15630080 | Traps CO2 during cell culture |
| L-carnitine | Sigma-Aldrich | C0283 | Supplemented for fatty acid oxidation |
| L-Glutamine | Sigma-Aldrich | g3126 | Used to make custom media |
| MEM alpha | Thermo | A10490 | Cell culture medium |
| Parafilm | Pecheney Plastic Packaging | PM998 | Used to seal cell culture dishes |
| Penicillin-Streptomycin | Thermo Fisher | 15140122 | Prevents contamination in cell culture |
| Perchloric Acid | Sigma-Aldrich | 244252 | Releases CO2 during metabolic assay |
| Pyruvate | Sigma-Aldrich | p5280 | Used to make custom media |
| Scintillation Counter | Beckman Coulter | LS6500 | Determines radioactivity from the filter paper |
| Scintillation Fluid | MP Biomedicals | 882453 | Absorb the energy emitted by RAMs and re-emit it as flashes of light |
| Scintillation Vial | Fisher Scientific | 03-337-1 | Reaction containers for scintillation fluid |
| Sodium carbonate | Sigma-Aldrich | S5761 | Balance buffer for medium |
| Sodium Hydroxide | Sigma-Aldrich | 58045 | Traps CO2 during metaboilc assay |
| Sodium oleate | SANTA CRUZ | SC-215879 | BSA conjugated fatty acid preparation |
| Vaccum filtration 1000 | TPP | 99950 | Filter cMEMα |
Referenzen
- Hargreaves, M., Spriet, L. L. Skeletal muscle energy metabolism during exercise. Nature Metabolism. 2 (9), 817-828 (2020).
- Jeukendrup, A. E. Regulation of fat metabolism in skeletal muscle. Annals of the New York Academy of Sciences. 967, 217-235 (2002).
- Randle, P. J., Garland, P. B., Hales, C. N., Newsholme, E. A. The glucose fatty-acid cycle. Its role in insulin sensitivity and the metabolic disturbances of diabetes mellitus. Lancet. 1 (7285), 785-789 (1963).
- Randle, P. J., Newsholme, E. A., Garland, P. B. Regulation of glucose uptake by muscle. 8. Effects of fatty acids, ketone bodies and pyruvate, and of alloxan-diabetes and starvation, on the uptake and metabolic fate of glucose in rat heart and diaphragm muscles. Biochemical Journal. 93 (3), 652-665 (1964).
- Taegtmeyer, H., Hems, R., Krebs, H. A. Utilization of energy-providing substrates in the isolated working rat heart. Biochemical Journal. 186 (3), 701-711 (1980).
- Cha, B. S., et al. Impaired fatty acid metabolism in type 2 diabetic skeletal muscle cells is reversed by PPARgamma agonists. American Journal of Physiology. Endocrinology and Metabolism. 289 (1), 151-159 (2005).
- Lee, W. C., Guntur, A. R., Long, F., Rosen, C. J. Energy metabolism of the osteoblast: implications for osteoporosis. Endocrine Reviews. 38 (3), 255-266 (2017).
- Li, B., et al. Both aerobic glycolysis and mitochondrial respiration are required for osteoclast differentiation. FASEB Journal. 34 (8), 11058-11067 (2020).
- Lee, W. C., Ji, X., Nissim, I., Long, F. Malic enzyme couples mitochondria with aerobic glycolysis in osteoblasts. Cell Reports. 32 (10), 108108 (2020).
- Kim, S. P., et al. Fatty acid oxidation by the osteoblast is required for normal bone acquisition in a sex- and diet-dependent manner. JCI Insight. 2 (16), 92704 (2017).
- Yu, Y., et al. Glutamine metabolism regulates proliferation and lineage allocation in skeletal stem cells. Cell Metabolism. 29 (4), 966-978 (2019).
- Karner, C. M., Esen, E., Okunade, A. L., Patterson, B. W., Long, F. Increased glutamine catabolism mediates bone anabolism in response to WNT signaling. The Journal of Clinical Investigation. 125 (2), 551-562 (2015).
- Takeshita, S., Kaji, K., Kudo, A. Identification and characterization of the new osteoclast progenitor with macrophage phenotypes being able to differentiate into mature osteoclasts. Journal of Bone and Mineral Research. 15 (8), 1477-1488 (2000).
- Itoh, Y., et al. Dichloroacetate effects on glucose and lactate oxidation by neurons and astroglia in vitro and on glucose utilization by brain in vivo. Proceedings of the National Academy of Sciences of the United States of America. 100 (8), 4879-4884 (2003).
- Oba, M., Baldwin, R. L. 4. t. h., Bequette, B. J. Oxidation of glucose, glutamate, and glutamine by isolated ovine enterocytes in vitro is decreased by the presence of other metabolic fuels. Journal of Animal Science. 82 (2), 479-486 (2004).
- Esen, E., Lee, S. Y., Wice, B. M., Long, F. PTH promotes bone anabolism by stimulating aerobic glycolysis via IGF signaling. Journal of Bone and Mineral Research. 30 (11), 1959-1968 (2015).
- Huynh, F. K., Green, M. F., Koves, T. R., Hirschey, M. D. Measurement of fatty acid oxidation rates in animal tissues and cell lines. Methods in Enzymology. 542, 391-405 (2014).
- Hodson, L., Skeaff, C. M., Fielding, B. A. Fatty acid composition of adipose tissue and blood in humans and its use as a biomarker of dietary intake. Progress in Lipid Research. 47 (5), 348-380 (2008).
- Abdelmagid, S. A., et al. Comprehensive profiling of plasma fatty acid concentrations in young healthy Canadian adults. PLoS One. 10 (2), 0116195 (2015).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten