Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Rektale Organoidmorphologieanalyse (ROMA): Ein diagnostischer Assay bei Mukoviszidose
In diesem Artikel
Zusammenfassung
Dieses Protokoll beschreibt die rektale Organoidmorphologieanalyse (ROMA), einen neuartigen diagnostischen Assay für Mukoviszidose (CF). Morphologische Merkmale, nämlich die Rundheit (Zirkularitätsindex, CI) und das Vorhandensein eines Lumens (Intensitätsverhältnis, IR), sind ein Maß für die CFTR-Funktion. Die Analyse von 189 Probanden zeigte eine perfekte Unterscheidung zwischen CF und Nicht-CF.
Zusammenfassung
Die Diagnose einer Mukoviszidose (CF) ist nicht immer einfach, insbesondere wenn die Schweißchloridkonzentration intermediär ist und/oder weniger als zwei krankheitsverursachende CFTR-Mutationen identifiziert werden können. Physiologische CFTR-Assays (nasale Potentialdifferenz, Darmstrommessung) wurden in den diagnostischen Algorithmus aufgenommen, sind aber nicht immer leicht verfügbar oder durchführbar (z. B. bei Säuglingen). Rektale Organoide sind 3D-Strukturen, die aus Stammzellen wachsen, die aus Krypten einer rektalen Biopsie isoliert wurden, wenn sie unter bestimmten Bedingungen kultiviert werden. Organoide von Nicht-CF-Probanden haben eine runde Form und ein flüssigkeitsgefülltes Lumen, da CFTR-vermittelter Chloridtransport Wasser in das Lumen treibt. Organoide mit defekter CFTR-Funktion quellen nicht an, behalten eine unregelmäßige Form und haben kein sichtbares Lumen. Unterschiede in der Morphologie zwischen CF- und Nicht-CF-Organoiden werden in der 'Rectal Organoid Morphology Analysis' (ROMA) als neuartiger CFTR-physiologischer Assay quantifiziert. Für den ROMA-Assay werden Organoide in 96-Well-Platten plattiert, mit Calcein gefärbt und in einem konfokalen Mikroskop abgebildet. Morphologische Unterschiede werden anhand von zwei Indizes quantifiziert: Der Zirkularitätsindex (CI) quantifiziert die Rundheit von Organoiden, und das Intensitätsverhältnis (IR) ist ein Maß für das Vorhandensein eines zentralen Lumens. Nicht-CF-Organoide haben im Vergleich zu CF-Organoiden ein hohes CI und einen niedrigen IR. ROMA indiziert perfekt 167 Probanden mit CF von 22 Probanden ohne CF, was ROMA zu einem ansprechenden physiologischen CFTR-Assay zur Unterstützung der CF-Diagnose macht. Rektale Biopsien können routinemäßig in jedem Alter in den meisten Krankenhäusern durchgeführt werden, und Gewebe kann an ein zentrales Labor für Organoidkultur und ROMA geschickt werden. In Zukunft könnte ROMA auch angewendet werden, um die Wirksamkeit von CFTR-Modulatoren in vitro zu testen. Das Ziel des vorliegenden Berichts ist es, die für ROMA verwendeten Methoden vollständig zu erklären, um die Replikation in anderen Labors zu ermöglichen.
Einleitung
Mukoviszidose (CF) ist eine autosomal-rezessive Erkrankung, die durch Mutationen im CF-Transmembran-Leitfähigkeitsregulatorgen (CFTR) verursacht wird. Das CFTR-Protein ist ein Chlorid- und Bikarbonatkanal, der die Hydratation mehrerer Epithelien gewährleistet1. CF ist eine hochbelastete, lebensverkürzende, multisystemische Erkrankung, die sich in erster Linie als Atemwegserkrankung manifestiert, aber auch den Magen-Darm-Trakt, die Bauchspeicheldrüse, die Leber und den Fortpflanzungstrakt betrifft2.
Krankheitsverursachende CFTR-Mutationen führen zu einer Abnahme der Menge oder Funktion von CFTR , was wiederum zu Schleimdehydrierung führt. Mehr als 2.000 Varianten im CFTR-Gen wurden beschrieben3, von denen nur 466 gründlich charakterisiert wurden4.
Eine Diagnose von CF kann gestellt werden, wenn entweder die Schweißchloridkonzentration (SCC) über dem Schwellenwert von 60 mmol/L liegt oder wenn zwei krankheitsverursachende CFTR-Mutationen (gemäß der CFTR2-Datenbank) identifiziert werden 4,5. Bei Patienten mit nur mittelstark erhöhtem (30-60 mmol/L) SCC, das bei etwa 4%-5% der Schweißtests auftritt6, und CFTR-Mutationen unterschiedlicher oder unbekannter klinischer Konsequenz kann die Diagnose weder bestätigt noch ausgeschlossen werden, selbst wenn sie CF-kompatible Symptome oder einen positiven neonatalen Screening-Test haben. Für diese Fälle wurden physiologische Second-Line-CFTR-Assays (Nasale Potential Difference (NPD) und Darmstrommessungen (ICM)) in den Diagnosealgorithmus aufgenommen. Diese Tests sind in den meisten Zentren nicht ohne weiteres verfügbar und auch nicht in jedem Alter durchführbar, insbesondere bei Säuglingen5.
Rektale Organoide sind 3D-Strukturen, die aus Lgr5(+) adulten Darmstammzellen aus Darmkrypten gezüchtet werden, die durch Rektalbiopsie gewonnen werden7. Organoide werden zunehmend in der biomedizinischen Forschung eingesetzt, etwa bei der Testung der Modulatorbehandlung bei CF8. Eine lebensfähige Biopsie kann entweder durch Absaugen oder Zangenbiopsie erreicht werden, ein Verfahren, das nur minimale Beschwerden verursacht und selbst bei Säuglingen mit niedrigen Komplikationsraten sicher ist9. Die aus den rektalen Biopsien isolierten Krypten werden in Stammzellen angereichert, und unter bestimmten Kulturbedingungen organisieren sich diese selbst zu rektalen Organoiden. Die Morphologie dieser Organoide wird durch die Expression und Funktion von CFTR bestimmt, das sich an der apikalen Membran von Epithelzellen befindet. Funktionelle CFTR lässt Chlorid und Wasser in das Organoidlumen eindringen, wodurch eine Quellung von Nicht-CF-Organoiden induziert wird. CF-Organoide quellen nicht an und haben kein sichtbares Lumen10,11.
Die rektale Organoidmorphologieanalyse (ROMA) ermöglicht die Unterscheidung zwischen CF- und Nicht-CF-Organoiden basierend auf diesen Unterschieden in der Organoidmorphologie. Nicht-CF-Organoide sind runder und haben ein sichtbares Lumen, während das Gegenteil für CF-Organoide gilt. Für diesen Assay werden patientenspezifische Organoide in 32 Vertiefungen einer 96-Well-Platte plattiert. Nach 1 Tag des Wachstums werden die Organoide mit Calceingrün gefärbt und in einem konfokalen Mikroskop abgebildet. Die Nicht-CF-Organoide zeigen eine kreisförmigere Form und einen weniger fluoreszierenden zentralen Teil, da das Lumen Flüssigkeit enthält und Calcein nur Zellen färbt. Diese Unterschiede in der Morphologie werden mit zwei ROMA-Indizes quantifiziert: Der Zirkularitätsindex (CI) quantifiziert die Rundheit von Organoiden, während das Intensitätsverhältnis (IR) ein Maß für das Vorhandensein oder Fehlen eines zentralen Lumens ist. In diesem Bericht beschreiben wir detailliert das Protokoll, um diese diskriminativen Indizes zu erhalten, um die Replikation der Technik zu ermöglichen.
Protokoll
Für alle Eingriffe mit menschlichem Gewebe wurde die Genehmigung durch die Ethikkommission Forschung UZ/KU Leuven (EC Research) eingeholt. Alle Untersuchungen wurden mit informierter Zustimmung und / oder Zustimmung von Eltern, Vertretern und / oder Patienten durchgeführt.
HINWEIS: Alle Verfahren mit rektalen Biopsien und Organoiden sollten in einem laminaren Fluss durchgeführt werden, um den Forscher vor biologischen Gefahren zu schützen und das Risiko einer Kontamination der Kulturen zu minimieren. Wie bei jedem Laborverfahren sollten Forscher jederzeit Laborkittel, Handschuhe und Schutzbrillen tragen, um Proben zu manipulieren.
1. Rektale Biopsie, Isolierung adulter Stammzellen aus Krypten und Organoidkultur
- Für diesen ersten Teil des Protokolls, einschließlich Medienproduktion und -nutzung, Organoidkultur, Spaltung, Expansion und Einfrieren für Biobanking, folgen Sie den beiden zuvor veröffentlichten Protokollen 8,12.
- Kurz gesagt, müssen folgende Schritte unternommen werden:
- Nehmen Sie drei bis vier rektale Biopsien mit einer Pinzette oder Absaugvorrichtung und sammeln Sie in einem sterilen Behälter (z. B. 1,5 ml Mikrozentrifugenröhrchen) mit dem Ad-DF+++ Medium, wie im oben genannten Protokoll beschrieben.
- Transport von Biopsien in ein zentrales Labor auf Eis oder bei 4 °C. Der Transport zu anderen Zentren ist möglich, und die Qualität wird nicht wesentlich beeinträchtigt, auch wenn der Transport bis zu 48 Stunden dauert. Wenn der Transport voraussichtlich 6 Stunden dauert, verwenden Sie ein 15-ml-konisches Röhrchen mit 6 mL Ad-DF+++ -Medium anstelle eines 1,5-ml-Mikrozentrifugenröhrchens.
- Waschen Sie die Biopsien in kaltem PBS, bis der Überstand klar ist, um die Ablagerungen und nicht-epithelialen Gewebe wie Fettgewebe zu entfernen.
- Inkubieren Sie die Biopsien mit EDTA (Endkonzentration 10 mM), um die Krypten zu lösen. Verkleiden Sie die Krypten in einer Basalmembranmatrix. Fügen Sie dem Medium in der ersten Woche der Kultivierung Breitbandantibiotika (Gentamicin 50 μg / ml und Vancomycin 50 μg / ml) hinzu, um eine bakterielle Kontamination zu verhindern.
- Wenn die Krypten knospen haben und geschlossen und vermehrt sind, führen Sie eine mechanische Spaltung durch, normalerweise nach 7 Tagen.
- Teilen Sie die resultierenden Organoide ungefähr alle 7 Tage. Auf diese Weise erweitern Sie die Kultur, frieren Backup-Proben in einer Biobank ein oder verwenden Sie die Organoide für Assays.
2. Organoid-Beschichtung für ROMA (Tag 1)
- Mechanische Spaltung der Organoide für die Beschichtung
- Sammeln Sie Organoide aus drei gut gewachsenen Vertiefungen von einer 24-Well-Platte, indem Sie zweimal mit kaltem Ad-DF+++ Medium waschen, sammeln Sie sie in einem 1,5-ml-Mikrozentrifugenröhrchen und beurteilen Sie sie unter dem Mikroskop12.
- Stellen Sie sicher, dass Organoide lebensfähig und von hoher Qualität sind, was normalerweise nach 5-7 Tagen Wachstum nach vorheriger Spaltung erreicht werden kann (Abbildung 1).
- Die Organoide werden gemäß dem oben genannten Protokoll 8,12 mechanisch aufgeteilt. Wiederholen Sie, bis die Mehrheit der Organoide, wie mit dem Hellfeldmikroskop beurteilt, im Vergleich zur ersten Beobachtung klein genug ist.
- Sammeln Sie die kleineren Organoide, die normalerweise der Sammlung von etwa den oberen 4/5 der Organoid-Medium-Lösung in einem neuen Mikrozentrifugenröhrchen entsprechen, da große Organoide aufgrund der Schwerkraft auf den Boden des Röhrchens sinken und kleine Organoide im oberen Teil der mittleren Säule suspendiert bleiben.
- Zentrifugieren Sie die Probe kleinerer Organoide bei 0,3 x 1000 g für 2 min und verwerfen Sie das Medium.
- Verdünnen Sie das Pellet kleinerer Organoide in 130 μL 40%-50% Basalmembranmatrix (verdünnt mit Ad-DF+++ medium12).
- Mit einer 200 μL Mikropipette gut resuspendieren.
- Beschichten Sie die Organoide in einer 96-Well-Platte
- Unter Verwendung einer 20-μL-Pipette werden Organoide in 32 Vertiefungen einer vorgewärmten 96-Well-Platte verkleidet. Stellen Sie sicher, dass jede Vertiefung einen 4-μL-Tropfen der im vorherigen Schritt hergestellten Organoid-Matrix-Lösung enthält.
- Die Organoide in der Lösung neigen aufgrund der schwerkraftabhängigen Abwärtsverschiebung dazu, ein Pellet am Boden des Rohres zu bilden. Um dies zu verhindern und eine gleichmäßige Beschichtung zu gewährleisten, resuspendieren Sie die Organoid-Matrix-Lösung regelmäßig mit einer 200-μL-Mikropipette (z. B. jedes Mal nach der Beschichtung von vier bis acht Vertiefungen).
- Legen Sie jeden Tropfen in die Mitte des Vertiefungs, um zu verhindern, dass der Tropfen zu den Rändern des Vertiefungs läuft, was die Bildqualität später verringern würde.
- Streben Sie etwa 30 Organoide pro Vertiefung an (mindestens 15, maximal 90) ohne überlappende Organoide.
- Denken Sie daran, dass diese Organoide über Nacht inkubiert werden müssen und während dieser Zeit leicht wachsen und sich überlappen können, wenn die Beschichtungsdichte zu hoch ist.
- Überprüfen Sie die Beschichtungsdichte mit einem Hellfeldmikroskop mit einem 5-fachen Vergrößerungsobjektiv, nachdem Sie die ersten ein oder zwei Vertiefungen plattiert haben.
- Wenn die Beschichtungsdichte zu hoch ist, wird die Organoid-Matrix-Lösung schrittweise mit der Zugabe der Basalmembranmatrix verdünnt, bis die gewünschte Beschichtungsdichte erreicht ist (Abbildung 2).
- Klopfen Sie die Platte nach dem Plattieren vorsichtig auf eine ebene Oberfläche, um sicherzustellen, dass sich die Mehrheit der Organoide in derselben Fokusebene befindet.
- Inkubieren Sie die Platte
- Inkubieren Sie die 96-Well-Platte bei 37 °C und 5% CO2 während 8-10 min, um die Gelierung der Basalmembranmatrix zu ermöglichen.
- Fügen Sie 50 μL humanes Dickdarm-Organoid-Medium +/+12 in jede Vertiefung und inkubieren Sie über Nacht für 16-24 h.
- Fügen Sie keine Forskolin- oder CFTR-Modulatoren hinzu, so dass die Organoide unter basalen Bedingungen wachsen.
3. Organoide Bildgebung mittels konfokaler Mikroskopie (Tag 2)
- Färben Sie die Organoide mit Calceingrün
- Bereiten Sie eine 1 mM Stammlösung von Calceingrün vor, indem Sie 50 μL Dimethylsulfoxid (DMSO) zu einer Durchstechflasche mit 50 μg Calceingrün hinzufügen.
- Bereiten Sie eine Arbeitslösung von Calceingrün vor, indem Sie 1,2 μL der Stammlösung zu 200 μL Ad-DF+++ (Konzentration 6 μM) hinzufügen.
- Geben Sie 5 μL dieser Calceinmischung in jede Vertiefung der 96-Well-Platte, in der Organoide plattiert wurden (endgültige Calceinkonzentration 0,6 μM). Berühren und dislozieren Sie den Matrixtropfen im Bohrloch nicht, wenn Sie diesen Schritt ausführen.
- Drehen und kippen Sie die Platte mit dem Deckel einige Male, um eine homogene Verteilung des Calceingrüns im gesamten Vertiefung zu gewährleisten.
- Inkubieren Sie die Platte erneut für 15-30 min bei 37 °C und 5% CO2 , um eine Färbung aller Organoide in den Vertiefungen sicherzustellen.
- Übertragen der Platte auf das konfokale Mikroskop
- Überführen Sie die Platte mit einem automatisierten Tisch und integriertem Inkubator auf das konfokale Mikroskop. Stellen Sie sicher, dass die Platte gut im Plattenhalter befestigt ist.
- Eine Inkubation bei 37 °C und 5% CO2 ist optional, aber aufgrund der kurzen Dauer (ca. 10 min) des Bildgebungsverfahrens nicht notwendig.
- Fokus auf die Organoide (Abbildung 3)
- Bestimmen Sie mit dem konfokalen Mikroskop manuell die optimale x/y-Position und den Fokus (z-Position) der Organoide in jeder Vertiefung und speichern Sie diese Positionen in der Bildgebungssoftware.
- Der beste Fokus impliziert eine möglichst scharfe Abgrenzung der Organoide und die Visualisierung des größtmöglichen Teils des Organoidtropfens. Wenn die Verteilung des Flecks nicht ausreichend ist, wiederholen Sie die Schritte 3.1.4 und 3.1.5 (z. B. weitere 5-10 Minuten inkubieren).
- Verwenden Sie Lebendzellbildgebungseinstellungen mit Emission bei 488 nm und Anregung bei 515 nm (spezifisch für die Visualisierung der Calceinfluoreszenz) und ein 5-faches LD-Vergrößerungsobjektiv.
- Nehmen Sie Organoidbilder der 32 Vertiefungen mit dem konfokalen Mikroskop auf (Abbildung 3 und Abbildung 4).
- Nehmen Sie Bilder unidirektional mit einer Auflösung von 1024 Pixel x 1024 Pixel (Pixelgröße 2,5 μm x 2,5 μm) und einer Tiefe von 16 Bit auf.
- Wählen Sie die Laserintensität und Masterverstärkung für eine optimale Visualisierung morphologischer Unterschiede zwischen CF- und Nicht-CF-Organoiden (z. B. Unterscheidung zwischen einem nicht gefärbten wassergefüllten zentralen Lumen (falls vorhanden) und einer gefärbten Zellgrenze).
HINWEIS: Organoide werden nicht korrekt abgegrenzt, wenn die Masterverstärkung und damit das Fluoreszenzsignal zu niedrig sind; Wenn Sie die Masterverstärkung zu hoch einstellen, erhalten Sie Bilder mit Organoiden mit homogener sehr hoher Signalintensität, wodurch die Abbildung subtilerer morphologischer Unterschiede verhindert wird (Abbildung 2). - Speichern Sie ein Bild pro Vertiefung für alle 32 Vertiefungen im Mikroskopformat und exportieren Sie sie als TIFF-Dateien.
4. Bildanalyse (Abbildung 5)
- Laden Sie die TIFF-Dateien in die Bildanalysesoftware.
- Führen Sie die erste Qualitätsprüfung anhand der vom Bediener festgelegten Ausschlusskriterien durch (Abbildung 2): viele differenzierte oder tote Strukturen oder Trümmer, unzureichende Beschichtungsdichte, zu viele (z. B. überlappende) oder zu wenige Organoide und unzureichende Fluoreszenzverteilung (Organoide nicht klar abgegrenzt, Hintergrundsignal zu hoch).
HINWEIS: Dieser Schritt kann auch vor dem Export von Bildern als TIFF-Dateien in Schritt 3.4 ausgeführt werden. - Vorbereiten von Bildern für die Analyse
- Kalibrieren Sie Bilder neu, sodass 1 Pixel 2,5 μm x 2,5 μm entspricht.
- Erstellen und öffnen Sie eine Netzwerkdaten (. ND) aller 32 Bilder für jede Organoidkultur, was die gleichzeitige Analyse aller 32 Bilder pro Subjekt ermöglicht.
- Abgrenzung der Organoide
- Beschreiben Sie Strukturen mit einer unteren Intensitätsschwelle von 4.500 und einer oberen Schwelle von 65.535 (Smooth and Clean-Funktionen aus; Funktion "Löcher füllen "; Separate Funktion bei x3).
HINWEIS: Dies grenzt fluoreszierende Strukturen ab, wobei Fülllöcher einschließlich des Lumens in der abgegrenzten Struktur vorhanden sind.
- Beschreiben Sie Strukturen mit einer unteren Intensitätsschwelle von 4.500 und einer oberen Schwelle von 65.535 (Smooth and Clean-Funktionen aus; Funktion "Löcher füllen "; Separate Funktion bei x3).
- Zählen Sie die Organoide
- Wählen Sie alle Strukturen ≥40 μm.
- Klicken Sie auf die Schaltfläche ND-Messung aktualisieren (jedes Mal, wenn eine Messung erforderlich ist); Die gezählten Organoide werden nummeriert.
HINWEIS: Dies entspricht der Gesamtzahl der Organoide in den 32 Vertiefungen, während kleine Trümmer, wie tote Zellen, ausgeschlossen sind.
- Messintensität und Zirkularität zur Berechnung der Indizes
- Wählen Sie alle Strukturen ≥60 μm aus und zählen Sie.
HINWEIS: Dies zählt die Organoide, die groß genug sind, um eine Morphologie zu zeigen, die entweder für CF oder Nicht-CF typisch ist. Organoide >40 μm und <60 μm sind klein und dicht, sowohl bei Nicht-CF als auch bei CF. - Entfernen Sie alle Strukturen, die den Rand des Bildes berühren. Tun Sie dies, um Organoide zu entfernen, die nicht vollständig sichtbar sind, da ihre Morphologie nicht genau quantifiziert werden kann.
- Erodieren Sie 1 Pixel (= 2,5 μm) vom Rand jeder ≥60 μm-Struktur. Dadurch wird der Halo der diffusen Calceinfluoreszenz, der die Organoide umgibt, entfernt.
- Messen Sie die mittlere Intensität jeder Struktur. Auf diese Weise wird die mittlere Fluoreszenz der Organoide gemessen.
- Selektieren Sie erneut alle Strukturen ≥60 μm und entfernen Sie alle Strukturen, die die Ränder berühren.
- Erodieren Sie 10 Pixel (= 25 μm) vom Rand jeder ≥60 μm-Struktur.
HINWEIS: Bei Nicht-CF-Organoiden erodiert dies den Zellrand und hinterlässt nur das Lumen. Bei CF-Organoiden erodiert dies den äußeren Teil des Organoids, der ungefähr die gleiche Fluoreszenz aufweist wie der verbleibende innere Teil. - Messen Sie die mittlere Intensität jeder erodierten Struktur. Auf diese Weise wird die mittlere Fluoreszenz des zentralen Teils der Organoide gemessen.
- Messen Sie die Kreisförmigkeit jeder Struktur. Dies entspricht der mittleren Zirkularität der Organoide.
- Wählen Sie alle Strukturen ≥60 μm aus und zählen Sie.
- Führen Sie die zweite Qualitätsprüfung durch: von der Software festgelegte Ausschlusskriterien. Schließen Sie den Satz von Bildern aus, wenn weniger als 50% der Organoide (definiert als Strukturen ≥40 μm) ≥60 μm sind, da genügend Organoide groß genug sein sollten, um eine für CF oder Nicht-CF typische Morphologie zu zeigen. Schließen Sie auch aus, wenn in den 32 Vertiefungen <500 oder >3.000 Organoide (definiert als Strukturen ≥40 μm) vorhanden sind.
5. Messen Sie die Indizes in der Imaging-Software (Abbildung 6)
- Messen Sie den Zirkularitätsindex (CI).
HINWEIS: Dies entspricht der in Schritt 4.6 gemessenen mittleren Zirkularität, d.h. der mittleren Zirkularität aller Organoide in allen 32 Vertiefungen. CI quantifiziert die Rundheit der Organoide, definiert als , die bei CF niedriger ist als bei Nicht-CF-Organoiden.
bei Nicht-CF-Organoiden. - Messen Sie das Intensitätsverhältnis (IR)
- Berechnen Sie den IR, indem Sie den Mittelwert der Intensitätsmessung nach dem Erodieren von 25 μm vom Rand jeder ≥60 μm-Struktur durch den Mittelwert der Intensitätsmessung nach dem Erodieren von 2,5 μm vom Rand jeder ≥60 μm-Struktur dividieren.
HINWEIS: IR misst das Vorhandensein oder Fehlen eines zentralen Lumens. IR ist gleich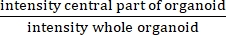 und ist bei CF höher als bei Nicht-CF-Organoiden.
und ist bei CF höher als bei Nicht-CF-Organoiden.
- Berechnen Sie den IR, indem Sie den Mittelwert der Intensitätsmessung nach dem Erodieren von 25 μm vom Rand jeder ≥60 μm-Struktur durch den Mittelwert der Intensitätsmessung nach dem Erodieren von 2,5 μm vom Rand jeder ≥60 μm-Struktur dividieren.
- Vereinfachen Sie den Analyseprozess
- Bereiten Sie ein Standardarbeitsblatt mit Tabellenkalkulationssoftware vor, um diese Indizes beim Kopieren der Daten automatisch zu berechnen (Abbildung 7).
- So berechnen Sie die IR:
- Führen Sie die Intensitätsmessung durch, nachdem Sie 2,5 μm vom Rand jeder ≥60 μm-Struktur erodiert haben. Verwenden Sie den Mittelwert für die Berechnung (Nenner).
- Führen Sie die Intensitätsmessung durch, nachdem Sie 25 μm vom Rand jeder ≥60 μm-Struktur erodiert haben. Verwenden Sie den Mittelwert für die Berechnung (Zähler).
- Um das CI zu berechnen, nehmen Sie die Zirkularität aller Organoide in allen Vertiefungen. Der Mittelwert entspricht dem KI.
HINWEIS: Wenn die Analyse großer Bildmengen erforderlich ist, kann der Bildanalyseprozess, wie in Abschnitt 4 beschrieben, halbautomatisiert werden, beginnend mit den kalibrierten ND-Dateien und Ausführen eines Makros, wobei alle Schritte kombiniert werden.
Ergebnisse
Organoide von 212 Probanden wurden während routinemäßiger klinischer Besuche gesammelt. Während oder nach der rektalen Biopsie traten keine unerwünschten Ereignisse auf. Organoide wurden von einem Forscher abgebildet, der für Probandenmerkmale wie Genotyp und klinische Informationen blind war. Aufgrund der schlechten Bildqualität wurden 23 Probanden ausgeschlossen. Beispiele für erfolgreiche und gescheiterte Organoidkulturen und Bildaufnahmen sind in Abbildung 2 zu sehen.
Diskussion
Wir stellen ein detailliertes Protokoll für die rektale Organoidmorphologieanalyse (ROMA) zur Verfügung. Die beiden mit ROMA, IR und CI berechneten Indizes unterschieden Organoide von Probanden mit CF von denen ohne CF mit perfekter Genauigkeit. ROMA könnte somit als neuartiger physiologischer CFTR-Assay fungieren, der SCC und andere derzeit verfügbare Testsergänzt 13,14,15.
Das Protokoll ist abh...
Offenlegungen
Diese Studie wurde von der belgischen CF-Patientenvereinigung "Mucovereniging/Association Muco", dem Research Grant der Belgian Society of Paediatrics BVK-SBP 2019 und einem Grant des UZ Leuven Fund for Translational Biomedical Research finanziert. Die Autoren erklären keinen Interessenkonflikt.
Danksagungen
Wir danken den Patienten und Eltern, die an dieser Studie teilgenommen haben. Wir danken Abida Bibi für alle Kultivierungsarbeiten mit den Organoiden. Wir danken Els Aertgeerts, Karolien Bruneel, Claire Collard, Liliane Collignon, Monique Delfosse, Anja Delporte, Nathalie Feyaerts, Cécile Lambremont, Lut Nieuwborg, Nathalie Peeters, Ann Raman, Pim Sansen, Hilde Stevens, Marianne Schulte, Els Van Ransbeeck, Christel Van de Brande, Greet Van den Eynde, Marleen Vanderkerken, Inge Van Dijck, Audrey Wagener, Monika Waskiewicz und Bernard Wenderickx für die logistische Unterstützung. Wir danken auch der Mucovereniging/Association Muco, insbesondere Stefan Joris und Dr. Jan Vanleeuwe, für ihre Unterstützung und Finanzierung. Wir danken allen Mitarbeitern des Belgian Organoid Project: Hedwige Boboli (CHR Citadelle, Lüttich, Belgien), Linda Boulanger (Universitätskliniken Leuven, Belgien), Georges Casimir (HUDERF, Brüssel, Belgien), Benedicte De Meyere (Universitätsklinikum Gent, Belgien), Elke De Wachter (Universitätsklinikum Brüssel, Belgien), Danny De Looze (Universitätsklinikum Gent, Belgien), Isabelle Etienne (CHU Erasme, Brüssel, Belgien), Laurence Hanssens (HUDERF, Brüssel), Christiane Knoop (CHU Erasme, Brüssel, Belgien), Monique Lequesne (Universitätsklinikum Antwerpen, Belgien), Vicky Nowé (GZA St. Vincentius Hospital Antwerpen), Dirk Staessen (GZA St. Vincentius Hospital Antwerpen), Stephanie Van Biervliet (Universitätsklinikum Gent, Belgien), Eva Van Braeckel (Universitätsklinikum Gent, Belgien), Kim Van Hoorenbeeck (Universitätsklinikum Antwerpen, Belgien), Eef Vanderhelst (Universitätsklinikum Brüssel, Belgien), Stijn Verhulst (Universitätsklinikum Antwerpen, Belgien), Stefanie Vincken (Universitätsklinikum Brüssel, Belgien).
Materialien
| Name | Company | Catalog Number | Comments |
| 1.5 mL microcentrifuge tubes | Sorenson | 17040 | |
| 15 mL conical tubes | VWR | 525-0605 | |
| 24 well plates | Corning | 3526 | |
| 96 well plates | Greiner | 655101 | |
| Brightfield microscope | Zeiss | Axiovert 40C | |
| Centrifuge | Eppendorf | 5702 | |
| CO2 incubator | Binder | CB160 | |
| Computer | Hewlett-Packard | Z240 | |
| Confocal microscope | Zeiss | LSM 800 | |
| Laminar flow hood | Thermo Fisher | 51025413 | |
| Material for organoid culture as detailed in previous protocol10 | |||
| Micropipettes (20, 200, and 1000 µL) | Eppendorf | 3123000039, 3123000055, 3123000063 | |
| Microsoft Excel | Microsoft | Microsoft Excel 2019 MSO 64-bit | Spreadsheet software |
| NIS-Elements Advanced Research Analysis Imaging Software | Nikon | v.5.02.00 | Imaging software |
| Pipette tips (20, 200, and 1000 µL) | Greiner | 774288, 775353, 750288 | |
| Zeiss Zen Blue software | Zeiss | v2.6 | Imaging software |
Referenzen
- Riordan, J. R., et al. Identification of the cystic fibrosis gene: Cloning and characterization of complementary DNA. Science. 245 (4922), 1066-1073 (1989).
- Castellani, C., et al. ECFS best practice guidelines: the 2018 revision. Journal of Cystic Fibrosis. 17 (2), 153-178 (2018).
- . CFTR2 Available from: https://www.cftr2.org/ (2022)
- Farrell, P. M., et al. Diagnosis of cystic fibrosis: Consensus guidelines from the cystic fibrosis foundation. The Journal of Pediatrics. 181, 4-15 (2017).
- Vermeulen, F., Lebecque, P., De Boeck, K., Leal, T. Biological variability of the sweat chloride in diagnostic sweat tests: A retrospective analysis. Journal of Cystic Fibrosis: Official Journal of the European Cystic Fibrosis Society. 16 (1), 30-35 (2017).
- Sato, T., et al. Long-term expansion of epithelial organoids from human colon, adenoma, adenocarcinoma, and Barrett's epithelium. Gastroenterology. 141 (5), 1762-1772 (2011).
- Boj, S. F., et al. Forskolin-induced swelling in intestinal organoids: An in vitro assay for assessing drug response in cystic fibrosis patients. Journal of Visualized Experiments: JoVE. (120), e55159 (2017).
- Friedmacher, F., Puri, P. Rectal suction biopsy for the diagnosis of Hirschsprung's disease: a systematic review of diagnostic accuracy and complications. Pediatric Surgery International. 31 (9), 821-830 (2015).
- Dekkers, J. F., et al. A functional CFTR assay using primary cystic fibrosis intestinal organoids. Nature Medicine. 19 (7), 939-945 (2013).
- Dekkers, J. F., et al. Characterizing responses to CFTR-modulating drugs using rectal organoids derived from subjects with cystic fibrosis. Science Translational Medicine. 8 (344), (2016).
- Vonk, A. M., et al. Protocol for application, standardization and validation of the forskolin-induced swelling assay in cystic fibrosis human colon organoids. STAR Protocols. 1 (1), 100019 (2020).
- Cuyx, S., et al. Rectal organoid morphology analysis (ROMA) as a promising diagnostic tool in cystic fibrosis. Thorax. 76 (11), 1146-1149 (2021).
- Wilschanski, M., et al. Mutations in the cystic fibrosis transmembrane regulator gene and in vivo transepithelial potentials. American Journal of Respiratory and Critical Care Medicine. 174 (7), 787-794 (2006).
- Derichs, N., et al. Intestinal current measurement for diagnostic classification of patients with questionable cystic fibrosis: validation and reference data. Thorax. 65 (7), 594-599 (2010).
- Ramalho, A. S., et al. Correction of CFTR function in intestinal organoids to guide treatment of Cystic Fibrosis. European Respiratory Journal. 57, 1902426 (2020).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten