Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Analyse de la morphologie organoïde rectale (ROMA): un test diagnostique dans la fibrose kystique
Dans cet article
Résumé
Ce protocole décrit l’analyse de la morphologie organoïde rectale (ROMA), un nouveau test diagnostique de la fibrose kystique (FK). Les caractéristiques morphologiques, à savoir la rondeur (indice de circularité, IC) et la présence d’une lumière (rapport d’intensité, IR), sont une mesure de la fonction CFTR. L’analyse de 189 sujets a montré une discrimination parfaite entre les FK et les non-FC.
Résumé
Le diagnostic de la fibrose kystique (FK) n’est pas toujours simple, en particulier lorsque la concentration de chlorure dans la sueur est intermédiaire et/ou que moins de deux mutations du CFTR causant la maladie peuvent être identifiées. Des tests physiologiques CFTR (différence de potentiel nasal, mesure du courant intestinal) ont été inclus dans l’algorithme diagnostique, mais ne sont pas toujours facilement disponibles ou réalisables (p. ex. chez les nourrissons). Les organoïdes rectaux sont des structures 3D qui se développent à partir de cellules souches isolées des cryptes d’une biopsie rectale lorsqu’elles sont cultivées dans des conditions spécifiques. Les organoïdes de sujets non atteints de fibrose kystique ont une forme ronde et une lumière remplie de liquide, car le transport du chlorure médié par CFTR conduit l’eau dans la lumière. Les organoïdes dont la fonction CFTR est défectueuse ne gonflent pas, conservant une forme irrégulière et n’ayant pas de lumière visible. Les différences de morphologie entre les organoïdes FK et non FK sont quantifiées dans l’analyse de la morphologie des organoïdes rectaux (ROMA) en tant que nouveau test physiologique CFTR. Pour le test ROMA, les organoïdes sont plaqués dans des plaques à 96 puits, colorés avec de la calcéine et imagés au microscope confocal. Les différences morphologiques sont quantifiées à l’aide de deux indices : l’indice de circularité (IC) quantifie la rondeur des organoïdes, et le rapport d’intensité (IR) est une mesure de la présence d’une lumière centrale. Les organoïdes non-FK ont un IC élevé et un IR faible par rapport aux organoïdes de la FK. Les index ROMA ont parfaitement discriminé 167 sujets atteints de mucoviscidose de 22 sujets sans mucoviscidose, ce qui fait de la ROMA un test physiologique CFTR attrayant pour aider au diagnostic de la mucoviscidose. Les biopsies rectales peuvent être effectuées systématiquement à tous les âges dans la plupart des hôpitaux et les tissus peuvent être envoyés à un laboratoire central pour la culture organoïde et la ROMA. À l’avenir, la ROMA pourrait également être appliquée pour tester l’efficacité des modulateurs CFTR in vitro. L’objectif du présent rapport est d’expliquer en détail les méthodes utilisées pour ROMA, afin de permettre la reproduction dans d’autres laboratoires.
Introduction
La fibrose kystique (FK) est une maladie autosomique récessive causée par des mutations du gène régulateur de la conductance transmembranaire de la mucoviscidose (CFTR). La protéine CFTR est un canal chlorure et bicarbonate, assurant l’hydratation de plusieurs épithéliums1. La FK est une maladie multisystémique à charge de morbidité élevée, qui raccourcit la vie, qui se manifeste principalement par une maladie respiratoire, mais qui affecte également le tractus gastro-intestinal, le pancréas, le foie et l’appareil reproducteur2.
Les mutations du CFTR causant la maladie entraînent une diminution de la quantité ou de la fonction de CFTR , provoquant à son tour une déshydratation du mucus. Plus de 2 000 variantes du gène CFTR ont été décrites3, dont seulement 466 ont été caractérisées de manière approfondie4.
Un diagnostic de FK peut être posé lorsque la concentration de chlorure dans la sueur (CSC) est supérieure au seuil de 60 mmol/L ou lorsque deux mutations du CFTR causant la maladie (selon la base de données CFTR2) sont identifiées 4,5. Chez les sujets présentant seulement un CSC moyennement élevé (30-60 mmol / L), qui se produit dans environ 4% à 5% des tests de sueur6, et des mutations CFTR de conséquence clinique variable ou inconnue, le diagnostic ne peut être confirmé ni exclu, même s’ils présentent des symptômes compatibles avec la FK ou un test de dépistage néonatal positif. Pour ces cas, des tests physiologiques CFTR de deuxième intention (différence de potentiel nasal (NPD) et mesures de courant intestinal (ICM)) ont été inclus dans l’algorithme de diagnostic. Ces tests ne sont pas facilement disponibles dans la plupart des centres ni réalisables à tous les âges, en particulier chez les nourrissons5.
Les organoïdes rectaux sont des structures 3D cultivées à partir de cellules souches intestinales adultes Lgr5(+) provenant de cryptes intestinales obtenues par biopsie rectale7. Les organoïdes sont de plus en plus utilisés dans la recherche biomédicale, comme l’essai d’un traitement modulateur dans la FC8. Une biopsie viable peut être obtenue par aspiration ou par forcepsie, une procédure qui ne provoque qu’un inconfort minimal et qui est sûre même chez les nourrissons, avec de faibles taux de complications9. Les cryptes isolées des biopsies rectales sont enrichies en cellules souches et, dans des conditions de culture spécifiques, celles-ci s’auto-organisent en organoïdes rectaux. La morphologie de ces organoïdes est déterminée par l’expression et la fonction du CFTR, situé au niveau de la membrane apicale des cellules épithéliales. Le CFTR fonctionnel permet au chlorure et à l’eau de pénétrer dans la lumière organoïde, induisant ainsi un gonflement des organoïdes non CF. Les organoïdes de la mucoviscidose ne gonflent pas et n’ont pas de lumière visible10,11.
L’analyse de la morphologie organoïde rectale (ROMA) permet la discrimination entre les organoïdes FK et non FK en fonction de ces différences de morphologie organoïde. Les organoïdes non-FK sont plus ronds et ont une lumière visible, alors que l’inverse est vrai pour les organoïdes FK. Pour ce test, les organoïdes spécifiques au patient sont plaqués dans 32 puits d’une plaque de 96 puits. Après 1 jour de croissance, les organoïdes sont colorés au vert de calcéine et imagés au microscope confocal. Les organoïdes non-CF montrent une forme plus circulaire et une partie centrale moins fluorescente, car la lumière contient du liquide et la calcéine ne colore que les cellules. Ces différences de morphologie sont quantifiées à l’aide de deux indices ROMA : l’indice de circularité (IC) quantifie la rondeur des organoïdes, tandis que le rapport d’intensité (IR) est une mesure de la présence ou de l’absence d’une lumière centrale. Dans ce rapport, nous décrivons en détail le protocole pour obtenir ces indices discriminatifs, afin de permettre la réplication de la technique.
Protocole
Pour toutes les procédures impliquant des tissus humains, l’approbation du comité d’éthique de la recherche UZ/KU Leuven (recherche CE) a été obtenue. Toutes les recherches ont été effectuées avec le consentement éclairé et/ou l’assentiment des parents, des représentants et/ou des patients.
REMARQUE : Toutes les procédures impliquant des biopsies rectales et des organoïdes doivent être effectuées dans un flux laminaire afin de protéger le chercheur contre tout danger biologique et de minimiser le risque de contamination des cultures. Comme pour toute procédure de laboratoire, les chercheurs devraient en tout temps porter des blouses de laboratoire, des gants et des lunettes de sécurité pour manipuler les échantillons.
1. Biopsie rectale, isolement de cellules souches adultes dans des cryptes et culture organoïde
- Pour cette première partie du protocole, y compris la production et l’utilisation des milieux, la culture organoïde, la division, l’expansion et la congélation pour les biobanques, suivez les deux protocoles 8,12 publiés précédemment.
- En bref, les mesures suivantes doivent être prises:
- Prélever trois à quatre biopsies rectales à l’aide d’une pince ou d’un dispositif d’aspiration et recueillir dans un contenant stérile (p. ex. tube microcentrifuge de 1,5 mL) avec le milieu Ad-DF+++ tel que décrit dans le protocole mentionné ci-dessus.
- Transporter les biopsies vers un laboratoire central sur glace ou à 4 °C. Le transport vers d’autres centres est possible et la qualité n’est pas affectée de manière significative, même lorsque le transport prend jusqu’à 48 h. Si le transport doit durer plus de 6 heures, utiliser un tube conique de 15 mL avec 6 mL de milieu Ad-DF+++ au lieu d’un tube microcentrifuge de 1,5 mL.
- Lavez les biopsies dans du PBS froid jusqu’à ce que le surnageant soit clair pour enlever les débris et les tissus non épithéliaux tels que le tissu adipeux.
- Incuber les biopsies avec de l’EDTA (concentration finale 10 mM) pour détacher les cryptes. Plaquer les cryptes dans une matrice membranaire basale. Ajouter des antibiotiques à large spectre (gentamicine 50 μg/mL et vancomycine 50 μg/mL) au milieu au cours de la première semaine de culture pour prévenir la contamination bactérienne.
- Lorsque les cryptes ont bourgeonné et sont fermées et proliférées, effectuez un fractionnement mécanique, généralement après 7 jours.
- Diviser les organoïdes résultants environ tous les 7 jours. De cette façon, développez la culture, congelez des échantillons de sauvegarde dans une biobanque ou utilisez les organoïdes pour les tests.
2. Placage organoïde pour ROMA (jour 1)
- Diviser mécaniquement les organoïdes pour le placage
- Prélever les organoïdes de trois puits bien développés dans une plaque de 24 puits en les lavant deux fois avec un milieu Ad-DF+++ froid, les recueillir dans un tube à microcentrifuger de 1,5 ml et les évaluer au microscope12.
- S’assurer que les organoïdes sont viables et de haute qualité, ce qui peut généralement être obtenu après 5 à 7 jours de croissance après avoir été fendus précédemment (Figure 1).
- Diviser mécaniquement les organoïdes selonle protocole 8,12 précité. Répéter jusqu’à ce que la majorité des organoïdes, tels qu’évalués à l’aide du microscope à fond clair, soient suffisamment petits par rapport à l’observation initiale.
- Recueillir les organoïdes plus petits, correspondant généralement à la collecte d’environ les 4/5 supérieurs de la solution organoïde-milieu dans un nouveau tube microcentrifugé, car les gros organoïdes coulent au fond du tube en raison de la gravité et les petits organoïdes restent suspendus dans la partie supérieure de la colonne moyenne.
- Centrifuger l’échantillon d’organoïdes plus petits à 0,3 x 1000 g pendant 2 min et éliminer le milieu.
- Diluer la pastille d’organoïdes plus petits dans 130 μL de matrice membranaire basale à 40%-50% (diluée avec un milieu Ad-DF+++12).
- Bien remettre en suspension à l’aide d’une micropipette de 200 μL.
- Plaquer les organoïdes dans une plaque de 96 puits
- À l’aide d’une pipette de 20 μL, les organoïdes des plaques dans 32 puits d’une plaque préchauffée de 96 puits. Assurez-vous que chaque puits contient une goutte de 4 μL de la solution à matrice organoïde produite à l’étape précédente.
- Les organoïdes dans la solution ont tendance à former une pastille au fond du tube en raison du déplacement vers le bas dépendant de la gravité. Pour éviter cela et assurer un placage uniforme, remettre régulièrement en suspension la solution à matrice organoïde à l’aide d’une micropipette de 200 μL (par exemple, chaque fois après le placage de quatre à huit puits).
- Plaquez chaque goutte au centre du puits pour empêcher la goutte de couler vers les bords du puits, ce qui réduirait la qualité de l’image plus tard.
- Visez environ 30 organoïdes par puits (minimum 15, maximum 90) sans chevauchement des organoïdes.
- Gardez à l’esprit que ces organoïdes devront être incubés pendant la nuit et se développeront légèrement pendant cette période, et pourraient commencer à se chevaucher si la densité de placage est trop élevée.
- Vérifiez la densité de placage à l’aide d’un microscope à fond clair avec un objectif de grossissement 5x après avoir plaqué le premier ou les deux premiers puits.
- Si la densité de placage est trop élevée, diluer progressivement la solution organoïde-matrice en ajoutant la matrice membranaire basale jusqu’à ce que la densité de placage souhaitée soit atteinte (figure 2).
- Après le placage, tapotez doucement la plaque sur une surface plane pour vous assurer que la majorité des organoïdes sont dans le même plan focal.
- Incuber l’assiette
- Incuber la plaque de 96 puits à 37 °C et 5% de CO2 pendant 8-10 min pour permettre la gélification de la matrice membranaire basale.
- Ajouter 50 μL de milieu organoïde du côlon humain +/+12 dans chaque puits et incuber pendant une nuit pendant 16-24 h.
- N’ajoutez pas de forskoline ni de modulateurs CFTR, de sorte que les organoïdes se développent dans des conditions basales.
3. Imagerie organoïde par microscopie confocale (jour 2)
- Colorer les organoïdes avec du vert calcéine
- Préparer une solution mère de 1 mM de vert de calcéine en ajoutant 50 μL de diméthylsulfoxyde (DMSO) dans un flacon contenant 50 μg de vert de calcéine.
- Préparer une solution de travail de vert de calcéine en ajoutant 1,2 μL de la solution mère à 200 μL d’Ad-DF+++ (concentration 6 μM).
- Ajouter 5 μL de ce mélange de calcéine à chaque puits de la plaque de 96 puits dans laquelle les organoïdes ont été plaqués (concentration finale de calcéine 0,6 μM). Ne touchez pas et ne disloquez pas la goutte de matrice à l’intérieur du puits lorsque vous effectuez cette étape.
- Faites pivoter et inclinez légèrement la plaque avec le couvercle plusieurs fois pour assurer une répartition homogène de la calcéine verte dans l’ensemble du puits.
- Incuber à nouveau la plaque pendant 15-30 min à 37 °C et 5% de CO2 pour assurer la coloration de tous les organoïdes dans les puits.
- Transférer la plaque au microscope confocal
- Transférez la plaque au microscope confocal avec une platine automatisée et un incubateur intégré. Assurez-vous que la plaque est bien fixée dans le porte-plaque.
- L’incubation à 37 °C et 5% de CO2 est facultative mais pas nécessaire, étant donné la courte durée (environ 10 min) du processus d’imagerie.
- Focus sur les organoïdes (Figure 3)
- À l’aide du microscope confocal, déterminez manuellement la position x/y optimale et la focalisation (position z) des organoïdes dans chaque puits, puis enregistrez ces positions dans le logiciel d’imagerie.
- La meilleure mise au point implique la délimitation la plus nette possible des organoïdes et la visualisation de la plus grande partie possible de la goutte organoïde. Si la répartition de la tache n’est pas adéquate, répétez les étapes 3.1.4 et 3.1.5 (incuber, par exemple, pendant 5 à 10 minutes supplémentaires).
- Utilisez des paramètres d’imagerie de cellules vivantes avec émission à 488 nm et excitation à 515 nm (spécifiques pour la visualisation de la fluorescence de la calcéine) et un objectif de grossissement DL 5x.
- Acquérir des images organoïdes des 32 puits avec le microscope confocal (Figure 3 et Figure 4).
- Prenez des images de manière unidirectionnelle avec une résolution de 1024 pixels x 1024 pixels (taille des pixels 2,5 μm x 2,5 μm) et une profondeur de 16 bits.
- Choisissez l’intensité laser et le gain principal pour une visualisation optimale des différences morphologiques entre les organoïdes FK et non CF (par exemple, discrimination entre une lumière centrale remplie d’eau non colorée (le cas échéant) et une bordure cellulaire colorée).
NOTE: Les organoïdes ne seront pas délimités correctement lorsque le gain principal et donc le signal de fluorescence sont trop faibles; Régler le gain maître trop haut se traduira par des images avec des organoïdes avec une intensité de signal homogène très élevée, empêchant l’imagerie de différences morphologiques plus subtiles (Figure 2). - Enregistrez une image par puits pour les 32 puits au format microscope et exportez-les sous forme de fichiers TIFF.
4. Analyse d’images (Figure 5)
- Chargez les fichiers TIFF dans le logiciel d’analyse d’image.
- Effectuer le premier contrôle de qualité en fonction des critères d’exclusion déterminés par l’opérateur (figure 2) : beaucoup de structures ou de débris différenciés ou morts, densité de placage inadéquate, trop (p. ex. chevauchement) ou trop peu d’organoïdes, et distribution de fluorescence inadéquate (organoïdes non clairement délimités, signal de fond trop élevé).
REMARQUE: Cette étape peut également être effectuée avant d’exporter des images en tant que fichiers TIFF à l’étape 3.4. - Préparer les images pour l’analyse
- Recalibrez les images de sorte que 1 pixel corresponde à 2,5 μm x 2,5 μm.
- Créez et ouvrez une donnée réseau (. ND) des 32 images pour chaque culture organoïde, permettant l’analyse simultanée des 32 images par sujet.
- Délimiter les organoïdes
- Délimiter les structures à l’aide d’un seuil d’intensité inférieur de 4 500 et d’un seuil supérieur de 65 535 (fonctions lisses et propres désactivées; Fonction de remplissage des trous ; Fonction séparée à x3).
REMARQUE: Cela délimite les structures fluorescentes, avec des trous de remplissage , y compris la lumière dans la structure délimitée si elle est présente.
- Délimiter les structures à l’aide d’un seuil d’intensité inférieur de 4 500 et d’un seuil supérieur de 65 535 (fonctions lisses et propres désactivées; Fonction de remplissage des trous ; Fonction séparée à x3).
- Compter les organoïdes
- Sélectionnez toutes les structures ≥40 μm.
- Cliquez sur le bouton Mettre à jour la mesure ND (chaque fois qu’une mesure est nécessaire) ; Les organoïdes comptés seront numérotés.
NOTE: Cela équivaut au nombre total d’organoïdes dans les 32 puits, tandis que les petits débris, tels que les cellules mortes, sont exclus.
- Mesurer l’intensité et la circularité pour le calcul des indices
- Sélectionnez toutes les structures ≥60 μm et comptez.
REMARQUE : Cela compte les organoïdes suffisamment grands pour montrer une morphologie typique de la mucoviscidose ou non. Les organoïdes >40 μm et <60 μm sont petits et denses, tant dans les non-FC que dans les FK. - Supprimez toutes les structures qui touchent les bordures de l’image. Faites cela pour éliminer les organoïdes qui ne sont pas complètement visibles, car leur morphologie ne peut pas être quantifiée avec précision.
- Éroder 1 pixel (= 2,5 μm) à partir de la bordure de chaque structure de ≥60 μm. Cela supprime le halo de fluorescence diffuse de calcéine entourant les organoïdes.
- Mesurer l’intensité moyenne de chaque structure. De cette façon, la fluorescence moyenne des organoïdes est mesurée.
- Sélectionnez à nouveau toutes les structures ≥60 μm et retirez toutes les structures qui touchent les bordures.
- Éroder 10 pixels (= 25 μm) de la bordure de chaque structure de ≥60 μm.
REMARQUE: Dans les organoïdes non-CF, cela érode la frontière cellulaire et ne laisse que la lumière. Dans les organoïdes CF, cela érode la partie externe de l’organoïde, qui a à peu près la même fluorescence que la partie interne qui reste. - Mesurer l’intensité moyenne de chaque structure érodée. De cette façon, la fluorescence moyenne de la partie centrale des organoïdes est mesurée.
- Mesurer la circularité de chaque structure. Cela correspond à la circularité moyenne des organoïdes.
- Sélectionnez toutes les structures ≥60 μm et comptez.
- Effectuer le deuxième contrôle qualité : critères d’exclusion déterminés par le logiciel. Exclure l’ensemble des images lorsque moins de 50 % des organoïdes (définis comme des structures ≥40 μm) mesurent ≥60 μm, car suffisamment d’organoïdes doivent être suffisamment grands pour montrer une morphologie typique de la mucoviscidose ou non. Exclure également lorsque < 500 ou >3 000 organoïdes (définis comme des structures ≥40 μm) sont présents dans les 32 puits.
5. Mesurer les indices dans le logiciel d’imagerie (Figure 6)
- Mesurer l’indice de circularité (IC).
NOTE: Cela correspond à la circularité moyenne mesurée à l’étape 4.6, qui est la circularité moyenne de tous les organoïdes dans les 32 puits. L’IC quantifie la rondeur des organoïdes, définie par , qui est plus faible dans la mucoviscidose que dans les organoïdes non mucovisques.
, qui est plus faible dans la mucoviscidose que dans les organoïdes non mucovisques. - Mesurer le rapport d’intensité (IR)
- Calculer l’IR en divisant la moyenne de la mesure de l’intensité après érosion de 25 μm de la frontière de chaque structure de ≥60 μm par la moyenne de la mesure de l’intensité après érosion de 2,5 μm à partir de la bordure de chaque structure de ≥60 μm.
REMARQUE: IR mesure la présence ou l’absence d’une lumière centrale. L’IR est égal à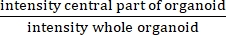 , et est plus élevé dans les organoïdes non CF que dans les organoïdes non-CF.
, et est plus élevé dans les organoïdes non CF que dans les organoïdes non-CF.
- Calculer l’IR en divisant la moyenne de la mesure de l’intensité après érosion de 25 μm de la frontière de chaque structure de ≥60 μm par la moyenne de la mesure de l’intensité après érosion de 2,5 μm à partir de la bordure de chaque structure de ≥60 μm.
- Simplifier le processus d’analyse
- Préparez une feuille de calcul standard à l’aide d’un tableur pour calculer automatiquement ces indices lors de la copie des données (figure 7).
- Pour calculer l’IR :
- Prenez la mesure de l’intensité après érosion de 2,5 μm à partir de la bordure de chaque structure de ≥60 μm. Utilisez la moyenne pour le calcul (dénominateur).
- Prenez la mesure de l’intensité après érosion de 25 μm à partir de la bordure de chaque structure de ≥60 μm. Utilisez la moyenne pour le calcul (numérateur).
- Pour calculer l’IC, prenez la circularité de tous les organoïdes dans tous les puits. La moyenne correspond à l’IC.
REMARQUE: Lorsque l’analyse de grands lots d’images est requise, le processus d’analyse d’image décrit dans la section 4 peut être semi-automatisé, en commençant par les fichiers ND calibrés et en exécutant une macro avec toutes les étapes combinées.
Résultats
Des organoïdes de 212 sujets ont été prélevés lors de visites cliniques de routine. Aucun événement indésirable n’est survenu pendant ou après la biopsie rectale. Les organoïdes ont été imagés par un chercheur aveugle aux caractéristiques du sujet telles que le génotype et les informations cliniques. En raison d’images de faible qualité, 23 sujets ont été exclus. Des exemples de cultures organoïdes réussies et ratées et d’acquisition d’images peuvent être vus à la figur...
Discussion
Nous fournissons un protocole détaillé pour l’analyse de la morphologie organoïde rectale (ROMA). Les deux indices calculés avec ROMA, IR et CI ont distingué les organoïdes des sujets atteints de mucoviscidose de ceux sans mucoviscidose avec une précision parfaite. ROMA pourrait donc fonctionner comme un nouveau test physiologique CFTR complémentaire au CSC et à d’autres tests actuellement disponibles13,14,15.
<...Déclarations de divulgation
Cette étude a été financée par l’association belge des patients atteints de mucoviscidose « Mucovereniging/Association Muco », la bourse de recherche de la Société belge de pédiatrie BVK-SBP 2019 et une subvention du Fonds UZ Leuven pour la recherche biomédicale translationnelle. Les auteurs ne déclarent aucun conflit d’intérêts.
Remerciements
Nous remercions les patients et les parents qui ont participé à cette étude. Nous remercions Abida Bibi pour tout le travail de culture avec les organoïdes. Nous remercions Els Aertgeerts, Karolien Bruneel, Claire Collard, Liliane Collignon, Monique Delfosse, Anja Delporte, Nathalie Feyaerts, Cécile Lambremont, Lut Nieuwborg, Nathalie Peeters, Ann Raman, Pim Sansen, Hilde Stevens, Marianne Schulte, Els Van Ransbeeck, Christel Van de Brande, Greet Van den Eynde, Marleen Vanderkerken, Inge Van Dijck, Audrey Wagener, Monika Waskiewicz et Bernard Wenderickx pour leur soutien logistique. Nous remercions également le Mucovereniging/Association Muco, et en particulier Stefan Joris et Dr. Jan Vanleeuwe, pour leur soutien et leur financement. Nous remercions tous les collaborateurs du Belgian Organoid Project: Hedwige Boboli (CHR Citadelle, Liège, Belgique), Linda Boulanger (Hôpitaux Universitaires de Louvain, Belgique), Georges Casimir (HUDERF, Bruxelles, Belgique), Benedicte De Meyere (Hôpital Universitaire de Gand, Belgique), Elke De Wachter (Hôpital Universitaire de Bruxelles, Belgique), Danny De Looze (Hôpital Universitaire de Gand, Belgique), Isabelle Etienne (CHU Erasme, Bruxelles, Belgique), Laurence Hanssens (HUDERF, Bruxelles), Christiane Knoop (CHU Erasme, Bruxelles, Belgique), Monique Lequesne (Hôpital universitaire d’Anvers, Belgique), Vicky Nowé (GZA Hôpital Saint-Vincentius Anvers), Dirk Staessen (GZA Hôpital Saint-Vincentius Anvers), Stephanie Van Biervliet (Hôpital universitaire de Gand, Belgique), Eva Van Braeckel (Hôpital universitaire de Gand, Belgique), Kim Van Hoorenbeeck (Hôpital universitaire d’Anvers, Belgique), Eef Vanderhelst (Hôpital universitaire de Bruxelles, Belgique), Stijn Verhulst (Hôpital universitaire d’Anvers, Belgique), Stefanie Vincken (Hôpital Universitaire de Bruxelles, Belgique).
matériels
| Name | Company | Catalog Number | Comments |
| 1.5 mL microcentrifuge tubes | Sorenson | 17040 | |
| 15 mL conical tubes | VWR | 525-0605 | |
| 24 well plates | Corning | 3526 | |
| 96 well plates | Greiner | 655101 | |
| Brightfield microscope | Zeiss | Axiovert 40C | |
| Centrifuge | Eppendorf | 5702 | |
| CO2 incubator | Binder | CB160 | |
| Computer | Hewlett-Packard | Z240 | |
| Confocal microscope | Zeiss | LSM 800 | |
| Laminar flow hood | Thermo Fisher | 51025413 | |
| Material for organoid culture as detailed in previous protocol10 | |||
| Micropipettes (20, 200, and 1000 µL) | Eppendorf | 3123000039, 3123000055, 3123000063 | |
| Microsoft Excel | Microsoft | Microsoft Excel 2019 MSO 64-bit | Spreadsheet software |
| NIS-Elements Advanced Research Analysis Imaging Software | Nikon | v.5.02.00 | Imaging software |
| Pipette tips (20, 200, and 1000 µL) | Greiner | 774288, 775353, 750288 | |
| Zeiss Zen Blue software | Zeiss | v2.6 | Imaging software |
Références
- Riordan, J. R., et al. Identification of the cystic fibrosis gene: Cloning and characterization of complementary DNA. Science. 245 (4922), 1066-1073 (1989).
- Castellani, C., et al. ECFS best practice guidelines: the 2018 revision. Journal of Cystic Fibrosis. 17 (2), 153-178 (2018).
- . CFTR2 Available from: https://www.cftr2.org/ (2022)
- Farrell, P. M., et al. Diagnosis of cystic fibrosis: Consensus guidelines from the cystic fibrosis foundation. The Journal of Pediatrics. 181, 4-15 (2017).
- Vermeulen, F., Lebecque, P., De Boeck, K., Leal, T. Biological variability of the sweat chloride in diagnostic sweat tests: A retrospective analysis. Journal of Cystic Fibrosis: Official Journal of the European Cystic Fibrosis Society. 16 (1), 30-35 (2017).
- Sato, T., et al. Long-term expansion of epithelial organoids from human colon, adenoma, adenocarcinoma, and Barrett's epithelium. Gastroenterology. 141 (5), 1762-1772 (2011).
- Boj, S. F., et al. Forskolin-induced swelling in intestinal organoids: An in vitro assay for assessing drug response in cystic fibrosis patients. Journal of Visualized Experiments: JoVE. (120), e55159 (2017).
- Friedmacher, F., Puri, P. Rectal suction biopsy for the diagnosis of Hirschsprung's disease: a systematic review of diagnostic accuracy and complications. Pediatric Surgery International. 31 (9), 821-830 (2015).
- Dekkers, J. F., et al. A functional CFTR assay using primary cystic fibrosis intestinal organoids. Nature Medicine. 19 (7), 939-945 (2013).
- Dekkers, J. F., et al. Characterizing responses to CFTR-modulating drugs using rectal organoids derived from subjects with cystic fibrosis. Science Translational Medicine. 8 (344), (2016).
- Vonk, A. M., et al. Protocol for application, standardization and validation of the forskolin-induced swelling assay in cystic fibrosis human colon organoids. STAR Protocols. 1 (1), 100019 (2020).
- Cuyx, S., et al. Rectal organoid morphology analysis (ROMA) as a promising diagnostic tool in cystic fibrosis. Thorax. 76 (11), 1146-1149 (2021).
- Wilschanski, M., et al. Mutations in the cystic fibrosis transmembrane regulator gene and in vivo transepithelial potentials. American Journal of Respiratory and Critical Care Medicine. 174 (7), 787-794 (2006).
- Derichs, N., et al. Intestinal current measurement for diagnostic classification of patients with questionable cystic fibrosis: validation and reference data. Thorax. 65 (7), 594-599 (2010).
- Ramalho, A. S., et al. Correction of CFTR function in intestinal organoids to guide treatment of Cystic Fibrosis. European Respiratory Journal. 57, 1902426 (2020).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon