È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Analisi della morfologia organoide rettale (ROMA): un saggio diagnostico nella fibrosi cistica
In questo articolo
Riepilogo
Questo protocollo descrive l'analisi morfologica degli organoidi rettali (ROMA), un nuovo test diagnostico per la fibrosi cistica (FC). Le caratteristiche morfologiche, vale a dire la rotondità (indice di circolarità, CI) e la presenza di un lume (rapporto di intensità, IR), sono una misura della funzione CFTR. L'analisi di 189 soggetti ha mostrato una perfetta discriminazione tra FC e non CF.
Abstract
La diagnosi di fibrosi cistica (FC) non è sempre semplice, specialmente quando la concentrazione di cloruro nel sudore è intermedia e/o possono essere identificate meno di due mutazioni CFTR che causano la malattia. I saggi fisiologici CFTR (differenza di potenziale nasale, misurazione della corrente intestinale) sono stati inclusi nell'algoritmo diagnostico, ma non sono sempre prontamente disponibili o fattibili (ad esempio, nei neonati). Gli organoidi rettali sono strutture 3D che crescono da cellule staminali isolate da cripte di una biopsia rettale quando coltivate in condizioni specifiche. Gli organoidi di soggetti non CF hanno una forma rotonda e un lume pieno di liquido, poiché il trasporto di cloruro mediato da CFTR guida l'acqua nel lume. Gli organoidi con funzione CFTR difettosa non si gonfiano, mantenendo una forma irregolare e non avendo lume visibile. Le differenze nella morfologia tra organoidi CF e non CF sono quantificate nell'"Analisi della morfologia degli organoidi rettali" (ROMA) come nuovo test fisiologico CFTR. Per il test ROMA, gli organoidi sono placcati in lastre a 96 pozzetti, colorati con calceina e ripresi in un microscopio confocale. Le differenze morfologiche sono quantificate utilizzando due indici: l'indice di circolarità (CI) quantifica la rotondità degli organoidi e il rapporto di intensità (IR) è una misura della presenza di un lume centrale. Gli organoidi non CF hanno un alto CI e un basso IR rispetto agli organoidi CF. Gli indici ROMA hanno perfettamente discriminato 167 soggetti con FC da 22 soggetti senza FC, rendendo ROMA un interessante test fisiologico CFTR per aiutare nella diagnosi di FC. Le biopsie rettali possono essere eseguite di routine a tutte le età nella maggior parte degli ospedali e il tessuto può essere inviato a un laboratorio centrale per la coltura di organoidi e ROMA. In futuro, ROMA potrebbe anche essere applicato per testare l'efficacia dei modulatori CFTR in vitro. Lo scopo del presente rapporto è quello di spiegare pienamente i metodi utilizzati per ROMA, per consentire la replica in altri laboratori.
Introduzione
La fibrosi cistica (FC) è una malattia autosomica recessiva causata da mutazioni nel gene CFTR (Transmembrane Conductance Regulator (CFTR). La proteina CFTR è un canale del cloruro e del bicarbonato, garantendo l'idratazione di diversi epiteli1. La FC è una malattia multisistemica ad alto carico, che accorcia la vita, che si manifesta principalmente come una malattia respiratoria, ma colpisce anche il tratto gastrointestinale, il pancreas, il fegato e il tratto riproduttivo2.
Le mutazioni di CFTR che causano malattie portano a una diminuzione della quantità o della funzione di CFTR, causando a loro volta disidratazione del muco. Sono state descritte più di 2.000 varianti del gene CFTR 3, di cui solo 466 sono state accuratamente caratterizzate4.
Una diagnosi di FC può essere fatta quando la concentrazione di cloruro di sudore (SCC) è superiore alla soglia di 60 mmol / L o quando vengono identificate due mutazioni CFTR che causano la malattia (secondo il database CFTR2) 4,5. Nei soggetti con SCC solo intermediamente elevata (30-60 mmol/L), che si verifica in circa il 4%-5% dei test del sudore6, e mutazioni CFTR di conseguenze cliniche variabili o sconosciute, la diagnosi non può essere confermata né esclusa, anche quando hanno sintomi compatibili con FC o un test di screening neonatale positivo. Per questi casi, nell'algoritmo diagnostico sono stati inclusi saggi fisiologici CFTR di seconda linea (differenza di potenziale nasale (NPD) e misurazioni della corrente intestinale (ICM). Questi test non sono prontamente disponibili nella maggior parte dei centri né fattibili a tutte le età, specialmente nei neonati5.
Gli organoidi rettali sono strutture 3D cresciute da cellule staminali intestinali adulte Lgr5(+) provenienti da cripte intestinali ottenute attraverso la biopsia rettale7. Gli organoidi vengono sempre più utilizzati nella ricerca biomedica, come il trattamento con modulatore di test in CF8. Una biopsia praticabile può essere ottenuta mediante aspirazione o pinza, una procedura che causa solo un disagio minimo ed è sicura anche nei neonati, con bassi tassi di complicanze9. Le cripte isolate dalle biopsie rettali sono arricchite in cellule staminali e, in specifiche condizioni di coltura, queste si auto-organizzano in organoidi rettali. La morfologia di questi organoidi è determinata dall'espressione e dalla funzione del CFTR, situato sulla membrana apicale delle cellule epiteliali. Il CFTR funzionale consente al cloruro e all'acqua di entrare nel lume degli organoidi, inducendo così il gonfiore degli organoidi non CF. Gli organoidi CF non si gonfiano e non hanno lume visibile10,11.
L'analisi della morfologia degli organoidi rettali (ROMA) consente la discriminazione tra organoidi CF e non CF sulla base di queste differenze nella morfologia degli organoidi. Gli organoidi non CF sono più rotondi e hanno un lume visibile, mentre il contrario è vero per gli organoidi CF. Per questo test, gli organoidi specifici del paziente sono placcati in 32 pozzetti di una piastra da 96 pozzetti. Dopo 1 giorno di crescita, gli organoidi sono colorati con verde calceina e ripresi in un microscopio confocale. Gli organoidi non-CF mostrano una forma più circolare e una parte centrale meno fluorescente, poiché il lume contiene fluido e le macchie di calceina solo le cellule. Queste differenze morfologiche sono quantificate utilizzando due indici ROMA: l'indice di circolarità (CI) quantifica la rotondità degli organoidi, mentre il rapporto di intensità (IR) è una misura della presenza o assenza di un lume centrale. In questo report, descriviamo in dettaglio il protocollo per ottenere questi indici discriminanti, per consentire la replica della tecnica.
Protocollo
Per tutte le procedure che coinvolgono tessuti umani, è stata ottenuta l'approvazione da parte del Comitato etico di ricerca UZ/KU Leuven (ricerca CE). Tutte le ricerche sono state eseguite con il consenso informato e / o l'assenso di genitori, rappresentanti e / o pazienti.
NOTA: Tutte le procedure che coinvolgono biopsie rettali e organoidi devono essere eseguite in un flusso laminare per proteggere il ricercatore da qualsiasi pericolo biologico e per ridurre al minimo il rischio di contaminazione delle colture. Come per qualsiasi procedura di laboratorio, i ricercatori dovrebbero sempre indossare camici da laboratorio, guanti e occhiali di sicurezza per manipolare i campioni.
1. Biopsia rettale, isolamento di cellule staminali adulte da cripte e coltura organoide
- Per questa parte iniziale del protocollo, compresa la produzione e l'utilizzo dei media, la coltura di organoidi, la scissione, l'espansione e il congelamento per il biobanking, seguire idue protocolli 8,12 precedentemente pubblicati.
- In breve, devono essere prese le seguenti misure:
- Effettuare da tre a quattro biopsie rettali con una pinza o un dispositivo di aspirazione e raccogliere in un contenitore sterile (ad esempio, una provetta da microcentrifuga da 1,5 ml) con il mezzo Ad-DF+++ come descritto nel protocollo sopra menzionato.
- Trasporto di biopsie in un laboratorio centrale su ghiaccio o a 4 °C. Il trasporto verso altri centri è possibile e la qualità non è influenzata in modo significativo anche quando il trasporto richiede fino a 48 ore. Se si prevede che il trasporto duri più di 6 ore, utilizzare un tubo conico da 15 mL con 6 mL di mezzo Ad-DF+++ invece di un tubo di microcentrifuga da 1,5 ml.
- Lavare le biopsie in PBS freddo fino a quando il surnatante è chiaro per rimuovere i detriti e i tessuti non epiteliali come il tessuto adiposo.
- Incubare le biopsie con EDTA (concentrazione finale 10 mM) per staccare le cripte. Placcare le cripte in una matrice di membrana basacolare. Aggiungere antibiotici ad ampio spettro (gentamicina 50 μg/mL e vancomicina 50 μg/mL) al terreno nella prima settimana di coltura per prevenire la contaminazione batterica.
- Quando le cripte sono germogliate e sono chiuse e proliferate, eseguire la scissione meccanica, di solito dopo 7 giorni.
- Dividere gli organoidi risultanti circa ogni 7 giorni. In questo modo, espandere la coltura, congelare campioni di backup in una biobanca o utilizzare gli organoidi per i test.
2. Placcatura organoide per ROMA (giorno 1)
- Dividere meccanicamente gli organoidi per la placcatura
- Raccogliere organoidi da tre pozzetti ben cresciuti da una piastra a 24 pozzetti lavandoli due volte con mezzo freddo Ad-DF+++, raccoglierli in una provetta da microcentrifuga da 1,5 ml e valutarli al microscopio12.
- Assicurarsi che gli organoidi siano vitali e di alta qualità, che di solito possono essere raggiunti dopo 5-7 giorni di crescita dopo essere stati precedentemente divisi (Figura 1).
- Dividere meccanicamente gli organoidi secondo il suddetto protocollo 8,12. Ripetere fino a quando la maggior parte degli organoidi, valutati utilizzando il microscopio a campo chiaro, sono abbastanza piccoli rispetto all'osservazione iniziale.
- Raccogliere gli organoidi più piccoli, di solito corrispondenti alla raccolta di circa i 4/5 superiori della soluzione organoide-mezzo in una nuova provetta da microcentrifuga, poiché i grandi organoidi affondano sul fondo del tubo a causa della gravità e i piccoli organoidi rimangono sospesi nella parte superiore della colonna media.
- Centrifugare il campione di organoidi più piccoli a 0,3 x 1000 g per 2 minuti ed eliminare il terreno.
- Diluire il pellet di organoidi più piccoli in 130 μL di matrice di membrana basale 40%-50% (diluita con mezzo Ad-DF+++12).
- Risospendere bene utilizzando una micropipetta da 200 μL.
- Impiattare gli organoidi in un piatto da 96 pozzetti
- Utilizzando una pipetta da 20 μL, gli organoidi della piastra in 32 pozzetti di una piastra preriscaldata a 96 pozzetti. Assicurarsi che ogni pozzetto contenga una goccia da 4 μL della soluzione di matrice organoide prodotta nella fase precedente.
- Gli organoidi nella soluzione tendono a formare un pellet sul fondo del tubo a causa dello spostamento verso il basso dipendente dalla gravità. Per evitare che ciò accada e garantire una placcatura uniforme, risospendere regolarmente la soluzione di matrice organoide utilizzando una micropipetta da 200 μL (ad esempio, ogni volta dopo aver placcato da quattro a otto pozzetti).
- Placca ogni goccia al centro del pozzo per evitare che la goccia scorra verso i bordi del pozzo, il che ridurrebbe la qualità dell'immagine in seguito.
- Puntare a circa 30 organoidi per pozzetto (minimo 15, massimo 90) senza organoidi sovrapposti.
- Tieni presente che questi organoidi dovranno essere incubati durante la notte e cresceranno leggermente durante questo periodo e potrebbero iniziare a sovrapporsi se la densità di placcatura è troppo alta.
- Controllare la densità di placcatura utilizzando un microscopio a campo chiaro con un obiettivo di ingrandimento 5x dopo aver placcato i primi uno o due pozzetti.
- Se la densità di placcatura è troppo elevata, diluire gradualmente la soluzione di matrice organoide con l'aggiunta della matrice della membrana basale fino a raggiungere la densità di placcatura desiderata (Figura 2).
- Dopo la placcatura, picchiettare delicatamente la piastra su una superficie piana per assicurarsi che la maggior parte degli organoidi si trovi sullo stesso piano focale.
- Incubare il piatto
- Incubare la piastra a 96 pozzetti a 37 °C e al 5% di CO2 per 8-10 minuti per consentire la gelificazione della matrice della membrana basale.
- Aggiungere 50 μL di terreno organoide del colon umano +/+12 in ciascun pozzetto e incubare durante la notte per 16-24 ore.
- Non aggiungere modulatori di forskolina né CFTR, quindi gli organoidi crescono in condizioni basali.
3. Imaging organoide mediante microscopia confocale (giorno 2)
- Colorare gli organoidi con calcein verde
- Preparare una soluzione madre da 1 mM di calcein verde aggiungendo 50 μL di dimetilsolfossido (DMSO) ad un flaconcino contenente 50 μg di calcein verde.
- Preparare una soluzione di lavoro di calcein verde aggiungendo 1,2 μL della soluzione madre a 200 μL di Ad-DF+++ (concentrazione 6 μM).
- Aggiungere 5 μL di questa miscela di calceina a ciascun pozzetto della piastra a 96 pozzetti in cui sono stati placcati gli organoidi (concentrazione finale di calceina 0,6 μM). Non toccare e dislocare la goccia della matrice all'interno del pozzetto quando si esegue questo passaggio.
- Ruotare e inclinare leggermente la piastra con il coperchio acceso alcune volte per garantire una distribuzione omogenea del verde calcein in tutto il pozzetto.
- Incubare nuovamente la piastra per 15-30 minuti a 37 °C e al 5% di CO2 per garantire la colorazione di tutti gli organoidi nei pozzetti.
- Trasferire la piastra al microscopio confocale
- Trasferire la piastra al microscopio confocale con uno stadio automatizzato e un incubatore integrato. Assicurarsi che la piastra sia ben fissata nel supporto della piastra.
- L'incubazione a 37 °C e al 5% di CO2 è facoltativa ma non necessaria, data la breve durata (circa 10 minuti) del processo di imaging.
- Focus sugli organoidi (Figura 3)
- Utilizzando il microscopio confocale, determinare manualmente la posizione x/y ottimale e la messa a fuoco (posizione z) degli organoidi in ciascun pozzetto e salvare queste posizioni nel software di imaging.
- La migliore messa a fuoco implica la delineazione più nitida possibile degli organoidi e la visualizzazione della maggior parte possibile della goccia organoide. Se la distribuzione della macchia non è adeguata, ripetere i passaggi 3.1.4 e 3.1.5 (incubare, ad esempio, per altri 5-10 minuti).
- Utilizzare impostazioni di imaging di cellule vive con emissione a 488 nm ed eccitazione a 515 nm (specifiche per la visualizzazione della fluorescenza della calceina) e un obiettivo di ingrandimento LD 5x.
- Acquisire immagini organoidi dei 32 pozzetti con il microscopio confocale (Figura 3 e Figura 4).
- Scatta immagini in modo unidirezionale con una risoluzione di 1024 pixel x 1024 pixel (dimensione pixel 2,5 μm x 2,5 μm) e profondità di 16 bit.
- Scegli l'intensità laser e il guadagno master per una visualizzazione ottimale delle differenze morfologiche tra organoidi CF e non CF (ad esempio, discriminazione tra un lume centrale pieno d'acqua non colorato (se presente) e un bordo cellulare colorato).
NOTA: Gli organoidi non saranno delineati correttamente quando il guadagno master e quindi il segnale di fluorescenza sono troppo bassi; impostando il guadagno master troppo alto si otterranno immagini con organoidi con intensità di segnale omogenea molto elevata, impedendo l'imaging di differenze morfologiche più sottili (Figura 2). - Salva un'immagine per pozzetto per tutti i 32 pozzetti nel formato microscopio ed esportali come file TIFF.
4. Analisi delle immagini (Figura 5)
- Caricare i file TIFF nel software di analisi delle immagini.
- Eseguire il primo controllo di qualità in base a criteri di esclusione determinati dall'operatore (Figura 2): molte strutture o detriti differenziati o morti, densità di placcatura inadeguata, troppi (ad esempio, sovrapposti) o troppo pochi organoidi e distribuzione della fluorescenza inadeguata (organoidi non chiaramente delineati, segnale di fondo troppo alto).
NOTA: questo passaggio può essere eseguito anche prima di esportare le immagini come file TIFF nel passaggio 3.4. - Preparare le immagini per l'analisi
- Ricalibrare le immagini, in modo che 1 pixel corrisponda a 2,5 μm x 2,5 μm.
- Creare e aprire un Dati di rete (. ND) di tutte le 32 immagini per ogni coltura organoide, consentendo l'analisi simultanea di tutte le 32 immagini per soggetto.
- Delinea gli organoidi
- Delinea le strutture utilizzando una soglia di intensità inferiore di 4.500 e una soglia superiore di 65.535 (funzioni Smooth e Clean disattivate; Funzione Fill Holes attivata; Funzione separata in x3).
NOTA: Questo delinea le strutture fluorescenti, con fori di riempimento che includono il lume nella struttura delineata, se presente.
- Delinea le strutture utilizzando una soglia di intensità inferiore di 4.500 e una soglia superiore di 65.535 (funzioni Smooth e Clean disattivate; Funzione Fill Holes attivata; Funzione separata in x3).
- Contare gli organoidi
- Selezionare tutte le strutture ≥40 μm.
- Fare clic sul pulsante Aggiorna misurazione ND (ogni volta che è necessaria una misurazione); Gli organoidi contati saranno numerati.
NOTA: Questo equivale al numero totale di organoidi nei 32 pozzi, mentre i piccoli detriti, come le cellule morte, sono esclusi.
- Misurare l'intensità e la circolarità per il calcolo degli indici
- Selezionare tutte le strutture ≥60 μm e contare.
NOTA: Questo conta gli organoidi abbastanza grandi da mostrare la morfologia tipica di CF o non CF. Gli organoidi >40 μm e <60 μm sono piccoli e densi, sia in non-CF che in CF. - Rimuovere tutte le strutture che toccano i bordi dell'immagine. Fai questo per rimuovere gli organoidi che non sono completamente visibili, poiché la loro morfologia non può essere quantificata con precisione.
- Erodere 1 pixel (= 2,5 μm) dal bordo di ciascuna struttura di ≥60 μm. Questo rimuove l'alone di fluorescenza di calceina diffusa che circonda gli organoidi.
- Misurare l'intensità media di ogni struttura. In questo modo, viene misurata la fluorescenza media degli organoidi.
- Selezionare nuovamente tutte le strutture ≥60 μm e rimuovere tutte le strutture che toccano i bordi.
- Erodere 10 pixel (= 25 μm) dal bordo di ogni struttura di ≥60 μm.
NOTA: Negli organoidi non-CF, questo erode il bordo cellulare e lascia solo il lume. Negli organoidi CF, questo erode la parte esterna dell'organoide, che ha all'incirca la stessa fluorescenza della parte interna che rimane. - Misurare l'intensità media di ogni struttura erosa. In questo modo, viene misurata la fluorescenza media della parte centrale degli organoidi.
- Misurare la circolarità di ogni struttura. Ciò corrisponde alla circolarità media degli organoidi.
- Selezionare tutte le strutture ≥60 μm e contare.
- Eseguire il secondo controllo di qualità: criteri di esclusione determinati dal software. Escludere l'insieme delle immagini quando meno del 50% degli organoidi (definiti come strutture ≥40 μm) sono ≥60 μm, poiché un numero sufficiente di organoidi dovrebbe essere abbastanza grande da mostrare la morfologia tipica della CF o della non CF. Escludere anche quando <500 o >3.000 organoidi (definiti come strutture ≥40 μm) sono presenti nei 32 pozzi.
5. Misurare gli indici nel software di imaging (Figura 6)
- Misurare l'indice di circolarità (CI).
NOTA: Corrisponde alla circolarità media misurata al punto 4.6, che è la circolarità media di tutti gli organoidi in tutti i 32 pozzetti. L'IC quantifica la rotondità degli organoidi, definita come , che è inferiore nella FC rispetto agli organoidi non CF
, che è inferiore nella FC rispetto agli organoidi non CF - Misurare il rapporto di intensità (IR)
- Calcolare l'IR dividendo la media della misurazione dell'intensità dopo aver eroso 25 μm dal bordo di ciascuna struttura di ≥60 μm per la media della misurazione dell'intensità dopo aver eroso 2,5 μm dal bordo di ciascuna struttura di ≥60 μm.
NOTA: IR misura la presenza o l'assenza di un lume centrale. IR è uguale a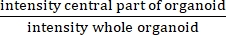 , ed è più alto in CF che in organoidi non CF.
, ed è più alto in CF che in organoidi non CF.
- Calcolare l'IR dividendo la media della misurazione dell'intensità dopo aver eroso 25 μm dal bordo di ciascuna struttura di ≥60 μm per la media della misurazione dell'intensità dopo aver eroso 2,5 μm dal bordo di ciascuna struttura di ≥60 μm.
- Semplifica il processo di analisi
- Preparare un foglio di lavoro standard utilizzando un software per fogli di calcolo per calcolare automaticamente questi indici dopo aver copiato i dati (Figura 7).
- Per calcolare l'IR:
- Prendi la misura dell'intensità dopo aver eroso 2,5 μm dal bordo di ciascuna struttura di ≥60 μm. Utilizzare la media per il calcolo (denominatore).
- Prendi la misura dell'intensità dopo aver eroso 25 μm dal bordo di ciascuna struttura di ≥60 μm. Utilizzare la media per il calcolo (numeratore).
- Per calcolare l'IC, prendi la circolarità di tutti gli organoidi in tutti i pozzi. La media corrisponde all'IC.
NOTA: quando è richiesta l'analisi di grandi lotti di immagini, il processo di analisi delle immagini descritto nella sezione 4 può essere semi-automatico, partendo dai file ND calibrati ed eseguendo una macro con tutti i passaggi combinati.
Risultati
Gli organoidi di 212 soggetti sono stati raccolti durante le visite cliniche di routine. Non si sono verificati eventi avversi durante o dopo la procedura di biopsia rettale. Gli organoidi sono stati fotografati da un ricercatore in cieco alle caratteristiche del soggetto come il genotipo e le informazioni cliniche. A causa delle immagini di bassa qualità, sono stati esclusi 23 soggetti. Esempi di colture organoidi riuscite e fallite e acquisizione di immagini possono essere visti nella Figura 2
Discussione
Forniamo un protocollo dettagliato per l'analisi della morfologia organoide rettale (ROMA). I due indici calcolati con ROMA, IR e CI, distinguevano gli organoidi dai soggetti con FC da quelli senza FC con perfetta precisione. ROMA potrebbe quindi funzionare come un nuovo test fisiologico CFTR complementare a SCC e ad altri test attualmente disponibili13,14,15.
Il protocollo dipende dall'uso di organoi...
Divulgazioni
Questo studio è stato finanziato dall'associazione belga di pazienti FC "Mucovereniging/Association Muco", dalla borsa di ricerca della Società belga di pediatria BVK-SBP 2019 e da una sovvenzione del Fondo UZ Leuven per la ricerca biomedica traslazionale. Gli autori non dichiarano alcun conflitto di interessi.
Riconoscimenti
Ringraziamo i pazienti e i genitori che hanno partecipato a questo studio. Ringraziamo Abida Bibi per tutto il lavoro di coltura con gli organoidi. Ringraziamo Els Aertgeerts, Karolien Bruneel, Claire Collard, Liliane Collignon, Monique Delfosse, Anja Delporte, Nathalie Feyaerts, Cécile Lambremont, Lut Nieuwborg, Nathalie Peeters, Ann Raman, Pim Sansen, Hilde Stevens, Marianne Schulte, Els Van Ransbeeck, Christel Van de Brande, Greet Van den Eynde, Marleen Vanderkerken, Inge Van Dijck, Audrey Wagener, Monika Waskiewicz e Bernard Wenderickx per il supporto logistico. Ringraziamo anche la Mucovereniging/Association Muco, e in particolare Stefan Joris e Dr. Jan Vanleeuwe, per il loro sostegno e finanziamento. Ringraziamo tutti i collaboratori del Belgian Organoid Project: Hedwige Boboli (CHR Citadelle, Liegi, Belgio), Linda Boulanger (University Hospitals Leuven, Belgio), Georges Casimir (HUDERF, Bruxelles, Belgio), Benedicte De Meyere (University Hospital Ghent, Belgio), Elke De Wachter (University Hospital Brussels, Belgio), Danny De Looze (University Hospital Ghent, Belgio), Isabelle Etienne (CHU Erasme, Bruxelles, Belgio), Laurence Hanssens (HUDERF, Bruxelles), Christiane Knoop (CHU Erasme, Bruxelles, Belgio), Monique Lequesne (Ospedale universitario di Anversa, Belgio), Vicky Nowé (GZA St. Vincentius Hospital Anversa), Dirk Staessen (GZA St. Vincentius Hospital Anversa), Stephanie Van Biervliet (University Hospital Ghent, Belgio), Eva Van Braeckel (University Hospital Ghent, Belgio), Kim Van Hoorenbeeck (University Hospital Antwerp, Belgio), Eef Vanderhelst (University Hospital Brussels, Belgio), Stijn Verhulst (University Hospital Antwerp, Belgio), Stefanie Vincken (Ospedale universitario di Bruxelles, Belgio).
Materiali
| Name | Company | Catalog Number | Comments |
| 1.5 mL microcentrifuge tubes | Sorenson | 17040 | |
| 15 mL conical tubes | VWR | 525-0605 | |
| 24 well plates | Corning | 3526 | |
| 96 well plates | Greiner | 655101 | |
| Brightfield microscope | Zeiss | Axiovert 40C | |
| Centrifuge | Eppendorf | 5702 | |
| CO2 incubator | Binder | CB160 | |
| Computer | Hewlett-Packard | Z240 | |
| Confocal microscope | Zeiss | LSM 800 | |
| Laminar flow hood | Thermo Fisher | 51025413 | |
| Material for organoid culture as detailed in previous protocol10 | |||
| Micropipettes (20, 200, and 1000 µL) | Eppendorf | 3123000039, 3123000055, 3123000063 | |
| Microsoft Excel | Microsoft | Microsoft Excel 2019 MSO 64-bit | Spreadsheet software |
| NIS-Elements Advanced Research Analysis Imaging Software | Nikon | v.5.02.00 | Imaging software |
| Pipette tips (20, 200, and 1000 µL) | Greiner | 774288, 775353, 750288 | |
| Zeiss Zen Blue software | Zeiss | v2.6 | Imaging software |
Riferimenti
- Riordan, J. R., et al. Identification of the cystic fibrosis gene: Cloning and characterization of complementary DNA. Science. 245 (4922), 1066-1073 (1989).
- Castellani, C., et al. ECFS best practice guidelines: the 2018 revision. Journal of Cystic Fibrosis. 17 (2), 153-178 (2018).
- . CFTR2 Available from: https://www.cftr2.org/ (2022)
- Farrell, P. M., et al. Diagnosis of cystic fibrosis: Consensus guidelines from the cystic fibrosis foundation. The Journal of Pediatrics. 181, 4-15 (2017).
- Vermeulen, F., Lebecque, P., De Boeck, K., Leal, T. Biological variability of the sweat chloride in diagnostic sweat tests: A retrospective analysis. Journal of Cystic Fibrosis: Official Journal of the European Cystic Fibrosis Society. 16 (1), 30-35 (2017).
- Sato, T., et al. Long-term expansion of epithelial organoids from human colon, adenoma, adenocarcinoma, and Barrett's epithelium. Gastroenterology. 141 (5), 1762-1772 (2011).
- Boj, S. F., et al. Forskolin-induced swelling in intestinal organoids: An in vitro assay for assessing drug response in cystic fibrosis patients. Journal of Visualized Experiments: JoVE. (120), e55159 (2017).
- Friedmacher, F., Puri, P. Rectal suction biopsy for the diagnosis of Hirschsprung's disease: a systematic review of diagnostic accuracy and complications. Pediatric Surgery International. 31 (9), 821-830 (2015).
- Dekkers, J. F., et al. A functional CFTR assay using primary cystic fibrosis intestinal organoids. Nature Medicine. 19 (7), 939-945 (2013).
- Dekkers, J. F., et al. Characterizing responses to CFTR-modulating drugs using rectal organoids derived from subjects with cystic fibrosis. Science Translational Medicine. 8 (344), (2016).
- Vonk, A. M., et al. Protocol for application, standardization and validation of the forskolin-induced swelling assay in cystic fibrosis human colon organoids. STAR Protocols. 1 (1), 100019 (2020).
- Cuyx, S., et al. Rectal organoid morphology analysis (ROMA) as a promising diagnostic tool in cystic fibrosis. Thorax. 76 (11), 1146-1149 (2021).
- Wilschanski, M., et al. Mutations in the cystic fibrosis transmembrane regulator gene and in vivo transepithelial potentials. American Journal of Respiratory and Critical Care Medicine. 174 (7), 787-794 (2006).
- Derichs, N., et al. Intestinal current measurement for diagnostic classification of patients with questionable cystic fibrosis: validation and reference data. Thorax. 65 (7), 594-599 (2010).
- Ramalho, A. S., et al. Correction of CFTR function in intestinal organoids to guide treatment of Cystic Fibrosis. European Respiratory Journal. 57, 1902426 (2020).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon