Method Article
Kombination von Clearing- und Fluoreszenzmikroskopie zur Visualisierung von Veränderungen der Genexpression und physiologischen Reaktionen auf Plasmodiophora brassicae
In diesem Artikel
Zusammenfassung
Das vorliegende Protokoll beschreibt eine optimierte Methode zur histologischen Beobachtung von Gallen, die durch Plasmodiophora brassicae induziert werden. Vibratomschnitte von Hypokotylen werden vor der Fluoreszenzbildgebung entfernt, um die Beteiligung von Transkriptionsfaktoren und Phytohormonen während des Krankheitsverlaufs zu untersuchen. Dieses Protokoll überwindet Einschränkungen bei der Einbettung von Harz und ermöglicht die Visualisierung fluoreszierender Proteine in planta .
Zusammenfassung
Die Infektion von Brassica-Pflanzen durch den bodenbürtigen Protisten Plasmodiophora brassicae führt zur Gallenbildung an den unterirdischen Organen. Die Bildung von Gallen erfordert eine zelluläre Reprogrammierung und Veränderungen im Stoffwechsel der infizierten Pflanze. Dies ist notwendig, um eine pathogenorientierte physiologische Senke zu etablieren, zu der die Wirtsnährstoffe umgeleitet werden. Für ein vollständiges Verständnis dieser speziellen Pflanzen-Pathogen-Interaktion und der Mechanismen, durch die Wirtswachstum und -entwicklung untergraben und umstrukturiert werden, ist es wichtig, die internen Veränderungen, die die Gallenbildung begleiten, mit zellulärer Auflösung zu verfolgen und zu beobachten. Methoden, die fluoreszierende Färbungen und fluoreszierende Proteine kombinieren, werden häufig eingesetzt, um anatomische und physiologische Reaktionen in Pflanzen zu untersuchen. Leider stellen die Größe der Gallen und ihre geringe Transparenz große Hürden bei der Durchführung von Beobachtungen unter dem Mikroskop dar. Darüber hinaus begrenzt die geringe Transparenz den Einsatz der Fluoreszenzmikroskopie zur Untersuchung des Fortschreitens und der Gallenbildung von Klumpwurzelerkrankungen. Dieser Artikel stellt eine optimierte Methode zur Fixierung und Reinigung von Gallen vor, um Epifluoreszenz und konfokale Mikroskopie zur Inspektion von P. brassicae-infizierten Gallen zu erleichtern. Ein Gewebereinigungsprotokoll für eine schnelle optische Reinigung wurde verwendet, gefolgt von Vibratomschnitten, um anatomische Veränderungen zu erkennen und die Genexpression mit Promotorfusionen und Reporterlinien zu lokalisieren, die mit fluoreszierenden Proteinen markiert waren. Diese Methode wird sich als nützlich erweisen, um zelluläre und physiologische Reaktionen in anderen durch Krankheitserreger ausgelösten Strukturen in Pflanzen zu untersuchen, wie Nematoden-induzierte Synzytien und Wurzelknoten sowie Blattgallen und Deformationen, die durch Insekten verursacht werden.
Einleitung
Pflanzen, die von Krankheitserregern oder Insekten befallen sind, können abnormale Strukturen (Organdeformationen oder Gallen) entwickeln, die es dem Eindringling ermöglichen, Nährstoffe aufzunehmen und sich zu vermehren1. Hier wurde ein effizienter histopathologischer Ansatz verfolgt, um die Veränderungen in Gallen zu untersuchen, die sich an den unterirdischen Teilen von Pflanzen entwickeln, die mit dem Protisten Plasmodiophora brassicae infiziert sind (Abbildung 1). Der kleine Faden, der mit diesem Erreger assoziiert ist, ergibt sich aus der Tatsache, dass P. brassicae-Sporen ihre Fähigkeit, in Pflanzen einzudringen, für viele Jahre behalten können. Beim großflächigen Anbau von Raps (Brassica napus) ist dies ein ernstes Problem, da wirtschaftliche Faktoren die Fruchtfolge einschränken und zu einer Ansammlung von Restsporen im Boden führen2. Die Resistenz von Raps gegen die durch P. brassicae verursachte Clubroot-Krankheit ist genetisch bedingt. Leider überlistet der Erreger Resistenzen oft aufgrund seiner Biologie und des engen genetischen Pools, aus dem Raps stammt. Daher ist es relevant geworden, die Reaktionen nach der Infektion in Wirtspflanzen und ihre Fähigkeit, das Fortschreiten der Krankheit zu verlangsamen oder die Entwicklung bestimmter Symptome zu verhindern, zu untersuchen.
Bei Clubroot-Erkrankungen wird der Schweregrad im Allgemeinen basierend auf der Entwicklung von Gallen und dem Grad der Schädigung des Wurzelsystems bewertet. Dies wird als Krankheitsindex-DI bezeichnet3. Es erfasst jedoch nicht vollständig die wahre Einschätzung dieser Pflanzen-Pathogen-Interaktion. Insbesondere geht es nicht darum, wie sich der Erreger in den Wurzeln verteilt und ob die Pflanze zurückhalten kann. P. brassicae Bewegung in seinen Geweben. Darüber hinaus ist es nicht leicht vorherzusagen, in welchem Ausmaß P. brassicae programmiert die unterirdische Organanatomie neu. Untersuchungen an der Modellanlage Arabidopsis thaliana haben gezeigt, dass P. brassicae Infektion führt zur Hemmung der Xylogenese (sowohl der Initiierungs- als auch der Reifungsschritt) und zur Verbesserung der Phloemdifferenzierung innerhalb der Gallen4,5. Darüber hinaus verlässt die Nachkommenschaft der kambialen Zellen in Wurzeln und Hypokotylen infizierter Pflanzen den mitotischen Zustand nicht und vermehrt sich länger als bei gesunden Pflanzen.6. Dieser Prozess bestimmt die endgültige Größe der Galle und bestimmt die Anzahl der pathogenen Sporen, die in der infizierten Pflanze produziert werden. P. brassicae-getriebene Entwicklungs-, metabolische und physiologische Reprogrammierung im Wirt ist sehr komplex7; Daher ist die Anwendung von Werkzeugen, die die Inspektion interner Veränderungen innerhalb von Gallen ermöglichen, entscheidend für die richtige Beurteilung dieser Wechselwirkung. Der Lebenszyklusverlauf von P. brassicae geht einher mit einer Reprogrammierung des Wirtszellstoffwechsels, die als Stärke- oder Lipidablagerung beobachtet werden kann7,8. Das Haupthindernis für die erfolgreiche Mikroskopie von Gallen liegt in ihrer geringen Transparenz. Aus diesem Grund stammt die Mehrheit der histologischen Proben, die keulenwurzelbedingte Veränderungen innerhalb der Gallen aufweisen, aus Fixierungstechniken (Wachs oder Harz), gefolgt von Mikrotomschnitten. Solche Ansätze wurden erfolgreich eingesetzt, um die Promotoraktivität für zahlreiche Gene zu lokalisieren, die in Klumpwurzelgallen aktiv sind.4,5 oder verschiedene Färbetechniken, die die Beobachtung von P. brassicae Lebenszyklusverlauf9. Es muss jedoch beachtet werden, dass die Fixierungs- und Einbettungsphasen zeitaufwendig sind und zu einem teilweisen oder vollständigen Auswaschen wichtiger Biomoleküle (z. B. Lipide) führen, was bestimmte Beobachtungen erheblich behindert. Vor kurzem P. brassicae Der Lebenszyklusverlauf im Wirt wurde mit Hilfe der Fluorescent In Situ Hybridization (FISH) visualisiert, bei der eine SABATH-artige Methyltransferase (PbBSMT) Genspezifische Sonde wurde verwendet, um ruhende Sporenbildung zu markieren10. Eine gute Alternative ist die Verwendung anderer fluoreszenzbasierter Methoden, bei denen die Autofluoreszenz einiger zellulärer Komponenten, die Aktivität von 5'- vorgeschalteten regulatorischen Regionen von Genen, die mit fluoreszierenden Proteinmarkern fusioniert sind, und die Akkumulation bestimmter fluoreszenzmarkierter Proteine beobachtet werden können. Neben der geringen Transparenz der Proben ist jedoch ein großer Nachteil solcher Objekte die Arbeit mit unfixierten Proben, was die Zeit, in der qualitativ hochwertige Bilder dokumentiert werden können, erheblich verkürzt. Im Jahr 2015 haben Kurihara et al.11 entwickelte ein Clearing-Reagenz, das die Konservierung fluoreszierender Proteine ermöglicht und die Transparenz von Pflanzengewebeproben erhöht. Darüber hinaus ist es mit zahlreichen histologischen Färbungen kompatibel. Vor kurzem wurde die gleiche Technik erfolgreich angewendet, um verschiedene Zellwandkomponenten in Pflanzengeweben sichtbar zu machen.12,13. Hier wurde dieses Protokoll verwendet, um verschiedene Aspekte der Klumpwurzelgallentwicklung zu analysieren. Der Workflow beginnt mit der Fixierung von Gallen, Vibratomschnitten, Gewebereinigung, Färbung und Fluoreszenzbildgebung. Je nach Bedarf, direkt oder nach einer bestimmten Färbung, können die resultierenden Schnitte einer Inspektion unter einem Epifluoreszenz- oder Konfokalmikroskop unterzogen werden. Diese Methode bietet eine effektive Lösung, um lokale Veränderungen in der Genexpression und physiologischen Reaktionen zu untersuchen, einschließlich des Phytohormonhaushalts und der Signalübertragung. Das Fortschreiten der Krankheit kann verfolgt werden, indem das Verteilungsmuster der ruhenden Sporen und die Reifungsdynamik betrachtet werden. Darüber hinaus kann das Protokoll leicht für die Abbildung von charakteristischen Veränderungen innerhalb P. brassicae infizierte Pflanzen, einschließlich der Hemmung der Xylogenese oder der Abwehrreaktionen der Wirtspflanze, die als lokale Verholzung in resistenten Genotypen sichtbar sind. Beispiele in diesem Protokoll stammen aus der Bildgebung, die auf der Arabidopsis thaliana Modell; Das Protokoll kann jedoch auch auf andere Kulturarten angewandt werden, die zur Brassicaceae Familie. Die unten beschriebene Methode wird zukünftige detaillierte Untersuchungen von Zellstrukturen und molekularen Veränderungen ermöglichen, die die Gallenbildung in P. brassicae-infizierte Pflanzen.
Der allgemeine Arbeitsablauf des Protokolls ist recht einfach, und alle Phasen der Gallenentwicklung können leicht abgebildet und charakterisiert werden (Abbildung 2). Da P. brassicae ein bodenbürtiger Krankheitserreger ist, müssen alle Experimente in bodenbasierten Systemen durchgeführt werden. Der Erreger bevorzugt saure Bedingungen; Daher müssen nicht kalkbehandelte Bodensubstrate verwendet werden. Obwohl P. brassicae keine Gefahr für den Menschen darstellt, handelt es sich ausschließlich um einen Pflanzenpathogen, der sich über Boden und Wasser ausbreiten kann. Daher müssen alle Teile der infizierten Pflanze sowie der Boden nach dem Experiment durch Autoklavieren oder durch Behandlung mit Bleichmittel zerstört werden.
Protokoll
1. Bedingungen für das Pflanzenwachstum
- Züchten Sie Arabidopsis thaliana Pflanzen in einem Kurztageslichtregime mit 9 h Licht bei 22 °C und 15 h Dunkelheit bei 20 °C und einer Bestrahlungsstärke von 120 μmol·m−2s−1 (gemessen als photosynthetisch aktive Strahlung, PAR auf Baldachinebene, siehe Materialtabelle).
2. Herstellung von Sporeninokulum
ANMERKUNG: Für Details siehe Fuchs et al.14.
- Zwei bis drei gefrorene Gallen aus Chinakohlpflanzen (Brassica rapa var. pekinensis Sorte "Granaat") in einem Mixer mit 300 mL autoklaviertem destilliertem Wasser homogenisieren und durch vier Schichten sterile Gaze filtern.
- Zentrifugieren Sie das Filtrat (bei 6.000 x g, für 5 min und bei 4 °C) und entfernen Sie die Stärkeschicht mechanisch mit einem Spatel vom Sporenpellet. Wiederholen Sie diesen Vorgang, bis der größte Teil der Stärke entfernt ist.
- Bestimmen Sie die Sporenkonzentration mit einem Hämozytometer13 , indem Sie die Anzahl der Sporen in 20 verschiedenen Feldbereichen zählen.
- Stellen Sie sicher, dass die Endkonzentration der zur Beimpfung verwendeten Sporensuspension 1 x 106 Sporen·ml−1 für Arabidopsis thaliana beträgt. Beimpfen Sie jede Pflanze 20 Tage nach der Keimung mit 2 ml kalibrierter Sporensuspension.
3. Gewebevorbereitung und Fixierung
- Bereiten Sie das Pflanzengewebe vor.
- Entfernen Sie vorsichtig Erde aus den Wurzelsystemen von Clubroot-infizierten und nicht infizierten Pflanzen. Gründlich mit Wasser reinigen und Hypokotylen und Gallen (Länge zwischen 0,5-2 cm) in Mikrozentrifugenröhrchen sammeln.
- Führen Sie eine PFA-Fixierung (4% Paraformaldehyd, PFA in 1x PBS + 0,01% Triton X-100) durch, indem Sie die folgenden Schritte ausführen.
- 4 g PFA-Pulver (siehe Materialtabelle) zu 100 ml PBS (phosphatgepufferte Kochsalzlösung, pH 7,4, Tabelle 1) durch Rühren bei 65 °C (nicht kochen) geben. Verwenden Sie KOH (10 M und 1 M) tropfenweise, um eine klare Lösung mit einem pH-Wert von etwa 11 zu erhalten.
- Stellen Sie den pH-Wert mit H2SO4 ein, um ihn auf 6,9 zu senken, und fügen Sie 0,01% (v/v) Triton X-100 hinzu, um die Fixierung zu verbessern. Aliquot die Lösung zur Lagerung bei −20 °C (nicht wieder einfrieren) oder bei 4 °C lagern.
- Führen Sie eine Gewebefixierung durch.
- Fixieren Sie die Proben in 200-500 μL PFA-Fixiermittel für 1 h bei Raumtemperatur durch Anlegen eines konstanten Vakuums (700 mbar) mit einer Vakuumpumpe. Stellen Sie sicher, dass das Objekt vollständig in die PFA-Lösung eingetaucht ist.
HINWEIS: Die Proben können einige Wochen bei 4 °C (Kühlschrank) gelagert und später zum Schneiden verwendet werden.
- Fixieren Sie die Proben in 200-500 μL PFA-Fixiermittel für 1 h bei Raumtemperatur durch Anlegen eines konstanten Vakuums (700 mbar) mit einer Vakuumpumpe. Stellen Sie sicher, dass das Objekt vollständig in die PFA-Lösung eingetaucht ist.
4. Einbettung und Schneiden von Gewebe
- Verwenden Sie 4% w/v Agarose für die Einbettung von Agarose von nicht infizierten Hypokotylen und infizierten Gallen. Kochen Sie die Lösung, um Agarose aufzulösen, und gießen Sie sie, wenn sie abkühlt, während sie noch viskos ist.
- Trocknen Sie das Objekt einige Sekunden lang leicht auf einem Gewebe, um überschüssiges PFA zu entfernen.
- Verwenden Sie Zahnstocher / Pinzetten, um das Pflanzenobjekt vorsichtig in Agarose einzubetten und zu orientieren. Um schräge Schnitte zu vermeiden, stellen Sie sicher, dass das Objekt richtig ausgerichtet und geformt ist, so dass seine Achse senkrecht zur Ebene der Vibratomklinge steht (für radiale Abschnitte).
- Um die Erstarrung der Agarose zu beschleunigen, stellen Sie die Petri- oder Multikulturplatte für 10 min auf 4 °C.
HINWEIS: Wenn das Objekt zu groß ist (wie im Fall größerer Gallen von Arabidopsis 21 Tage nach der Impfung [DPI]), kann das Objekt auch ohne Einbettung in Agarose geschnitten werden.
5. Vibratom-Schnitte
HINWEIS: Das Vibratom ist mit einer vibrierenden Rasierklinge ausgestattet, um Pflanzenorgane / Gewebe zu durchschneiden. Die Vibrationsgeschwindigkeit, die Amplitude, der Winkel der Klinge und die Profildicke sind Parameter, die eingestellt werden können (siehe Materialtabelle).
- Schneiden und formen Sie das Objekt vorsichtig mit einer Klinge innerhalb eines Agaroseblocks und notieren Sie die bevorzugte Ausrichtung für das Schneiden.
- Kleben Sie den Agaroseblock/die große Galle, um ihn mit Cyanacrylat/Sofortkleber und Abdeckband richtig auf dem Probenhalter zu montieren (siehe Materialtabelle).
- Halten Sie bei Arabidopsis-Hypokotylen und Gallen (frühe Stadien ) die Dicke der Abschnitte zwischen 30-40 μm, um Bilder in guter Qualität zu erhalten.
HINWEIS: Für die Analyse der späteren Stadien der Gallenprogression kann die Dicke der Abschnitte zwischen 50-80 μm betragen. - Verzichten Sie darauf, dünnere Abschnitte zu schneiden, da dies die Gallenprobe aufgrund der Vibrationen der beweglichen Klinge zerstören kann.
- Passen Sie die Dicke für Abschnitte, Geschwindigkeit und Vibrationsamplitude entsprechend der Dicke und Größe der Galle (40 μm oder 60 μm) an.
- Fügen Sie destilliertes Wasser in das Wasserbad und beginnen Sie mit dem Schneiden.
- Sammeln Sie Abschnitte vorsichtig mit einer Pinzette oder einem Pinsel und geben Sie sie in ein Mikrozentrifugenröhrchen, das 1 ml 1x PBS-Puffer (pH 7,4) enthält.
HINWEIS: Je höher die Schwingungsamplitude, desto besser die Schnittqualität. Bei nicht infizierten Hypokotylen oder Gallen, die keine reifen Ruhesporen enthalten, kann die Vibrationsamplitude bei 1,2 mm mit einer Geschwindigkeit von 0,60 mm·s−1 gehalten werden. Bei großen Gallen mit teilweisem Verlust der zellulären Integrität und sichtbaren Anzeichen einer Sporenreifung sollte die Vibrationsamplitude mit einer Geschwindigkeit von 0,45 mm·s−1 auf 0,55 mm verringert werden.
6. Probenreinigung
- Entfernen Sie 1x PBS aus dem Mikrozentrifugenröhrchen und fügen Sie 200-500 μL Clearinglösung hinzu (Tabelle 1).
- Lagern Sie die Proben von nun an bei Raumtemperatur im Dunkeln. Stellen Sie sicher, dass die Objekte immer in die Clearinglösung eingetaucht sind.
- Ersetzen Sie die Clearing-Lösung durch die neue, wenn sich ihre Farbe nach einiger Zeit während der Probenverarbeitung ändert.
HINWEIS: Die Farbe der Reinigungslösung kann aufgrund der Probenverarbeitung gelblich werden. In einem solchen Fall wird empfohlen, die Lösung zu ersetzen, um die Reinigung von Geweben zu verbessern.
7. Färbeverfahren
- Für die Calcofluor-Weißfärbung der Zellwände (zum Austragen von Pflanzenzellen) bereiten Sie 5% v/v Calcofluor-Weißfärbung (siehe Materialtabelle) in der Reinigungslösung vor. Im Dunkeln mindestens 5 min einfärben.
- Zum Färben von Lipiden mit Nilrot (zum Färben ruhender Sporen und Öltröpfchen in infizierten Zellen) 1 mg/ml der Brühe (siehe Materialtabelle) in Clearinglösung vorbereiten. Verdünnen Sie es weiter, um 1:99 zu erhalten. Im Dunkeln mindestens 10 min einfärben.
- Für die einfache Fuchsin-Ligninfärbung 0,2% w/v der Färbung (siehe Materialtabelle) in Reinigungslösung vorbereiten. Im Dunkeln mindestens 10 min einfärben.
HINWEIS: Während der Doppelfärbung werden die Proben zunächst 10 Minuten lang mit Nilrot gefärbt, bevor Calcofluor White aufgetragen wird. Die überschüssigen Färbelösungen wurden nach jedem Schritt entfernt. Wenn das Objekt überfleckt erscheint, entfernen Sie den überschüssigen Fleck, indem Sie ihn mit der Reinigungslösung waschen. Verdünnen Sie die Flecken bei Bedarf mit der Reinigungslösung. Färben Sie die Probe vor der Calcofluor-Färbung immer mit Nilrot / Basic Fuchsin. Eine erste Färbung mit Calcofluor kann eine korrekte Färbung der Sporen durch Nilrot verhindern.
8. Mikroskopie
- Montieren Sie geräumte Schnitte auf den Mikroskopieobjektträger und beobachten Sie sie unter einem Epifluoreszenz- oder Konfokalmikroskop (siehe Materialtabelle). Verwenden Sie Clearing-Lösung als Montagemedium, um ein Austrocknen der Probe zu verhindern.
- Verwenden Sie mehrere Aufnahmemodi, um mehr als ein Fluoreszenzspektrum gleichzeitig abzubilden.
ANMERKUNG: Die im vorliegenden Protokoll verwendeten Anregungs-/Emissionsspektren lauten wie folgt: für Nilrot 553/636 nm, für Xylemautofluoreszenz 380/475 nm, für Basic Fuchsin 561/650 nm, für Calcofluorweiß 405/475 nm, für erRFP 585/608 nm und für GFP 488/509 nm (Tabelle 2).
Ergebnisse
Mit Nilrot, das Lipide und Suberin färbt, ist es möglich, die pathogenen Sporen zu sehen, die Lipide enthalten (Abbildung 3A, B). Daher können durch doppelte Färbung gestochen scharfe Bilder erhalten werden, um das Muster der Krankheitserregerverteilung innerhalb der Gallen zu betrachten. Die Gegenfärbung mit Calcofluorweiß erzeugt Kontrast und hilft, die Xylementwicklung gleichzeitig mit der P. brassicae-Reifung zu verfolgen (Abbildung 3B).
Die Bildung und Entwicklung von Xylem könnte auch durch Beobachtung der Autofluoreszenz in ungefärbten Proben (Abbildung 4A) oder durch die Verwendung von Färbungen wie Basic Fuchsin überprüft werden, die eine fluoreszenzbasierte Abbildung von Lignin ermöglichen (Abbildung 4B).
Mit dieser Methode könnte man Genexpressionsänderungen oder Reaktionen auf Wachstumsregulatoren verfolgen. Ein perfektes Beispiel ist, wo Arabidopsis-Pflanzen, die das pHCA2:erRFP-Konstrukt beherbergen, verwendet wurden, um die Genexpression von HIGH CAMBIAL ACTIVITY 2 (HCA2) im Phloemgewebe in Clubroot-Gallen zu visualisieren. HCA2-Genaktivität wurde zuvor in meristematisch aktiven Kambium- und Phloemlinienzellen gefunden15. Hier lokalisiert es sich mit dem Phloem in den späten Stadien der P. brassicae-getriebenen Gallentwicklung, und seine Aktivität spiegelt wider, wie P. brassicae die Phloemkomplexität erhöht (Abbildung 5). Das resultierende Bild zeigt die Phloemproliferation im späten Stadium der Gallenentwicklung, wenn das Kambium fragmentiert wird. Abbildung 5A zeigt einen nicht geklärten Handabschnitt der Galle, während Abbildung 5B klarere und lokalisierte fluoreszierende Signale zeigt, die durch Vibratomschnitte gefolgt von Gewebereinigung erhalten werden. Die Objekte wurden mit Calcofluorweiß gegengefärbt. Abbildung 6 vergleicht dieses Bild (Abbildung 6B) mit einer ähnlichen Region, die in harzeingebetteten und mikrotomgeschnittenen Gallen (Abbildung 6A) bei 21 DPI dargestellt ist. Differentielle Cytokininantworten zwischen infizierten und nicht infizierten Pflanzen wurden durch Überprüfung der Expression des TCS:GFP (Two Component Signalling) Markers16 in sich entwickelnden Gallen bewertet (Abbildung 7). Bei der Abbildung schwacher GFP-Signale in Gallen und Geweben mit sekundären Verdickungen ist es wichtig zu beachten, dass während der Bildgebung auch ein zusätzliches Hintergrundsignal aufgrund der Autofluoreszenz reifer Xylemzellen erfasst wird.

Abbildung 1: Symptome der Clubroot-Krankheit bei Raps (B. napus) und Arabidopsis thaliana (Columbia-0) bei 26 DPI mit Plasmodiophora brassicae-Sporen . Im Verlauf der Erkrankung entwickeln sich große Gallen auf dem gesamten Wurzelsystem, wodurch es extrem spröde wird. Es schließt mit der Freisetzung von Sporen in den umgebenden Boden, um zukünftige Infektionen zu fördern. Die oberen Teile des Pflanzenkörpers zeigen auch Anzeichen von schlechtem Wachstum und Entwicklung. Schließlich erliegen die infizierten Pflanzen den verheerenden Auswirkungen auf den Wachstumsstoffwechsel und die Entwicklung, sobald das Wurzelsystem vollständig geschädigt ist und die Pflanze die Krankheit nicht mehr bewältigen kann. Der Maßstabsbalken stellt 1 cm dar. Das -INF steht für Mock-inoculated, während +INF für P. brassicae-inoculated plants steht. Bei dieser Gelegenheit wird vor der Bodenentfernung ein Bild von Rapspflanzen zur Verfügung gestellt, um gesunde Wurzelsysteme zu präsentieren. Nach dem Waschen werden nur das Hypokotyl und der obere Teil der Wurzel gesammelt. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.

Abbildung 2: Der allgemeine Workflow. Gewaschene Arabidopsis-Wurzelsysteme werden (A) seziert, (B) fixiert, (C) in Agarose eingebettet, (D) montiert und (E) auf dem Vibratom geschnitten. Die resultierenden Objekte werden einer Gewebereinigung unterzogen (3 Tage bis mehrere Wochen bei RT im Dunkeln, abhängig von Gewebetyp und Dicke). (F) Entfernte Objekte können dann gefärbt und unter dem Mikroskop inspiziert werden. (G) Zusammenfassung des Workflows. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
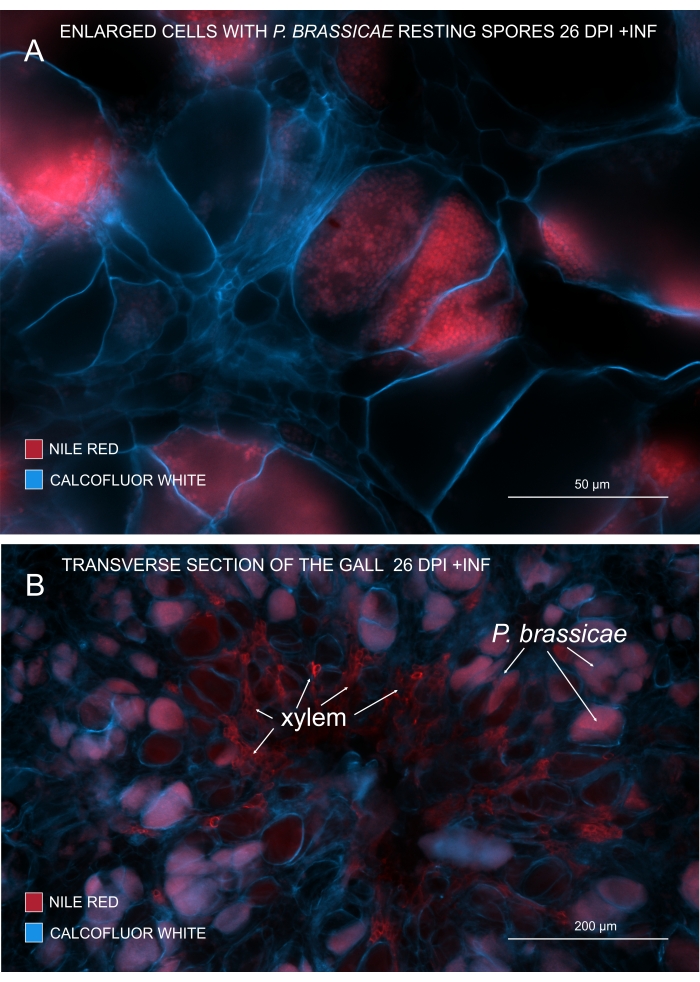
Abbildung 3: Markierung von Erregersporen mit Nilrot-Färbung. (A,B) Nilrot färbt Lipide in ruhenden Sporen, was perfekt funktioniert, um die Reifung von P. brassicae zu verfolgen. (A) Vergrößerte Zellen, die von P. brassicae besiedelt und mit pathogenen Sporen gefüllt sind. (B) Reife Xylemzellen werden auch durch Nilrot gefärbt. Der Schnitt wurde mit Calcofluorweiß gegengefärbt, um den Aufwand der Wirtszellen zu sehen (A: Objektivlinse = 20x und Schnittdicke = 60 μm; B: Objektiv = 5x und Querschnittsdicke = 60 μm). Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
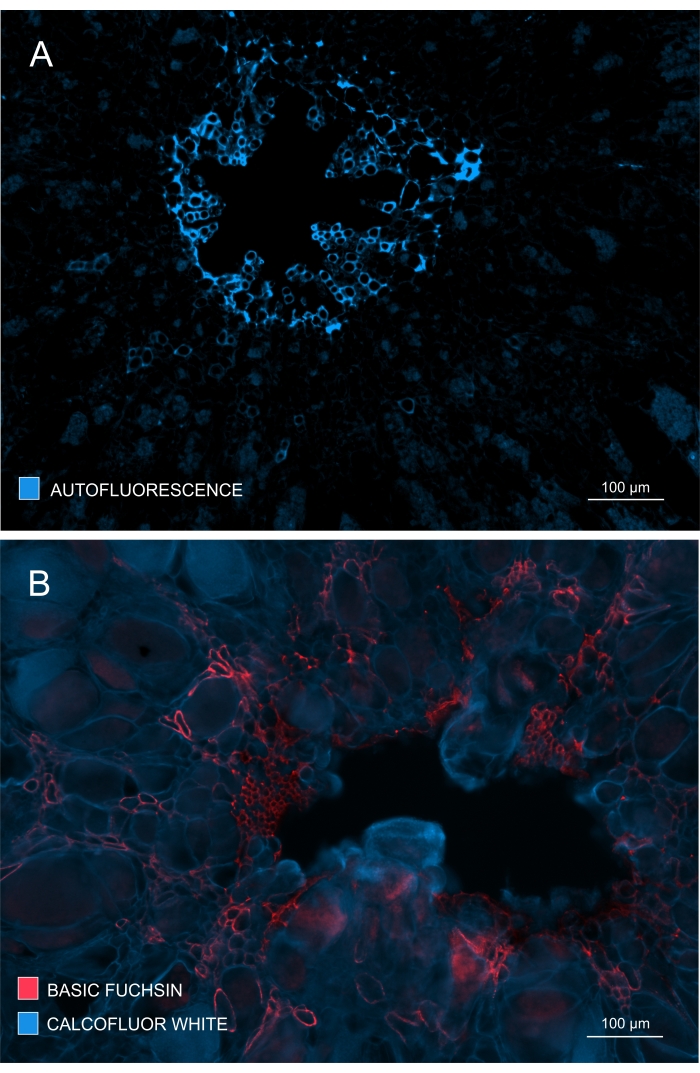
Abbildung 4: Verfolgung des Ausmaßes der Xylementwicklung und -reifung. (A) Lignin gibt starke Autofluoreszenz bei Anregung mit UV; Daher kann reifes Xylem relativ leicht diskriminiert werden. (B) Die doppelte Färbung mit Basic Fuchsin und Calcofluor führt zu besseren Ergebnissen, da alle Zellen mit dem letzteren Farbstoff gefärbt werden, während reifes Xylem deutlich mit Basic Fuchsin gefärbt wird. Auf diese Weise liefert die Doppelfärbung Bilder mit verbessertem Kontrast, die eine spürbare Hemmung der Xylogenese darstellen (A: Objektivlinse = 10x und Schnittdicke = 60 μm; B: Objektiv = 10x und Querschnittsdicke = 60 μm). Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
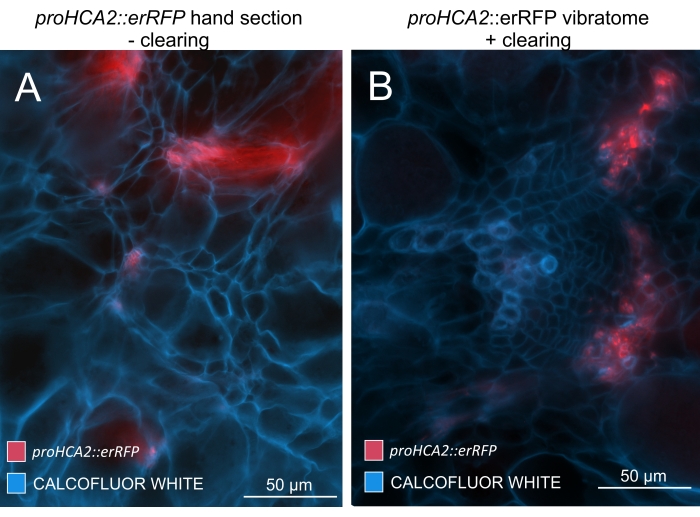
Abbildung 5: Phloem-spezifisches Signal für das HCA2-Gen in Hypokotylen von P. brassicae-infizierten Pflanzen bei 21 DPI. Die Promotoraktivität für proHCA2::erRFP mit transgener Arabidopsis thaliana ist in (A) und (B) zu sehen. Unterschiede können zwischen einem nicht geklärten Handabschnitt in Panel (A) beobachtet werden, in dem das erRFP-Signal aufgrund von Überlagerung und überlappenden Zellschichten diffus erscheint, insbesondere in unebenen Handabschnitten. Auf der anderen Seite zeigt (B) einen Vibratomschnitt nach der Gewebereinigung, bei dem das erRFP-Signal die Phloemzellen in einem reifen Gefäßbündel präzise markiert (Objektivlinse = 20x und Schnittdicke = 60 μm). Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
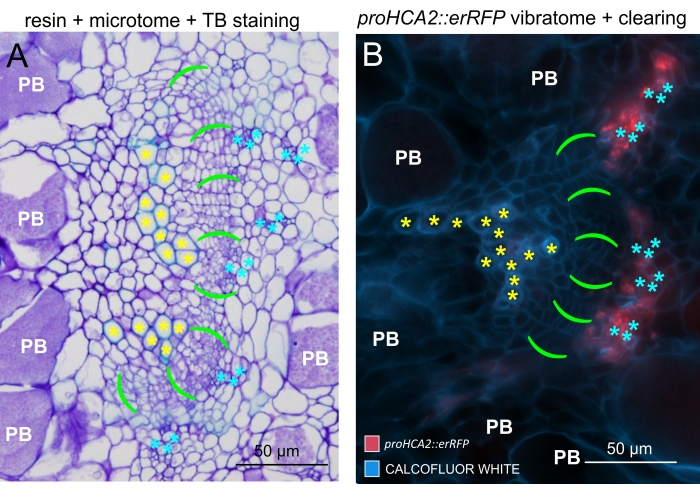
Abbildung 6: Vergleich zwischen TB-, Harz-eingebetteten und mikrotom-geschnittenen Gallen mit Hilfe von Fluoreszenz. (A) Ein Vergleich zwischen Bildern von Toluidinblau (TB), harzeingebetteten und mikrotomgeschnittenen Gallen und (B) einem repräsentativen Objekt (ebenfalls in Abbildung 5B dargestellt), das mit Hilfe von Fluoreszenz aufgenommen wurde. Xylemzellen sind mit gelben Sternchen gekennzeichnet, der kambiale Bereich mit leuchtend grünen Klammern, Phloem mit Cyan-Sternchen und Plasmodiophora brassicae-besiedelte Zellen mit einem weißen PB-Symbol. Harzeingebettete Abschnitte (A) bieten eine gute Auflösung für die Untersuchung der Verteilung ruhender Sporen in hypertrophierten Organen, des Grades der Krankheitsprogression und anderer Prozesse wie der lokalen Verholzung in resistenten Pflanzen. Das hier beschriebene Protokoll (B) ermöglicht jedoch die sensitive Beobachtung der Genexpression oder Proteinakkumulation und die Visualisierung anderer physiologischer Veränderungen und wichtiger Moleküle wie Lipide (in Sporen). Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
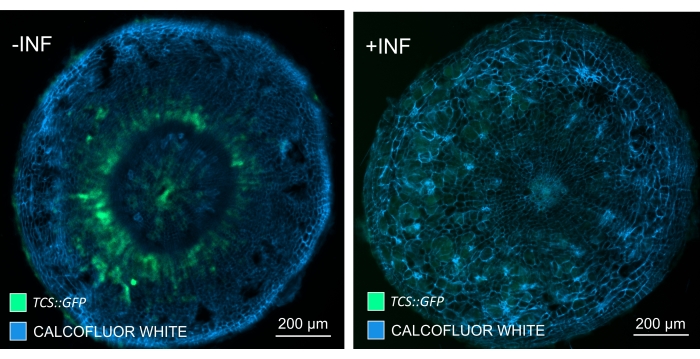
Abbildung 7: Verfolgung von Cytokinin-Signalantworten in Hypokotylen von nicht infizierten (-INF) und P. brassicae-infizierten (+INF) Arabidopsis-Pflanzen bei 16 DPI. TCS::GFP-Marker wurde zur Charakterisierung von Planta-Cytokinin-Antworten verwendet. Vibratomschnitte wurden einer Reinigungsbehandlung unterzogen, gefolgt von einer Färbung mit Calcofluorweiß. Basierend auf dem Bild scheinen die Cytokinin-Reaktionen bei 16 DPI in infizierten Gallen (rechtes Bild) weitgehend vermindert zu sein, während sie stark bleiben, insbesondere im Phloempool (Zellen, die sich schließlich differenzieren, um Phloemgewebe zu bilden), in nicht infizierten Pflanzen (linkes Bild) (Objektivlinse = 5x und Dicke = 30 μm). Bestimmte Konzentrationen von Xylem-Autofluoreszenz können ebenfalls sichtbar sein. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
| Clearing-Lösung (toxisch) | ||
| Komponenten | Prozentsatz (%) | in 100 mL destilliertem Wasser |
| Xylitol | 10% | 10g |
| Natriumdesoxycholat | 15% | 15g |
| Harnstoff | 25% | 25g |
| 10x PBS (phosphatgepufferte Kochsalzlösung) | 1x PBS (100 ml) | |
| NaCl | 8 g | 10 mL 10x PBS + 90 mL destilliertes Wasser |
| Kcl | 0,2 g | |
| KH2PO4 | 0,24 g | |
| Na2HPO4 · 2H2O | 1,81 g | |
| destilliertes Wasser | 100 ml | |
| Ph | pH-Wert mit HCl auf 7,4 eingestellt | |
| Autoklav und bei 4 °C lagern. | ||
Tabelle 1: Zusammensetzung der Clearing-Lösung und der phosphatgepufferten Kochsalzlösung (PBS).
| Fluoreszierende Färbung/ Tag | Anregungs-/Emissionswellenlängen | Verwendetes Mikroskopfilterset |
| Nilrot | 553/636 nm | Filterset 43 |
| Xylem Autofluoreszenz | 380/475 nm | Filterset 49 |
| Calcofluor Weiß | 405/475 nm | Filterset 49 |
| Basic Fuchsin | 561/650 nm | Filterset 43 |
| erRFP | 585/608 nm | Filterset 43 |
| GFP | 488/509 nm | Filterset 38 |
Tabelle 2: Für die vorliegende Studie ausgewählte Anregungs-/Emissionsspektren.
Diskussion
Das Auftragen der Reinigungslösung auf vibratomgeschnittene Abschnitte von Gallen verbessert sicherlich die Fähigkeit, die biotrophe Interaktion zwischen P. brassicae und der Wirtspflanze zu untersuchen. Obwohl das Clearing-Protokoll sogar für Handabschnitte gilt, funktioniert es besser mit Vibratomabschnitten. Die Fixierung von Proben in PFA-Fixiermittel ist ein kritischer Schritt im Protokoll, da die Proben dann einige Tage bei 4 ° C gelagert werden können, bevor mit dem Schneiden fortgefahren wird. Dies bietet die Flexibilität, Proben für einen begrenzten Zeitraum zu lagern, ohne die Expression und Konservierung fluoreszierender Proteine während der Fixierung zu beeinträchtigen.
Nilrot (in DMSO oder Methanol) ist aufgrund seiner Hydrophobie, die Harz auflöst und in Harz eingebettete Abschnitte zerstört17, nicht mit Harzabschnitten kompatibel. Daher erweisen sich Vibratomschnitte als hilfreich für die Untersuchung der Erregerverteilung und ihres Lebenszyklus in sich entwickelnden Gallen, wobei die Nilrotfärbung leicht verwendet werden kann.
Die in diesem Protokoll verwendete Clearing-Lösung ist sehr vielseitig12, so dass eine Vielzahl von Fluoreszenzfärbungen in verschiedenen Kombinationen verwendet werden kann, um verschiedene Biomoleküle / Komponenten der Zellwände (Suberin, Lignin, Cellulose und Chitin in Pilzinteraktionen) zu färben. Es ist auch möglich, Abschnitte fluoreszierender GFP-Markerlinien gegenzufärben und dadurch die Promotoraktivität oder das Proteinakkumulationsmuster mit der Anwesenheit des Erregers in bestimmten Zellen oder Regionen der Galle zu korrelieren. Die Hintergrundautofluoreszenz von Xylem und mit Krankheitserregern gefüllten Riesenzellen konnte jedoch auch nach dem Clearing-Protokoll nicht eliminiert werden. Dies stellt eine Einschränkung für die Betrachtung fluoreszierender Marker in den späteren Stadien der Gallenbildung dar, insbesondere bei Verwendung eines Epifluoreszenzmikroskops und der Abbildung schwacher Signale.
Aufgrund der geringen Expression/Akkumulation von Fluoreszenzsignalen sind Transkriptionsfaktoren schwer zu erkennen, aber mit dieser Technik ist es möglich, zufriedenstellende Bilder für sie zu erhalten. Insgesamt erweitert die Kombination von Vibratomschnitten mit dem Gewebereinigungsansatz das Toolkit für histologische Beobachtungen komplexer Gallengewebe. Die Flexibilität dieses Protokolls erleichtert den Prozess der Gewebefixierung und reduziert die Zeit, die für das Schneiden und Imaging frischer Gewebeproben benötigt wird, um fluoreszierende Proteine und Promotoraktivitäten zu beobachten. Mit weiteren Verbesserungen und durch die Verwendung anderer Fluoreszenzfarbstoffe, die für verschiedene Biomoleküle spezifisch sind, wird diese Methode größere Fortschritte in histologischen Studien und der Bildanalyse von dichten, undurchsichtigen Geweben mit komplexer Gewebeorganisation markieren. In jüngster Zeit hat sich die vorgestellte Gewebereinigungsmethode zu einem beliebten und weit verbreiteten Protokoll entwickelt, um die gleichzeitige Erfassung verschiedener fluoreszierender Signale zu kombinieren undzu ermöglichen 11,12,13. Zukünftige Entwicklungen und Modifikationen solcher Techniken werden die Bildauflösung für die Beobachtung von Pflanzen-Pathogen-Interaktionen auf zellulärer Ebene erheblich verbessern.
Offenlegungen
Die Autoren haben nichts offenzulegen.
Danksagungen
Die Arbeit wurde vom National Science Centre Poland OPUS17 grant No. 2019/33/B/NZ9/00751 "Long distance Vascular Coordination in Plants Infected by Plasmodiophora brassicae" unterstützt. Wir danken Prof. Yrjö Helariutta (Sainsbury Laboratory, University of Cambridge) für die Bereitstellung der proHCA2::erRFP-Linie .
Materialien
| Name | Company | Catalog Number | Comments |
| 2N Sulfuric acid (H2SO4) | Roth | UN2796 | pH adjustment |
| Agarose | PRONA | BGQT100 | Embedding |
| Basic Fuchsin | BIOSHOP | BSF410.5 | Fluorescent dye |
| Calcofluor White | Sigma Aldrich | 18909-100ML-F | Fluorescent dye |
| Commercial Bleach | Domestos | ||
| Cyanoacrylate/ Instant glue | Kropelka | Adhesive | |
| Dimethyl Sulfoxide (DMSO) | BIOSHOP | DMS555.500 | Solvent |
| Epifluorescence microscope | Carl Zeiss M2 automated epifluorescence microscope with Colibri LED system | Carl Zeiss M2 | Carl Zeiss Filter Set filter set 38, 43, 49 used |
| Fully automated Vibratome | Leica | VT1200 S | |
| Lightmeter /Photometer | LI-COR Biosciences | LI-250A + LI-190R quantum sensor | For measuring light intensity within the 400-700nm (PAR) waveband |
| Masking tape | For sticking agarose block on mould | ||
| Murashige & Skoog Medium (MS Medium) | Duchefa Biochemie | MO222.0050 | Plant Growth Medium |
| Nile Red | Sigma Aldrich | N3013-100MG | Fluorescent dye |
| Paraformaldehyde PFA | Sigma Aldrich | 158127-100G | Fixative |
| Potassium Chloride (KCl) | POCH | 739740114 | PBS component |
| Potassium Hydroxide (KOH) | Sigma Aldrich | P1767-250G | pH adjustment |
| Potassium Phosphate Monobasic (KH2PO4) | BIOSHOP | PPM302.500 | PBS component |
| Sodium chloride (NaCl) | BIOSHOP | SOD001.1 | PBS component |
| Sodium Deoxycholate | Sigma Aldrich | D6750-25G | Clearing Solution |
| Sodium Phosphate Dibasic (Na2HPO4 · 2H2O) | POCH | 799490116 | PBS component |
| Triton X-100 | BIOSHOP | TRX506.100 | Fixative |
| Urea | Sigma Aldrich | U5378-100G | Clearing Solution |
| Vacuum/Pressure pump and Dessicator | Welch by Gardner Denver | 2522C-02 | For Vacuum Infilteration |
| Xylitol | Sigma Aldrich | X3375-25G | Clearing Solution (componenet) |
Referenzen
- Harris, M. O., Pitzschke, A. Plants make galls to accommodate foreigners: Some are friends, most are foes. New Phytologist. 225 (5), 1852-1872 (2020).
- Peng, G., et al. Crop rotation, cultivar resistance, and fungicides/biofungicides for managing clubroot (Plasmodiophora brassicae) on canola. Canadian Journal of Plant Pathology. 36 (1), 99-112 (2014).
- Siemens, J., Nagel, M., Ludwig-Müller, J., Sacristán, M. D. The interaction of Plasmodiophora brassicae and Arabidopsis thaliana: Parameters for disease quantification and screening of mutant lines. Journal of Phytopathology. 150 (11-12), 592-605 (2002).
- Malinowski, R., Smith, J. A., Fleming, A. J., Scholes, J. D., Rolfe, S. A. Gall formation in clubroot-infected Arabidopsis results from an increase in existing meristematic activities of the host but is not essential for the completion of the pathogen life cycle. The Plant Journal. 71 (2), 226-238 (2012).
- Walerowski, P., et al. Clubroot disease stimulates early steps of phloem differentiation and recruits SWEET sucrose transporters within developing galls. The Plant Cell. 30 (12), 3058-3073 (2018).
- Olszak, M., et al. Transcriptional profiling identifies critical steps of cell cycle reprogramming necessary for Plasmodiophora brassicae-driven gall formation in Arabidopsis. Plant Journal. 97 (4), 715-729 (2019).
- Malinowski, R., Truman, W., Blicharz, S. Genius architect or clever thief-How Plasmodiophora brassicae reprograms host development to establish a pathogen-oriented physiological sink. Molecular Plant-Microbe Interactions. 32 (10), 1259-1266 (2019).
- Bi, K., et al. Integrated omics study of lipid droplets from Plasmodiophora brassicae. Scientific Reports. 6, 36965 (2016).
- Schuller, A., Ludwig-Müller, J. Histological methods to detect the clubroot pathogen Plasmodiophora brassicae during its complex life cycle. Plant Pathology. 65 (8), 1223-1237 (2016).
- Badstöber, J., Gachon, C. M. M., Ludwig-Müller, J., Sandbichler, A. M., Neuhauser, S. Demystifying biotrophs: FISHing for mRNAs to decipher plant and algal pathogen-host interaction at the single cell level. Scientific Reports. 10, 14269 (2020).
- Kurihara, D., Mizuta, Y., Sato, Y., Higashiyama, T. ClearSee: A rapid optical clearing reagent for whole-plant fluorescence imaging. Development. 142 (23), 4168-4179 (2015).
- Ursache, R., Andersen, T. G., Marhavý, P., Geldner, N. A protocol for combining fluorescent proteins with histological stains for diverse cell wall components. The Plant Journal. 93 (2), 399-412 (2018).
- Sexauer, M., Shen, D., Schön, M., Andersen, T. G., Markmann, K. Visualizing polymeric components that define distinct root barriers across plant lineages. Development. 148 (23), (2021).
- Fuchs, H., Sacristan, M. Identification of a gene in Arabidopsis thaliana controlling resistance to clubroot (Plasmodiophora brassicae) and characterization of the resistance response. Molecular Plant-Microbe Interactions. 9 (2), 91-97 (1996).
- Guo, Y., Qin, G., Gu, H., Qu, L. -JDof56HCA2, a Dof transcription factor gene, regulates interfascicular cambium formation and vascular tissue development in Arabidopsis. The Plant Cell. 21 (11), 3518-3534 (2009).
- Liu, J., Müller, B., Kleine-Vehn, J., Sauer, M. Imaging TCSn::GFP, a Synthetic Cytokinin Reporter, in Arabidopsis thaliana. Plant Hormones: Methods and Protocols. , 81-90 (2017).
- Suzuki, M., Shinohara, Y., Fujimoto, T., Taatjes, D. J., Roth, J. Histochemical Detection of Lipid Droplets in Cultured Cells. Cell Imaging Techniques: Methods and Protocols. , 483-491 (2013).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten