Method Article
Funktionelle Charakterisierung und Visualisierung ösophagealer Fibroblasten mittels Organoid-Cokulturen
In diesem Artikel
Zusammenfassung
Organoid-Fibroblasten-Kokulturen bieten ein Modell zur Untersuchung der In-vivo-Stammzellnische . In dieser Arbeit wird ein Protokoll für ösophageale Organoid-Fibroblasten-Kokulturen beschrieben. Zusätzlich wird die Whole-Mount-Bildgebung verwendet, um die Fibroblasten-Organoid-Interaktion sichtbar zu machen.
Zusammenfassung
Epitheliale Stamm- und Vorläuferzellen tragen zur Bildung und Aufrechterhaltung der Epithelbarriere während des gesamten Lebens bei. Die meisten Stamm- und Vorläuferzellpopulationen sind an anatomisch unterschiedlichen Orten versteckt, was exklusive Interaktionen mit Nischensignalen ermöglicht, die die Stammzellität erhalten. Während die Entwicklung von epithelialen Organoidkulturen ein leistungsfähiges Werkzeug zum Verständnis der Rolle von Stamm- und Vorläuferzellen bei Homöostase und Krankheit darstellt, fehlt die Interaktion innerhalb der Nischenumgebung weitgehend, was die Identifizierung von Faktoren, die das Verhalten von Stammzellen beeinflussen, erschwert. Fibroblasten spielen eine Schlüsselrolle bei der Steuerung des Schicksals von Epithelstamm und Vorläufer. In dieser Arbeit wird ein umfassendes Organoid-Fibroblasten-Kokulturprotokoll vorgestellt, das die Abgrenzung von Fibroblasten-Subpopulationen in der Erneuerung und Differenzierung von Ösophagusvorläuferzellen ermöglicht. In diesem Protokoll wird eine Methode beschrieben, um sowohl Epithelzellen als auch Fibroblasten parallel aus der Speiseröhre zu isolieren. Es werden verschiedene fluoreszenzaktivierte Zellsortierstrategien beschrieben, um sowohl die ösophagealen Vorläuferzellen als auch die Fibroblasten-Subpopulationen aus transgenen Reporter- oder Wildtyp-Mäusen zu isolieren. Dieses Protokoll bietet einen vielseitigen Ansatz, der an die Isolierung spezifischer Fibroblasten-Subpopulationen angepasst werden kann. Die Etablierung und Passage von ösophagealen epithelialen Organoid-Monokulturen ist in diesem Protokoll enthalten und ermöglicht einen direkten Vergleich mit dem Co-Kultursystem. Darüber hinaus wird ein 3D-Clearing-Ansatz beschrieben, der eine detaillierte Bildanalyse von Epithel-Fibroblasten-Interaktionen ermöglicht. Insgesamt beschreibt dieses Protokoll eine vergleichende und relativ hohe Durchsatzmethode zur Identifizierung und zum Verständnis von Nischenkomponenten von Ösophagusstammzellen in vitro.
Einleitung
Organoide werden als 3D-In-vitro-Assays verwendet, um Stamm- und Vorläuferzellen zu charakterisieren und die Signalsignale zu verstehen, die von den zellulären Komponenten der Stammzellnischeabgeleitet werden 1,2,3,4. Ösophagusorganoide der Maus wurden erstmals 2014 beschrieben, und mehrere Arbeiten haben Wachstumsfaktoren wie R-Spondin (RSPO), NOGGIN und epidermalen Wachstumsfaktor (EGF) identifiziert, die für die Aufrechterhaltung und Passage von ösophagealen Organoiden erforderlich sind 5,6,7, wobei argumentiert wird, dass ähnliche Signalsignale für in vivo erforderlich sind Erneuerung der Vorläuferzellen. Wachstumsfaktoren werden jedoch häufig in unphysiologischen Konzentrationen hinzugefügt, was zu organoiden Wachstumsbedingungen führt, die nicht unbedingt die In-vivo-Signalumgebung widerspiegeln.
Fibroblasten sind heterogene Stromazellpopulationen, die die Eigenschaften von Vorläuferzellen in vielen Stammzellnischen unterstützen8. Die Kombination von epithelialen Vorläuferzellen und Fibroblasten in derselben Organoidkultur ermöglicht die Bildung von Organoiden in reduzierten Konzentrationen exogen supplementierter Wachstumsfaktoren. Organoide Kokultursysteme aus intestinalen und hepatischen Epithelien werden beschrieben, aber ein Protokoll zur Etablierung ösophagealer Organoid-Fibroblasten-Kokulturen steht noch aus 9,10,11.
In diesem Protokoll werden zwei fluoreszenzaktivierte Zellsortierungsstrategien (FACS) für Fibroblasten aus der Speiseröhre beschrieben, die entweder transgene PdgfrαH2BeGFP-Mäuse 12 oder Wildtyp-Mäuse mit klassischer Antikörperfärbung verwenden. Verschiedene Subpopulationen von Fibroblasten können mit Hilfe von Markern der Zelloberfläche isoliert werden, was dem Protokoll Flexibilität verleiht. Darüber hinaus wird ein 3D-Fluoreszenz-Bildgebungsverfahren verwendet, das die Organoid-Morphologie bewahrt, um Fibroblasten-Organoid-Wechselwirkungen zu charakterisieren. Organoid-Clearing bietet eine schnelle Methode, um die Lichteindringtiefe in die Organoide zu erhöhen, die Visualisierung von Organoid-Fibroblasten-Verbindungen zu verbessern und die Rekapitulation der Organoidstruktur in ihrer Gesamtheit zu ermöglichen. Dieses Protokoll kombiniert die Ösophagus-Organoid-Co-Kultur mit einer Whole-Mount-Imaging-Strategie und ermöglicht so die funktionelle Charakterisierung der Fibroblasten-Organoid-Interaktion.
Protokoll
Tierversuche für diese Studie wurden von Stockholms Norra djurförsöksetiska nämnd genehmigt (ethische Genehmigung Nr. 14051-2019). Die Tiere wurden unter pathogenfreien Bedingungen nach den Empfehlungen der Federation of European Laboratory Animal Science Association untergebracht.
1. Vorbereitung
- Tauen Sie die enzymatischen Stammlösungen, die für die Dissoziation verwendet werden (siehe Materialtabelle), auf Eis auf. Tauen Sie eine aliquote wachstumsfaktorreduzierte (GFR) Basalmembranmatrix (Matrix) bei 4 °C auf.
- Bereiten Sie das Nährmedium vor. Verwenden Sie das in Tabelle 1 beschriebene Medium für Organoidkulturen und Kokulturen und bereiten Sie es vor, bevor Sie mit dem Protokoll beginnen.
2. Dissektion und Trennung von Ösophagusepithel und Stroma
Anmerkungen: Stellen Sie sicher, dass alle Instrumente, die für die Präparation und Gewebeaufbereitung verwendet werden, steril sind. Bereiten Sie 2 ml Dissoziationslösung (siehe Materialtabelle) in Hanks' balancierter Salzlösung (HBSS) pro drei Speiseröhren vor.
- Verwenden Sie einen Mausstamm Ihrer Wahl, z. B. C57BL/6J-Mäuse. Die Entwicklung des Ösophagus bei Mäusen endet nach dem postnatalen Tag (p) 70, daher wird empfohlen, Mäuse zu verwenden, die älter als p70sind 13. Verwenden Sie vier oder fünf Mäuse, da sie genügend Material liefern, um 8-10 Organoid-Kokulturen zu etablieren.
HINWEIS: Die Effizienz der Organoidbildung nimmt mit dem Alter der Mäuse ab. Wenn bestimmte Fibroblasten-Subpopulationen von Interesse sind, ist es möglich, dass die geringe Fibroblastenausbeute die Anzahl der Organoid-Kokulturen einschränkt, die etabliert werden können. - Verwenden Sie genetisch veränderte (z. B. PdgfrαH2BeGFP) oder Wildtyp-Mäuse (WT) zur Isolierung von Fibroblastenpopulationen. Wenn Sie WT-Mäuse verwenden, führen Sie eine Antikörperfärbung durch, um bestimmte Fibroblasten-Subpopulationen aus dem Stroma über FACS zu isolieren.
- Euthanasieren Sie die Mäuse durch CO2 - Erstickung. Präparieren Sie die Speiseröhre mit einer Pinzette und einer Sezierschere. Um die gesamte Speiseröhre zu entfernen, schneiden Sie das distale Ende der Speiseröhre direkt über dem Magen und das proximale Ende am Anfang der Luftröhre durch. Tauchen Sie die Speiseröhre in PBS und legen Sie sie auf Eis.
- Die Muscularis externa wird mechanisch mit einem Präpariermikroskop (Gesamtvergrößerungsbereich von 8x-40x) und einer Pinzette entfernt. Halten Sie das distale Ende der präparierten Speiseröhre mit einer Pinzette fest und verwenden Sie die andere Pinzette, um den Muskel vom distalen zum proximalen Ende der präparierten Speiseröhre zu greifen und zu ziehen. Entfernen und entsorgen Sie die Muskelschicht.
- Öffnen Sie die Speiseröhre in Längsrichtung. Dies funktioniert am besten mit einer Mikrodissektions-Federschere mit Kugelspitze, um Gewebeschäden zu vermeiden. Halten Sie ein Ende der Speiseröhre fest, um die Kugel der Federschere in das Lumen der Speiseröhre einzuführen, und schneiden Sie die Speiseröhre auf, während Sie sich am Ende festhalten.
- Legen Sie die Speiseröhre in ein 1,5-ml-Mikrozentrifugenröhrchen oder eine 24-Well-Platte. Die geöffnete Speiseröhre wird in 0,5 mg/ml Thermolysin in HBSS bei 37 °C auf einem Rocker-Shaker für 15 min inkubiert. Tauchen Sie die Speiseröhre vollständig in Thermolysinlösung.
HINWEIS: Das Volumen der verwendeten Thermolysinlösung hängt von der Vertiefung oder der Röhrchengröße ab. Mehrere Speiseröhren können in denselben Brunnen oder dieselbe Röhre gelegt und untergetaucht werden. - Nehmen Sie die Speiseröhre aus der Thermolysinlösung. Verwenden Sie ein Präpariermikroskop, um das Ösophagusepithel vorsichtig vom Stroma zu trennen. Fassen Sie mit einer feinen Pinzette sowohl die Epithelseite als auch die Stromaseite des Gewebes und ziehen Sie sie langsam auseinander, um die beiden Schichten zu trennen.
HINWEIS: Das Stroma ist an seinem weißen und undurchsichtigen Aussehen im Gegensatz zum transparenten Epithel zu erkennen. Das Stroma enthält die Lamina propria und die submuköse Schicht. - Die Epithel- und Stromaschicht werden in zwei separate 1,5-ml-Mikrozentrifugenröhrchen mit 200 μl Dissoziationslösung in HBSS überführt. Auf Eis legen.
3. Isolierung von ösophagealen Vorläuferzellen
HINWEIS: Die Isolierung von ösophagealen Vorläuferzellen (Schritt 2) und Fibroblasten (Schritt 3) kann gleichzeitig durchgeführt werden. Bereiten Sie ein 50-ml-Röhrchen mit 1 % FBS in HBSS (1 % FBS) vor.
- Das abgetrennte Ösophagusepithel aus dem 1,5-ml-Mikrozentrifugenröhrchen (Schritt 2.8) in eine saubere Petrischale überführen und mit einem scharfen Skalpell zerkleinern. Sammeln Sie das zerkleinerte Gewebe mit 200 μl Dissoziationslösung aus der Petrischale und übertragen Sie es zurück in das 1,5-ml-Mikrozentrifugenröhrchen.
Anmerkungen: Das Epithel ist richtig zerkleinert, wenn die Stücke mit einer 200-μl-Pipettenspitze wieder in das 1,5-ml-Mikrozentrifugenröhrchen eingesetzt werden können. - Geben Sie 800 μl frische Dissoziationslösung in das 1,5-ml-Mikrozentrifugenröhrchen auf ein Gesamtvolumen von 1 ml.
Anmerkungen: Es ist wichtig, ein ausreichendes Volumen der Dissoziationslösung hinzuzufügen. Pro ein bis drei Speiseröhren wird 1 ml Lösung empfohlen. Skalieren Sie, wenn mehrere Speiseröhren gleichzeitig verarbeitet werden. - Legen Sie das Röhrchen mit der zerkleinerten Epithelschicht für 60 min auf einen Rocker-Shaker bei 37 °C. Pipettieren Sie die Lösung etwa 20 Mal alle 15 Minuten mit einer 200-μl-Pipettenspitze auf und ab, um die Verdauung zu verbessern.
- Nach 60 min mit einer 200-μl-Pipettenspitze weitere 20 Mal auf und ab pipettieren. Die Lösung wird durch ein 40-μm-Zellsieb in ein neues 1,5-ml-Mikrozentrifugenröhrchen geleitet. Bei 300 x g für 10 min bei 4 °C zentrifugieren.
Anmerkungen: Befeuchten Sie das Zellsieb mit 1% FBS, bevor Sie die Zellen abseihen, um die Haftung der Zellen am Filter zu minimieren. Das Epithel wird nicht vollständig verdaut und Gewebestücke sind noch sichtbar. Eine längere Inkubation verringert jedoch das Überleben der Zellen und führt nicht zu einer höheren Ausbeute an lebensfähigen Zellen. - Verwerfen Sie den Überstand, indem Sie überschüssige Flüssigkeit mit einer 1-ml-Pipette entfernen, und resuspendieren Sie das Pellet in 1 ml 1%iger FBS. Bei 300 x g für 10 min bei 4 °C zentrifugieren.
- Während der Zentrifugation wird die konjugierte Antikörpermischung für FACS von Ösophagusvorläuferzellen vorbereitet.
- Mischen Sie 1 μl CD324-PE-Cy7 (ECADHERIN) und CD104-A647 (INTEGRIN-β4) in 200 μl 1% FBS pro eine Million Zellen.
HINWEIS: 1 μl Antikörper (200 μl Endvolumen) ist ausreichend für ein oder zwei Ösophagen. Wenn Sie mehrere Ösophagi auf einmal verarbeiten, erhöhen Sie das Volumen der Antikörper-Färbelösung.
- Das Pellet wird in 200 μl Antikörpermischung resuspendiert und in ein Durchflusszytometrieröhrchen überführt. Halten Sie die Zellsuspensionen nach der Zugabe von fluoreszierenden Antikörpern im Dunkeln, um ein Ausbleichen des Signals zu vermeiden. Inkubieren Sie die Zellen für 30 min bei 4 °C. Fügen Sie 3 ml 1 % FBS hinzu und zentrifugieren Sie 10 Minuten lang bei 4 °C bei 300 x g und resuspendieren Sie die Zellen dann in mindestens 200 μl 1 % FBS.
HINWEIS: 500 μl 1% FBS werden für bis zu vier oder fünf Speiseröhren verwendet. Erhöhen Sie das Volumen, wenn mehr Speiseröhren gleichzeitig verarbeitet werden, so dass eine FACS-Flussrate von 100-300 Ereignissen/s erreicht werden kann. Mehr Ereignisse/s verringern die Sortiereffizienz, und eine Erhöhung der Flussrate verringert das Überleben der Zellen. - Fügen Sie den Marker für die Färbung toter Zellen in einer Endkonzentration von 1:10.000 5 Minuten vor der FACS-Sortierung hinzu, um lebende Zellen zu isolieren. Sortieren Sie die Vorläuferzellen mit einer FACS-Maschine (siehe Abbildung 1 für die Gating-Strategie). Die Zellen werden in 1,5-ml-Mikrozentrifugenröhrchen gesammelt, die mit 200 μl basischem Organoidmedium gefüllt sind (Tabelle 1).
4. Isolierung von Fibroblasten aus der Stromaschicht
- Die Stomaschicht wird in einem 1,5-ml-Röhrchen mit 200 μl Dissoziationslösung (Schritt 2.8) mit einer Sezierschere in feine Stücke geschnitten. Das Gewebe ist richtig zerkleinert, sobald die Lösung mit einer 200-μl-Pipettenspitze auf und ab pipettiert werden kann.
- Geben Sie 800 μl frische Dissoziationslösung in das 1,5-ml-Mikrozentrifugenröhrchen auf ein Gesamtvolumen von 1 ml.
Anmerkungen: Es ist wichtig, ein ausreichendes Volumen der Dissoziationslösung hinzuzufügen. Pro ein bis drei Speiseröhren wird 1 ml Lösung empfohlen. Skalieren Sie, wenn mehrere Speiseröhren gleichzeitig verarbeitet werden. - Legen Sie die Tube für 30 min auf einen Rocker-Shaker bei 37 °C. Nach 15 Minuten pipettieren Sie die Lösung mit einer 200-μl-Pipettenspitze etwa 20 Mal auf und ab, um die Verdauung zu verbessern.
- Nach 30 Minuten enzymatischem Aufschluss pipettieren Sie weitere 20 Mal mit einer 200-μl-Pipettenspitze auf und ab. Die Lösung wird durch ein 70-μm-Zellsieb in ein neues 1,5-ml-Mikrozentrifugenröhrchen abgeseiht. Bei 300 x g für 10 min bei 4 °C zentrifugieren.
Anmerkungen: Befeuchten Sie das Zellsieb mit 1% FBS, bevor Sie die Zellen abseihen, um die Anhaftung der Zellen am Filter zu minimieren. - Verwerfen Sie den Überstand, indem Sie überschüssige Flüssigkeit mit einer 1-ml-Pipette entfernen, und resuspendieren Sie das Pellet in 1 ml 1%iger FBS. Bei 300 x g für 10 min bei 4 °C zentrifugieren.
HINWEIS: Bei Verwendung eines genetisch veränderten Mausstamms, der fluoreszenzmarkierte Fibroblasten enthält, sind Antikörperfärbungen optional. Wenn keine Antikörperfärbung erforderlich ist, fahren Sie mit Schritt 3.7 fort und überführen Sie die Probe in ein Durchflusszytometrieröhrchen.- Während der Zentrifugation wird die konjugierte Antikörpermischung für die FACS-Isolierung von Fibroblasten vorbereitet. Mischen Sie 1 μl CD26-APC (DPP4) in 200 μl 1 % FBS pro eine Million Zellen.
HINWEIS: 1 μl Antikörper reicht für ein oder zwei Speiseröhren. Wenn Sie mehrere Ösophagi auf einmal verarbeiten, erhöhen Sie das Volumen der Antikörper-Färbelösung.
- Während der Zentrifugation wird die konjugierte Antikörpermischung für die FACS-Isolierung von Fibroblasten vorbereitet. Mischen Sie 1 μl CD26-APC (DPP4) in 200 μl 1 % FBS pro eine Million Zellen.
- Das Pellet wird in 200 μl konjugierter Antikörpermischung in 1% FBS resuspendiert und in ein Durchflusszytometrieröhrchen überführt. Inkubieren Sie die Zellen für 30 min bei 4 °C.
- Fügen Sie 3 ml 1% FBS hinzu und zentrifugieren Sie bei 300 x g für 10 min bei 4 °C. Resuspendieren Sie die Zellen in mindestens 200 μl 1 % FBS.
HINWEIS: 500 μl 1% FBS werden für bis zu vier oder fünf Speiseröhren verwendet. Erhöhen Sie das Volumen, wenn mehr Speiseröhren gleichzeitig verarbeitet werden, so dass eine Flussrate von 100-300 Ereignissen/s erreicht werden kann. Mehr Ereignisse/s verringern die Sortiereffizienz, und eine Erhöhung der Flussrate verringert das Überleben der Zellen. Nach der Zugabe von fluoreszierenden Antikörpern sollten die Zellsuspensionen dunkel gehalten werden, um ein Ausbleichen des Signals zu vermeiden. - Fügen Sie den Marker für die Färbung toter Zellen in einer Endkonzentration von 1:10.000 5 Minuten vor der FACS-Sortierung hinzu, um lebende Zellen zu isolieren. Sortieren Sie die Zellen mit einem FACS-Gerät (siehe Abbildung 1 für die Gating-Strategie). Die Zellen werden in 1,5-ml-Mikrozentrifugenröhrchen gesammelt, die mit 200 μl basischem Organoidmedium gefüllt sind (Tabelle 1).
5. Etablierung und Kultivierung von Ösophagusorganoiden
HINWEIS: Vorwarmes ER-niedrig (Organoid-Co-Kultur), ENR-Medium (Organoid) (Beschreibung siehe Tabelle 1) und eine 48-Well-Platte bei 37 °C. Legen Sie die aufgetaute Matrix (vorbereitet in Schritt 1) aliquot auf Eis. Es wird empfohlen, die hier bereitgestellte Matrix (siehe Materialtabelle) für die Ösophagus-Organoidkultur der Maus zu verwenden, da andere Matrixmarken die Effizienz der Organoidbildung negativ beeinflussen.
- Für die organoide Co-Kultur werden die sortierten Epithelzellen und Fibroblasten im Verhältnis 1:2 in einem Röhrchen gemischt. Verwenden Sie für jede Matrixkuppel 5.000 Epithelzellen und 10.000 Fibroblasten. Wenn Sie sich auf weitere Kuppeln vorbereiten, fügen Sie ein Vielfaches von 5.000 Epithelzellen und 10.000 Fibroblasten in ein Röhrchen hinzu. Verwenden Sie für Organoidkulturen ohne Fibroblasten 5.000 Epithelzellen pro Matrixkuppel.
- Zentrifugieren Sie die gemischte Zellpopulation bei 300 x g für 5 min. Entsorgen Sie den Überstand, indem Sie ihn vorsichtig mit einer 200-μl-Pipette entfernen.
- Waschen Sie die Zellen einmal, indem Sie das Pellet in kaltem basischem Organoidmedium resuspendieren und 5 Minuten lang bei 300 x g zentrifugieren. Legen Sie die Zellen auf Eis.
- Bereiten Sie eine Mischung vor, die zu 80 % aus Matrix und zu 20 % aus kaltem basischem Organoidmedium besteht. Alles auf Eis legen, während sich die Matrix bei Raumtemperatur (RT) verfestigt.
- Entsorgen Sie den Überstand nach der Zentrifugation, indem Sie ihn vorsichtig mit einer 200-μl-Pipette entfernen. Resuspendieren Sie die Zellen in 10 μl Matrix-Mix/Dome und legen Sie sie wieder auf Eis.
- Nehmen Sie die vorgewärmte 48-Well-Platte aus dem 37 °C-Inkubator und stellen Sie mit einer 20-μl-Pipette eine Matrixkuppel pro Well her. Jede Kuppel enthält 10 μl 80%ige Matrix, 5.000 Epithelzellen und 10.000 Fibroblasten. Übertragen Sie die Platte kopfüber in einen Inkubator und lassen Sie die Kuppeln weitere 20-30 Minuten bei 37 °C erstarren.
HINWEIS: Eine Abnahme des Matrixkuppelvolumens und/oder eine Zunahme der Zellzahl wirkt sich auf die Effizienz der Organoidbildung aus. - Geben Sie 200 μl vorgewärmtes ER-Low-Medium (Tabelle 1) zu den Matrixkuppeln, die Organoid-Kokulturen enthalten, und ENR-Medium (Tabelle 1) zu den jeweiligen Matrixkuppeln, die nur epitheliale Organoide enthalten, und stellen Sie die Platte in den Inkubator.
- Züchten Sie die Organoide bei 37 °C und 5% CO2. Ergänzen Sie das Medium in den ersten 2 Tagen mit 10 μM Rock-Inhibitor (Y-27623). Gesteinsinhibitoren verhindern den stressinduzierten Zelltod und erhöhen die Chance auf eine erfolgreiche Etablierung von Organoidkulturen.
- Erneuern Sie das Medium alle 2-3 Tage. Stellen Sie sicher, dass das Medium warm ist, um die Dissoziation der temperaturempfindlichen Matrix zu verhindern.
- Führen Sie Analysen des Experiments 6 bis 8 Tage nach der Beschichtung durch. Die Organoide können bis zu 14 Tage in Kultur aufbewahrt werden. Um Tag 14 herum wird ein Verlust der Kuppelintegrität beobachtet.
6. Passage von Organoiden
HINWEIS: Die Weitergabe von Organoiden, die in Co-Kultur gezüchtet wurden, führt zum Verlust von Fibroblasten. Daher wird empfohlen, ENR-Medium für alle Organoide zu verwenden, wenn sie durchgelassen werden. ENR, PBS und eine 48-Well-Platte bei 37 °C vorwärmen.
- Entfernen Sie das Medium und waschen Sie die Vertiefung mit der Matrixkuppel mit vorgewärmtem PBS. Fügen Sie 200 μl kalte 0,25%ige Trypsinlösung hinzu und pipettieren Sie auf und ab, um die Matrixkuppel aufzubrechen.
Anmerkungen: Die Verwendung von kaltem 0,25%igem Trypsin wird empfohlen, da die Matrix temperaturempfindlich ist und dies beim Aufbrechen der Matrixkuppeln hilft. - Inkubieren Sie die Organoide mit Trypsin bei 37 °C für ~20 min. Nach 10 Minuten auf und ab pipettieren, um die Dissoziation der Organoide zu erhöhen. Überwachen Sie den Dissoziationsprozess alle 5-10 Minuten mit einem Mikroskop. Da Trypsin die Lebensfähigkeit der Zellen verringert, kann dies helfen, den idealen Dissoziationszeitpunkt zu bestimmen.
- Nach 20 Minuten pipettieren Sie die Organoide mit einer 200-μl-Pipette auf und ab, um die Organoide zu dissoziieren. Sammeln Sie die Zellen in einem 1,5-ml-Mikrozentrifugenröhrchen, fügen Sie 1 ml basisches organoides Medium hinzu und zentrifugieren Sie bei 300 x g für 5 Minuten bei RT.
- Optional: Um die besten Wachstumsbedingungen für Organoide zu gewährleisten und sicherzustellen, dass neue Organoide aus einzelnen Zellen gewonnen werden, werden die Zellen durch ein vorbenetztes 40-μm-Zellsieb gesiebt. Das Filtern der Zellsuspension führt zur Entfernung von Organoidkernen und Zellklumpen, die schwer zu dissoziieren sind.
- Bereiten Sie eine Matrixmischung vor, die zu 80 % aus Matrix und zu 20 % aus kaltem basischem Organoidmedium besteht. Legen Sie alles auf Eis, während sich die Matrix bei RT verfestigt.
- Entsorgen Sie den Überstand, indem Sie ihn vorsichtig mit einer 200-μl-Pipette entfernen, resuspendieren Sie die Zellen in 10 μl Matrix-Mix/Dome und legen Sie die Mischung wieder auf Eis.
Anmerkungen: Organoide können je nach Kuppeldichte im Verhältnis 1:5 bis 1:10 gespalten werden. Epithelzellen können auch bei 5.000 Zellen/Dome gezählt und neu plattiert werden. - Nehmen Sie die vorgewärmte 48-Well-Platte aus dem 37 °C-Inkubator und machen Sie eine Kuppel pro Well. Übertragen Sie die Platte kopfüber in einen Inkubator und lassen Sie die Kuppeln weitere 20-30 Minuten bei 37 °C erstarren.
- Geben Sie 200 μl vorgewärmtes ENR-Medium in die entsprechenden Organoidkuppeln. Ergänzen Sie das Medium in den ersten 2 Tagen mit 10 μM Rock-Inhibitor (Y-27623).
- Erneuern Sie das Medium alle 2-3 Tage. Stellen Sie sicher, dass das Medium warm ist, um eine Dissoziation der temperaturempfindlichen Matrix zu verhindern.
7. Organoid-Verarbeitung für die Färbung ganzer Einrichter
Anmerkungen: Beschichten Sie die Spitzen und Röhrchen vor Gebrauch mit 10 % FBS in PBS, um zu vermeiden, dass Organoide an Kunststoffen haften. Bei Pipettenspitzen reicht es aus, vor der Verwendung der Spitze ein- oder zweimal in 10%iger FBS/PBS-Lösung auf und ab zu pipettieren. Füllen Sie bei Röhrchen das Röhrchen mit 10 % FBS/PBS und entfernen Sie dann die Lösung.
- Entfernen Sie das Organoidmedium und geben Sie 200 μl eiskaltes PBS in die Matrixkuppeln. Legen Sie den Teller für 5-10 Minuten auf Eis.
- Pipettieren Sie auf und ab und übertragen Sie die Lösung in 0,6-ml-Mikrozentrifugenröhrchen, die nicht haften. 30-60 s bei 100 x g kurz zentrifugieren, damit sich die Organoide am Boden absetzen können.
Anmerkungen: Übermäßiges Pipettieren und langes Zentrifugieren brechen die Organoide auf und stören die Fibroblasten-Organoid-Interaktion. - Entfernen Sie überschüssige Flüssigkeit und fügen Sie eiskaltes PBS hinzu. 30-60 s bei 100 x g kurz zentrifugieren, damit sich die Organoide am Boden absetzen können.
- Entfernen Sie überschüssige Flüssigkeit und fixieren Sie das Organoid mit 200 μl kaltem 4%igem Formaldehyd in PBS-Lösung für 30 Minuten auf Eis.
ACHTUNG: Formaldehyd ist giftig und muss immer in einem chemischen Abzug verwendet werden. Nitrilhandschuhe, Schutzbrillen und Laborkittel müssen immer getragen werden. - Durch die Fixierung des Organoids sinken sie ab, und eine Zentrifugation ist nicht mehr erforderlich. Lassen Sie die Organoide sinken, indem Sie das Röhrchen aufrecht stellen und 2-3 Minuten warten, entfernen Sie das Formaldehyd und fügen Sie 500 μl kaltes PBS hinzu, um das Formaldehyd wegzuwaschen.
- Lassen Sie die Organoide einsinken, entfernen Sie das überschüssige PBS und fügen Sie 500 μl frisches, kaltes PBS hinzu. Lassen Sie die Organoide einsinken, entfernen Sie das überschüssige PBS und fügen Sie 500 μl Blockierungspuffer hinzu (5 % normales Eselsserum, 1 % BSA und 0,5 % Triton X-100 in PBS). Legen Sie den Schlauch für 60 min bei RT auf einen Rocker-Shaker.
HINWEIS: Das Blockieren kann auch über Nacht (O/N) bei 4 °C erfolgen. - Lassen Sie die Organoide absinken, entfernen Sie den Blockierungspuffer und resuspendieren Sie die Organoide in 200 μl Blockierungspuffer mit Primärantikörpern (siehe Materialtabelle). Halten Sie die Organoide auf einem Wippen-Shaker O/N bei 4 °C.
HINWEIS: Um die Färbung von Kernproteinen, niedrig exprimierten Proteinen oder Antikörpern, die eine unspezifische Färbung aufweisen, zu verbessern, kann die Inkubationszeit um 1 oder 2 Tage bei 4 °C verlängert werden. Die Platzierung der Organoide auf einem Rocker-Shaker ist unerlässlich, da dies verhindert, dass die Organoide verklumpen, und die Färbeeffizienz erhöht. - Lassen Sie die Organoide einsinken, entfernen Sie die primäre Antikörpermischung und waschen Sie die Organoide mit 500 μl 0,02 % Triton X-100 in PBS (0,02 % Tx) für 60 Minuten bei RT. Wiederholen Sie dies dreimal.
Anmerkungen: Wenn eine längere Inkubation der primären Antikörper erforderlich ist, fügen Sie einen Waschschritt mit 0,02 % Tx in PBS O/N bei 4 °C hinzu. - Lassen Sie die Organoide einsinken, entfernen Sie den Waschpuffer und fügen Sie 200 μl fluoreszenzkonjugierten Sekundärantikörper in den Blockierungspuffer O/N bei 4 °C hinzu. Halten Sie die Organoide nach der Zugabe von fluoreszierenden Sekundärantikörpern im Dunkeln, um ein Ausbleichen des Signals zu vermeiden.
- Lassen Sie die Organoide einsinken, entfernen Sie die sekundäre Antikörpermischung und waschen Sie die Organoide mit 500 μl 0,02 % Tx für 60 min bei RT.
- Bei Bedarf werden die Organoide mit 200 μl DAPI-Lösung (0,25 μg/ml) für die Kernfärbung gegengefärbt. Inkubieren Sie die Proben für 60 min bei RT auf einem Rocker-Shaker.
- Lassen Sie die Organoide einsinken, entfernen Sie die DAPI-Lösung und waschen Sie die Organoide in 500 μl PBS für 15 Minuten bei RT. Wiederholen Sie den Vorgang dreimal.
- Lassen Sie die Organoide einsinken und entfernen Sie die überschüssige Flüssigkeit. Fügen Sie den Organoiden 10 μl Klärlösung hinzu und inkubieren Sie sie 15 Minuten lang bei RT.
Anmerkungen: Die Clearinglösung ist zähflüssig, schneiden Sie daher den äußeren Teil einer 20-μl-Pipettenspitze ab, bevor Sie die Clearinglösung pipettieren. Eine weitere Clearing-Lösung kann hinzugefügt werden, wenn ein größerer Abstandshalter für die Bildgebung verwendet wird. Die Einbettlösung kann anstelle der Reinigungslösung verwendet werden, wenn die Organoide nicht gereinigt werden. Die Clearing-Lösung reduziert den Hintergrund bei der Bildgebung und erhöht die Abbildungstiefe. - Legen Sie einen doppelseitigen, klebrigen 4-Well-Abstandshalter von 0,05 mm auf einen Objektträger. Geben Sie die 10 μl Klärlösung mit den Organoiden in eine Vertiefung und legen Sie ein Deckglas auf den Abstandshalter.
HINWEIS: Organoide können auch ohne die Verwendung eines Abstandshalters montiert werden. Der Abstandshalter hält die Organoidform intakt und verhindert, dass die Organoide abgeflacht werden. - Halten Sie den Objektträger auf RT O/N, um die Organoide zu entfernen. Für eine Langzeitlagerung bewahren Sie die Objektträger bei 4 °C auf. Erfassen Sie Bilder mit einem konfokalen Mikroskopiesystem.
Ergebnisse
Die Speiseröhre ist in verschiedene Schichten unterteilt: Epithel, Lamina propria, Submukosa und Muscularis externa (Abbildung 1A). Fibroblasten befinden sich in der Submukosa und der Lamina propria, die als Stroma bezeichnet werden. Bei diesem Protokoll wird die Muscularis externa mechanisch entfernt (Abbildung 1B), was nicht zu einem Verlust der im Stroma befindlichen Fibroblasten (PdgfrαH2BeGFP+) führt (Abbildung 1C). Vor der Dissoziation wird das Epithel vom Stroma getrennt, so dass zwei Gewebesegmente entstehen (Abbildung 1B). Die Trennung der beiden Schichten bietet die Möglichkeit, die Dissoziationszeit für die robustere Epithelschicht im Vergleich zur fragilen Stromaschicht zu verlängern. Auf diese Weise wird ein effizientes Isolationsprotokoll etabliert, das sowohl lebensfähige epitheliale Vorläuferzellen als auch stromale Fibroblasten liefert (Abbildung 1B). Die Vorläuferzellen des Ösophagus werden anhand ihrer hohen INTEGRIN-β4- und E-CADHERIN-Expression sortiert (Abbildung 1C,D).
Subpopulationen von Fibroblasten können mit Hilfe verschiedener Marker isoliert werden. In diesem Protokoll wird eine Strategie zur Isolierung von Fibroblasten vorgestellt, die auf den häufig verwendeten Fibroblastenmarkern PDGFRα und DPP4 (CD26) basiert. Die Isolierung entweder durch die PdgfrαH2BeGFP-Reporterexpression oder den DPP4-Antikörper zeigt, dass etwa 50% der isolierten Zellen Fibroblasten sind (Abbildung 1E,F). Darüber hinaus sind 70% der PDGFRα+-Fibroblasten DPP4+, was darauf hindeutet, dass eine weitgehend überlappende, aber nicht identische Fibroblastenpopulation erhalten wird. Nach der Isolierung von Epithel- und Stromazellpopulationen werden Ösophagusvorläuferzellen entweder allein oder zusammen mit Fibroblasten in einer Matrixkuppel kultiviert. Um den Beitrag von Fibroblasten zur Organoidbildung zu untersuchen, wird die Co-Kultur in einem wachstumsfaktorreduzierten Medium beibehalten (Abbildung 1G).
Ösophagus-Vorläuferzellen bilden Organoide in Gegenwart von EGF, NOGGIN und RSPO (ENR). Entfernen von NOGGIN und Reduzierung der RSPO-Menge (25 ng/μl; ERniedrig) ist ausreichend, um die Bildung von Organoiden zu verhindern (Abbildung 2A). Interessanterweise stellt die Zugabe von DPP4+- oder PDGFRα+-Fibroblasten zu den Vorläuferzellen des Ösophagus imER-Low-Medium die Fähigkeit zur Organoidbildung wieder her, was eine unterstützende Funktion für beide Fibroblastenpopulationen zeigt (Abbildung 2A-D). Die Visualisierung des PdgfrαH2BeGFP-Transgens zeigt, dass Fibroblasten während der Organoidbildung in engem Kontakt mit den epithelialen Vorläuferzellen stehen (Abbildung 2A). An Tag 6 sind PdgfrαH2BeGFP+ Fibroblasten noch reichlich in der Co-Kultur vorhanden. Fibroblasten sind in der gesamten Kuppel vorhanden, in der Nähe und Berührung der Organoide (vollständiger Pfeil) oder an den Organoiden befestigt (Pfeilspitze; Abbildung 2D).
Die Färbung des gesamten Mount zeigt eine 3D-Darstellung der Wechselwirkung der Fibroblasten mit den Organoiden (Abbildung 3). Es ist zwar möglich, das gesamte Einbettungsprotokoll ohne die Verwendung einer Clearing-Lösung durchzuführen, aber es verringert die Transparenz und die Laserdurchdringung des Organoids (Abbildung 3B, z-Ansicht). Bei der Montage von Organoiden hilft der Abstandshalter, die Organoidmorphologie zu erhalten. Im Gegensatz dazu flacht das Deckglas direkt auf den Organoiden (ohne Abstandshalter) die Organoide ab und führt zum Verlust der Organoidstruktur (Abbildung 3A,B).
Sowohl die DPP4+- als auch die PDGFRα+-Fibroblasten sind um die Organoide gewickelt (Abbildung 3C, Video1 und Video 2). Die Differenzierung von ösophagealen Organoiden kann anhand verschiedener Marker beurteilt werden. Abbildung 4 zeigt, dass das bereitgestellte Färbeprotokoll sowohl für leichter zu färbende Keratine (KRT14/13) als auch für schwieriger zu färbende Transkriptionsfaktoren (TRP63/KLF4) geeignet ist. Das Co-Kultur-Protokoll erzeugt Organoide mit einem ähnlichen Differenzierungsmuster, wie es in vivo13,14 gezeigt wurde und wie es in Organoiden beobachtet wurde, die in ENR-Medium gezüchtet wurden. KRT14+ oder TRP63+ Vorläuferzellen bilden die äußere Schicht und KRT13+ oder KLF4+ differenzierte Zellen orientieren sich nach innen.
Dieses Protokoll bietet ein Werkzeug, um die Nische der Ösophagusstammzellen in vitro zu untersuchen und die Interaktion zwischen Organoiden und Fibroblasten sichtbar zu machen. Durch die Implementierung eines Protokolls zur Isolierung von Fibroblasten mit Hilfe von Antikörpern ist die Methode anpassungsfähig und kann zur Untersuchung von Fibroblasten-Subpopulationen verwendet werden, ohne dass transgene Mäuse benötigt werden.
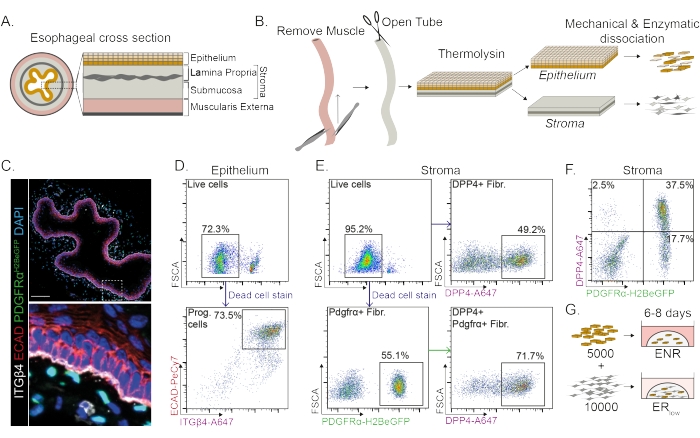
Abbildung 1: Isolierung von Vorläuferzellen und Fibroblasten-Subpopulationen aus der Speiseröhre. (A) Schematische Übersicht über die verschiedenen Schichten in der Speiseröhre. Das Stroma enthält die Lamina propria und die Submukosa. (B) Schematische Übersicht über das Isolationsprotokoll. Der Muskel (Muscularis externa) wird mechanisch mit einer Pinzette entfernt; Die verbleibende Speiseröhre wird aufgeschnitten und in Thermolysin inkubiert, um die Epithelschicht vom Stroma zu trennen. Epithel und Stroma werden getrennt, mechanisch zerkleinert und enzymatisch zu einzelligen Suspensionen verdaut. Dissoziierte Zellen werden dann gefärbt und für FACS präpariert. (C) Querschnitt durch die Speiseröhre, der von der Muscularis externa abgestreift wurde, zeigt PdgfrαH2BeGFP+ Fibroblasten im Stroma. Die doppelt positiven Zellen INTEGRIN-β4 (ITGβ4) und E-CADHERIN (ECAD) sind die epithelialen Vorläuferzellen der Speiseröhre. Maßstabsbalken = 100 μm. (D) Repräsentatives durchflusszytometrisches Diagramm der Epithelzellisolierung, das den prozentualen Anteil lebender Zellen (oberes Bild) aller Einzelzellen zeigt. Das untere Bild zeigt den prozentualen Anteil der isolierten ITGβ4+ ECAD+ Vorläuferzellen (Prog.) aus allen lebenden Zellen. (E) Repräsentatives durchflusszytometrisches Diagramm der Stromazellisolierung, das den prozentualen Anteil lebender Zellen zeigt (oberes linkes Feld). Repräsentative durchflusszytometrische Diagramme, die den prozentualen Anteil der isolierten DPP4+-Fibroblasten (Fibr.; oben rechts) und Pdgfrα+-Fibroblasten (unten links) an allen lebenden Zellen zeigen. 70% der Pdgfrα+ Fibroblasten sind ebenfalls DPP4+ (unten rechts). (F) Repräsentatives durchflusszytometrisches Diagramm des Stromas mit DPP4+-Zellen (2,5 %), DPP4+-PDGFRα+-Zellen (37,5 %) und reinen PDGFRα+-Zellen (17,7 %). Die Prozentsätze beziehen sich auf alle lebenden Zellen. (G) Nur Epithelzellen werden in Gegenwart von 50 ng/μL EGF, 100 ng/μL NOGGIN und 250 ng/μL RSPO (ENR) oder zusammen mit Fibroblasten in Gegenwart von EGF und einer niedrigen Konzentration von RSPO (25 ng/μL) in eine Matrixkuppel plattiert. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
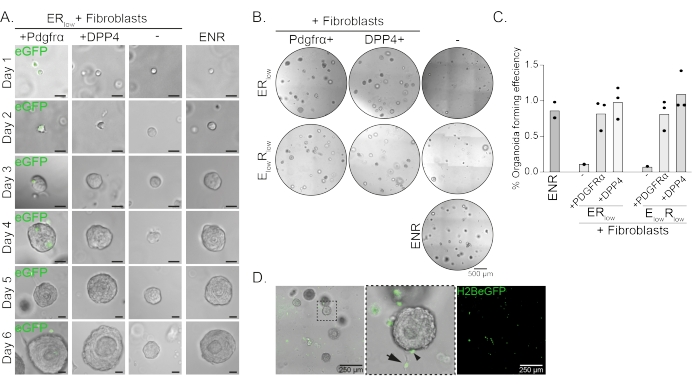
Abbildung 2: Repräsentative Ergebnisse von Organoid-Kokulturen. (A) Hellfeldbilder, die das Wachstum der Organoide von Tag 1 bis Tag 6 zeigen. Die Hellfeldbilder mit den Organoiden, die mit PdgfrαH2BeGFP+ Fibroblasten kokultiviert wurden, zeigen auch das nukleäre eGFP-Signal. Maßstabsbalken = 25 μm. (B) Hellfeldbilder der gesamten Matrixkuppel an Tag 6. Die linke Spalte zeigt Organoid-Co-Kulturen, die in Gegenwart von Pdgfrα+-Fibroblasten in ER-niedrigem oderE-niedrigem R-Low-Medium gezüchtet wurden. Die mittlere Säule zeigt Organoid-Kokulturen, die in Gegenwart von DPP4+-Fibroblasten inER-niedrigem oderE-niedrigemR-Low-Medium gezüchtet wurden. Die rechte Spalte zeigt organoide Monokulturen, die in ENR-Medium gezüchtet wurden. ENR-Medium = EGF (50 ng/μl), NOGGIN (100 ng/μl) und RSPO (250 ng/μl). ERniedrig = EGF und 25 ng/μL RSPO. E niedrigRniedrig = 5 ng/μL EGF und 25 ng/μL RSPO. Maßstabsbalken = 500 μm. (C) Diagramm mit der Effizienz der Organoidbildung (%) (d. h. der Prozentsatz der Zellen, die unter verschiedenen Kulturbedingungen Organoide bilden). Jeder Punkt stellt eine Matrixkuppel dar, und der Balken stellt den Mittelwert aller Punkte pro Bedingung dar. (D) Hellfeld- und Fluoreszenzbild von Tag-6-Organoiden, die mit PdgfrαH2BeGFP+-Fibroblasten kokultiviert wurden. PdgfrαH2BeGFP+-Fibroblasten sind in der gesamten Kuppel vorhanden, an die Organoide gebunden (Pfeilspitze) und nicht gebunden, aber in Kontakt mit den Organoiden (vollständiger Pfeil). Maßstabsbalken = 250 μm. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
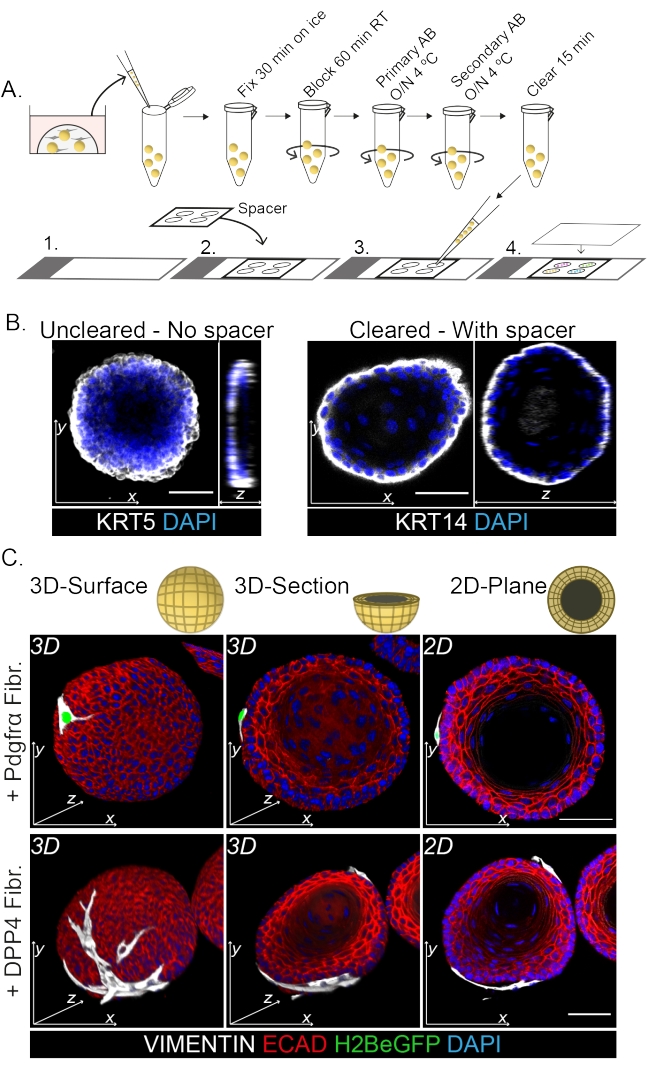
Abbildung 3: Whole-Mount-Färbeprotokoll zur Untersuchung von Fibroblasten-Organoid-Wechselwirkungen . (A) Schematische Übersicht über das gesamte Mount-Immunfluoreszenzprotokoll. AB = Antikörper. (B) Immunfluoreszenzbild einer ungeklärten Ganzbettfärbung, die eine verringerte Transparenz und Durchdringung des Laserlichts im Vergleich zu den geklärten Organoiden zeigt. Das Fehlen eines Abstandshalters führt zu einer Abflachung des Organoids und zu einem Verlust der Organoidmorphologie. (C) Ganzbettbilder der kokultivierten Organoide zeigen 3D-Oberflächen der Organoide mit VIMENTIN+ Fibroblasten (Fibr.), die um das Organoid gewickelt sind und in engem Kontakt mit ihm stehen. 3D-Querschnitte und 2D-Ebenenbilder zeigen das Lumen des Organoids. Maßstabsbalken = 50 μm. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
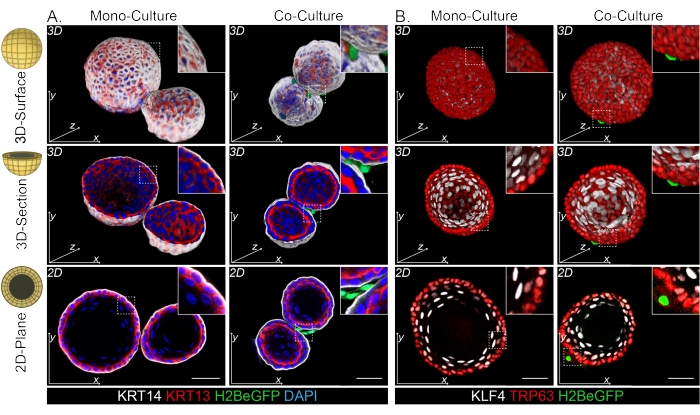
Abbildung 4: Whole-Mount-Bilder zeigen unterschiedliche basale und suprabasale Zellpopulationen . (A) Whole-Mount-Färbung von mono- und kokultivierten Organoiden mit PdgfrαH2BeGFP+-Fibroblasten mit KRT14+ Basalzellen in der äußeren Schicht und KRT13+ differenzierten suprabasalen Zellen. Maßstabsbalken = 50 μm. (B) Ganzbettfärbung von mono- und kokultivierten Organoiden mit PdgfrαH2BeGFP+ Fibroblasten mit TRP63+ Basalzellen in der äußeren Schicht und KLF4+ differenzierten suprabasalen Zellen. Maßstabsbalken = 50 μm. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
Tabelle 1: Tabelle, in der die Bestandteile der Organoid-Nährmedien beschrieben sind. Bitte klicken Sie hier, um diese Tabelle herunterzuladen.
Video 1: PdgfrαH2BeGFP+ Fibroblast umhüllt und in engem Kontakt mit dem Organoid. Das Video begleitet die obere Tafel von Abbildung 3C. Der Maßstabsbalken in Abbildung 3C beträgt 50 μm und das Organoid hat einen Durchmesser von ~120 μm. VIMENTIN ist in Weiß, E-CADHERIN in Rot, PdgfrαH2BeGFP in Grün und DAPI in Blau dargestellt. Bitte klicken Sie hier, um dieses Video herunterzuladen.
Video 2: DPP4+-Fibroblasten, die um das Organoid gewickelt sind und in engem Kontakt mit ihm stehen. Das Video ist dem unteren Bereich von Abbildung 3C beigefügt. Der Maßstabsbalken in Abbildung 3C beträgt 50 μm und das Organoid hat einen Durchmesser von ~120 μm. VIMENTIN ist in weiß, E-CADHERIN in rot und DAPI in blau dargestellt. Bitte klicken Sie hier, um dieses Video herunterzuladen.
Diskussion
Das hier vorgestellte Protokoll etabliert ein in vitro Modell zur Untersuchung funktioneller ösophagealer Epithel-Fibroblasten-Interaktionen.
Die Epithelschicht ist vom Stroma getrennt, was ein optimiertes Dissoziationsprotokoll sowohl für das epitheliale als auch für das stromale Kompartiment ermöglicht. Trotz Optimierung des epithelialen Dissoziationsprotokolls bleiben Gewebeklumpen sichtbar. Wenn Sie alle 15 Minuten kräftig auf und ab pipettieren, verringert sich die Anzahl und Größe der Klumpen erheblich. Andere Protokolle verwenden Trypsin, um die Epithelschicht weiter zu dissoziieren 5,6. Hier wird die Verwendung von Trypsin oder eine weitere Verlängerung der Dissoziationszeit nicht empfohlen, da dies tendenziell zu einer verringerten Lebensfähigkeit der Epithelzellen und einer verminderten Effizienz der Organoidbildung führt. Im Gegensatz zum Epithel ist das Stroma leicht dissoziierbar, und 30 min in Dissoziationslösung führen zu einer einzelligen Suspension mit ~90% Fibroblastenviabilität (Abbildung 1E). Der Ausschluss des epithelial-stomalen Trennschritts im Protokoll verlängert die Dissoziationszeit erheblich, was zu einer verringerten Lebensfähigkeit der Fibroblasten und einer geringeren Ausbeute an Epithelzellen führt. Darüber hinaus bietet die Trennung des Epithels vom Stroma die Möglichkeit, die Zellzahlen jeder Population zu bestimmen und Epithelzellen und Fibroblasten aus verschiedenen Mauslinien beim Aufbau der Kokulturen zu mischen.
Die Untersuchung der Funktion von Fibroblasten auf das Organoidwachstum ist eine häufig verwendete Methode in der Stammzellbiologie 9,10,11,15,16. Etablierte Co-Kultivierungsmedien sind entweder DMEM, das mit 10 % fetalem Kälberserum (FCS)9,15 oder wachstumsfaktorreduziertem Medium10,16 ergänzt wird. In diesem Protokoll wird das wachstumsfaktorreduzierte Medium verwendet, um die Bedingungen in der In-vivo-Stammzellnische nachzuahmen, in der die Fibroblasten weitgehend ruhen. FCS ist ein wachstumsfaktorreiches Serum, das zu einer Aktivierung und Proliferation von Fibroblasten in den Kokulturen führt, was wahrscheinlich einem Fibroblastenzellzustand entspricht, der sich vom In-vivo-Zustand unterscheidet. Durch den Ausschluss von FCS und die Reduzierung von Wachstumsfaktoren, so dass das Medium allein (ERlow) das Organoidwachstum nicht unterstützt und die Proliferation der Fibroblasten nicht stimuliert, ist es möglich, die Wirkung der Fibroblasten auf das Organoidwachstum zu isolieren. In diesem Medium wird NOGGIN entfernt und RSPO auf ein Minimum reduziert (10% RPSO). Sowohl NOGGIN als auch RSPO haben sich als essentiell für das Wachstum von Organoiden in der Speiseröhre erwiesen6. EGF wurde im Co-Kulturmedium beibehalten, da es das Organoidwachstum selbst nicht unterstützt. Fibroblasten sind aber auch in der Lage, das Organoidwachstum in einem EGF-reduzierten Medium (Elow Rlow; Abbildung 2B,D).
Organoide Kokulturen können nicht durch Passage aufrechterhalten werden, da Fibroblasten während der Trypsinisierung verloren gehen. Die Organoid-Passage wurde jedoch in das Protokoll aufgenommen, da ösophageale Organoide beibehalten, erweitert und für weitere Experimente als Monokulturen verwendet werden können. Weitergegebene Organoide aus Monokulturen können verwendet werden, um Kokulturen mit frisch isolierten Fibroblasten aufzubauen. Ein Nachteil bei der Verwendung von Primärzellen ist die Anzahl der Mäuse, die benötigt werden, um mehrere Organoid-Kokulturen aufzubauen. Wenn man sich auf kleine Subpopulationen von Fibroblasten konzentriert, ist die Anzahl der erhaltenen Kokulturen begrenzt. In anderen Protokollen werden Fibroblasten zunächst in Kultur vermehrt, bevor sie zum Aufbau von Organoid-Kokulturen verwendet werden10. Fibroblasten verändern jedoch ihre Morphologie und Identität während der Passage, wie die Verwendung von primären Haut- und kardialen Fibroblasten zeigt17,18. Die konventionelle 2D-Passage von ösophagealen Fibroblasten führt sowohl zu Veränderungen der Morphologie als auch des Phänotyps, was zeigt, dass die In-vitro-Anreicherung von Fibroblasten nicht für Kokulturen geeignet ist, die darauf abzielen, die endogene Stammzellnische zu phänokopieren.
Die Whole-Mount-Färbung bietet ein Werkzeug zur Aufrechterhaltung und Visualisierung der Fibroblasten-Organoid-Interaktion. Es sollte beachtet werden, dass zwar nicht alle Organoide direkt an Fibroblasten gebunden sind, die meisten Organoide jedoch in Kontakt mit Fibroblasten stehen (siehe Abbildung 2C). Um die Wechselwirkungen zwischen Epithel und Fibroblasten aufrechtzuerhalten, ist es wichtig, die Organoide mit Vorsicht zu behandeln und heftiges Pipettieren, Vortexing und Hochgeschwindigkeitsschleudern zu vermeiden. Eine optimale Fixierung ist wichtig, um die 3D-Gewebearchitektur aufrechtzuerhalten und die endogene Fluoreszenz zu erhalten. Eine 30-minütige Fixierung reicht aus, um das H2BeGFP-Signal zu erhalten, und ist optimal für die in diesem Protokoll verwendeten Antikörper, dies kann jedoch zwischen den verwendeten Fluorophoren und Antikörpern variieren. Das Löschen der Organoide reduziert die Lichtstreuung und verbessert die Visualisierung der gesamten 3D-Struktur erheblich. Da die Organoide klein sind, ist die Reinigung einfach und schnell; Die Abbildung ganzer Organoide mit konfokaler Laser-Scanning-Mikroskopie kann jedoch zeitaufwändig sein, da mehrere Z-Stapel hergestellt werden müssen. Konfokale Mikroskope, wie die rotierende Scheibe, können verwendet werden, um die Bildgebungszeit zu verkürzen.
Insgesamt bieten ösophageale Organoide, die in Gegenwart von Fibroblasten gezüchtet werden, ein wertvolles Werkzeug, um Aspekte der Nische der ösophagusalen Stammzellen zu verstehen. Darüber hinaus bietet das Whole Mount Clearing eine zugängliche Methode, um die Wechselwirkung zwischen Fibroblasten und Organoiden zu visualisieren.
Offenlegungen
Die Autoren erklären, dass kein Interessenkonflikt besteht.
Danksagungen
Diese Studie wurde vom ERC StG TroyCAN (851241) unterstützt. E.E. ist Postdoktorandin des Cancerfonden. M.G. ist Ragnar Söderberg Fellow und Cancerfonden Junior Investigator. Wir sind dankbar für die technische Unterstützung durch die Kerneinrichtungen des Karolinska-Instituts, einschließlich der Biomedicum Flow Cytometry Core Facility, des Biomedicum Imaging Core (BIC) und der Comparative Medicine Biomedicum (KMB) Tiereinrichtung. Wir danken den Mitgliedern des Genander-Labors für die sorgfältige Lektüre und Kommentierung des Protokolls.
Materialien
| Name | Company | Catalog Number | Comments |
| B-27 Supplement (50X), serum free | Thermo Fisher (Gibco) | 17504001 | |
| Corning Matrigel Growth Factor Reduced (GFR) Basement Membrane Matrix | fisher scientific | 356231 | |
| Dimethyl sulfoxide | Sigma-Aldrich | 276855-100ML | |
| DMEM/F-12 | Thermo Fisher (Gibco) | 11320074 | |
| DPBS | Thermo Fisher (Gibco) | 14190250 | |
| Fetal Bovine Serum | Sigma-Aldrich | F7524 | |
| GlutaMAX Supplement | Thermo Fisher (Gibco) | 35050061 | |
| HBSS, no calcium, no magnesium, no phenol red | Thermo Fisher (Gibco) | 14175-129 | |
| Normal Donkey Serum | Jackson Immuno | 017-000-121 | |
| Penicillin-Streptomycin (10,000 U/mL) | Thermo Fisher (Gibco) | 15140122 | |
| Triton X-100 solution | Merck | 93443-100ML | |
| Trypsin-EDTA (0.25%), phenol red | Thermo Fisher (Gibco) | 25200-056 | |
| Chemicals, Peptides, and recombinant proteins | |||
| DAPI Solution | Thermo Fisher | 62248 | |
| Dissociation solution: 0.25 mg/ml Liberase TM, 0.25 mg/ml Dnase in HBSS | |||
| Dnase I | Sigma-Aldrich | 11284932001 | |
| Formaldehyde, 37%, with 10-15% methanol | Sigma-Aldrich | 252549-1L | |
| Liberase | Sigma-Aldrich | 5401127001 | |
| N-Acetyl-cysteine | Sigma-Aldrich | A9165-25G | |
| Noggin murine | Peprotech | 250-38 | |
| RapiClear 1.47 | SunJin Lab | #RC147001 | |
| Recombinant Mouse EGF Protein, CF | R&D systems | 2028-EG-200 | |
| R-spondin-1 murine | Peprotech | 315-32 | |
| SYTOX Blue Dead Cell Stain | Thermo Fisher | S34857 | |
| Thermolysin | Sigma-Aldrich | T7902-25MG | |
| Y-27632 dihydrochloride | Sigma-Aldrich | Y0503-5MG | |
| Plastic & Glassware | |||
| Corning Sterile Cell Strainers 40um | VWR | 15360801 | |
| Corning Sterile Cell Strainers 70um | VWR | 431751 | |
| Menzel Deckgläser/ cover slips | Thermo Fisher | Q10143263NR15 | |
| SafeSeal reaction tube, 1.5 mL, PP | Sarstedt | 72.706 | |
| Snap Cap Low Retention Microcentrifuge Tubes 0.6 mL | Thermo Fisher | 3446 | |
| SuperFrost Slides | VWR | 631-9483 | |
| Tools | |||
| 0.05 mm 4 circular well iSpacer | SunJin Lab | #IS204 | |
| Dumont #5 forceps, biology tip | F.S.T | 11252-20 | |
| ImmEdge Pen | VectorLaboratories | H-4000 | |
| Spring Scissors Angled to Side Ball Tip 8mm Cutting Edge | F.S.T | 15033-09 | |
| Instruments | |||
| Confocal microscope Stellaris 5 | Leica | ||
| Dissection microscope ZEISS Stemi 305 | Zeiss | ||
| FACS ARIA III | BD Biosciences | ||
| Conjugated Antibodies for FACS | |||
| Alexa Fluor 647 anti-mouse CD104 Antibody Clone: 346-11A | 123608 | 123608 | |
| APC anti-mouse CD26 (DPP-4) Antibody | H194-112 | H194-112 | |
| PE/Cy7 anti-mouse/human CD324 (E-Cadherin) Antibody | 147310 | 147310 | |
| Antibodies for Immunofluorescence | |||
| CD104 (ß-integrin 4) Clone: 346-11A | BioLegend | 553745 | |
| Cytokeratin 14 | Acris Antibodies (AbD Serotec) | BP5009 | |
| Cytokeratin13 Clone: EPR3671 | abcam | ab92551 | |
| E-cadherin (CD324) Clone: 2.40E+11 | Cell Signaling Technology | 3195 | |
| Keratin 5 Polyclonal Chicken Antibody, Purified Clone: Poly9059 | BioLegend | 905901 | |
| p63 Clone: 4a4 | abcam | ab735 | |
| Recombinant Anti-KLF4 antibod Clone: EPR20753-25 | abcam | ab214666 | |
| Vimentin | Sigma-Aldrich | AB5733 | |
| Secondary antibodies | |||
| Donkey anti-species* antibodies with fluorophore of choice | Jackson Immuno |
Referenzen
- Sachs, N., et al. Long-term expanding human airway organoids for disease modeling. The EMBO Journal. 38 (4), 100300 (2019).
- Lohmussaar, K., et al. Patient-derived organoids model cervical tissue dynamics and viral oncogenesis in cervical cancer. Cell Stem Cell. 28 (8), 1380-1396 (2021).
- Sato, T., et al. Single Lgr5 stem cells build crypt-villus structures in vitro without a mesenchymal niche. Nature. 459 (7244), 262-265 (2009).
- Smukler, S. R., et al. The adult mouse and human pancreas contain rare multipotent stem cells that express insulin. Cell Stem Cell. 8 (3), 281-293 (2011).
- Zheng, B., et al. A new murine esophageal organoid culture method and organoid-based model of esophageal squamous cell neoplasia. iScience. 24 (12), 103440 (2021).
- DeWard, A. D., Cramer, J., Lagasse, E. Cellular heterogeneity in the mouse esophagus implicates the presence of a nonquiescent epithelial stem cell population. Cell Reports. 9 (2), 701-711 (2014).
- Kasagi, Y., et al. The esophageal organoid system reveals functional interplay between notch and cytokines in reactive epithelial changes. Cellular and Molecular Gastroenterology and Hepatology. 5 (3), 333-352 (2018).
- Plikus, M. V., et al. Fibroblasts: Origins, definitions, and functions in health and disease. Cell. 184 (15), 3852-3872 (2021).
- McCarthy, N., et al. Distinct mesenchymal cell populations generate the essential intestinal BMP signaling gradient. Cell Stem Cell. 26 (3), 391-402 (2020).
- Cordero-Espinoza, L., et al. Dynamic cell contacts between periportal mesenchyme and ductal epithelium act as a rheostat for liver cell proliferation. Cell Stem Cell. 28 (11), 1907-1921 (2021).
- Pastula, A., et al. Three-dimensional gastrointestinal organoid culture in combination with nerves or fibroblasts: a method to characterize the gastrointestinal stem cell niche. Stem Cells International. 2016, 3710836 (2016).
- Hamilton, T. G., Klinghoffer, R. A., Corrin, P. D., Soriano, P. Evolutionary divergence of platelet-derived growth factor alpha receptor signaling mechanisms. Molecular and Cellular Biology. 23 (11), 4013-4025 (2003).
- McGinn, J., et al. A biomechanical switch regulates the transition towards homeostasis in oesophageal epithelium. Nature Cell Biology. 23 (5), 511-525 (2021).
- Zhang, Y., Bailey, D., Yang, P., Kim, E., Que, J. The development and stem cells of the esophagus. Development. 148 (6), (2021).
- Ohlund, D., et al. Distinct populations of inflammatory fibroblasts and myofibroblasts in pancreatic cancer. The Journal of Experimental Medicine. 214 (3), 579-596 (2017).
- Pentinmikko, N., et al. Notum produced by Paneth cells attenuates regeneration of aged intestinal epithelium. Nature. 571 (7765), 398-402 (2019).
- Janson, D., Rietveld, M., Willemze, R., El Ghalbzouri, A. Effects of serially passaged fibroblasts on dermal and epidermal morphogenesis in human skin equivalents. Biogerontology. 14 (2), 131-140 (2013).
- Landry, N. M., Rattan, S. G., Dixon, I. M. C. An improved method of maintaining primary murine cardiac fibroblasts in two-dimensional cell culture. Scientific Reports. 9 (1), 12889 (2019).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten