Method Article
Caractérisation fonctionnelle et visualisation de fibroblastes œsophagiens à l’aide de co-cultures organoïdes
Dans cet article
Résumé
Les co-cultures organoïdes-fibroblastes fournissent un modèle pour étudier la niche des cellules souches in vivo . Ici, un protocole pour les co-cultures organoïdes-fibroblastes œsophagiennes est décrit. De plus, l’imagerie de monture entière est utilisée pour visualiser l’interaction fibroblaste-organoïde.
Résumé
Les cellules souches épithéliales et progénitrices contribuent à la formation et au maintien de la barrière épithéliale tout au long de la vie. La plupart des populations de cellules souches et progénitrices sont cachées dans des endroits anatomiquement distincts, ce qui permet des interactions exclusives avec des signaux de niche qui maintiennent la souche. Alors que le développement de cultures organoïdes épithéliales fournit un outil puissant pour comprendre le rôle des cellules souches et progénitrices dans l’homéostasie et la maladie, l’interaction dans l’environnement de niche est largement absente, entravant ainsi l’identification des facteurs influençant le comportement des cellules souches. Les fibroblastes jouent un rôle clé dans la direction du destin de la tige épithéliale et des progéniteurs. Ici, un protocole complet de co-culture organoïde-fibroblaste permettant la délimitation des sous-populations de fibroblastes dans le renouvellement et la différenciation des cellules progénitrices de l’œsophage est présenté. Dans ce protocole, une méthode pour isoler à la fois les cellules épithéliales et les fibroblastes en parallèle de l’œsophage est décrite. Des stratégies distinctes de tri cellulaire activées par fluorescence pour isoler à la fois les cellules progénitrices de l’œsophage ainsi que les sous-populations de fibroblastes de souris rapporteures transgéniques ou de souris de type sauvage sont décrites. Ce protocole fournit une approche polyvalente qui peut être adaptée pour permettre l’isolement de sous-populations spécifiques de fibroblastes. L’établissement et la transmission de monocultures organoïdes épithéliales œsophagiennes sont inclus dans ce protocole, ce qui permet une comparaison directe avec le système de co-culture. En outre, une approche de nettoyage 3D permettant une analyse détaillée des images des interactions épithélio-fibroblastes est décrite. Collectivement, ce protocole décrit une méthode comparative et à débit relativement élevé pour identifier et comprendre les composants de niche des cellules souches œsophagiennes in vitro.
Introduction
Les organoïdes sont utilisés comme tests 3D in vitro pour caractériser les cellules souches et progénitrices, ainsi que pour comprendre les signaux de signalisation dérivés des composants cellulaires de la niche des cellules souches 1,2,3,4. Les organoïdes œsophagiens de souris ont été décrits pour la première fois en 2014 et plusieurs articles ont identifié des facteurs de croissance, tels que la R-Spondine (RSPO), NOGGIN et le facteur de croissance épidermique (EGF), nécessaires au maintien et au passage des organoïdes œsophagiens 5,6,7, faisant valoir que des signaux de signalisation similaires sont nécessaires pour in vivo renouvellement des cellules progénitrices. Cependant, des facteurs de croissance sont généralement ajoutés à des concentrations non physiologiques, ce qui entraîne des conditions de croissance organoïdes qui ne reflètent pas nécessairement l’environnement de signalisation in vivo.
Les fibroblastes sont des populations de cellules stromales hétérogènes qui soutiennent les propriétés des cellules progénitrices dans de nombreuses niches de cellules souches8. La combinaison de cellules progénitrices épithéliales et de fibroblastes dans la même culture organoïde permet la formation d’organoïdes à des concentrations réduites de facteurs de croissance exogènes. Des systèmes de co-culture organoïdes provenant d’épithélium intestinal et hépatique sont décrits, mais un protocole pour établir des co-cultures organoïdes-fibroblastes œsophagiennes est toujours en suspens 9,10,11.
Dans ce protocole, deux stratégies de tri cellulaire activé par fluorescence (FACS) pour les fibroblastes de l’œsophage, utilisant soit des souris transgéniques PdgfrαH2BeGFP 12 ou des souris de type sauvage avec une coloration classique par anticorps, sont décrites. Différentes sous-populations de fibroblastes peuvent être isolées à l’aide de marqueurs de surface cellulaire de choix, offrant ainsi une flexibilité au protocole. De plus, une technique d’imagerie par fluorescence 3D préservant la morphologie organoïde est utilisée pour caractériser les interactions fibroblastes-organoïdes. Le nettoyage organoïde fournit une méthode rapide pour augmenter la profondeur de pénétration de la lumière dans les organoïdes, améliorant la visualisation des connexions organoïdes-fibroblastes et permettant la récapitulation de la structure organoïde dans son intégralité. Ce protocole combine la co-culture organoïde œsophagienne avec une stratégie d’imagerie complète de montage, permettant une caractérisation fonctionnelle de l’interaction fibroblaste-organoïde.
Protocole
Les expériences sur les animaux pour cette étude ont été approuvées par Stockholms Norra djurförsöksetiska nämnd (permis éthique n° 14051-2019). Les animaux ont été logés dans des conditions exemptes d’agents pathogènes conformément aux recommandations de la Federation of European Laboratory Animal Science Association.
1. Préparation
- Décongeler les solutions mères enzymatiques utilisées pour la dissociation (voir le tableau des matériaux) sur la glace. Décongeler une partie aliquote de la matrice membranaire basale (matrice) réduite du facteur de croissance (DFG) à 4 °C.
- Préparer le milieu de culture. Utiliser le milieu décrit dans le tableau 1 pour les cultures organoïdes et les co-cultures et le préparer avant de commencer le protocole.
2. Dissection et séparation de l’épithélium œsophagien et du stroma
REMARQUE : Assurez-vous que tous les instruments utilisés pour la dissection et le traitement des tissus sont stériles. Préparer 2 mL de solution de dissociation (voir le tableau des matières) dans la solution saline équilibrée de Hanks (HBSS) pour trois œsophages.
- Utilisez une souche de souris de votre choix, telle que les souris C57BL/6J. Le développement œsophagien chez la souris se termine après le jour postnatal (p) 70, il est donc recommandé d’utiliser des souris plus âgées que p7013. Utilisez quatre ou cinq souris car elles fournissent suffisamment de matériel pour établir 8 à 10 co-cultures organoïdes.
REMARQUE: L’efficacité de formation des organoïdes diminue avec l’âge des souris. Si des sous-populations spécifiques de fibroblastes sont intéressantes, il est possible que le faible rendement en fibroblastes limite le nombre de co-cultures organoïdes qui peuvent être établies. - Utiliser des souris génétiquement modifiées (p. ex. PdgfrαH2BeGFP) ou de type sauvage (WT) pour isoler les populations de fibroblastes. Lorsque vous utilisez des souris WT, effectuez une coloration des anticorps pour isoler des sous-populations de fibroblastes spécifiques du stroma via FACS.
- Euthanasier les souris par asphyxie au CO2 . Disséquer l’œsophage à l’aide d’une pince et de ciseaux à dissection. Pour enlever tout l’œsophage, coupez l’extrémité distale de l’œsophage directement au-dessus de l’estomac et l’extrémité proximale au début de la trachée. Immergez l’œsophage dans le PBS et mettez-le sur la glace.
- Retirer mécaniquement la musculeuse externe à l’aide d’un microscope à dissection (plage de grossissement total de 8x-40x) et d’une pince. Tenez l’extrémité distale de l’œsophage disséqué à l’aide d’une paire de forceps et utilisez l’autre forceps pour saisir et tirer le muscle de l’extrémité distale à l’extrémité proximale de l’œsophage disséqué. Retirez et jetez la couche musculaire.
- Ouvrez l’œsophage longitudinalement. Cela fonctionne mieux en utilisant des ciseaux à ressort de microdissection avec une pointe de bille pour prévenir les dommages aux tissus. Tenez une extrémité de l’œsophage afin d’insérer la boule des ciseaux à ressort dans la lumière de l’œsophage et coupez l’œsophage tout en tenant l’extrémité.
- Placez l’œsophage dans un tube microcentrifuge de 1,5 mL ou une plaque de 24 puits. Incuber l’œsophage ouvert dans de la thermolysine à 0,5 mg/mL dans HBSS à 37 °C sur un agitateur à bascule pendant 15 min. Immergez complètement l’œsophage dans une solution de thermolysine.
REMARQUE: Le volume de solution de thermolysine utilisé dépend de la taille du puits ou du tube. Plusieurs œsophages peuvent être placés et immergés dans le même puits ou tube. - Retirez l’œsophage de la solution de thermolysine. Utilisez un microscope à dissection pour séparer soigneusement l’épithélium œsophagien du stroma. À l’aide de pinces fines, saisissez à la fois le côté épithélial et le côté stroma du tissu et séparez-les lentement pour séparer les deux couches.
NOTE: Le stroma est identifié par son aspect blanc et opaque, contrairement à l’épithélium transparent. Le stroma contient la lamina propria et la couche sous-muqueuse. - Transférer la couche épithéliale et stromale dans deux tubes microcentrifugeux distincts de 1,5 mL avec 200 μL de solution de dissociation dans HBSS. Mettez sur la glace.
3. Isolement des cellules progénitrices de l’œsophage
REMARQUE: L’isolement des cellules progénitrices de l’œsophage (étape 2) et des fibroblastes (étape 3) peut être effectué simultanément. Préparer un tube de 50 ml de FBS à 1 % dans HBSS (1 % FBS).
- Transférer l’épithélium œsophagien séparé du tube microcentrifuge de 1,5 ml (étape 2.8) dans une boîte de Petri propre et utiliser un scalpel tranchant pour hacher. Prélever le tissu haché de la boîte de Petri avec 200 μL de solution de dissociation et le transférer dans le tube microcentrifuge de 1,5 mL.
REMARQUE : L’épithélium est haché correctement lorsque les morceaux peuvent être replacés dans le tube microcentrifuge de 1,5 mL à l’aide d’un embout de pipette de 200 μL. - Ajouter 800 μL de solution de dissociation fraîche au tube microcentrifuge de 1,5 mL jusqu’à un volume total de 1 mL.
NOTE: Il est important d’ajouter un volume suffisant de solution de dissociation. Pour un à trois œsophages, 1 mL de solution est recommandé. Passez à l’échelle lorsque plusieurs œsophages sont traités à la fois. - Placer le tube avec la couche épithéliale émincée sur un agitateur à bascule à 37 °C pendant 60 min. Pipeter la solution de haut en bas environ 20 fois à l’aide d’un embout de pipette de 200 μL toutes les 15 minutes pour améliorer la digestion.
- Après 60 min, montez et descendez encore 20 fois à l’aide d’une pointe de pipette de 200 μL. Passez la solution à travers une crépine cellulaire de 40 μm dans un nouveau tube microcentrifuge de 1,5 mL. Centrifuger à 300 x g pendant 10 min à 4 °C.
REMARQUE: Mouillez la crépine cellulaire avec 1% FBS avant de filtrer les cellules pour minimiser l’adhérence des cellules au filtre. L’épithélium ne sera pas complètement digéré et des morceaux de tissu seront toujours visibles. Une incubation plus longue diminuera toutefois la survie des cellules et n’entraînera pas un rendement plus élevé de cellules viables. - Jeter le surnageant en retirant l’excès de liquide à l’aide d’une pipette de 1 mL et remettre la pastille en suspension dans 1 mL de FBS à 1 %. Centrifuger à 300 x g pendant 10 min à 4 °C.
- Pendant la centrifugation, préparer le mélange d’anticorps conjugués pour le FACS des cellules progénitrices de l’œsophage.
- Mélanger 1 μL de CD324-PE-Cy7 (ECADHERIN) et CD104-A647 (INTEGRIN-β4) dans 200 μL de FBS à 1 % par million de cellules.
REMARQUE : 1 μL d’anticorps (volume final de 200 μL) est suffisant pour un ou deux œsophages. Lors du traitement de plusieurs œsophages à la fois, augmentez le volume de la solution de coloration d’anticorps.
- Remettez la pastille en suspension dans 200 μL de mélange d’anticorps et transférez-la dans un tube de cytométrie en flux. Après avoir ajouté des anticorps fluorescents, gardez les suspensions cellulaires dans l’obscurité pour éviter le blanchiment du signal. Incuber les cellules pendant 30 min à 4 °C. Ajouter 3 mL de FBS à 1 % et centrifuger à 300 x g pendant 10 min à 4 °C, puis remettre les cellules en suspension dans un minimum de 200 μL de FBS à 1 %.
REMARQUE : On utilise 500 μL de FBS à 1 % pour un maximum de quatre ou cinq œsophages. Augmenter le volume lorsque plusieurs œsophages sont traités à la fois, de sorte qu’un débit FACS de 100-300 événements / s peut être atteint. Plus d’événements/s diminueront l’efficacité du tri, et une augmentation du débit diminuera la survie des cellules. - Ajouter un marqueur de coloration de cellules mortes à une concentration finale de 1:10 000, 5 minutes avant le tri FACS pour isoler les cellules vivantes. Triez les cellules progénitrices à l’aide d’une machine FACS (voir la figure 1 pour la stratégie de contrôle). Recueillir les cellules dans des tubes microcentrifugés de 1,5 mL remplis de 200 μL de milieu organoïde basique (tableau 1).
4. Isolement des fibroblastes de la couche stromale
- Couper la couche stomale en petits morceaux dans un tube de 1,5 mL contenant 200 μL de solution de dissociation (étape 2.8) à l’aide de ciseaux à dissection. Le tissu est correctement haché une fois que la solution peut être pipetée de haut en bas à l’aide d’un embout de pipette de 200 μL.
- Ajouter 800 μL de solution de dissociation fraîche au tube microcentrifuge de 1,5 mL jusqu’à un volume total de 1 mL.
NOTE: Il est important d’ajouter un volume suffisant de solution de dissociation. Pour un à trois œsophages, 1 mL de solution est recommandé. Passez à l’échelle lorsque plusieurs œsophages sont traités à la fois. - Placer le tube sur un agitateur à bascule à 37 °C pendant 30 min. Après 15 minutes, pipeter la solution de haut en bas environ 20 fois à l’aide d’un embout de pipette de 200 μL pour améliorer la digestion.
- Après 30 minutes de digestion enzymatique, pipeter de haut en bas 20 fois à l’aide d’un embout de pipette de 200 μL. Filtrer la solution à travers une crépine cellulaire de 70 μm dans un nouveau tube microcentrifuge de 1,5 mL. Centrifuger à 300 x g pendant 10 min à 4 °C.
REMARQUE: Mouillez la crépine cellulaire avec 1% FBS avant de filtrer les cellules pour minimiser l’adhérence des cellules au filtre. - Jeter le surnageant en retirant l’excès de liquide à l’aide d’une pipette de 1 mL et remettre la pastille en suspension dans 1 mL de FBS à 1 %. Centrifuger à 300 x g pendant 10 min à 4 °C.
REMARQUE: Lors de l’utilisation d’une souche de souris génétiquement modifiée contenant des fibroblastes marqués par fluorescence, les colorations d’anticorps sont facultatives. Si aucune coloration d’anticorps n’est nécessaire, passez à l’étape 3.7 et transférez l’échantillon dans un tube de cytométrie en flux.- Pendant la centrifugation, préparer le mélange d’anticorps conjugués pour l’isolement FACS des fibroblastes. Mélanger 1 μL de CD26-APC (DPP4) dans 200 μL de FBS à 1 % par million de cellules.
REMARQUE : 1 μL d’anticorps est suffisant pour un ou deux œsophages. Lors du traitement de plusieurs œsophages à la fois, augmentez le volume de la solution de coloration d’anticorps.
- Pendant la centrifugation, préparer le mélange d’anticorps conjugués pour l’isolement FACS des fibroblastes. Mélanger 1 μL de CD26-APC (DPP4) dans 200 μL de FBS à 1 % par million de cellules.
- Resuspendre la pastille dans 200 μL de mélange d’anticorps conjugués dans du FBS à 1% et transférer dans un tube de cytométrie en flux. Incuber les cellules pendant 30 min à 4 °C.
- Ajouter 3 mL de FBS à 1 % et centrifuger à 300 x g pendant 10 min à 4 °C. Remettez les cellules en suspension dans un minimum de 200 μL de 1% de FBS.
REMARQUE : On utilise 500 μL de FBS à 1 % pour un maximum de quatre ou cinq œsophages. Augmentez le volume lorsque plusieurs œsophages sont traités à la fois, de sorte qu’un débit de 100 à 300 événements/s peut être atteint. Plus d’événements/s diminueront l’efficacité du tri, et l’augmentation du débit diminuera la survie des cellules. Après l’ajout d’anticorps fluorescents, les suspensions cellulaires doivent être maintenues sombres pour éviter le blanchiment du signal. - Ajouter un marqueur de coloration de cellules mortes à une concentration finale de 1:10 000, 5 minutes avant le tri FACS pour isoler les cellules vivantes. Triez les cellules à l’aide d’une machine FACS (voir la figure 1 pour la stratégie de contrôle). Recueillir les cellules dans des tubes microcentrifugés de 1,5 mL remplis de 200 μL de milieu organoïde basique (tableau 1).
5. Etablissement et culture des organoïdes œsophagiens
NOTA : PréchaufferER bas (co-culture organoïde), milieu ENR (organoïde) (voir le tableau 1 pour la description) et une plaque de 48 puits à 37 °C. Placer la matrice décongelée (préparée à l’étape 1) sur de la glace. Il est recommandé d’utiliser la matrice fournie ici (voir le tableau des matériaux) pour la culture organoïde œsophagienne de souris, car d’autres marques de matrice affectent négativement l’efficacité de la formation organoïde.
- Pour la co-culture organoïde, mélanger les cellules épithéliales triées et les fibroblastes dans un rapport de 1:2 dans un tube. Pour chaque dôme matriciel, utilisez 5 000 cellules épithéliales et 10 000 fibroblastes. Lorsque vous vous préparez à plus de dômes, ajoutez un multiple de 5 000 cellules épithéliales et 10 000 fibroblastes dans un tube. Pour les cultures organoïdes sans fibroblastes, utilisez 5 000 cellules épithéliales par dôme matriciel.
- Centrifuger la population de cellules mixtes à 300 x g pendant 5 min. Jetez le surnageant en le retirant délicatement à l’aide d’une pipette de 200 μL.
- Laver les cellules une fois en remettant la pastille en suspension dans un milieu organoïde basique froid et centrifuger à 300 x g pendant 5 min. Placez les cellules sur de la glace.
- Préparer un mélange composé de 80% de matrice et de 20% de milieu organoïde basique froid. Placez le tout sur la glace pendant que la matrice se solidifie à température ambiante (RT).
- Jeter le surnageant après centrifugation en le retirant délicatement à l’aide d’une pipette de 200 μL. Remettez les cellules en suspension dans 10 μL de mélange matriciel/dôme et remettez-les sur de la glace.
- Prenez la plaque préchauffée de 48 puits de l’incubateur à 37 °C et fabriquez un dôme matriciel par puits à l’aide d’une pipette de 20 μL. Chaque dôme contient 10 μL de matrice à 80%, 5 000 cellules épithéliales et 10 000 fibroblastes. Transférer la plaque à l’envers dans un incubateur et laisser les dômes se solidifier pendant encore 20-30 minutes à 37 ° C.
REMARQUE: Une diminution du volume du dôme de la matrice et / ou une augmentation du nombre de cellules affectera l’efficacité de formation des organoïdes. - Ajouter 200 μL de milieu ERfaible préchauffé (tableau 1) aux dômes matriciels contenant des co-cultures organoïdes et un milieu ENR (tableau 1) aux dômes matriciels respectifs contenant uniquement des organoïdes épithéliaux et placer la plaque dans l’incubateur.
- Cultiver les organoïdes à 37 °C et 5% de CO2. Pendant les 2 premiers jours, compléter le milieu avec un inhibiteur de roche de 10 μM (Y-27623). L’inhibiteur de roche empêche la mort cellulaire induite par le stress et augmente les chances d’une mise en place réussie de cultures organoïdes.
- Rafraîchir le milieu tous les 2-3 jours. Assurez-vous que le milieu est chaud pour éviter la dissociation de la matrice sensible à la température.
- Effectuer des analyses de l’expérience 6 à 8 jours après le placage. Les organoïdes peuvent être conservés en culture jusqu’à 14 jours. Vers le jour 14, une perte d’intégrité du dôme est observée.
6. Passage des organoïdes
NOTE: La transmission d’organoïdes cultivés en co-culture entraîne la perte de fibroblastes. Par conséquent, il est recommandé d’utiliser le milieu ENR pour tous les organoïdes lors du passage. Préchauffer ENR, PBS et une plaque de 48 puits à 37 °C.
- Retirer le milieu et laver le puits contenant le dôme de matrice avec du PBS préchauffé. Ajouter 200 μL de solution froide de trypsine à 0,25% et pipeter de haut en bas pour casser le dôme matriciel.
REMARQUE: L’utilisation de trypsine froide à 0,25% est recommandée, car la matrice est sensible à la température, ce qui aide à décomposer les dômes de la matrice. - Incuber les organoïdes avec de la trypsine à 37 °C pendant ~20 min. Pipeter de haut en bas après 10 min pour augmenter la dissociation des organoïdes. Surveillez le processus de dissociation avec un microscope toutes les 5-10 minutes. Puisque la trypsine diminue la viabilité cellulaire, cela peut aider à identifier le temps de dissociation idéal.
- Après 20 min, pipeter les organoïdes de haut en bas avec une pipette de 200 μL pour dissocier les organoïdes. Recueillir les cellules dans un tube microcentrifuge de 1,5 mL, ajouter 1 mL de milieu organoïde basique et centrifuger à 300 x g pendant 5 min à TA.
- Facultatif : Pour garantir les meilleures conditions de croissance des organoïdes et que les nouveaux organoïdes sont dérivés de cellules individuelles, filtrer les cellules à l’aide d’une passoire cellulaire pré-mouillée de 40 μm. La filtration de la suspension cellulaire entraîne l’élimination des noyaux organoïdes et des amas cellulaires difficiles à dissocier.
- Préparer un mélange matriciel composé de 80 % de milieu organoïde basique à matrice et de 20 % de milieu organoïde basique froid. Placez le tout sur la glace, car la matrice se solidifie à RT.
- Jeter le surnageant en le retirant délicatement avec une pipette de 200 μL, remettre les cellules en suspension dans 10 μL de mélange matriciel/dôme et remettre le mélange sur de la glace.
REMARQUE: Les organoïdes peuvent être divisés à un rapport de 1: 5 à 1: 10, en fonction de la densité du dôme. Les cellules épithéliales peuvent également être comptées et replaquées à 5 000 cellules / dôme. - Prenez la plaque préchauffée de 48 puits de l’incubateur à 37 °C et faites un dôme par puits. Transférer la plaque à l’envers dans un incubateur et laisser les dômes se solidifier pendant encore 20-30 minutes à 37 ° C.
- Ajouter 200 μL de milieu ENR préchauffé aux dômes organoïdes respectifs. Compléter le milieu avec un inhibiteur de roche de 10 μM (Y-27623) pendant les 2 premiers jours.
- Rafraîchir le milieu tous les 2-3 jours. Assurez-vous que le milieu est chaud pour éviter la dissociation de la matrice sensible à la température.
7. Traitement organoïde pour la coloration de montage entier
REMARQUE: Enduisez les embouts et les tubes de 10% FBS de PBS avant utilisation pour éviter que les organoïdes n’adhèrent aux plastiques. Pour les pointes de pipette, il suffit de pipeter une ou deux fois de haut en bas dans une solution FBS/PBS à 10% avant d’utiliser la pointe. Pour les tubes, remplissez le tube avec 10% FBS / PBS, puis retirez la solution.
- Retirez le milieu organoïde et ajoutez 200 μL de PBS glacé aux dômes de la matrice. Placez l’assiette sur de la glace pendant 5-10 min.
- Pipeter de haut en bas et transférer la solution dans des tubes non adhérents microcentrifuges de 0,6 mL. Centrifuger brièvement pendant 30-60 s à 100 x g pour laisser les organoïdes se déposer dans le fond.
NOTE: Un pipetage excessif et une longue centrifugation cassent les organoïdes et perturbent l’interaction fibroblaste-organoïde. - Retirez l’excès de liquide et ajoutez du PBS glacé. Centrifuger brièvement pendant 30-60 s à 100 x g pour laisser les organoïdes se déposer dans le fond.
- Retirer l’excès de liquide et fixer l’organoïde avec 200 μL de formaldéhyde froid à 4% dans une solution de PBS pendant 30 minutes sur glace.
ATTENTION : Le formaldéhyde est toxique et doit toujours être utilisé dans une hotte chimique. Des gants en nitrile, des lunettes de sécurité et des blouses de laboratoire doivent toujours être portés. - La fixation de l’organoïde les fait couler et la centrifugation n’est plus nécessaire. Laissez les organoïdes couler en plaçant le tube à la verticale et attendez 2-3 minutes, retirez le formaldéhyde et ajoutez 500 μL de PBS froid pour éliminer le formaldéhyde.
- Laissez couler les organoïdes, retirez l’excès de PBS et ajoutez 500 μL de PBS frais froid. Laissez couler les organoïdes, retirez l’excès de PBS et ajoutez 500 μL de tampon bloquant (5% de sérum d’âne normal, 1% de BSA et 0,5% de Triton X-100 dans PBS). Mettez le tube sur un rocker-shaker pendant 60 min à RT.
REMARQUE: Le blocage peut également être effectué pendant la nuit (O/N) à 4 °C. - Laisser couler les organoïdes, retirer le tampon de blocage et remettre les organoïdes en suspension dans 200 μL de tampon bloquant avec des anticorps primaires (voir le tableau des matières). Maintenir les organoïdes sur un rocker-shaker O/N à 4 °C.
NOTE: Pour améliorer la coloration des protéines nucléaires, des protéines faiblement exprimées ou des anticorps qui présentent une coloration non spécifique, le temps d’incubation peut être augmenté pendant 1 ou 2 jours à 4 ° C. Placer les organoïdes sur un agitateur à bascule est essentiel car cela empêche les organoïdes de s’agglutiner et augmente l’efficacité de la coloration. - Laisser couler les organoïdes, retirer le mélange d’anticorps primaires et laver les organoïdes en utilisant 500 μL de Triton X-100 à 0,02% dans du PBS (0,02% Tx) pendant 60 minutes à TA. Répéter trois fois.
REMARQUE: Lorsque l’incubation d’anticorps primaires est plus longue, ajouter une étape de lavage avec 0,02% Tx dans PBS O / N à 4 ° C. - Laisser couler les organoïdes, retirer le tampon de lavage et ajouter 200 μL d’anticorps secondaire conjugué à la fluorescence dans le tampon de blocage O/N à 4 °C. Après avoir ajouté des anticorps secondaires fluorescents, gardez les organoïdes dans l’obscurité pour éviter le blanchiment du signal.
- Laisser couler les organoïdes, retirer le mélange d’anticorps secondaires et laver les organoïdes en utilisant 500 μL de Tx à 0,02% pendant 60 minutes à TA. Répéter trois fois.
- Contre-colorer les organoïdes avec 200 μL de solution DAPI (0,25 μg/mL) pour la coloration nucléaire si nécessaire. Incuber les échantillons pendant 60 min à TA sur un rocker-shaker.
- Laisser couler les organoïdes, retirer la solution DAPI et laver les organoïdes dans 500 μL de PBS pendant 15 min à TA. Répéter trois fois.
- Laissez les organoïdes couler et retirez tout excès de liquide. Ajouter 10 μL de solution de clairage aux organoïdes et incuber pendant 15 min à TA.
REMARQUE: La solution de nettoyage est visqueuse, coupez donc la partie extérieure d’une pointe de pipette de 20 μL avant de pipeter la solution de nettoyage. Une solution de nettoyage supplémentaire peut être ajoutée si une entretoise plus grande est utilisée pour l’imagerie. La solution de montage peut être utilisée à la place de la solution de nettoyage lorsque les organoïdes ne sont pas nettoyés. La solution de nettoyage réduit l’arrière-plan lors de l’imagerie et augmente la profondeur d’imagerie. - Placez une entretoise collante à 4 puits double face de 0,05 mm sur une lame de microscope. Ajouter les 10 μL de solution de nettoyage avec les organoïdes dans un puits et placer un couvercle sur le dessus de l’entretoise.
REMARQUE: Les organoïdes peuvent également être montés sans l’utilisation d’une entretoise. L’entretoise maintient la forme organoïde intacte et empêche les organoïdes d’être aplatis. - Gardez la lame à RT O/N pour effacer les organoïdes. Pour un stockage à long terme, maintenez les lames à 4 °C. Acquérir des images à l’aide d’un système de microscopie confocale.
Résultats
L’œsophage est divisé en différentes couches : épithélium, lamina propria, sous-muqueuse et musculeuse externe (figure 1A). Les fibroblastes résident dans la sous-muqueuse et la lamina propria, appelée stroma. Dans ce protocole, la musculeuse externe est enlevée mécaniquement (Figure 1B), ce qui n’entraîne pas de perte de fibroblastes (PdgfrαH2BeGFP+) résidant dans le stroma (Figure 1C). Avant la dissociation, l’épithélium est séparé du stroma, ce qui donne deux segments tissulaires (Figure 1B). La séparation des deux couches offre la possibilité d’augmenter le temps de dissociation de la couche épithéliale plus robuste par rapport à la couche stromale fragile. De cette façon, un protocole d’isolement efficace produisant à la fois des cellules progénitrices épithéliales viables ainsi que des fibroblastes stromaux est établi (Figure 1B). Les cellules progénitrices œsophagiennes sont triées en fonction de leur expression élevée en INTEGRIN-β4 et E-CADHERIN (Figure 1C,D).
Les sous-populations de fibroblastes peuvent être isolées à l’aide de marqueurs distincts. Dans ce protocole, une stratégie d’isolement des fibroblastes basée sur les marqueurs de fibroblastes couramment utilisés PDGFRα et DPP4 (CD26) est fournie. L’isolement par l’expression du rapporteur PdgfrαH2BeGFP ou l’anticorps DPP4 montre qu’environ 50% des cellules isolées sont des fibroblastes (Figure 1E,F). De plus, 70 % des fibroblastes PDGFRα+ sont DPP4+, ce qui indique qu’une population de fibroblastes qui se chevauche largement, mais pas identique, est obtenue. Après avoir isolé les populations de cellules épithéliales et stromales, les cellules progénitrices de l’œsophage sont cultivées seules ou avec des fibroblastes dans un dôme matriciel. Pour étudier la contribution des fibroblastes à la formation d’organoïdes, la co-culture est maintenue dans un milieu de croissance réduit (Figure 1G).
Les cellules progénitrices œsophagiennes forment des organoïdes en présence d’EGF, NOGGIN et RSPO (ENR). Élimination de NOGGIN et réduction de la quantité de RSPO (25 ng/μL; ERfaible) est suffisant pour empêcher la formation d’organoïdes (figure 2A). Fait intéressant, l’ajout de fibroblastes DPP4+ ou PDGFRα+ aux cellules progénitrices œsophagiennes dans le milieufaible ER restaure la capacité de formation d’organoïdes, démontrant une fonction de soutien pour les deux populations de fibroblastes (Figure 2A-D). La visualisation du transgène PdgfrαH2BeGFP montre que les fibroblastes sont en contact étroit avec les cellules progénitrices épithéliales lors de la formation organoïde (Figure 2A). Au jour 6, les fibroblastes PdgfrαH2BeGFP+ sont encore abondamment présents dans la co-culture. Les fibroblastes sont présents dans tout le dôme, près et en contact avec les organoïdes (flèche pleine), ou attachés aux organoïdes (pointe de flèche; Figure 2D).
La coloration de l’ensemble du support montre une représentation 3D de l’interaction des fibroblastes avec les organoïdes (Figure 3). Bien qu’il soit possible d’effectuer l’ensemble du protocole de montage sans utiliser de solution de nettoyage, cela diminue la transparence et la pénétration laser de l’organoïde (Figure 3B, z-view). Lors du montage d’organoïdes, l’entretoise aide à maintenir la morphologie organoïde. En revanche, le placage de la lamelle de couverture directement sur les organoïdes (sans entretoise) aplatit les organoïdes et entraîne une perte de structure organoïde (Figure 3A,B).
Les fibroblastes DPP4+ et PDGFRα+ sont enroulés autour des organoïdes (Figure 3C, Vidéo1 et Vidéo 2). La différenciation des organoïdes œsophagiens peut être évaluée à l’aide de différents marqueurs. La figure 4 montre que le protocole de coloration fourni convient aux kératines plus faciles à colorer (KRT14/13) ainsi qu’aux facteurs de transcription plus difficiles à colorer (TRP63/KLF4). Le protocole de co-culture génère des organoïdes avec un schéma de différenciation similaire, comme démontré in vivo13,14 et comme on l’a vu dans les organoïdes cultivés en milieu ENR ; Les cellules progénitrices KRT14+ ou TRP63+ forment la couche externe et les cellules différenciées KRT13+ ou KLF4+ s’orientent vers l’intérieur.
Ce protocole fournit un outil pour étudier la niche des cellules souches œsophagiennes in vitro et visualise l’interaction entre les organoïdes et les fibroblastes. En mettant en œuvre un protocole pour l’isolement des fibroblastes à l’aide d’anticorps, la méthode est adaptable et peut être utilisée pour étudier les sous-populations de fibroblastes sans avoir besoin de souris transgéniques.
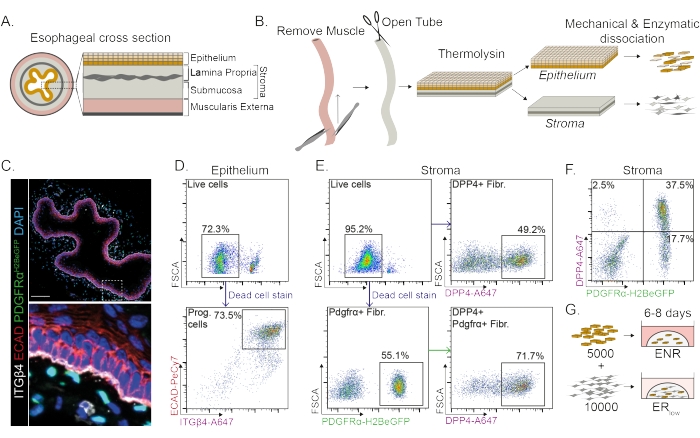
Figure 1 : Isolement des cellules progénitrices et des sous-populations de fibroblastes de l’œsophage. (A) Vue d’ensemble schématique des différentes couches de l’œsophage. Le stroma contient la lamina propria et la sous-muqueuse. B) Vue d’ensemble schématique du protocole d’isolement. Le muscle (musculeux externe) est enlevé mécaniquement à l’aide d’une pince; L’œsophage restant est ouvert et incubé dans de la thermolysine pour séparer la couche épithéliale du stroma. L’épithélium et le stroma sont séparés, hachés mécaniquement et digérés enzymatiquement en suspensions unicellulaires. Les cellules dissociées sont ensuite colorées et préparées pour la FACS. (C) Coupe transversale de l’œsophage arrachée de la musculeuse externe montrant des fibroblastes PdgfrαH2BeGFP+ dans le stroma. Les cellules doubles positives de l’intégrine-β4 (ITGβ4) et de la e-cadhérine (ECAD) sont les cellules progénitrices épithéliales de l’œsophage. Barre d’échelle = 100 μm. (D) Diagramme représentatif de cytométrie en flux de l’isolement des cellules épithéliales montrant le pourcentage de cellules vivantes (panneau supérieur) de toutes les cellules individuelles. Le panneau inférieur montre le pourcentage de cellules progénitrices ITGβ4+ ECAD+ (Prog.) isolées de toutes les cellules vivantes. (E) Diagramme représentatif de cytométrie en flux de l’isolement des cellules stromales montrant le pourcentage de cellules vivantes (panneau supérieur gauche). Graphiques représentatifs de cytométrie en flux montrant le pourcentage de fibroblastes DPP4+ isolés (Fibr.; panneau supérieur droit) et de fibroblastes Pdgfrα+ (panneau inférieur gauche) de toutes les cellules vivantes. 70% des fibroblastes Pdgfrα+ sont également DPP4+ (panneau inférieur droit). (F) Tracé représentatif de la cytométrie en flux du stroma montrant les cellules DPP4+ seulement (2,5%), les cellules DPP4+ PDGFRα+ (37,5%) et les cellules PDGFRα+ seules (17,7%). Les pourcentages sont de toutes les cellules vivantes. (G) Les cellules épithéliales seules sont plaquées dans un dôme matriciel en présence de 50 ng/μL d’EGF, 100 ng/μL de NOGGIN et 250 ng/μL RSPO (ENR), ou avec des fibroblastes en présence d’EGF et d’une faible concentration de RSPO (25 ng/μL). Veuillez cliquer ici pour voir une version agrandie de cette figure.
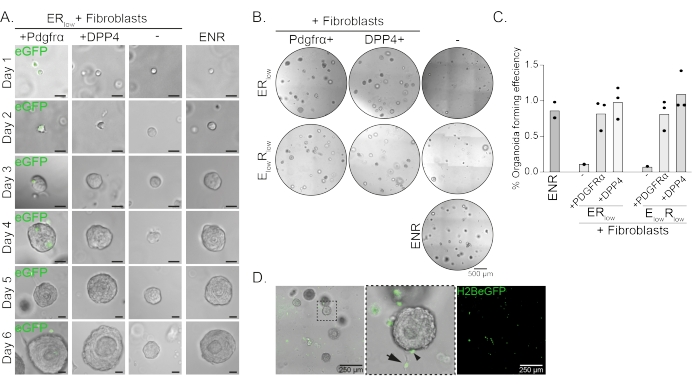
Figure 2 : Résultats représentatifs des co-cultures organoïdes. (A) Images en fond clair montrant la croissance des organoïdes du jour 1 au jour 6. Les images en fond clair avec les organoïdes co-cultivés avec les fibroblastes PdgfrαH2BeGFP+ montrent également le signal eGFP nucléaire. Barre d’échelle = 25 μm. (B) Images en fond clair de l’ensemble du dôme de la matrice au jour 6. La colonne de gauche montre des co-cultures organoïdes cultivées en présence de fibroblastes Pdgfrα+ dans un milieu ER faibleou Efaible Rbas. La colonne du milieu montre les co-cultures organoïdes cultivées en présence de fibroblastes DPP4+ dans un milieu ER faibleou Efaible Rbas. La colonne de droite montre les monocultures organoïdes cultivées en milieu ENR. milieu ENR = EGF (50 ng/μL), NOGGIN (100 ng/μL) et RSPO (250 ng/μL). ERfaible = EGF et 25 ng/μL RSPO. E faibleRfaible = 5 ng/μL EGF et 25 ng/μL RSPO. Barre d’échelle = 500 μm. (C) Graphique montrant l’efficacité de formation des organoïdes (%) (c.-à-d. le pourcentage de cellules formant des organoïdes dans différentes conditions de culture). Chaque point représente un dôme matriciel et la barre représente la moyenne de tous les points par condition. (D) Image en fond clair et fluorescente des organoïdes du jour 6 co-cultivés avec des fibroblastes PdgfrαH2BeGFP+. Les fibroblastes PdgfrαH2BeGFP+ sont présents dans tout le dôme, attachés aux organoïdes (pointe de flèche) et non attachés mais en contact avec les organoïdes (flèche pleine). Barre d’échelle = 250 μm. Veuillez cliquer ici pour voir une version agrandie de cette figure.
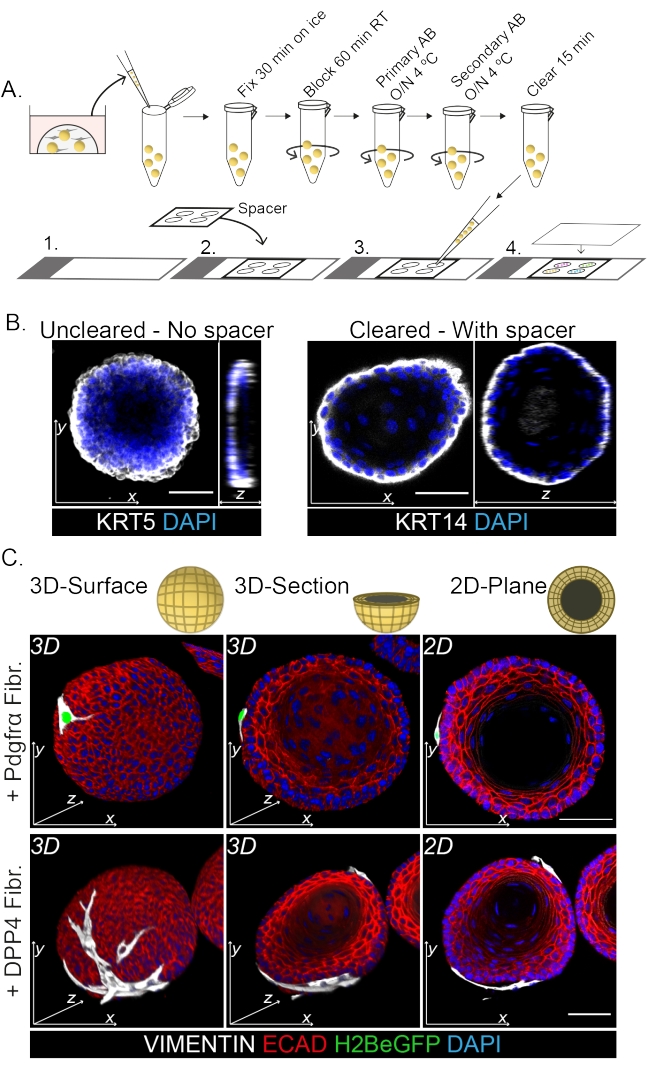
Figure 3 : Protocole de coloration du montage entier pour l’étude des interactions fibroblastes-organoïdes. (A) Vue d’ensemble schématique de l’ensemble du protocole d’immunofluorescence du monture. AB = anticorps. (B) Image d’immunofluorescence de la coloration de monture entière non effacée montrant une diminution de la transparence et de la pénétration de la lumière laser par rapport aux organoïdes effacés. L’absence d’espaceur entraîne l’aplatissement de l’organoïde et la perte de morphologie organoïde. (C) Des images de montage entier des organoïdes en co-culture montrent les surfaces 3D des organoïdes avec des fibroblastes VIMENTIN+ (Fibr.) enroulés autour et en contact étroit avec l’organoïde. Les coupes transversales 3D et les images planes 2D montrent la lumière de l’organoïde. Barre d’échelle = 50 μm. Veuillez cliquer ici pour voir une version agrandie de cette figure.
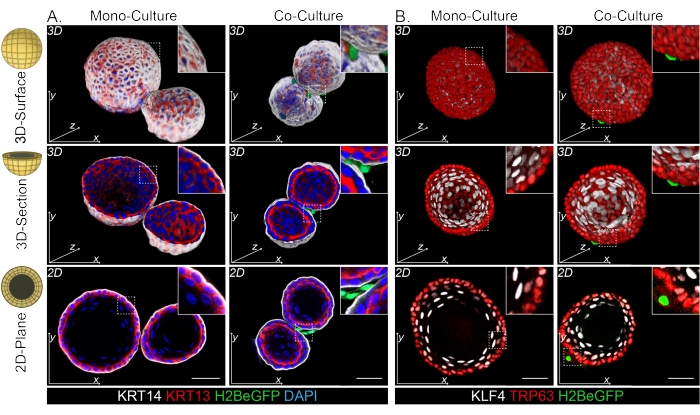
Figure 4 : Les images de monture entière révèlent des populations distinctes de cellules basales et suprabasales. (A) Coloration en monture entière d’organoïdes mono et co-cultivés avec des fibroblastes PdgfrαH2BeGFP+ montrant des cellules basales KRT14+ dans la couche externe et des cellules suprabasales différenciées KRT13+. Barre d’échelle = 50 μm. (B) Coloration sur montage entier d’organoïdes mono et co-cultivés avec des fibroblastes PdgfrαH2BeGFP+ montrant des cellules basales TRP63+ dans la couche externe et des cellules suprabasales différenciées KLF4+. Barre d’échelle = 50 μm. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Tableau 1 : Tableau décrivant les composants des milieux de culture organoïdes. Veuillez cliquer ici pour télécharger ce tableau.
Vidéo 1: PdgfrαH2BeGFP+ fibroblaste enroulé autour et en contact étroit avec l’organoïde. La vidéo accompagne le panneau supérieur de la figure 3C. La barre d’échelle de la figure 3C est de 50 μm et l’organoïde a un diamètre de ~120 μm. VIMENTIN est représenté en blanc, E-CADHERIN en rouge, PdgfrαH2BeGFP en vert et DAPI en bleu. Veuillez cliquer ici pour télécharger cette vidéo.
Vidéo 2 : Fibroblaste DPP4+ enroulé autour et en contact étroit avec l’organoïde. La vidéo accompagne le panneau inférieur de la figure 3C. La barre d’échelle de la figure 3C est de 50 μm et l’organoïde a un diamètre de ~120 μm. VIMENTIN est représenté en blanc, E-CADHERIN en rouge et DAPI en bleu. Veuillez cliquer ici pour télécharger cette vidéo.
Discussion
Le protocole présenté ici établit un modèle in vitro pour étudier les interactions fonctionnelles entre l’épithélium et les fibroblastes de l’œsophage.
La couche épithéliale est séparée du stroma, ce qui permet un protocole de dissociation optimisé pour le compartiment épithélial et stromal. Malgré l’optimisation du protocole de dissociation épithéliale, des amas tissulaires restent apparents. Pipeter de haut en bas vigoureusement toutes les 15 minutes diminue considérablement le nombre et la taille des touffes. D’autres protocoles utilisent la trypsine pour dissocier davantage la couche épithéliale 5,6. Ici, l’utilisation de trypsine, ou l’augmentation du temps de dissociation, n’est pas recommandée, car cela tend à entraîner une diminution de la viabilité des cellules épithéliales et de l’efficacité de la formation organoïde. Contrairement à l’épithélium, le stroma est facilement dissociable, et 30 min en solution de dissociation donnent une suspension unicellulaire avec ~90% de viabilité des fibroblastes (Figure 1E). L’exclusion de l’étape de séparation épithéliale-stomale dans le protocole augmente considérablement le temps de dissociation, ce qui entraîne une diminution de la viabilité des fibroblastes et un rendement plus faible des cellules épithéliales. De plus, la séparation de l’épithélium du stroma permet de déterminer le nombre de cellules de chaque population et de mélanger les cellules épithéliales et les fibroblastes de différentes lignées de souris lors de la mise en place des co-cultures.
L’étude de la fonction des fibroblastes sur la croissance organoïde est une méthode couramment utilisée en biologie des cellules souches 9,10,11,15,16. Les milieux de coculture établis sont soit du DMEM complété par 10% de sérum de veau fœtal (FCS)9,15 ou un milieu réduit en facteur de croissance10,16. Dans ce protocole, le milieu réduit du facteur de croissance est utilisé pour imiter les conditions dans la niche des cellules souches in vivo, où les fibroblastes sont en grande partie au repos. Le FCS est un sérum riche en facteur de croissance qui entraîne l’activation et la prolifération des fibroblastes dans les co-cultures, correspondant probablement à un état cellulaire de fibroblastes distinct de l’état in vivo. En excluant le FCS et en réduisant les facteurs de croissance, de sorte que le milieu seul (ERfaible) ne favorise pas la croissance organoïde et ne stimule pas la prolifération des fibroblastes, il est possible d’isoler l’effet des fibroblastes sur la croissance organoïde. Dans ce milieu, NOGGIN est supprimé et RSPO réduit au minimum (10% RPSO). Il a été démontré que NOGGIN et RSPO sont essentiels à la croissance des organoïdes œsophagiens6. L’EGF a été retenu dans le milieu de coculture, car il ne supporte pas la croissance organoïde par lui-même. Cependant, les fibroblastes sont également capables de soutenir la croissance organoïde dans un milieu réduit en EGF (E faible Rfaible; Figure 2B, D).
Les co-cultures organoïdes ne peuvent pas être maintenues par passage car les fibroblastes sont perdus lors de la trypsinisation. Cependant, le passage organoïde a été inclus dans le protocole puisque les organoïdes œsophagiens peuvent être maintenus, étendus et utilisés pour d’autres expériences en mono-cultures. Les organoïdes passés issus de monocultures peuvent être utilisés pour mettre en place des co-cultures avec des fibroblastes fraîchement isolés. Un inconvénient de l’utilisation de cellules primaires est le nombre de souris nécessaires pour mettre en place plusieurs co-cultures organoïdes. Lorsque l’on se concentre sur de petites sous-populations de fibroblastes, le nombre de co-cultures obtenues est limité. Dans d’autres protocoles, les fibroblastes sont d’abord développés en culture avant de les utiliser pour mettre en place des co-cultures organoïdes10. Cependant, les fibroblastes changent de morphologie et d’identité au cours du passage, comme le montrent l’utilisation de fibroblastes primitifs de la peau et du cœur17,18. La transmission 2D conventionnelle des fibroblastes œsophagiens entraîne à la fois des changements de morphologie et de phénotype, démontrant que l’enrichissement in vitro des fibroblastes ne convient pas aux co-cultures visant à phénocopier la niche des cellules souches endogènes.
La coloration de montage entier fournit un outil pour maintenir et visualiser l’interaction fibroblaste-organoïde. Il convient de noter que, bien que tous les organoïdes n’aient pas de fibroblastes directement attachés, la plupart des organoïdes sont en contact avec des fibroblastes (voir la figure 2C). Pour maintenir les interactions épithéliale-fibroblaste, il est important de manipuler les organoïdes avec soin et d’éviter le pipetage vigoureux, le vortex et la rotation à grande vitesse. Une fixation optimale est importante pour maintenir l’architecture tissulaire 3D, ainsi que pour maintenir la fluorescence endogène. Une fixation de 30 minutes suffit à retenir le signal H2BeGFP et est optimale pour les anticorps utilisés dans ce protocole, mais cela peut varier entre les fluorophores et les anticorps utilisés. Le nettoyage des organoïdes réduit la diffusion de la lumière et améliore considérablement la visualisation de l’ensemble de la structure 3D. Comme les organoïdes sont petits, le défrichage est facile et rapide; cependant, l’imagerie d’organoïdes entiers à l’aide de la microscopie confocale à balayage laser peut prendre beaucoup de temps, car plusieurs piles Z doivent être fabriquées. Les microscopes confocaux, comme le disque rotatif, peuvent être utilisés pour réduire le temps d’imagerie.
Dans l’ensemble, les organoïdes œsophagiens cultivés en présence de fibroblastes constituent un outil précieux pour comprendre certains aspects de la niche des cellules souches œsophagiennes. De plus, le nettoyage de monture entière fournit une méthode accessible pour visualiser l’interaction entre les fibroblastes et les organoïdes.
Déclarations de divulgation
Les auteurs ne déclarent aucun conflit d’intérêts.
Remerciements
Cette étude a été soutenue par ERC StG TroyCAN (851241). E.E. est un associé postdoctoral de Cancerfonden. M.G. est boursier Ragnar Söderberg et chercheur junior Cancerfonden. Nous sommes reconnaissants de l’assistance technique des installations principales du Karolinska Institutet, y compris l’installation centrale de cytométrie en flux Biomedicum, la centrale d’imagerie Biomedicum (BIC) et l’installation vétérinaire Comparative Medicine Biomedicum (KMB). Nous remercions les membres du laboratoire Genander d’avoir lu attentivement et commenté le protocole.
matériels
| Name | Company | Catalog Number | Comments |
| B-27 Supplement (50X), serum free | Thermo Fisher (Gibco) | 17504001 | |
| Corning Matrigel Growth Factor Reduced (GFR) Basement Membrane Matrix | fisher scientific | 356231 | |
| Dimethyl sulfoxide | Sigma-Aldrich | 276855-100ML | |
| DMEM/F-12 | Thermo Fisher (Gibco) | 11320074 | |
| DPBS | Thermo Fisher (Gibco) | 14190250 | |
| Fetal Bovine Serum | Sigma-Aldrich | F7524 | |
| GlutaMAX Supplement | Thermo Fisher (Gibco) | 35050061 | |
| HBSS, no calcium, no magnesium, no phenol red | Thermo Fisher (Gibco) | 14175-129 | |
| Normal Donkey Serum | Jackson Immuno | 017-000-121 | |
| Penicillin-Streptomycin (10,000 U/mL) | Thermo Fisher (Gibco) | 15140122 | |
| Triton X-100 solution | Merck | 93443-100ML | |
| Trypsin-EDTA (0.25%), phenol red | Thermo Fisher (Gibco) | 25200-056 | |
| Chemicals, Peptides, and recombinant proteins | |||
| DAPI Solution | Thermo Fisher | 62248 | |
| Dissociation solution: 0.25 mg/ml Liberase TM, 0.25 mg/ml Dnase in HBSS | |||
| Dnase I | Sigma-Aldrich | 11284932001 | |
| Formaldehyde, 37%, with 10-15% methanol | Sigma-Aldrich | 252549-1L | |
| Liberase | Sigma-Aldrich | 5401127001 | |
| N-Acetyl-cysteine | Sigma-Aldrich | A9165-25G | |
| Noggin murine | Peprotech | 250-38 | |
| RapiClear 1.47 | SunJin Lab | #RC147001 | |
| Recombinant Mouse EGF Protein, CF | R&D systems | 2028-EG-200 | |
| R-spondin-1 murine | Peprotech | 315-32 | |
| SYTOX Blue Dead Cell Stain | Thermo Fisher | S34857 | |
| Thermolysin | Sigma-Aldrich | T7902-25MG | |
| Y-27632 dihydrochloride | Sigma-Aldrich | Y0503-5MG | |
| Plastic & Glassware | |||
| Corning Sterile Cell Strainers 40um | VWR | 15360801 | |
| Corning Sterile Cell Strainers 70um | VWR | 431751 | |
| Menzel Deckgläser/ cover slips | Thermo Fisher | Q10143263NR15 | |
| SafeSeal reaction tube, 1.5 mL, PP | Sarstedt | 72.706 | |
| Snap Cap Low Retention Microcentrifuge Tubes 0.6 mL | Thermo Fisher | 3446 | |
| SuperFrost Slides | VWR | 631-9483 | |
| Tools | |||
| 0.05 mm 4 circular well iSpacer | SunJin Lab | #IS204 | |
| Dumont #5 forceps, biology tip | F.S.T | 11252-20 | |
| ImmEdge Pen | VectorLaboratories | H-4000 | |
| Spring Scissors Angled to Side Ball Tip 8mm Cutting Edge | F.S.T | 15033-09 | |
| Instruments | |||
| Confocal microscope Stellaris 5 | Leica | ||
| Dissection microscope ZEISS Stemi 305 | Zeiss | ||
| FACS ARIA III | BD Biosciences | ||
| Conjugated Antibodies for FACS | |||
| Alexa Fluor 647 anti-mouse CD104 Antibody Clone: 346-11A | 123608 | 123608 | |
| APC anti-mouse CD26 (DPP-4) Antibody | H194-112 | H194-112 | |
| PE/Cy7 anti-mouse/human CD324 (E-Cadherin) Antibody | 147310 | 147310 | |
| Antibodies for Immunofluorescence | |||
| CD104 (ß-integrin 4) Clone: 346-11A | BioLegend | 553745 | |
| Cytokeratin 14 | Acris Antibodies (AbD Serotec) | BP5009 | |
| Cytokeratin13 Clone: EPR3671 | abcam | ab92551 | |
| E-cadherin (CD324) Clone: 2.40E+11 | Cell Signaling Technology | 3195 | |
| Keratin 5 Polyclonal Chicken Antibody, Purified Clone: Poly9059 | BioLegend | 905901 | |
| p63 Clone: 4a4 | abcam | ab735 | |
| Recombinant Anti-KLF4 antibod Clone: EPR20753-25 | abcam | ab214666 | |
| Vimentin | Sigma-Aldrich | AB5733 | |
| Secondary antibodies | |||
| Donkey anti-species* antibodies with fluorophore of choice | Jackson Immuno |
Références
- Sachs, N., et al. Long-term expanding human airway organoids for disease modeling. The EMBO Journal. 38 (4), 100300 (2019).
- Lohmussaar, K., et al. Patient-derived organoids model cervical tissue dynamics and viral oncogenesis in cervical cancer. Cell Stem Cell. 28 (8), 1380-1396 (2021).
- Sato, T., et al. Single Lgr5 stem cells build crypt-villus structures in vitro without a mesenchymal niche. Nature. 459 (7244), 262-265 (2009).
- Smukler, S. R., et al. The adult mouse and human pancreas contain rare multipotent stem cells that express insulin. Cell Stem Cell. 8 (3), 281-293 (2011).
- Zheng, B., et al. A new murine esophageal organoid culture method and organoid-based model of esophageal squamous cell neoplasia. iScience. 24 (12), 103440 (2021).
- DeWard, A. D., Cramer, J., Lagasse, E. Cellular heterogeneity in the mouse esophagus implicates the presence of a nonquiescent epithelial stem cell population. Cell Reports. 9 (2), 701-711 (2014).
- Kasagi, Y., et al. The esophageal organoid system reveals functional interplay between notch and cytokines in reactive epithelial changes. Cellular and Molecular Gastroenterology and Hepatology. 5 (3), 333-352 (2018).
- Plikus, M. V., et al. Fibroblasts: Origins, definitions, and functions in health and disease. Cell. 184 (15), 3852-3872 (2021).
- McCarthy, N., et al. Distinct mesenchymal cell populations generate the essential intestinal BMP signaling gradient. Cell Stem Cell. 26 (3), 391-402 (2020).
- Cordero-Espinoza, L., et al. Dynamic cell contacts between periportal mesenchyme and ductal epithelium act as a rheostat for liver cell proliferation. Cell Stem Cell. 28 (11), 1907-1921 (2021).
- Pastula, A., et al. Three-dimensional gastrointestinal organoid culture in combination with nerves or fibroblasts: a method to characterize the gastrointestinal stem cell niche. Stem Cells International. 2016, 3710836 (2016).
- Hamilton, T. G., Klinghoffer, R. A., Corrin, P. D., Soriano, P. Evolutionary divergence of platelet-derived growth factor alpha receptor signaling mechanisms. Molecular and Cellular Biology. 23 (11), 4013-4025 (2003).
- McGinn, J., et al. A biomechanical switch regulates the transition towards homeostasis in oesophageal epithelium. Nature Cell Biology. 23 (5), 511-525 (2021).
- Zhang, Y., Bailey, D., Yang, P., Kim, E., Que, J. The development and stem cells of the esophagus. Development. 148 (6), (2021).
- Ohlund, D., et al. Distinct populations of inflammatory fibroblasts and myofibroblasts in pancreatic cancer. The Journal of Experimental Medicine. 214 (3), 579-596 (2017).
- Pentinmikko, N., et al. Notum produced by Paneth cells attenuates regeneration of aged intestinal epithelium. Nature. 571 (7765), 398-402 (2019).
- Janson, D., Rietveld, M., Willemze, R., El Ghalbzouri, A. Effects of serially passaged fibroblasts on dermal and epidermal morphogenesis in human skin equivalents. Biogerontology. 14 (2), 131-140 (2013).
- Landry, N. M., Rattan, S. G., Dixon, I. M. C. An improved method of maintaining primary murine cardiac fibroblasts in two-dimensional cell culture. Scientific Reports. 9 (1), 12889 (2019).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon