Method Article
Caratterizzazione funzionale e visualizzazione di fibroblasti esofagei mediante co-colture organoidi
In questo articolo
Riepilogo
Le co-colture organoide-fibroblasti forniscono un modello per studiare la nicchia delle cellule staminali in vivo . Qui viene descritto un protocollo per le co-colture organoide-fibroblasti esofagee. Inoltre, l'imaging a montaggio intero viene utilizzato per visualizzare l'interazione fibroblasto-organoide.
Abstract
Le cellule staminali epiteliali e progenitrici contribuiscono alla formazione e al mantenimento della barriera epiteliale per tutta la vita. La maggior parte delle popolazioni di cellule staminali e progenitrici sono nascoste in posizioni anatomicamente distinte, consentendo interazioni esclusive con segnali di nicchia che mantengono la staminalità. Mentre lo sviluppo di colture organoidi epiteliali fornisce un potente strumento per comprendere il ruolo delle cellule staminali e progenitrici nell'omeostasi e nella malattia, l'interazione all'interno dell'ambiente di nicchia è in gran parte assente, ostacolando così l'identificazione dei fattori che influenzano il comportamento delle cellule staminali. I fibroblasti svolgono un ruolo chiave nel dirigere il destino del tronco epiteliale e del progenitore. Qui viene presentato un protocollo completo di co-coltura organoide-fibroblasti che consente la delineazione delle sottopopolazioni di fibroblasti nel rinnovamento e nella differenziazione delle cellule progenitrici esofagee. In questo protocollo, viene descritto un metodo per isolare sia le cellule epiteliali che i fibroblasti in parallelo dall'esofago. Sono delineate distinte strategie di selezione cellulare attivate dalla fluorescenza per isolare sia le cellule progenitrici esofagee che le sottopopolazioni di fibroblasti da topi transgenici reporter o wild-type. Questo protocollo fornisce un approccio versatile che può essere adattato per adattarsi all'isolamento di specifiche sottopopolazioni di fibroblasti. La creazione e il passaggio di monocolture organoidi epiteliali esofagee è incluso in questo protocollo, consentendo un confronto diretto con il sistema di co-coltura. Inoltre, viene descritto un approccio di compensazione 3D che consente un'analisi dettagliata delle immagini delle interazioni epiteliali-fibroblasti. Collettivamente, questo protocollo descrive un metodo comparativo e relativamente ad alto rendimento per identificare e comprendere i componenti di nicchia delle cellule staminali esofagee in vitro.
Introduzione
Gli organoidi sono utilizzati come saggi 3D in vitro per caratterizzare le cellule staminali e progenitrici, nonché per comprendere i segnali di segnalazione derivati dai componenti cellulari della nicchia delle cellule staminali 1,2,3,4. Gli organoidi esofagei di topo sono stati descritti per la prima volta nel 2014 e diversi articoli hanno identificato fattori di crescita, come R-Spondin (RSPO), NOGGIN e fattore di crescita epidermico (EGF), necessari per mantenere e passare gli organoidi esofagei 5,6,7, sostenendo che segnali di segnalazione simili sono necessari per in vivo Rinnovamento delle cellule progenitrici. Tuttavia, i fattori di crescita sono comunemente aggiunti in concentrazioni non fisiologiche, con conseguenti condizioni di crescita organoide che non riflettono necessariamente l'ambiente di segnalazione in vivo.
I fibroblasti sono popolazioni eterogenee di cellule stromali che supportano le proprietà delle cellule progenitrici in molte nicchie di cellule staminali8. La combinazione di cellule progenitrici epiteliali e fibroblasti nella stessa coltura organoide consente la formazione di organoidi in concentrazioni ridotte di fattori di crescita integrati esogenamente. Sono descritti sistemi di co-coltura organoidi da epiteli intestinali ed epatici, ma un protocollo per stabilire co-colture organoidi-fibroblasti esofagei è ancora in sospeso 9,10,11.
In questo protocollo, vengono delineate due strategie di selezione cellulare attivata dalla fluorescenza (FACS) per i fibroblasti dall'esofago, utilizzando topi transgenici PdgfrαH2BeGFP 12 o topi wild-type con colorazione anticorpale classica. Diverse sottopopolazioni di fibroblasti possono essere isolate utilizzando marcatori di superficie cellulare di scelta, fornendo così flessibilità al protocollo. Inoltre, una tecnica di imaging a fluorescenza 3D che preserva la morfologia organoide viene utilizzata per caratterizzare le interazioni fibroblasto-organoide. La pulizia degli organoidi fornisce un metodo rapido per aumentare la profondità di penetrazione della luce negli organoidi, migliorando la visualizzazione delle connessioni organoide-fibroblasti e consentendo la ricapitolazione della struttura organoide nella sua interezza. Questo protocollo combina la co-coltura di organoidi esofagei con una strategia di imaging a montaggio completo, consentendo la caratterizzazione funzionale dell'interazione fibroblasto-organoide.
Protocollo
Gli esperimenti sugli animali per questo studio sono stati approvati da Stockholms Norra djurförsöksetiska nämnd (permesso etico n. 14051-2019). Gli animali sono stati alloggiati in condizioni prive di agenti patogeni secondo le raccomandazioni della Federation of European Laboratory Animal Science Association.
1. Preparazione
- Scongelare le soluzioni enzimatiche utilizzate per la dissociazione (vedi Tabella dei materiali) su ghiaccio. Scongelare un'aliquota della matrice di membrana basale (matrice) ridotta dal fattore di crescita (GFR) a 4 °C.
- Preparare il terreno di coltura. Utilizzare il terreno descritto nella Tabella 1 per le colture organoidi e le co-colture e prepararlo prima di iniziare il protocollo.
2. Dissezione e separazione dell'epitelio esofageo e dello stroma
NOTA: Assicurarsi che tutti gli strumenti utilizzati per la dissezione e la lavorazione dei tessuti siano sterili. Preparare 2 ml di soluzione di dissociazione (vedere Tabella dei materiali) nella soluzione salina bilanciata di Hanks (HBSS) per tre esofagi.
- Utilizzare un ceppo di topo di scelta, come i mouse C57BL / 6J. Lo sviluppo esofageo nei topi termina dopo il giorno post-natale (p) 70, quindi si consiglia di utilizzare topi più vecchi di p7013. Utilizzare quattro o cinque topi in quanto forniscono materiale sufficiente per stabilire 8-10 co-colture organoidi.
NOTA: L'efficienza della formazione di organoidi diminuisce con l'età dei topi. Se specifiche sottopopolazioni di fibroblasti sono di interesse, è possibile che la bassa resa di fibroblasti stia limitando il numero di co-colture organoidi che possono essere stabilite. - Utilizzare topi geneticamente modificati (ad esempio, PdgfrαH2BeGFP) o wild-type (WT) per l'isolamento delle popolazioni di fibroblasti. Quando si utilizzano topi WT, eseguire la colorazione anticorpale per isolare specifiche sottopopolazioni di fibroblasti dallo stroma tramite FACS.
- Eutanasia dei topi mediante asfissia da CO2 . Sezionare l'esofago usando una pinza e forbici da dissezione. Per rimuovere l'intero esofago, tagliare l'estremità distale dell'esofago direttamente sopra lo stomaco e l'estremità prossimale all'inizio della trachea. Immergere l'esofago in PBS e metterlo sul ghiaccio.
- Rimuovere meccanicamente la muscolaris externa utilizzando un microscopio a dissezione (intervallo di ingrandimento totale di 8x-40x) e una pinza. Tenere l'estremità distale dell'esofago sezionato usando un paio di pinze e usare l'altra pinza per afferrare e tirare il muscolo dall'estremità distale all'estremità prossimale dell'esofago sezionato. Rimuovere e scartare lo strato muscolare.
- Aprire l'esofago longitudinalmente. Questo funziona meglio usando forbici a molla di microdissezione con una punta a sfera per prevenire danni ai tessuti. Tenere un'estremità dell'esofago per inserire la palla delle forbici a molla nel lume dell'esofago e tagliare l'esofago mentre si tiene fino alla fine.
- Posizionare l'esofago in un tubo da microcentrifuga da 1,5 ml o in una piastra a 24 pozzetti. Incubare l'esofago aperto in 0,5 mg/mL di termolisina in HBSS a 37 °C su un rocker-shaker per 15 minuti. Immergere completamente l'esofago in soluzione di termolisina.
NOTA: Il volume della soluzione di termolisina utilizzata dipende dalla dimensione del pozzo o del tubo. Diversi esofagi possono essere posizionati e immersi nello stesso pozzo o tubo. - Estrarre l'esofago dalla soluzione di termolisina. Utilizzare un microscopio a dissezione per separare accuratamente l'epitelio esofageo dallo stroma. Usando una pinza sottile, afferrare sia il lato epiteliale che il lato stroma del tessuto e separarli lentamente per separare i due strati.
NOTA: Lo stroma è identificato dal suo aspetto bianco e opaco, in contrasto con l'epitelio trasparente. Lo stroma contiene la lamina propria e lo strato sottomucoso. - Trasferire lo strato epiteliale e stromale in due provette separate da microcentrifuga da 1,5 mL con 200 μL di soluzione di dissociazione in HBSS. Mettere in ghiaccio.
3. Isolamento delle cellule progenitrici esofagee
NOTA: L'isolamento delle cellule progenitrici esofagee (fase 2) e dei fibroblasti (fase 3) può essere eseguita contemporaneamente. Preparare una provetta da 50 mL di FBS all'1% in HBSS (1% FBS).
- Trasferire l'epitelio esofageo separato dal tubo di microcentrifuga da 1,5 mL (fase 2.8) in una capsula di Petri pulita e utilizzare un bisturi affilato per tritare. Raccogliere il tessuto tritato dalla capsula di Petri con 200 μL di soluzione di dissociazione e trasferirlo nuovamente nella provetta da microcentrifuga da 1,5 ml.
NOTA: L'epitelio viene tritato correttamente quando i pezzi possono essere ricollocati nella provetta da microcentrifuga da 1,5 ml utilizzando una punta per pipetta da 200 μL. - Aggiungere 800 μL di soluzione di dissociazione fresca alla provetta da microcentrifuga da 1,5 mL per ottenere un volume totale di 1 mL.
NOTA: è importante aggiungere un volume sufficiente di soluzione di dissociazione. Per uno a tre esofagi, si raccomanda 1 mL di soluzione. Scalare quando più esofagi vengono elaborati contemporaneamente. - Posizionare il tubo con lo strato epiteliale tritato su un rocker-shaker a 37 °C per 60 minuti. Pipettare la soluzione su e giù circa 20 volte utilizzando una punta di pipetta da 200 μL ogni 15 minuti per migliorare la digestione.
- Dopo 60 minuti, pipettare su e giù altre 20 volte utilizzando una punta da 200 μL. Far passare la soluzione attraverso un filtro cellulare da 40 μm in una nuova provetta da microcentrifuga da 1,5 ml. Centrifugare a 300 x g per 10 minuti a 4 °C.
NOTA: Bagnare il filtro cellulare con FBS all'1% prima di filtrare le celle per ridurre al minimo l'aderenza delle celle al filtro. L'epitelio non sarà completamente digerito e pezzi di tessuto saranno ancora visibili. Un'incubazione più lunga, tuttavia, diminuirà la sopravvivenza cellulare e non si tradurrà in una maggiore resa di cellule vitali. - Scartare il surnatante rimuovendo il liquido in eccesso con una pipetta da 1 mL e risospendere il pellet in 1 mL di FBS all'1%. Centrifugare a 300 x g per 10 minuti a 4 °C.
- Durante la centrifugazione, preparare la miscela di anticorpi coniugati per FACS delle cellule progenitrici esofagee.
- Mescolare 1 μL di CD324-PE-Cy7 (ECADERINA) e CD104-A647 (INTEGRINA-β4) in 200 μL di FBS all'1% per milione di cellule.
NOTA: 1 μL di anticorpo (200 μL di volume finale) è sufficiente per uno o due esofagi. Quando si elaborano più esofagi contemporaneamente, aumentare il volume della soluzione di colorazione anticorpale.
- Risospendere il pellet in 200 μL di miscela anticorpale e trasferirlo in un tubo citometrico a flusso. Dopo aver aggiunto anticorpi fluorescenti, mantenere le sospensioni cellulari al buio per evitare lo sbiancamento del segnale. Incubare le cellule per 30 minuti a 4 °C. Aggiungere 3 mL di FBS all'1% e centrifugare a 300 x g per 10 minuti a 4 °C, quindi risospendere le cellule in un minimo di 200 μL dell'1% di FBS.
NOTA: 500 μL di FBS all'1% vengono utilizzati per un massimo di quattro o cinque esofagi. Aumentare il volume quando vengono elaborati più esofagi contemporaneamente, in modo da ottenere una portata FACS di 100-300 eventi / s. Più eventi diminuiranno l'efficienza di selezione e un aumento della portata diminuirà la sopravvivenza cellulare. - Aggiungere il marcatore della colorazione delle cellule morte a una concentrazione finale di 1:10.000, 5 minuti prima dello smistamento FACS per isolare le cellule vive. Ordinare le cellule progenitrici usando una macchina FACS (vedere la Figura 1 per la strategia di gating). Raccogliere le cellule in provette da microcentrifuga da 1,5 mL riempite con 200 μL di terreno organoide basico (Tabella 1).
4. Isolamento dei fibroblasti dallo strato stromale
- Tagliare lo strato di stomi in pezzi fini in un tubo da 1,5 mL contenente 200 μL di soluzione di dissociazione (fase 2.8) usando le forbici da dissezione. Il tessuto viene opportunamente tritato una volta che la soluzione può essere pipettata su e giù utilizzando una punta di pipetta da 200 μL.
- Aggiungere 800 μL di soluzione di dissociazione fresca alla provetta da microcentrifuga da 1,5 mL per ottenere un volume totale di 1 mL.
NOTA: è importante aggiungere un volume sufficiente di soluzione di dissociazione. Per uno a tre esofagi, si raccomanda 1 mL di soluzione. Scalare quando più esofagi vengono elaborati contemporaneamente. - Posizionare il tubo su un rocker-shaker a 37 °C per 30 minuti. Dopo 15 minuti, pipettare la soluzione su e giù circa 20 volte utilizzando una punta da 200 μL per migliorare la digestione.
- Dopo 30 minuti di digestione enzimatica, pipettare su e giù altre 20 volte utilizzando una punta da 200 μL. Filtrare la soluzione attraverso un filtro cellulare da 70 μm in una nuova provetta da microcentrifuga da 1,5 ml. Centrifugare a 300 x g per 10 minuti a 4 °C.
NOTA: Bagnare il filtro cellulare con FBS all'1% prima di filtrare le celle per ridurre al minimo l'aderenza delle cellule al filtro. - Scartare il surnatante rimuovendo il liquido in eccesso con una pipetta da 1 mL e risospendere il pellet in 1 mL di FBS all'1%. Centrifugare a 300 x g per 10 minuti a 4 °C.
NOTA: Quando si utilizza un ceppo di topo geneticamente modificato contenente fibroblasti marcati con farina, le colorazioni anticorpali sono facoltative. Se non sono necessarie colorazioni anticorpali, continuare con il punto 3.7 e trasferire il campione in un tubo citometrico a flusso.- Durante la centrifugazione, preparare la miscela di anticorpi coniugati per l'isolamento FACS dei fibroblasti. Mescolare 1 μL di CD26-APC (DPP4) in 200 μL di FBS all'1% per un milione di cellule.
NOTA: 1 μL di anticorpo è sufficiente per uno o due esofagi. Quando si elaborano più esofagi contemporaneamente, aumentare il volume della soluzione di colorazione anticorpale.
- Durante la centrifugazione, preparare la miscela di anticorpi coniugati per l'isolamento FACS dei fibroblasti. Mescolare 1 μL di CD26-APC (DPP4) in 200 μL di FBS all'1% per un milione di cellule.
- Risospendere il pellet in 200 μL di miscela di anticorpi coniugati in FBS all'1% e trasferirlo in un tubo di citometria a flusso. Incubare le cellule per 30 minuti a 4 °C.
- Aggiungere 3 mL di FBS all'1% e centrifugare a 300 x g per 10 minuti a 4 °C. Risospendere le cellule in un minimo di 200 μL dell'1% di FBS.
NOTA: 500 μL di FBS all'1% vengono utilizzati per un massimo di quattro o cinque esofagi. Aumentare il volume quando vengono elaborati più esofagi contemporaneamente, in modo da ottenere una portata di 100-300 eventi / s. Più eventi diminuiranno l'efficienza di selezione e l'aumento della portata diminuirà la sopravvivenza cellulare. Dopo aver aggiunto anticorpi fluorescenti, le sospensioni cellulari devono essere mantenute scure per evitare lo sbiancamento del segnale. - Aggiungere il marcatore della colorazione delle cellule morte a una concentrazione finale di 1:10.000, 5 minuti prima dello smistamento FACS per isolare le cellule vive. Ordinare le celle utilizzando una macchina FACS (vedere la Figura 1 per la strategia di gating). Raccogliere le cellule in provette da microcentrifuga da 1,5 mL riempite con 200 μL di terreno organoide basico (Tabella 1).
5. Istituzione e cultura degli organoidi esofagei
NOTA: ER precaldobasso (cocoltura organoide), terreno ENR (organoide) (vedere Tabella 1 per la descrizione) e una piastra a 48 pozzetti a 37 °C. Porre l'aliquota della matrice scongelata (preparata al punto 1) sul ghiaccio. Si raccomanda di utilizzare la matrice fornita qui (vedi Tabella dei materiali) per la coltura di organoidi esofagei di topo, poiché altre marche di matrice influenzano negativamente l'efficienza di formazione degli organoidi.
- Per la co-coltura organoide, mescolare le cellule epiteliali ordinate e i fibroblasti in un rapporto di 1: 2 in un tubo. Per ogni cupola della matrice, utilizzare 5.000 cellule epiteliali e 10.000 fibroblasti. Quando ti prepari per più cupole, aggiungi un multiplo di 5.000 cellule epiteliali e 10.000 fibroblasti a un tubo. Per le colture organoidi senza fibroblasti, utilizzare 5.000 cellule epiteliali per cupola della matrice.
- Centrifugare la popolazione di cellule miste a 300 x g per 5 minuti. Eliminare il surnatante rimuovendolo con cautela con una pipetta da 200 μL.
- Lavare le celle una volta risospendendo il pellet in mezzo organoide basico freddo e centrifugare a 300 x g per 5 minuti. Metti le celle sul ghiaccio.
- Preparare una miscela composta per l'80% da matrice e per il 20% da un mezzo organoide basico freddo. Mettere tutto sul ghiaccio mentre la matrice si solidifica a temperatura ambiente (RT).
- Eliminare il surnatante dopo la centrifugazione rimuovendolo accuratamente con una pipetta da 200 μL. Risospendere le cellule in 10 μL di miscela/cupola della matrice e rimettere sul ghiaccio.
- Prelevare la piastra preriscaldata a 48 pozzetti dall'incubatore a 37 °C e realizzare una cupola a matrice per pozzetto utilizzando una pipetta da 20 μL. Ogni cupola contiene 10 μL di matrice all'80%, 5.000 cellule epiteliali e 10.000 fibroblasti. Trasferire la piastra capovolta in un'incubatrice e lasciare solidificare le cupole per altri 20-30 minuti a 37 °C.
NOTA: Una diminuzione del volume della cupola della matrice e/o un aumento del numero di celle influenzerà l'efficienza di formazione degli organoidi. - Aggiungere 200 μLdi mezzo ER basso preriscaldato (Tabella 1) alle cupole della matrice contenenti co-colture organoidi e mezzo ENR (Tabella 1) alle rispettive cupole della matrice contenenti solo organoidi epiteliali e posizionare la piastra nell'incubatore.
- Coltivare gli organoidi a 37 °C e 5% di CO2. Per i primi 2 giorni, integrare il terreno con 10 μM di inibitore di roccia (Y-27623). L'inibitore della roccia previene la morte cellulare indotta dallo stress e aumenta la possibilità di una creazione di successo di colture organoidi.
- Aggiorna il mezzo ogni 2-3 giorni. Assicurarsi che il mezzo sia caldo per evitare la dissociazione della matrice sensibile alla temperatura.
- Eseguire analisi dell'esperimento da 6 a 8 giorni dopo la placcatura. Gli organoidi possono essere tenuti in coltura per un massimo di 14 giorni. Intorno al giorno 14, si osserva la perdita dell'integrità della cupola.
6. Passaggio degli organoidi
NOTA: Il passaggio di organoidi coltivati in co-coltura provoca la perdita di fibroblasti. Pertanto, si raccomanda di utilizzare il mezzo ENR per tutti gli organoidi durante il passaggio. Preriscaldare ENR, PBS e una piastra a 48 pozzetti a 37 °C.
- Rimuovere il mezzo e lavare il pozzetto contenente la cupola della matrice con PBS preriscaldato. Aggiungere 200 μL di soluzione fredda di tripsina allo 0,25% e pipettare su e giù per rompere la cupola della matrice.
NOTA: Si raccomanda l'uso di tripsina fredda allo 0,25%, poiché la matrice è sensibile alla temperatura e questo aiuta a rompere le cupole della matrice. - Incubare gli organoidi con tripsina a 37 °C per ~20 min. Pipettare su e giù dopo 10 minuti per aumentare la dissociazione degli organoidi. Monitorare il processo di dissociazione con un microscopio ogni 5-10 minuti. Poiché la tripsina diminuisce la vitalità cellulare, questo può aiutare a identificare il tempo di dissociazione ideale.
- Dopo 20 minuti, pipettare gli organoidi su e giù con una pipetta da 200 μL per dissociare gli organoidi. Raccogliere le cellule in una provetta da microcentrifuga da 1,5 ml, aggiungere 1 ml di terreno organoide di base e centrifugare a 300 x g per 5 minuti a RT.
- Facoltativo: per garantire le migliori condizioni di crescita degli organoidi e che i nuovi organoidi siano derivati da singole cellule, filtrare le cellule attraverso un filtro cellulare pre-bagnato da 40 μm. Il filtraggio della sospensione cellulare provoca la rimozione di nuclei organoidi e grumi cellulari difficili da dissociare.
- Preparare una miscela di matrice composta per l'80% da matrice e per il 20% da mezzo organoide basico freddo. Metti tutto sul ghiaccio, mentre la matrice si solidifica a RT.
- Scartare il surnatante rimuovendolo con cautela con una pipetta da 200 μL, risospendere le cellule in 10 μL di miscela/cupola della matrice e rimettere la miscela sul ghiaccio.
NOTA: Gli organoidi possono essere suddivisi in rapporto da 1:5 a 1:10, a seconda della densità della cupola. Le cellule epiteliali possono anche essere contate e ri-placcate a 5.000 cellule / cupola. - Prelevare la piastra preriscaldata a 48 pozzetti dall'incubatore a 37 °C e fare una cupola per pozzetto. Trasferire la piastra capovolta in un'incubatrice e lasciare solidificare le cupole per altri 20-30 minuti a 37 °C.
- Aggiungere 200 μL di mezzo ENR preriscaldato alle rispettive cupole organoidi. Integrare il terreno con 10 μM di inibitore di roccia (Y-27623) per i primi 2 giorni.
- Aggiorna il mezzo ogni 2-3 giorni. Assicurarsi che il mezzo sia caldo per evitare la dissociazione della matrice sensibile alla temperatura.
7. Lavorazione organoide per la colorazione dell'intero montaggio
NOTA: Rivestire le punte e i tubi con il 10% di FBS in PBS prima dell'uso per evitare che organoidi aderiscano alla plastica. Per le punte delle pipette, è sufficiente pipettare una o due volte su e giù in soluzione FBS/PBS al 10% prima di utilizzare la punta. Per i tubi, riempire il tubo con FBS / PBS al 10% e quindi rimuovere la soluzione.
- Rimuovere il mezzo organoide e aggiungere 200 μL di PBS ghiacciato alle cupole della matrice. Posizionare il piatto sul ghiaccio per 5-10 minuti.
- Pipettare su e giù e trasferire la soluzione in provette non aderenti da microcentrifuga da 0,6 ml. Centrifugare brevemente per 30-60 s a 100 x g per far sedimentare gli organoidi sul fondo.
NOTA: L'eccesso di pipettaggio e la lunga centrifugazione rompono gli organoidi e interrompono l'interazione fibroblasto-organoide. - Rimuovere il liquido in eccesso e aggiungere PBS ghiacciato. Centrifugare brevemente per 30-60 s a 100 x g per far sedimentare gli organoidi sul fondo.
- Rimuovere il liquido in eccesso e fissare l'organoide con 200 μL di formaldeide fredda al 4% in soluzione PBS per 30 minuti su ghiaccio.
ATTENZIONE: La formaldeide è tossica e deve essere sempre utilizzata in una cappa aspirante chimica. I guanti Nitril, gli occhiali di sicurezza e i camici da laboratorio devono essere sempre indossati. - La fissazione dell'organoide li fa affondare e la centrifugazione non è più necessaria. Lasciare affondare gli organoidi posizionando il tubo in posizione verticale e attendere 2-3 minuti, rimuovere la formaldeide e aggiungere 500 μL di PBS freddo per lavare via la formaldeide.
- Lasciare affondare gli organoidi, rimuovere il PBS in eccesso e aggiungere 500 μL di PBS freddo fresco. Lasciare affondare gli organoidi, rimuovere il PBS in eccesso e aggiungere 500 μL di tampone bloccante (5% siero normale dell'asino, 1% BSA e 0,5% Triton X-100 in PBS). Metti il tubo su un rocker-shaker per 60 minuti a RT.
NOTA: Il blocco può essere effettuato anche durante la notte (O/N) a 4 °C. - Lasciare affondare gli organoidi, rimuovere il tampone bloccante e risospendere gli organoidi in 200 μL di tampone bloccante con anticorpi primari (vedi Tabella dei materiali). Conservare gli organoidi su un rocker-shaker O/N a 4 °C.
NOTA: Per migliorare la colorazione delle proteine nucleari, delle proteine a bassa espressione o degli anticorpi che mostrano una colorazione aspecifica, il tempo di incubazione può essere aumentato per 1 o 2 giorni a 4 °C. Posizionare gli organoidi su un rocker-shaker è essenziale in quanto impedisce agli organoidi di aggregarsi e aumenta l'efficienza di colorazione. - Lasciare affondare gli organoidi, rimuovere la miscela di anticorpi primari e lavare gli organoidi usando 500 μL di Triton X-100 allo 0,02% in PBS (0,02% Tx) per 60 minuti a RT. Ripetere tre volte.
NOTA: Quando è necessaria un'incubazione di anticorpi primari più lunga, aggiungere una fase di lavaggio con Tx 0,02% in PBS O/N a 4 °C. - Lasciare affondare gli organoidi, rimuovere il tampone di lavaggio e aggiungere 200 μL di anticorpo secondario coniugato con fluorescenza nel tampone O/N bloccante a 4 °C. Dopo aver aggiunto anticorpi secondari fluorescenti, tenere gli organoidi al buio per evitare lo sbiancamento del segnale.
- Lasciare affondare gli organoidi, rimuovere la miscela di anticorpi secondari e lavare gli organoidi usando 500 μL di Tx allo 0,02% per 60 minuti a RT. Ripetere tre volte.
- Controcolorare gli organoidi con 200 μL di soluzione DAPI (0,25 μg/mL) per la colorazione nucleare, se necessario. Incubare i campioni per 60 minuti a RT su un rocker-shaker.
- Lasciare affondare gli organoidi, rimuovere la soluzione DAPI e lavare gli organoidi in 500 μL di PBS per 15 minuti a RT. Ripetere tre volte.
- Lascia affondare gli organoidi e rimuovi tutto il liquido in eccesso. Aggiungere 10 μL di soluzione limpida agli organoidi e incubare per 15 minuti a RT.
NOTA: la soluzione di clearing è viscosa, quindi tagliare la parte esterna di una punta di pipetta da 20 μL prima di pipettare la soluzione di clearing. È possibile aggiungere più soluzioni di compensazione se per l'imaging viene utilizzato un distanziatore più grande. La soluzione di montaggio può essere utilizzata al posto della soluzione di compensazione quando non si eliminano gli organoidi. La soluzione di compensazione riduce lo sfondo durante l'imaging e aumenta la profondità di imaging. - Posizionare un distanziatore a 4 pozzetti appiccicoso a doppia faccia da 0,05 mm su un vetrino da microscopio. Aggiungere i 10 μL di soluzione limpida con gli organoidi in un pozzetto e posizionare un vetrino di copertura sopra il distanziatore.
NOTA: Gli organoidi possono anche essere montati senza l'uso di un distanziatore. Il distanziatore mantiene intatta la forma dell'organoide e impedisce che gli organoidi vengano appiattiti. - Tenere il vetrino su RT O/N per eliminare gli organoidi. Per una conservazione a lungo termine, conservare i vetrini a 4 °C. Acquisire immagini utilizzando un sistema di microscopia confocale.
Risultati
L'esofago è diviso in diversi strati: epitelio, lamina propria, sottomucosa e muscularis externa (Figura 1A). I fibroblasti risiedono all'interno della sottomucosa e della lamina propria, indicata come stroma. In questo protocollo, la muscularis externa viene rimossa meccanicamente (Figura 1B), il che non porta a una perdita di fibroblasti (PdgfrαH2BeGFP+) residenti nello stroma (Figura 1C). Prima della dissociazione, l'epitelio viene separato dallo stroma risultando in due segmenti di tessuto (Figura 1B). La separazione dei due strati offre l'opportunità di aumentare il tempo di dissociazione per lo strato epiteliale più robusto rispetto allo strato stromale fragile. In questo modo, viene stabilito un protocollo di isolamento efficiente che produce sia cellule progenitrici epiteliali vitali che fibroblasti stromali (Figura 1B). Le cellule progenitrici esofagee sono classificate in base alla loro elevata espressione di INTEGRIN-β4 ed E-CADHERIN (Figura 1C,D).
Le sottopopolazioni di fibroblasti possono essere isolate utilizzando marcatori distinti. In questo protocollo, viene fornita una strategia per l'isolamento dei fibroblasti basata sui marcatori di fibroblasti comunemente usati PDGFRα e DPP4 (CD26). L'isolamento mediante l'espressione del reporter PdgfrαH2BeGFP o l'anticorpo DPP4 mostra che circa il 50% delle cellule isolate sono fibroblasti (Figura 1E,F). Inoltre, il 70% dei fibroblasti PDGFRα+ sono DPP4+, indicando che si ottiene una popolazione di fibroblasti in gran parte sovrapposta, ma non identica. Dopo aver isolato le popolazioni di cellule epiteliali e stromali, le cellule progenitrici esofagee vengono coltivate da sole o insieme ai fibroblasti in una cupola a matrice. Per studiare il contributo dei fibroblasti alla formazione di organoidi, la co-coltura viene mantenuta in un terreno ridotto dal fattore di crescita (Figura 1G).
Le cellule progenitrici esofagee formano organoidi in presenza di EGF, NOGGIN e RSPO (ENR). Rimozione di NOGGIN e riduzione della quantità di RSPO (25 ng/μL; ERbasso) è sufficiente per prevenire la formazione di organoidi (Figura 2A). È interessante notare che l'aggiunta di fibroblasti DPP4+ o PDGFRα+ alle cellule progenitrici esofageenel mezzo basso ER ripristina la capacità di formazione degli organoidi, dimostrando una funzione di supporto per entrambe le popolazioni di fibroblasti (Figura 2A-D). La visualizzazione del transgene PdgfrαH2BeGFP mostra che i fibroblasti sono in stretto contatto con le cellule progenitrici epiteliali durante la formazione degli organoidi (Figura 2A). Al giorno 6, i fibroblasti PdgfrαH2BeGFP+ sono ancora abbondantemente presenti nella co-coltura. I fibroblasti sono presenti in tutta la cupola, vicino e toccando gli organoidi (freccia piena), o attaccati agli organoidi (punta di freccia; Figura 2D).
La colorazione a montaggio intero mostra una rappresentazione 3D dell'interazione dei fibroblasti con gli organoidi (Figura 3). Mentre è possibile eseguire l'intero protocollo di montaggio senza l'uso di una soluzione di clearing, diminuisce la trasparenza e la penetrazione laser dell'organoide (Figura 3B, z-view). Quando si montano gli organoidi, il distanziatore aiuta a mantenere la morfologia degli organoidi. Al contrario, placcando il coprislip direttamente sugli organoidi (senza distanziatore) appiattisce gli organoidi e provoca la perdita della struttura organoide (Figura 3A,B).
Entrambi i fibroblasti DPP4+ e PDGFRα+ sono avvolti attorno agli organoidi (Figura 3C, Video1 e Video 2). La differenziazione degli organoidi esofagei può essere valutata utilizzando diversi marcatori. La Figura 4 mostra che il protocollo di colorazione fornito è adatto per cheratine più facili da colorare (KRT14/13) e fattori di trascrizione più difficili da colorare (TRP63/KLF4). Il protocollo di co-coltura genera organoidi con un modello di differenziazione simile, come dimostrato in vivo13,14 e come visto negli organoidi coltivati in mezzo ENR; Le cellule progenitrici KRT14+ o TRP63+ formano lo strato esterno e le cellule differenziate KRT13+ o KLF4+ si orientano verso l'interno.
Questo protocollo fornisce uno strumento per studiare la nicchia delle cellule staminali esofagee in vitro e visualizza l'interazione tra organoidi e fibroblasti. Implementando un protocollo per l'isolamento dei fibroblasti utilizzando anticorpi, il metodo è adattabile e può essere utilizzato per studiare sottopopolazioni di fibroblasti senza la necessità di topi transgenici.
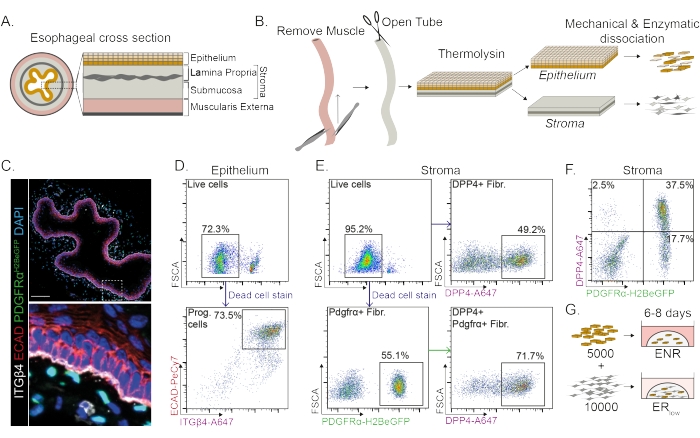
Figura 1: Isolamento delle cellule progenitrici e delle sottopopolazioni di fibroblasti dall'esofago . (A) Panoramica schematica dei diversi strati dell'esofago. Lo stroma contiene la lamina propria e la sottomucosa. (B) Panoramica schematica del protocollo di isolamento. Il muscolo (muscularis externa) viene rimosso meccanicamente usando una pinza; L'esofago rimanente viene tagliato e incubato in termolisina per separare lo strato epiteliale dallo stroma. L'epitelio e lo stroma sono separati, tritati meccanicamente e digeriti enzimaticamente in sospensioni monocellulari. Le cellule dissociate vengono quindi colorate e preparate per FACS. (C) Sezione trasversale dell'esofago strappato dal muscolo esterno che mostra fibroblasti PdgfrαH2BeGFP+ nello stroma. Le cellule doppie positive di INTEGRIN-β4 (ITGβ4) e E-CADERINA (ECAD) sono le cellule progenitrici epiteliali dell'esofago. Barra di scala = 100 μm. (D) Grafico rappresentativo della citometria a flusso dell'isolamento delle cellule epiteliali che mostra la percentuale di cellule vive (pannello superiore) da tutte le singole cellule. Il pannello inferiore mostra la percentuale di cellule progenitrici (Prog.) ITGβ4+ ECAD+ isolate da tutte le cellule vive. (E) Grafico rappresentativo della citometria a flusso dell'isolamento delle cellule stromali che mostra la percentuale di cellule vive (pannello in alto a sinistra). Grafici rappresentativi della citometria a flusso che mostrano la percentuale di fibroblasti DPP4+ isolati (Fibr.; pannello in alto a destra) e fibroblasti Pdgfrα+ (pannello in basso a sinistra) di tutte le cellule vive. Il 70% dei fibroblasti Pdgfrα+ sono anche DPP4+ (pannello in basso a destra). (F) Grafico rappresentativo della citometria a flusso dello stroma che mostra solo cellule DPP4+ (2,5%), cellule DPP4+ PDGFRα+ (37,5%) e solo cellule PDGFRα+ (17,7%). Le percentuali sono di tutte le cellule vive. (G) Le cellule solo epiteliali sono placcate in una cupola a matrice in presenza di 50 ng/μL EGF, 100 ng/μL NOGGIN e 250 ng/μL RSPO (ENR), o insieme a fibroblasti in presenza di EGF e una bassa concentrazione di RSPO (25 ng/μL). Fare clic qui per visualizzare una versione ingrandita di questa figura.
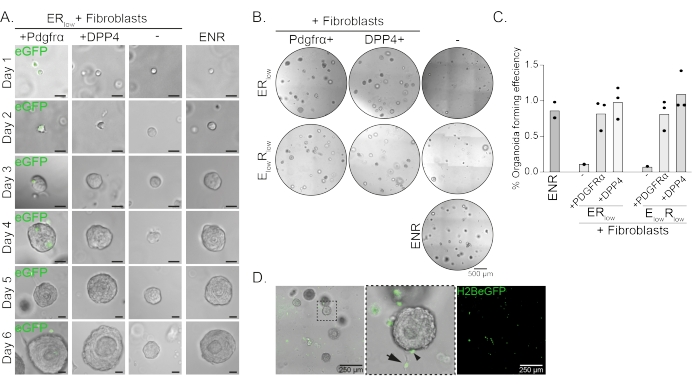
Figura 2: Risultati rappresentativi delle cocolture di organoidi. (A) Immagini in campo chiaro che mostrano la crescita degli organoidi dal giorno 1 al giorno 6. Le immagini in campo chiaro con gli organoidi co-coltivati con fibroblasti PdgfrαH2BeGFP+ mostrano anche il segnale nucleare eGFP. Barra di scala = 25 μm. (B) Immagini in campo chiaro dell'intera cupola della matrice al giorno 6. La colonna di sinistra mostra co-colture organoidi coltivate in presenza di fibroblasti Pdgfrα+ in ER basso o Ebasso Rbasso medio. La colonna centrale mostra co-colture organoidi coltivate in presenza di fibroblasti DPP4+ in ER basso o E bassoRbasso medio. La colonna di destra mostra monocolture organoidi coltivate in mezzo ENR. Mezzo ENR = EGF (50 ng/μL), NOGGIN (100 ng/μL) e RSPO (250 ng/μL). ERbasso = EGF e 25 ng/μL RSPO. E bassoRbasso = 5 ng/μL EGF e 25 ng/μL RSPO. Barra di scala = 500 μm. (C) Grafico che mostra l'efficienza di formazione degli organoidi (%) (cioè la percentuale di cellule che formano organoidi in diverse condizioni di coltura). Ogni punto rappresenta una cupola a matrice e la barra rappresenta la media di tutti i punti per condizione. (D) Immagine in campo chiaro e fluorescente di organoidi del giorno 6 co-coltivati con fibroblasti PdgfrαH2BeGFP+. I fibroblasti PdgfrαH2BeGFP+ sono presenti in tutta la cupola, attaccati agli organoidi (punta di freccia) e non attaccati ma a contatto con gli organoidi (freccia completa). Barra di scala = 250 μm. Fare clic qui per visualizzare una versione ingrandita di questa figura.
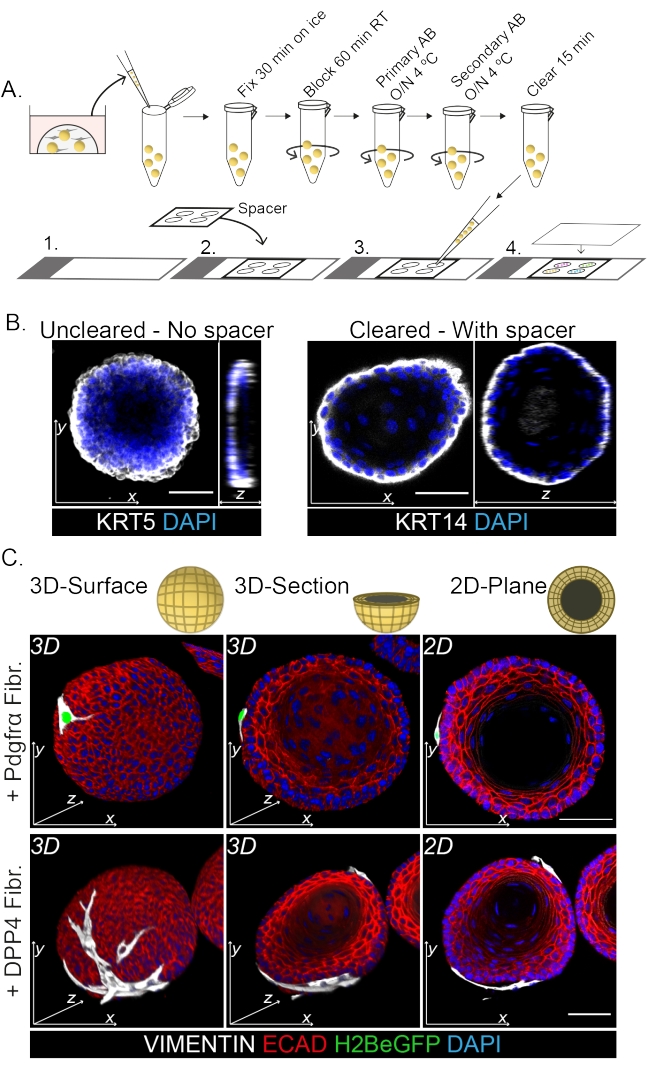
Figura 3: Protocollo di colorazione a montaggio intero per lo studio delle interazioni fibroblasto-organoide . (A) Panoramica schematica dell'intero protocollo di immunofluorescenza a monte. AB = anticorpo. (B) Immagine di immunofluorescenza della colorazione dell'intera montatura non cancellata che mostra una diminuzione della trasparenza e della penetrazione della luce laser rispetto agli organoidi eliminati. L'assenza di un distanziatore provoca l'appiattimento dell'organoide e la perdita della morfologia organoide. (C) Le immagini a montaggio intero degli organoidi co-coltivati mostrano superfici 3D degli organoidi con fibroblasti VIMENTINA + (Fibr.) avvolti intorno e a stretto contatto con l'organoide. Le sezioni trasversali 3D e le immagini del piano 2D mostrano il lume dell'organoide. Barra di scala = 50 μm. Fare clic qui per visualizzare una versione ingrandita di questa figura.
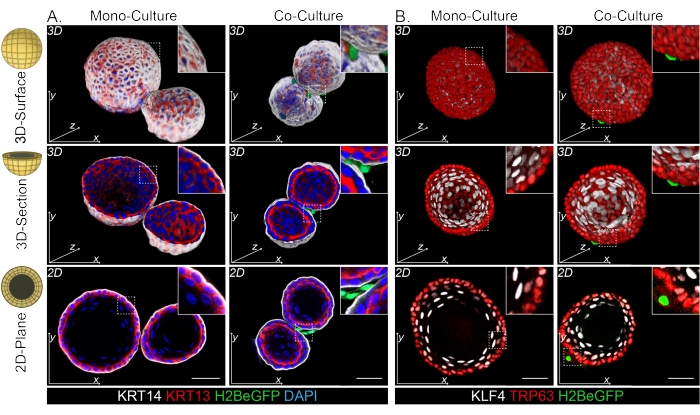
Figura 4: Le immagini a montaggio intero rivelano distinte popolazioni di cellule basali e soprabasali. (A) Colorazione a montaggio intero di organoidi mono- e co-coltivati con fibroblasti PdgfrαH2BeGFP+ che mostrano cellule basali KRT14+ nello strato esterno e cellule soprabasali differenziate KRT13+. Barra di scala = 50 μm. (B) Colorazione a montaggio intero di organoidi mono- e co-coltivati con fibroblasti PdgfrαH2BeGFP+ che mostrano cellule basali TRP63+ nello strato esterno e cellule soprabasali differenziate KLF4+. Barra di scala = 50 μm. Fare clic qui per visualizzare una versione ingrandita di questa figura.
Tabella 1: Tabella che descrive i componenti dei terreni di coltura organoidi. Clicca qui per scaricare questa tabella.
Video 1: Fibroblasto PdgfrαH2BeGFP+ avvolto intorno e a stretto contatto con l'organoide. Il video accompagna il pannello superiore della Figura 3C. La barra della scala nella Figura 3C è di 50 μm e l'organoide ha un diametro di ~ 120 μm. VIMENTINA è mostrato in bianco, E-CADHERIN in rosso, PdgfrαH2BeGFP in verde e DAPI in blu. Clicca qui per scaricare questo video.
Video 2: Fibroblasto DPP4+ avvolto intorno e a stretto contatto con l'organoide. Il video accompagna il pannello inferiore della Figura 3C. La barra della scala nella Figura 3C è di 50 μm e l'organoide ha un diametro di ~ 120 μm. VIMENTIN è mostrato in bianco, E-CADHERIN in rosso e DAPI in blu. Clicca qui per scaricare questo video.
Discussione
Il protocollo qui presentato stabilisce un modello in vitro per lo studio delle interazioni funzionali epiteliali-fibroblasti esofagei.
Lo strato epiteliale è separato dallo stroma, consentendo un protocollo di dissociazione ottimizzato sia per il compartimento epiteliale che per quello stromale. Nonostante l'ottimizzazione del protocollo di dissociazione epiteliale, i grumi di tessuto rimangono evidenti. Il pipettaggio su e giù vigorosamente ogni 15 minuti diminuisce sostanzialmente il numero e le dimensioni dei grumi. Altri protocolli utilizzano la tripsina per dissociare ulteriormente lo strato epiteliale 5,6. Qui, l'uso di tripsina, o aumentare ulteriormente il tempo di dissociazione, non è raccomandato, in quanto ciò tende a provocare una diminuzione della vitalità delle cellule epiteliali e dell'efficienza di formazione degli organoidi. In contrasto con l'epitelio, lo stroma è facilmente dissociato e 30 minuti in soluzione di dissociazione si traduce in una sospensione unicellulare con ~ 90% di vitalità dei fibroblasti (Figura 1E). L'esclusione della fase di separazione epiteliale-stomale nel protocollo aumenta sostanzialmente il tempo di dissociazione, con conseguente diminuzione della vitalità dei fibroblasti e una minore resa delle cellule epiteliali. Inoltre, separare l'epitelio dallo stroma offre l'opportunità di determinare il numero di cellule di ciascuna popolazione e mescolare cellule epiteliali e fibroblasti di diverse linee di topo durante la creazione delle co-colture.
Lo studio della funzione dei fibroblasti sulla crescita organoide è un metodo comunemente usato nella biologia delle cellule staminali 9,10,11,15,16. I terreni di co-coltura consolidati sono DMEM integrato con siero fetale di vitello al 10% (FCS)9,15 o con fattore di crescita ridotto10,16. In questo protocollo, il mezzo ridotto del fattore di crescita viene utilizzato per imitare le condizioni nella nicchia delle cellule staminali in vivo, dove i fibroblasti sono in gran parte quiescenti. FCS è un siero ricco di fattori di crescita che provoca l'attivazione e la proliferazione dei fibroblasti nelle co-colture, probabilmente corrispondente a uno stato cellulare di fibroblasti distinto dallo stato in vivo. Escludendo la FCS e riducendo i fattori di crescita, in modo che il mezzo da solo (ERbasso) non supporti la crescita organoide e non stimoli la proliferazione dei fibroblasti, è possibile isolare l'effetto dei fibroblasti sulla crescita degli organoidi. In questo mezzo, NOGGIN viene rimosso e RSPO ridotto al minimo (10% RPSO). Sia NOGGIN che RSPO hanno dimostrato di essere essenziali per la crescita degli organoidi esofagei6. L'EGF è stato mantenuto nel mezzo di cocoltura, in quanto non supporta di per sé la crescita degli organoidi. Tuttavia, i fibroblasti sono anche in grado di supportare la crescita organoide in un mezzo ridotto di EGF (E basso Rbasso; Figura 2B,D).
Le co-colture organoidi non possono essere sostenute attraverso il passaggio poiché i fibroblasti vengono persi durante la tripsinizzazione. Tuttavia, il passaggio degli organoidi è stato incluso nel protocollo poiché gli organoidi esofagei possono essere mantenuti, ampliati e utilizzati per ulteriori esperimenti come mono-colture. Gli organoidi passati da monocolture possono essere utilizzati per creare co-colture con fibroblasti appena isolati. Uno svantaggio dell'uso di cellule primarie è il numero di topi necessari per creare più co-colture di organoidi. Quando ci si concentra su piccole sottopopolazioni di fibroblasti, il numero di co-colture ottenute è limitato. In altri protocolli, i fibroblasti vengono prima espansi in coltura prima di utilizzarli per creare co-colture organoidi10. Tuttavia, i fibroblasti cambiano morfologia e identità durante il passaggio, come dimostrato utilizzando fibroblasti primari cutanei e cardiaci17,18. Il passaggio 2D convenzionale dei fibroblasti esofagei provoca cambiamenti sia di morfologia che di fenotipo, dimostrando che l'arricchimento in vitro dei fibroblasti non è adatto per le co-colture che mirano a fenocopiare la nicchia delle cellule staminali endogene.
La colorazione a montaggio intero fornisce uno strumento per mantenere e visualizzare l'interazione fibroblasto-organoide. Va notato che, mentre non tutti gli organoidi avranno fibroblasti direttamente attaccati ad essi, la maggior parte degli organoidi sono in contatto con fibroblasti (vedi Figura 2C). Per mantenere le interazioni epiteliali-fibroblasti, è importante maneggiare gli organoidi con cura ed evitare il pipettaggio vigoroso, il vortice e la rotazione ad alta velocità. La fissazione ottimale è importante per mantenere l'architettura tissutale 3D e mantenere la fluorescenza endogena. Una fissazione di 30 minuti è sufficiente per mantenere il segnale H2BeGFP ed è ottimale per gli anticorpi utilizzati in questo protocollo, tuttavia questo potrebbe variare tra i fluorofori e gli anticorpi utilizzati. La pulizia degli organoidi riduce la dispersione della luce e migliora sostanzialmente la visualizzazione dell'intera struttura 3D. Poiché gli organoidi sono piccoli, la pulizia è facile e veloce; tuttavia, l'imaging di interi organoidi utilizzando la microscopia confocale a scansione laser può richiedere molto tempo, poiché è necessario creare più stack Z. I microscopi confocali, come il disco rotante, possono essere utilizzati per ridurre i tempi di imaging.
Nel complesso, gli organoidi esofagei cresciuti in presenza di fibroblasti forniscono uno strumento prezioso per comprendere aspetti della nicchia delle cellule staminali esofagee. Inoltre, la compensazione dell'intera montatura fornisce un metodo accessibile per visualizzare l'interazione tra fibroblasti e organoidi.
Divulgazioni
Gli autori non dichiarano alcun conflitto di interessi.
Riconoscimenti
Questo studio è stato sostenuto dal CER StG TroyCAN (851241). E.E. è un associato post-dottorato Cancerfonden. M.G. è Ragnar Söderberg Fellow e Cancerfonden Junior Investigator. Siamo grati per l'assistenza tecnica delle strutture principali del Karolinska Institutet, tra cui la Biomedicum Flow Cytometry Core Facility, il Biomedicum Imaging Core (BIC) e la struttura per animali Comparative Medicine Biomedicum (KMB). Ringraziamo i membri del laboratorio Genander per aver attentamente letto e commentato il protocollo.
Materiali
| Name | Company | Catalog Number | Comments |
| B-27 Supplement (50X), serum free | Thermo Fisher (Gibco) | 17504001 | |
| Corning Matrigel Growth Factor Reduced (GFR) Basement Membrane Matrix | fisher scientific | 356231 | |
| Dimethyl sulfoxide | Sigma-Aldrich | 276855-100ML | |
| DMEM/F-12 | Thermo Fisher (Gibco) | 11320074 | |
| DPBS | Thermo Fisher (Gibco) | 14190250 | |
| Fetal Bovine Serum | Sigma-Aldrich | F7524 | |
| GlutaMAX Supplement | Thermo Fisher (Gibco) | 35050061 | |
| HBSS, no calcium, no magnesium, no phenol red | Thermo Fisher (Gibco) | 14175-129 | |
| Normal Donkey Serum | Jackson Immuno | 017-000-121 | |
| Penicillin-Streptomycin (10,000 U/mL) | Thermo Fisher (Gibco) | 15140122 | |
| Triton X-100 solution | Merck | 93443-100ML | |
| Trypsin-EDTA (0.25%), phenol red | Thermo Fisher (Gibco) | 25200-056 | |
| Chemicals, Peptides, and recombinant proteins | |||
| DAPI Solution | Thermo Fisher | 62248 | |
| Dissociation solution: 0.25 mg/ml Liberase TM, 0.25 mg/ml Dnase in HBSS | |||
| Dnase I | Sigma-Aldrich | 11284932001 | |
| Formaldehyde, 37%, with 10-15% methanol | Sigma-Aldrich | 252549-1L | |
| Liberase | Sigma-Aldrich | 5401127001 | |
| N-Acetyl-cysteine | Sigma-Aldrich | A9165-25G | |
| Noggin murine | Peprotech | 250-38 | |
| RapiClear 1.47 | SunJin Lab | #RC147001 | |
| Recombinant Mouse EGF Protein, CF | R&D systems | 2028-EG-200 | |
| R-spondin-1 murine | Peprotech | 315-32 | |
| SYTOX Blue Dead Cell Stain | Thermo Fisher | S34857 | |
| Thermolysin | Sigma-Aldrich | T7902-25MG | |
| Y-27632 dihydrochloride | Sigma-Aldrich | Y0503-5MG | |
| Plastic & Glassware | |||
| Corning Sterile Cell Strainers 40um | VWR | 15360801 | |
| Corning Sterile Cell Strainers 70um | VWR | 431751 | |
| Menzel Deckgläser/ cover slips | Thermo Fisher | Q10143263NR15 | |
| SafeSeal reaction tube, 1.5 mL, PP | Sarstedt | 72.706 | |
| Snap Cap Low Retention Microcentrifuge Tubes 0.6 mL | Thermo Fisher | 3446 | |
| SuperFrost Slides | VWR | 631-9483 | |
| Tools | |||
| 0.05 mm 4 circular well iSpacer | SunJin Lab | #IS204 | |
| Dumont #5 forceps, biology tip | F.S.T | 11252-20 | |
| ImmEdge Pen | VectorLaboratories | H-4000 | |
| Spring Scissors Angled to Side Ball Tip 8mm Cutting Edge | F.S.T | 15033-09 | |
| Instruments | |||
| Confocal microscope Stellaris 5 | Leica | ||
| Dissection microscope ZEISS Stemi 305 | Zeiss | ||
| FACS ARIA III | BD Biosciences | ||
| Conjugated Antibodies for FACS | |||
| Alexa Fluor 647 anti-mouse CD104 Antibody Clone: 346-11A | 123608 | 123608 | |
| APC anti-mouse CD26 (DPP-4) Antibody | H194-112 | H194-112 | |
| PE/Cy7 anti-mouse/human CD324 (E-Cadherin) Antibody | 147310 | 147310 | |
| Antibodies for Immunofluorescence | |||
| CD104 (ß-integrin 4) Clone: 346-11A | BioLegend | 553745 | |
| Cytokeratin 14 | Acris Antibodies (AbD Serotec) | BP5009 | |
| Cytokeratin13 Clone: EPR3671 | abcam | ab92551 | |
| E-cadherin (CD324) Clone: 2.40E+11 | Cell Signaling Technology | 3195 | |
| Keratin 5 Polyclonal Chicken Antibody, Purified Clone: Poly9059 | BioLegend | 905901 | |
| p63 Clone: 4a4 | abcam | ab735 | |
| Recombinant Anti-KLF4 antibod Clone: EPR20753-25 | abcam | ab214666 | |
| Vimentin | Sigma-Aldrich | AB5733 | |
| Secondary antibodies | |||
| Donkey anti-species* antibodies with fluorophore of choice | Jackson Immuno |
Riferimenti
- Sachs, N., et al. Long-term expanding human airway organoids for disease modeling. The EMBO Journal. 38 (4), 100300(2019).
- Lohmussaar, K., et al. Patient-derived organoids model cervical tissue dynamics and viral oncogenesis in cervical cancer. Cell Stem Cell. 28 (8), 1380-1396 (2021).
- Sato, T., et al. Single Lgr5 stem cells build crypt-villus structures in vitro without a mesenchymal niche. Nature. 459 (7244), 262-265 (2009).
- Smukler, S. R., et al. The adult mouse and human pancreas contain rare multipotent stem cells that express insulin. Cell Stem Cell. 8 (3), 281-293 (2011).
- Zheng, B., et al. A new murine esophageal organoid culture method and organoid-based model of esophageal squamous cell neoplasia. iScience. 24 (12), 103440(2021).
- DeWard, A. D., Cramer, J., Lagasse, E. Cellular heterogeneity in the mouse esophagus implicates the presence of a nonquiescent epithelial stem cell population. Cell Reports. 9 (2), 701-711 (2014).
- Kasagi, Y., et al. The esophageal organoid system reveals functional interplay between notch and cytokines in reactive epithelial changes. Cellular and Molecular Gastroenterology and Hepatology. 5 (3), 333-352 (2018).
- Plikus, M. V., et al. Fibroblasts: Origins, definitions, and functions in health and disease. Cell. 184 (15), 3852-3872 (2021).
- McCarthy, N., et al. Distinct mesenchymal cell populations generate the essential intestinal BMP signaling gradient. Cell Stem Cell. 26 (3), 391-402 (2020).
- Cordero-Espinoza, L., et al. Dynamic cell contacts between periportal mesenchyme and ductal epithelium act as a rheostat for liver cell proliferation. Cell Stem Cell. 28 (11), 1907-1921 (2021).
- Pastula, A., et al. Three-dimensional gastrointestinal organoid culture in combination with nerves or fibroblasts: a method to characterize the gastrointestinal stem cell niche. Stem Cells International. 2016, 3710836(2016).
- Hamilton, T. G., Klinghoffer, R. A., Corrin, P. D., Soriano, P. Evolutionary divergence of platelet-derived growth factor alpha receptor signaling mechanisms. Molecular and Cellular Biology. 23 (11), 4013-4025 (2003).
- McGinn, J., et al. A biomechanical switch regulates the transition towards homeostasis in oesophageal epithelium. Nature Cell Biology. 23 (5), 511-525 (2021).
- Zhang, Y., Bailey, D., Yang, P., Kim, E., Que, J. The development and stem cells of the esophagus. Development. 148 (6), (2021).
- Ohlund, D., et al. Distinct populations of inflammatory fibroblasts and myofibroblasts in pancreatic cancer. The Journal of Experimental Medicine. 214 (3), 579-596 (2017).
- Pentinmikko, N., et al. Notum produced by Paneth cells attenuates regeneration of aged intestinal epithelium. Nature. 571 (7765), 398-402 (2019).
- Janson, D., Rietveld, M., Willemze, R., El Ghalbzouri, A. Effects of serially passaged fibroblasts on dermal and epidermal morphogenesis in human skin equivalents. Biogerontology. 14 (2), 131-140 (2013).
- Landry, N. M., Rattan, S. G., Dixon, I. M. C. An improved method of maintaining primary murine cardiac fibroblasts in two-dimensional cell culture. Scientific Reports. 9 (1), 12889(2019).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon