Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Messung lokaler Gewebebelastungen in Sehnen mittels digitaler Open-Source-Bildkorrelation
In diesem Artikel
Zusammenfassung
In diesem Artikel wird ein Open-Source-Algorithmus zur digitalen Bildkorrelationsalgorithmus zur Messung lokaler 2D-Gewebestämme in Sehnenexplantaten beschrieben. Die Genauigkeit der Technik wurde mit mehreren Techniken validiert und steht der Öffentlichkeit zur Verfügung.
Zusammenfassung
Es besteht ein erhebliches wissenschaftliches Interesse daran, die Stämme zu verstehen, denen Sehnenzellen in situ ausgesetzt sind, und wie diese Stämme den Gewebeumbau beeinflussen. Basierend auf diesem Interesse wurden verschiedene Analysetechniken entwickelt, um lokale Gewebestämme innerhalb von Sehnenexplantaten während der Belastung zu messen. In mehreren Fällen wurde jedoch nicht über die Genauigkeit und Empfindlichkeit dieser Techniken berichtet, und keiner der Algorithmen ist öffentlich verfügbar. Dies hat die breitere Messung lokaler Gewebestämme in Sehnenexplantaten erschwert. Ziel dieser Arbeit war es daher, ein validiertes Analysewerkzeug zur Messung lokaler Gewebestämme in Sehnenexplantaten zu entwickeln, das leicht verfügbar und einfach zu bedienen ist. Insbesondere wurde ein öffentlich zugänglicher ALDIC-Algorithmus (Augmented-Lagrangeian Digital Image Correlation) für die Messung von 2D-Dehnungen angepasst, indem die Verschiebungen von Zellkernen innerhalb der Achillessehnen der Maus unter einachsiger Spannung verfolgt wurden. Zusätzlich wurde die Genauigkeit der berechneten Dehnungen durch die Analyse digital transformierter Bilder sowie durch den Vergleich der Dehnungen mit Werten validiert, die mit einer unabhängigen Technik (d.h. photogebleichten Linien) bestimmt wurden. Schließlich wurde eine Technik in den Algorithmus integriert, um das Referenzbild unter Verwendung des berechneten Verschiebungsfeldes zu rekonstruieren, mit dem die Genauigkeit des Algorithmus in Abwesenheit bekannter Dehnungswerte oder einer sekundären Messtechnik beurteilt werden kann. Der Algorithmus ist in der Lage, Dehnungen bis zu 0,1 mit einer Genauigkeit von 0,00015 zu messen. Die Technik zum Vergleich eines rekonstruierten Referenzbildes mit dem tatsächlichen Referenzbild identifizierte erfolgreich Proben, die fehlerhafte Daten enthielten, und zeigte an, dass in Proben mit guten Daten etwa 85% des Verschiebungsfeldes genau waren. Schließlich stimmten die in den Achillessehnen der Maus gemessenen Stämme mit der bisherigen Literatur überein. Daher ist dieser Algorithmus ein sehr nützliches und anpassungsfähiges Werkzeug zur genauen Messung lokaler Gewebebelastungen in Sehnen.
Einleitung
Sehnen sind mechanoempfindliche Gewebe, die sich als Reaktion auf mechanische Belastungen anpassen und degenerieren 1,2,3,4. Aufgrund der Rolle, die mechanische Reize in der Sehnenzellbiologie spielen, besteht ein großes Interesse daran, die Belastungen zu verstehen, denen Sehnenzellen in der nativen Gewebeumgebung während der Belastung ausgesetzt sind. Es wurden verschiedene experimentelle und analytische Techniken entwickelt, um lokale Gewebebelastungen in Sehnen zu messen. Dazu gehören 2D/3D-Analysen der digitalen Bildkorrelation (DIC) von Oberflächendehnungen unter Verwendung von Speckle-Mustern oder photogebleichten Linien (PBLs)5,6,7,8, die Messung der Änderungen des Schwerpunkt-zu-Schwerpunkt-Abstands einzelner Kerne innerhalb des Gewebes9,10 und eine neuere Vollfeld-3D-DIC-Methode, die Bewegungen außerhalb der Ebene und 3D-Verformungen berücksichtigt 11 . Die Genauigkeit und Empfindlichkeit dieser Techniken wurde jedoch nur in wenigen Fällen berichtet, und keine dieser Techniken wurde öffentlich zugänglich gemacht, was die weit verbreitete Einführung und Nutzung dieser Techniken erschwert.
Ziel dieser Arbeit war es, ein validiertes Analysewerkzeug zur Messung lokaler Gewebestämme in Sehnenexplantaten zu schaffen, das leicht verfügbar und einfach zu bedienen ist. Die gewählte Methode basiert auf einem öffentlich zugänglichen ALDIC-Algorithmus (Augmented-Lagrangeian Digital Image Correlation), der in MATLAB geschrieben wurde und von Yang und Bhattacharya12 entwickelt wurde. Dieser Algorithmus wurde für die Analyse von Sehnenproben angepasst und validiert, indem er auf digital transformierte Bilder angewendet und die in tatsächlichen Sehnenproben gemessenen Dehnungen mit den Ergebnissen aus photogebleichten Linien verglichen wurde. Darüber hinaus wurde eine zusätzliche Funktionalität in den Algorithmus implementiert, um die Genauigkeit des berechneten Verschiebungsfeldes auch ohne bekannte Dehnungswerte oder eine sekundäre Messtechnik zu bestätigen. Daher ist dieser Algorithmus ein sehr nützliches und anpassungsfähiges Werkzeug zur genauen Messung lokaler 2D-Gewebestämme in Sehnen.
Protokoll
Diese Studie wurde vom Institutional Animal Care and Use Committee der Pennsylvania State University genehmigt.
1. Gewebevorbereitung
- Für dieses Protokoll werden die Achillessehnen von 2-4 Monate alten männlichen C57BL/6-Mäusen entnommen.
HINWEIS: Es können auch verschiedene Sehnen oder Bänder von Mäusen oder anderen Kleintieren verwendet werden.- Machen Sie einen Schnitt in der Haut oberflächlich zur Achillessehne, um die Plantarissehne und das umgebende Bindegewebe freizulegen. Entfernen Sie sie dann mit einer chirurgischen Klinge.
- Trennen Sie den freiliegenden Musculus soleus und den Musculus gastrocnemius vom Hinterbein und kratzen Sie sie vorsichtig mit der chirurgischen Klinge von der Achillessehne ab
- Trennen Sie das Fersenbein vom Rest des Fußes mit einem Schneidradaufsatz an einem Rotationswerkzeug.
- Färben Sie das Gewebe in 1,5 ml einer 5 μg/ml-Lösung von 5-(4,6-Dichlortriazinyl)aminofluorescein (DTAF) und 0,1 M Natriumbicarbonatpuffer für 20 Minuten auf einem rotierenden Mischer bei Raumtemperatur. Diese Lösung färbt Proteine (z. B. extrazelluläre Matrix) im Gewebe ein.
HINWEIS: Während dieses Zeitraums von 20 Minuten sollte Schritt 1.3 abgeschlossen sein. - Bereiten Sie eine 1:1.000-Lösung von DRAQ5 in phosphatgepufferter Kochsalzlösung (PBS) vor, um die Kerne zu färben. Verwenden Sie einen Wirbelmischer, um die Lösung zu homogenisieren.
- Nach der 20-minütigen Inkubationszeit in Schritt 1.2 wird das Gewebe aus der DTAF-Lösung in die DRAQ5-Lösung überführt und 10 Minuten lang bei Raumtemperatur in einem dunklen Raum inkubiert.
2. Sehnenbelastung und Bildaufnahme
HINWEIS: Dieses Protokoll erfordert eine Zugvorrichtung, die auf einem konfokalen Mikroskop montiert werden kann. Für diese Studie wurde die von Peterson und Szczesny13 beschriebene Mikrozugvorrichtung verwendet.
- Legen Sie die Sehne in die Griffe der Zugbelastungsvorrichtung. Messen Sie vor der Montage der Griffe in der Ladevorrichtung mit digitalen Messschiebern den Abstand zwischen dem Fersenbeinaufsatz und dem gegenüberliegenden Griff. Dieser Abstand ist die Spanngliedlänge.
- Alternativ können Sie die Griffe vor dem Einführen der Sehne in die Ladevorrichtung montieren und in Kontakt drücken, um die Position des Nullhubmotors zu definieren. Die Verschiebung der Motoren nach dem Einsetzen der Sehne könnte zu einer potenziell genaueren Griff-zu-Griff-Messlänge führen.
- Montieren Sie die Griffe in der Ladevorrichtung, die PBS enthält, um die Gewebehydratation aufrechtzuerhalten. Richten Sie die Sehne so gut wie möglich an der x-Achse oder der y-Achse der Mikroskopbilder aus, so dass die x-Dehnungs- und y-Dehnungsausgänge des Algorithmus mit den Sehnenachsen übereinstimmen.
HINWEIS: In dieser Studie wurden die Sehnen auf die x-Achse ausgerichtet. Wenn es nicht möglich ist, die Sehne perfekt mit den Bildachsen auszurichten, können die x-Dehnungs- und y-Dehnungsausgänge des Algorithmus unter Verwendung der Standard-Dehnungstransformationsgleichungen14 so transformiert werden, dass sie sich an den Längs-/Senkrechtachsen der Sehne ausrichten. - Spannglied mit 1 g Spannung vorspannen und, falls gewünscht, zyklisch belastet werden, um die Probe vorzukonditionieren. In diesem Protokoll wurde keine Vorkonditionierung verwendet, da das Studienziel darin bestand, die gemessenen lokalen Gewebestämme zu validieren, anstatt die Gewebematerialeigenschaften zu messen. Wenn Interesse an der Messung der makroskaligen Materialeigenschaften besteht, die von der Belastungshistorie abhängig sind, wird eine Vorkonditionierung empfohlen. Tragen Sie nach der Vorkonditionierung und Erholung erneut eine Vorspannung von 1 g auf.
- Falls gewünscht, photobleichen Sie einen Satz von vier Linien im Abstand von 80 μm im mittleren Bereich des Gewebes (siehe Peterson und Szczesny13 für weitere Details).
HINWEIS: Die photogebleichten Linien wurden verwendet, um die Messungen des ALDIC-Algorithmus zu validieren, und sind für die Durchführung des ALDIC selbst nicht erforderlich. Die Anzahl und der Abstand der Linien können angepasst werden, und die Position der Linien sollte so gewählt werden, dass Artefakte in der Stichprobe vermieden werden, die die Klarheit der Linie verringern würden. - Wiederholen Sie den Photobleaching-Vorgang am linken und rechten Ende des Gewebes in der Nähe der Griffe.
- Nehmen Sie mit dem konfokalen Mikroskop volumetrische Bilder (x,y: 1,25 μm/Pixel, z: 2,5 μm/Pixel) der DTAF- und DRAQ5-Fluoreszenz bei 1 g Vorspannung auf.
- Führen Sie eine Dehnungsrampe mit einer Dehnung von 0,5 %/s bis 2 % durch. Beachten Sie, dass die Dehnungsrate und die inkrementelle Dehnungsgröße angepasst werden können.
- Lassen Sie das Gewebe 10 Minuten lang entspannen.
HINWEIS: Die Dauer der Spannungsrelaxation sollte so gewählt werden, dass die Probe während der Bildaufnahme einer annähernd quasistatischen Belastung ausgesetzt ist. Um zu bestimmen, ob die Dauer der Spannungsrelaxation akzeptabel ist, bestimmen Sie die Steigung der Kraft-Zeit-Kurve während der letzten Minute der Spannungsentspannung (ergänzende Abbildung 1) und multiplizieren Sie diese Steigung mit der gesamten Bilddauer. In dieser Studie änderte sich die Kraft, die bei der größten Dehnungserhöhung ausgeübt wurde, nie um mehr als 5%. - Machen Sie ein weiteres volumetrisches Bild des Gewebes nach der Verformung.
- Wiederholen Sie die Schritte 2.7-2.9, bis die gewünschte Enddehnung erreicht ist. In dieser Arbeit wurde ein endgültiger Dehnungswert von 12% gewählt.
3. Bildverarbeitung
- Verwenden Sie ImageJ oder Fiji, um maximale Z-Projektionen für jedes volumetrische Bild des DRAQ5-Kanals (Kernkanal) zu erstellen. Dies dient als 2D-gesprenkelte Bilder für das ALDIC.
- Speichern Sie die Z-Projektionen mit maximaler Intensität als .tiff-Dateien, und benennen Sie sie gemäß der folgenden Namenskonvention.
- Verwenden Sie eine Zahl als erstes Zeichen des Bildnamens.
- Die Nummer muss der Reihenfolge entsprechen, in der die Bilder bei der Dehnungsanalyse berücksichtigt werden. Beispielsweise sollte das erste Bild mit eins und das zweite Bild mit zwei beginnen. Es können verschiedene Zahlen gewählt werden, die jedoch nacheinander ansteigen müssen. Ein Beispiel für eine Namenskonvention lautet wie folgt: "0_Experiment1_MaxZProjection".
- Speichern Sie alle umbenannten Z-Projektionen mit maximaler Intensität in einem Ordner.
4. Installation und Anwendung des Codes für die Photobleich-Linienanalyse
HINWEIS: Diese Schritte sind nur erforderlich, wenn die Genauigkeit des ALDIC-Algorithmus mithilfe von photogebleichten Linien bestätigt werden soll. Der Code berechnet die lokale Gewebebelastung als die durchschnittliche normalisierte Änderung des Abstands zwischen den einzelnen photogebleichten Linien innerhalb des photogebleichten Liniensatzes. In dieser Studie wurden dann die durchschnittlichen lokalen Werte über alle photogebleichten Liniensätze (d. h. in der Mitte und am linken/rechten Ende) gemittelt, um einen einzigen durchschnittlichen lokalen Gewebedehnungswert für jede Probe zu bestimmen. Dieser Wert wurde dann verwendet, um die Genauigkeit des ALDIC-Algorithmus abzuschätzen.
- Laden Sie den Ordner "PBL Code" von GitHub (https://github.com/Szczesnytendon/TendonStrainCalc) herunter und verschieben Sie den gesamten Inhalt in das Arbeitsverzeichnis in MATLAB.
- Öffnen Sie das MATLAB-Skript "Micro_Mech_Template.m".
- Drücken Sie auf Ausführen und wählen Sie eine der Bilddateien aus, die die volumetrischen Bilder enthalten. Bei den volumetrischen Bildern kann es sich um einen der folgenden Dateitypen handeln: .lsm, .tiff, .nd2.
- Die Software lädt automatisch alle Bilder in den Ordner und zeigt ein projiziertes Bild des volumetrischen Referenzbildes an. Wenn Sie dazu aufgefordert werden, klicken Sie mit der linken Maustaste , um Mehrpunktlinien zu erstellen, die das linke und rechte Ende der Probe nachzeichnen. Klicken Sie mit der rechten Maustaste , um eine Zeile zu beenden. Wenn die Eingabe verarbeitet wurde und die Kanten korrekt sind, drücken Sie OK , um das Ergebnis zu übernehmen.
- Zeichnen Sie eine zufällige diagonale Linie als Referenzlinie über die Stichprobe, wenn Sie dazu aufgefordert werden.
- Geben Sie die Anzahl der erzeugten photogebleichten Linien ein, und verfolgen Sie die photogebleichten Linien mit mehrpunktigen Linien.
- Wenn das Ergebnis akzeptabel ist, akzeptieren Sie es. Wenn das Ergebnis fehlerhaft ist, passen Sie es an und verarbeiten Sie es erneut.
- Wiederholen Sie Schritt 4.2 für alle Bilder und verschieben Sie alle Bilder der nachgezeichneten Linien in einen einzigen Ordner.
- Öffnen Sie das Skript "Micro_Mech_Strain.m".
- Drücken Sie Ausführen , um den Code auszuführen, und wählen Sie eines der gespeicherten Bilder aus, auf denen die photogebleichten Linien nachgezeichnet sind.
- Vergewissern Sie sich, dass die ausgewählten Begleitbilder korrekt sind, sobald das Bild ausgewählt wurde, indem Sie auf OK klicken.
5. Digital transformierte Bilder erstellen
HINWEIS: Diese Schritte sind nur erforderlich, wenn die Genauigkeit des ALDIC-Algorithmus mithilfe digital transformierter Bilder bestätigt werden soll. Diese Bilder simulieren homogene 2D-Dehnungsfelder bekannter Größe, indem sie das Referenzbild künstlich transformieren.
- Laden Sie den Code "Digital_strain.m" von GitHub (https://github.com/Szczesnytendon/TendonStrainCalc) herunter.
- Öffnen Sie den Code, und führen Sie ihn aus.
- Wenn Sie dazu aufgefordert werden, geben Sie die gewünschten Werte für die maximal angelegte Dehnung, das angewendete Dehnungsinkrement und das Poisson-Verhältnis ein. Drücken Sie OK.
ANMERKUNG: Für dieses Experiment betrug die maximal angewandte Dehnung 0,1 (10 %), das angewandte Dehnungsinkrement 0,02 (2 %) und es wurde ein Poisson-Verhältnis von 1 verwendet, das mit den experimentellen Daten des Sehnenzugversuchs15,16 übereinstimmt. Der Code verwendet die eingebettete MATLAB-Funktion imwarp und die Eingabewerte (z. B. Dehnungsinkremente, Poisson-Verhältnis), um die digital transformierten Bilder zu erstellen. - Wenn Sie dazu aufgefordert werden, wählen Sie das unverformte Referenzbild aus.
- Für jedes Dehnungsinkrement wird eine Überlagerung des Referenzbildes und des transformierten Bildes angezeigt. Das transformierte Bild wird im Verzeichnis unter dem Titel "DigitallyTransformedX%Strain" gespeichert, wobei X das Dehnungsinkrement ist.
6. Installation und Anwendung des Dehnungsberechnungs- und Validierungscodes
- Laden Sie den Ordner "Strain Calculation and Validation Code" von GitHub (https://github.com/Szczesnytendon/TendonStrainCalc) herunter und verschieben Sie den gesamten Inhalt in das MATLAB-Arbeitsverzeichnis
- Installieren Sie einen mex C/C++ Compiler nach Yang und Bhattacharya12. Die Schritte sind im Folgenden zusammengefasst.
- Überprüfen Sie MATLAB, um festzustellen, ob ein mex C/C++-Compiler installiert wurde, indem Sie "mex -setup" in das MATLAB-Befehlsfenster eingeben und die Eingabetaste drücken.
- Wenn ein Fehler angezeigt wird, der darauf hinweist, dass ein Compiler nicht unterstützt wird oder nicht vorhanden ist, fahren Sie mit Schritt 6.3 und Schritt 6.4 fort.
- Wenn kein Fehler vorliegt, fahren Sie mit Schritt 6.5 fort
- Um einen mex C/C++-Compiler herunterzuladen, gehen Sie zu "https:/tdm-gcc.tdragon.net/" und wählen Sie den TDM-gcc-Compiler aus.
- Installieren Sie den heruntergeladenen Compiler an einem bekannten Speicherort.
- Kehren Sie zum MATLAB-Befehlsfenster zurück und geben Sie Folgendes ein: "setenv("MW_MINGW64_LOC","[Geben Sie hier Ihren Installationspfad ein]")". Dies könnte z.B. "setenv("MW_MINGW64_LOC","C:\TDM-GCC-64")". Wenn dieser Befehl erfolgreich ausgeführt wird, ist der mex-Compiler ordnungsgemäß installiert.
- Geben Sie das Funktionsskript "main_aldic.m" ein und ändern Sie Zeile 22 so, dass sie mit dem in Schritt 6.5 ausgeführten Befehl übereinstimmt.
- Öffnen Sie das Skript "Strain_calc_and_validate.m".
- Drücken Sie Ausführen , um die Bildanalyse zu starten.
- Wenn Sie dazu aufgefordert werden, ändern Sie die Werte für die ALDIC-Parameter wie gewünscht.
HINWEIS: Die Fenstergröße sollte das 0,25- bis 1-fache der Teilmengengröße betragen. Weitere Informationen zur Parameterauswahl finden Sie im Online-Benutzerhandbuch: (https://www.researchgate.net/publication/344796296_Augmented_Lagrangian_Digital
_Image_Correlation_AL-DIC_Code_Manual).- Die folgenden Werte wurden in dieser Studie verwendet:
Größe der Teilmenge (Pixel): 20
Fenstergröße (Pixel): 10
Methode zur Lösung von ALDIC: Finite Differenz (1)
Paralleles Rechnen wurde nicht verwendet (1)
Methode zur Berechnung der anfänglichen Schätzung: Multigrid-Suche basierend auf der Bildpyramide (0)
- Die folgenden Werte wurden in dieser Studie verwendet:
- Wenn Sie dazu aufgefordert werden, aktivieren Sie das Kontrollkästchen "Ja", damit der Algorithmus automatisch den Mittelwert, die Standardabweichung und die 2D-Karten für die gewünschte Sammlung von Variablen (z. B. x-Dehnung, y-Dehnung, Schubdehnung, fehlerhafte Bereiche usw.) speichert. Wählen Sie aus, welche Variablen gespeichert werden sollen, und drücken Sie OK.
- Wenn Sie dazu aufgefordert werden, ändern Sie die Parameter wie gewünscht.
- Die folgenden Werte wurden in diesem Experiment verwendet:
Umgebungspunkte zur Berechnung der Dehnung (numP): 12
Korrelationskoeffizient für die Identifizierung schlechter Regionen (corr_threshold): 0,5
Größe des Subbereichs (Pixel) für die Analyse fehlerhafter Bereiche (Subsize): 32
- Die folgenden Werte wurden in diesem Experiment verwendet:
- Wenn Sie dazu aufgefordert werden, wählen Sie den Ordner aus, der die umbenannten Z-Projektionen mit maximaler Intensität enthält. Beachten Sie, dass die Software automatisch inkrementelles ALDIC durchführt, um die Dehnungsfelder der verformten Bilder zu bestimmen. Das heißt, jedes deformierte Bild dient als neues "Referenz"-Bild für das nächste deformierte Bild. Dies verbessert die Genauigkeit der Ergebnisse (ergänzende Abbildung 2) im Vergleich zur Durchführung von kumulativem ALDIC, bei dem jedes deformierte Bild mit dem ursprünglichen Referenzbild (0% Dehnung) verglichen wird. Um eine kumulative Analyse durchzuführen, laden Sie die Bilder, wählen Sie jedoch nur das ursprüngliche Referenzbild und das deformierte Bild aus, das Sie interessieren.
HINWEIS: Die normale Dehnung wird als λ - 1 berechnet, wobei λ die Gewebedehnung ist. Die Gewebedehnung wird nach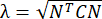 berechnet, wobei N = [1 0]T oder [0 1]T für die x-Richtung bzw. y-Richtung und C = F T F ist, wobei F der Verformungsgradient ist, der unter Verwendung von "numP"-Punkten berechnet wird, die jeden vom ALDIC-Algorithmus ausgegebenen Datenpunkt umgeben. Die Schubdehnung wird berechnet als
berechnet, wobei N = [1 0]T oder [0 1]T für die x-Richtung bzw. y-Richtung und C = F T F ist, wobei F der Verformungsgradient ist, der unter Verwendung von "numP"-Punkten berechnet wird, die jeden vom ALDIC-Algorithmus ausgegebenen Datenpunkt umgeben. Die Schubdehnung wird berechnet als 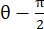 , wobei
, wobei 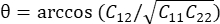 .
. - Wenn Sie dazu aufgefordert werden, klicken Sie mit der linken Maustaste, um ein Vier-Punkt-Polygon zu erstellen, um den Bereich zu definieren, der für die Messung der Dehnungen von Interesse ist. Beginnen Sie mit dem Punkt in der oberen linken Ecke, und weisen Sie die nachfolgenden Punkte im Uhrzeigersinn zu.
HINWEIS: Die im MATLAB-Arbeitsbereich gespeicherte Variable "Storage" enthält alle Werte für die durchschnittliche x-Dehnung, die x-Dehnungsstandardabweichung, die durchschnittliche y-Dehnung, die y-Dehnungsstandardabweichung, die durchschnittliche Scherdehnung, die Scherdehnungsstandardabweichung und den Prozentsatz der fehlerhaften Bereiche. Die schlechten Regionen werden gemäß der Korrelationskoeffizientenanalyse innerhalb der in Schritt 6.13 ausgewählten Region von Interesse definiert. Der Ordner "NuclearTrackingResults" (der durch Anpassen der Zeilen 555 und 556 umbenannt werden kann) speichert alle in Schritt 6.10 angegebenen Plots. Dieser Ordner enthält auch eine Tabellenkalkulationsdatei mit dem Namen "Ergebnisse", in der alle in Schritt 6.10 angegebenen Mittelwerte und Standardabweichungen gespeichert sind.
Ergebnisse
Vor der Analyse der Dehnungsfelder in realen Gewebeproben wurde das ALDIC-Protokoll zunächst anhand von digital gespannten/transformierten Bildern von Kernen in den Achillessehnen der Maus validiert. Insbesondere wurden die Bilder transformiert, um digitale gleichmäßige Dehnungen in x-Richtung von 2 %, 4 %, 6 %, 8 % und 10 % Dehnung mit einem simulierten Poisson-Verhältnis von 115,16 zu erzeugen. Die Genauigkeit des ALDIC-Algorithmus wurde dann bewertet, inde...
Diskussion
Das Ziel dieser Arbeit war es, eine quelloffene, validierte Methode zur Messung der 2D-Dehnungsfelder in Spanngliedern unter Zugbelastung bereitzustellen. Das Fundament der Software basierte auf einem öffentlich zugänglichen ALDIC-Algorithmus12. Dieser Algorithmus wurde in einen größeren MATLAB-Code eingebettet, der die Funktionalität einer inkrementellen (im Vergleich zur kumulativen) Dehnungsanalyse umfasste. Dieser angepasste Algorithmus wurde dann auf den Zugversuch von Spanngliedern ange...
Offenlegungen
Alle Autoren haben keine Interessenkonflikte offenzulegen.
Danksagungen
Diese Arbeit wurde von den National Institutes of Health (R21 AR079095) und der National Science Foundation (2142627) finanziert.
Materialien
| Name | Company | Catalog Number | Comments |
| 5-DTAF (5-(4,6-Dichlorotriazinyl) Aminofluorescein), single isomer | ThermoFisher | D16 | |
| Calipers | Mitutoyo | 500-196-30 | |
| Confocal Microscope | Nikon | A1R HD | |
| Corning LSE Vortex Mixer | Coning | 6775 | |
| DRAQ5 Fluorescent Probe Solution (5 mM) | ThermoFisher | 62554 | |
| MATLAB | MathWorks | R2022b | |
| Tensile Loading Device | N/A | N/A | Tensile loading device described in Peterson et al, 2020. (ref 13) |
| Tube Revolver Rotator | ThermoFisher | 88881001 |
Referenzen
- Devkota, A. C. Distributing a fixed amount of cyclic loading to tendon explants over longer periods induces greater cellular and mechanical responses. Journal of Orthopaedic Research. 11 (4), 1609-1612 (2007).
- Sun, H. B., et al. Cycle-dependent matrix remodeling gene expression response in fatigue-loaded rat patellar tendons. Journal of Orthopaedic Research. 28 (10), 1380-1386 (2010).
- Shepherd, J. H., Screen, H. R. C. Fatigue loading of tendon. International Journal of Experimental Pathology. 94 (4), 260-270 (2013).
- Paschall, L., Pedaprolu, K., Carrozzi, S., Dhawan, A., Szczesny, S. Mechanical stimulation as both the cause and the cure of tendon and ligament injuries. Regenerative Rehabilitation: From Basic Science to the Clinic. , 359-386 (2022).
- Andarawis-Puri, N., Ricchetti, E. T., Soslowsky, L. J. Rotator cuff tendon strain correlates with tear propagation. Journal of Biomechanics. 42 (2), 158-163 (2009).
- Cheng, V. W. T., Screen, H. R. C. The micro-structural strain response of tendon. Journal of Materials Science. 42 (21), 8957-8965 (2007).
- Luyckx, T., et al. Digital image correlation as a tool for three-dimensional strain analysis in human tendon tissue. Journal of Experimental Orthopaedics. 1 (1), 7 (2014).
- Duncan, N. A., Bruehlmann, S. B., Hunter, C. J., Shao, X., Kelly, E. J. In situ cell-matrix mechanics in tendon fascicles and seeded collagen gels: Implications for the multiscale design of biomaterials. Computer Methods in Biomechanics and Biomedical Engineering. 17 (1), 39-47 (2014).
- Arnoczky, S. P., Lavagnino, M., Whallon, J. H., Hoonjan, A. In situ cell nucleus deformation in tendons under tensile load; A morphological analysis using confocal laser microscopy. Journal of Orthopaedic Research. 20 (1), 29-35 (2002).
- Screen, H. R. C., Bader, D. L., Lee, D. A., Shelton, J. C. Local strain measurement within tendon. Strain. 40 (4), 157-163 (2004).
- Fung, A. K., Paredes, J. J., Andarawis-Puri, N. Novel image analysis methods for quantification of in situ 3-D tendon cell and matrix strain. Journal of Biomechanics. 67, 184-189 (2018).
- Yang, J., Bhattacharya, K. Augmented Lagrangian digital image correlation. Experimental Mechanics. 59 (2), 187-205 (2019).
- Peterson, B. E., Szczesny, S. E. Dependence of tendon multiscale mechanics on sample gauge length is consistent with discontinuous collagen fibrils. Acta Biomaterialia. 117, 302-309 (2020).
- Humphrey, J. D., O'Rourke, S. L. . An Introduction to Biomechanics. , (2015).
- Reese, S. P., Weiss, J. A. Tendon fascicles exhibit a linear correlation between Poisson's ratio and force during uniaxial stress relaxation. Journal of Biomechanical Engineering. 135 (3), 34501 (2013).
- Ahmadzadeh, H., Freedman, B. R., Connizzo, B. K., Soslowsky, L. J., Shenoy, V. B. Micromechanical poroelastic finite element and shear-lag models of tendon predict large strain dependent Poisson's ratios and fluid expulsion under tensile loading. Acta Biomaterialia. 22, 83-91 (2015).
- Szczesny, S. E., Elliott, D. M. Interfibrillar shear stress is the loading mechanism of collagen fibrils in tendon. Acta Biomaterialia. 10 (6), 2582-2590 (2014).
- Han, W. M., et al. Macro- to microscale strain transfer in fibrous tissues is heterogeneous and tissue-specific. Biophysical Journal. 105 (3), 807-817 (2013).
- Pedaprolu, K., Szczesny, S. E. A novel, open-source, low-cost bioreactor for load-controlled cyclic loading of tendon explants. Journal of Biomechanical Engineering. 144 (8), 084505 (2022).
- Gatt, R., et al. Negative Poisson's ratios in tendons: An unexpected mechanical response. Acta Biomaterialia. 24, 201-208 (2015).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten