Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Mesure des souches tissulaires locales dans les tendons via une corrélation d’images numériques open source
Dans cet article
Résumé
Cet article décrit un algorithme de corrélation d’images numériques open source pour mesurer les souches tissulaires 2D locales dans les explants de tendons. La précision de la technique a été validée à l’aide de plusieurs techniques, et elle est disponible pour un usage public.
Résumé
Il existe un intérêt scientifique considérable pour comprendre les souches que les cellules tendineuses subissent in situ et comment ces souches influencent le remodelage tissulaire. Sur la base de cet intérêt, plusieurs techniques analytiques ont été développées pour mesurer les déformations tissulaires locales dans les explants de tendons pendant la charge. Cependant, dans plusieurs cas, l’exactitude et la sensibilité de ces techniques n’ont pas été rapportées, et aucun des algorithmes n’est accessible au public. Cela a rendu difficile la mesure plus généralisée des souches tissulaires locales dans les explants de tendons. Par conséquent, l’objectif de cet article était de créer un outil d’analyse validé pour mesurer les souches tissulaires locales dans les explants de tendons, facilement disponible et facile à utiliser. Plus précisément, un algorithme de corrélation d’images numériques de Lagrangien augmentée (ALDIC) accessible au public a été adapté pour mesurer les souches 2D en suivant les déplacements des noyaux cellulaires dans les tendons d’Achille de souris sous tension uniaxiale. De plus, la précision des souches calculées a été validée en analysant des images transformées numériquement, ainsi qu’en comparant les souches avec des valeurs déterminées à partir d’une technique indépendante (c.-à-d. des lignes photoblanchies). Enfin, une technique a été incorporée dans l’algorithme pour reconstruire l’image de référence à l’aide du champ de déplacement calculé, qui peut être utilisé pour évaluer la précision de l’algorithme en l’absence de valeurs de déformation connues ou d’une technique de mesure secondaire. L’algorithme est capable de mesurer des déformations jusqu’à 0,1 avec une précision de 0,00015. La technique de comparaison d’une image de référence reconstruite avec l’image de référence réelle a permis d’identifier les échantillons qui avaient des données erronées et a indiqué que, dans les échantillons avec de bonnes données, environ 85% du champ de déplacement était précis. Enfin, les souches mesurées dans les tendons d’Achille de souris étaient cohérentes avec la littérature antérieure. Par conséquent, cet algorithme est un outil très utile et adaptable pour mesurer avec précision les souches tissulaires locales dans les tendons.
Introduction
Les tendons sont des tissus mécanosensibles qui s’adaptent et dégénèrent en réponse à la charge mécanique 1,2,3,4. En raison du rôle que jouent les stimuli mécaniques dans la biologie des cellules tendineuses, il existe un grand intérêt pour la compréhension des souches que les cellules tendineuses subissent dans l’environnement tissulaire natif pendant le chargement. Plusieurs techniques expérimentales et analytiques ont été développées pour mesurer les souches tissulaires locales dans les tendons. Il s’agit notamment d’analyses de corrélation d’images numériques (DIC) 2D/3D de déformations de surface utilisant des motifs de mouchetures ou des lignes photoblanchies (PBL)5,6,7,8, la mesure des changements dans la distance centroïde-centroïde des noyaux individuels dans le tissu 9,10, et une méthode DIC 3D en champ complet récente qui tient compte du mouvement hors plan et des déformations 3D 11 . Cependant, l’exactitude et la sensibilité de ces techniques n’ont été signalées que dans quelques cas, et aucune de ces techniques n’a été rendue publique, ce qui rend difficile l’adoption et l’utilisation généralisées de ces techniques.
L’objectif de ce travail était de créer un outil d’analyse validé pour mesurer les souches tissulaires locales dans les explants de tendons, facilement disponible et facile à utiliser. La méthode choisie est basée sur un algorithme de corrélation d’images numériques en lagrangienne augmentée (ALDIC) accessible au public écrit en MATLAB et développé par Yang et Bhattacharya12. Cet algorithme a été adapté pour analyser des échantillons de tendons et validé en l’appliquant à des images transformées numériquement et en comparant les déformations mesurées dans des échantillons de tendons réels aux résultats obtenus à partir de lignes photoblanchies. En outre, des fonctionnalités supplémentaires ont été implémentées dans l’algorithme pour confirmer la précision du champ de déplacement calculé même en l’absence de valeurs de déformation connues ou d’une technique de mesure secondaire. Par conséquent, cet algorithme est un outil très utile et adaptable pour mesurer avec précision les souches tissulaires 2D locales dans les tendons.
Protocole
Cette étude a été approuvée par le Pennsylvania State University Institutional Animal Care and Use Committee.
1. Préparation des tissus
- Pour ce protocole, récoltez les tendons d’Achille de souris mâles C57BL/6 âgées de 2 à 4 mois.
REMARQUE: Différents tendons ou ligaments de souris ou d’autres petits animaux peuvent également être utilisés.- Faites une incision à la peau superficielle au tendon d’Achille pour exposer le tendon plantaire et le tissu conjonctif environnant. Ensuite, retirez-les à l’aide d’une lame chirurgicale.
- Séparez les muscles soléaires et gastrocnémiens exposés du membre postérieur et grattez-les soigneusement du tendon d’Achille avec la lame chirurgicale
- Séparez le calcanéum du reste du pied avec une attache de roue de coupe sur un outil rotatif.
- Colorer le tissu dans 1,5 mL d’une solution de 5 μg/mL de 5-(4,6-dichlorotriazinyl)aminofluorescéine (DTAF) et de tampon de bicarbonate de sodium 0,1 M pendant 20 min sur un mélangeur rotatif à température ambiante. Cette solution colore les protéines (par exemple, la matrice extracellulaire) dans le tissu.
REMARQUE : Au cours de cette période de 20 minutes, l’étape 1.3 doit être complétée. - Préparer une solution 1:1 000 de DRAQ5 dans une solution saline tamponnée au phosphate (PBS) pour colorer les noyaux. Utilisez un mélangeur vortex pour homogénéiser la solution.
- Après la période d’incubation de 20 minutes à l’étape 1.2, transférer le tissu de la solution DTAF à la solution DRAQ5 et incuber dans un espace sombre pendant 10 minutes à température ambiante.
2. Chargement des tendons et acquisition d’images
REMARQUE: Ce protocole nécessite un dispositif de traction qui peut être monté sur un microscope confocal. Pour cette étude, le dispositif de microtraction décrit par Peterson et Szczesny13 a été utilisé.
- Placer le tendon dans les poignées du dispositif de charge de traction. Avant de monter les poignées dans le dispositif de chargement, utilisez des étriers numériques pour mesurer la distance entre l’attache calcanéum et la poignée opposée. Cette distance est la longueur de la jauge du tendon.
- Vous pouvez également monter les poignées dans le dispositif de chargement avant d’insérer le tendon et pousser en contact pour définir la position du moteur à déplacement zéro. Le déplacement des moteurs après l’insertion du tendon pourrait fournir une longueur de jauge de poignée à poignée potentiellement plus précise.
- Montez les poignées dans le dispositif de chargement, qui contient du PBS pour maintenir l’hydratation des tissus. Aligner le tendon le mieux possible avec l’axe x ou l’axe y des images du microscope afin que les sorties de déformation x et de déformation y de l’algorithme correspondent aux axes tendineux.
REMARQUE: Dans cette étude, les tendons étaient alignés avec l’axe des abscisses. S’il n’est pas possible d’aligner parfaitement le tendon avec les axes d’image, les sorties de déformation x et de déformation y de l’algorithme peuvent être transformées pour s’aligner sur les axes longitudinal/perpendiculaire du tendon à l’aide d’équations de transformation de déformation standard14. - Précharger le tendon avec 1 g de tension et, si désiré, appliquer une charge cyclique pour préconditionner l’échantillon. Dans ce protocole, aucun préconditionnement n’a été utilisé puisque l’objectif de l’étude était de valider les souches tissulaires locales mesurées plutôt que de mesurer les propriétés du matériau tissulaire. S’il y a un intérêt à mesurer les propriétés du matériau à l’échelle macroscopique, qui dépendent de l’historique de chargement, le préconditionnement est recommandé. Après le préconditionnement et la récupération, réappliquer une précharge de 1 g.
- Si vous le souhaitez, photoblanchir un ensemble de quatre lignes espacées de 80 μm dans la région centrale du tissu (voir Peterson et Szczesny13 pour plus de détails).
REMARQUE: Les lignes photoblanchies ont été utilisées pour valider les mesures de l’algorithme ALDIC et ne sont pas nécessaires pour effectuer l’ALDIC lui-même. Le nombre et l’espacement des lignes peuvent être ajustés, et l’emplacement des lignes doit être choisi pour éviter tout artefact dans l’échantillon qui diminuerait la clarté de la ligne. - Répétez la procédure de photoblanchiment aux extrémités gauche et droite du tissu près des poignées.
- À l’aide du microscope confocal, acquérir des images volumétriques (x,y : 1,25 μm/pixel, z : 2,5 μm/pixel) de la fluorescence DTAF et DRAQ5 à 1 g de précharge.
- Effectuer une rampe de déformation à 0,5 %/s à 2 % de déformation. Notez que la vitesse de déformation et l’amplitude de déformation incrémentielle peuvent être ajustées.
- Laissez le tissu stresser se détendre pendant 10 min.
REMARQUE : La durée de relaxation des contraintes doit être choisie de telle sorte que l’échantillon soit soumis à une charge quasi quasi statique pendant l’acquisition de l’image. Pour déterminer si la durée de relaxation des contraintes est acceptable, déterminez la pente de la courbe force-temps pendant la dernière minute de relaxation des contraintes (figure supplémentaire 1) et multipliez cette pente par la durée totale d’imagerie. Dans cette étude, la force appliquée au plus grand incrément de déformation n’a jamais changé de plus de 5%. - Prenez une autre image volumétrique du tissu après déformation.
- Répétez les étapes 2.7-2.9 jusqu’à ce que la contrainte finale souhaitée soit atteinte. Dans cet article, une valeur de déformation finale de 12 % a été choisie.
3. Traitement de l’image
- Utilisez ImageJ ou Fiji pour créer des projections z maximales de chaque image volumétrique du canal DRAQ5 (nucléaire). Cela servira d’images mouchetées 2D pour l’ALDIC.
- Enregistrez les projections z d’intensité maximale en tant que fichiers .tiff et nommez-les selon la convention d’affectation de noms suivante.
- Utilisez un nombre comme premier caractère du nom de l’image.
- Faites correspondre le numéro à l’ordre dans lequel les images seront prises en compte lors de l’analyse de la déformation. Par exemple, la première image doit commencer par un, et la seconde image doit commencer par deux. Différents nombres peuvent être choisis, mais ils doivent augmenter séquentiellement. Voici un exemple de convention de nommage : « 0_Experiment1_MaxZProjection ».
- Enregistrez toutes les projections z d’intensité maximale renommées dans un dossier.
4. Installation et application du code d’analyse de ligne photoblanchie
REMARQUE: Ces étapes ne sont nécessaires que si l’on souhaite confirmer l’exactitude de l’algorithme ALDIC à l’aide de lignes photoblanchies. Le code calcule la souche tissulaire locale comme le changement moyen normalisé de distance entre chaque ligne photoblanchie dans l’ensemble de lignes photoblanchies. Dans cette étude, les valeurs locales moyennes ont ensuite été moyennées pour tous les ensembles de lignes photoblanchies (c.-à-d. au centre et aux extrémités gauche / droite) afin de déterminer une seule valeur moyenne de souche tissulaire locale pour chaque échantillon. Cette valeur a ensuite été utilisée pour estimer la précision de l’algorithme ALDIC.
- Téléchargez le dossier « PBL Code » depuis GitHub (https://github.com/Szczesnytendon/TendonStrainCalc) et déplacez tout le contenu vers le répertoire de travail dans MATLAB.
- Ouvrez le script MATLAB « Micro_Mech_Template.m ».
- Appuyez sur Exécuter, puis sélectionnez l’un des fichiers image contenant les images volumétriques. Les images volumétriques peuvent être de l’un des types de fichiers suivants : .lsm, .tiff, .nd2.
- Le logiciel chargera automatiquement toutes les images du dossier et affichera une image projetée de l’image volumétrique de référence. Lorsque vous y êtes invité, cliquez avec le bouton gauche de la souris pour créer des lignes multipoints qui tracent les extrémités gauche et droite de l’exemple. Cliquez avec le bouton droit pour terminer une ligne. Une fois l’entrée traitée, si les bords sont corrects, appuyez sur OK pour accepter le résultat.
- Tracez une diagonale aléatoire sur l’échantillon comme ligne de référence lorsque vous y êtes invité.
- Entrez le nombre de lignes photoblanchies créées et tracez les lignes photoblanchies avec des lignes multipoints.
- Si le résultat est acceptable, acceptez-le. Si le résultat est erroné, ajustez-le et retraitez-le.
- Répétez l’étape 4.2 pour toutes les images et déplacez toutes les images des lignes tracées dans un seul dossier.
- Ouvrez le script « Micro_Mech_Strain.m ».
- Appuyez sur Exécuter pour exécuter le code et sélectionnez l’une des images enregistrées où les lignes photoblanchies sont tracées.
- Vérifiez que les images d’accompagnement sélectionnées sont correctes une fois l’image sélectionnée en appuyant sur OK.
5. Créer des images transformées numériquement
REMARQUE: Ces étapes ne sont nécessaires que si l’on souhaite confirmer l’exactitude de l’algorithme ALDIC à l’aide d’images transformées numériquement. Ces images simulent des champs de déformation 2D homogènes d’une ampleur connue en transformant artificiellement l’image de référence.
- Téléchargez le code « Digital_strain.m » depuis GitHub (https://github.com/Szczesnytendon/TendonStrainCalc).
- Ouvrez et exécutez le code.
- Lorsque vous y êtes invité, insérez les valeurs souhaitées pour la déformation maximale appliquée, l’incrément de déformation appliquée et le rapport de Poisson. Appuyez sur OK.
REMARQUE : Pour cette expérience, la déformation maximale appliquée était de 0,1 (10 %), l’augmentation de la déformation appliquée était de 0,02 (2 %) et un rapport de Poisson de 1 a été utilisé, ce qui est cohérent avec les données expérimentales des essais de traction tendineux15,16. Le code utilise la fonction MATLAB intégrée imwarp et les valeurs d’entrée (par exemple, les incréments de déformation, le rapport de Poisson) pour créer les images transformées numériquement. - Lorsque vous y êtes invité, sélectionnez l’image de référence non déformée.
- Pour chaque incrément de déformation, une superposition de l’image de référence et de l’image transformée est affichée. L’image transformée sera enregistrée dans le répertoire sous le titre « DigitallyTransformedX%Strain », où X est l’incrément de déformation.
6. Installation et application du code de calcul et de validation des contraintes
- Téléchargez le dossier « Strain Calculation and Validation Code » depuis GitHub (https://github.com/Szczesnytendon/TendonStrainCalc) et déplacez tout le contenu vers le répertoire de travail MATLAB
- Installez un compilateur C/C++ mex selon Yang et Bhattacharya12. Les étapes sont résumées ci-dessous.
- Vérifiez MATLAB pour voir si un compilateur C/C++ mex a été installé en tapant « mex -setup » dans la fenêtre de commande MATLAB et en appuyant sur Entrée.
- Si une erreur s’affiche indiquant qu’un compilateur n’est pas pris en charge ou présent, passez aux étapes 6.3 et 6.4.
- Si aucune erreur n’est présente, passez à l’étape 6.5
- Pour télécharger un compilateur C/C++ mex, allez dans « https:/tdm-gcc.tdragon.net/ », et choisissez le compilateur TDM-gcc.
- Installez le compilateur téléchargé à un emplacement connu.
- Revenez à la fenêtre de commande MATLAB et tapez : « setenv(« MW_MINGW64_LOC »,"[Saisissez votre chemin d’installation ici] ») ». Par exemple, cela pourrait être « setenv(« MW_MINGW64_LOC »,"C:\TDM-GCC-64 ») ». Si cette commande s’exécute correctement, le compilateur mex est correctement installé.
- Entrez le script de fonction « main_aldic.m » et modifiez la ligne 22 pour qu’elle corresponde à la commande exécutée à l’étape 6.5.
- Ouvrez le script « Strain_calc_and_validate.m ».
- Appuyez sur Exécuter pour commencer l’analyse de l’image.
- Lorsque vous y êtes invité, modifiez les valeurs des paramètres ALDIC comme vous le souhaitez.
Remarque : La taille de la fenêtre doit être 0,25 à 1 fois la taille du sous-ensemble. Pour plus d’informations sur les choix de paramètres, reportez-vous au manuel d’utilisation en ligne : (https://www.researchgate.net/publication/344796296_Augmented_Lagrangian_Digital
_Image_Correlation_AL à DIC_Code_Manual).- Les valeurs suivantes ont été utilisées dans cette étude :
Taille du sous-ensemble (pixels): 20
Taille de la fenêtre (pixels): 10
Méthode pour résoudre ALDIC: différence finie (1)
Le calcul parallèle n’a pas été utilisé (1)
Méthode de calcul de l’estimation initiale: recherche multigrille basée sur la pyramide d’images (0)
- Les valeurs suivantes ont été utilisées dans cette étude :
- Lorsque vous y êtes invité, cochez la case « Oui » pour que l’algorithme enregistre automatiquement la valeur moyenne, l’écart-type et les cartes 2D pour la collection de variables souhaitée (par exemple, déformation x, déformation y, déformation de cisaillement, régions défectueuses, etc.). Sélectionnez les variables à enregistrer, puis appuyez sur OK.
- Lorsque vous y êtes invité, modifiez les paramètres comme vous le souhaitez.
- Les valeurs suivantes ont été utilisées dans cette expérience :
Points environnants pour calculer la déformation (numP): 12
Coefficient de corrélation pour l’identification des régions défectueuses (corr_threshold) : 0,5
Taille de la sous-région (pixels) pour l’analyse des régions incorrectes (sous-taille) : 32
- Les valeurs suivantes ont été utilisées dans cette expérience :
- Lorsque vous y êtes invité, sélectionnez le dossier qui contient les projections z d’intensité maximale renommées. Notez que le logiciel effectue automatiquement un ALDIC incrémentiel pour déterminer les champs de déformation des images déformées. C’est-à-dire que chaque image déformée sert de nouvelle image « de référence » pour l’image déformée suivante. Cela améliore la précision des résultats (figure supplémentaire 2) par rapport à l’exécution d’ALDIC cumulatifs, où chaque image déformée est comparée à l’image de référence originale (déformation de 0%). Pour effectuer une analyse cumulative, chargez les images mais sélectionnez uniquement l’image de référence d’origine et l’image déformée qui vous intéresse.
NOTE: La déformation normale est calculée comme λ - 1, où λ est l’étirement tissulaire. L’étirement tissulaire est calculé selon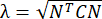 , où N = [1 0]T ou [0 1]T pour la direction x et la direction y, respectivement, et C = F T F, où F est le gradient de déformation calculé à l’aide de points « numP » entourant chaque sortie de point de données par l’algorithme ALDIC. La déformation de cisaillement est calculée comme suit
, où N = [1 0]T ou [0 1]T pour la direction x et la direction y, respectivement, et C = F T F, où F est le gradient de déformation calculé à l’aide de points « numP » entourant chaque sortie de point de données par l’algorithme ALDIC. La déformation de cisaillement est calculée comme suit 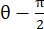 : , où
: , où 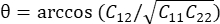 .
. - Lorsque vous y êtes invité, cliquez avec le bouton gauche de la souris pour créer un polygone à quatre points afin de définir la région d’intérêt pour mesurer les déformations. Commencez par le point dans le coin supérieur gauche et attribuez les points suivants dans le sens des aiguilles d’une montre.
REMARQUE : la variable « Storage » enregistrée dans l’espace de travail MATLAB contient toutes les valeurs de la déformation x moyenne, de l’écart type de la déformation x, de la déformation y moyenne, de l’écart type de la déformation y, de la déformation de cisaillement moyenne, de l’écart type de la déformation de cisaillement et du pourcentage de régions défectueuses. Les régions défectueuses sont définies selon l’analyse du coefficient de corrélation au sein de la région d’intérêt sélectionnée à l’étape 6.13. Le dossier « NuclearTrackingResults » (qui peut être renommé en ajustant les lignes 555 et 556) stocke tous les tracés spécifiés à l’étape 6.10. Ce dossier contient également un fichier de feuille de calcul nommé « Résultats », qui stocke toutes les moyennes et les écarts-types spécifiés à l’étape 6.10.
Résultats
Avant d’analyser les champs de déformation dans des échantillons de tissus réels, le protocole ALDIC a d’abord été validé à l’aide d’images déformées / transformées numériquement de noyaux dans les tendons d’Achille de souris. Plus précisément, les images ont été transformées pour produire numériquement des déformations uniformes dans la direction x de 2%, 4%, 6%, 8% et 10% de déformation avec un rapport de Poisson simulé de 115,16. ...
Discussion
L’objectif de cet article était de fournir une méthode open source validée pour mesurer les champs de déformation 2D dans les tendons sous charge de traction. La base du logiciel était basée sur un algorithme ALDIC accessible au public12. Cet algorithme a été intégré dans un code MATLAB plus volumineux avec la fonctionnalité supplémentaire d’analyse de contrainte incrémentielle (par opposition à cumulative). Cet algorithme adapté a ensuite été appliqué à l’essai de tractio...
Déclarations de divulgation
Tous les auteurs n’ont aucun conflit d’intérêts à divulguer.
Remerciements
Ce travail a été financé par les National Institutes of Health (R21 AR079095) et la National Science Foundation (2142627).
matériels
| Name | Company | Catalog Number | Comments |
| 5-DTAF (5-(4,6-Dichlorotriazinyl) Aminofluorescein), single isomer | ThermoFisher | D16 | |
| Calipers | Mitutoyo | 500-196-30 | |
| Confocal Microscope | Nikon | A1R HD | |
| Corning LSE Vortex Mixer | Coning | 6775 | |
| DRAQ5 Fluorescent Probe Solution (5 mM) | ThermoFisher | 62554 | |
| MATLAB | MathWorks | R2022b | |
| Tensile Loading Device | N/A | N/A | Tensile loading device described in Peterson et al, 2020. (ref 13) |
| Tube Revolver Rotator | ThermoFisher | 88881001 |
Références
- Devkota, A. C. Distributing a fixed amount of cyclic loading to tendon explants over longer periods induces greater cellular and mechanical responses. Journal of Orthopaedic Research. 11 (4), 1609-1612 (2007).
- Sun, H. B., et al. Cycle-dependent matrix remodeling gene expression response in fatigue-loaded rat patellar tendons. Journal of Orthopaedic Research. 28 (10), 1380-1386 (2010).
- Shepherd, J. H., Screen, H. R. C. Fatigue loading of tendon. International Journal of Experimental Pathology. 94 (4), 260-270 (2013).
- Paschall, L., Pedaprolu, K., Carrozzi, S., Dhawan, A., Szczesny, S. Mechanical stimulation as both the cause and the cure of tendon and ligament injuries. Regenerative Rehabilitation: From Basic Science to the Clinic. , 359-386 (2022).
- Andarawis-Puri, N., Ricchetti, E. T., Soslowsky, L. J. Rotator cuff tendon strain correlates with tear propagation. Journal of Biomechanics. 42 (2), 158-163 (2009).
- Cheng, V. W. T., Screen, H. R. C. The micro-structural strain response of tendon. Journal of Materials Science. 42 (21), 8957-8965 (2007).
- Luyckx, T., et al. Digital image correlation as a tool for three-dimensional strain analysis in human tendon tissue. Journal of Experimental Orthopaedics. 1 (1), 7 (2014).
- Duncan, N. A., Bruehlmann, S. B., Hunter, C. J., Shao, X., Kelly, E. J. In situ cell-matrix mechanics in tendon fascicles and seeded collagen gels: Implications for the multiscale design of biomaterials. Computer Methods in Biomechanics and Biomedical Engineering. 17 (1), 39-47 (2014).
- Arnoczky, S. P., Lavagnino, M., Whallon, J. H., Hoonjan, A. In situ cell nucleus deformation in tendons under tensile load; A morphological analysis using confocal laser microscopy. Journal of Orthopaedic Research. 20 (1), 29-35 (2002).
- Screen, H. R. C., Bader, D. L., Lee, D. A., Shelton, J. C. Local strain measurement within tendon. Strain. 40 (4), 157-163 (2004).
- Fung, A. K., Paredes, J. J., Andarawis-Puri, N. Novel image analysis methods for quantification of in situ 3-D tendon cell and matrix strain. Journal of Biomechanics. 67, 184-189 (2018).
- Yang, J., Bhattacharya, K. Augmented Lagrangian digital image correlation. Experimental Mechanics. 59 (2), 187-205 (2019).
- Peterson, B. E., Szczesny, S. E. Dependence of tendon multiscale mechanics on sample gauge length is consistent with discontinuous collagen fibrils. Acta Biomaterialia. 117, 302-309 (2020).
- Humphrey, J. D., O'Rourke, S. L. . An Introduction to Biomechanics. , (2015).
- Reese, S. P., Weiss, J. A. Tendon fascicles exhibit a linear correlation between Poisson's ratio and force during uniaxial stress relaxation. Journal of Biomechanical Engineering. 135 (3), 34501 (2013).
- Ahmadzadeh, H., Freedman, B. R., Connizzo, B. K., Soslowsky, L. J., Shenoy, V. B. Micromechanical poroelastic finite element and shear-lag models of tendon predict large strain dependent Poisson's ratios and fluid expulsion under tensile loading. Acta Biomaterialia. 22, 83-91 (2015).
- Szczesny, S. E., Elliott, D. M. Interfibrillar shear stress is the loading mechanism of collagen fibrils in tendon. Acta Biomaterialia. 10 (6), 2582-2590 (2014).
- Han, W. M., et al. Macro- to microscale strain transfer in fibrous tissues is heterogeneous and tissue-specific. Biophysical Journal. 105 (3), 807-817 (2013).
- Pedaprolu, K., Szczesny, S. E. A novel, open-source, low-cost bioreactor for load-controlled cyclic loading of tendon explants. Journal of Biomechanical Engineering. 144 (8), 084505 (2022).
- Gatt, R., et al. Negative Poisson's ratios in tendons: An unexpected mechanical response. Acta Biomaterialia. 24, 201-208 (2015).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon